Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Estimación de la sensibilidad estructural de regiones intrínsecamente desordenadas en respuesta al estrés hiperosmótico en células vivas mediante FRET
* Estos autores han contribuido por igual
En este artículo
Resumen
Las regiones intrínsecamente desordenadas (IDR) son dominios proteicos flexibles que modifican su conformación en respuesta a los cambios ambientales. La transferencia de energía por resonancia de fluorescencia de conjunto (FRET) puede estimar las dimensiones de las proteínas en diferentes condiciones. Presentamos un enfoque FRET para evaluar la sensibilidad estructural de IDR en células vivas de Saccharomyces cerevisiae bajo estrés hiperosmótico.
Resumen
Las regiones intrínsecamente desordenadas (IDR, por sus siglas en inglés) son dominios proteicos que participan en procesos celulares cruciales. Durante las condiciones de estrés, las propiedades fisicoquímicas del entorno celular cambian, lo que afecta directamente al conjunto conformacional de las IDR. Los exámenes exhaustivos son inherentemente sensibles a las perturbaciones ambientales. El estudio de cómo las propiedades fisicoquímicas de la célula regulan el conjunto conformacional de las IDR es esencial para comprender el control ambiental de su función. Aquí, describimos un método paso a paso para medir la sensibilidad estructural de las IDR en células vivas de Saccharomyces cerevisiae en respuesta a condiciones de estrés hiperosmótico. Presentamos el uso de la transferencia de energía por resonancia fluorescente de conjunto (FRET) para estimar cómo cambian las dimensiones globales de las IDR durante un aumento progresivo del estrés hiperosmótico impuesto a las células con cualquier osmolito. Además, proporcionamos un script para procesar las mediciones de fluorescencia y comparar la sensibilidad estructural para diferentes IDR. Siguiendo este método, los investigadores pueden obtener información valiosa sobre los cambios conformacionales que experimentan las IDR en el complejo entorno intracelular al cambiar de entorno.
Introducción
Las regiones intrínsecamente desordenadas (IDR) son componentes críticos en los procesos celulares1. En combinación con los dominios estructurados, los IDR son esenciales para las funciones de las proteínas. La composición de aminoácidos de los IDR está sesgada, representada principalmente por residuos cargados, hidrófilos y pequeños. Debido a esta propiedad, los IDR se consideran dominios de baja complejidad 2,3. Numerosas IDR han atraído la atención, principalmente porque estas regiones juegan un papel crucial en las condiciones patológicas, particularmente las enfermedades neurodegenerativas. Estas enfermedades se caracterizan por el autoensamblaje y el posterior depósito extracelular o intracelular de IDR en las neuronas4. Ejemplos de tales IDR incluyen amiloide-β (Aβ) en la enfermedad de Alzheimer, huntingtina (HTT) en la enfermedad de Huntington y proteína de unión al ADN-43 de TAR (TDP-43) y fusionada en el sarcoma (FUS) en la esclerosis lateral amiotrófica y la demencia frontotemporal4. El estudio de los reordenamientos estructurales de las IDR en el contexto de la enfermedad se ha mejorado significativamente mediante métodos espectroscópicos, incluida la transferencia de energía por resonancia de fluorescencia (FRET).
La naturaleza hidrofílica y extendida de los IDR los hace extremadamente sensibles a los cambios en las propiedades fisicoquímicas del entorno de la solución5. El grado en que el conjunto conformacional de IDR es modificado por el medio ambiente se denomina sensibilidad estructural 5,6,7. Se pueden utilizar diferentes técnicas para estudiar la conformación y la dinámica de las IDR, incluyendo el dicroísmo circular (CD) y la dispersión de rayos X de ángulo pequeño (SAXS)8,9. Desafortunadamente, la CD y el SAXS requieren grandes cantidades de proteínas purificadas, por lo que no son apropiados para estudios en células. Por el contrario, FRET es una técnica que mide la intensidad de fluorescencia de dos moléculas fluorescentes que marcan específicamente una IDR, lo que significa que pueden ser monitoreadas en mezclas complejas como las células vivas10. La medición dinámica de la sensibilidad estructural de las IDR en células vivas es necesaria para comprender cómo el entorno regula la conformación y la función del proteoma desordenado.
FRET es un método potente para cuantificar la sensibilidad estructural de las IDR, así como de las proteínas globulares y multidominio en células vivas. El método requiere una construcción que consiste en un IDR de interés intercalado entre dos proteínas fluorescentes (FP), conocido como par FRET. Para este protocolo, sugerimos el uso de mCerulean3 como FP donante y Citrino como FP aceptor, debido a su amplio rango dinámico, en comparación con otros MF reportados en un estudio previo sobre la sensibilidad de lasIDRs 6. La FRET se ha explotado previamente para medir la sensibilidad estructural de una IDR de una planta en diferentes contextos celulares6. Además, esta técnica ha sido utilizada para caracterizar las dimensiones proteicas globales de las IDR por diferentes grupos de investigación tanto in vitro como in vivo 5,11.
Aquí, describimos el método FRET de conjunto para estudiar la sensibilidad estructural de las IDR en células vivas de levadura (Saccharomyces cerevisiae). Mostramos resultados representativos que se basan en una IDR de planta llamada AtLEA4-5. AtLEA4-5 está desordenada en solución, pero se pliega en α-hélice cuando se induce el hacinamiento macromolecular in vitro12. AtLEA4-5 es un buen modelo de referencia para este método porque es relativamente pequeño (158 residuos), desordenado y sensible a la perturbación ambiental como se ha reportado in silico e in vitro 6,12. El método presentado aquí se puede escalar para enfoques de alto rendimiento porque las células de levadura son fáciles de cultivar y el tratamiento se aplica en pequeños volúmenes. Además, pequeñas modificaciones del protocolo pueden aplicarse a otros sistemas celulares como bacterias y células vegetales6. El protocolo se puede realizar en cualquier laboratorio de biología molecular con acceso a un lector de microplacas con modo fluorescencia, un equipo disponible en la mayoría de las instituciones de investigación.
Protocolo
1. Construcción de plásmido
- Amplifique el marco de lectura abierto (ORF) que codifica la IDR deseada mediante la reacción en cadena de la polimerasa (PCR). No incluya el codón de parada, ya que el ORF estará flanqueado por los genes que codifican las proteínas fluorescentes. Para la amplificación, diseñe cebadores con los sitios de restricción SacI (5') y BglII (3').
NOTA: Para la sección de resultados representativos, utilizamos AtLEA4-5 como IDR seleccionado. Amplificamos el ORF de AtLEA4-5 a partir del plásmido12 pTrc99A-AtLEA4-5. - Digerir el producto de PCR ORF por restricción consecutiva con las enzimas SacI y BglII13.
- Obtenga el plásmido comercial pDRFLIP38-AtLEA4-5 (#178189) de Addgene (https://www.addgene.org/178189/).
- Llevar a cabo una restricción consecutiva utilizando las enzimas SacI y BglII para eliminar el ORF AtLEA4-5 del plásmido pDRFLIP38-AtLEA4-5, dejando un plásmido abierto que contiene el par FRET13.
- Purificar los fragmentos de ADN digeridos mediante la recuperación en gel de una electroforesis en gel de agarosa14. Ligar los fragmentos restringidos utilizando la ADN ligasa15.
- Transformar la reacción de clonación en células competentes de Escherichia coli (DH5α o cepas relacionadas) y seleccionar placas de Luria-Bertani (LB) que contengan 50 μg/mL de ampicilina16. Cultivar durante la noche (ON) a 37 °C.
- Aislar y cultivar al menos cinco colonias transformadas en una nueva placa y genotipo por PCR17. Purificar el ADN plasmídico de una colonia transformada positiva utilizando métodos estándar de minipreparación18.
- Verificar la extracción e integridad del ADN plasmídico mediante electroforesis en gel de agarosa19. Verifique la clonación correcta de la construcción utilizando la secuenciaciónSanger 20.
2. Expresión de plásmidos en células de levadura
NOTA: Utilice técnicas asépticas estándar para realizar los siguientes pasos. Utilice una campana de flujo laminar de cultivo o un encendedor.
- Inocular una veta de S. cerevisiae BJ5465 (ATCC: 208289) en 3 mL de medio de levadura peptona dextrosa (YPD) y crecer a 30 °C y 200 rpm.
NOTA: BJ5465 es una cepa deficiente en proteasa (CH1) recomendada para trabajar con IDR. Se pueden probar y utilizar otras cepas de S. cerevisiae . - Centrifugar 1 ml de cultivo de levadura saturada (OD600 ~ 3-4) durante la noche a 14.000 x g durante 1 min. Retire con cuidado el sobrenadante mediante pipeteo.
- Vuelva a suspender el gránulo en 1 ml de tampón Tris-EDTA (TE) (pH 7,5) moviendo suavemente el tubo. Centrifugar a 14.000 x g durante 1 min y retirar con cuidado el sobrenadante.
- Vuelva a suspender el gránulo en 500 μL de tampón Lazy Bones (40% p/v PEG 3.350; 100 mM de acetato de litio; 10 mM de Tris-HCl; 1 mM de EDTA; pH 7,5) mediante pipeteo. Añadir 25 μL de ADN de esperma de salmón hervido (100 °C durante 5 min, seguido de enfriamiento en hielo durante 5 min) a las células de levadura resuspendidas.
- Agregue 100 ng del ADN plasmídico a la mezcla y haga vórtice la mezcla durante 1 minuto para asegurar una mezcla completa. Deje reposar la mezcla a temperatura ambiente durante 1-2 h.
- Calentar la mezcla de levadura a 42 °C durante 12 minutos, luego enfriar en hielo durante otros 12 minutos. Centrifugar el tubo a 14 000 x g durante 1 min y retirar con cuidado el sobrenadante.
- Vuelva a suspender el gránulo en 1 ml de tampón TE (pH 7,5). Colocar 100 μL de las células transformadas en placas de medio de abandono sintético de levadura sin uracilo (SD-ura) suplementadas con agar 15 g/L.
- Incubar las placas a 30 °C durante 2-3 días (Figura 1). Raye al menos cinco transformadores de levadura en una nueva placa y crezca ON.
3. Validación de transformantes de levadura
- Elija una pequeña porción de cada candidato transformante raspando suavemente y resuspendiendo 5 μL de NaOH de 20 mM en tubos de PCR individuales.
- Calentar la muestra a 99 °C durante 10 min en un termociclador para lisar las células y liberar el ADN en la solución. Este paso es crucial para preparar la plantilla de ADN para la PCR.
- Transfiera 1 μl de la muestra hervida a una mezcla de reacción de PCR separada que contenga los cebadores y reactivos de PCR adecuados para el genotipado. Se utilizaron los siguientes cebadores: Imprimación directa: 5'-AAATATACCCCAGCCTCGATCTAGA-3'. Imprimación inversa: 5'-GTAATACGACTCACTATAGGGCG-3'.
- Configure una reacción de PCR y valide la presencia del plásmido correcto mediante electroforesis en gel de agarosa. Seleccione un transformador para realizar más experimentos.
4. Preparación del cultivo de células de levadura para el ensayo FRET
- Inocular una veta del transformante de levadura seleccionado en 3 mL de medio líquido 1x SD-Ura.
NOTA: Realice este procedimiento en una campana de flujo laminar y material estéril. - Cultivar a 30 °C, 200 rpm durante al menos 12 h para alcanzar la saturación. Mida el diámetro exterior600. El crecimiento debe estar comprendido entre OD600 = 1,0-2,0.
NOTA: Si OD600 es inferior a 1.0, dé a las células más tiempo de incubación para crecer. Si OD600 supera 2.0, no continúe y reinicie desde el paso 4.1.
5. Preparación de células de levadura para el ensayo FRET
- Recoja 2 ml del cultivo de levadura durante la noche y centrifugue a 14.000 x g a temperatura ambiente. Retire con cuidado el sobrenadante mediante pipeteo.
- Resuspender el pellet en 1 mL de tampón 50 mM de ácido etanosulfónico (MES) 2-(N-Morfolino) (pH 6).
- Centrifugar a 14.000 x g a temperatura ambiente. Retire el sobrenadante.
- Repita los pasos 5.2 y 5.3. Vuelva a suspender las células de levadura en 2 ml de tampón MES, pH 6 y vierta el volumen en un depósito de reactivos.
6. Configuración de las mediciones de fluorescencia
NOTA: Se considera que el presente escaneo se realiza en un lector de microplacas con modo fluorescencia.
- Para los ajustes básicos, configure el instrumento de la siguiente manera: Tipo de medición: espectro de intensidad de fluorescencia (FI); Nombre de la microplaca: Greiner 96 F-bottom.
- Para los ajustes ópticos, ajuste el instrumento de la siguiente manera: No. de puntos de escaneo de longitud de onda: 91; Longitud de onda de excitación: 433 nm; Ancho de banda de excitación: 10 nm; Longitud de onda de emisión: 460 - 550; Ancho de paso: 1 nm; Anchura de banda de emisión: 10 nm; Altura focal obtenida por autofoco; Altura focal: 5 mm; Longitud de onda utilizada para la ganancia: 490 nm.
- Para los ajustes generales, ajuste el instrumento de la siguiente manera: Tiempo de estabilización: 0,1 s; Dirección de lectura: unidireccional, horizontal de izquierda a derecha, de arriba a abajo.
NOTA: La ganancia introducida manualmente debe ajustarse para cada medición utilizando el pozo que se espera que muestre los niveles de intensidad de fluorescencia más altos a lo largo de la exploración de longitud de onda.
7. Preparación de soluciones hipertónicas y mediciones de fluorescencia
- Prepare soluciones de 0 M, 0,2 M, 0,4 M, 0,6 M, 0,8 M, 1 M, 1,5 M y 2 M de NaCl en una placa negra de fondo transparente de 96 pocillos. El volumen final en cada pocillo será de 200 μL (150 μL de solución de osmolito + 50 μL de suspensión de células de levadura).
NOTA: Se pueden utilizar otros osmolitos para inducir estrés hiperosmótico. Todas las soluciones deben prepararse en tampón MES de 50 mM pH 6. - Cargue 150 μL de tampón MES de 50 mM, pH 6 en los tres primeros pocillos de la fila A (A1, A2, A3) y en los pocillos subsiguientes, agregue 150 μL de la solución correspondiente en orden creciente de izquierda a derecha (Figura 2). Para probar todas las concentraciones sugeridas, continúe la carga en la fila B.
NOTA: Este ajuste considera las mediciones de 3 réplicas técnicas para cada concentración. - Utilice una micropipeta de 12 canales para transferir 50 μL de células de levadura lavadas a cada pocillo. Las células de levadura tienden a sedimentarse en el fondo del embalse. Asegúrese de volver a suspender bien la suspensión de levadura pipeteando hacia arriba y hacia abajo antes de transferir las células.
- Cargue las celdas en la placa de 96 pocillos que contiene las diferentes soluciones y mezcle bien pipeteando hacia arriba y hacia abajo al menos 4 veces.
- Mida inmediatamente los espectros de emisión de intensidad de fluorescencia para los pocillos deseados en un lector de microplacas con la configuración de especificación del paso 6. Repita el procedimiento para cada IDR que se vaya a probar en el ensayo FRET.
- Paso opcional. Si está disponible, mida un constructo solo para donantes (pDRFLIP38-mCerulean3) siguiendo los pasos descritos aquí.
NOTA: Se recomienda realizar este paso para calcular la eficiencia de FRET de cada condición. Si el lector no puede realizar este paso, FRET se puede calcular utilizando el método de relación FRET. Ambos métodos para los cálculos de FRET se explican en los pasos 8 y 9. - Realice al menos tres réplicas en tres días independientes para cada construcción.
8. Tratamiento de datos mediante el método de eficiencia FRET
NOTA: Para los lectores con conocimientos del lenguaje de programación R, proporcionamos un conjunto de scripts de R para realizar el procesamiento de datos descrito en esta sección. Los guiones se pueden encontrar en https://github.com/Kaz-bits/cuevaslab-procotols/tree/main/FRET. Siga las instrucciones del archivo README.
- Exporte datos como archivos .xlsx desde el lector de microplacas.
- Normalice los valores de intensidad de fluorescencia en cada longitud de onda de escaneo hasta el punto isosmejor del par FRET utilizado.
NOTA: Este paso es necesario para comparar los espectros de todas las condiciones probadas. El punto isosmejor del par mCerulean3 y Citrine FRET es de 515 nm. Por ejemplo, obtenga la relación de los valores de intensidad de fluorescencia de 460 nm/515 nm, 461 nm/515 nm, 462 nm/515 nm, etc. - Calcule la media de cada réplica técnica una vez normalizados los valores de fluorescencia. En total, debe haber una columna para cada condición de estrés hiperosmótico.
- Visualice todos los espectros de intensidad de fluorescencia en el mismo gráfico (Figura 3). Cada espectro debe mostrar una combinación de los espectros de emisión de fluorescencia de mCerulean3 y Citrino individuales. En casos raros, cuando la eficiencia FRET es 0, solo se observará el espectro de emisión de fluorescencia del donante, o cuando la eficiencia FRET es 1, solo se observará la fluorescencia del aceptor. El pico de emisión de fluorescencia para mCerulean3 (donante) es de 475 nm, y el pico de emisión de fluorescencia para Citrine (aceptor) es de 525 nm.
- Determine si la medición de fluorescencia muestra un cambio FRET típico en respuesta al estrés hiperosmótico. Si la conformación de la IDR es sensible al tratamiento, esto se reflejará como un cambio en la eficiencia de FRET. Un cambio típico en la eficiencia de FRET combinará una disminución en la intensidad de fluorescencia del donante con un aumento en la intensidad de fluorescencia del aceptor o viceversa.
- Cuantificar la eficiencia de FRET (EFRET). Obtener los valores máximos normalizados del donante (mCerulean3) (475 nm/515 nm) para el constructo IDR y el constructo solo donante para cada condición de estrés hiperosmótico. Utilice los valores máximos medios normalizados de las réplicas técnicas. Realice esta operación para cada una de las tres réplicas independientes. Calcule EFRET con la siguiente fórmula:
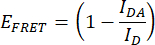
Donde IDA es la relación 475 nm/515 nm de la construcción IDR (construcción de pares FRET), e ID es la relación 475 nm/515 nm de la construcción solo donante. - Compare las eficiencias de FRET. Normalice los valores de EFRET de cada condición de estrés hiperosmótico con elE FRET de la condición sin estrés (por ejemplo, 0 M NaCl). Realice comparaciones utilizando un diagrama de caja o un diagrama de curva suave. Realizar un ANOVA de un factor seguido de una prueba estadística de Tukey post hoc (p-valores: *p < 0,05, **p < 0,01, ***p < 0,001, ****p < 0,0001).
9. Tratamiento de los datos mediante el método de la relación FRET
NOTA: Si no se pueden adquirir mediciones solo del donante, realice el método de proporción FRET. Este método compara la relación FRET en las diferentes condiciones de estrés hiperosmótico. Los pasos 7.1 a 7.5 deben realizarse antes de los pasos de esta sección para validar un comportamiento típico de FRET. Para los lectores con conocimientos del lenguaje de programación R, proporcionamos un conjunto de scripts de R para realizar el procesamiento de datos descrito en esta sección. Los guiones se pueden encontrar en https://github.com/Kaz-bits/cuevaslab-procotols/tree/main/FRET. Siga las instrucciones del archivo README.
- Extraiga datos a 475 nm y 525 nm del archivo xlsx exportado previamente desde el lector de microplacas. Estos valores corresponden al pico de emisión de fluorescencia para mCerulean3 (donante, DxDm) y al pico de emisión de fluorescencia para Citrino (aceptor, DxAm) cuando se excita el fluoróforo donante (433 nm).
- Normalice los valores de intensidad de fluorescencia a 475 nm y 525 nm al punto isosmejor del par FRET utilizado. El punto isosmejor del par mCerulean3 y Citrine FRET es de 515 nm. Obtener la relación FRET (DxAm/DxDm).
- Compara las proporciones de FRET. Normalice los valores de DxAm/DxDm de cada condición de estrés hiperosmótico a la media de DxAm/DxDm de la condición sin estrés (por ejemplo, 0 M NaCl). Realice comparaciones utilizando un diagrama de caja o un diagrama de curva suave (Figura 4 y Figura 5). Realizar un ANOVA de un factor seguido de una prueba estadística de Tukey post hoc (p-valores: *p < 0,05, **p < 0,01, ***p < 0,001, ****p < 0,0001).
Resultados
Después de transformar las células de levadura con el plásmido pDRFLIP38-AtLEA4-5, se observó la fluorescencia de los transformantes positivos con un transiluminador de luz azul y un filtro (Figura 1). La preparación de las diferentes soluciones para inducir el estrés hiperosmótico requiere mucho tiempo, por lo que sugerimos seguir la plantilla de 96 pocillos de la Figura 2. Inmediatamente después del tratamiento de estrés hiperosmótico con concentraci...
Discusión
El método que aquí se presenta ofrece una forma de obtener información sobre cómo las dimensiones globales del conjunto de exámenes exhaustivos detectan y responden a las perturbaciones ambientales. Este método se basa en una construcción codificada genéticamente y no requiere componentes adicionales más allá de una expresión estable de plásmido en células de levadura, lo que lo hace adaptable para aplicaciones potenciales en otros tipos de células. Además, es versátil para explorar otras perturbaciones f...
Divulgaciones
Los autores declaran no tener conflictos de intereses.
Agradecimientos
Agradecemos a los miembros del laboratorio Cuevas-Velázquez por la revisión crítica del manuscrito. Este trabajo contó con el apoyo del Programa de Apoyo a Proyectos de Investigación e Innovación Tecnológica, Dirección General de Asuntos del Personal Académico, Universidad Nacional Autónoma de México (UNAM-PAPIIT) proyecto número IA203422; Consejo Nacional de Humanidades, Ciencias y Tecnología (CONAHCYT), proyecto número 252952; y Programa de Apoyo a la Investigación y el Posgrado, Facultad de Química, Universidad Nacional Autónoma de México, Subvención 5000-9182. CET (CVU 1083636) y CAPD (CVU 1269643) reconocen a CONAHCYT por su M.Sc. Beca.
Materiales
| Name | Company | Catalog Number | Comments |
| 96-well plate | Greiner Bio-One | 655096 | |
| Agar | Sigma-Aldrich | 5040 | |
| BglII | New England BioLabs | R0144S | |
| BJ5465 cells | American Type Culture Collection | 208289 | |
| Buffer MES 50 mM | Sigma-Aldrich | M8250 | |
| Buffer Tris-HCl 10 mM | Invitrogen | 15506017 | |
| EDTA 1 mM | Merck | 108452 | |
| Falcon tubes | Corning | 352057 | |
| LB media | Sigma-Aldrich | L2897 | |
| Lithium acetate 0.1 M | Sigma-Aldrich | L6883 | |
| Low Melt Agarose | GOLDBIO | A-204-25 | |
| Microcentrifuge | eppendorf | 5452000010 | |
| Miniprep kit | ZymoPure | D4210 | |
| NaOH 0.02 M | Merck | 106462 | |
| PEG 3,350 40% | Sigma-Aldrich | 1546547 | |
| plasmid pDRFLIP38-AtLEA4-5 | addgene | 178189 | |
| Plate reader | BMG LABTECH | CLARIOstar Plus | |
| SacI | New England BioLabs | R3156S | |
| Salmon sperm DNA 2 mg/mL | Thermo Fisher Scientific | 15632011 | |
| SD-Ura | Sigma-Aldrich | Y1501 | |
| Sodium cloride | Sigma-Aldrich | S9888 | |
| Taq polymesare | Promega | M5123 | |
| Transiluminator | Accuris instruments | E4000 | |
| UV-Visible spectrophotometer | Thermo Fisher Scientific | Biomate3 | |
| YPD media | Sigma-Aldrich | Y1500 |
Referencias
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nat Rev Mol Cell Biol. 16 (1), 18-29 (2015).
- Covarrubias, A. A., Romero-Pérez, P. S., Cuevas-Velazquez, C. L., Rendón-Luna, D. F. The functional diversity of structural disorder in plant proteins. Arch Biochem Biophys. 680, 108229 (2019).
- Ahmed, S. S., et al. Characterization of intrinsically disordered regions in proteins informed by human genetic diversity. PLoS Comput Biol. 18 (3), e1009911 (2022).
- Birol, M., Melo, A. M. Untangling the conformational polymorphism of disordered proteins associated with neurodegeneration at the single-molecule level. Front Mol Neurosci. 12, 309 (2019).
- Moses, D., et al. Revealing the hidden sensitivity of intrinsically disordered proteins to their chemical environment. J Phys Chem Lett. 11 (23), 10131-10136 (2020).
- Cuevas-Velazquez, C. L., et al. Intrinsically disordered protein biosensor tracks the physical-chemical effects of osmotic stress on cells. Nat Commun. 12 (1), 5438 (2021).
- Holehouse, A. S., Sukenik, S. Controlling structural bias in intrinsically disordered proteins using solution space scanning. J Chem Theory Comput. 16 (3), 1794-1805 (2020).
- Martin, E. W., Hopkins, J. B., Mittag, T. Small-angle X-ray scattering experiments of monodisperse intrinsically disordered protein samples close to the solubility limit. Methods Enzymol. 646, 185-222 (2021).
- Miles, A. J., Drew, E. D., Wallace, B. A. DichroIDP: a method for analyses of intrinsically disordered proteins using circular dichroism spectroscopy. Commun Biol. 6 (1), 823 (2023).
- Kaminski, C. F., Rees, E. J., Schierle, G. S. K. A quantitative protocol for intensity-based live cell FRET imaging. Method Mol Biol. 1076, 445-454 (2014).
- Moses, D., et al. Structural biases in disordered proteins are prevalent in the cell. bioRxiv. , (2022).
- Cuevas-Velazquez, C. L., Saab-Rincón, G., Reyes, J. L., Covarrubias, A. A. The Unstructured N-terminal Region of Arabidopsis Group 4 Late Embryogenesis Abundant (LEA) Proteins Is Required for Folding and for Chaperone-like Activity under Water Deficit. J Biol Chem. 291 (20), 10893-10903 (2016).
- JoVE Science Education Database. . Restriction enzyme digests. , (2023).
- JoVE Science Education Database. . Gel purification. , (2023).
- JoVE Science Education Database. . DNA ligation reactions. , (2023).
- JoVE Science Education Database. . Bacterial transformation using heat Ssock and competent cells. , (2023).
- JoVE Science Education Database. . PCR: Principle, instrumentation, and applications. , (2023).
- JoVE Science Education Database. . Plasmid purification. , (2023).
- JoVE Science Education Database. . DNA gel electrophoresis. , (2023).
- JoVE Core Molecular Biology. Sanger/chain termination sequencing using dideoxynucleotides - Concept Available from: https://app.jove.com/science-education/v/12020/sanger-sequencing (2023)
- Theillet, F. X., et al. Physicochemical properties of cells and their effects on intrinsically disordered proteins (IDPs). Chem Rev. 114 (13), 6661-6714 (2014).
- Brutscher, B., et al. NMR methods for the study of instrinsically disordered proteins structure, dynamics, and interactions: General overview and practical guidelines. Adv Exp Med Biol. 870, 49-122 (2015).
- Metskas, L. A., Rhoades, E. Single-molecule FRET of intrinsically disordered proteins. Annu Rev Phys Chem. 71, 391-414 (2020).
- Roebroek, T., et al. Simultaneous readout of multiple FRET pairs using photochromism. Nat Commun. 12 (1), 2005 (2021).
- Algar, W. R., Hildebrandt, N., Vogel, S. S., Medintz, I. L. FRET as a biomolecular research tool - understanding its potential while avoiding pitfalls. Nat Methods. 16 (9), 815-829 (2019).
- Lyon, A. S., Peeples, W. B., Rosen, M. K. A framework for understanding the functions of biomolecular condensates across scales. Nat Rev Mol Cell Biol. 22 (3), 215-235 (2021).
- Belott, C., Janis, B., Menze, M. A. Liquid-liquid phase separation promotes animal desiccation tolerance. Proc Natl Acad Sci U S A. 117 (44), 27676-27684 (2020).
- Miermont, A., et al. Severe osmotic compression triggers a slowdown of intracellular signaling, which can be explained by molecular crowding. Proc Natl Acad Sci U S A. 110 (14), 5725-5730 (2013).
- Saito, H., Posas, F. Response to hyperosmotic stress. Genetics. 192 (2), 289-318 (2012).
- Selenko, P., Wagner, G. Looking into live cells with in-cell NMR spectroscopy. J Struct Biol. 158 (2), 244-253 (2007).
- Cattani, J., Subramaniam, V., Drescher, M. Room-temperature in-cell EPR spectroscopy: alpha-Synuclein disease variants remain intrinsically disordered in the cell. Phys Chem Chem Phys. 19 (28), 18147-18151 (2017).
- Beveridge, R., Chappuis, Q., Macphee, C., Barran, P. Mass spectrometry methods for intrinsically disordered proteins. Analyst. 138 (1), 32-42 (2013).
- Beveridge, R., et al. Ion mobility mass spectrometry uncovers the impact of the patterning of oppositely charged residues on the conformational distributions of intrinsically disordered proteins. J Am Chem Soc. 141 (12), 4908-4918 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados