È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Stima della sensibilità strutturale di regioni intrinsecamente disordinate in risposta allo stress iperosmotico in cellule viventi utilizzando FRET
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Le regioni intrinsecamente disordinate (IDR) sono domini proteici flessibili che modificano la loro conformazione in risposta ai cambiamenti ambientali. Il trasferimento di energia di risonanza di fluorescenza d'insieme (FRET) può stimare le dimensioni delle proteine in diverse condizioni. Presentiamo un approccio FRET per valutare la sensibilità strutturale dell'IDR in cellule viventi di Saccharomyces cerevisiae in condizioni di stress iperosmotico.
Abstract
Le regioni intrinsecamente disordinate (IDR) sono domini proteici che partecipano a processi cellulari cruciali. Durante le condizioni di stress, le proprietà fisico-chimiche dell'ambiente cellulare cambiano, influenzando direttamente l'insieme conformazionale degli IDR. Gli esami approfonditi sono intrinsecamente sensibili alle perturbazioni ambientali. Studiare come le proprietà fisico-chimiche della cellula regolano l'insieme conformazionale degli IDR è essenziale per comprendere il controllo ambientale della loro funzione. Qui, descriviamo un metodo passo-passo per misurare la sensibilità strutturale degli IDR in cellule viventi di Saccharomyces cerevisiae in risposta a condizioni di stress iperosmotico. Presentiamo l'uso del trasferimento di energia di risonanza di fluorescenza d'insieme (FRET) per stimare come cambiano le dimensioni globali degli IDR durante un progressivo aumento dello stress iperosmotico imposto alle cellule con qualsiasi osmolito. Inoltre, forniamo uno script per l'elaborazione delle misure di fluorescenza e il confronto della sensibilità strutturale per diversi IDR. Seguendo questo metodo, i ricercatori possono ottenere preziose informazioni sui cambiamenti conformazionali che gli IDR subiscono nel complesso ambiente intracellulare al variare degli ambienti.
Introduzione
Le regioni intrinsecamente disordinate (IDR) sono componenti fondamentali nei processi cellulari1. In combinazione con i domini strutturati, gli IDR sono essenziali per le funzioni proteiche. La composizione amminoacidica degli IDR è distorta, rappresentata principalmente da residui carichi, idrofili e di piccole dimensioni. A causa di questa proprietà, gli esami approfonditi sono considerati domini a bassa complessità 2,3. Numerosi esami approfonditi hanno attirato l'attenzione, principalmente perché queste regioni svolgono un ruolo cruciale nelle condizioni patologiche, in particolare nelle malattie neurodegenerative. Tali malattie sono caratterizzate dall'auto-assemblaggio e dalla successiva deposizione extracellulare o intracellulare di IDR nei neuroni4. Esempi di tali IDR includono l'amiloide-β (Aβ) nella malattia di Alzheimer, l'huntingtina (HTT) nella malattia di Huntington e la proteina-43 legante il DNA TAR (TDP-43) e fusa nel sarcoma (FUS) nella sclerosi laterale amiotrofica e nella demenza frontotemporale4. Lo studio dei riarrangiamenti strutturali degli IDR nel contesto della malattia è stato significativamente migliorato da metodi spettroscopici, tra cui il trasferimento di energia di risonanza di fluorescenza (FRET).
La natura idrofila ed estesa degli IDR li rende estremamente sensibili ai cambiamenti nelle proprietà fisico-chimiche dell'ambiente della soluzione5. Il grado in cui l'insieme conformazionale degli IDR viene modificato dall'ambiente è chiamato sensibilità strutturale 5,6,7. Diverse tecniche possono essere utilizzate per studiare la conformazione e la dinamica degli IDR, tra cui il dicroismo circolare (CD) e lo scattering di raggi X a piccolo angolo (SAXS)8,9. Sfortunatamente, CD e SAXS richiedono grandi quantità di proteine purificate, quindi non sono appropriati per gli studi sulle cellule. Al contrario, la FRET è una tecnica che misura l'intensità della fluorescenza di due molecole fluorescenti che marcano specificamente un IDR, il che significa che possono essere monitorate in miscele complesse come le cellule viventi10. Misurare dinamicamente la sensibilità strutturale degli IDR nelle cellule viventi è necessario per comprendere come l'ambiente regola la conformazione e la funzione del proteoma disordinato.
La FRET è un metodo potente per quantificare la sensibilità strutturale degli IDR, nonché delle proteine globulari e multidominio nelle cellule viventi. Il metodo richiede un costrutto costituito da un IDR di interesse inserito tra due proteine fluorescenti (FP), noto come coppia FRET. Per questo protocollo, suggeriamo l'uso di mCerulean3 come FP donatore e Citrine come FP accettore, a causa del loro ampio range dinamico, rispetto ad altri FP riportati in un precedente studiosulla sensibilità 6 degli IDR. La FRET è stata precedentemente sfruttata per misurare la sensibilità strutturale di una pianta IDR in diversi contesti cellulari6. Inoltre, questa tecnica è stata utilizzata per caratterizzare le dimensioni proteiche complessive degli IDR da diversi gruppi di ricerca sia in vitro che in vivo 5,11.
In questo articolo, descriviamo il metodo FRET d'insieme per studiare la sensibilità strutturale degli IDR in cellule di lievito viventi (Saccharomyces cerevisiae). Mostriamo risultati rappresentativi che si basano su un IDR di pianta chiamato AtLEA4-5. AtLEA4-5 è disordinato in soluzione, ma si ripiega in α-elica quando l'affollamento macromolecolare è indotto in vitro12. AtLEA4-5 è un buon modello di riferimento per questo metodo perché è relativamente piccolo (158 residui), disordinato e sensibile alle perturbazioni ambientali come riportato in silico e in vitro 6,12. Il metodo qui presentato può essere scalato per approcci ad alto rendimento perché le cellule di lievito sono facili da coltivare e il trattamento viene applicato in piccoli volumi. Inoltre, piccole modifiche al protocollo possono essere applicate ad altri sistemi cellulari come batteri e cellule vegetali6. Il protocollo può essere eseguito in qualsiasi laboratorio di biologia molecolare con accesso a un lettore di micropiastre con modalità fluorescenza, un'apparecchiatura disponibile nella maggior parte degli istituti di ricerca.
Protocollo
1. Costrutto plasmidico
- Amplificare il frame di lettura aperto (ORF) che codifica per l'IDR desiderato mediante reazione a catena della polimerasi (PCR). Non includere il codone di stop poiché l'ORF sarà affiancato dai geni che codificano le proteine fluorescenti. Per l'amplificazione, progettare primer con i siti di restrizione SacI (5') e BglII (3').
NOTA: Per la sezione dei risultati rappresentativi, abbiamo utilizzato AtLEA4-5 come IDR selezionato. Abbiamo amplificato l'ORF di AtLEA4-5 dal plasmide pTrc99A-AtLEA4-512. - Digerire il prodotto ORF PCR mediante restrizione consecutiva con enzimi SacI e BglII13.
- Ottenere il plasmide commerciale pDRFLIP38-AtLEA4-5 (#178189) da Addgene (https://www.addgene.org/178189/).
- Eseguire una restrizione consecutiva utilizzando gli enzimi SacI e BglII per rimuovere l'ORF AtLEA4-5 dal plasmide pDRFLIP38-AtLEA4-5, lasciando un plasmide aperto contenente la coppia FRET13.
- Purificare i frammenti di DNA digeriti utilizzando il recupero del gel da un'elettroforesi su gel di agarosio14. Legare i frammenti ristretti utilizzando la DNA ligasi15.
- Trasformare la reazione di clonaggio in cellule competenti di Escherichia coli (DH5α o ceppi correlati) e selezionare in piastre Luria-Bertani (LB) contenenti 50 μg/mL di ampicillina16. Coltivare durante la notte (ON) a 37 °C.
- Isolare e far crescere almeno cinque colonie trasformate in una nuova piastra e genotipo mediante PCR17. Purificare il DNA plasmidico da una colonia trasformata positiva utilizzando metodi standard di miniprep18.
- Verificare l'estrazione e l'integrità del DNA plasmidico utilizzando l'elettroforesi su gel di agarosio19. Verificare la corretta clonazione del costrutto utilizzando la sequenza Sanger20.
2. Espressione plasmidica in cellule di lievito
NOTA: Utilizzare tecniche asettiche standard per eseguire i seguenti passaggi. Utilizzare una cappa a flusso laminare per colture o un accendino.
- Inoculare una striscia di S. cerevisiae BJ5465 (ATCC: 208289) strain in 3 mL di terreno di lievito peptone destrosio (YPD) e far crescere a 30 °C e 200 rpm.
NOTA: BJ5465 è un ceppo carente di proteasi (CH1) raccomandato per lavorare con gli IDR. Altri ceppi di S. cerevisiae possono essere testati e utilizzati. - Centrifugare 1 mL di coltura di lievito saturo durante la notte (OD600 ~ 3-4) a 14.000 x g per 1 min. Rimuovere con cautela il surnatante mediante pipettaggio.
- Risospendere il pellet in 1 mL di tampone Tris-EDTA (TE) (pH 7,5) agitando delicatamente la provetta. Centrifugare a 14.000 x g per 1 minuto e rimuovere con cautela il surnatante.
- Risospendere il pellet in 500 μL di tampone Lazy Bones (40% p/v PEG 3.350; 100 mM di acetato di litio; 10 mM Tris-HCl; 1 mM EDTA; pH 7,5) mediante pipettaggio. Aggiungere 25 μL di DNA spermatico di salmone bollito (100 °C per 5 min, seguito da raffreddamento su ghiaccio per 5 min) alle cellule di lievito risospese.
- Aggiungere 100 ng di DNA plasmidico alla miscela e vorticare la miscela per 1 minuto per garantire una miscelazione accurata. Lasciare riposare il composto a temperatura ambiente per 1-2 ore.
- Fate fare uno shock termico alla miscela di lievito a 42 °C per 12 min, poi raffreddate su ghiaccio per altri 12 min. Centrifugare la provetta a 14 000 x g per 1 minuto e rimuovere con cautela il surnatante.
- Risospendere il pellet in 1 mL di tampone TE (pH 7,5). Piastra 100 μL delle cellule trasformate su piastre di lievito Synthetic dropout Medium senza uracile (SD-ura) integrato con 15 g/L di agar.
- Incubare le piastre a 30 °C per 2-3 giorni (Figura 1). Strisciare almeno cinque trasformatori di lievito in una nuova piastra e crescere.
3. Validazione dei trasformatori del lievito
- Prelevare una piccola porzione di ciascun candidato trasformante raschiando delicatamente e risospendendo in 5 μL di NaOH da 20 mM in singole provette PCR.
- Riscaldare il campione a 99 °C per 10 minuti in un termociclatore per lisare le cellule e rilasciare il DNA nella soluzione. Questo passaggio è fondamentale per preparare il modello di DNA per la PCR.
- Trasferire 1 μL del campione bollito in una miscela di reazioni PCR separata contenente i primer e i reagenti PCR appropriati per la genotipizzazione. Abbiamo utilizzato i seguenti primer: Primer in avanti: 5'-AAATATACCCCAGCCTCGATCTAGA-3'. Primer inverso: 5'-GTAATACGACTCACTATAGGGCG-3'.
- Impostare una reazione PCR e convalidare la presenza del plasmide corretto utilizzando l'elettroforesi su gel di agarosio. Seleziona un trasformatore per ulteriori esperimenti.
4. Preparazione di colture cellulari di lievito per il saggio FRET
- Inoculare una striscia del trasformante di lievito selezionato in 3 ml di terreno liquido 1x SD-Ura.
NOTA: Eseguire questa procedura in una cappa a flusso laminare e materiale sterile. - Coltivare a 30 °C, 200 giri/min per almeno 12 ore fino a raggiungere la saturazione. Misurare il diametro esterno600. La crescita dovrebbe essere compresa tra OD600 = 1,0-2,0.
NOTA: Se OD600 è inferiore a 1,0, dare alle cellule più tempo di incubazione per crescere. Se OD600 supera 2.0, non procedere e ricominciare dal punto 4.1.
5. Preparazione di cellule di lievito per il saggio FRET
- Raccogliere 2 ml di coltura di lievito durante la notte e centrifugare a 14.000 x g a temperatura ambiente. Rimuovere con cautela il surnatante mediante pipettaggio.
- Risospendere il pellet in 1 mL di tampone 50 mM di acido 2-(N-morfolino) etansolfonico (MES) (pH 6).
- Centrifugare a 14.000 x g a temperatura ambiente. Rimuovere il surnatante.
- Ripetere i passaggi 5.2 e 5.3. Risospendere le cellule di lievito in 2 mL di tampone MES, pH 6 e versare il volume in un serbatoio di reagenti.
6. Impostazione delle misure di fluorescenza
NOTA: La presente scansione si considera eseguita in un lettore di micropiastre con modalità fluorescenza.
- Per le impostazioni di base, impostare lo strumento come segue: Tipo di misurazione: spettro dell'intensità della fluorescenza (FI); Nome micropiastra: Greiner 96 F-bottom.
- Per le impostazioni ottiche, impostare lo strumento come segue: No. di punti di scansione della lunghezza d'onda: 91; Lunghezza d'onda di eccitazione: 433 nm; Larghezza di banda di eccitazione: 10 nm; Lunghezza d'onda di emissione: 460 - 550; Larghezza passo: 1 nm; Larghezza di banda di emissione: 10 nm; Altezza focale ottenuta con l'autofocus; Altezza focale: 5 mm; Lunghezza d'onda utilizzata per il guadagno: 490 nm.
- Per le impostazioni generali, impostare lo strumento come segue: Tempo di assestamento: 0,1 s; Direzione di lettura: unidirezionale, orizzontale da sinistra a destra, dall'alto verso il basso.
NOTA: Il guadagno inserito manualmente deve essere regolato per ogni misurazione utilizzando il pozzetto previsto per visualizzare i livelli di intensità di fluorescenza più elevati durante la scansione della lunghezza d'onda.
7. Preparazione di soluzioni ipertoniche e misure di fluorescenza
- Preparare soluzioni di NaCl 0 M, 0,2 M, 0,4 M, 0,6 M, 0,8 M, 1 M, 1,5 M e 2 M NaCl in una piastra nera a fondo trasparente a 96 pozzetti. Il volume finale in ciascun pozzetto sarà di 200 μL (150 μL di soluzione di osmolita + 50 μL di sospensione di cellule di lievito).
NOTA: Altri osmoliti possono essere utilizzati per indurre stress iperosmotico. Tutte le soluzioni devono essere preparate in tampone MES da 50 mM pH 6. - Caricare 150 μL di tampone MES da 50 mM, pH 6 nei primi tre pozzetti della fila A (A1, A2, A3) e nei pozzetti successivi, aggiungere 150 μL della soluzione corrispondente in ordine crescente da sinistra a destra (Figura 2). Per testare tutte le concentrazioni suggerite, continuare il carico nella riga B.
NOTA: Questa impostazione considera le misurazioni di 3 repliche tecniche per ogni concentrazione. - Utilizzare una micropipetta a 12 canali per trasferire 50 μL di cellule di lievito lavate in ciascun pozzetto. Le cellule di lievito tendono a sedimentare sul fondo del serbatoio. Assicurarsi di risospendere accuratamente la sospensione di lievito pipettando su e giù prima di trasferire le cellule.
- Caricare le celle nella piastra a 96 pozzetti contenente le diverse soluzioni e miscelare bene pipettando su e giù almeno 4 volte.
- Misurare immediatamente gli spettri di emissione dell'intensità della fluorescenza per i pozzetti desiderati in un lettore per micropiastre con le impostazioni delle specifiche del passaggio 6. Ripetere la procedura per ogni esame approfondito da testare nel test FRET.
- Passaggio facoltativo. Se disponibile, misurare un costrutto solo donatore (pDRFLIP38-mCerulean3) seguendo i passaggi descritti qui.
NOTA: Si consiglia di eseguire questo passaggio per calcolare l'efficienza FRET di ciascuna condizione. Se il lettore non è in grado di eseguire questo passaggio, è possibile calcolare FRET utilizzando il metodo del rapporto FRET. Entrambi i metodi per i calcoli FRET sono spiegati nei passaggi 8 e 9. - Eseguire almeno tre repliche in tre giorni indipendenti per ogni costrutto.
8. Trattamento dei dati con il metodo dell'efficienza FRET
NOTA: per i lettori con conoscenza del linguaggio di programmazione R, viene fornito un set di script R per eseguire l'elaborazione dei dati descritta in questa sezione. Gli script sono disponibili all'indirizzo https://github.com/Kaz-bits/cuevaslab-procotols/tree/main/FRET. Seguire le istruzioni contenute nel file README.
- Esportare i dati come file .xlsx dal lettore di micropiastre.
- Normalizzare i valori di intensità della fluorescenza ad ogni lunghezza d'onda di scansione al punto isosbestico della coppia FRET utilizzata.
NOTA: Questo passaggio è necessario per confrontare gli spettri di tutte le condizioni testate. Il punto isosbestico della coppia mCerulean3 e Frit citrino è 515 nm. Ad esempio, ottenere il rapporto tra i valori di intensità della fluorescenza di 460 nm/515 nm, 461 nm/515 nm, 462 nm/515 nm e così via. - Calcolare la media per ogni replica tecnica una volta normalizzati i valori di fluorescenza. In totale, dovrebbe esserci una colonna per ogni condizione di stress iperosmotico.
- Visualizzare tutti gli spettri di intensità della fluorescenza nello stesso grafico (Figura 3). Ogni spettro dovrebbe mostrare una combinazione degli spettri di emissione di fluorescenza dei singoli mCerulean3 e Citrine. In rari casi, dove l'efficienza FRET è 0, verrà osservato solo lo spettro di emissione della fluorescenza del donatore, o quando l'efficienza FRET è 1, verrà osservata solo la fluorescenza dell'accettore. Il picco di emissione di fluorescenza per mCerulean3 (donatore) è di 475 nm e il picco di emissione di fluorescenza per Citrine (accettore) è di 525 nm.
- Determinare se la misurazione della fluorescenza mostra una tipica variazione di FRET in risposta allo stress iperosmotico. Se la conformazione dell'IDR è sensibile al trattamento, ciò si rifletterà come un cambiamento nell'efficienza del FRET. Un tipico cambiamento nell'efficienza del FRET accoppia una diminuzione dell'intensità di fluorescenza del donatore con un aumento dell'intensità di fluorescenza dell'accettore o viceversa.
- Quantificare l'efficienza del FRET (EFRET). Ottenere i valori di picco normalizzati del donatore (mCerulean3) (475 nm/515 nm) per il costrutto IDR e il costrutto del solo donatore per ogni condizione di stress iperosmotico. Utilizzare i valori di picco medi normalizzati delle repliche tecniche. Eseguire questa operazione per ognuna delle tre repliche indipendenti. Calcola EFRET con la seguente formula:
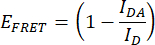
Dove IDA è il rapporto 475 nm/515 nm dal costrutto IDR (costrutto di coppia FRET) e ID è il rapporto 475 nm/515 nm dal costrutto del solo donatore. - Confronta le efficienze FRET. Normalizzare i valori di EFRET di ogni condizione di stress iperosmotico all'EFRET della condizione di non stress (ad esempio, 0 M NaCl). Effettuare confronti utilizzando un box plot o un grafico a curva liscia. Eseguire un'ANOVA unidirezionale seguita da un test statistico Tukey post hoc (valori p: *p < 0,05, **p < 0,01, ***p < 0,001, ****p < 0,0001).
9. Trattamento dei dati con il metodo del rapporto FRET
NOTA: Se non è possibile acquisire misurazioni con solo donatore, eseguire il metodo del rapporto FRET. Questo metodo confronta il rapporto FRET tra le diverse condizioni di stress iperosmotico. I passaggi da 7.1 a 7.5 devono essere eseguiti prima dei passaggi di questa sezione per convalidare un comportamento FRET tipico. Per i lettori con conoscenza del linguaggio di programmazione R, viene fornito un set di script R per eseguire l'elaborazione dei dati descritta in questa sezione. Gli script sono disponibili all'indirizzo https://github.com/Kaz-bits/cuevaslab-procotols/tree/main/FRET. Seguire le istruzioni contenute nel file README.
- Estrarre i dati a 475 nm e 525 nm dal file xlsx precedentemente esportato dal lettore di micropiastre. Questi valori corrispondono al picco di emissione di fluorescenza per mCerulean3 (donatore, DxDm) e al picco di emissione di fluorescenza per Citrino (accettore, DxAm) quando il fluoroforo donatore è eccitato (433 nm).
- Normalizzare i valori di intensità della fluorescenza a 475 nm e 525 nm al punto isosbestico della coppia FRET utilizzata. Il punto isosbestico della coppia mCerulean3 e Frit citrino è 515 nm. Ottenere il rapporto FRET (DxAm/DxDm).
- Confronta i rapporti FRET. Normalizzare i valori di DxAm/DxDm di ogni condizione di stress iperosmotico alla media di DxAm/DxDm della condizione di non stress (ad esempio, 0 M NaCl). Effettuare confronti utilizzando un box plot o un grafico a curva liscia (Figura 4 e Figura 5). Eseguire un'ANOVA unidirezionale seguita da un test statistico di Tukey post hoc (valori p: *p < 0,05, **p < 0,01, ***p < 0,001, ****p < 0,0001).
Risultati
Dopo aver trasformato le cellule di lievito con il plasmide pDRFLIP38-AtLEA4-5, è stata osservata la fluorescenza dei trasformanti positivi con un transilluminatore a luce blu e un filtro (Figura 1). La preparazione delle diverse soluzioni per indurre lo stress iperosmotico richiede molto tempo, quindi suggeriamo di seguire il modello a 96 pozzetti della Figura 2. Immediatamente dopo il trattamento da stress iperosmotico con concentrazioni variabili di cloruro ...
Discussione
Il metodo qui presentato offre un modo per ottenere informazioni su come le dimensioni globali dell'insieme degli IDR percepiscono e rispondono alle perturbazioni ambientali. Questo metodo si basa su un costrutto geneticamente codificato e non richiede componenti aggiuntivi oltre a un'espressione stabile del plasmide nelle cellule di lievito, il che lo rende adattabile per potenziali applicazioni in altri tipi di cellule. Inoltre, è versatile per esplorare altre perturbazioni fisico-chimiche che le cellule eucariotiche ...
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Ringraziamo i membri del laboratorio Cuevas-Velázquez per la revisione critica del manoscritto. Questo lavoro è stato sostenuto dal Programa de Apoyo a Proyectos de Investigación e Innovación Tecnológica, dalla Dirección General de Asuntos del Personal Académico, dall'Universidad Nacional Autónoma de México (UNAM-PAPIIT) numero di progetto IA203422; Consejo Nacional de Humanidades, Ciencias y Tecnología (CONAHCYT), progetto numero 252952; e Programa de Apoyo a la Investigación y el Posgrado, Facultad de Química, Universidad Nacional Autónoma de México, Grant 5000-9182. CET (CVU 1083636) e CAPD (CVU 1269643) riconoscono CONAHCYT per la loro borsa di studio M.Sc.
Materiali
| Name | Company | Catalog Number | Comments |
| 96-well plate | Greiner Bio-One | 655096 | |
| Agar | Sigma-Aldrich | 5040 | |
| BglII | New England BioLabs | R0144S | |
| BJ5465 cells | American Type Culture Collection | 208289 | |
| Buffer MES 50 mM | Sigma-Aldrich | M8250 | |
| Buffer Tris-HCl 10 mM | Invitrogen | 15506017 | |
| EDTA 1 mM | Merck | 108452 | |
| Falcon tubes | Corning | 352057 | |
| LB media | Sigma-Aldrich | L2897 | |
| Lithium acetate 0.1 M | Sigma-Aldrich | L6883 | |
| Low Melt Agarose | GOLDBIO | A-204-25 | |
| Microcentrifuge | eppendorf | 5452000010 | |
| Miniprep kit | ZymoPure | D4210 | |
| NaOH 0.02 M | Merck | 106462 | |
| PEG 3,350 40% | Sigma-Aldrich | 1546547 | |
| plasmid pDRFLIP38-AtLEA4-5 | addgene | 178189 | |
| Plate reader | BMG LABTECH | CLARIOstar Plus | |
| SacI | New England BioLabs | R3156S | |
| Salmon sperm DNA 2 mg/mL | Thermo Fisher Scientific | 15632011 | |
| SD-Ura | Sigma-Aldrich | Y1501 | |
| Sodium cloride | Sigma-Aldrich | S9888 | |
| Taq polymesare | Promega | M5123 | |
| Transiluminator | Accuris instruments | E4000 | |
| UV-Visible spectrophotometer | Thermo Fisher Scientific | Biomate3 | |
| YPD media | Sigma-Aldrich | Y1500 |
Riferimenti
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nat Rev Mol Cell Biol. 16 (1), 18-29 (2015).
- Covarrubias, A. A., Romero-Pérez, P. S., Cuevas-Velazquez, C. L., Rendón-Luna, D. F. The functional diversity of structural disorder in plant proteins. Arch Biochem Biophys. 680, 108229 (2019).
- Ahmed, S. S., et al. Characterization of intrinsically disordered regions in proteins informed by human genetic diversity. PLoS Comput Biol. 18 (3), e1009911 (2022).
- Birol, M., Melo, A. M. Untangling the conformational polymorphism of disordered proteins associated with neurodegeneration at the single-molecule level. Front Mol Neurosci. 12, 309 (2019).
- Moses, D., et al. Revealing the hidden sensitivity of intrinsically disordered proteins to their chemical environment. J Phys Chem Lett. 11 (23), 10131-10136 (2020).
- Cuevas-Velazquez, C. L., et al. Intrinsically disordered protein biosensor tracks the physical-chemical effects of osmotic stress on cells. Nat Commun. 12 (1), 5438 (2021).
- Holehouse, A. S., Sukenik, S. Controlling structural bias in intrinsically disordered proteins using solution space scanning. J Chem Theory Comput. 16 (3), 1794-1805 (2020).
- Martin, E. W., Hopkins, J. B., Mittag, T. Small-angle X-ray scattering experiments of monodisperse intrinsically disordered protein samples close to the solubility limit. Methods Enzymol. 646, 185-222 (2021).
- Miles, A. J., Drew, E. D., Wallace, B. A. DichroIDP: a method for analyses of intrinsically disordered proteins using circular dichroism spectroscopy. Commun Biol. 6 (1), 823 (2023).
- Kaminski, C. F., Rees, E. J., Schierle, G. S. K. A quantitative protocol for intensity-based live cell FRET imaging. Method Mol Biol. 1076, 445-454 (2014).
- Moses, D., et al. Structural biases in disordered proteins are prevalent in the cell. bioRxiv. , (2022).
- Cuevas-Velazquez, C. L., Saab-Rincón, G., Reyes, J. L., Covarrubias, A. A. The Unstructured N-terminal Region of Arabidopsis Group 4 Late Embryogenesis Abundant (LEA) Proteins Is Required for Folding and for Chaperone-like Activity under Water Deficit. J Biol Chem. 291 (20), 10893-10903 (2016).
- JoVE Science Education Database. . Restriction enzyme digests. , (2023).
- JoVE Science Education Database. . Gel purification. , (2023).
- JoVE Science Education Database. . DNA ligation reactions. , (2023).
- JoVE Science Education Database. . Bacterial transformation using heat Ssock and competent cells. , (2023).
- JoVE Science Education Database. . PCR: Principle, instrumentation, and applications. , (2023).
- JoVE Science Education Database. . Plasmid purification. , (2023).
- JoVE Science Education Database. . DNA gel electrophoresis. , (2023).
- JoVE Core Molecular Biology. Sanger/chain termination sequencing using dideoxynucleotides - Concept Available from: https://app.jove.com/science-education/v/12020/sanger-sequencing (2023)
- Theillet, F. X., et al. Physicochemical properties of cells and their effects on intrinsically disordered proteins (IDPs). Chem Rev. 114 (13), 6661-6714 (2014).
- Brutscher, B., et al. NMR methods for the study of instrinsically disordered proteins structure, dynamics, and interactions: General overview and practical guidelines. Adv Exp Med Biol. 870, 49-122 (2015).
- Metskas, L. A., Rhoades, E. Single-molecule FRET of intrinsically disordered proteins. Annu Rev Phys Chem. 71, 391-414 (2020).
- Roebroek, T., et al. Simultaneous readout of multiple FRET pairs using photochromism. Nat Commun. 12 (1), 2005 (2021).
- Algar, W. R., Hildebrandt, N., Vogel, S. S., Medintz, I. L. FRET as a biomolecular research tool - understanding its potential while avoiding pitfalls. Nat Methods. 16 (9), 815-829 (2019).
- Lyon, A. S., Peeples, W. B., Rosen, M. K. A framework for understanding the functions of biomolecular condensates across scales. Nat Rev Mol Cell Biol. 22 (3), 215-235 (2021).
- Belott, C., Janis, B., Menze, M. A. Liquid-liquid phase separation promotes animal desiccation tolerance. Proc Natl Acad Sci U S A. 117 (44), 27676-27684 (2020).
- Miermont, A., et al. Severe osmotic compression triggers a slowdown of intracellular signaling, which can be explained by molecular crowding. Proc Natl Acad Sci U S A. 110 (14), 5725-5730 (2013).
- Saito, H., Posas, F. Response to hyperosmotic stress. Genetics. 192 (2), 289-318 (2012).
- Selenko, P., Wagner, G. Looking into live cells with in-cell NMR spectroscopy. J Struct Biol. 158 (2), 244-253 (2007).
- Cattani, J., Subramaniam, V., Drescher, M. Room-temperature in-cell EPR spectroscopy: alpha-Synuclein disease variants remain intrinsically disordered in the cell. Phys Chem Chem Phys. 19 (28), 18147-18151 (2017).
- Beveridge, R., Chappuis, Q., Macphee, C., Barran, P. Mass spectrometry methods for intrinsically disordered proteins. Analyst. 138 (1), 32-42 (2013).
- Beveridge, R., et al. Ion mobility mass spectrometry uncovers the impact of the patterning of oppositely charged residues on the conformational distributions of intrinsically disordered proteins. J Am Chem Soc. 141 (12), 4908-4918 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon