Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Оценка структурной чувствительности внутренне неупорядоченных участков в ответ на гиперосмотический стресс в живых клетках с помощью FRET
В этой статье
Резюме
Внутренне неупорядоченные области (IDR) представляют собой гибкие белковые домены, которые изменяют свою конформацию в ответ на изменения окружающей среды. Ансамблевый флуоресцентный резонансный перенос энергии (FRET) позволяет оценивать размеры белка в различных условиях. Мы представляем подход FRET для оценки структурной чувствительности РДЭ в живых клетках Saccharomyces cerevisiae в условиях гиперосмотического стресса.
Аннотация
Внутренне неупорядоченные области (IDR) — это белковые домены, которые участвуют в важнейших клеточных процессах. В стрессовых условиях изменяются физико-химические свойства клеточной среды, что напрямую влияет на конформационный ансамбль РДЭ. РДЭ по своей природе чувствительны к возмущениям окружающей среды. Изучение того, как физико-химические свойства клетки регулируют конформационный ансамбль РДЭ, имеет важное значение для понимания экологического контроля их функции. В данной статье мы опишем пошаговый метод измерения структурной чувствительности РДЭ в живых клетках Saccharomyces cerevisiae в ответ на гиперосмотические стрессовые условия. Мы представляем использование ансамбля флуоресцентного резонансного переноса энергии (FRET) для оценки того, как изменяются глобальные размеры РДЭ при прогрессирующем увеличении гиперосмотического стресса, наложенного на клетки с любым осмолитом. Кроме того, мы предоставляем скрипт для обработки флуоресцентных измерений и сравнения структурной чувствительности для разных РДЭ. Следуя этому методу, исследователи могут получить ценную информацию о конформационных изменениях, которые претерпевают IDR в сложной внутриклеточной среде при изменении окружающей среды.
Введение
Внутренне неупорядоченные области (РДЭ) являются важнейшими компонентами клеточных процессов1. В сочетании со структурированными доменами IDR имеют важное значение для белковых функций. Аминокислотный состав РДЭ смещен, представлен в основном заряженными, гидрофильными и мелкими остатками. Из-за этого свойства IDR считаются доменами низкой сложности 2,3. Многочисленные РДЭ привлекли к себе внимание, в первую очередь потому, что эти регионы играют решающую роль в патологических состояниях, особенно нейродегенеративных заболеваниях. Такие заболевания характеризуются самосборкой и последующим внеклеточным или внутриклеточным отложением РДЭ в нейронах4. Примерами таких РДЭ являются амилоид-β (Aβ) при болезни Альцгеймера, гентингтин (HTT) при болезни Хантингтона, а также TAR-ДНК-связывающий белок-43 (TDP-43) и слитый при саркоме (FUS) при боковом амиотрофическом склерозе и лобно-височной деменции4. Изучение структурных перестроек РДЭ в контексте заболевания было значительно расширено спектроскопическими методами, в том числе флуоресцентно-резонансным переносом энергии (FRET).
Гидрофильная и расширенная природа РДЭ делает их чрезвычайно чувствительными к изменениям физико-химических свойств растворной среды5. Степень, в которой конформационный ансамбль РДЭ модифицируется окружающей средой, называется структурной чувствительностью 5,6,7. Для изучения конформации и динамики РДЭ можно использовать различные методы, включая круговой дихроизм (КД) и малоугловое рассеяние рентгеновского излучения (SAXS)8,9. К сожалению, CD и SAXS требуют большого количества очищенных белков, поэтому они не подходят для исследований в клетках. В отличие от этого, FRET представляет собой метод, который измеряет интенсивность флуоресценции двух флуоресцентных молекул, которые специфически маркируют одну IDR, что означает, что их можно контролировать в сложных смесях, таких как живые клетки10. Динамическое измерение структурной чувствительности РДЭ в живых клетках необходимо для понимания того, как окружающая среда регулирует конформацию и функцию неупорядоченного протеома.
FRET является мощным методом количественной оценки структурной чувствительности IDR, а также глобулярных и мультидоменных белков в живых клетках. Для этого метода требуется конструкция, состоящая из интересующего IDR, зажатого между двумя флуоресцентными белками (FP), известных как пара FRET. Для этого протокола мы предлагаем использовать mCerulean3 в качестве донорного FP и Citrine в качестве акцепторного FP из-за их большого динамического диапазона по сравнению с другими FP, о которых сообщалось в предыдущем исследовании чувствительности IDR6. Ранее FRET использовался для измерения структурной чувствительности РДК растений в различных клеточных контекстах6. Кроме того, этот метод был использован для характеристики общих размеров белка IDR различными исследовательскими группами как in vitro, так и in vivo 5,11.
В данной работе мы описываем метод ансамбля FRET для изучения структурной чувствительности РДЭ в клетках живых дрожжей (Saccharomyces cerevisiae). Мы показываем репрезентативные результаты, основанные на РДЭ растений под названием AtLEA4-5. AtLEA4-5 неупорядочен в растворе, но сворачивается в α-спираль, когда макромолекулярная скученность индуцируется in vitro12. AtLEA4-5 является хорошей эталонной моделью для этого метода, поскольку он относительно мал (158 остатков), неупорядочен и чувствителен к возмущениям окружающей среды, о чем сообщалось in silico и in vitro 6,12. Представленный здесь метод может быть масштабирован для высокопроизводительных подходов, поскольку дрожжевые клетки легко выращивать, а обработка применяется в небольших объемах. Кроме того, небольшие модификации протокола могут быть применены к другим клеточным системам, таким как бактерии и растительные клетки6. Протокол может быть выполнен в любой молекулярно-биологической лаборатории, имеющей доступ к микропланшетному ридеру с режимом флуоресценции, оборудованию, имеющемуся в большинстве научно-исследовательских учреждений.
протокол
1. Плазмидная конструкция
- С помощью полимеразной цепной реакции (ПЦР) усилите открытую рамку считывания (ORF), которая кодирует желаемый IDR. Не включайте стоп-кодон, так как ORF будет окружен генами, кодирующими флуоресцентные белки. Для усиления разработайте праймеры с сайтами ограничения SacI (5') и BglII (3').
ПРИМЕЧАНИЕ: Для раздела репрезентативных результатов мы использовали AtLEA4-5 в качестве выбранного IDR. Мы амплифицировали КИН AtLEA4-5 из плазмиды pTrc99A-AtLEA4-512. - Расщепление продукта ПЦР ORF путем последовательного ограничения ферментами SacI и BglII13.
- Получение коммерческой плазмиды pDRFLIP38-AtLEA4-5 (#178189) из Addgene (https://www.addgene.org/178189/).
- Проводят последовательное ограничение с использованием ферментов SacI и BglII для удаления ORF AtLEA4-5 из плазмиды pDRFLIP38-AtLEA4-5, оставляя открытую плазмиду, содержащую пару FRET13.
- Очищают расщепленные фрагменты ДНК с помощью гелевого восстановления из электрофореза в агарозном геле14. Лигируют ограниченные фрагменты с помощью ДНК-лигазы15.
- Трансформируют реакцию клонирования в компетентные клетки Escherichia coli (DH5α или родственные штаммы) и отбирают в планшетах Лурии-Бертани (LB), содержащих 50 мкг/мл ампициллина16. Выращивайте в течение ночи (ON) при 37 °C.
- Изолируйте и вырастите не менее пяти трансформированных колоний в новой пластине и генотипе с помощью ПЦР17. Очищают плазмидную ДНК от положительно трансформированной колонии с помощью стандартных методов miniprep18.
- Проверяют экстракцию и целостность плазмидной ДНК с помощью электрофореза в агарозном геле19. Проверьте правильность клонирования конструкции с помощью секвенирования по Сэнгеру20.
2. Экспрессия плазмид в клетках дрожжей
ПРИМЕЧАНИЕ: Используйте стандартные асептические методы для выполнения следующих шагов. Используйте культуральный ламинарный колпак или зажигалку.
- Инокуляцию штамма S. cerevisiae BJ5465 (ATCC: 208289) в 3 мл среды дрожжевой пептон-декстрозы (YPD) и выращивают при 30 °C и 200 об/мин.
ПРИМЕЧАНИЕ: BJ5465 - это штамм с дефицитом протеазы (CH1), рекомендованный для работы с IDR. Другие штаммы S. cerevisiae могут быть протестированы и использованы. - Центрифуга 1 мл ночной насыщенной (OD600 ~ 3-4) культуры дрожжей при 14 000 x g в течение 1 мин. Осторожно удалите надосадочную жидкость пипетированием.
- Повторно суспендируйте гранулу в 1 мл буфера Tris-EDTA (TE) (pH 7,5), осторожно щелкнув пробиркой. Центрифугу при 14 000 x g в течение 1 мин и осторожно удалите надосадочную жидкость.
- Ресуспендант гранулы в 500 мкл буфера Lazy Bones (40% с ПЭГ 3,350; 100 мМ ацетата лития; 10 мМ Tris-HCl; 1 мМ ЭДТА; pH 7,5) путем пипетирования. Добавьте 25 мкл кипяченой (100 °C в течение 5 мин с последующим охлаждением на льду в течение 5 мин) ДНК сперматозоидов лосося (2 мг/мл) к ресуспендированным дрожжевым клеткам.
- Добавьте в смесь 100 нг плазмидной ДНК и перемешайте смесь в течение 1 минуты, чтобы обеспечить тщательное перемешивание. Дайте смеси постоять при комнатной температуре в течение 1-2 часов.
- Дрожжевая смесь нагревается при температуре 42 °C в течение 12 минут, затем охлаждается на льду еще 12 минут. Центрифугируют пробирку при 14 000 x g в течение 1 мин и осторожно удаляют надосадочную жидкость.
- Ресуспендируйте гранулу в 1 мл буфера TE (pH 7,5). Планшет 100 мкл трансформированных клеток на дрожжевой синтетической отсеточной среде без урацила (SD-ura) с добавлением 15 г/л агара.
- Инкубируют планшеты при 30 °C в течение 2-3 дней (рисунок 1). Процедите не менее пяти дрожжевых трансформантов в новую пластину и выращивайте дальше.
3. Валидация дрожжевых трансформантов
- Выберите небольшую порцию каждого кандидата-трансформанта, осторожно соскабливая и ресуспендируя в 5 мкл 20 мМ NaOH в отдельные пробирки для ПЦР.
- Нагрейте образец до 99 °C в течение 10 минут в амплификаторе, чтобы лизировать клетки и выпустить ДНК в раствор. Этот шаг имеет решающее значение для подготовки матрицы ДНК для ПЦР.
- Переносят 1 мкл кипяченого образца в отдельную реакционную смесь ПЦР, содержащую соответствующие праймеры и ПЦР-реагенты для генотипирования. Мы использовали следующие праймеры: Форвардный праймер: 5'-AAATATACCCCCCCCTCGATCTAGA-3'. Обратная капсюль: 5'-GTAATACGACTCACTATAGGGCG-3'.
- Настройте реакцию ПЦР и подтвердите наличие правильной плазмиды с помощью электрофореза в агарозном геле. Выберите один трансформатор для дальнейших экспериментов.
4. Подготовка культуры дрожжевых клеток к FRET анализу
- Высейте полоску выбранного дрожжевого трансформанта в 3 мл жидкой среды 1x SD-Ura.
ПРИМЕЧАНИЕ: Выполняйте эту процедуру в ламинарном вытяжном шкафу и стерильном материале. - Выращивать при температуре 30 °C, 200 об/мин не менее 12 часов до насыщения. Измерьте наружный диаметр600. Рост должен находиться в пределахOD 600 = 1.0-2.0.
ПРИМЕЧАНИЕ: Если OD600 ниже 1,0, дайте клеткам больше времени для инкубации. Если наружный диаметр600 превышает 2,0, не продолжайте и перезапустите с шага 4.1.
5. Подготовка дрожжевых клеток к анализу FRET
- Соберите 2 мл ночной дрожжевой культуры и центрифугируйте при 14 000 x g при комнатной температуре. Осторожно удалите надосадочную жидкость пипетированием.
- Ресуспендируйте гранулу в 1 мл буфера 50 мМ 2 (N-морфолино) этансульфоновой кислоты (MES) (pH 6).
- Центрифуга при 14 000 x g при комнатной температуре. Удалите надосадочную жидкость.
- Повторите шаги 5.2 и 5.3. Дрожжевые клетки ресуспендировать в 2 мл буфера MES, рН 6 и перелить объем в резервуар с реагентом.
6. Настройка флуоресцентных измерений
ПРИМЕЧАНИЕ: Данное сканирование считается выполненным в микропланшетном ридере с режимом флуоресценции.
- Для основных настроек установите прибор следующим образом: Тип измерения: Спектр интенсивности флуоресценции (FI); Название микропластины: Greiner 96 F-bottom.
- Для настройки оптики установите прибор следующим образом: Нет. точек развертки длин волн: 91; Длина волны возбуждения: 433 нм; Полоса возбуждения: 10 нм; Длина волны излучения: 460 - 550; Ширина шага: 1 нм; Полоса пропускания излучения: 10 нм; Фокусная высота, полученная автофокусом; Фокусное расстояние: 5 мм; Длина волны, используемая для усиления: 490 нм.
- Для общих настроек установите прибор следующим образом: Время установления: 0,1 с; Направление чтения: однонаправленное, горизонтальное слева направо, сверху вниз.
ПРИМЕЧАНИЕ: Коэффициент усиления, введенный вручную, должен быть отрегулирован для каждого измерения с использованием скважины, которая, как ожидается, будет отображать самые высокие уровни интенсивности флуоресценции на протяжении всего сканирования длины волны.
7. Приготовление гипертонических растворов и флуоресцентные измерения
- Приготовьте растворы 0 М, 0,2 М, 0,4 М, 0,6 М, 0,8 М, 1 М, 1,5 М и 2 М NaCl в 96-луночном черном планшете с прозрачным дном. Окончательный объем в каждой лунке составит 200 мкл (150 мкл раствора осмолита + 50 мкл суспензии дрожжевых клеток).
ПРИМЕЧАНИЕ: Другие осмолиты могут быть использованы для индуцирования гиперосмотического стресса. Все растворы должны быть приготовлены в буфере MES 50 мМ с рН 6. - Загрузите 150 мкл 50 мМ буфера MES, рН 6 в первые три лунки ряда А (А1, А2, А3) и в последующие лунки добавьте 150 мкл соответствующего раствора в порядке возрастания слева направо (рисунок 2). Чтобы проверить все предложенные концентрации, продолжайте загрузку в ряду B.
ПРИМЕЧАНИЕ: Эта настройка учитывает измерения 3 технических повторов для каждой концентрации. - С помощью 12-канальной микропипетки перенесите 50 мкл промытых дрожжевых клеток в каждую лунку. Дрожжевые клетки имеют свойство оседать на дно водоема. Обязательно тщательно ресуспендируйте дрожжевую суспензию, пипетируя вверх и вниз перед переносом клеток.
- Поместите тензодатчики в 96-луночную пластину, содержащую различные растворы, и хорошо перемешайте, пипетируя вверх и вниз не менее 4 раз.
- Немедленно измерьте спектры интенсивности излучения флуоресценции для желаемых лунок в микропланшетном считывателе с настройками спецификации шага 6. Повторите процедуру для каждого IDR, который будет проверяться в анализе FRET.
- Необязательный шаг. Если возможно, измерьте конструкцию, предназначенную только для доноров (pDRFLIP38-mCerulean3), выполнив действия, описанные здесь.
ПРИМЕЧАНИЕ: Мы рекомендуем выполнить этот шаг, чтобы рассчитать эффективность FRET для каждого условия. Если считыватель не может выполнить этот шаг, FRET можно рассчитать с помощью метода коэффициента FRET. Оба метода расчета FRET описаны на шагах 8 и 9. - Выполните по крайней мере три репликации в течение трех независимых дней для каждой конструкции.
8. Обработка данных по методу эффективности FRET
ПРИМЕЧАНИЕ: Для читателей, знакомых с языком программирования R, мы предоставляем набор скриптов R для обработки данных, описанных в этом разделе. Скрипты можно найти на https://github.com/Kaz-bits/cuevaslab-procotols/tree/main/FRET. Следуйте инструкциям в файле README.
- Экспортируйте данные в виде .xlsx файлов из считывателя микропланшетов.
- Нормализовать значения интенсивности флуоресценции на каждой длине волны сканирования до изобестовой точки используемой пары FRET.
ПРИМЕЧАНИЕ: Этот шаг необходим для сравнения спектров всех тестируемых условий. Изобестическая точка пары mCerulean3 и Citrine FRET составляет 515 нм. Например, получить отношение значений интенсивности флуоресценции 460 нм/515 нм, 461 нм/515 нм, 462 нм/515 нм и так далее. - Вычислите среднее значение для каждой технической репликации после нормализации значений флуоресценции. Всего для каждого состояния гиперосмотического стресса должна быть своя колонка.
- Визуализируйте все спектры интенсивности флуоресценции на одном графике (рис. 3). Каждый спектр должен отображать комбинацию спектров флуоресцентного излучения отдельных mCerulean3 и Citrine. В редких случаях, когда эффективность FRET равна 0, будет наблюдаться только спектр излучения флуоресценции донора, или, когда эффективность FRET равна 1, будет наблюдаться только флуоресценция акцептора. Пик флуоресцентного излучения для mCerulean3 (донор) составляет 475 нм, а пик флуоресцентного излучения для Citrine (акцептор) — 525 нм.
- Определите, показывает ли измерение флуоресценции типичное изменение FRET в ответ на гиперосмотическое напряжение. Если конформация IDR чувствительна к лечению, это отразится как изменение эффективности FRET. Типичное изменение эффективности FRET связано со снижением интенсивности флуоресценции донора с увеличением интенсивности флуоресценции акцептора или наоборот.
- Количественная оценка эффективности FRET (EFRET). Получить нормализованные пиковые значения донора (mCerulean3) (475 нм/515 нм) для конструкции IDR и конструкции только для донора для каждого состояния гиперосмотического стресса. Используйте средние нормализованные пиковые значения технических репликаций. Выполните эту операцию для каждой из трех независимых репликаций. Рассчитайте EFRET по следующей формуле:
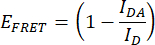
Где IDA — отношение 475 нм/515 нм от конструкции IDR (конструкция пары FRET), а ID — отношение 475 нм/515 нм от конструкции только для доноров. - Сравните эффективность FRET. Нормализуйте значения EFRET каждого гиперосмотического стрессового состояния к EFRET нестрессового состояния (например, 0 M NaCl). Проводите сравнения с помощью ящичковой диаграммы или графика с гладкой кривой. Выполните односторонний ANOVA с последующим статистическим тестом Тьюки post hoc (p-значения: *p < 0,05, **p < 0,01, ***p < 0,001, ****p < 0,0001).
9. Обработка данных методом коэффициента FRET
ПРИМЕЧАНИЕ: Если измерения только с помощью доноров не могут быть получены, выполните метод коэффициента FRET. Этот метод сравнивает соотношение FRET в различных условиях гиперосмотического стресса. Шаги с 7.1 по 7.5 должны быть выполнены перед шагами этого раздела, чтобы проверить типичное поведение FRET. Для читателей, знакомых с языком программирования R, мы предоставляем набор скриптов R для выполнения обработки данных, описанных в этом разделе. Скрипты можно найти на https://github.com/Kaz-bits/cuevaslab-procotols/tree/main/FRET. Следуйте инструкциям в файле README.
- Извлечение данных на длинах волн 475 нм и 525 нм из файла xlsx, ранее экспортированного из считывателя микропланшетов. Эти значения соответствуют пику флуоресцентного излучения для mCerulean3 (донор, DxDm) и пику флуоресцентного излучения для цитрина (акцептор, DxAm) при возбуждении донорного флуорофора (433 нм).
- Нормализовать значения интенсивности флуоресценции на длинах волны 475 нм и 525 нм до изобестовой точки используемой пары FRET. Изобестическая точка пары mCerulean3 и Citrine FRET составляет 515 нм. Получаем соотношение FRET (DxAm/DxDm).
- Сравните коэффициенты FRET. Нормализуйте значения DxAm/DxDm для каждого гиперосмотического стрессового состояния к среднему значению DxAm/DxDm для нестрессового состояния (например, 0 M NaCl). Проведите сравнение, используя ящичковую диаграмму или график плавной кривой (рис. 4 и рис. 5). Выполните односторонний анализ дисперсии с последующим статистическим тестом Тьюки post hoc (p-значения: *p < 0,05, **p < 0,01, ***p < 0,001, ****p < 0,0001).
Результаты
После трансформации дрожжевых клеток плазмидой pDRFLIP38-AtLEA4-5 наблюдалась флуоресценция положительных трансформантов с помощью трансиллюминатора синего света и фильтра (рис. 1). Приготовление различных растворов для индуцирования гиперосмотического стресса занимает мно...
Обсуждение
Представленный здесь метод позволяет получить представление о том, как глобальные измерения ансамбля РДЭ воспринимают и реагируют на возмущения окружающей среды. Этот метод основан на генетически закодированной конструкции и не требует никаких дополнительных компонентов, кроме ста?...
Раскрытие информации
Авторы заявляют об отсутствии конфликта интересов.
Благодарности
Благодарим сотрудников лаборатории Куэваса-Веласкеса за критическое рецензирование рукописи. Эта работа была поддержана Programa de Apoyo a Proyectos de Investigación e Innovación Tecnológica, Dirección General de Asuntos del Personal Académico, Universidad Nacional Autónoma de México (UNAM-PAPIIT), проект No IA203422; Consejo Nacional de Humanidades, Ciencias y Tecnología (CONAHCYT), проект номер 252952; и Programa de Apoyo a la Investigación y el Posgrado, Facultad de Química, Universidad Nacional Autónoma de México, Grant 5000-9182. CET (CVU 1083636) и CAPD (CVU 1269643) благодарят CONAHCYT за стипендию M.Sc.
Материалы
| Name | Company | Catalog Number | Comments |
| 96-well plate | Greiner Bio-One | 655096 | |
| Agar | Sigma-Aldrich | 5040 | |
| BglII | New England BioLabs | R0144S | |
| BJ5465 cells | American Type Culture Collection | 208289 | |
| Buffer MES 50 mM | Sigma-Aldrich | M8250 | |
| Buffer Tris-HCl 10 mM | Invitrogen | 15506017 | |
| EDTA 1 mM | Merck | 108452 | |
| Falcon tubes | Corning | 352057 | |
| LB media | Sigma-Aldrich | L2897 | |
| Lithium acetate 0.1 M | Sigma-Aldrich | L6883 | |
| Low Melt Agarose | GOLDBIO | A-204-25 | |
| Microcentrifuge | eppendorf | 5452000010 | |
| Miniprep kit | ZymoPure | D4210 | |
| NaOH 0.02 M | Merck | 106462 | |
| PEG 3,350 40% | Sigma-Aldrich | 1546547 | |
| plasmid pDRFLIP38-AtLEA4-5 | addgene | 178189 | |
| Plate reader | BMG LABTECH | CLARIOstar Plus | |
| SacI | New England BioLabs | R3156S | |
| Salmon sperm DNA 2 mg/mL | Thermo Fisher Scientific | 15632011 | |
| SD-Ura | Sigma-Aldrich | Y1501 | |
| Sodium cloride | Sigma-Aldrich | S9888 | |
| Taq polymesare | Promega | M5123 | |
| Transiluminator | Accuris instruments | E4000 | |
| UV-Visible spectrophotometer | Thermo Fisher Scientific | Biomate3 | |
| YPD media | Sigma-Aldrich | Y1500 |
Ссылки
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nat Rev Mol Cell Biol. 16 (1), 18-29 (2015).
- Covarrubias, A. A., Romero-Pérez, P. S., Cuevas-Velazquez, C. L., Rendón-Luna, D. F. The functional diversity of structural disorder in plant proteins. Arch Biochem Biophys. 680, 108229 (2019).
- Ahmed, S. S., et al. Characterization of intrinsically disordered regions in proteins informed by human genetic diversity. PLoS Comput Biol. 18 (3), e1009911 (2022).
- Birol, M., Melo, A. M. Untangling the conformational polymorphism of disordered proteins associated with neurodegeneration at the single-molecule level. Front Mol Neurosci. 12, 309 (2019).
- Moses, D., et al. Revealing the hidden sensitivity of intrinsically disordered proteins to their chemical environment. J Phys Chem Lett. 11 (23), 10131-10136 (2020).
- Cuevas-Velazquez, C. L., et al. Intrinsically disordered protein biosensor tracks the physical-chemical effects of osmotic stress on cells. Nat Commun. 12 (1), 5438 (2021).
- Holehouse, A. S., Sukenik, S. Controlling structural bias in intrinsically disordered proteins using solution space scanning. J Chem Theory Comput. 16 (3), 1794-1805 (2020).
- Martin, E. W., Hopkins, J. B., Mittag, T. Small-angle X-ray scattering experiments of monodisperse intrinsically disordered protein samples close to the solubility limit. Methods Enzymol. 646, 185-222 (2021).
- Miles, A. J., Drew, E. D., Wallace, B. A. DichroIDP: a method for analyses of intrinsically disordered proteins using circular dichroism spectroscopy. Commun Biol. 6 (1), 823 (2023).
- Kaminski, C. F., Rees, E. J., Schierle, G. S. K. A quantitative protocol for intensity-based live cell FRET imaging. Method Mol Biol. 1076, 445-454 (2014).
- Moses, D., et al. Structural biases in disordered proteins are prevalent in the cell. bioRxiv. , (2022).
- Cuevas-Velazquez, C. L., Saab-Rincón, G., Reyes, J. L., Covarrubias, A. A. The Unstructured N-terminal Region of Arabidopsis Group 4 Late Embryogenesis Abundant (LEA) Proteins Is Required for Folding and for Chaperone-like Activity under Water Deficit. J Biol Chem. 291 (20), 10893-10903 (2016).
- JoVE Science Education Database. . Restriction enzyme digests. , (2023).
- JoVE Science Education Database. . Gel purification. , (2023).
- JoVE Science Education Database. . DNA ligation reactions. , (2023).
- JoVE Science Education Database. . Bacterial transformation using heat Ssock and competent cells. , (2023).
- JoVE Science Education Database. . PCR: Principle, instrumentation, and applications. , (2023).
- JoVE Science Education Database. . Plasmid purification. , (2023).
- JoVE Science Education Database. . DNA gel electrophoresis. , (2023).
- JoVE Core Molecular Biology. Sanger/chain termination sequencing using dideoxynucleotides - Concept Available from: https://app.jove.com/science-education/v/12020/sanger-sequencing (2023)
- Theillet, F. X., et al. Physicochemical properties of cells and their effects on intrinsically disordered proteins (IDPs). Chem Rev. 114 (13), 6661-6714 (2014).
- Brutscher, B., et al. NMR methods for the study of instrinsically disordered proteins structure, dynamics, and interactions: General overview and practical guidelines. Adv Exp Med Biol. 870, 49-122 (2015).
- Metskas, L. A., Rhoades, E. Single-molecule FRET of intrinsically disordered proteins. Annu Rev Phys Chem. 71, 391-414 (2020).
- Roebroek, T., et al. Simultaneous readout of multiple FRET pairs using photochromism. Nat Commun. 12 (1), 2005 (2021).
- Algar, W. R., Hildebrandt, N., Vogel, S. S., Medintz, I. L. FRET as a biomolecular research tool - understanding its potential while avoiding pitfalls. Nat Methods. 16 (9), 815-829 (2019).
- Lyon, A. S., Peeples, W. B., Rosen, M. K. A framework for understanding the functions of biomolecular condensates across scales. Nat Rev Mol Cell Biol. 22 (3), 215-235 (2021).
- Belott, C., Janis, B., Menze, M. A. Liquid-liquid phase separation promotes animal desiccation tolerance. Proc Natl Acad Sci U S A. 117 (44), 27676-27684 (2020).
- Miermont, A., et al. Severe osmotic compression triggers a slowdown of intracellular signaling, which can be explained by molecular crowding. Proc Natl Acad Sci U S A. 110 (14), 5725-5730 (2013).
- Saito, H., Posas, F. Response to hyperosmotic stress. Genetics. 192 (2), 289-318 (2012).
- Selenko, P., Wagner, G. Looking into live cells with in-cell NMR spectroscopy. J Struct Biol. 158 (2), 244-253 (2007).
- Cattani, J., Subramaniam, V., Drescher, M. Room-temperature in-cell EPR spectroscopy: alpha-Synuclein disease variants remain intrinsically disordered in the cell. Phys Chem Chem Phys. 19 (28), 18147-18151 (2017).
- Beveridge, R., Chappuis, Q., Macphee, C., Barran, P. Mass spectrometry methods for intrinsically disordered proteins. Analyst. 138 (1), 32-42 (2013).
- Beveridge, R., et al. Ion mobility mass spectrometry uncovers the impact of the patterning of oppositely charged residues on the conformational distributions of intrinsically disordered proteins. J Am Chem Soc. 141 (12), 4908-4918 (2019).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены