このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
FRETを用いた生細胞の高浸透圧ストレスに応答した内在性変性領域の構造感受性の推定
要約
天然変性領域(IDR)は、環境変化に応答してコンフォメーションを変化させる柔軟なタンパク質ドメインです。アンサンブル蛍光共鳴エネルギー移動(FRET)は、さまざまな条件下でタンパク質の寸法を推定できます。高浸透圧ストレス下で生きた 出芽酵母 細胞のIDR構造感受性を評価するためのFRETアプローチを提示します。
要約
天然変性領域(IDR)は、重要な細胞プロセスに関与するタンパク質ドメインです。ストレス状態では、細胞環境の物理化学的特性が変化し、IDRのコンフォメーションアンサンブルに直接影響します。IDRは本質的に環境の摂動に敏感です。細胞の物理化学的性質がIDRの立体構造アンサンブルをどのように制御するかを研究することは、その機能の環境制御を理解するために不可欠です。ここでは、高浸透圧ストレス条件に応答して、生きた出 芽酵母 細胞におけるIDRの構造感受性を測定するための段階的な方法について説明します。アンサンブル蛍光共鳴エネルギー移動(FRET)を使用して、浸透圧を有する細胞に課せられる高浸透圧ストレスの漸進的な増加中にIDRの全体的な寸法がどのように変化するかを推定します。さらに、蛍光測定を処理し、異なるIDRの構造感度を比較するためのスクリプトを提供しています。この方法に従うことで、研究者は、環境の変化に応じてIDRが複雑な細胞内環境で受ける立体構造の変化に関する貴重な洞察を得ることができます。
概要
天然変性領域(IDR)は、細胞プロセスにおける重要な構成要素です1。構造化ドメインと組み合わせて、IDRはタンパク質機能に不可欠です。IDRのアミノ酸組成は偏っており、主に荷電残基、親水性残基、および小残基で表されます。この特性により、IDRは複雑度の低いドメインと見なされる2,3。多くのIDRが注目されているのは、これらの領域が病態、特に神経変性疾患において重要な役割を果たしていることが主な理由です。このような疾患は、自己組織化とそれに続くニューロンにおけるIDRの細胞外または細胞内沈着を特徴とする4。例えば、アルツハイマー病のアミロイドβ(Aβ)、ハンチントン病のハンチンチン(HTT)、筋萎縮性側索硬化症や前頭側頭型認知症のTAR DNA結合タンパク質-43(TDP-43)や肉腫で融合したタンパク質(FUS)などが挙げられる4。疾患におけるIDRの構造再配列の研究は、蛍光共鳴エネルギー移動(FRET)などの分光法によって大幅に強化されています。
IDRの親水性と伸長性により、溶液環境の物理化学的特性の変化に非常に敏感になります5。IDRの立体配座アンサンブルが環境によって変化する度合いを構造感度5,6,7と呼ぶ。IDRの立体構造とダイナミクスの研究には、円二色性(CD)や小角X線散乱(SAXS)など、さまざまな手法が用いられます8,9。残念ながら、CDやSAXSは精製タンパク質を大量に必要とするため、細胞での研究には適していません。対照的に、FRETは、1つのIDRを特異的に標識する2つの蛍光分子の蛍光強度を測定する技術であり、生細胞などの複雑な混合物でそれらをモニターすることができることを意味する10。生細胞におけるIDRの構造感受性を動的に測定することは、環境が無秩序なプロテオームのコンフォメーションと機能をどのように制御しているかを理解するために必要です。
FRETは、生細胞内のIDR、球状タンパク質、マルチドメインタンパク質の構造感受性を定量化するための強力な方法です。この方法では、FRETペアとして知られる2つの蛍光タンパク質(FP)の間に挟まれた目的のIDRからなるコンストラクトが必要です。このプロトコルでは、IDRの感度に関する以前の研究で報告された他のFPと比較して、ダイナミックレンジが広いため、ドナーFPとしてmCerulean3を、アクセプターFPとしてシトリンを使用することを提案します6。FRETは、異なる細胞環境における植物IDRの構造的感受性を測定するために以前に利用されてきました6。さらに、この手法は、in vitroとin vivoの両方でさまざまな研究グループによってIDRの全体的なタンパク質寸法を特徴付けるために使用されています5,11。
ここでは、生きた酵母(Saccharomyces cerevisiae)細胞におけるIDRの構造感受性を研究するためのアンサンブルFRET法について説明します。AtLEA4-5と呼ばれるプラントIDRに基づく代表的な結果を示します。AtLEA4-5は溶液中で無秩序であるが、in vitroで高分子クラウディングが誘導されるとαヘリックスに折り畳まれる12。AtLEA4-5は、in silicoおよびin vitroで報告されているように、比較的小さく(158残基)、無秩序で環境摂動に敏感であるため、この方法の優れた参照モデルです6,12。ここで紹介する方法は、酵母細胞は増殖しやすく、処理量も少量であるため、ハイスループットアプローチに拡張できます。さらに、プロトコルへの小さな修正は、細菌および植物細胞6などの他の細胞系に適用することができる。このプロトコルは、ほとんどの研究機関で利用可能な機器である蛍光モードを備えたマイクロプレートリーダーにアクセスできる分子生物学研究室で行うことができます。
プロトコル
1. プラスミドコンストラクト
- ポリメラーゼ連鎖反応(PCR)によって目的のIDRをコードするオープンリーディングフレーム(ORF)を増幅します。終止コドンは、蛍光タンパク質をコードする遺伝子に挟まれるため、含めないでください。増幅には、 SacI(5')および BglII(3')の制限部位を持つプライマーを設計します。
注:代表的な結果のセクションでは、選択したIDRとしてAtLEA4-5を使用しました。pTrc99A-AtLEA4-5プラスミド12からAtLEA4-5のORFを増幅した。 - SacIおよびBglII酵素による連続制限によりORF PCR産物を消化する13。
- 市販のプラスミド pDRFLIP38-AtLEA4-5 (#178189) を Addgene (https://www.addgene.org/178189/) から入手します。
- SacIおよびBglII酵素を用いて連続的な制限を行い、pDRFLIP38-AtLEA4-5プラスミドからAtLEA4-5 ORFを除去し、FRETペアを含むオープンプラスミドを残します13。
- アガロースゲル電気泳動からのゲル回収を用いて、消化されたDNA断片を精製する14.DNAリガーゼ15を用いて制限された断片をライゲーションする。
- クローニング反応をコンピテント大 腸菌 細胞(DH5αまたは関連株)に形質転換し、50 μg/mLのアンピシリンを含むLuria-Bertani(LB)プレートで選択します16。37°Cで一晩(ON)成長させます。
- PCR17によって新しいプレートと遺伝子型で少なくとも5つの形質転換コロニーを分離し、増殖させます。標準的なミニプレップ法を用いて、ポジティブ形質転換コロニーからプラスミドDNAを精製する18。
- アガロースゲル電気泳動を用いてプラスミドDNAの抽出と完全性を検証します19.サンガーシーケンシング20を用いて、コンストラクトの正しいクローニングを確認します。
2. 酵母細胞におけるプラスミド発現
メモ: 標準的な無菌技術を使用して、次の手順を実行します。培養層流フードまたはライターを使用してください。
- 出芽酵母BJ5465(ATCC:208289)株を酵母ペプトンデキストロース(YPD)培地3 mLに接種し、30°C、200rpmでON増殖させます。
注:BJ5465は、IDRの取り扱いに推奨されるプロテアーゼ欠損(CH1)株です。他の 出芽酵母 株は、試験して使用することができます。 - 一晩飽和(OD600 ~3-4)酵母培養液1mLを14,000× g で1分間遠心分離する。ピペッティングで上清を慎重に除去します。
- チューブを静かにフリックして、ペレットを1 mLのTris-EDTA(TE)緩衝液(pH 7.5)に再懸濁します。14,000 x g で1分間遠心分離し、上清を慎重に除去します。
- ペレットを500 μLのLazy Bonesバッファー(40%w/v PEG 3,350;100 mM酢酸リチウム;10 mM Tris-HCl;1 mM EDTA;pH 7.5)にピペッティングで再懸濁します。再懸濁した酵母細胞に、25 μLの茹でたサケ精子DNA(100°Cで5分間、続いて氷上で5分間冷却)を加えます。
- 100 ng のプラスミド DNA を混合物に加え、混合物を 1 分間ボルテックスして、完全に混合します。混合物を室温で1〜2時間放置します。
- 酵母混合物を42°Cで12分間ヒートショックし、氷上でさらに12分間冷却します。チューブを14,000 x g で1分間遠心分離し、上清を慎重に除去します。
- ペレットを1 mLのTE緩衝液(pH 7.5)に再懸濁します。形質転換した細胞100 μLを、15 g/L寒天培地を添加したウラシル(SD-ura)プレートを添加せずに酵母合成ドロップアウト培地に播種します。
- プレートを30°Cで2〜3日間インキュベートします(図1)。新しいプレートに少なくとも5つの酵母形質転換体をストリークし、成長させます。
3. 酵母形質転換体のバリデーション
- 5 μL の 20 mM NaOH を静かに掻き取り、個々の PCR チューブに再懸濁することにより、各形質転換候補のごく一部をピックします。
- サンプルをサーマルサイクラーで99°Cで10分間加熱して細胞を溶解し、DNAを溶液中に放出します。このステップは、PCR用のDNAテンプレートを調製するために重要です。
- 煮沸したサンプル1 μLを、ジェノタイピングに適したプライマーとPCR試薬を含む別のPCR反応ミックスに移します。以下のプライマーを使用した:フォワードプライマー:5'-AAATATACCCCAGCCTCGATCTAGA-3'。リバースプライマー:5'-GTAATACGACTCACTATAGGGCG-3'。
- PCR反応をセットアップし、アガロースゲル電気泳動を使用して正しいプラスミドの存在を検証します。さらなる実験のために形質転換体を1つ選択します。
4. FRETアッセイのための酵母細胞培養液の調製
- 選択した酵母形質転換体のストリークを、3 mLの液体1x SD-Ura培地に接種します。
注意: この手順は、層流フードと滅菌材料で行ってください。 - 30°C、200rpmで少なくとも12時間成長させ、飽和させます。OD600を測定します。成長率はOD600 = 1.0-2.0の間になります。
注:OD600 が1.0未満の場合は、細胞が増殖するまでのインキュベーション時間を長くしてください。OD600 が 2.0 を超える場合は、続行せずに手順 4.1 からやり直してください。
5. FRETアッセイのための酵母細胞の調製
- 2 mLの酵母培養液を回収し、室温で14,000 x g で遠心分離します。ピペッティングで上清を慎重に除去します。
- ペレットを50 mM 2-(N-モルホリノ)エタンスルホン酸(MES)緩衝液1 mL(pH 6)に再懸濁します。
- 室温で14,000 x g で遠心分離します。上澄みを取り除きます。
- 手順 5.2 と 5.3 を繰り返します。酵母細胞を2 mLのMES緩衝液(pH 6)に再懸濁し、試薬リザーバーに注ぎます。
6. 蛍光測定の設定
注:本スキャンは、蛍光モードのマイクロプレートリーダーで行われるものと考えられます。
- 基本設定については、機器を次のように設定します。 測定タイプ:蛍光強度(FI)スペクトル。マイクロプレート名:Greiner 96 F-bottom。
- 光学系設定の場合、機器を次のように設定します。 いいえ。波長スキャンポイント数:91;励起波長:433nm;励起帯域幅:10 nm;発光波長:460 - 550;ステップ幅:1 nm;発光帯域幅:10nm;オートフォーカスによって得られた焦点高。焦点高:5 mm;ゲインに使用される波長:490nm。
- 一般設定では、機器を次のように設定します:セトリング時間:0.1秒。読み取り方向:一方向、水平左から右、上から下。
注:手動で入力したゲインは、波長スキャン全体で最高の蛍光強度レベルを表示すると予想されるウェルを使用して、測定ごとに調整する必要があります。
7. 高張溶液の調製と蛍光測定
- 0 M、0.2 M、0.4 M、0.6 M、0.8 M、1 M、1.5 M、2 M NaClの溶液を96ウェル透明底の黒色プレートに調製します。各ウェルの最終容量は200μL(浸透圧溶液150μL+酵母細胞懸濁液50μL)です。
注:他の浸透圧は、高浸透圧ストレスを誘発するために使用できます。すべての溶液は、50 mM MES緩衝液(pH 6)で調製する必要があります。 - 列Aの最初の3つのウェル(A1、A2、A3)にpH 6の50 mM MESバッファー150 μLをロードし、後続のウェルに、対応する溶液150 μLを左から右に順に加えます(図2)。提案されたすべての濃度をテストするには、行Bでローディングを続けます。
注:この設定では、各濃度について3つのテクニカルレプリケートの測定値が考慮されます。 - 12チャンネルのマイクロピペットを使用して、洗浄した酵母細胞50 μLを各ウェルに移します。酵母細胞は貯水池の底に沈殿する傾向があります。細胞を移す前に、ピペッティングで上下にピペッティングして酵母懸濁液を完全に再懸濁してください。
- さまざまな溶液を含む96ウェルプレートにセルをロードし、少なくとも4回ピペッティングしてよく混合します。
- ステップ6の仕様設定を使用して、マイクロプレートリーダーで目的のウェルの蛍光強度発光スペクトルを直ちに測定します。FRETアッセイで検査するすべてのIDRについて、この手順を繰り返します。
- 省略可能な手順。可能な場合は、ここで説明する手順に従って、ドナーのみのコンストラクト(pDRFLIP38-mCerulean3)を測定します。
注: この手順を実行して、各条件の FRET 効率を計算することをお勧めします。リーダーがこの手順を実行できない場合は、FRET比法を使用してFRETを計算できます。FRET計算の2つの方法については、手順8と9で説明します。 - 各構成体について、3 つの独立した日に少なくとも 3 回の反復を実行します。
8. FRET効率化法によるデータ処理
注: R プログラミング言語の知識を持つ読者向けに、このセクションで説明するデータ処理を実行するための一連の R スクリプトが用意されています。スクリプトは https://github.com/Kaz-bits/cuevaslab-procotols/tree/main/FRET にあります。README ファイルの指示に従ってください。
- マイクロプレートリーダーからデータを.xlsxファイルとしてエクスポートします。
- すべてのスキャン波長における蛍光強度値を、使用するFRETペアのアイソベスト点に正規化します。
注:この手順は、テストしたすべての条件のスペクトルを比較するために必要です。mCerulean3とCitrine FRETペアのアイソベスト点は515 nmです。例えば、460 nm/515 nm、461 nm/515 nm、462 nm/515 nmなどの蛍光強度値の比を求めます。 - 蛍光値が正規化されたら、すべてのテクニカルレプリケートの平均を計算します。合計で、高浸透圧ストレスのすべての状態の列があるはずです。
- すべての蛍光強度スペクトルを同じプロットで可視化します(図3)。各スペクトルは、個々のmCerulean3とCitrineの蛍光発光スペクトルの組み合わせを表示する必要があります。稀に、FRET効率が0の場合、ドナー蛍光発光スペクトルのみが観察され、FRET効率が1の場合、アクセプター蛍光のみが観察されます。mCerulean3(ドナー)の蛍光発光ピークは475 nm、シトリン(アクセプター)の蛍光発光ピークは525 nmです。
- 蛍光測定で、高浸透圧ストレスに応答した典型的なFRETの変化が見られるかどうかを確認します。IDRのコンフォメーションが治療に敏感な場合、これはFRET効率の変化として反映されます。FRET効率の典型的な変化は、ドナーの蛍光強度の低下とアクセプターの蛍光強度の増加、またはその逆を伴います。
- FRET効率(EFRET)を定量化します。高浸透圧ストレスのすべての条件について、IDRコンストラクトおよびドナーのみのコンストラクトについて、ドナー(mCerulean3)正規化ピーク値(475 nm/515 nm)を取得します。テクニカル反復の平均正規化ピーク値を使用します。この操作は、3 つの独立した反復のそれぞれに対して実行します。EFRET は、次の式で計算します。
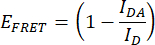
ここで、IDA はIDRコンストラクト(FRETペアコンストラクト)からの比475 nm/515 nmであり、ID はドナーのみのコンストラクトからの比475 nm/515 nmである。 - FRETの効率を比較します。すべての高浸透圧ストレス条件の EFRET 値を、非ストレス条件のEFRET 値(たとえば、0 M NaCl)に正規化します。箱ひげ図または滑らかな曲線プロットを使用して比較を行います。一元配置分散分析の後に事後テューキー統計的検定を実行します(p値:*p < 0.05、**p < 0.01、***p < 0.001、****p < 0.0001)。
9. FRET比法によるデータ処理
注:ドナーのみの測定値を取得できない場合は、FRET比法を実行してください。この方法では、さまざまな高浸透圧ストレス条件でFRET比を比較します。手順 7.1 から 7.5 は、このセクションの手順の前に実行して、一般的な FRET の動作を検証する必要があります。R プログラミング言語の知識を持つ読者向けに、このセクションで説明するデータ処理を実行するための一連の R スクリプトが用意されています。スクリプトは https://github.com/Kaz-bits/cuevaslab-procotols/tree/main/FRET にあります。README ファイルの指示に従ってください。
- 475 nm および 525 nm のデータを、以前にマイクロプレートリーダーからエクスポートした xlsx ファイルから抽出します。これらの値は、mCerulean3(ドナー、DxDm)の蛍光発光ピークと、ドナー蛍光色素が励起されたとき(433 nm)のシトリン(アクセプター、DxAm)の蛍光発光ピークに対応します。
- 475 nm および 525 nm の蛍光強度値を、使用した FRETペアのアイソベスト点に正規化します。mCerulean3とCitrine FRETペアのアイソベスト点は515 nmです。FRET比(DxAm/DxDm)を求めます。
- FRET比を比較します。すべての高浸透圧ストレス条件の DxAm/DxDm 値を、非ストレス条件の平均 DxAm/DxDm に正規化します (たとえば、0 M NaCl)。箱ひげ図または滑らかな曲線プロットを使用して比較を行います(図4 および 図5)。一元配置分散分析とそれに続く事後テューキー統計的検定を実行します(p値:*p < 0.05、**p < 0.01、***p < 0.001、****p < 0.0001)。
結果
酵母細胞をpDRFLIP38-AtLEA4-5プラスミドで形質転換した後、青色光トランスイルミネーターとフィルターで正の形質転換体の蛍光を観察しました(図1)。高浸透圧ストレスを誘発するためのさまざまな溶液の調製には時間がかかるため、 図 2 の 96 ウェルテンプレートに従うことをお勧めします。さまざまな濃度の塩化ナトリウムによる高浸透圧ストレ?...
ディスカッション
ここで紹介する手法は、IDRのアンサンブルのグローバルな次元が環境摂動をどのように感知し、応答するかについての洞察を得る方法を提供します。この方法は、遺伝的にコードされたコンストラクトに依存しており、酵母細胞でのプラスミドの安定発現以外に追加の成分を必要としないため、他の細胞タイプでの潜在的なアプリケーションに適応できます。さらに、真核細胞がライフサイ?...
開示事項
著者は利益相反がないことを宣言します。
謝辞
Cuevas-Velazquez研究室のメンバーには、原稿の批判的なレビューに感謝します。この研究は、Programa de Apoyo a Proyectos de Investigación e Innovación Tecnológica、Dirección General de Asuntos del Personal Académico、Universidad Nacional Autónoma de México(UNAM-PAPIIT)プロジェクト番号IA203422の支援を受けました。Consejo Nacional de Humanidades, Ciencias y Tecnología (CONAHCYT), プロジェクト番号 252952;Programa de Apoyo a la Investigación y el Posgrado, Facultad de Química, Universidad Nacional Autónoma de México, Grant 5000-9182.CET(CVU 1083636)およびCAPD(CVU 1269643)は、CONAHCYTの M.Sc 奨学金を認めています。
資料
| Name | Company | Catalog Number | Comments |
| 96-well plate | Greiner Bio-One | 655096 | |
| Agar | Sigma-Aldrich | 5040 | |
| BglII | New England BioLabs | R0144S | |
| BJ5465 cells | American Type Culture Collection | 208289 | |
| Buffer MES 50 mM | Sigma-Aldrich | M8250 | |
| Buffer Tris-HCl 10 mM | Invitrogen | 15506017 | |
| EDTA 1 mM | Merck | 108452 | |
| Falcon tubes | Corning | 352057 | |
| LB media | Sigma-Aldrich | L2897 | |
| Lithium acetate 0.1 M | Sigma-Aldrich | L6883 | |
| Low Melt Agarose | GOLDBIO | A-204-25 | |
| Microcentrifuge | eppendorf | 5452000010 | |
| Miniprep kit | ZymoPure | D4210 | |
| NaOH 0.02 M | Merck | 106462 | |
| PEG 3,350 40% | Sigma-Aldrich | 1546547 | |
| plasmid pDRFLIP38-AtLEA4-5 | addgene | 178189 | |
| Plate reader | BMG LABTECH | CLARIOstar Plus | |
| SacI | New England BioLabs | R3156S | |
| Salmon sperm DNA 2 mg/mL | Thermo Fisher Scientific | 15632011 | |
| SD-Ura | Sigma-Aldrich | Y1501 | |
| Sodium cloride | Sigma-Aldrich | S9888 | |
| Taq polymesare | Promega | M5123 | |
| Transiluminator | Accuris instruments | E4000 | |
| UV-Visible spectrophotometer | Thermo Fisher Scientific | Biomate3 | |
| YPD media | Sigma-Aldrich | Y1500 |
参考文献
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nat Rev Mol Cell Biol. 16 (1), 18-29 (2015).
- Covarrubias, A. A., Romero-Pérez, P. S., Cuevas-Velazquez, C. L., Rendón-Luna, D. F. The functional diversity of structural disorder in plant proteins. Arch Biochem Biophys. 680, 108229 (2019).
- Ahmed, S. S., et al. Characterization of intrinsically disordered regions in proteins informed by human genetic diversity. PLoS Comput Biol. 18 (3), e1009911 (2022).
- Birol, M., Melo, A. M. Untangling the conformational polymorphism of disordered proteins associated with neurodegeneration at the single-molecule level. Front Mol Neurosci. 12, 309 (2019).
- Moses, D., et al. Revealing the hidden sensitivity of intrinsically disordered proteins to their chemical environment. J Phys Chem Lett. 11 (23), 10131-10136 (2020).
- Cuevas-Velazquez, C. L., et al. Intrinsically disordered protein biosensor tracks the physical-chemical effects of osmotic stress on cells. Nat Commun. 12 (1), 5438 (2021).
- Holehouse, A. S., Sukenik, S. Controlling structural bias in intrinsically disordered proteins using solution space scanning. J Chem Theory Comput. 16 (3), 1794-1805 (2020).
- Martin, E. W., Hopkins, J. B., Mittag, T. Small-angle X-ray scattering experiments of monodisperse intrinsically disordered protein samples close to the solubility limit. Methods Enzymol. 646, 185-222 (2021).
- Miles, A. J., Drew, E. D., Wallace, B. A. DichroIDP: a method for analyses of intrinsically disordered proteins using circular dichroism spectroscopy. Commun Biol. 6 (1), 823 (2023).
- Kaminski, C. F., Rees, E. J., Schierle, G. S. K. A quantitative protocol for intensity-based live cell FRET imaging. Method Mol Biol. 1076, 445-454 (2014).
- Moses, D., et al. Structural biases in disordered proteins are prevalent in the cell. bioRxiv. , (2022).
- Cuevas-Velazquez, C. L., Saab-Rincón, G., Reyes, J. L., Covarrubias, A. A. The Unstructured N-terminal Region of Arabidopsis Group 4 Late Embryogenesis Abundant (LEA) Proteins Is Required for Folding and for Chaperone-like Activity under Water Deficit. J Biol Chem. 291 (20), 10893-10903 (2016).
- JoVE Science Education Database. . Restriction enzyme digests. , (2023).
- JoVE Science Education Database. . Gel purification. , (2023).
- JoVE Science Education Database. . DNA ligation reactions. , (2023).
- JoVE Science Education Database. . Bacterial transformation using heat Ssock and competent cells. , (2023).
- JoVE Science Education Database. . PCR: Principle, instrumentation, and applications. , (2023).
- JoVE Science Education Database. . Plasmid purification. , (2023).
- JoVE Science Education Database. . DNA gel electrophoresis. , (2023).
- JoVE Core Molecular Biology. Sanger/chain termination sequencing using dideoxynucleotides - Concept Available from: https://app.jove.com/science-education/v/12020/sanger-sequencing (2023)
- Theillet, F. X., et al. Physicochemical properties of cells and their effects on intrinsically disordered proteins (IDPs). Chem Rev. 114 (13), 6661-6714 (2014).
- Brutscher, B., et al. NMR methods for the study of instrinsically disordered proteins structure, dynamics, and interactions: General overview and practical guidelines. Adv Exp Med Biol. 870, 49-122 (2015).
- Metskas, L. A., Rhoades, E. Single-molecule FRET of intrinsically disordered proteins. Annu Rev Phys Chem. 71, 391-414 (2020).
- Roebroek, T., et al. Simultaneous readout of multiple FRET pairs using photochromism. Nat Commun. 12 (1), 2005 (2021).
- Algar, W. R., Hildebrandt, N., Vogel, S. S., Medintz, I. L. FRET as a biomolecular research tool - understanding its potential while avoiding pitfalls. Nat Methods. 16 (9), 815-829 (2019).
- Lyon, A. S., Peeples, W. B., Rosen, M. K. A framework for understanding the functions of biomolecular condensates across scales. Nat Rev Mol Cell Biol. 22 (3), 215-235 (2021).
- Belott, C., Janis, B., Menze, M. A. Liquid-liquid phase separation promotes animal desiccation tolerance. Proc Natl Acad Sci U S A. 117 (44), 27676-27684 (2020).
- Miermont, A., et al. Severe osmotic compression triggers a slowdown of intracellular signaling, which can be explained by molecular crowding. Proc Natl Acad Sci U S A. 110 (14), 5725-5730 (2013).
- Saito, H., Posas, F. Response to hyperosmotic stress. Genetics. 192 (2), 289-318 (2012).
- Selenko, P., Wagner, G. Looking into live cells with in-cell NMR spectroscopy. J Struct Biol. 158 (2), 244-253 (2007).
- Cattani, J., Subramaniam, V., Drescher, M. Room-temperature in-cell EPR spectroscopy: alpha-Synuclein disease variants remain intrinsically disordered in the cell. Phys Chem Chem Phys. 19 (28), 18147-18151 (2017).
- Beveridge, R., Chappuis, Q., Macphee, C., Barran, P. Mass spectrometry methods for intrinsically disordered proteins. Analyst. 138 (1), 32-42 (2013).
- Beveridge, R., et al. Ion mobility mass spectrometry uncovers the impact of the patterning of oppositely charged residues on the conformational distributions of intrinsically disordered proteins. J Am Chem Soc. 141 (12), 4908-4918 (2019).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved