É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Estimativa da Sensibilidade Estrutural de Regiões Intrinsecamente Desordenadas em Resposta ao Estresse Hiperosmótico em Células Vivas Utilizando FRET
Neste Artigo
Resumo
Regiões intrinsecamente desordenadas (IDRs) são domínios proteicos flexíveis que modificam sua conformação em resposta a mudanças ambientais. A transferência de energia por ressonância de fluorescência em conjunto (FRET) pode estimar as dimensões da proteína sob diferentes condições. Apresentamos uma abordagem FRET para avaliar a sensibilidade estrutural da IDR em células vivas de Saccharomyces cerevisiae sob estresse hiperosmótico.
Resumo
Regiões intrinsecamente desordenadas (IDRs) são domínios proteicos que participam de processos celulares cruciais. Durante as condições de estresse, as propriedades físico-químicas do ambiente celular mudam, impactando diretamente o conjunto conformacional das IDRs. As IDRs são inerentemente sensíveis a perturbações ambientais. Estudar como as propriedades físico-químicas da célula regulam o conjunto conformacional de IDRs é essencial para a compreensão do controle ambiental de sua função. Aqui, descrevemos um método passo-a-passo para medir a sensibilidade estrutural de IDRs em células vivas de Saccharomyces cerevisiae em resposta a condições de estresse hiperosmótico. Apresentamos o uso da transferência de energia por ressonância de fluorescência de conjunto (FRET) para estimar como as dimensões globais das IDRs mudam durante um aumento progressivo do estresse hiperosmótico imposto às células com qualquer osmólito. Além disso, fornecemos um script para processar medições de fluorescência e comparar a sensibilidade estrutural para diferentes IDRs. Seguindo esse método, os pesquisadores podem obter informações valiosas sobre as mudanças conformacionais que as IDRs sofrem no complexo meio intracelular ao mudar ambientes.
Introdução
Regiões intrinsecamente desordenadas (IDRs) são componentes críticos nos processos celulares1. Em combinação com domínios estruturados, as IDRs são essenciais para as funções proteicas. A composição de aminoácidos das IDRs é tendenciosa, representada principalmente por resíduos carregados, hidrofílicos e pequenos. Devido a essa propriedade, os IDRs são considerados domínios de baixa complexidade 2,3. Numerosas IDRs têm chamado a atenção, principalmente porque essas regiões desempenham um papel crucial em condições patológicas, particularmente doenças neurodegenerativas. Tais doenças caracterizam-se pela automontagem e subsequente deposição extracelular ou intracelular de IDRs nosneurônios4. Exemplos dessas IDRs incluem β amiloide (Aβ) na doença de Alzheimer, huntingtina (HTT) na doença de Huntington e proteína ligadora de DNA TAR-43 (TDP-43) e fusionadas em sarcoma (FUS) na esclerose lateral amiotrófica e demência frontotemporal4. O estudo dos rearranjos estruturais das IDRs no contexto da doença tem sido significativamente aprimorado por métodos espectroscópicos, incluindo a transferência de energia por ressonância de fluorescência (FRET).
A natureza hidrofílica e estendida das IDRs as torna extremamente sensíveis a mudanças nas propriedades físico-químicas do ambiente de solução5. O grau pelo qual o conjunto conformacional das IDRs é modificado pelo ambiente é chamado de sensibilidade estrutural 5,6,7. Diferentes técnicas podem ser utilizadas para estudar a conformação e a dinâmica das IDRs, incluindo dicroísmo circular (CD) e espalhamento de raios X a baixos ângulos (SAXS)8,9. Infelizmente, CD e SAXS requerem grandes quantidades de proteínas purificadas, por isso não são apropriados para estudos em células. Em contraste, o FRET é uma técnica que mede a intensidade de fluorescência de duas moléculas fluorescentes que marcam especificamente uma IDR, o que significa que elas podem ser monitoradas em misturas complexas, como células vivas10. Medir dinamicamente a sensibilidade estrutural das IDRs em células vivas é necessário para entender como o ambiente regula a conformação e a função do proteoma desordenado.
FRET é um método poderoso para quantificar a sensibilidade estrutural de IDRs, bem como proteínas globulares e multidomínios em células vivas. O método requer uma construção que consiste em um IDR de interesse prensado entre duas proteínas fluorescentes (FP), conhecido como par FET. Para esse protocolo, sugerimos o uso de mCerulean3 como FP doador e Citrino como PF receptor, devido à sua grande faixa dinâmica, em comparação com outros FPs relatados em estudo anterior sobre sensibilidade dasRRD6. O FRET já foi explorado para medir a sensibilidade estrutural de uma planta IDR em diferentes contextos celulares6. Além disso, essa técnica tem sido utilizada para caracterizar as dimensões proteicas globais das IDRs por diferentes grupos de pesquisa, tanto in vitro quanto in vivo5,11.
Aqui, descrevemos o método de FRET de conjunto para estudar a sensibilidade estrutural de IDRs em células de levedura viva (Saccharomyces cerevisiae). Mostramos resultados representativos que são baseados em uma planta IDR chamada AtLEA4-5. AtLEA4-5 é desordenado em solução, mas dobra-se em α-hélice quando o apinhamento macromolecular é induzido in vitro12. O AtLEA4-5 é um bom modelo de referência para esse método por ser relativamente pequeno (158 resíduos), desordenado e sensível a perturbações ambientais, como relatado in silico e in vitro6,12. O método apresentado aqui pode ser dimensionado para abordagens de alto rendimento, pois as células de levedura são fáceis de cultivar, e o tratamento é aplicado em pequenos volumes. Além disso, pequenas modificações no protocolo podem ser aplicadas a outros sistemas celulares, como bactérias e célulasvegetais6. O protocolo pode ser realizado em qualquer laboratório de biologia molecular com acesso a um leitor de microplacas com modo fluorescência, equipamento disponível na maioria das instituições de pesquisa.
Protocolo
1. Construção do plasmídeo
- Amplificar o quadro de leitura aberto (ORF) que codifica o IDR desejado por reação em cadeia da polimerase (PCR). Não inclua o códon stop, pois a ORF será ladeada pelos genes que codificam as proteínas fluorescentes. Para a amplificação, foram projetados primers com os sítios de restrição SacI (5') e BglII (3').
NOTA: Para a seção de resultados representativos, usamos AtLEA4-5 como o IDR selecionado. Amplificamos a ORF de AtLEA4-5 a partir do plasmídeo pTrc99A-AtLEA4-512. - Digerir o produto da PCR ORF por restrição consecutiva com as enzimas SacI e BglII13.
- Obter o plasmídeo comercial pDRFLIP38-AtLEA4-5 (#178189) da Addgene (https://www.addgene.org/178189/).
- Realizar uma restrição consecutiva usando as enzimas SacI e BglII para remover o AtLEA4-5 ORF do plasmídeo pDRFLIP38-AtLEA4-5, deixando um plasmídeo aberto contendo o par FRET13.
- Purificar os fragmentos de DNA digeridos usando a recuperação em gel de eletroforese em gel de agarose14. Ligate os fragmentos restritos usando DNA ligase15.
- Transformar a reação de clonagem em células competentes de Escherichia coli (DH5α ou cepas relacionadas) e selecionar em placas de Luria-Bertani (LB) contendo 50 μg/mL de ampicilina16. Crescer durante a noite (ON) a 37 °C.
- Isolar e cultivar pelo menos cinco colônias transformadas em nova placa e genótipo por PCR17. Purificar o DNA plasmidial de uma colônia transformada positiva usando métodos padrão de miniprep18.
- Verificar a extração e integridade do DNA plasmidial utilizando eletroforese em gel de agarose19. Verifique a clonagem correta da construção usando o sequenciamento de Sanger20.
2. Expressão plasmidial em células de levedura
Observação : use técnicas assépticas padrão para executar as seguintes etapas. Use uma capela de fluxo laminar de cultura ou um isqueiro.
- Inocular uma faixa de S. cerevisiae BJ5465 (ATCC: 208289) em 3 mL de meio de levedura peptona dextrose (YPD) e crescer ON a 30 °C e 200 rpm.
NOTA: BJ5465 é uma cepa deficiente em protease (CH1) recomendada para trabalhar com IDRs. Outras cepas de S. cerevisiae podem ser testadas e utilizadas. - Centrifugar 1 mL da cultura de levedura saturada durante a noite (OD600 ~ 3-4) a 14.000 x g por 1 min. Remova cuidadosamente o sobrenadante por pipetagem.
- Ressuspender o pellet em 1 mL de tampão Tris-EDTA (TE) (pH 7,5) agitando suavemente o tubo. Centrifugar a 14.000 x g por 1 min e remover cuidadosamente o sobrenadante.
- Ressuspender o pellet em 500 μL de tampão Lazy Bones (40% p/v PEG 3.350; 100 mM acetato de lítio; 10 mM Tris-HCl; 1 mM EDTA; pH 7,5) por pipetagem. Adicionar 25 μL de DNA de espermatozoides de salmão fervido (100 °C por 5 min, seguido de resfriamento no gelo por 5 min) às células de levedura ressuspensas.
- Adicione 100 ng do DNA plasmidial à mistura e vórtice a mistura por 1 min para garantir uma mistura completa. Deixe a mistura repousar à temperatura ambiente por 1-2 h.
- Choque térmico da mistura de levedura a 42 °C por 12 min, depois resfriar no gelo por mais 12 min. Centrifugar o tubo a 14 000 x g durante 1 min e remover cuidadosamente o sobrenadante.
- Ressuspender o pellet em 1 mL de tampão TE (pH 7,5). Placa 100 μL das células transformadas em meio de levedura sintética sem uracila (SD-ura) suplementada com ágar 15 g/L.
- Incubar as placas a 30 °C por 2-3 dias (Figura 1). Passe pelo menos cinco transformadores de levedura em uma nova placa e cresça ON.
3. Validação de transformadores de levedura
- Escolha uma pequena porção de cada candidato a transformador raspando suavemente e ressuspendendo em 5 μL de NaOH 20 mM em tubos de PCR individuais.
- Aquecer a amostra a 99 °C durante 10 minutos num termociclador para lisar as células e libertar o ADN na solução. Esta etapa é crucial para preparar o molde de DNA para PCR.
- Transferir 1 μL da amostra fervida para uma mistura separada de reacções de PCR contendo os primers e reagentes de PCR adequados para genotipagem. Foram utilizados os seguintes primers: Primer Forward: 5'-AAATATACCCCAGCCTCGATCTAGA-3'. Primer reverso: 5'-GTAATACGACTCACTATAGGGCG-3'.
- Configure uma reação de PCR e valide a presença do plasmídeo correto usando eletroforese em gel de agarose. Selecione um transformador para experimentos adicionais.
4. Preparação de cultura de células de levedura para ensaio de FRET
- Inocular uma faixa do transformador de levedura selecionado em 3 mL de meio líquido 1x SD-Ura.
OBS: Faça este procedimento em uma capela de fluxo laminar e material estéril. - Crescer a 30 °C, 200 rpm por pelo menos 12 h até atingir a saturação. Meça o OD600. O crescimento deve ficar entre OD600 = 1,0-2,0.
Observação : se OD600 é menor que 1,0, dê às células mais tempo de incubação para crescer. Se o OD600 exceder 2.0, não prossiga e reinicie a partir da etapa 4.1.
5. Preparação de células de levedura para ensaio de FRET
- Recolher 2 ml da cultura de levedura durante a noite e centrifugar a 14.000 x g à temperatura ambiente. Remova cuidadosamente o sobrenadante por pipetagem.
- Ressuspender o pellet em 1 mL de tampão 50 mM de ácido etanossulfônico (MES) 2-(N-Morpholino) (pH 6).
- Centrífuga a 14.000 x g à temperatura ambiente. Remova o sobrenadante.
- Repita as etapas 5.2 e 5.3. Ressuspender as células de levedura em 2 mL de tampão MES, pH 6 e despejar o volume em um reservatório de reagente.
6. Configuração das medições de fluorescência
NOTA: A presente varredura é considerada realizada em um leitor de microplacas com modo de fluorescência.
- Para configurações básicas, defina o instrumento da seguinte forma: Tipo de medição: Espectro de intensidade de fluorescência (FI); Nome da microplaca: Greiner 96 F-bottom.
- Para configurações ópticas, defina o instrumento da seguinte forma: Não. dos pontos de varredura de comprimento de onda: 91; Comprimento de onda de excitação: 433 nm; Largura de banda de excitação: 10 nm; Comprimento de onda de emissão: 460 - 550; Largura do passo: 1 nm; Largura de banda de emissão: 10 nm; Altura focal obtida por autofoco; Altura focal: 5 mm; Comprimento de onda utilizado para ganho: 490 nm.
- Para configurações gerais, defina o instrumento da seguinte forma: Tempo de liquidação: 0,1 s; Direção de leitura: unidirecional, horizontal da esquerda para a direita, de cima para baixo.
NOTA: O ganho inserido manualmente deve ser ajustado para cada medição usando o poço esperado para exibir os níveis mais altos de intensidade de fluorescência em toda a varredura de comprimento de onda.
7. Preparação de soluções hipertônicas e medidas de fluorescência
- Prepare soluções de NaCl 0 M, 0,2 M, 0,4 M, 0,6 M, 0,8 M, 1 M, 1,5 M e 2 M em uma placa preta de fundo claro de 96 poços. O volume final em cada poço será de 200 μL (150 μL de solução de osmólito + 50 μL de suspensão de células de levedura).
NOTA: Outros osmolitos podem ser usados para induzir estresse hiperosmótico. Todas as soluções devem ser preparadas em tampão MES 50 mM pH 6. - Carregar 150 μL de tampão MES 50 mM, pH 6 nos três primeiros poços da linha A (A1, A2, A3) e nos poços subsequentes, adicionar 150 μL da solução correspondente em ordem crescente da esquerda para a direita (Figura 2). Para testar todas as concentrações sugeridas, continue o carregamento na linha B.
NOTA: Esta configuração considera as medições de 3 réplicas técnicas para cada concentração. - Use uma micropipeta de 12 canais para transferir 50 μL de células de levedura lavadas para cada poço. As células de levedura tendem a sedimentar para o fundo do reservatório. Certifique-se de ressuspender a suspensão de levedura completamente, pipetando para cima e para baixo antes de transferir as células.
- Carregue células na placa de 96 poços contendo as diferentes soluções e misture bem pipetando para cima e para baixo pelo menos 4x.
- Meça os espectros de emissão de intensidade de fluorescência imediatamente para os poços desejados em um leitor de microplacas com as configurações de especificação da etapa 6. Repita o procedimento para cada IDR a ser testado no ensaio FET.
- Etapa opcional. Se disponível, meça uma construção somente para doadores (pDRFLIP38-mCerulean3) seguindo as etapas descritas aqui.
NOTA: Recomendamos executar esta etapa para calcular a eficiência do FRET de cada condição. Se o leitor não puder executar essa etapa, o FRET pode ser calculado usando o método da razão FET. Ambos os métodos para cálculos de FRET são explicados nas etapas 8 e 9. - Execute pelo menos três réplicas em três dias independentes para cada construção.
8. Processamento de dados usando o método de eficiência FRET
NOTA: Para leitores com conhecimento da linguagem de programação R, fornecemos um conjunto de scripts R para executar o processamento de dados descrito nesta seção. Os scripts podem ser encontrados em https://github.com/Kaz-bits/cuevaslab-procotols/tree/main/FRET. Siga as instruções no arquivo LEIA-ME.
- Exporte dados como arquivos .xlsx do leitor de microplacas.
- Normalize os valores de intensidade de fluorescência em cada comprimento de onda de varredura até o ponto isosbestico do par de FRET usado.
NOTA: Esta etapa é necessária para comparar os espectros de todas as condições testadas. O ponto isosbestico do par mCerulean3 e Citrino FRET é de 515 nm. Por exemplo, obtenha a razão dos valores de intensidade de fluorescência de 460 nm/515 nm, 461 nm/515 nm, 462 nm/515 nm, e assim por diante. - Calcule a média para cada réplica técnica assim que os valores de fluorescência forem normalizados. No total, deve haver uma coluna para cada condição de estresse hiperosmótico.
- Visualize todos os espectros de intensidade de fluorescência no mesmo gráfico (Figura 3). Cada espectro deve exibir uma combinação dos espectros de emissão de fluorescência de cada mCerulean3 e Citrino. Em casos raros, onde a eficiência de FRET é 0, apenas o espectro de emissão de fluorescência do doador será observado, ou quando a eficiência de FRET é 1, apenas a fluorescência do receptor será observada. O pico de emissão de fluorescência para mCerulean3 (doador) é de 475 nm, e o pico de emissão de fluorescência para Citrino (aceitador) é de 525 nm.
- Determine se a medição de fluorescência exibe uma alteração típica de FRET em resposta ao estresse hiperosmótico. Se a conformação da IDR for sensível ao tratamento, isso refletirá como uma mudança na eficiência do FET. Uma mudança típica na eficiência do FRET acoplará uma diminuição na intensidade de fluorescência do doador com um aumento na intensidade de fluorescência do receptor ou vice-versa.
- Quantificar a eficiência do FRIT (EFRET). Obter os valores de pico normalizados do doador (mCerulean3) (475 nm/515 nm) para o construto IDR e o construto somente doador para cada condição de estresse hiperosmótico. Use os valores médios de pico normalizados das réplicas técnicas. Execute essa operação para cada uma das três réplicas independentes. Calcule EFRET com a seguinte fórmula:
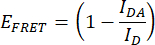
Onde IDA é a razão 475 nm/515 nm da construção IDR (construção do par FET), e ID é a razão 475 nm/515 nm da construção somente doador. - Compare as eficiências do FET. Normalizar os valores deE FRET de cada condição de estresse hiperosmótico para oFRET E da condição sem estresse (por exemplo, NaCl 0 M). Faça comparações usando um gráfico de caixa ou um gráfico de curva suave. Realizar uma ANOVA One-way seguida de um teste estatístico post hoc de Tukey (valores de p: *p < 0,05, **p < 0,01, ***p < 0,001, ****p < 0,0001).
9. Processamento de dados usando o método da razão FRET
NOTA: Se não for possível obter medições somente para doadores, execute o método da razão FET. Este método compara a razão FRET entre as diferentes condições de estresse hiperosmótico. As etapas 7.1 a 7.5 devem ser executadas antes das etapas desta seção para validar um comportamento típico de FET. Para leitores com conhecimento da linguagem de programação R, fornecemos um conjunto de scripts R para realizar o processamento de dados descrito nesta seção. Os scripts podem ser encontrados em https://github.com/Kaz-bits/cuevaslab-procotols/tree/main/FRET. Siga as instruções no arquivo LEIA-ME.
- Extraia dados a 475 nm e 525 nm do arquivo xlsx exportado anteriormente do leitor de microplacas. Esses valores correspondem ao pico de emissão de fluorescência para mCerulean3 (doador, DxDm) e ao pico de emissão de fluorescência para Citrino (receptor, DxAm) quando o fluoróforo doador está excitado (433 nm).
- Normalizar os valores de intensidade de fluorescência a 475 nm e 525 nm até o ponto isosbestico do par de FRET utilizado. O ponto isosbestico do par mCerulean3 e Citrino FRET é de 515 nm. Obter a razão FRET (DxAm/DxDm).
- Compare as razões de FET. Normalizar os valores de DxAm/DxDm de cada condição de estresse hiperosmótico para a média de DxAm/DxDm da condição sem estresse (por exemplo, NaCl 0 M). Faça comparações usando um gráfico de caixa ou um gráfico de curvas suaves (Figura 4 e Figura 5). Realizar uma ANOVA One-way seguida de um teste estatístico post hoc de Tukey (valores de p: *p < 0,05, **p < 0,01, ***p < 0,001, ****p < 0,0001).
Resultados
Após a transformação das células de levedura com o plasmídeo pDRFLIP38-AtLEA4-5, a fluorescência dos transformadores positivos foi observada com um transiluminador de luz azul e um filtro (Figura 1). O preparo das diferentes soluções para induzir estresse hiperosmótico é demorado, por isso sugerimos seguir o molde de 96 poços da Figura 2. Imediatamente após o tratamento do estresse hiperosmótico com concentrações variáveis de cloreto de sódio, o...
Discussão
O método apresentado aqui oferece uma maneira de obter insights sobre como as dimensões globais do conjunto de IDRs sentem e respondem a perturbações ambientais. Este método baseia-se em uma construção geneticamente codificada e não requer componentes adicionais além de uma expressão estável de plasmídio em células de levedura, tornando-o adaptável para aplicações potenciais em outros tipos celulares. Além disso, é versátil para explorar outras perturbações físico-químicas que as células eucariót...
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
Agradecemos aos membros do laboratório Cuevas-Velazquez pela revisão crítica do manuscrito. Este trabalho foi apoiado pelo Projeto IA203422 Programa de Apoyo a Proyectos de Investigación e Innovación Tecnológica, Dirección General de Asuntos del Personal Académico, Universidad Nacional Autónoma de México (UNAM-PAPIIT); Consejo Nacional de Humanidades, Ciencias y Tecnología (CONAHCYT), projeto número 252952; e Programa de Apoyo a la Investigación y el Posgrado, Facultad de Química, Universidad Nacional Autónoma de México, Grant 5000-9182. CET (CVU 1083636) e CAPD (CVU 1269643) reconhecem a CONAHCYT por sua bolsa de M.Sc.
Materiais
| Name | Company | Catalog Number | Comments |
| 96-well plate | Greiner Bio-One | 655096 | |
| Agar | Sigma-Aldrich | 5040 | |
| BglII | New England BioLabs | R0144S | |
| BJ5465 cells | American Type Culture Collection | 208289 | |
| Buffer MES 50 mM | Sigma-Aldrich | M8250 | |
| Buffer Tris-HCl 10 mM | Invitrogen | 15506017 | |
| EDTA 1 mM | Merck | 108452 | |
| Falcon tubes | Corning | 352057 | |
| LB media | Sigma-Aldrich | L2897 | |
| Lithium acetate 0.1 M | Sigma-Aldrich | L6883 | |
| Low Melt Agarose | GOLDBIO | A-204-25 | |
| Microcentrifuge | eppendorf | 5452000010 | |
| Miniprep kit | ZymoPure | D4210 | |
| NaOH 0.02 M | Merck | 106462 | |
| PEG 3,350 40% | Sigma-Aldrich | 1546547 | |
| plasmid pDRFLIP38-AtLEA4-5 | addgene | 178189 | |
| Plate reader | BMG LABTECH | CLARIOstar Plus | |
| SacI | New England BioLabs | R3156S | |
| Salmon sperm DNA 2 mg/mL | Thermo Fisher Scientific | 15632011 | |
| SD-Ura | Sigma-Aldrich | Y1501 | |
| Sodium cloride | Sigma-Aldrich | S9888 | |
| Taq polymesare | Promega | M5123 | |
| Transiluminator | Accuris instruments | E4000 | |
| UV-Visible spectrophotometer | Thermo Fisher Scientific | Biomate3 | |
| YPD media | Sigma-Aldrich | Y1500 |
Referências
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nat Rev Mol Cell Biol. 16 (1), 18-29 (2015).
- Covarrubias, A. A., Romero-Pérez, P. S., Cuevas-Velazquez, C. L., Rendón-Luna, D. F. The functional diversity of structural disorder in plant proteins. Arch Biochem Biophys. 680, 108229 (2019).
- Ahmed, S. S., et al. Characterization of intrinsically disordered regions in proteins informed by human genetic diversity. PLoS Comput Biol. 18 (3), e1009911 (2022).
- Birol, M., Melo, A. M. Untangling the conformational polymorphism of disordered proteins associated with neurodegeneration at the single-molecule level. Front Mol Neurosci. 12, 309 (2019).
- Moses, D., et al. Revealing the hidden sensitivity of intrinsically disordered proteins to their chemical environment. J Phys Chem Lett. 11 (23), 10131-10136 (2020).
- Cuevas-Velazquez, C. L., et al. Intrinsically disordered protein biosensor tracks the physical-chemical effects of osmotic stress on cells. Nat Commun. 12 (1), 5438 (2021).
- Holehouse, A. S., Sukenik, S. Controlling structural bias in intrinsically disordered proteins using solution space scanning. J Chem Theory Comput. 16 (3), 1794-1805 (2020).
- Martin, E. W., Hopkins, J. B., Mittag, T. Small-angle X-ray scattering experiments of monodisperse intrinsically disordered protein samples close to the solubility limit. Methods Enzymol. 646, 185-222 (2021).
- Miles, A. J., Drew, E. D., Wallace, B. A. DichroIDP: a method for analyses of intrinsically disordered proteins using circular dichroism spectroscopy. Commun Biol. 6 (1), 823 (2023).
- Kaminski, C. F., Rees, E. J., Schierle, G. S. K. A quantitative protocol for intensity-based live cell FRET imaging. Method Mol Biol. 1076, 445-454 (2014).
- Moses, D., et al. Structural biases in disordered proteins are prevalent in the cell. bioRxiv. , (2022).
- Cuevas-Velazquez, C. L., Saab-Rincón, G., Reyes, J. L., Covarrubias, A. A. The Unstructured N-terminal Region of Arabidopsis Group 4 Late Embryogenesis Abundant (LEA) Proteins Is Required for Folding and for Chaperone-like Activity under Water Deficit. J Biol Chem. 291 (20), 10893-10903 (2016).
- JoVE Science Education Database. . Restriction enzyme digests. , (2023).
- JoVE Science Education Database. . Gel purification. , (2023).
- JoVE Science Education Database. . DNA ligation reactions. , (2023).
- JoVE Science Education Database. . Bacterial transformation using heat Ssock and competent cells. , (2023).
- JoVE Science Education Database. . PCR: Principle, instrumentation, and applications. , (2023).
- JoVE Science Education Database. . Plasmid purification. , (2023).
- JoVE Science Education Database. . DNA gel electrophoresis. , (2023).
- JoVE Core Molecular Biology. Sanger/chain termination sequencing using dideoxynucleotides - Concept Available from: https://app.jove.com/science-education/v/12020/sanger-sequencing (2023)
- Theillet, F. X., et al. Physicochemical properties of cells and their effects on intrinsically disordered proteins (IDPs). Chem Rev. 114 (13), 6661-6714 (2014).
- Brutscher, B., et al. NMR methods for the study of instrinsically disordered proteins structure, dynamics, and interactions: General overview and practical guidelines. Adv Exp Med Biol. 870, 49-122 (2015).
- Metskas, L. A., Rhoades, E. Single-molecule FRET of intrinsically disordered proteins. Annu Rev Phys Chem. 71, 391-414 (2020).
- Roebroek, T., et al. Simultaneous readout of multiple FRET pairs using photochromism. Nat Commun. 12 (1), 2005 (2021).
- Algar, W. R., Hildebrandt, N., Vogel, S. S., Medintz, I. L. FRET as a biomolecular research tool - understanding its potential while avoiding pitfalls. Nat Methods. 16 (9), 815-829 (2019).
- Lyon, A. S., Peeples, W. B., Rosen, M. K. A framework for understanding the functions of biomolecular condensates across scales. Nat Rev Mol Cell Biol. 22 (3), 215-235 (2021).
- Belott, C., Janis, B., Menze, M. A. Liquid-liquid phase separation promotes animal desiccation tolerance. Proc Natl Acad Sci U S A. 117 (44), 27676-27684 (2020).
- Miermont, A., et al. Severe osmotic compression triggers a slowdown of intracellular signaling, which can be explained by molecular crowding. Proc Natl Acad Sci U S A. 110 (14), 5725-5730 (2013).
- Saito, H., Posas, F. Response to hyperosmotic stress. Genetics. 192 (2), 289-318 (2012).
- Selenko, P., Wagner, G. Looking into live cells with in-cell NMR spectroscopy. J Struct Biol. 158 (2), 244-253 (2007).
- Cattani, J., Subramaniam, V., Drescher, M. Room-temperature in-cell EPR spectroscopy: alpha-Synuclein disease variants remain intrinsically disordered in the cell. Phys Chem Chem Phys. 19 (28), 18147-18151 (2017).
- Beveridge, R., Chappuis, Q., Macphee, C., Barran, P. Mass spectrometry methods for intrinsically disordered proteins. Analyst. 138 (1), 32-42 (2013).
- Beveridge, R., et al. Ion mobility mass spectrometry uncovers the impact of the patterning of oppositely charged residues on the conformational distributions of intrinsically disordered proteins. J Am Chem Soc. 141 (12), 4908-4918 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados