Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Ensayo basado en transferencia de energía por resonancia de bioluminiscencia (BRET) para medir las interacciones de CRAF con proteínas 14-3-3 en células vivas
En este artículo
Resumen
Este protocolo describe un ensayo basado en BRET para medir las interacciones de la quinasa CRAF con las proteínas 14-3-3 en células vivas. El protocolo describe los pasos para la preparación de las celdas, la lectura de las emisiones BRET y el análisis de datos. También se presenta un ejemplo de resultado con la identificación de los controles adecuados y la resolución de problemas para la optimización del ensayo.
Resumen
El CRAF es un efector primario de las GTPasas RAS y desempeña un papel fundamental en la tumorigénesis de varios cánceres impulsados por KRAS. Además, el CRAF es un punto caliente para las mutaciones de la línea germinal, que se ha demostrado que causan la RASopatía del desarrollo, síndrome de Noonan. Todas las quinasas RAF contienen múltiples sitios de unión dependientes de la fosforilación para las proteínas reguladoras 14-3-3. La unión diferencial de 14-3-3 a estos sitios desempeña un papel esencial en la formación de dímeros activos de RAF en la membrana plasmática en condiciones de señalización y en el mantenimiento de la autoinhibición de RAF en condiciones de reposo. Comprender cómo se regulan estas interacciones y cómo se pueden modular es fundamental para identificar nuevos enfoques terapéuticos que se dirijan a la función de la RAF. Aquí, describo un ensayo basado en la transferencia de energía por resonancia de bioluminiscencia (BRET) para medir las interacciones de CRAF con proteínas 14-3-3 en células vivas. En concreto, este ensayo mide las interacciones de CRAF fusionado con un donante de nanoluciferasa y 14-3-3 fusionado con un aceptor de etiquetas Halo, donde la interacción de RAF y 14-3-3 da lugar a la transferencia de energía de donante a aceptor y a la generación de la señal BRET. El protocolo muestra además que esta señal puede ser interrumpida por mutaciones que se ha demostrado que evitan la unión de 14-3-3 a cada uno de sus sitios de acoplamiento RAF de alta afinidad. Este protocolo describe los procedimientos para sembrar, transfectar y replantear las células, junto con instrucciones detalladas para leer las emisiones de BRET, realizar análisis de datos y confirmar los niveles de expresión de proteínas. Además, se proporcionan ejemplos de resultados de ensayos, junto con pasos de optimización y solución de problemas.
Introducción
Las quinasas RAF (ARAF, BRAF y CRAF) son los efectores directos de las GTPasas RAS y los miembros iniciadores de la cascada de quinasas RAF-MEK-ERK pro-proliferativa/pro-supervivencia. Estudios recientes han demostrado que la expresión de CRAF desempeña un papel clave en la tumorigénesis de varios cánceres impulsados por KRAS, incluido el cáncer de pulmón de células no pequeñas y el adenocarcinoma ductal de páncreas 1,2,3,4,5. Además, las mutaciones de la línea germinal en CRAF causan una forma particularmente grave de la RASopatía (síndrome de Noonan) 6,7. Comprender la regulación del CRAF es fundamental para desarrollar enfoques terapéuticos exitosos que se dirijan a su función en las células.
Todas las quinasas RAF se pueden dividir en dos dominios funcionales, un dominio catalítico C-terminal (CAT) y un dominio regulador N-terminal (REG), que controla su actividad (Figura 1A)8. El dominio REG abarca el dominio de unión RAS (RBD), el dominio rico en cisteína (CRD) y una región rica en serina/treonina (rica en S/T). En particular, la región rica en S/T contiene el sitio N', que se une a 14-3-3 de una manera dependiente de la fosforilación (S259 en CRAF; Figura 1A) 8. El dominio CAT abarca el dominio quinasa, junto con un segundo sitio de acoplamiento 14-3-3 de alta afinidad, denominado sitio C' (S621 en CRAF; Figura 1A) 8. La unión diferencial de las proteínas diméricas 14-3-3 a los sitios N' y C', junto con la CRD, desempeña un papel crítico tanto en la activación como en la inhibición de RAF 9,10,11,12,13. En condiciones normales de señalización, la activación de RAF se inicia mediante su reclutamiento a la membrana plasmática por RAS, lo que le permite formar dímeros activos, de los cuales el heterodímero BRAF-CRAF es la forma activa predominante14,15. Los ensayos bioquímicos con BRAF y CRAF, junto con estructuras de microscopía electrónica criogénica (Cryo-EM) de BRAF dimérico, indican que un dímero 14-3-3 estabiliza los dímeros activos de RAF al unirse simultáneamente al sitio C' de ambos protómeros de RAF (Figura 1B)9,13,16,17. Por el contrario, los estudios han demostrado que, en condiciones de reposo, la RAF adopta una confirmación citosólica autoinhibida, donde el dominio REG se une al dominio CAT e inhibe su actividad 12,18,19,20. Este estado cerrado se estabiliza mediante un dímero 14-3-3 unido al sitio CRD y N' en el dominio REG y al sitio C' en el dominio CAT (Figura 1B)10,13,21. En BRAF, este modelo está respaldado por estructuras Cryo-EM recientes de monómeros BRAF autoinhibidos y por nuestros estudios bioquímicos previos 10,12,13,21,22. Sin embargo, mientras que se ha demostrado que el 14-3-3 desempeña un papel inhibidor en la regulación23 del CRAF, un estado autoinhibido similar al BRAF puede desempeñar un papel menor en la regulación12 del CRAF; por lo tanto, se requieren más estudios para aclarar los mecanismos por los cuales las proteínas 14-3-3 regulan la actividad de CRAF. La regulación mediada por 14-3-3 de las quinasas RAF requiere una gran cantidad de eventos de fosforilación y desfosforilación de RAF, la unión a varias proteínas reguladoras e interacciones con la membrana plasmática8. Por lo tanto, es fundamental que las interacciones de 14-3-3-RAF se midan en condiciones fisiológicamente relevantes y en presencia de una bicapa lipídica intacta.
Para abordar este problema, se utilizó la tecnología NanoBRET (en adelante denominada N-BRET; consulte la Tabla de materiales para obtener detalles del kit) para desarrollar un ensayo basado en la proximidad para medir las interacciones de CRAF con las proteínas 14-3-3 en células vivas (Figura 1C). Este sistema basado en BRET mide las interacciones de dos proteínas de interés (POI), donde una proteína se marca con un donante de nanoluciferasa (Nano) y la otra con una etiqueta Halo, para su marcaje con el ligando aceptor de energía Halo61822,24. La interacción de las proteínas de interés da como resultado la transferencia de energía del donante al aceptor, que a su vez genera la señal BRET (Figura 1C). La proteína donante Nano extremadamente brillante (emisión (em) 460 nm) y el ligando Halo618 (em 618 nm) proporcionan una mayor separación espectral y sensibilidad que el BRET convencional, lo que lo convierte en una plataforma ideal para estudiar interacciones más débiles y detectar cambios sutiles en la unión24. De hecho, previamente desarrollamos un ensayo basado en N-BRET para medir las interacciones autoinhibitorias de los dominios RAF REG y CAT, que fue esencial para la caracterización de un panel de mutaciones de RASopathy en el BRAF CRD y demostró la importancia crítica de este dominio para mantener la autoinhibición y prevenir la activación constitutiva de BRAF12.
El ensayo descrito aquí mide las interacciones de CRAF, fusionado con una etiqueta Nano N-terminal (Nano-CRAF), y la isoforma zeta de 14-3-3 fusionada con la etiqueta Halo C-terminal (14-3-3ζ-Halo; Figura 1C). Demostramos que las interacciones de Nano-CRAF con 14-3-3ζ-Halo generan una señal BRET robusta, que a su vez puede ser interrumpida por mutaciones que impiden la unión de 14-3-3 al sitio N' (S259A) y/o al sitio C' (S621A). El siguiente protocolo proporciona pasos detallados para realizar, optimizar y solucionar problemas de este ensayo.
Protocolo
NOTA: Este ensayo se realiza en celdas de 293 pies. Una línea epitelial bien caracterizada y fácilmente transfectable derivada de células embrionarias de riñón humano. Una sola placa de cultivo confluente de 10 cm de estas células suele proporcionar suficientes células para sembrar 20 pocillos de placas de cultivo de tejidos de 6 pocillos. Los pasos 1 a 3 deben realizarse utilizando técnica estéril en una cabina de seguridad biológica.
1. Siembra de células (Día 1)
NOTA: En este paso, las células se separan de la(s) placa(s) de cultivo de tejidos, se cuentan y se siembran en placas de cultivo de tejidos de 6 pocillos para la transfección en el paso 2 (Figura 2).
- Aspire el medio de las células en el plato de 10 cm. Lave las células con 5 mL de solución salina tamponada con fosfato (PBS) y aspire.
- Añadir 1 mL de ácido tripsina-etilendiaminotetraacético (EDTA) e incubar durante 3-5 min a 37 °C para separar las células de la placa.
- Añada 9 mL del medio eagle modificado completo de Dulbecco (DMEM) a las células para neutralizar la tripsina y pipetee hacia arriba y hacia abajo repetidamente para generar una suspensión unicelular homogénea.
- Transfiera inmediatamente 20 μL de células a un tubo de 1,7 mL y mezcle con 20 μL de tinción de azul de tripano. Cuente las células utilizando un contador de células automatizado (Tabla de materiales) o un hemocitómetro.
- Diluya las células a 2 x 105 células/ml en un medio DMEM completo y añada 2 ml a cada pocillo de una placa de cultivo de tejidos de 6 pocillos (4 x 105 células/pocillo). Incubar las células a 37 °C y 10% de CO2 durante la noche.
NOTA: Se recomienda utilizar celdas de 293 pies que hayan pasado menos de 20x. Hemos encontrado previamente que el uso de celdas con un mayor número de pasajes puede resultar en una reducción de las proporciones BRET y una mayor variabilidad de la señal de pozo a pocillo.
2. Transfección celular (Día 2)
NOTA: Aquí, las células se transfectan con las construcciones de expresión pCMV5-NanoLuc-CRAF y pCMV5-14-3-3ζ-Halo, junto con el vector vacío pCDNA3.1 (Figura 2).
- Antes de la transfección, diluir los plásmidos N-BRET a 5 ng/μL y pCDNA3.1 a 100 ng/μL, y numerar un juego de tubos estériles de 1.7 mL.
- Agregue 100 μL de medio de transfección (consulte la Tabla de materiales para obtener más detalles) a cada tubo, junto con 5 ng de pCMV5-NanoLuc-CRAF, 10 ng de pCMV5-14-3-3ζ y 200 ng de PCDNA3.1.
- Añada 2 μL de reactivo de transfección (consulte la Tabla de materiales para obtener más detalles) y vórtice suavemente para mezclar. Centrifugar los tubos por pulsos brevemente en una microcentrífuga para asegurarse de que todo el líquido se recoge en el fondo de los tubos y, a continuación, incubar a unos 25 °C durante 15 minutos.
- Añadir complejos de transfección gota a gota a las células e incubar a 37 °C/10% CO2 durante 16-20 h para permitir que se expresen las proteínas marcadas con Halo y Nano.
NOTA: La adición de un vector vacío (pCDNA3.1) de ADN portador es esencial para lograr una alta eficiencia de transfección de las construcciones de expresión de N-BRET. La falta de adición de vector vacío da lugar a niveles reducidos de expresión de las proteínas 14-3-3ζ-Halo y Nano-CRAF y, a su vez, da lugar a proporciones BRET débiles e inconsistentes, como se ha comentado anteriormente22.
3. Rechapado de células (Día 3)
NOTA: En este paso, las células se transfieren a una placa de 384 pocillos y el ligando Halo 618 (+ligando; Tabla de Materiales) o DMSO (+vehículo) para leer las emisiones BRET en el paso 4. Las células restantes se transfieren a placas de cultivo frescas de 6 pocillos para el análisis de Western blot en el Paso 5 (Figura 2).
- Reúna los siguientes materiales y prepare el área de trabajo como se describe a continuación.
- Usando un baño de agua a 37 °C, precaliente la tripsina-EDTA, junto con los medios de ensayo sin suero y los medios de ensayo suplementados con un 10% de FBS (consulte la Tabla de materiales para obtener más detalles).
- En función del número de muestras que se van a medir, etiquete previamente tres juegos de tubos estériles de 1,7 ml (juego 1-3) y un juego de tubos estériles de fondo cónico de 15 ml. Equipe una centrífuga de cubo oscilante con insertos de tubo de 15 mL y preenfriamiento a 4 °C.
- Coloque los siguientes elementos en la campana de cultivo de tejidos: depósitos de reactivos, placas de cultivo de tejidos de 384 pocillos, placas de cultivo de tejidos de 6 pocillos, una pipeta y puntas multicanal, portaobjetos/cámaras de recuento de células y tinción de azul de tripán (Tabla de materiales), junto con los juegos de tubos 1-3 de 1,7 ml.
- Coseche y cuente las celdas como se describe a continuación.
- Aspire el medio de las placas de 6 pocillos y agregue 250 μL de tripsina-EDTA a las células. Incubar las placas de 6 pocillos a 37 °C hasta que las células comiencen a desprenderse (3-5 min).
- Agregue 1 ml de medio de ensayo suplementado con FBS al 10% a cada pocillo para neutralizar la tripsina y pipetear hacia arriba y hacia abajo vigorosamente para generar una suspensión unicelular.
- Transfiera 1 mL de la suspensión celular a los tubos de 15 mL preetiquetados. Agregue otro 1 mL de medio de ensayo suplementado con FBS al 10% a cada uno de los tubos de 15 mL.
- Invierta los tubos 5 veces para mezclar y transfiera inmediatamente 20 μL de suspensión celular al juego de tubos 1.
- Centrífuga en la centrífuga de cubeta oscilante preenfriada durante 5 minutos a ~250 x g. Durante la etapa de centrifugación, mezcle 20 μL de tinción de células de azul de tripano con las células del juego de tubos 1 y luego cuente las células usando un contador de células automatizado o un hemocitómetro. Es típico un rendimiento de 6-8 x 105 células/mL.
- Retire los tubos de 15 ml de la centrífuga y aspire los medios de los gránulos de la celda. Vuelva a suspender los gránulos celulares a 2 x 105 células/ml en un medio de ensayo sin suero y pipetee enérgicamente para generar una suspensión de una sola célula.
NOTA: El uso de medios de ensayo sin suero se utiliza para poner en modo inactivo las vías normales de señalización celular.
- Vuelva a colocar las celdas en placas de 384 pocillos y 6 pocillos como se describe a continuación.
- Invierta los tubos de 15 ml varias veces para asegurar una suspensión celular homogénea y transfiera 500 μL a los tubos de 1,7 ml del juego 2 y del juego 3.
- Añada 0,5 μL de DMSO (+vehículo) al conjunto 2 y 0,5 μL de ligando Halo 618 (+ligando) al juego de tubos 3 y pipetea para mezclar.
- Transfiera las suspensiones de celdas +vehículo y +ligando a pozos separados de depósitos de reactivos. Utilizando la pipeta multicanal (Tabla de materiales), transfiera 40 μL de la suspensión de celda +vehículo desde los depósitos de reactivos a los pocillos cuadruplicados de la placa de cultivo de 384 pocillos. Repita este paso para las suspensiones de células +ligando.
- Transfiera las células restantes a placas de cultivo frescas de 6 pocillos. Incubar las placas de 384 pocillos y 6 pocillos durante la noche a 37 °C y 5% deCO2.
4. Lectura de las emisiones BRET (Día 4)
NOTA: En este paso, el sustrato de nanoluciferasa (consulte la Tabla de materiales para obtener más detalles) se agrega a las células en la placa de cultivo de 384 pocillos y se leen las emisiones del aceptor N-BRET (618 nm) y del donante (460 nm) (Figura 2). A continuación, se calculan los ratios BRET corregidos.
- Precalentar los medios de ensayo sin suero en un baño de agua a 37 °C y descongelar el sustrato de nanoluciferasa a 25 °C. Diluir el sustrato de nanoluciferasa 1:100 en un medio de ensayo sin suero y transferir a un depósito de reactivo. Prepare suficiente cantidad de esta mezcla para agregar 10 μL a cada pocillo de la placa de 384 pocillos, más un 10%-15% de volumen adicional.
- Utilizando una pipeta multicanal (Tabla de Materiales), transfiera 10 μL de la mezcla de sustrato a cada uno de los pocillos que contienen células en la placa de cultivo de 384 pocillos. Gire suavemente la placa durante 1 minuto, ya sea manualmente o con un agitador orbital.
- Inserte la placa de 384 pocillos en el lector de placas multimodo y registre las emisiones de 460 nm y 618 nm para todos los pocillos que contienen celdas.
NOTA: Para el lector de placas multimodo utilizado para este paso (consulte la Tabla de materiales para obtener más detalles), se utilizó una altura de lectura de 6,5 mm, con una medición de tiempo de lectura de 0,1 s, sin embargo, puede ser necesaria una mayor optimización si se utilizan otros lectores de placas. - Calcule las proporciones BRET brutas para +vehículo y +ligando en unidades miliBRET (mBU) individualmente, utilizando la siguiente ecuación:
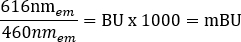
- Calcule la relación BRET corregida restando la relación BRET +vehículo de la del +ligando (Tabla suplementaria 1).
NOTA: Al comparar los resultados de múltiples experimentos, las proporciones BRET corregidas se pueden agrupar y normalizar al promedio del par N-BRET de tipo salvaje (WT), que consta de Nano-CRAFWT y 14-3-3ζWT-Halo (Tabla suplementaria 1).
5. Confirmación de los niveles de expresión proteica (Días 4 y 5)
NOTA: En este paso, las células de las placas de 6 pocillos se lisan y los niveles de expresión proteica de las proteínas Nano-CRAF y 14-3-3ζ-Halo se determinan mediante análisis de Western blot utilizando anticuerpos específicos para las etiquetas Halo y Nano. (Figura 2).
- Añadir inhibidores de la proteasa y la fosfatasa al tampón de lisis NP40 (Tabla de materiales), lo que permite 200 μL de tampón de lisis por muestra.
- Aspire los medios de las placas de 6 pocillos que contienen células. Lavar las celdas una vez con 1 mL de PBS frío y aspirar.
- Añadir 125 μL de tampón de lisis NP40 a cada pocillo e incubar placas de 6 pocillos a 4 °C en una plataforma oscilante durante 15 min para lisar las células.
- Transfiera los lisados a tubos de 1,7 ml sobre hielo y centrifuga a 20.000 x g durante 10 min a 4 °C. Vuelva a colocar los lisados aclarados en hielo y determine la concentración de proteínas utilizando ensayos disponibles comercialmente, como los ensayos de Bradford o de ácido bicinconínico (BCA).
- Normalice la concentración de proteínas en todas las muestras transfiriendo un volumen adecuado de lisado y tampón de lisis NP40 a tubos nuevos de 1,7 ml hasta un volumen total de 100 μl.
- Hervir 5 veces el tampón de muestra de proteínas (Tabla de materiales) durante 1 minuto y añadir 25 μL a cada muestra. Hervir las muestras durante 5-6 minutos y luego centrifugar los tubos por pulsos brevemente para asegurarse de que toda la muestra se acumule en la base del tubo.
- Cargue 25 μL de muestra en cada pocillo de geles de proteínas duplicados (Gel 1 y Gel 2) y luego transfiera las proteínas a membranas de nitrocelulosa o PVDF utilizando procedimientos estándar de Western blot, como se describió anteriormente22.
- Bloquee las membranas en BSA-PBS al 3% a 25 °C durante 30 min y luego incube las membranas durante la noche con el anticuerpo Halo (dilución 1:1,000; Gel 1) y anticuerpo nanoluciferasa o CRAF (dilución 1:500; Gel 2) en solución salina tamponada con tris, suplementado con Tween-20 al 0,2% (TBST; Tabla de Materiales).
- Lave las membranas 1 vez durante 5 min en 10 mL de TBST y luego incube a temperatura ambiente durante 1 h con el anticuerpo secundario HRP anti-ratón, diluido 1:10.000 en TBST.
- Lave las membranas 3 veces en una plataforma oscilante en 10 mL de TBST a 25 °C durante 5 min cada una. Retire el TBST y visualice las bandas de proteínas utilizando reactivos ECL y un procesador de película de rayos X u otro sistema de imagen adecuado.
Resultados
Cuando se realiza como se describe en este protocolo (Figura 2), la interacción de Nano-CRAFWT y 14-3-3ζ-Halo debería producir proporciones BRET corregidas de 50-60 mBU (Figura 3A; Cuadro complementario 1). CRAF contiene dos sitios de acoplamiento 14-3-3 dependientes de fosforilación, el sitio N' y el sitio C' (Figura 1)8. Por lo tanto, los controles apropiados para reducir la ...
Discusión
Estudios previos han demostrado que las proteínas 14-3-3 desempeñan un papel fundamental tanto en la activación como en la inhibición de las quinasas RAF. Comprender cómo se regulan estos eventos de unión y los efectos de la modulación de estas interacciones en la señalización de RAF y la oncogénesis impulsada por RAF puede descubrir nuevas vulnerabilidades terapéuticas que se dirigen a la función de CRAF. Sin embargo, el ciclo de activación de Raf está respaldado por una gran cantidad de proteínas asociad...
Divulgaciones
Nada que revelar.
Agradecimientos
Este proyecto ha sido financiado en parte con fondos federales del Instituto Nacional del Cáncer, Institutos Nacionales de Salud, bajo el número de proyecto ZIA BC 010329.
Materiales
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| HaloTag® mouse monoclonal antibody | Promega | G9211 | Antibody for detecting HaloTag tagged proteins by immunoblot |
| NanoLuc® mouse monoclonal antibody | R&D Systems | MAB10026 | Antibody for detecting Nano-tagged proteins by immunoblot |
| CRAF mouse monoclonal antibody (E10) | Santa Crus Biotechnology | sc-7267 | Antibody directly detecting CRAF proteins by immunoblot |
| ECL anti-mouse HRP secondary antibody | Amersham | NA931-1ML | Secondary HRP conjugated mouse antibody (from sheep) |
| Reagents | |||
| X-tremeGENE™ 9 | Roche/Sigma | 6365809001 | |
| NanoBRET™ kit | Promega | N1661 | NanoBRET kit containing Halo 618 ligand and NanoGlo (nanoluciferase) substrate |
| DPBS, without Ca++ and Mg++ | Quality Biologicals | 114-057-101 | |
| Trypsin-EDTA (0.05%), phenol red | Life Technologies | 25300120 | |
| DMEM cell culture media | Life Technologies | 11995073 | High glucose, L-glutamine, phenol red, sodium pyruvate; without HEPES, suppliment media with 10% FBS, 2 mM L-glutamine and 100U penicillin-streptomycin |
| L-Glutamine (200 mM) | Life Technologies | 25030164 | |
| Penicillin-Streptomycin (10,000 U/mL) | Life Technologies | 15140163 | |
| Opti-MEM™ I reduced serum media | Gibco | 31985062 | For cell transfection |
| Opti-MEM reduced serum media, no phenol red | Gibco | 11058021 | For replating cells on Day 3. Supplement with 2 mM L-glutamine and 100U penicillin-streptomycin, along with 10% FBS (where indicated). |
| Invitrogen Trypan Blue Stain | Thermo Scientific | T10282 | |
| NP40 lysis buffer | N/A | N/A | 20 mM Tris (pH 8.0), 137mM NaCl, 10% glycerol, NP40 alternative (Milipore, Cat# 492016). Store at 4 degrees C.. Add the following protease and phosphatase immediately prior to use: 20 µM leupeptin, 0.5 mM sodium orthovanidate, 0.15 U/mL, 1mM PMSF. |
| 5x gel sample buffer | N/A | N/A | 240 mM Tris (pH 8.0), 9.5% SDS, 30% glycerol, 500mM DTT, 3mM bromophenol blue. Store at -20 degrees C. |
| Cell lines | |||
| 293FT cells (human) | Thermo Scientific | R70007 | |
| DNA vectors | |||
| pCMV5-Nano-CRAF WT and mutant | N/A | N/A | |
| pCMV5-14-3-3ζ-Halo | N/A | N/A | |
| Equipment | |||
| EnVision 2104 Multimode Plate Reader | PerkinElmer 2104 | 2104-0010 | 600LP NanoBRET & M460/50 nm NanoBRET emmisions filters, Luminescence 404 mirror, 6.5 mm measurement height and 0.1 s measurement time |
| Invitrogen Countess™ II Automated Cell Counter | Thermo Scientific | AMQAX1000 | |
| ThermoFisher E1-ClipTip™ Multichannel Pipettor | Thermo Scientific | 4672070 | |
| Software | |||
| GraphPad Prism (version 10.0.3) | GraphPad | www.graphpad.com | |
| Other | |||
| ThermoFisher ClipTip Multichannel pipette tips | Thermo Scientific | 94410153 | |
| Reagent Reservoir, 25 mL Divided, Sterile | Thomas Scientific | 1228K16 | |
| Perkin Elmer 384-well CulturPlate™ | PerkinElmer | 6007680 | White, polystyrene, tissue culture treated |
| Countess Cell Counting Chamber Slides | Thermo Scientific | C10228 |
Referencias
- Blasco, R. B., et al. c-Raf, but not B-Raf, is essential for development of K-Ras oncogene-driven non-small cell lung carcinoma. Cancer Cell. 19, 652-663 (2011).
- Blasco, M. T., et al. Complete regression of advanced pancreatic ductal adenocarcinomas upon combined inhibition of EGFR and C-RAF. Cancer Cell. 35, 573-587 (2019).
- Karreth, F. A., Frese, K. K., DeNicola, G. M., Baccarini, M., Tuveson, D. A. C-Raf is required for the initiation of lung cancer by K-Ras(G12D). Cancer Discov. 1, 128-136 (2011).
- Lito, P., et al. Disruption of CRAF-mediated MEK activation is required for effective MEK inhibition in KRAS mutant tumors. Cancer Cell. 25, 697-710 (2014).
- Sanclemente, M., et al. c-RAF ablation induces regression of advanced Kras/Trp53 mutant lung adenocarcinomas by a mechanism independent of MAPK signaling. Cancer Cell. 33, 217-228 (2018).
- Razzaque, M. A., et al. Germline gain-of-function mutations in RAF1 cause Noonan syndrome. Nat Genet. 39, 1013-1017 (2007).
- Pandit, B., et al. Gain-of-function RAF1 mutations cause Noonan and LEOPARD syndromes with hypertrophic cardiomyopathy. Nat Genet. 39, 1007-1012 (2007).
- Terrell, E. M., Morrison, D. K. Ras-mediated activation of the Raf family kinases. Cold Spring Harb Perspect Med. 9 (1), 033746 (2019).
- Kondo, Y., et al. Cryo-EM structure of a dimeric B-Raf:14-3-3 complex reveals asymmetry in the active sites of B-Raf kinases. Science. 366, 109-115 (2019).
- Park, E., et al. Architecture of autoinhibited and active BRAF-MEK1-14-3-3 complexes. Nature. 575 (7783), 545-550 (2019).
- Tzivion, G., Luo, Z., Avruch, J. A dimeric 14-3-3 protein is an essential cofactor for Raf kinase activity. Nature. 394, 88-92 (1998).
- Spencer-Smith, R., et al. RASopathy mutations provide functional insight into the BRAF cysteine-rich domain and reveal the importance of autoinhibition in BRAF regulation. Mol Cell. 82, 4262-4276 (2022).
- Martinez Fiesco, J. A., Durrant, D. E., Morrison, D. K., Zhang, P. Structural insights into the BRAF monomer-to-dimer transition mediated by RAS binding. Nat Commun. 13, 486 (2022).
- Freeman, A. K., Ritt, D. A., Morrison, D. K. The importance of Raf dimerization in cell signaling. Small GTPases. 4, 180-185 (2013).
- Freeman, A. K., Ritt, D. A., Morrison, D. K. Effects of Raf dimerization and its inhibition on normal and disease-associated Raf signaling. Mol Cell. 49, 751-758 (2013).
- Rushworth, L. K., Hindley, A. D., O'Neill, E., Kolch, W. Regulation and role of Raf-1/B-Raf heterodimerization. Mol Cell Biol. 26, 2262-2272 (2006).
- Garnett, M. J., Rana, S., Paterson, H., Barford, D., Marais, R. Wild-type and mutant B-RAF activate C-RAF through distinct mechanisms involving heterodimerization. Mol Cell. 20, 963-969 (2005).
- Tran, N. H., Wu, X., Frost, J. A. B-Raf and Raf-1 are regulated by distinct autoregulatory mechanisms. J Biol Chem. 280, 16244-16253 (2005).
- Chong, H., Guan, K. L. Regulation of Raf through phosphorylation and N terminus-C terminus interaction. J Biol Chem. 278, 36269-36276 (2003).
- Cutler, R. E., Stephens, R. M., Saracino, M. R., Morrison, D. K. Autoregulation of the Raf-1 serine/threonine kinase. Proc Natl Acad Sci U S A. 95, 9214-9219 (1998).
- Park, E., et al. Cryo-EM structure of a RAS/RAF recruitment complex. Nat Commun. 14, 4580 (2023).
- Spencer-Smith, R., Morrison, D. K. Protocol for measuring BRAF autoinhibition in live cells using a proximity-based NanoBRET assay. STAR Protoc. 4, 102461 (2023).
- Clark, G. J., et al. 14-3-3 zeta negatively regulates raf-1 activity by interactions with the Raf-1 cysteine-rich domain. J Biol Chem. 272, 20990-20993 (1997).
- Machleidt, T., et al. NanoBRET--A novel BRET platform for the analysis of protein-protein interactions. ACS Chem Biol. 10, 1797-1804 (2015).
- Hekman, M., et al. Dynamic changes in C-Raf phosphorylation and 14-3-3 protein binding in response to growth factor stimulation: differential roles of 14-3-3 protein binding sites. J Biol Chem. 279, 14074-14086 (2004).
- Hatzivassiliou, G., et al. RAF inhibitors prime wild-type RAF to activate the MAPK pathway and enhance growth. Nature. 464, 431-435 (2010).
- Bondzi, C., Grant, S., Krystal, G. W. A novel assay for the measurement of Raf-1 kinase activity. Oncogene. 19, 5030-5033 (2000).
- Spencer-Smith, R., et al. Inhibition of RAS function through targeting an allosteric regulatory site. Nat Chem Biol. 13 (1), 62-68 (2016).
- Roy, S., et al. 14-3-3 facilitates Ras-dependent Raf-1 activation in vitro and in vivo. Mol Cell Biol. 18, 3947-3955 (1998).
- Durrant, D. E., et al. Development of a high-throughput NanoBRET screening platform to identify modulators of the RAS/RAF interaction. Mol Cancer Ther. 20, 1743-1754 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados