Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Biolumineszenz-Resonanz-Energietransfer (BRET)-basierter Assay zur Messung der Wechselwirkungen von CRAF mit 14-3-3-Proteinen in lebenden Zellen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt einen BRET-basierten Assay zur Messung der Wechselwirkungen der CRAF-Kinase mit 14-3-3-Proteinen in lebenden Zellen. Das Protokoll beschreibt die Schritte zur Vorbereitung der Zellen, zum Ablesen der BRET-Emissionen und zur Datenanalyse. Ein Beispielergebnis mit der Identifizierung geeigneter Kontrollen und der Fehlerbehebung für die Assay-Optimierung wird ebenfalls vorgestellt.
Zusammenfassung
CRAF ist ein primärer Effektor von RAS-GTPasen und spielt eine entscheidende Rolle bei der Tumorgenese mehrerer KRAS-getriebener Krebsarten. Darüber hinaus ist CRAF ein Hotspot für Keimbahnmutationen, die nachweislich die entwicklungsbedingte RASopathie, das Noonan-Syndrom, verursachen. Alle RAF-Kinasen enthalten mehrere phosphorylierungsabhängige Bindungsstellen für 14-3-3-regulatorische Proteine. Die differentielle Bindung von 14-3-3 an diese Stellen spielt eine wesentliche Rolle bei der Bildung aktiver RAF-Dimere an der Plasmamembran unter Signalbedingungen und bei der Aufrechterhaltung der RAF-Autoinhibition unter Ruhebedingungen. Zu verstehen, wie diese Wechselwirkungen reguliert werden und wie sie moduliert werden können, ist entscheidend für die Identifizierung neuer therapeutischer Ansätze, die auf die RAF-Funktion abzielen. Hier beschreibe ich einen auf Biolumineszenz-Resonanzenergietransfer (BRET) basierenden Assay zur Messung der Wechselwirkungen von CRAF mit 14-3-3-Proteinen in lebenden Zellen. Insbesondere misst dieser Assay die Wechselwirkungen von CRAF, das mit einem Nano-Luciferase-Donor fusioniert ist, und 14-3-3, das mit einem Halo-Tag-Akzeptor fusioniert ist, wobei die Interaktion von RAF und 14-3-3 zu einem Energietransfer zwischen Donor und Akzeptor und der Erzeugung des BRET-Signals führt. Das Protokoll zeigt ferner, dass dieses Signal durch Mutationen gestört werden kann, die nachweislich die Bindung von 14-3-3 an jede seiner hochaffinen RAF-Andockstellen verhindern. Dieses Protokoll beschreibt die Verfahren für die Aussaat, Transfektion und Neubeschichtung der Zellen sowie detaillierte Anweisungen zum Ablesen von BRET-Emissionen, zur Durchführung der Datenanalyse und zur Bestätigung der Proteinexpressionsniveaus. Darüber hinaus werden Beispiel-Assay-Ergebnisse sowie Optimierungs- und Fehlerbehebungsschritte bereitgestellt.
Einleitung
RAF-Kinasen (ARAF, BRAF und CRAF) sind die direkten Effektoren der RAS-GTPasen und die initiierenden Mitglieder der pro-proliferativen/überlebensfördernden RAF-MEK-ERK-Kinase-Kaskade. Jüngste Studien haben gezeigt, dass die CRAF-Expression eine Schlüsselrolle bei der Tumorgenese mehrerer KRAS-getriebener Krebsarten spielt, darunter nicht-kleinzelliger Lungenkrebs und duktales Adenokarzinomder Bauchspeicheldrüse 1,2,3,4,5. Darüber hinaus verursachen Keimbahn-CRAF-Mutationen eine besonders schwere Form der RASopathie, das Noonan-Syndrom 6,7. Das Verständnis der CRAF-Regulation ist entscheidend für die Entwicklung erfolgreicher therapeutischer Ansätze, die auf seine Funktion in Zellen abzielen.
Alle RAF-Kinasen können in zwei funktionelle Domänen unterteilt werden, eine C-terminale katalytische (CAT) Domäne und eine N-terminale regulatorische (REG) Domäne, die ihre Aktivität steuert (Abbildung 1A)8. Die REG-Domäne umfasst die RAS-Bindungsdomäne (RBD), die Cystein-reiche Domäne (CRD) und eine serin/Threonin-reiche Region (S/T-reich). Bemerkenswert ist, dass die S/T-reiche Region die N'-Stelle enthält, die phosphorylierungsabhängig an 14-3-3 bindet (S259 in CRAF; Abbildung 1A) 8. Die CAT-Domäne umfasst die Kinase-Domäne zusammen mit einer zweiten hochaffinen 14-3-3-Andockstelle, die als C'-Stelle bezeichnet wird (S621 in CRAF; Abbildung 1A) 8. Die differentielle Bindung von dimeren 14-3-3-Proteinen an die N'- und C'-Stellen spielt zusammen mit der CRD eine entscheidende Rolle sowohl bei der RAF-Aktivierung als auch bei der Hemmung 9,10,11,12,13. Unter normalen Signalbedingungen wird die RAF-Aktivierung durch seine Rekrutierung an die Plasmamembran durch RAS initiiert, wodurch aktive Dimere gebildet werden können, von denen das BRAF-CRAF-Heterodimer die vorherrschende aktive Formist 14,15. Biochemische Assays mit BRAF und CRAF sowie kryogene Elektronenmikroskopie-Strukturen (Kryo-EM) von dimerem BRAF deuten darauf hin, dass ein 14-3-3-Dimer aktive RAF-Dimere stabilisiert, indem es gleichzeitig an die C'-Stelle beider RAF-Protomere bindet (Abbildung 1B)9,13,16,17. Umgekehrt haben Studien gezeigt, dass RAF unter Ruhebedingungen eine zytosolische, autoinhibierte Bestätigung annimmt, bei der die REG-Domäne an die CAT-Domäne bindet und deren Aktivität hemmt 12,18,19,20. Dieser geschlossene Zustand wird durch ein 14-3-3-Dimer stabilisiert, das an die CRD- und N'-Stelle in der REG-Domäne und an die C'-Stelle in der CAT-Domäne gebunden ist (Abbildung 1B)10,13,21. Bei BRAF wird dieses Modell durch neuere Kryo-EM-Strukturen von autoinhibierten BRAF-Monomeren und durch unsere früheren biochemischen Studienunterstützt 10,12,13,21,22. Während jedoch gezeigt wurde, dass 14-3-3 eine hemmende Rolle bei der CRAF-Regulationspielt 23, könnte ein BRAF-ähnlicher autoinhibierter Zustand eine geringere Rolle bei der CRAF-Regulation spielen12; daher sind weitere Studien erforderlich, um die Mechanismen zu klären, durch die 14-3-3-Proteine die CRAF-Aktivität regulieren. Die 14-3-3-vermittelte Regulation von RAF-Kinasen erfordert eine Vielzahl von RAF-Phosphorylierungs- und Dephosphorylierungsereignissen, die Bindung an verschiedene regulatorische Proteine und Wechselwirkungen mit der Plasmamembran8. Daher ist es wichtig, dass 14-3-3-RAF-Wechselwirkungen unter physiologisch relevanten Bedingungen und in Gegenwart einer intakten Lipiddoppelschicht gemessen werden.
Um dieses Problem zu lösen, wurde die NanoBRET-Technologie (im Folgenden als N-BRET bezeichnet; siehe Materialtabelle für Details zum Kit) eingesetzt, um einen näherungsbasierten Assay zur Messung der Wechselwirkungen von CRAF mit 14-3-3-Proteinen in lebenden Zellen zu entwickeln (Abbildung 1C). Dieses BRET-basierte System misst die Wechselwirkungen von zwei Proteinen von Interesse (POI), wobei ein Protein mit einem Nanoluciferase (Nano)-Donor und das andere mit einem Halo-Tag markiert wird, um mit dem Halo618-Energieakzeptorliganden22,24 markiert zu werden. Die Interaktion der interessierenden Proteine führt zu einem Energietransfer von Donor zu Akzeptor, der wiederum das BRET-Signal erzeugt (Abbildung 1C). Das extrem helle Nano-Donorprotein (Emission (em) 460 nm) und der Halo618-Ligand (em 618 nm) bieten eine größere spektrale Trennung und Empfindlichkeit als herkömmliche BRET, was es zu einer idealen Plattform für die Untersuchung schwächerer Wechselwirkungen und den Nachweis subtiler Änderungen in der Bindungmacht 24. In der Tat haben wir zuvor einen N-BRET-basierten Assay zur Messung der autoinhibitorischen Wechselwirkungen der RAF REG- und CAT-Domänen entwickelt, der für die Charakterisierung einer Reihe von RASopathie-Mutationen in der BRAF-CRD unerlässlich war und die entscheidende Bedeutung dieser Domäne für die Aufrechterhaltung der Autoinhibition und die Verhinderung der konstitutiven BRAF-Aktivierung demonstrierte12.
Der hier beschriebene Assay misst die Wechselwirkungen von CRAF, das mit einem N-terminalen Nano-Tag (Nano-CRAF) fusioniert ist, und der Zeta-Isoform von 14-3-3, die mit einem C-terminalen Halo-Tag fusioniert ist (14-3-3ζ-Halo; Abbildung 1C). Wir zeigen, dass die Wechselwirkungen von Nano-CRAF mit 14-3-3ζ-Halo ein robustes BRET-Signal erzeugen, das wiederum durch Mutationen gestört werden kann, die eine Bindung von 14-3-3 an die N'-Stelle (S259A) und/oder die C'-Stelle (S621A) verhindern. Das folgende Protokoll enthält detaillierte Schritte zur Durchführung, Optimierung und Fehlerbehebung dieses Assays.
Protokoll
HINWEIS: Dieser Assay wird in 293FT-Zellen durchgeführt. Eine gut charakterisierte und leicht transfizierbare Epithellinie, die aus menschlichen embryonalen Nierenzellen gewonnen wird. Eine einzelne konfluente 10-cm-Kulturschale dieser Zellen liefert in der Regel genügend Zellen für die Aussaat von 20 Wells mit 6-Well-Gewebekulturplatten. Die Schritte 1-3 müssen in steriler Technik in einer biologischen Sicherheitswerkbank durchgeführt werden.
1. Aussaat der Zellen (Tag 1)
HINWEIS: In diesem Schritt werden die Zellen von der/den Gewebekulturschale(n) gelöst, gezählt und in 6-Well-Gewebekulturplatten für die Transfektion in Schritt 2 ausgesät (Abbildung 2).
- Aspirieren Sie das Medium aus den Zellen in der 10-cm-Schale. Waschen Sie die Zellen mit 5 mL phosphatgepufferter Kochsalzlösung (PBS) und aspirieren Sie sie.
- 1 ml Trypsin-Ethylendiamintetraessigsäure (EDTA) zugeben und 3-5 Minuten bei 37 °C inkubieren, um die Zellen von der Schale zu lösen.
- Geben Sie 9 ml vollständiges Dulbecco's modifiziertes Eagle-Medium (DMEM) zu den Zellen, um Trypsin zu neutralisieren, und pipettieren Sie wiederholt auf und ab, um eine homogene Einzelzellsuspension zu erzeugen.
- Sofort 20 μl Zellen in ein 1,7-ml-Röhrchen umfüllen und mit 20 μl Trypanblau-Färbung mischen. Zählen Sie die Zellen entweder mit einem automatisierten Zellzähler (Table of Materials) oder einem Hämozytometer.
- Verdünnen Sie die Zellen auf 2 x 105 Zellen/ml in vollständigem DMEM-Medium und geben Sie 2 mL in jede Vertiefung einer 6-Well-Gewebekulturplatte (4 x 10,5 Zellen/Well). Die Zellen werden über Nacht bei 37 °C und 10 % CO2 inkubiert.
HINWEIS: Es wird empfohlen, 293FT-Zellen zu verwenden, die weniger als 20x durchlaufen wurden. Wir haben bereits festgestellt, dass die Verwendung von Zellen mit größeren Passagenzahlen zu reduzierten BRET-Verhältnissen und einer erhöhten Variabilität des Well-to-Well-Signals führen kann.
2. Zelltransfektion (Tag 2)
HINWEIS: Hier werden die Zellen mit den Expressionskonstrukten pCMV5-NanoLuc-CRAF und pCMV5-14-3-3ζ-Halo zusammen mit dem leeren Vektor pCDNA3.1 transfiziert (Abbildung 2).
- Vor der Transfektion verdünnen Sie die N-BRET-Plasmide auf 5 ng/μl und pCDNA3.1 auf 100 ng/μl und nummerieren Sie einen Satz steriler 1,7-ml-Röhrchen.
- Geben Sie 100 μl Transfektionsmedium (siehe Materialtabelle für Details) zusammen mit 5 ng pCMV5-NanoLuc-CRAF, 10 ng pCMV5-14-3-3ζ und 200 ng PCDNA3.1 in jedes Röhrchen.
- Fügen Sie 2 μl Transfektionsreagenz hinzu (siehe Materialtabelle für Details) und wirbeln Sie es vorsichtig zum Mischen. Schleudern Sie die Röhrchen kurz in einer Mikrozentrifuge, um sicherzustellen, dass die gesamte Flüssigkeit am Boden der Röhrchen gesammelt wird, und inkubieren Sie dann 15 Minuten lang bei etwa 25 °C.
- Transfektionskomplexe tropfenweise in die Zellen geben und 16-20 h bei 37 °C/10 % CO2 inkubieren, damit die Halo- und Nano-markierten Proteine exprimiert werden können.
HINWEIS: Die Zugabe einer Träger-DNA mit einem leeren Vektor (pCDNA3.1) ist unerlässlich, um eine hohe Transfektionseffizienz der N-BRET-Expressionskonstrukte zu erreichen. Das Versäumnis, einen leeren Vektor hinzuzufügen, führt zu einer verringerten Expression der 14-3-3ζ-Halo- und Nano-CRAF-Proteine und führt wiederum zu schwachen und inkonsistenten BRET-Verhältnissen, wie zuvor diskutiert22.
3. Neubeschichtung der Zellen (Tag 3)
HINWEIS: In diesem Schritt werden die Zellen auf eine 384-Well-Platte übertragen und entweder Halo 618-Ligand (+Ligand; Tabelle der Materialien) oder DMSO (+Fahrzeug) wird zum Ablesen der BRET-Emissionen in Schritt 4 hinzugefügt. Die verbleibenden Zellen werden für die Western-Blot-Analyse in Schritt 5 auf frische 6-Well-Kulturplatten überführt (Abbildung 2).
- Sammeln Sie die folgenden Materialien und bereiten Sie den Arbeitsbereich wie unten beschrieben vor.
- In einem 37 °C warmen Wasserbad wird Trypsin-EDTA zusammen mit serumfreien Assay-Medien und 10 % FBS-supplementierten Assay-Medien vorerwärmt (siehe Materialtabelle für Details).
- Basierend auf der Anzahl der zu messenden Proben werden drei Sätze sterile 1,7-ml-Röhrchen (Set 1-3) und ein Satz sterile 15-ml-Röhrchen mit konischem Boden voretikettiert. Rüsten Sie eine Schwenkbecherzentrifuge mit 15-ml-Röhrcheneinsätzen aus und kühlen Sie sie auf 4 °C vor.
- Legen Sie die folgenden Gegenstände in die Gewebekulturhaube: Reagenzienreservoire, 384-Well-Gewebekulturplatten, 6-Well-Gewebekulturplatten, eine Mehrkanalpipette und -spitzen, Objektträger/Kammern für die Zellzählung und Trypanblau-Färbung (Materialtabelle) sowie die 1,7-ml-Röhrchen-Sets 1-3.
- Ernten und zählen Sie die Zellen wie unten beschrieben.
- Aspirieren Sie das Medium aus den 6-Well-Platten und geben Sie 250 μl Trypsin-EDTA in die Zellen. Inkubieren Sie die 6-Well-Platten bei 37 °C, bis sich die Zellen abzulösen beginnen (3-5 Minuten).
- Geben Sie 1 ml 10 % FBS-supplementiertes Assay-Medium in jede Vertiefung, um das Trypsin zu neutralisieren, und pipettieren Sie kräftig auf und ab, um eine Einzelzellsuspension zu erzeugen.
- Übertragen Sie 1 ml der Zellsuspension in die vormarkierten 15-ml-Röhrchen. Geben Sie weitere 1 ml 10 % FBS-supplementiertes Testmedium in jedes der 15 ml-Röhrchen.
- Drehen Sie die Röhrchen 5x um, um sie zu mischen, und überführen Sie sofort 20 μl Zellsuspension in das Röhrchenset 1.
- Zentrifuge in der vorgekühlten Schwenkschaufelzentrifuge für 5 min bei ~250 x g. Mischen Sie während des Zentrifugationsschritts 20 μl Trypanblau-Färbung mit den Zellen im Röhrchensatz 1 und zählen Sie die Zellen dann entweder mit einem automatisierten Zellzähler oder einem Hämozytometer. Typisch ist eine Ausbeute von 6-8 x 105 Zellen/ml.
- Nehmen Sie die 15-ml-Röhrchen aus der Zentrifuge und saugen Sie das Medium aus den Zellpellets an. Resuspendieren Sie die Zellpellets auf 2 x 105 Zellen/ml in serumfreien Assay-Medien und pipettieren Sie kräftig, um eine Einzelzellsuspension zu erzeugen.
HINWEIS: Die Verwendung von serumfreien Assay-Medien wird verwendet, um normale zelluläre Signalwege zum Stillstand zu bringen.
- Plattieren Sie die Zellen in 384-Well- und 6-Well-Platten wie unten beschrieben.
- Drehen Sie die 15-ml-Röhrchen mehrmals um, um eine homogene Zellsuspension zu gewährleisten, und übertragen Sie 500 μl auf 1,7 ml-Röhrchen Set 2 und Set 3.
- 0,5 μl DMSO (+Vehikel) zu Satz 2 und 0,5 μl Halo 618 Ligand (+Ligand) zu Satz 3 des Röhrchens hinzufügen und zum Mischen pipettieren.
- Übertragen Sie die Suspensionen der +Vehikel- und +Ligandenzellen in separate Vertiefungen mit Reagenzreservoiren. Übertragen Sie mit der Mehrkanalpipette (Materialtabelle) 40 μl der +Vehikel-Zellsuspension aus den Reagenzreservoiren in vierfache Vertiefungen der 384-Well-Kulturplatte. Wiederholen Sie diesen Schritt für die +Ligandenzellsuspensionen.
- Übertragen Sie die restlichen Zellen in frische 6-Well-Kulturplatten. Die 384-Well- und 6-Well-Platten über Nacht bei 37 °C und 5 % CO2 inkubieren.
4. Ablesen der BRET-Emissionen (Tag 4)
HINWEIS: In diesem Schritt wird das Nanoluziferase-Substrat (siehe Materialtabelle für Details) zu den Zellen in der 384-Well-Kulturplatte hinzugefügt und die N-BRET-Akzeptor- (618 nm) und Donor-Emissionen (460 nm) werden abgelesen (Abbildung 2). Anschließend werden die korrigierten BRET-Verhältnisse berechnet.
- Erwärmen Sie serumfreie Assay-Medien in einem 37 °C warmen Wasserbad und tauen Sie das Nanoluziferase-Substrat bei 25 °C auf. Verdünnen Sie das Nanoluziferase-Substrat 1:100 in serumfreiem Assay-Medium und überführen Sie es in ein Reagenzreservoir. Bereiten Sie genug von dieser Mischung vor, um 10 μl in jede Vertiefung der 384-Well-Platte plus 10 % bis 15 % zusätzliches Volumen zu geben.
- Übertragen Sie mit einer Mehrkanalpipette (Materialtabelle) 10 μl der Substratmischung in jede der Vertiefungen, die Zellen in der 384-Well-Kulturplatte enthalten. Drehen Sie die Platte vorsichtig 1 Minute lang, entweder manuell oder mit einem Orbitalschüttler.
- Setzen Sie die 384-Well-Platte in den Multimode-Plattenleser ein und zeichnen Sie die 460-nm- und 618-nm-Emissionen für alle Wells auf, die Zellen enthalten.
HINWEIS: Für den für diesen Schritt verwendeten Multimode-Plattenleser (siehe Materialtabelle für Details) wurde eine Lesehöhe von 6,5 mm mit einer Lesezeitmessung von 0,1 s verwendet. Bei Verwendung anderer Plattenleser können jedoch weitere Optimierungen erforderlich sein. - Berechnen Sie die rohen BRET-Verhältnisse für +Fahrzeug und +Ligand in MilliBRET-Einheiten (mBU) einzeln, indem Sie die folgende Gleichung verwenden:
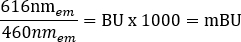
- Berechnen Sie das korrigierte BRET-Verhältnis, indem Sie das +Fahrzeug-BRET-Verhältnis von dem des +Liganden subtrahieren (Ergänzende Tabelle 1).
HINWEIS: Beim Vergleich der Ergebnisse mehrerer Experimente können die korrigierten BRET-Verhältnisse gepoolt und auf den Durchschnitt des Wildtype (WT) N-BRET-Paares, bestehend aus Nano-CRAFWT und 14-3-3ζWT-Halo, normiert werden (Ergänzende Tabelle 1).
5. Bestätigung der Proteinexpressionsniveaus (Tage 4 und 5)
HINWEIS: In diesem Schritt werden die Zellen in den 6-Well-Platten lysiert und die Proteinexpressionsniveaus der Nano-CRAF- und 14-3-3ζ-Halo-Proteine durch Western-Blot-Analyse unter Verwendung von Antikörpern, die für die Halo- und Nano-Tags spezifisch sind, bestimmt. (Abbildung 2).
- Fügen Sie dem NP40-Lysepuffer Protease- und Phosphatase-Inhibitoren hinzu (Materialtabelle), sodass 200 μl Lysepuffer pro Probe vorhanden sind.
- Aspirieren Sie Medien von 6-Well-Platten, die Zellen enthalten. Waschen Sie die Zellen einmal mit 1 mL kaltem PBS und aspirieren Sie.
- Geben Sie 125 μl NP40-Lysepuffer in jede Vertiefung und inkubieren Sie 6-Well-Platten bei 4 °C auf einer Wippplattform für 15 Minuten, um die Zellen zu lysieren.
- Lysate auf Eis in 1,7 mL Röhrchen überführen und bei 20.000 x g für 10 min bei 4 °C zentrifugieren. Legen Sie die geklärten Lysate wieder auf Eis und bestimmen Sie die Proteinkonzentration mit kommerziell erhältlichen Assays, wie z. B. Bradford- oder Bicinchoninsäure (BCA)-Assays.
- Normalisieren Sie die Proteinkonzentration in allen Proben, indem Sie ein angemessenes Volumen an Lysat und NP40-Lysepuffer auf frische 1,7-ml-Röhrchen auf ein Gesamtvolumen von 100 μl übertragen.
- Kochen Sie 5x Proteinprobenpuffer (Materialtabelle) für 1 min und fügen Sie jeder Probe 25 μl hinzu. Kochen Sie die Proben 5-6 Minuten lang und zentrifugieren Sie die Röhrchen dann kurz, um sicherzustellen, dass sich die gesamte Probe im Boden des Röhrchens sammelt.
- Laden Sie 25 μl Probe in jede Vertiefung mit doppelten Proteingelen (Gel 1 und Gel 2) und übertragen Sie dann Proteine auf Nitrozellulose- oder PVDF-Membranen unter Verwendung von Standard-Western-Blotting-Verfahren, wie zuvor beschrieben22.
- Blockieren Sie die Membranen in 3 % BSA-PBS bei 25 °C für 30 Minuten und inkubieren Sie die Membranen dann über Nacht mit Halo-Antikörpern (1:1.000 Verdünnung; Gel 1) und entweder Nanoluziferase oder CRAF-Antikörper (1:500 Verdünnung; Gel 2) in Tris-gepufferter Kochsalzlösung, ergänzt mit 0,2 % Tween-20 (TBST; Tabelle der Materialien).
- Waschen Sie die Membranen 1x für 5 min in 10 mL TBST und inkubieren Sie dann 1 h lang bei Raumtemperatur mit Anti-Maus-HRP-Sekundärantikörper, verdünnt 1:10.000 in TBST.
- Membranen 3x auf einer Wippplattform in 10 mL TBST bei 25 °C für jeweils 5 min waschen. Entfernen Sie TBST und visualisieren Sie Proteinbanden mit ECL-Reagenzien und entweder einem Röntgenfilmprozessor oder einem anderen geeigneten Bildgebungssystem.
Ergebnisse
Wenn die Wechselwirkung von Nano-CRAFWT und 14-3-3ζ-Halo wie in diesem Protokoll beschrieben durchgeführt wird, sollte sie korrigierte BRET-Verhältnisse von 50-60 mBU erzeugen (Abbildung 3A; Ergänzende Tabelle 1). CRAF enthält zwei phosphorylierungsabhängige 14-3-3-Andockstellen, die N'-Stelle und die C'-Stelle (Abbildung 1)8. Daher umfassen geeignete Kontrollen zur Verringer...
Diskussion
Frühere Studien haben gezeigt, dass 14-3-3-Proteine eine entscheidende Rolle sowohl bei der Aktivierung als auch bei der Hemmung von RAF-Kinasen spielen. Das Verständnis, wie diese Bindungsereignisse reguliert werden und welche Auswirkungen die Modulation dieser Wechselwirkungen auf die RAF-Signalgebung und die RAF-gesteuerte Onkogenese hat, könnte neue therapeutische Schwachstellen aufdecken, die auf die CRAF-Funktion abzielen. Der Raf-Aktivierungszyklus wird jedoch durch eine Vielzahl von assoziierten Proteinen, pos...
Offenlegungen
Nichts offenzulegen.
Danksagungen
Dieses Projekt wurde teilweise mit Bundesmitteln des National Cancer Institute, National Institutes of Health, unter der Projektnummer ZIA BC 010329 finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| HaloTag® mouse monoclonal antibody | Promega | G9211 | Antibody for detecting HaloTag tagged proteins by immunoblot |
| NanoLuc® mouse monoclonal antibody | R&D Systems | MAB10026 | Antibody for detecting Nano-tagged proteins by immunoblot |
| CRAF mouse monoclonal antibody (E10) | Santa Crus Biotechnology | sc-7267 | Antibody directly detecting CRAF proteins by immunoblot |
| ECL anti-mouse HRP secondary antibody | Amersham | NA931-1ML | Secondary HRP conjugated mouse antibody (from sheep) |
| Reagents | |||
| X-tremeGENE™ 9 | Roche/Sigma | 6365809001 | |
| NanoBRET™ kit | Promega | N1661 | NanoBRET kit containing Halo 618 ligand and NanoGlo (nanoluciferase) substrate |
| DPBS, without Ca++ and Mg++ | Quality Biologicals | 114-057-101 | |
| Trypsin-EDTA (0.05%), phenol red | Life Technologies | 25300120 | |
| DMEM cell culture media | Life Technologies | 11995073 | High glucose, L-glutamine, phenol red, sodium pyruvate; without HEPES, suppliment media with 10% FBS, 2 mM L-glutamine and 100U penicillin-streptomycin |
| L-Glutamine (200 mM) | Life Technologies | 25030164 | |
| Penicillin-Streptomycin (10,000 U/mL) | Life Technologies | 15140163 | |
| Opti-MEM™ I reduced serum media | Gibco | 31985062 | For cell transfection |
| Opti-MEM reduced serum media, no phenol red | Gibco | 11058021 | For replating cells on Day 3. Supplement with 2 mM L-glutamine and 100U penicillin-streptomycin, along with 10% FBS (where indicated). |
| Invitrogen Trypan Blue Stain | Thermo Scientific | T10282 | |
| NP40 lysis buffer | N/A | N/A | 20 mM Tris (pH 8.0), 137mM NaCl, 10% glycerol, NP40 alternative (Milipore, Cat# 492016). Store at 4 degrees C.. Add the following protease and phosphatase immediately prior to use: 20 µM leupeptin, 0.5 mM sodium orthovanidate, 0.15 U/mL, 1mM PMSF. |
| 5x gel sample buffer | N/A | N/A | 240 mM Tris (pH 8.0), 9.5% SDS, 30% glycerol, 500mM DTT, 3mM bromophenol blue. Store at -20 degrees C. |
| Cell lines | |||
| 293FT cells (human) | Thermo Scientific | R70007 | |
| DNA vectors | |||
| pCMV5-Nano-CRAF WT and mutant | N/A | N/A | |
| pCMV5-14-3-3ζ-Halo | N/A | N/A | |
| Equipment | |||
| EnVision 2104 Multimode Plate Reader | PerkinElmer 2104 | 2104-0010 | 600LP NanoBRET & M460/50 nm NanoBRET emmisions filters, Luminescence 404 mirror, 6.5 mm measurement height and 0.1 s measurement time |
| Invitrogen Countess™ II Automated Cell Counter | Thermo Scientific | AMQAX1000 | |
| ThermoFisher E1-ClipTip™ Multichannel Pipettor | Thermo Scientific | 4672070 | |
| Software | |||
| GraphPad Prism (version 10.0.3) | GraphPad | www.graphpad.com | |
| Other | |||
| ThermoFisher ClipTip Multichannel pipette tips | Thermo Scientific | 94410153 | |
| Reagent Reservoir, 25 mL Divided, Sterile | Thomas Scientific | 1228K16 | |
| Perkin Elmer 384-well CulturPlate™ | PerkinElmer | 6007680 | White, polystyrene, tissue culture treated |
| Countess Cell Counting Chamber Slides | Thermo Scientific | C10228 |
Referenzen
- Blasco, R. B., et al. c-Raf, but not B-Raf, is essential for development of K-Ras oncogene-driven non-small cell lung carcinoma. Cancer Cell. 19, 652-663 (2011).
- Blasco, M. T., et al. Complete regression of advanced pancreatic ductal adenocarcinomas upon combined inhibition of EGFR and C-RAF. Cancer Cell. 35, 573-587 (2019).
- Karreth, F. A., Frese, K. K., DeNicola, G. M., Baccarini, M., Tuveson, D. A. C-Raf is required for the initiation of lung cancer by K-Ras(G12D). Cancer Discov. 1, 128-136 (2011).
- Lito, P., et al. Disruption of CRAF-mediated MEK activation is required for effective MEK inhibition in KRAS mutant tumors. Cancer Cell. 25, 697-710 (2014).
- Sanclemente, M., et al. c-RAF ablation induces regression of advanced Kras/Trp53 mutant lung adenocarcinomas by a mechanism independent of MAPK signaling. Cancer Cell. 33, 217-228 (2018).
- Razzaque, M. A., et al. Germline gain-of-function mutations in RAF1 cause Noonan syndrome. Nat Genet. 39, 1013-1017 (2007).
- Pandit, B., et al. Gain-of-function RAF1 mutations cause Noonan and LEOPARD syndromes with hypertrophic cardiomyopathy. Nat Genet. 39, 1007-1012 (2007).
- Terrell, E. M., Morrison, D. K. Ras-mediated activation of the Raf family kinases. Cold Spring Harb Perspect Med. 9 (1), 033746 (2019).
- Kondo, Y., et al. Cryo-EM structure of a dimeric B-Raf:14-3-3 complex reveals asymmetry in the active sites of B-Raf kinases. Science. 366, 109-115 (2019).
- Park, E., et al. Architecture of autoinhibited and active BRAF-MEK1-14-3-3 complexes. Nature. 575 (7783), 545-550 (2019).
- Tzivion, G., Luo, Z., Avruch, J. A dimeric 14-3-3 protein is an essential cofactor for Raf kinase activity. Nature. 394, 88-92 (1998).
- Spencer-Smith, R., et al. RASopathy mutations provide functional insight into the BRAF cysteine-rich domain and reveal the importance of autoinhibition in BRAF regulation. Mol Cell. 82, 4262-4276 (2022).
- Martinez Fiesco, J. A., Durrant, D. E., Morrison, D. K., Zhang, P. Structural insights into the BRAF monomer-to-dimer transition mediated by RAS binding. Nat Commun. 13, 486 (2022).
- Freeman, A. K., Ritt, D. A., Morrison, D. K. The importance of Raf dimerization in cell signaling. Small GTPases. 4, 180-185 (2013).
- Freeman, A. K., Ritt, D. A., Morrison, D. K. Effects of Raf dimerization and its inhibition on normal and disease-associated Raf signaling. Mol Cell. 49, 751-758 (2013).
- Rushworth, L. K., Hindley, A. D., O'Neill, E., Kolch, W. Regulation and role of Raf-1/B-Raf heterodimerization. Mol Cell Biol. 26, 2262-2272 (2006).
- Garnett, M. J., Rana, S., Paterson, H., Barford, D., Marais, R. Wild-type and mutant B-RAF activate C-RAF through distinct mechanisms involving heterodimerization. Mol Cell. 20, 963-969 (2005).
- Tran, N. H., Wu, X., Frost, J. A. B-Raf and Raf-1 are regulated by distinct autoregulatory mechanisms. J Biol Chem. 280, 16244-16253 (2005).
- Chong, H., Guan, K. L. Regulation of Raf through phosphorylation and N terminus-C terminus interaction. J Biol Chem. 278, 36269-36276 (2003).
- Cutler, R. E., Stephens, R. M., Saracino, M. R., Morrison, D. K. Autoregulation of the Raf-1 serine/threonine kinase. Proc Natl Acad Sci U S A. 95, 9214-9219 (1998).
- Park, E., et al. Cryo-EM structure of a RAS/RAF recruitment complex. Nat Commun. 14, 4580 (2023).
- Spencer-Smith, R., Morrison, D. K. Protocol for measuring BRAF autoinhibition in live cells using a proximity-based NanoBRET assay. STAR Protoc. 4, 102461 (2023).
- Clark, G. J., et al. 14-3-3 zeta negatively regulates raf-1 activity by interactions with the Raf-1 cysteine-rich domain. J Biol Chem. 272, 20990-20993 (1997).
- Machleidt, T., et al. NanoBRET--A novel BRET platform for the analysis of protein-protein interactions. ACS Chem Biol. 10, 1797-1804 (2015).
- Hekman, M., et al. Dynamic changes in C-Raf phosphorylation and 14-3-3 protein binding in response to growth factor stimulation: differential roles of 14-3-3 protein binding sites. J Biol Chem. 279, 14074-14086 (2004).
- Hatzivassiliou, G., et al. RAF inhibitors prime wild-type RAF to activate the MAPK pathway and enhance growth. Nature. 464, 431-435 (2010).
- Bondzi, C., Grant, S., Krystal, G. W. A novel assay for the measurement of Raf-1 kinase activity. Oncogene. 19, 5030-5033 (2000).
- Spencer-Smith, R., et al. Inhibition of RAS function through targeting an allosteric regulatory site. Nat Chem Biol. 13 (1), 62-68 (2016).
- Roy, S., et al. 14-3-3 facilitates Ras-dependent Raf-1 activation in vitro and in vivo. Mol Cell Biol. 18, 3947-3955 (1998).
- Durrant, D. E., et al. Development of a high-throughput NanoBRET screening platform to identify modulators of the RAS/RAF interaction. Mol Cancer Ther. 20, 1743-1754 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten