Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Modelo inmunocompetente de intestino en chip para analizar las respuestas inmunitarias de la mucosa intestinal
* Estos autores han contribuido por igual
En este artículo
Resumen
Nuestro protocolo detallado describe la creación y el uso del modelo avanzado de intestino en chip, que simula la mucosa intestinal humana con estructuras 3D y varios tipos de células, lo que permite un análisis en profundidad de las respuestas inmunitarias y las funciones celulares en respuesta a la colonización microbiana.
Resumen
Se ha desarrollado un modelo avanzado de intestino en chip que recrea estructuras epiteliales organotípicas en 3D, vellosidades y criptas. El modelo inmunocompetente incluye células endoteliales de la vena umbilical humana (HUVEC), células epiteliales intestinales Caco-2, macrófagos residentes en el tejido y células dendríticas, que se autoorganizan dentro del tejido, reflejando las características de la mucosa intestinal humana. Un aspecto único de esta plataforma es su capacidad para integrar células inmunitarias primarias humanas circulantes, lo que mejora la relevancia fisiológica. El modelo está diseñado para investigar la respuesta del sistema inmunitario intestinal a la colonización e infección bacteriana y fúngica. Debido al tamaño de su cavidad ampliada, el modelo ofrece diversas lecturas funcionales, como ensayos de permeación, liberación de citocinas e infiltración de células inmunitarias, y es compatible con la medición de inmunofluorescencia de estructuras 3D formadas por la capa de células epiteliales. Por lo tanto, proporciona información completa sobre la diferenciación y la función celular. La plataforma de intestino en chip ha demostrado su potencial para dilucidar interacciones complejas entre sustitutos de una microbiota viva y tejido huésped humano dentro de una plataforma de biochip microfisiológico perfundido.
Introducción
Los sistemas Organ-on-Chip (OoC) representan una técnica emergente de cultivo celular en 3D que es capaz de cerrar la brecha entre el cultivo celular 2D convencional y los modelos animales. Las plataformas OoC suelen consistir en uno o más compartimentos que contienen células específicas de tejido cultivadas en una amplia gama de andamios, como membranas o hidrogeles1. Los modelos son capaces de imitar una o más funciones organotípicas definidas. Las bombas permiten la perfusión microfluídica continua del medio de cultivo celular para la eliminación de productos de desecho celular, el suministro de factores de nutrición y crecimiento para mejorar la diferenciación celular y la recreación de condiciones esenciales in vivo. Con la integración de células inmunitarias, los sistemas OoC pueden imitar la respuesta inmunitaria humana in vitro2. Hasta la fecha, se ha presentado una amplia gama de órganos y unidades funcionales1. Estos sistemas incluyen modelos de la vasculatura3, el pulmón4, el hígado 2,5 y el intestino6 que pueden facilitarse para las pruebas de drogas 5,7 y los estudios de infección 6,8.
Aquí presentamos un modelo de intestino humano en chip que integra células epiteliales humanas formando una topografía organotípica en 3D de estructuras en forma de vellosidades y criptas combinadas con un revestimiento endotelial y macrófagos residentes en los tejidos. El modelo se cultiva en un biochip perfundido microfluídicamente en formato de portaobjetos microscópico. Cada biochip consta de dos cavidades microfluídicas separadas. Cada cavidad está dividida por una membrana porosa de tereftalato de polietileno (PET) en una cámara superior e inferior. La membrana en sí también sirve como andamio para que las células crezcan a cada lado. Los poros de la membrana permiten la diafonía celular y la migración celular entre las capas celulares. Se puede acceder a cada cámara por dos puertos hembra del tamaño de una cerradura Luer. Opcionalmente, un puerto adicional del tamaño de una cerradura mini-luer puede proporcionar acceso a la cámara superior o inferior (Figura 1).
La plataforma OoC ofrece una serie de lecturas que se pueden obtener de un solo experimento. El intestino en chip está diseñado para combinar el cultivo de células 3D perfundidas, el análisis de efluentes y la microscopía de fluorescencia para evaluar la expresión de marcadores celulares, las tasas de metabolización, la respuesta inmunitaria, la colonización e infección microbiana y la función de barrera 3,6,8. El modelo incluye células inmunitarias residentes en los tejidos y el contacto directo de microorganismos vivos con el tejido del huésped, lo que supone una ventaja en comparación con otros modelos publicados9. Además, las células epiteliales se autoorganizan en estructuras tridimensionales que proporcionan una interfaz fisiológicamente relevante para la colonización con una microbiota viva6.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Este protocolo requiere acceso a ~20 mL de sangre fresca por biochip de donantes sanos para aislar monocitos humanos primarios. Todos los donantes dieron su consentimiento informado por escrito para participar en este estudio, que fue aprobado por el comité de ética del Hospital Universitario de Jena (número de permiso 2018-1052-BO). Para obtener más información sobre los materiales, consulte la Tabla de materiales. Para obtener detalles sobre la composición de todas las soluciones y medios, consulte la Tabla 1.
1. Observaciones generales sobre el manejo de biochips
- Separe con cuidado una tira de depósitos y separe las tapas con un cuchillo caliente para obtener depósitos y tapas individuales. Ensancha el orificio de la tapa para que el tubo de silicona quede bien ajustado.
- El tubo de silicona tiene un diámetro interior de 0,5 mm, es asimétrico y está separado en lados más largos (20 cm) y más cortos (12 cm) por dos tapones peristálticos de la bomba. Ensamble dos tubos de cada simetría por biochip mediante la conexión de un tubo a un conector de bloqueo Luer macho y la tapa en el lado opuesto del tubo. También ensamble cuatro depósitos por biochip.
NOTA: Prepare los tubos y los depósitos con anticipación y esterilícelos en autoclave antes de usarlos. Como el tubo de silicona tiene una vida útil limitada, cámbielo después de 3 a 5 experimentos. Para ciertos intereses de investigación, como las pruebas de medicamentos, se recomienda preparar nuevos tubos para cada experimento. Muchos pasos de este protocolo se ejecutan en paralelo; consulte la figura de descripción general, que destaca los diferentes pasos realizados en un día, como se muestra en la Figura 2.
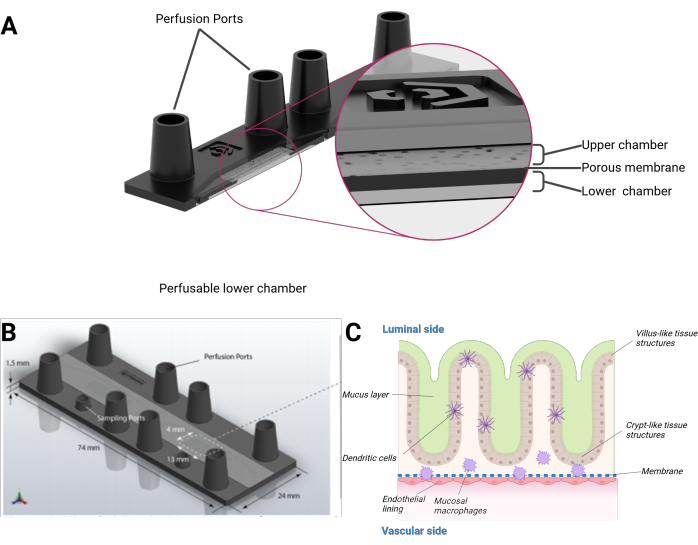
Figura 1: Representación esquemática del modelo de intestino en chip. (A) El biochip se presenta en una vista transversal. (B) La dimensión de todo el biochip, así como de la membrana de PET plana y extraíble, es visible. El volumen total de la cámara superior, incluidos los puertos del tamaño de la cerradura Luer hembra, es de 290 μL y 270 μL para la cámara inferior, respectivamente. (C) Se puede ver una composición esquemática del biochip intestinal, el epitelio de crecimiento tridimensional que se asemeja a estructuras en forma de vellosidades y criptas que incluyen células inmunitarias diferenciadas y una capa de moco. El otro lado de la membrana PET está cubierto por una monocapa endotelial. Haga clic aquí para ver una versión más grande de esta figura.
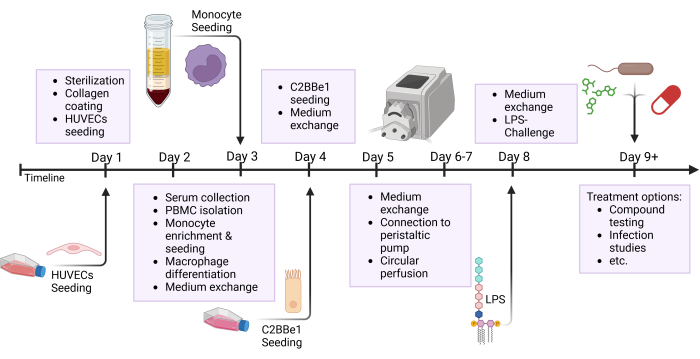
Figura 2: Resumen esquemático de la línea de tiempo de construcción del modelo y la configuración experimental. Esta figura muestra la descripción general esquemática del protocolo presentado. Los procedimientos importantes, como la siembra de células y el desafío epitelial con LPS, se indican con flechas. Abreviaturas: HUVECs = células endoteliales venosas umbilicales humanas; LPS = lipopolisacárido. Haga clic aquí para ver una versión más grande de esta figura.
2. Esterilización de biochips
- Llene una placa de Petri de vidrio estéril, que posea un diámetro de 15 cm, con un 70% de etanol sin desnaturalizar. Coloque el biochip en el interior de modo que todos los puertos del biochip queden completamente cubiertos por la solución de etanol.
- Dos veces por puerto, extraiga 1 ml de etanol al 70% a través de todas las cámaras del chip. Incubar durante 45-60 min a temperatura ambiente (RT).
NOTA: Asegúrese de que no quede aire atrapado dentro del biochip. A partir de este momento, no debe entrar aire en el sistema de biochip y las cavidades deben permanecer llenas de líquido. - Retire el etanol de la placa de Petri y reemplácelo con agua estéril de doble destilación (ddH2O) hasta que todos los puertos estén completamente cubiertos. De nuevo, dos veces por puerto, extraiga 1 mL deddH2O a través de la cavidad del biochip. Actualice el ddH2O en la placa de Petri y repita el procedimiento.
- Retire todo el líquido de la placa de Petri. A partir de ahora, mantenga los biochips completamente esterilizados dentro de la placa de Petri cerrada siempre que estén fuera de un entorno estéril. Agregue un pequeño depósito (por ejemplo, la tapa de un tubo de 50 ml) de 2-5 mL ddH2O a la placa de Petri para reducir la evaporación del líquido dentro del biochip.
NOTA: Los biochips se pueden esterilizar con hasta 3 días de anticipación si se mantienen en un ambiente estéril hasta su uso. Esto permite flexibilidad en la carga de trabajo en un solo día.
3. Cosecha y siembra de HUVECs
NOTA: Las células endoteliales venosas umbilicales humanas (HUVECs) fueron aisladas de cordones umbilicales como se publicó anteriormente10.
- Antes de sembrar los HUVEC, cubra la membrana con colágeno humano IV. Para ello, prepare una dilución 1:100 de una solución madre de colágeno (Tabla 1) en solución salina tamponada con fosfato de Dulbecco que contiene magnesio y calcio (PBS +/+). Añada 350 μL de la solución madre diluida a la cámara correspondiente. Incubar durante 5 min en RT.
NOTA: Si manipula el biochip en la campana estéril, le recomendamos que coloque un pañuelo estéril debajo para recoger el exceso de medio. - Enjuague todas las cámaras dos veces con 350 μL de PBS +/+ para lavar el colágeno y el ácido acético restantes. A continuación, añada 350 μL de medio de crecimiento de células endoteliales (EC) a cada cámara.
NOTA: A partir de aquí, los biochips están listos para la siembra de células y se pueden almacenar a 37 °C hasta su uso. - Utilice HUVEC en los pasajes 1-3 con un 80-90% de confluencia celular. Cultive HUVEC en medio EC que contenga una mezcla de suplementos definida proporcionada por el fabricante. En la Figura 3A se presenta una imagen representativa de campo claro de un cultivo celular HUVEC.
NOTA: Dependiendo del donante, los HUVEC de los pasajes superiores pueden comenzar a desdiferenciarse y no pueden formar de manera confiable una monocapa densa y confluente dentro del biochip. El uso de antibióticos, es decir, 100 U/mL de penicilina y 100 μg/mL de estreptomicina, es opcional, pero se recomienda como complemento del medio CE para prevenir la contaminación microbiana. - Retire el medio de cultivo celular de un matraz de cultivo celular T25 y lave las células suavemente con 3-5 mL de solución salina tamponada con fosfato de Dulbecco sin magnesio ni calcio (PBS -/-). Retire el PBS -/- y agregue 1 mL de reactivo de disociación de tripsina (Tabla 1). Incubar durante 5 min a 37 °C hasta que las células se desprendan del matraz de cultivo celular.
- Transfiera las células desprendidas a un tubo utilizando 9 mL de suero fetal bovino (FBS) al 5% en PBS -/-. Centrifugar a 350 × g durante 5 min a RT. Retirar el sobrenadante, resuspender en 1 mL de medio EC y determinar el número de células. Ajuste la concentración de células a 0,4 × 106 células por 150 μL (siembra en la cámara inferior) o por 250 μL (siembra en la cámara superior).
- Agregue el volumen respectivo de celdas a la cámara. Si siembra en la cámara inferior, cierre todos los puertos e inmediatamente coloque el biochip boca abajo para que las células caigan sobre la membrana PET. Incubar los biochips en una incubadora humidificada a 37 °C y 5% deCO2.
- Realice un cambio de medio de la cámara que contiene HUVEC con 350 μL de medio EC después de 24 h. El medio en la cámara opuesta no necesita ser reemplazado.
4. Recolección de suero humano y aislamiento de monocitos derivados de células mononucleares de sangre periférica (PBMC)
NOTA: Las PBMC se aislaron como se describe en Mosig et al.11.
- Extraer sangre venosa humana de donantes sanos. Por biochip, obtenga un mínimo de 10 ml de sangre completa en tubos de recolección de sangre que contengan silicato para la recolección de suero. Después de la coagulación completa, centrifugar los tubos de extracción de sangre a 2.500 × g durante 10 min en RT. Recoja el suero, la alícuota y almacene a -20 °C hasta su uso posterior.
- Obtener un mínimo de 10 mL de sangre entera del mismo donante en tubos de recolección de sangre que contengan EDTA para el aislamiento de PBMC. Mezcle suavemente la sangre no coagulada 1:1 con iso-tampón (Tabla 1) por inversión y coloque lentamente 35 mL de esta mezcla sobre 15 mL de un medio de gradiente de densidad con una densidad de 1.077 g/mL en un tubo de 50 mL.
- Centrifugar a 800 × g durante 20 min sin freno en RT. Retire con cuidado la capa de células inmunitarias resultante, que aparece en la parte superior del medio de gradiente de densidad, y transfiérala a un nuevo tubo de 50 mL. Llene hasta 50 ml con iso-buffer frío y lave las células mediante una inversión suave.
- Centrifugar a 200 × g durante 8 min sin freno a 4 °C. Deseche el sobrenadante y vuelva a suspender en 10 mL de iso-tampón por gradiente de densidad. Opcional: Agrupe las PBMC de un donante si se ejecutan varios gradientes en paralelo.
- Centrifugar a 150 × g durante 8 min a 4 °C. Deseche el sobrenadante y vuelva a suspender el pellet en 10 mL de isotampón por gradiente de densidad. Repita el paso de centrifugación 4.4. Finalmente, desechar el sobrenadante y resuspender las células en 2 mL de medio de diferenciación de monocitos (Tabla 1).
NOTA: La adición de M-CSF y GM-CSF refuerza la diferenciación de los monocitos aislados hacia macrófagos derivados de monocitos y células dendríticas derivadas de monocitos (en combinación con lipopolisacárido [LPS], que se agrega en un punto posterior de este protocolo). El uso de antibióticos, es decir, 100 U/mL de penicilina y 100 μg/mL de estreptomicina, es opcional, pero se recomienda como complemento del medio para prevenir la contaminación microbiana. - Determine el número de células y la semilla ~ 10 ×10 6 células por pocillo de una placa de 6 pocillos en 2 mL de medio de diferenciación de monocitos (Tabla 1). Incubar en una incubadora humidificada a 37 °C durante 1 h para permitir la fijación de los monocitos al plástico de la placa de 6 pocillos.
- Deseche cuidadosamente el sobrenadante y lávese 2 veces con 2 mL precalentados de medio celular hematopoyético para eliminar las células no unidas. Incubar a 37 °C durante otras 24 h en medio de diferenciación de monocitos.
- Para recolectar los monocitos, deseche cuidadosamente el sobrenadante y lávese una vez con 2 mL de PBS precalentado -/-. Véase la Figura 3B para ver un ejemplo de una imagen de campo claro del cultivo de monocitos en este punto. A continuación, incubar las células durante 7 min en 1 mL de reactivo de desprendimiento de monocitos precalentado (Tabla 1) a 37 °C para forzar el desprendimiento de los monocitos del plástico de la placa de 6 pocillos.
- Transfiera los monocitos desprendidos a un tubo de baja fijación. Opcional: para lograr un mayor rendimiento celular, lave cuidadosamente la placa de 6 pocillos varias veces con PBS -/-.
- Centrifugar a 300 × g durante 8 min a RT. Deseche el sobrenadante y vuelva a suspender en medio acondicionado por la CE (Tabla 1). Determine el número de células y ajuste la concentración de células a 0,1 × 106 células por 150 μL (siembra en la cavidad inferior) o por 250 μL (siembra en la cavidad superior).
NOTA: Sea cuidadoso en todos los pasos del aislamiento de las células inmunitarias y reduzca las fuerzas de cizallamiento para evitar la activación de las células inmunitarias. Al establecer este aislamiento, verifique la pureza de los monocitos derivados de PBMC (por ejemplo, mediante citometría de flujo). Más del 95% de todas las células deben ser positivas para marcadores típicos de monocitos, como CD14.
5. Siembra de monocitos
- Realice un intercambio de medio en la cámara que contiene HUVEC con 350 μL de medio acondicionado CE precalentado.
- Añada 150 μL (cámara inferior) o 250 μL (cámara superior) de la suspensión de monocitos preparada (ver paso 4.10) a la misma cámara. Si se siembra en la cámara inferior, cierre todos los puertos e inmediatamente coloque el biochip boca abajo para que las células caigan sobre la capa HUVEC. Incubar el biochip en una incubadora humidificada a 37 °C y 5% deCO2.
- Realizar un intercambio de medio en la cámara que contiene monocitos HUVEC + con 350 μL de medio acondicionado CE cada 24 h.
6. Cosecha y siembra de C2BBe1
NOTA: El borde del cepillo Caco-2 que expresa las celdas 1 (C2BBe1)12 se utiliza hasta el paso 35 y se toma de matraces con un 80-90% de confluencia. En la Figura 3C se presenta una imagen representativa de campo claro de un cultivo C2BBe1.
- Cultivar células C2BBe1 en medio C2 (Tabla 1).
NOTA: El uso de antibióticos, es decir, 20 μg/mL de gentamicina, es opcional, pero se recomienda como suplemento del medio C2 para prevenir la contaminación microbiana. - Retire el medio de cultivo celular de un matraz de cultivo celular T25 y lave las células suavemente con 3-5 mL de PBS -/-. Retire el PBS -/- y agregue 1 mL de reactivo de disociación de tripsina (Tabla 1). Incubar durante 5 min a 37 °C hasta que las células se desprendan del matraz de cultivo celular.
- Transfiera las células desprendidas a un tubo utilizando 9 mL de suero fetal bovino (FBS) al 5% en PBS -/-. Centrifugar a 350 × g durante 5 min en RT. Retirar el sobrenadante, resuspender en 1 mL de medio C2 y determinar el número de células. Ajuste la concentración de células a 0,5 × 106 células por 150 μL (siembra en la cámara inferior) o por 250 μL (siembra en la cámara superior).
- Antes de sembrar el C2BBe1, lave suavemente la cámara respectiva con 350 μL de medio C2.
- Añada 150 μL (cámara inferior) o 250 μL (cámara superior) de la suspensión C2BBe1 preparada (consulte el paso 6.3) a la cámara correspondiente. Si siembra en la cámara inferior, cierre todos los puertos e inmediatamente coloque el biochip boca abajo para que las células caigan sobre la membrana PET. Incubar los biochips en una incubadora humidificada a 37 °C y 5% deCO2.

Figura 3: Morfología celular de HUVECs, monocitos y C2BBe1 antes de la siembra en el biochip. Esta figura muestra imágenes representativas de campo claro de las diferentes fuentes celulares utilizadas en todo el protocolo. Las imágenes se tomaron con un microscopio de campo claro inverso con un aumento de 10x. Todos los tipos de células, (A) HUVECs, (B) monocitos y (C) C2BBe1 se cultivaron en cultivo celular monocapa 2D como se describe en sus secciones específicas del protocolo. Barras de escala = 200 μm. Abreviaturas: HUVECs = células endoteliales venosas umbilicales humanas; PBMC = células mononucleares de sangre periférica. Haga clic aquí para ver una versión más grande de esta figura.
7. Conexión a bomba peristáltica y perfusión circular
- Prepare una incubadora vacía con la adición de una bomba peristáltica. Limpie a fondo todas las áreas de la incubadora y la bomba con desinfectante para proporcionar un entorno casi estéril.
NOTA: Las bombas peristálticas pueden producir mucho calor mientras funcionan. En incubadoras bien aisladas o laboratorios con aire acondicionado deficiente, el número de bombas utilizables por incubadora puede ser limitado, ya que las incubadoras tienden a sobrecalentarse. Dos bombas peristálticas por incubadora deberían ser suficientes. - Antes de conectar los tubos esterilizados al biochip, enjuague cada tubo con 700 μL de PBS +/+ seguido de 500 μL de medio C2 o medio acondicionado con CE. Prepare un tubo de cada simetría por medio (consulte el paso 1.2). Utilice el tubo con la distancia corta desde el bloqueo luer hasta el tapón de la bomba peristáltica para la cavidad izquierda y el tubo con la otra simetría para la cavidad derecha.
- Recupere el biochip de la incubadora y realice un intercambio de medio con 350 μL para cada cámara. Retire todos los enchufes y llene todos los puertos hasta la parte superior.
- Comenzando en la cavidad izquierda, conecte el primer tubo al puerto derecho de la cámara superior insertando el adaptador Luer Lock en el puerto del biochip. Luego, conecte el segundo tubo al puerto izquierdo de la cámara inferior. Repita este procedimiento para la cavidad microfluídica correcta.
- Tome un depósito y agregue una pequeña gota de medio de cultivo celular al fondo del depósito. Luego, inserte el depósito en el lado opuesto del primer tubo y repita para la otra cámara. Una vez que todos los puertos estén conectados a un tubo o depósito, llene los depósitos con 3,5 ml de medio de cultivo celular.
- Coloque el lado suelto del tubo, que tiene la tapa adjunta, en la parte superior del depósito para cerrar el sistema microfluídico de cada cámara. En este estado, transporte el biochip a la bomba peristáltica.
NOTA: Dependiendo de la distancia a la incubadora y del entorno del laboratorio, se puede utilizar una caja previamente limpia y esterilizada en autoclave para transferir las fichas a la incubadora. - Utilice los tapones peristálticos de la bomba para conectar el tubo a la bomba. Conecte cada tubería a la bomba peristáltica de tal manera que el medio fluya desde el depósito hacia la cavidad, hacia la tubería y, a través de la bomba, de regreso al depósito (Figura 4). El depósito sirve como trampa de burbujas en la perfusión circular y evita que el aire quede atrapado en el sistema. Perfundir cada cámara con un caudal de 50 μL/min, lo que da como resultado un esfuerzo cortante de 0,013 dyn/cm2 en la cámara superior y de 0,006 dyn/cm2 en la cámara inferior8.
NOTA: Si el medio de las cavidades inferior y superior se mueve en direcciones opuestas, se puede lograr un mayor crecimiento tridimensional del tejido intestinal13. Por lo tanto, los depósitos de las cavidades superior e inferior se colocan en lados opuestos (Figura 4). La perfusión circular reduce la cantidad de medio de cultivo celular necesario, pero podría resultar en el enriquecimiento de citocinas y metabolitos. Si se desea, también es posible una perfusión lineal del biochip. - Perfundir el biochip durante 72 h a 37 °C y 5% de CO2.

Figura 4: Biochip conectado a la bomba peristáltica. Se presenta un ejemplo de un biochip conectado a una bomba peristáltica. Las células epiteliales C2BBe1 se cultivan en la cámara inferior (el medio C2 rojo se encuentra en los reservorios de la parte delantera), mientras que las HUVEC se cultivan en la cámara superior (el medio acondicionado de EC amarillento se encuentra en los reservorios de la parte posterior). Los diferentes medios de cultivo celular no se mezclan debido a la función de barrera del tejido cultivado. El biochip está conectado a la bomba peristáltica de tal manera que el medio fluye desde el depósito hasta la cavidad. Desde aquí, el medio fluye de regreso al depósito a través de la tubería a través de la bomba. Haga clic aquí para ver una versión más grande de esta figura.
8. Acondicionamiento de LPS de la barrera epitelial
- Después de 72 h de preperfusión, detenga la bomba peristáltica y retire la tapa conectada al tubo de cada depósito. Colóquelo sobre un pañuelo de papel estéril junto a la bomba.
- Retire todo el medio y llene los depósitos con 2 ml de medio recién preparado. Para el lado epitelial que contiene las células C2BBe1, agregue 100 ng/mL de LPS al medio.
NOTA: El LPS aumenta la función de barrera del tejido, estimula a los macrófagos derivados de monocitos para que migren al tejido epitelial y permite la diferenciación de células dendríticas derivadas de monocitos. - Vuelva a conectar el tubo y las tapas al depósito y continúe la perfusión circular a un caudal de 50 μL/min durante 24 h adicionales.
NOTA: A partir de este momento, el modelo de chip se puede utilizar en experimentos, pruebas de compuestos o estudios de infección. Recomendamos un cambio medio de 2 mL por depósito cada 24 h.
9. Acceso al tejido para diferentes métodos de lectura
- Recoger los sobrenadantes del medio de cultivo celular de los depósitos en todo momento de la perfusión. Abra el depósito y recoja el volumen deseado (consulte los pasos 8.1-8.3). Utilice estos sobrenadantes para la detección de metabolitos, citocinas u otras moléculas.
- Para acceder al tejido, utilice un bisturí para hacer un corte preciso a lo largo del exterior de la cámara superior y retire la lámina de unión para abrir la cavidad microfluídica. El tejido del modelo de biochip intestinal ya es accesible. Corta con cuidado a lo largo del exterior de la cámara microfluídica para separar la membrana del biochip. Recoja la membrana que contiene el tejido con pinzas.
PRECAUCIÓN: Tenga en cuenta la colocación de los dedos durante este paso y trabaje con cuidado para evitar accidentes. Se recomienda el uso de guantes resistentes a los cortes. - Alternativamente, recoja las células de capas separadas dentro del biochip usando soluciones enzimáticas, es decir, tripsina o células lisadas usando tampones que contienen Triton X-100.
10. Evaluación de la permeabilidad mediante difusión de dextrano FITC
NOTA: La función de barrera del tejido se puede analizar mediante un ensayo de permeabilidad al dextrano FITC después de la desconexión de la bomba peristáltica. La evaluación de la permeabilidad al dextrano FITC fue adaptada de Deinhardt-Emmer et al.4.
- Prepare una solución madre de isotiocianato de fluoresceína (FITC)-dextrano (peso molecular de 3-5 kDa, Tabla 1).
- Vacíe los depósitos y desconecte el chip de la perfusión.
- Realice un intercambio de medio en la cámara superior e inferior con medio libre de rojo fenol.
NOTA: Este paso no es necesario si ya se utilizó un medio libre de rojo de fenol durante el experimento. - Añadir 350 μL de 1 mg/mL de solución de FITC-dextrano a la cámara que contiene las células C2BBe1.
- Cierre los puertos e incube el chip durante 60 minutos a 37 °C con el lado epitelial hacia arriba.
- Después del tiempo de incubación, recoja el medio de cultivo de ambas cámaras del chip por separado y guárdelo a 4 °C, protegido de la luz hasta la medición.
- Para la medición, prepare una curva estándar en medio C2 y en medio acondicionado CE sin rojo de fenol en el rango de 1.000 μg/mL a 0 μg/mL de FITC-dextrano con 11 diluciones seriadas consecutivas 1:2.
- Transfiera 200 μL de cada muestra a una placa negra de 96 pocillos con un fondo transparente. Mida la fluorescencia con un lector de microplacas a una longitud de onda de excitación de 495 nm y una longitud de onda de emisión de 517 nm.
- Utilice la curva estándar para calcular la concentración de dextrano FITC de las muestras y, por lo tanto, el coeficiente de permeabilidad.
11. Tinción de inmunofluorescencia
NOTA: El tejido vivo se puede investigar microscópicamente. Para facilitar el manejo, recomendamos el desprendimiento del biochip de la bomba peristáltica y el uso de objetivos de larga distancia en un microscopio invertido. Como análisis de punto final, el tejido se puede fijar dentro del biochip para procedimientos como la tinción de inmunofluorescencia.
- Detenga la bomba peristáltica y abra los depósitos de todas las cavidades. Vacíe los depósitos y desconecte los tubos, así como los depósitos del biochip.
- Dos veces por cámara, lavar las cavidades microfluídicas con 500 μL de PBS frío +/+. Añadir 500 μL de metanol helado a todas las cavidades e incubar durante 15 min a -20 °C. A continuación, dos veces por cavidad, lavar la cámara microfluídica con 500 μL de PBS+/+.
NOTA: Otros métodos de fijación, como la fijación con paraformaldehído al 4% o el fijador de Carnoy, también son adecuados. Después de la fijación, los chips pueden almacenarse a 4 °C o proceder directamente a la tinción con fluorescencia inmune. PRECAUCIÓN: Los productos químicos fijadores como el metanol o el paraformaldehído son tóxicos. Realice las tareas respectivas bajo una campana extractora y recoja los residuos en consecuencia. - Abra el chip como se describe en el paso 9.2 para acceder al tejido. Cortar la membrana PET que contiene tejido en hasta tres pedazos para teñir en paralelo con diferentes inmunopaneles.
- Transfiera cada una de las piezas de la membrana a una placa separada de 24 pocillos que contenga una solución de bloqueo y permeabilización (Tabla 1) utilizando pinzas de precisión. Asegúrese de que la capa celular de interés siempre mire hacia arriba durante todo el proceso de tinción. Incubar las piezas de membrana durante 30 min en RT.
NOTA: Los mejores resultados de tinción se obtienen cuando el suero coincide con el anticuerpo secundario. Por ejemplo, si se obtienen anticuerpos secundarios de especies de cabras, recomendamos el uso de suero de cabra normal. - Transfiera las piezas de la membrana a un portaobjetos de vidrio limpio dentro de una cámara húmeda. Prepare el panel de anticuerpos primarios en la solución de tinción (Tabla 1) y agregue 50 μL a cada pieza de membrana. Incubar durante la noche a 4 °C.
NOTA: La concentración óptima de anticuerpos y la eficacia de la tinción pueden diferir entre los fabricantes y los clones. Recomendamos probar previamente los paneles de tinción en un cultivo celular 2D. - Después de la incubación, transfiera las muestras a una placa de 24 pocillos y lave suavemente las membranas durante 3 x 5 min con solución de lavado (Tabla 1).
- Nuevamente, transfiera las piezas de la membrana a un portaobjetos de vidrio limpio dentro de una cámara húmeda. Preparar el panel de anticuerpos secundarios en solución de tinción (Tabla 1) y añadir 50 μL a cada pieza de membrana. Si es necesario, agregue una contratinción nuclear como 4',6-diamidino-2-fenilindol (DAPI) o Hoechst. Incubar durante 30 min en RT.
NOTA: Mientras trabaja con fluoróforos, mantenga las muestras protegidas de la luz para evitar el fotoblanqueo y aumentar la calidad de la imagen. - Después de la incubación, transfiera las muestras a una placa de 24 pocillos y lave suavemente las membranas 2 x 5 min con solución de lavado (Tabla 1). Luego, lavar una vez con PBS +/+ durante 5 min.
- Monte las piezas de membrana en un portaobjetos de vidrio limpio utilizando un medio de montaje de fluorescencia y un cubreobjetos. Almacenar a 4 °C hasta obtener imágenes microscópicas.
Access restricted. Please log in or start a trial to view this content.
Resultados
Estos resultados representativos muestran las distintas capas de tejido del modelo de intestino en chip. Se tiñen con inmunofluorescencia como se describe en la sección 11 del protocolo. Las imágenes se tomaron con un microscopio de epifluorescencia o fluorescencia confocal como pilas z y se procesaron en una proyección ortogonal. Consulte la Tabla de materiales para obtener detalles sobre la configuración microscópica y el software. La figura 5 muestra la capa vascula...
Access restricted. Please log in or start a trial to view this content.
Discusión
El protocolo presentado detalla los pasos necesarios para generar un modelo de intestino en chip inmunocompetente. Describimos técnicas específicas y posibles métodos de lectura, como la microscopía de inmunofluorescencia, el análisis de citocinas y metabolitos, la citometría de flujo, el análisis genético y de proteínas y la medición de la permeabilidad.
El modelo descrito consiste en HUVECs primarios, macrófagos derivados de monocitos y células dendríticas derivadas de monocitos...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
M.R. es director general de Dynamic42 GmbH y posee acciones en la empresa. A.S.M. es asesor científico de Dynamic 42 GmbH y posee acciones en la empresa.
Agradecimientos
El trabajo contó con el apoyo financiero del Centro de Investigación Colaborativa PolyTarget 1278 (proyecto número 316213987) para que V.D.W. y A.S.M. A.F. y A.S.M. reconozcan además el apoyo financiero del Clúster de Excelencia "Balance of the Microverse" en el marco de la Estrategia de Excelencia de Alemania - EXC 2051 - Project-ID 690 390713860. Queremos agradecer a Astrid Tannert y al Laboratorio de Biofotónica e Imagen de Jena (JBIL) por proporcionarnos acceso a su microscopio de barrido láser confocal ZEISS LSM980. La Figura 1C y la Figura 2 se crearon con Biorender.com.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 96-well plate black, clear bottom | Thermo Fisher | 10000631 | Consumables |
| Acetic acid | Roth | 3738.4 | Chemicals |
| Alexa Fluor 488 AffiniPure, donkey, anti-mouse IgG (H+L) | Jackson Immuno Research | 715-545-150 | Secondary Antibody Vascular Staining and Epithelial Staining |
| Alexa Fluor 647 AffiniPure, donkey, anti-rabbit IgG (H+L) | Jackson Immuno Research | 711-605-152 | Secondary Antibody Epithelial Staining |
| Alexa Fluor 647, donkey, anti-rabbit IgG (H+L) | Thermo Fisher Scientific, Invitrogen | A31573 | Secondary Antibody Vascular Staining |
| Axiocam ERc5s camera | Zeiss | 426540-9901-000 | Technical equipment |
| Basal Medium MV, phenol red-free | Promocell | C-22225 | Cell culture consumables |
| Biochip | Dynamic 42 | BC002 | Microfluidic consumables |
| BSA fraction V | Gibco | 15260-037 | Cell culture consumables |
| C2BBe1 (clone of Caco-2) | ATCC | CRL-2102 | Epithelial Cell Source |
| Chloroform | Sigma | C2432 | Chemicals |
| CO2 Incubator | Heracell | 150i | Technical equipment |
| Collagen IV from human placenta | Sigma-Aldrich | C5533 | Cell culture consumables |
| Coverslips (24 x 40 mm; #1.5) | Menzel-Gläser | 15747592 | Consumables |
| Cy3 AffiniPure, donkey, anti-goat IgG (H+L) | Jackson Immuno Research | 705-165-147 | Secondary Antibody Vascular Staining |
| Cy3 AffiniPure, donkey, anti-rat IgG (H+L) | Jackson Immuno Research | 712-165-150 | Secondary Antibody Epithelial Staining |
| Descosept PUR | Dr.Schuhmacher | 00-323-100 | Cell culture consumables |
| DMEM high glucose | Gibco | 41965-062 | Cell culture consumables |
| DMEM high glucose w/o phenol red | Gibco | 31053028 | Cell culture consumables |
| DPBS (-/-) | Gibco | 14190-169 | Cell culture consumables |
| DPBS (+/+) | Gibco | 14040-133 | Cell culture consumables |
| EDTA solution | Invitrogen | 15575-038 | Cell culture consumables |
| Endothelial Cell Growth Medium | Promocell | C-22020 | Cell culture consumables |
| Endothelial Cell Growth Medium supplement mix | Promocell | C-39225 | Cell culture consumables |
| Ethanol 96%, undenatured | Nordbrand-Nordhausen | 410 | Chemicals |
| Fetal bovine Serum | invitrogen | 10270106 | Cell culture consumables |
| Fluorescein isothiocyanate (FITC)-dextran (3-5 kDa) | Sigma Aldrich | FD4-100MG | Chemicals |
| Fluorescent Mounting Medium | Dako | S3023 | Chemicals |
| Gentamycin (10mg/mL) | Sigma Aldrich | G1272 | Cell culture consumables |
| GlutaMAX Supplement (100x) | Gibco | 35050061 | Cell culture consumables |
| Histopaque | Sigma-Aldrich | 10771 | Cell culture consumables |
| Hoechst (bisBenzimid) H33342 | Sigma-Aldrich | 14533 | Epithelial Staining |
| Holotransferrin (5mg/mL) Transferrin, Holo, Human Plasma | Millipore | 616397 | Cell culture consumables |
| Human recombinant GM-CSF | Peprotech | 300-30 | Cell culture consumables |
| Human recombinant M-CSF | Peprotech | 300-25 | Cell culture consumables |
| Illumination device | Zeiss | HXP 120 C | Fluorescence Microscope Setup |
| Laser Scanning Microscope | Zeiss | CLSM980 | Fluorescence Microscope Setup |
| Lidocain hydrochloride | Sigma-Aldrich | L5647 | Cell culture consumables |
| Lipopolysaccharide (LPS) | Sigma | L2630 | Cell culture consumables |
| Loftex Wipes | Loftex | 1250115 | Consumables |
| Low attachment tubes (PS, 5 mL) | Falcon | 352052 | Consumables |
| Luer adapter for the top cap (M) | Mo Bi Tec | M3003 | Microfluidic consumables |
| Male mini luer plugs, row of four,PP, opaque | Microfluidic chipshop | 09-0556-0336-09 | Microfluidic consumables |
| MEM Non-Essential Amino Acids Solution | Gibco | 11140 | Cell culture consumables |
| Methanol | Roth | 8388.2 | Chemicals |
| Microscope | Zeiss | Axio Observer 5 | Fluorescence Microscope Setup |
| Microscope slides | Menzel | MZ-0002 | Consumables |
| Monoclonal, mouse, anti-human CD68 Antibody (KP1) | Thermo Fisher Scientific, Invitrogen | 14-0688-82 | Primary Antibody Vascular Staining |
| Monoclonal, rat, anti-human E-Cadherin antibody (DECMA-1) | Sigma-Aldrich, Millipore | MABT26 | Primary Antibody Epithelial Staining |
| Multiskan Go plate reader | Thermo Fisher | 51119300 | Technical equipment |
| Normal donkey serum | Biozol | LIN-END9010-10 | Chemicals |
| Optical Sectioning | Zeiss | ApoTome | Fluorescence Microscope Setup |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140-122 | Cell culture consumables |
| Plugs | Cole Parmer | GZ-45555-56 | Microfluidic consumables |
| Polyclonal, goat, anti-human VE-Cadherin Antibody | R&D Systems | AF938 | Primary Antibody Vascular Staining |
| Polyclonal, rabbit, anti-human Von Willebrand Factor Antibody | Dako | A0082 | Primary Antibody Vascular Staining |
| Polyclonal, rabbit, anti-human ZO-1 antibody | Thermo Fisher Scientific, Invitrogen | 61-7300 | Primary Antibody Epithelial Staining |
| Power Supply Microscope | Zeiss | Eplax Vp232 | Fluorescence Microscope Setup |
| Primovert microscope | Zeiss | 415510-1101-000 | Technical equipment |
| Reglo ICC peristaltic pump | Ismatec | ISM4412 | Technical equipment |
| SAHA (Vorinostat) | Sigma Aldrich | SML0061-25MG | Chemicals |
| Saponin | Fluka | 47036 | Chemicals |
| S-Monovette, 7.5 mL Z-Gel | Sarstedt | 01.1602 | Consumables |
| S-Monovette, 9.0 mL K3E | Sarstedt | 02.1066.001 | Consumables |
| Sodium Pyruvate | Gibco | 11360-088 | Cell culture consumables |
| Tank 4.5 mL | ChipShop | 10000079 | Microfluidic consumables |
| Trypane blue stain 0.4% | Invitrogen | T10282 | Cell culture consumables |
| Trypsin | Gibco | 11538876 | Cell culture consumables |
| Tubing | Dynamic 42 | ST001 | Microfluidic consumables |
| Tweezers (Präzisionspinzette DUMONT abgewinkelt Inox08, 5/45, 0,06 mm) | Roth | K343.1 | Consumables |
| Wheat Germ Agglutinin (WGA) | Thermo Fisher Scientific, Invitrogen | W32464 | Epithelial Staining |
| X-VIVO 15 | Lonza | BE02-060F | Cell culture consumables, Hematopoietic cell medium |
| Zellkultur Multiwell Platten, 24 Well, sterile | Greiner Bio-One | 662 160 | Consumables |
| Zellkultur Multiwell Platten, 6 Well, sterile | Greiner Bio-One | 657 160 | Consumables |
| Zen Blue Software | Zeiss | Version 3.7 | Microscopy Software |
Referencias
- Alonso-Roman, R., et al. Organ-on-chip models for infectious disease research. Nat Microbiol. 9 (4), 891-904 (2024).
- Fahrner, R., Groger, M., Settmacher, U., Mosig, A. S. Functional integration of natural killer cells in a microfluidically perfused liver on-a-chip model. BMC Res Notes. 16 (1), 285(2023).
- Raasch, M., et al. Microfluidically supported biochip design for culture of endothelial cell layers with improved perfusion conditions. Biofabrication. 7 (1), 015013(2015).
- Deinhardt-Emmer, S., et al. Co-infection with Staphylococcus aureus after primary influenza virus infection leads to damage of the endothelium in a human alveolus-on-a-chip model. Biofabrication. 12 (2), 025012(2020).
- Kaden, T., et al. Generation & characterization of expandable human liver sinusoidal endothelial cells and their application to assess hepatotoxicity in an advanced in vitro liver model. Toxicology. 483, 153374(2023).
- Maurer, M., et al. A three-dimensional immunocompetent intestine-on-chip model as in vitro platform for functional and microbial interaction studies. Biomaterials. 220, 119396(2019).
- Hoang, T. N. M., et al. Invasive aspergillosis-on-chip: A quantitative treatment study of human aspergillus fumigatus infection. Biomaterials. 283, 121420(2022).
- Kaden, T., et al. Modeling of intravenous caspofungin administration using an intestine-on-chip reveals altered Candida albicans microcolonies and pathogenicity. Biomaterials. 307, 122525(2024).
- Shah, P., et al. A microfluidics-based in vitro model of the gastrointestinal human-microbe interface. Nat Commun. 7, 11535(2016).
- Jaffe, E. A., Nachman, R. L., Becker, C. G., Minick, C. R. Culture of human endothelial cells derived from umbilical veins. Identification by morphologic and immunologic criteria. J Clin Invest. 52 (11), 2745-2756 (1973).
- Mosig, S., et al. Different functions of monocyte subsets in familial hypercholesterolemia: Potential function of cd14+ cd16+ monocytes in detoxification of oxidized ldl. FASEB J. 23 (3), 866-874 (2009).
- Peterson, M., Mooseker, M. Characterization of the enterocyte-like brush border cytoskeieton of the c2bbe clones of the human intestinal cell line, caco-2. J Cell Sci. 102, Pt 3 581-600 (1992).
- Shin, W., Hinojosa, C. D., Ingber, D. E., Kim, H. J. Human intestinal morphogenesis controlled by transepithelial morphogen gradient and flow-dependent physical cues in a microengineered gut-on-a-chip. iScience. 15, 391-406 (2019).
- Kim, H. J., Ingber, D. E. Gut-on-a-chip microenvironment induces human intestinal cells to undergo villus differentiation. Integr Biol (Camb). 5 (9), 1130-1140 (2013).
- Kim, H. J., Huh, D., Hamilton, G., Ingber, D. E. Human gut-on-a-chip inhabited by microbial flora that experiences intestinal peristalsis-like motions and flow. Lab Chip. 12 (12), 2165-2174 (2012).
- Karra, N., Fernandes, J., James, J., Swindle, E. J., Morgan, H. The effect of membrane properties on cell growth in an 'airway barrier on a chip'. Organs-on-a-Chip. 5, 10025(2023).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados