Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Immunkompetentes Darm-on-Chip-Modell zur Analyse der Immunantworten der Darmschleimhaut
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Unser detailliertes Protokoll beschreibt die Erstellung und Verwendung des fortschrittlichen Darm-on-Chip-Modells, das die menschliche Darmschleimhaut mit 3D-Strukturen und verschiedenen Zelltypen simuliert und eine eingehende Analyse von Immunantworten und zellulären Funktionen als Reaktion auf mikrobielle Besiedlung ermöglicht.
Zusammenfassung
Es wurde ein fortschrittliches Darm-on-Chip-Modell entwickelt, das epitheliale 3D-organotypische zottenartige und kryptenartige Strukturen nachbildet. Das immunkompetente Modell umfasst humane Nabelvenendothelzellen (HUVEC), Caco-2-Darmepithelzellen, geweberesidente Makrophagen und dendritische Zellen, die sich selbst im Gewebe organisieren und die Eigenschaften der menschlichen Darmschleimhaut widerspiegeln. Ein einzigartiger Aspekt dieser Plattform ist ihre Fähigkeit, zirkulierende menschliche primäre Immunzellen zu integrieren, was die physiologische Relevanz erhöht. Das Modell soll die Reaktion des intestinalen Immunsystems auf bakterielle und pilzliche Besiedlung und Infektion untersuchen. Aufgrund seiner vergrößerten Kavitätengröße bietet das Modell vielfältige funktionelle Messwerte wie Permeationsassays, Zytokinfreisetzung und Immunzellinfiltration und ist kompatibel mit der Immunfluoreszenzmessung von 3D-Strukturen, die von der Epithelzellschicht gebildet werden. Dabei bietet es umfassende Einblicke in die Zelldifferenzierung und -funktion. Die Darm-on-Chip-Plattform hat ihr Potenzial bei der Aufklärung komplexer Wechselwirkungen zwischen Surrogaten einer lebenden Mikrobiota und menschlichem Wirtsgewebe innerhalb einer mikrophysiologischen durchbluteten Biochip-Plattform unter Beweis gestellt.
Einleitung
Organ-on-Chip (OoC)-Systeme stellen eine aufstrebende Technik der 3D-Zellkultur dar, die in der Lage ist, die Lücke zwischen herkömmlichen 2D-Zellkulturen und Tiermodellen zu schließen. OoC-Plattformen bestehen in der Regel aus einem oder mehreren Kompartimenten, die gewebespezifische Zellen enthalten, die auf einer Vielzahl von Gerüsten wie Membranen oder Hydrogelen gezüchtetwerden 1. Die Modelle sind in der Lage, eine oder mehrere definierte organotypische Funktionen nachzuahmen. Pumpen ermöglichen die kontinuierliche mikrofluidische Perfusion von Zellkulturmedium zur Entfernung zellulärer Abfallprodukte, zur Versorgung mit Nährstoffen und Wachstumsfaktoren für eine verbesserte zelluläre Differenzierung und zur Wiederherstellung wesentlicher In-vivo-Bedingungen. Durch die Integration von Immunzellen können OoC-Systeme die menschliche Immunantwort in vitro nachahmen 2. Bisher wurde ein breites Spektrum an Organen und Funktionseinheiten vorgestellt1. Zu diesen Systemen gehören Modelle des Gefäßsystems3, der Lunge4, der Leber 2,5 und des Darms6, die für Arzneimitteltests 5,7 und Infektionsstudien 6,8 ermöglicht werden können.
Wir präsentieren hier ein humanes Darm-on-Chip-Modell, das humane Epithelzellen integriert, die eine organotypische 3D-Topographie von zottenartigen und kryptenartigen Strukturen bilden, kombiniert mit einer Endothelauskleidung und geweberesidenten Makrophagen. Das Modell wird in einem mikrofluidisch perfundierten Biochip in Form eines mikroskopischen Objektträgers kultiviert. Jeder Biochip besteht aus zwei separaten mikrofluidischen Kavitäten. Jeder Hohlraum ist durch eine poröse Polyethylenterephthalat (PET)-Membran in eine obere und eine untere Kammer unterteilt. Die Membran selbst dient auch als Gerüst für das Wachstum der Zellen auf jeder Seite. Die Poren der Membran ermöglichen zelluläres Übersprechen und Zellmigration zwischen den Zellschichten. Jede Kammer ist über zwei weibliche Luer-Lock-Anschlüsse zugänglich. Optional kann ein zusätzlicher Mini-Luer-Anschluss in der Größe eines Locks den Zugang zur oberen oder unteren Kammer ermöglichen (Abbildung 1).
Die OoC-Plattform bietet eine Reihe von Messwerten, die aus einem einzigen Experiment gewonnen werden können. Der Darm-on-Chip ist auf die Kombination von perfundierter 3D-Zellkultur, Abwasseranalyse und Fluoreszenzmikroskopie zugeschnitten, um die Zellmarkerexpression, die Metabolisierungsraten, die Immunantwort, die mikrobielle Besiedlung und Infektion sowie die Barrierefunktion zu beurteilen 3,6,8. Das Modell umfasst geweberesidente Immunzellen und den direkten Kontakt lebender Mikroorganismen mit dem Wirtsgewebe, was im Vergleich zu anderen veröffentlichten Modellen einen Vorteil darstellt9. Darüber hinaus organisieren sich Epithelzellen selbst zu dreidimensionalen Strukturen, die eine physiologisch relevante Schnittstelle für die Besiedlung mit einer lebenden Mikrobiota darstellen6.
Protokoll
Dieses Protokoll erfordert den Zugang zu ~20 ml frischem Blut pro Biochip von gesunden Spendern, um primäre menschliche Monozyten zu isolieren. Alle Spenderinnen und Spender gaben eine schriftliche, informierte Einwilligung zur Teilnahme an dieser Studie, die von der Ethikkommission des Universitätsklinikums Jena genehmigt wurde (Zulassungsnummer 2018-1052-BO). Weitere Informationen zu den Materialien finden Sie in der Materialtabelle. Einzelheiten zur Zusammensetzung aller Lösungen und Medien finden Sie in Tabelle 1.
1. Allgemeine Bemerkungen zur Handhabung von Biochips
- Trennen Sie vorsichtig einen Streifen von Behältern und lösen Sie die Deckel mit einem erhitzten Messer, um einzelne Behälter und Deckel zu erhalten. Erweitern Sie das Loch des Deckels, damit der Silikonschlauch eng anliegt.
- Der Silikonschlauch hat einen Innendurchmesser von 0,5 mm, ist asymmetrisch und wird durch zwei peristaltische Pumpenstopfen in eine längere (20 cm) und eine kürzere (12 cm) Seite getrennt. Montieren Sie zwei Röhrchen jeder Symmetrie pro Biochip, indem Sie ein Röhrchen an einem männlichen Luer-Lock-Connecter befestigen und den Deckel auf der gegenüberliegenden Seite des Röhrchens anbringen. Bauen Sie außerdem vier Reservoirs pro Biochip zusammen.
HINWEIS: Bereiten Sie Schläuche und Reservoirs im Voraus vor und sterilisieren Sie sie vor der Verwendung durch Autoklavieren. Da der Silikonschlauch eine begrenzte Lebensdauer hat, tauschen Sie den Schlauch nach 3-5 Versuchen aus. Für bestimmte Forschungsinteressen, wie z. B. Drogentests, wird empfohlen, für jedes Experiment neue Schläuche vorzubereiten. Viele Schritte dieses Protokolls laufen parallel ab. Siehe die Übersichtsabbildung, die die verschiedenen Schritte zeigt, die an einem Tag ausgeführt wurden, wie in Abbildung 2 gezeigt.
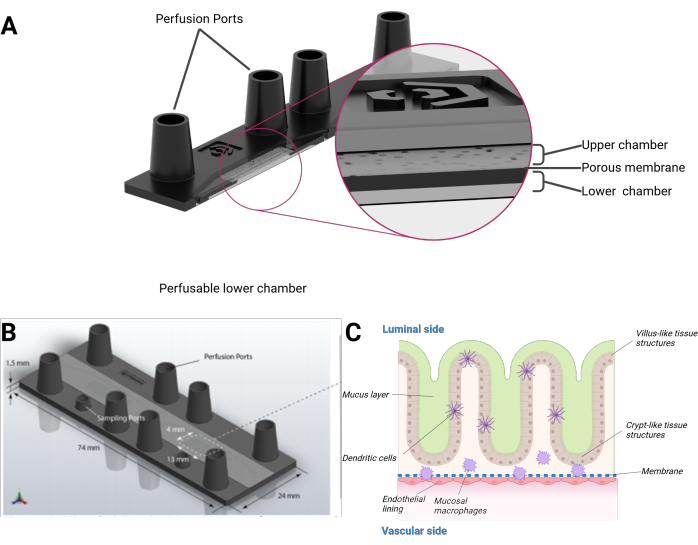
Abbildung 1: Schematische Darstellung des Darm-on-Chip-Modells. (A) Der Biochip wird in einer Querschnittsansicht dargestellt. (B) Die Abmessungen des gesamten Biochips sowie der flachen, abnehmbaren PET-Membran sind sichtbar. Das Gesamtvolumen der oberen Kammer einschließlich der weiblichen Luer-Lock-Ports beträgt 290 μl bzw. 270 μl für die untere Kammer. (C) Zu sehen ist ein schematischer Aufbau des Darm-Biochips, des dreidimensionalen Auswuchsepithels, das zottenartigen und kryptenartigen Strukturen ähnelt, einschließlich differenzierter Immunzellen und einer Schleimschicht. Die andere Seite der PET-Membran ist von einer endothelialen Monoschicht bedeckt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
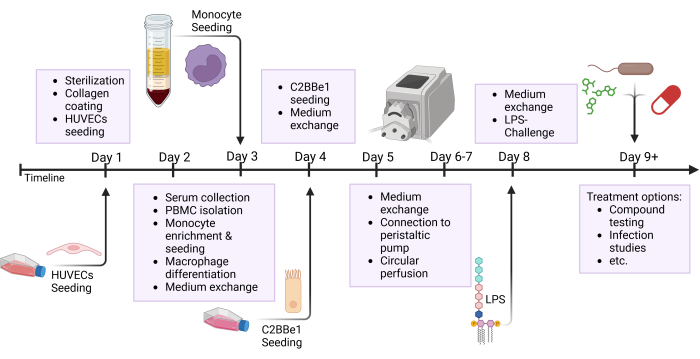
Abbildung 2: Schematischer Überblick über den Zeitplan für den Modellaufbau und den Versuchsaufbau. Diese Abbildung zeigt die schematische Übersicht des vorgestellten Protokolls. Wichtige Vorgänge, wie die Aussaat von Zellen und die epitheliale Provokation mit LPS sind durch Pfeile gekennzeichnet. Abkürzungen: HUVECs = humane nabelvenöse Endothelzellen; LPS = Lipopolysaccharid. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
2. Sterilisation von Biochips
- Eine sterile Petrischale aus Glas mit einem Durchmesser von 15 cm wird zu 70 % mit unvergälltem Ethanol gefüllt. Platzieren Sie den Biochip so, dass alle Anschlüsse des Biochips vollständig von der Ethanollösung bedeckt sind.
- Ziehen Sie zweimal pro Port 1 ml 70%iges Ethanol durch alle Kammern des Chips. 45-60 min bei Raumtemperatur (RT) inkubieren.
HINWEIS: Stellen Sie sicher, dass keine Luft im Biochip eingeschlossen ist. Ab diesem Zeitpunkt sollte keine Luft mehr in das Biochip-System eindringen und die Hohlräume sollten mit Flüssigkeit gefüllt bleiben. - Entfernen Sie das Ethanol aus der Petrischale und ersetzen Sie es durch steriles doppelt destilliertes Wasser (ddH2O), bis alle Anschlüsse vollständig bedeckt sind. Ziehen Sie wiederum zweimal pro Port 1 mL ddH2O durch den Biochip-Hohlraum. Frischen Sie das ddH2O in der Petrischale auf und wiederholen Sie den Vorgang.
- Entfernen Sie alle Flüssigkeiten aus der Petrischale. Bewahren Sie danach die vollständig sterilisierten Biochips in der geschlossenen Petrischale auf, wenn sie sich außerhalb einer sterilen Umgebung befinden. Geben Sie ein kleines Reservoir (z. B. den Deckel eines 50-ml-Röhrchens) mit 2-5 mL ddH2O in die Petrischale, um die Verdunstung von Flüssigkeit im Biochip zu reduzieren.
HINWEIS: Biochips können bis zu 3 Tage im Voraus sterilisiert werden, wenn sie bis zur Verwendung in einer sterilen Umgebung aufbewahrt werden. Dies ermöglicht eine flexible Arbeitsbelastung an einem einzigen Tag.
3. Ernte und Aussaat der HUVECs
HINWEIS: Die humanen venösen Nabelzellenzellen (HUVECs) wurden aus Nabelschnüren isoliert, wie vor10 veröffentlicht.
- Vor der Aussaat der HUVECs die Membran mit humanem Kollagen IV beschichten. Bereiten Sie dazu eine 1:100-Verdünnung einer Kollagen-Stammlösung (Tabelle 1) in Dulbeccos phosphatgepufferter Kochsalzlösung vor, die Magnesium und Calcium enthält (PBS +/+). Geben Sie 350 μl der verdünnten Stammlösung in die entsprechende Kammer. 5 min bei RT inkubieren.
HINWEIS: Wenn Sie den Biochip in der sterilen Haube handhaben, empfehlen wir, ein steriles Tuch darunter zu legen, um überschüssiges Medium aufzufangen. - Spülen Sie alle Kammern zweimal mit 350 μL PBS +/+, um das restliche Kollagen und die Essigsäure auszuwaschen. Geben Sie dann 350 μl Endothelzellwachstumsmedium (EC) in jede Kammer.
HINWEIS: Von hier aus sind die Biochips bereit für die Zellaussaat und können bis zur Verwendung bei 37 °C gelagert werden. - Verwenden Sie HUVECs an den Passagen 1-3 bei 80-90% Zellkonfluenz. Kultivieren Sie HUVECs in EC-Medium, das eine vom Hersteller bereitgestellte definierte Ergänzungsmischung enthält. Ein repräsentatives Hellfeldbild einer HUVEC-Zellkultur ist in Abbildung 3A dargestellt.
HINWEIS: Je nach Spender können HUVECs mit höheren Passagen beginnen, sich zu dedifferenzieren und können keine dichte und konfluente Monoschicht im Biochip bilden. Die Verwendung von Antibiotika, d. h. 100 μg/ml Penicillin und 100 μg/ml Streptomycin, ist fakultativ, wird aber als Ergänzung zum EC-Medium empfohlen, um eine mikrobielle Kontamination zu vermeiden. - Nehmen Sie das Zellkulturmedium aus einem T25-Zellkulturkolben und waschen Sie die Zellen vorsichtig mit 3-5 mL der phosphatgepufferten Kochsalzlösung von Dulbecco ohne Magnesium und Calcium (PBS -/-). Entfernen Sie das PBS -/- und fügen Sie 1 ml Trypsin-Dissoziationsreagenz hinzu (Tabelle 1). 5 min bei 37 °C inkubieren, bis sich die Zellen vom Zellkulturkolben lösen.
- Übertragen Sie die abgelösten Zellen in ein Röhrchen mit 9 ml 5 % fötalem Rinderserum (FBS) in PBS -/-. Bei 350 × g für 5 min bei RT zentrifugieren. Den Überstand entfernen, in 1 mL EC-Medium resuspendieren und die Anzahl der Zellen bestimmen. Stellen Sie die Zellkonzentration auf 0,4 × 106 Zellen pro 150 μl (Aussaat in der unteren Kammer) oder pro 250 μl (Aussaat in der oberen Kammer) ein.
- Geben Sie das entsprechende Zellvolumen in die Kammer. Wenn Sie in der unteren Kammer säen, schließen Sie alle Anschlüsse und positionieren Sie den Biochip sofort auf dem Kopf, so dass die Zellen auf die PET-Membran fallen. Inkubieren Sie die Biochips in einem befeuchteten Inkubator bei 37 °C und 5 % CO2.
- Führen Sie nach 24 h einen Medienaustausch der HUVEC-haltigen Kammer mit 350 μL EC-Medium durch. Das Medium in der gegenüberliegenden Kammer muss nicht ausgetauscht werden.
4. Humane Serumentnahme und Isolierung von mononukleären Zellen des peripheren Blutes (PBMC) aus Monozyten
HINWEIS: Die PBMCs wurden isoliert, wie in Mosig et al.11 beschrieben.
- Entnahme von humanem venösem Blut von gesunden Spendern. Beschaffen Sie pro Biochip mindestens 10 ml Vollblut in silikathaltigen Blutentnahmeröhrchen für die Serumentnahme. Nach vollständiger Koagulation zentrifugieren Sie die Blutentnahmeröhrchen bei 2.500 × g für 10 min bei RT. Entnehmen Sie das Serum, aliquot und lagern Sie es bis zur weiteren Verwendung bei -20 °C.
- Beschaffen Sie mindestens 10 ml Vollblut vom selben Spender in EDTA-haltigen Blutentnahmeröhrchen für die PBMC-Isolierung. Mischen Sie das nicht koagulierte Blut vorsichtig 1:1 mit Isopuffer (Tabelle 1) durch Inversion und schichten Sie langsam 35 ml dieser Mischung auf 15 ml eines Dichtegradientenmediums mit einer Dichte von 1,077 g/ml in einem 50 ml-Röhrchen.
- Zentrifugieren Sie bei 800 × g für 20 min ohne Bremse bei RT. Die resultierende Immunzellschicht, die auf dem Dichtegradientenmedium erscheint, wird vorsichtig herausgezogen und in ein neues 50-ml-Röhrchen überführt. Füllen Sie bis zu 50 mL mit kaltem Iso-Puffer und waschen Sie die Zellen durch sanfte Inversion.
- Bei 200 × g für 8 min ohne Bremse bei 4 °C zentrifugieren. Der Überstand wird verworfen und in 10 ml Isopuffer pro Dichtegradient resuspendiert. Optional: Poolen Sie PBMCs eines Donors, wenn mehrere Gradienten parallel ausgeführt werden.
- Bei 150 × g 8 min bei 4 °C zentrifugieren. Der Überstand wird verworfen und das Pellet in 10 ml Isopuffer pro Dichtegradient resuspendiert. Der Zentrifugationsschritt 4.4 wird wiederholt. Zum Schluss wird der Überstand verworfen und die Zellen in 2 ml Monozyten-Differenzierungsmedium resuspendiert (Tabelle 1).
HINWEIS: Die Zugabe von M-CSF und GM-CSF erzwingt die Differenzierung der isolierten Monozyten zu Monozyten-abgeleiteten Makrophagen und von Monozyten abgeleiteten dendritischen Zellen (in Kombination mit Lipopolysaccharid [LPS], das zu einem späteren Zeitpunkt dieses Protokolls hinzugefügt wird). Die Verwendung von Antibiotika, d. h. 100 μg/ml Penicillin und 100 μg/ml Streptomycin, ist fakultativ, wird aber als Ergänzung des Mediums empfohlen, um eine mikrobielle Kontamination zu verhindern. - Bestimmen Sie die Zellzahl und den Seed ~10 × 106 Zellen pro Well einer 6-Well-Platte in 2 ml Monozyten-Differenzierungsmedium (Tabelle 1). In einem befeuchteten Inkubator bei 37 °C für 1 h inkubieren, um die Monozyten an den Kunststoff der 6-Well-Platte anheften zu können.
- Entsorgen Sie den Überstand vorsichtig und waschen Sie ihn 2x mit vorgewärmten 2 ml hämatopoetischem Zellmedium, um die ungebundenen Zellen zu entfernen. Bei 37 °C für weitere 24 h in Monozyten-Differenzierungsmedium inkubieren.
- Um die Monozyten zu gewinnen, entsorgen Sie den Überstand vorsichtig und waschen Sie ihn einmal mit 2 mL vorgewärmtem PBS -/-. In Abbildung 3B finden Sie ein Beispiel für ein Hellfeldbild der Monozytenkultur an dieser Stelle. Inkubieren Sie die Zellen dann 7 Minuten lang in 1 ml vorgewärmtem Monozytenablösungsreagenz (Tabelle 1) bei 37 °C, um die Ablösung der Monozyten vom Kunststoff der 6-Well-Platte zu erzwingen.
- Übertragen Sie die abgelösten Monozyten in ein niedrig bindendes Röhrchen. Optional: Um eine höhere Zellausbeute zu erzielen, waschen Sie die 6-Well-Platte mehrmals vorsichtig mit PBS -/-.
- Bei 300 × g für 8 min bei RT zentrifugieren. Den Überstand verwerfen und in EC-konditioniertem Medium resuspendieren (Tabelle 1). Bestimmen Sie die Zellzahl und stellen Sie die Zellkonzentration auf 0,1 × 106 Zellen pro 150 μL (Aussaat in der unteren Kavität) oder pro 250 μL (Aussaat in der oberen Kavität) ein.
HINWEIS: Seien Sie bei allen Schritten der Immunzellisolierung vorsichtig und reduzieren Sie die Scherkräfte, um eine Aktivierung der Immunzellen zu verhindern. Bei der Etablierung dieser Isolierung ist die Reinheit der PBMC-abgeleiteten Monozyten zu überprüfen (z. B. durch Durchflusszytometrie). Mehr als 95% aller Zellen sollten positiv für typische Monozytenmarker wie CD14 sein.
5. Aussaat von Monozyten
- In der HUVEC-haltigen Kammer wird ein Medienaustausch mit 350 μL vorgewärmtem EC-konditioniertem Medium durchgeführt.
- 150 μl (untere Kammer) oder 250 μl (obere Kammer) der vorbereiteten Monozytensuspension (siehe Schritt 4.10) in dieselbe Kammer geben. Wenn Sie in die untere Kammer säen, schließen Sie alle Anschlüsse und positionieren Sie den Biochip sofort auf dem Kopf, damit die Zellen auf die HUVEC-Schicht fallen. Inkubieren Sie den Biochip in einem befeuchteten Inkubator bei 37 °C und 5 % CO2.
- In der HUVEC + Monozyten-haltigen Kammer wird alle 24 h ein Mediumaustausch mit 350 μl EC-konditioniertem Medium durchgeführt.
6. C2BBe1-Ernte und Aussaat
HINWEIS: Die Caco-2-Bürstenrand, die die Zellen 1 (C2BBe1)12 exprimieren, werden bis zum Durchgang 35 verwendet und aus Kolben mit einer Konfluenz von 80-90 % entnommen. Ein repräsentatives Hellfeldbild einer C2BBe1-Kultur ist in Abbildung 3C dargestellt.
- C2BBe1-Zellen in C2-Medium kultivieren (Tabelle 1).
HINWEIS: Die Verwendung von Antibiotika, d. h. 20 μg/ml Gentamicin, ist optional, wird aber als Ergänzung des C2-Mediums empfohlen, um eine mikrobielle Kontamination zu vermeiden. - Entfernen Sie das Zellkulturmedium aus einem T25-Zellkulturkolben und waschen Sie die Zellen vorsichtig mit 3-5 mL PBS -/-. Entfernen Sie das PBS -/- und fügen Sie 1 ml Trypsin-Dissoziationsreagenz hinzu (Tabelle 1). 5 min bei 37 °C inkubieren, bis sich die Zellen vom Zellkulturkolben lösen.
- Übertragen Sie die abgelösten Zellen in ein Röhrchen, indem Sie 9 ml 5% fötales Rinderserum (FBS) in PBS -/- verwenden. Bei 350 × g für 5 min bei RT zentrifugieren. Den Überstand entfernen, in 1 mL C2-Medium resuspendieren und die Anzahl der Zellen bestimmen. Stellen Sie die Zellkonzentration auf 0,5 × 106 Zellen pro 150 μl (Aussaat in der unteren Kammer) oder pro 250 μl (Aussaat in der oberen Kammer) ein.
- Waschen Sie vor der Aussaat des C2BBe1 die jeweilige Kammer vorsichtig mit 350 μL C2-Medium.
- 150 μL (untere Kammer) bzw. 250 μL (obere Kammer) der vorbereiteten C2BBe1-Suspension (siehe Schritt 6.3) in die jeweilige Kammer geben. Wenn Sie in die untere Kammer säen, schließen Sie alle Anschlüsse und positionieren Sie den Biochip sofort auf dem Kopf, damit die Zellen auf die PET-Membran fallen. Inkubieren Sie die Biochips in einem befeuchteten Inkubator bei 37 °C und 5 % CO2.

Abbildung 3: Zellmorphologie von HUVECs, Monozyten und C2BBe1 vor der Aussaat im Biochip. Diese Abbildung zeigt repräsentative Hellfeldbilder der verschiedenen Zellquellen, die im gesamten Protokoll verwendet werden. Die Bilder wurden mit einem umgekehrten Hellfeldmikroskop mit 10-facher Vergrößerung aufgenommen. Alle Zelltypen, (A) HUVECs, (B) Monozyten und (C) C2BBe1 wurden in 2D-Monolayer-Zellkulturen kultiviert, wie in den jeweiligen Protokollabschnitten beschrieben. Maßstabsleisten = 200 μm. Abkürzungen: HUVECs = humane nabelvenöse Endothelzellen; PBMCs = mononukleäre Zellen des peripheren Blutes. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
7. Anschluss an Peristaltikpumpe und Zirkularperfusion
- Bereiten Sie einen leeren Inkubator mit einer Schlauchpumpe vor. Reinigen Sie alle Bereiche des Inkubators und der Pumpe gründlich mit Desinfektionsmittel, um eine quasi-sterile Umgebung zu schaffen.
HINWEIS: Schlauchpumpen können während der Arbeit viel Wärme erzeugen. In gut isolierten Brutschränken oder schlecht klimatisierten Laboren kann die Anzahl der nutzbaren Pumpen pro Brutkasten begrenzt sein, da Brutschränke zur Überhitzung neigen. Zwei Schlauchpumpen pro Inkubator sollten ausreichend sein. - Bevor Sie die sterilisierten Röhrchen an den Biochip anschließen, spülen Sie jeden Schlauch mit 700 μl PBS +/+, gefolgt von 500 μl C2-Medium oder EC-konditioniertem Medium. Bereiten Sie einen Schlauch für jede Symmetrie pro Medium vor (siehe Schritt 1.2). Verwenden Sie den Schlauch mit dem kurzen Abstand vom Luer-Lock zum peristaltischen Pumpenstopfen für die linke Kavität und den Schlauch mit der anderen Symmetrie für die rechte Kavität.
- Entnehmen Sie den Biochip aus dem Inkubator und führen Sie einen Medienaustausch mit 350 μL für jede Kammer durch. Entfernen Sie alle Stecker und füllen Sie alle Anschlüsse bis ganz nach oben.
- Beginnen Sie am linken Hohlraum und verbinden Sie den ersten Schlauch mit dem rechten Anschluss der oberen Kammer, indem Sie den Luer-Lock-Adapter in den Anschluss des Biochips einführen. Verbinden Sie dann den zweiten Schlauch mit dem linken Anschluss der unteren Kammer. Wiederholen Sie diesen Vorgang für den richtigen mikrofluidischen Hohlraum.
- Nehmen Sie ein Reservoir und geben Sie einen kleinen Tropfen Zellkulturmedium auf den Boden des Reservoirs. Setzen Sie dann das Reservoir auf die gegenüberliegende Seite des ersten Schlauchs ein und wiederholen Sie den Vorgang für die andere Kammer. Sobald alle Anschlüsse mit einem Schlauch oder Reservoir verbunden sind, füllen Sie die Reservoirs mit 3,5 ml Zellkulturmedium.
- Legen Sie die lose Seite des Schlauchs, an der der Deckel befestigt ist, auf das Reservoir, um das mikrofluidische System jeder Kammer zu schließen. Transportieren Sie in diesem Zustand den Biochip zur Schlauchpumpe.
HINWEIS: Abhängig von der Entfernung zum Inkubator und der Laborumgebung kann eine zuvor gereinigte und autoklavierte Box verwendet werden, um die Chips in den Inkubator zu überführen. - Verwenden Sie die Schlauchpumpenstopfen, um den Schlauch mit der Pumpe zu verbinden. Schließen Sie jeden Schlauch so an die Schlauchpumpe an, dass das Medium aus dem Reservoir in den Hohlraum, in den Schlauch und über die Pumpe zurück in das Reservoir fließt (Abbildung 4). Das Reservoir dient als Blasenfalle in der zirkulären Perfusion und verhindert, dass Luft im System eingeschlossen wird. Jede Kammer wird mit einer Durchflussrate von 50 μL/min durchblutet, was zu einer Scherspannung von 0,013 dyn/cm2 in der oberen Kammer und 0,006 dyn/cm2 in der unteren Kammerführt 8.
HINWEIS: Wird das Medium der unteren und oberen Höhle in entgegengesetzte Richtungen bewegt, kann ein höheres dreidimensionales Auswachsen des Darmgewebes erreicht werden13. Daher sind die Reservoire des oberen und unteren Hohlraums auf gegenüberliegenden Seiten angeordnet (Abbildung 4). Die zirkuläre Perfusion reduziert die Menge an benötigtem Zellkulturmedium, könnte aber möglicherweise zu einer Anreicherung von Zytokinen und Metaboliten führen. Auf Wunsch ist auch eine lineare Perfusion des Biochips möglich. - Perfusionieren Sie den Biochip 72 h lang bei 37 °C und 5 % CO2.

Abbildung 4: Biochip, der mit der Schlauchpumpe verbunden ist. Vorgestellt wird ein Beispiel für einen Biochip, der mit einer Schlauchpumpe verbunden ist. Epithel-C2BBe1-Zellen werden in der unteren Kammer kultiviert (rotes C2-Medium befindet sich in den Reservoiren vorne), während die HUVECs in der oberen Kammer kultiviert werden (gelbliches EC-konditioniertes Medium befindet sich in den Reservoirs auf der Rückseite). Die verschiedenen Zellkulturmedien vermischen sich aufgrund der Barrierefunktion des gezüchteten Gewebes nicht. Der Biochip wird so mit der Peristaltikpumpe verbunden, dass das Medium aus dem Reservoir in den Hohlraum fließt. Von hier fließt das Medium durch die Schläuche über die Pumpe zurück in das Reservoir. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
8. LPS-Konditionierung der Epithelbarriere
- Nach 72 Stunden Präperfusion stoppen Sie die Peristaltikpumpe und entfernen Sie den Deckel, der mit dem Schlauch jedes Reservoirs verbunden ist. Legen Sie es auf ein steriles Tuch neben der Pumpe.
- Entfernen Sie das gesamte Medium und füllen Sie die Behälter mit 2 mL frisch zubereitetem Medium. Für die epitheliale Seite, die die C2BBe1-Zellen enthält, geben Sie 100 ng/mL LPS in das Medium.
HINWEIS: Das LPS erhöht die Barrierefunktion des Gewebes, stimuliert die von Monozyten abgeleiteten Makrophagen, in das Epithelgewebe zu migrieren, und ermöglicht die Differenzierung von dendritischen Zellen aus Monozyten. - Schließen Sie die Schläuche und Deckel wieder an das Reservoir an und setzen Sie die zirkuläre Perfusion bei einer Flussrate von 50 μl/min für weitere 24 Stunden fort.
HINWEIS: Ab diesem Zeitpunkt kann das Chip-Modell in Experimenten, Tests von Verbindungen oder Infektionsstudien verwendet werden. Wir empfehlen einen Medienaustausch von 2 mL pro Reservoir alle 24 h.
9. Zugang zum Gewebe für verschiedene Auslesemethoden
- Sammeln Sie zu jedem Zeitpunkt der Perfusion Überstände des Zellkulturmediums aus den Reservoiren. Öffnen Sie den Behälter und sammeln Sie das gewünschte Volumen (siehe Schritte 8.1-8.3). Verwenden Sie diese Überstände zum Nachweis von Metaboliten, Zytokinen oder anderen Molekülen.
- Um an das Gewebe zu gelangen, verwenden Sie ein Skalpell, um einen präzisen Schnitt entlang der Außenseite der oberen Kammer zu machen, und entfernen Sie die Klebefolie, um den mikrofluidischen Hohlraum zu öffnen. Das Gewebe des Darm-Biochip-Modells ist nun zugänglich. Schneiden Sie vorsichtig an der Außenseite der mikrofluidischen Kammer entlang, um die Membran vom Biochip zu lösen. Entnehmen Sie die gewebehaltige Membran mit einer Pinzette.
ACHTUNG: Achten Sie bei diesem Schritt auf die Fingerplatzierung und arbeiten Sie vorsichtig, um Unfälle zu vermeiden. Schnittschutzhandschuhe werden empfohlen. - Alternativ können die Zellen mit Enzymlösungen, d. h. Trypsin oder Zellen, die mit Triton X-100-haltigen Puffern lysiert wurden, aus separaten Schichten im Inneren des Biochips entnommen werden.
10. Permeabilitätsbewertung mittels FITC-Dextran-Diffusion
HINWEIS: Die Barrierefunktion des Gewebes kann über einen FITC-Dextran-Permeabilitätsassay nach Abschaltung der Peristaltikpumpe analysiert werden. Die Bewertung der Durchlässigkeit von FITC-Dextran wurde von Deinhardt-Emmer et al.4 adaptiert.
- Eine Stammlösung aus Fluoresceinisothiocyanat (FITC)-Dextran (Molekulargewicht von 3-5 kDa, Tabelle 1) herstellen.
- Leeren Sie die Behälter und trennen Sie den Chip von der Perfusion.
- Führen Sie in der oberen und unteren Kammer einen Medienaustausch mit phenolrotfreiem Medium durch.
HINWEIS: Dieser Schritt ist nicht erforderlich, wenn während des Versuchs bereits phenolrotfreies Medium verwendet wurde. - 350 μl 1 mg/ml FITC-Dextran-Lösung in die Kammer geben, die die C2BBe1-Zellen enthält.
- Schließen Sie die Öffnungen und inkubieren Sie den Chip 60 Minuten lang bei 37 °C mit der Epithelseite nach oben.
- Nach der Inkubationszeit wird das Nährmedium aus beiden Kammern des Chips getrennt entnommen und bis zur Messung bei 4 °C lichtgeschützt gelagert.
- Für die Messung ist eine Standardkurve in C2-Medium und dem EC-konditionierten Medium ohne Phenolrot im Bereich von 1.000 μg/mL bis 0 μg/mL FITC-Dextran mit 11 aufeinanderfolgenden 1:2 Serienverdünnungen zu erstellen.
- Übertragen Sie 200 μl jeder Probe in eine schwarze 96-Well-Platte mit klarem Boden. Messen Sie die Fluoreszenz mit einem Mikroplatten-Reader bei einer Anregungswellenlänge von 495 nm und einer Emissionswellenlänge von 517 nm.
- Verwenden Sie die Standardkurve, um die FITC-Dextran-Konzentration der Proben und damit den Permeabilitätskoeffizienten zu berechnen.
11. Immunfluoreszenz-Färbung
HINWEIS: Das lebende Gewebe kann mikroskopisch untersucht werden. Für eine einfachere Handhabung empfehlen wir die Abtrennung des Biochips von der Peristaltikpumpe und die Verwendung von Fernobjektiven an einem inversen Mikroskop. Als Endpunktanalyse kann das Gewebe im Biochip fixiert werden, z. B. für die Immunfluoreszenzfärbung.
- Stoppen Sie die Schlauchpumpe und öffnen Sie die Reservoirs aller Kavitäten. Leeren Sie die Reservoirs und trennen Sie die Schläuche sowie die Reservoirs vom Biochip.
- Waschen Sie die mikrofluidischen Kavitäten zweimal pro Kammer mit 500 μl kaltem PBS +/+. 500 μl eiskaltes Methanol in alle Kavitäten geben und 15 min bei -20 °C inkubieren. Waschen Sie dann die mikrofluidische Kammer zweimal pro Kavität mit 500 μl PBS +/+.
HINWEIS: Andere Fixierungsmethoden, wie z.B. die Fixierung mit 4% Paraformaldehyd oder Carnoy's Fixiermittel, sind ebenfalls geeignet. Nach der Fixierung können die Chips bei 4 °C gelagert werden oder direkt zur Immunfluoreszenzfärbung übergehen. ACHTUNG: Fixierende Chemikalien wie Methanol oder Paraformaldehyd sind giftig. Führen Sie die jeweiligen Aufgaben unter einem Abzug durch und sammeln Sie den Müll entsprechend. - Öffnen Sie den Chip wie in Schritt 9.2 beschrieben, um an das Gewebe zu gelangen. Schneiden Sie die gewebehaltige PET-Membran in bis zu drei Stücke, um sie parallel mit verschiedenen Immunopanels zu färben.
- Übertragen Sie jedes der Membranstücke mit einer Präzisionspinzette auf eine separate 24-Well-Platte, die eine Blockierungs- und Permeabilisierungslösung enthält (Tabelle 1). Stellen Sie sicher, dass die interessierende Zellschicht während des gesamten Färbeprozesses immer nach oben zeigt. Inkubieren Sie die Membranstücke 30 Minuten lang bei RT.
HINWEIS: Die besten Färbeergebnisse werden erzielt, wenn das Serum auf den Sekundärantikörper abgestimmt wird. Werden z.B. Sekundärantikörper von Ziegenarten gewonnen, empfehlen wir die Verwendung von normalem Ziegenserum. - Übertragen Sie die Membranstücke auf einen sauberen Objektträger in einer Feuchtkammer. Bereiten Sie das primäre Antikörperpanel in der Färbelösung vor (Tabelle 1) und fügen Sie jedem Membranstück 50 μl hinzu. Über Nacht bei 4 °C inkubieren.
HINWEIS: Die optimale Antikörperkonzentration und Färbewirksamkeit kann je nach Hersteller und Klon unterschiedlich sein. Wir empfehlen, die Färbeplatten vorher in einer 2D-Zellkultur zu testen. - Nach der Inkubation werden die Proben auf eine 24-Well-Platte umgefüllt und die Membranen 3 x 5 min lang vorsichtig mit Waschlösung gewaschen (Tabelle 1).
- Übertragen Sie die Membranstücke erneut auf einen sauberen Objektträger in einer Feuchtkammer. Bereiten Sie das sekundäre Antikörperpanel in Färbelösung vor (Tabelle 1) und fügen Sie jedem Membranstück 50 μl hinzu. Falls erforderlich, fügen Sie eine nukleare Gegenfärbung wie 4',6-Diamidino-2-phenylindol (DAPI) oder Hoechst hinzu. 30 Minuten bei RT inkubieren.
HINWEIS: Bewahren Sie bei der Arbeit mit Fluorophoren die Proben vor Licht geschützt auf, um Photobleiche zu verhindern und die Bildqualität zu verbessern. - Nach der Inkubation werden die Proben auf eine 24-Well-Platte umgefüllt und die Membranen 2 x 5 min vorsichtig mit Waschlösung gewaschen (Tabelle 1). Dann einmal mit PBS +/+ für 5 min waschen.
- Montieren Sie die Membranstücke mit einem Fluoreszenz-Eindeckmedium und einem Deckglas auf einen sauberen Objektträger. Bis zur mikroskopischen Bildgebung bei 4 °C lagern.
Ergebnisse
Diese repräsentativen Ergebnisse zeigen die unterschiedlichen Gewebeschichten des Darm-on-Chip-Modells. Sie sind immunfluoreszenzgefärbt, wie in Protokollabschnitt 11 beschrieben. Die Bilder wurden mit einem Epifluoreszenz- oder konfokalen Fluoreszenzmikroskop als z-Stacks aufgenommen und zu einer orthogonalen Projektion verarbeitet. In der Materialtabelle finden Sie Details zum mikroskopischen Aufbau und zur Software. Abbildung 5 zeigt die Gefäßschicht, eine barrierebil...
Diskussion
Das vorgestellte Protokoll beschreibt die notwendigen Schritte zur Generierung eines immunkompetenten Darm-on-Chip-Modells. Wir haben spezifische Techniken und mögliche Auslesemethoden wie Immunfluoreszenzmikroskopie, Zytokin- und Metabolitenanalyse, Durchflusszytometrie, Protein- und Genanalyse sowie Permeabilitätsmessung beschrieben.
Das beschriebene Modell besteht aus primären HUVECs, Monozyten-abgeleiteten Makrophagen und von Monozyten abgeleiteten dendritischen Zellen, die mit einer 3D...
Offenlegungen
M.R. ist Geschäftsführer der Dynamic42 GmbH und hält Anteile an dem Unternehmen. A.S.M. ist wissenschaftlicher Berater der Dynamic 42 GmbH und hält Anteile an dem Unternehmen.
Danksagungen
Die Arbeiten wurden finanziell unterstützt durch den Sonderforschungsbereich PolyTarget 1278 (Projektnummer 316213987) an V.D.W. und A.S.M. A.F. und A.S.M. würdigen ferner die finanzielle Unterstützung durch den Exzellenzcluster "Balance of the Microverse" im Rahmen der Exzellenzstrategie des Bundes und der Länder - EXC 2051 - Projekt-ID 690 390713860. Wir danken Astrid Tannert und dem Jena Biophotonic and Imaging Laboratory (JBIL) dafür, dass sie uns Zugang zu ihrem konfokalen Laser-Scanning-Mikroskop ZEISS LSM980 zur Verfügung gestellt haben. Abbildung 1C und Abbildung 2 wurden mit Biorender.com erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| 96-well plate black, clear bottom | Thermo Fisher | 10000631 | Consumables |
| Acetic acid | Roth | 3738.4 | Chemicals |
| Alexa Fluor 488 AffiniPure, donkey, anti-mouse IgG (H+L) | Jackson Immuno Research | 715-545-150 | Secondary Antibody Vascular Staining and Epithelial Staining |
| Alexa Fluor 647 AffiniPure, donkey, anti-rabbit IgG (H+L) | Jackson Immuno Research | 711-605-152 | Secondary Antibody Epithelial Staining |
| Alexa Fluor 647, donkey, anti-rabbit IgG (H+L) | Thermo Fisher Scientific, Invitrogen | A31573 | Secondary Antibody Vascular Staining |
| Axiocam ERc5s camera | Zeiss | 426540-9901-000 | Technical equipment |
| Basal Medium MV, phenol red-free | Promocell | C-22225 | Cell culture consumables |
| Biochip | Dynamic 42 | BC002 | Microfluidic consumables |
| BSA fraction V | Gibco | 15260-037 | Cell culture consumables |
| C2BBe1 (clone of Caco-2) | ATCC | CRL-2102 | Epithelial Cell Source |
| Chloroform | Sigma | C2432 | Chemicals |
| CO2 Incubator | Heracell | 150i | Technical equipment |
| Collagen IV from human placenta | Sigma-Aldrich | C5533 | Cell culture consumables |
| Coverslips (24 x 40 mm; #1.5) | Menzel-Gläser | 15747592 | Consumables |
| Cy3 AffiniPure, donkey, anti-goat IgG (H+L) | Jackson Immuno Research | 705-165-147 | Secondary Antibody Vascular Staining |
| Cy3 AffiniPure, donkey, anti-rat IgG (H+L) | Jackson Immuno Research | 712-165-150 | Secondary Antibody Epithelial Staining |
| DAPI (4',6-Diamidin-2-phenylindol, Dilactate) | Thermo Fisher Scientific, Invitrogen | D3571 | Vascular and Epithelial Staining |
| Descosept PUR | Dr.Schuhmacher | 00-323-100 | Cell culture consumables |
| DMEM high glucose | Gibco | 41965-062 | Cell culture consumables |
| DMEM high glucose w/o phenol red | Gibco | 31053028 | Cell culture consumables |
| DPBS (-/-) | Gibco | 14190-169 | Cell culture consumables |
| DPBS (+/+) | Gibco | 14040-133 | Cell culture consumables |
| EDTA solution | Invitrogen | 15575-038 | Cell culture consumables |
| Endothelial Cell Growth Medium | Promocell | C-22020 | Cell culture consumables |
| Endothelial Cell Growth Medium supplement mix | Promocell | C-39225 | Cell culture consumables |
| Ethanol 96%, undenatured | Nordbrand-Nordhausen | 410 | Chemicals |
| Fetal bovine Serum | invitrogen | 10270106 | Cell culture consumables |
| Fluorescein isothiocyanate (FITC)-dextran (3-5 kDa) | Sigma Aldrich | FD4-100MG | Chemicals |
| Fluorescent Mounting Medium | Dako | S3023 | Chemicals |
| Gentamycin (10mg/mL) | Sigma Aldrich | G1272 | Cell culture consumables |
| GlutaMAX Supplement (100x) | Gibco | 35050061 | Cell culture consumables |
| Histopaque | Sigma-Aldrich | 10771 | Cell culture consumables |
| Hoechst (bisBenzimid) H33342 | Sigma-Aldrich | 14533 | Epithelial Staining |
| Holotransferrin (5mg/mL) Transferrin, Holo, Human Plasma | Millipore | 616397 | Cell culture consumables |
| Human recombinant GM-CSF | Peprotech | 300-30 | Cell culture consumables |
| Human recombinant M-CSF | Peprotech | 300-25 | Cell culture consumables |
| Illumination device | Zeiss | HXP 120 C | Fluorescence Microscope Setup |
| Laser Scanning Microscope | Zeiss | CLSM980 | Fluorescence Microscope Setup |
| Lidocain hydrochloride | Sigma-Aldrich | L5647 | Cell culture consumables |
| Lipopolysaccharide (LPS) | Sigma | L2630 | Cell culture consumables |
| Loftex Wipes | Loftex | 1250115 | Consumables |
| Low attachment tubes (PS, 5 mL) | Falcon | 352052 | Consumables |
| Luer adapter for the top cap (M) | Mo Bi Tec | M3003 | Microfluidic consumables |
| Male mini luer plugs, row of four,PP, opaque | Microfluidic chipshop | 09-0556-0336-09 | Microfluidic consumables |
| MEM Non-Essential Amino Acids Solution | Gibco | 11140 | Cell culture consumables |
| Methanol | Roth | 8388.2 | Chemicals |
| Microscope | Zeiss | Axio Observer 5 | Fluorescence Microscope Setup |
| Microscope slides | Menzel | MZ-0002 | Consumables |
| Monoclonal, mouse, anti-human CD68 Antibody (KP1) | Thermo Fisher Scientific, Invitrogen | 14-0688-82 | Primary Antibody Vascular Staining |
| Monoclonal, rat, anti-human E-Cadherin antibody (DECMA-1) | Sigma-Aldrich, Millipore | MABT26 | Primary Antibody Epithelial Staining |
| Multiskan Go plate reader | Thermo Fisher | 51119300 | Technical equipment |
| Normal donkey serum | Biozol | LIN-END9010-10 | Chemicals |
| Optical Sectioning | Zeiss | ApoTome | Fluorescence Microscope Setup |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140-122 | Cell culture consumables |
| Plugs | Cole Parmer | GZ-45555-56 | Microfluidic consumables |
| Polyclonal, goat, anti-human VE-Cadherin Antibody | R&D Systems | AF938 | Primary Antibody Vascular Staining |
| Polyclonal, rabbit, anti-human Von Willebrand Factor Antibody | Dako | A0082 | Primary Antibody Vascular Staining |
| Polyclonal, rabbit, anti-human ZO-1 antibody | Thermo Fisher Scientific, Invitrogen | 61-7300 | Primary Antibody Epithelial Staining |
| Power Supply Microscope | Zeiss | Eplax Vp232 | Fluorescence Microscope Setup |
| Primovert microscope | Zeiss | 415510-1101-000 | Technical equipment |
| Reglo ICC peristaltic pump | Ismatec | ISM4412 | Technical equipment |
| SAHA (Vorinostat) | Sigma Aldrich | SML0061-25MG | Chemicals |
| Saponin | Fluka | 47036 | Chemicals |
| S-Monovette, 7.5 mL Z-Gel | Sarstedt | 01.1602 | Consumables |
| S-Monovette, 9.0 mL K3E | Sarstedt | 02.1066.001 | Consumables |
| Sodium Pyruvate | Gibco | 11360-088 | Cell culture consumables |
| Tank 4.5 mL | ChipShop | 10000079 | Microfluidic consumables |
| Trypane blue stain 0.4% | Invitrogen | T10282 | Cell culture consumables |
| Trypsin | Gibco | 11538876 | Cell culture consumables |
| Tubing | Dynamic 42 | ST001 | Microfluidic consumables |
| Tweezers (Präzisionspinzette DUMONT abgewinkelt Inox08, 5/45, 0,06 mm) | Roth | K343.1 | Consumables |
| Wheat Germ Agglutinin (WGA) | Thermo Fisher Scientific, Invitrogen | W32464 | Epithelial Staining |
| X-VIVO 15 | Lonza | BE02-060F | Cell culture consumables, Hematopoietic cell medium |
| Zellkultur Multiwell Platten, 24 Well, sterile | Greiner Bio-One | 662 160 | Consumables |
| Zellkultur Multiwell Platten, 6 Well, sterile | Greiner Bio-One | 657 160 | Consumables |
| Zen Blue Software | Zeiss | Version 3.7 | Microscopy Software |
Referenzen
- Alonso-Roman, R., et al. Organ-on-chip models for infectious disease research. Nat Microbiol. 9 (4), 891-904 (2024).
- Fahrner, R., Groger, M., Settmacher, U., Mosig, A. S. Functional integration of natural killer cells in a microfluidically perfused liver on-a-chip model. BMC Res Notes. 16 (1), 285 (2023).
- Raasch, M., et al. Microfluidically supported biochip design for culture of endothelial cell layers with improved perfusion conditions. Biofabrication. 7 (1), 015013 (2015).
- Deinhardt-Emmer, S., et al. Co-infection with Staphylococcus aureus after primary influenza virus infection leads to damage of the endothelium in a human alveolus-on-a-chip model. Biofabrication. 12 (2), 025012 (2020).
- Kaden, T., et al. Generation & characterization of expandable human liver sinusoidal endothelial cells and their application to assess hepatotoxicity in an advanced in vitro liver model. Toxicology. 483, 153374 (2023).
- Maurer, M., et al. A three-dimensional immunocompetent intestine-on-chip model as in vitro platform for functional and microbial interaction studies. Biomaterials. 220, 119396 (2019).
- Hoang, T. N. M., et al. Invasive aspergillosis-on-chip: A quantitative treatment study of human aspergillus fumigatus infection. Biomaterials. 283, 121420 (2022).
- Kaden, T., et al. Modeling of intravenous caspofungin administration using an intestine-on-chip reveals altered Candida albicans microcolonies and pathogenicity. Biomaterials. 307, 122525 (2024).
- Shah, P., et al. A microfluidics-based in vitro model of the gastrointestinal human-microbe interface. Nat Commun. 7, 11535 (2016).
- Jaffe, E. A., Nachman, R. L., Becker, C. G., Minick, C. R. Culture of human endothelial cells derived from umbilical veins. Identification by morphologic and immunologic criteria. J Clin Invest. 52 (11), 2745-2756 (1973).
- Mosig, S., et al. Different functions of monocyte subsets in familial hypercholesterolemia: Potential function of cd14+ cd16+ monocytes in detoxification of oxidized ldl. FASEB J. 23 (3), 866-874 (2009).
- Peterson, M., Mooseker, M. Characterization of the enterocyte-like brush border cytoskeieton of the c2bbe clones of the human intestinal cell line, caco-2. J Cell Sci. 102, 581-600 (1992).
- Shin, W., Hinojosa, C. D., Ingber, D. E., Kim, H. J. Human intestinal morphogenesis controlled by transepithelial morphogen gradient and flow-dependent physical cues in a microengineered gut-on-a-chip. iScience. 15, 391-406 (2019).
- Kim, H. J., Ingber, D. E. Gut-on-a-chip microenvironment induces human intestinal cells to undergo villus differentiation. Integr Biol (Camb). 5 (9), 1130-1140 (2013).
- Kim, H. J., Huh, D., Hamilton, G., Ingber, D. E. Human gut-on-a-chip inhabited by microbial flora that experiences intestinal peristalsis-like motions and flow. Lab Chip. 12 (12), 2165-2174 (2012).
- Karra, N., Fernandes, J., James, J., Swindle, E. J., Morgan, H. The effect of membrane properties on cell growth in an 'airway barrier on a chip'. Organs-on-a-Chip. 5, 10025 (2023).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten