È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Modello immunocompetente Intestino-on-Chip per l'analisi delle risposte immunitarie della mucosa intestinale
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Il nostro protocollo dettagliato delinea la creazione e l'uso del modello avanzato di intestino su chip, che simula la mucosa intestinale umana con strutture 3D e vari tipi di cellule, consentendo un'analisi approfondita delle risposte immunitarie e delle funzioni cellulari in risposta alla colonizzazione microbica.
Abstract
È stato sviluppato un modello avanzato di intestino su chip che ricrea strutture epiteliali 3D organotipiche simili a villi e cripte. Il modello immunocompetente comprende le cellule endoteliali della vena ombelicale umana (HUVEC), le cellule epiteliali intestinali Caco-2, i macrofagi residenti nei tessuti e le cellule dendritiche, che si auto-organizzano all'interno del tessuto, rispecchiando le caratteristiche della mucosa intestinale umana. Un aspetto unico di questa piattaforma è la sua capacità di integrare le cellule immunitarie primarie umane circolanti, migliorando la rilevanza fisiologica. Il modello è progettato per studiare la risposta del sistema immunitario intestinale alla colonizzazione e all'infezione batterica e fungina. Grazie alle dimensioni della cavità ingrandite, il modello offre diverse letture funzionali come saggi di permeazione, rilascio di citochine e infiltrazione di cellule immunitarie ed è compatibile con la misurazione in immunofluorescenza di strutture 3D formate dallo strato di cellule epiteliali. Fornisce informazioni complete sulla differenziazione e la funzione cellulare. La piattaforma intestino su chip ha dimostrato il suo potenziale nel chiarire le complesse interazioni tra surrogati di un microbiota vivente e tessuto ospite umano all'interno di una piattaforma di biochip microfisiologica perfusa.
Introduzione
I sistemi Organ-on-Chip (OoC) rappresentano una tecnica emergente di coltura cellulare 3D in grado di colmare il divario tra la coltura cellulare 2D convenzionale e i modelli animali. Le piattaforme OoC sono tipicamente costituite da uno o più compartimenti contenenti cellule tessuto-specifiche cresciute su un'ampia gamma di scaffold come membrane o idrogel1. I modelli sono in grado di imitare una o più funzioni organotipiche definite. Le pompe consentono la perfusione microfluidica continua del terreno di coltura cellulare per la rimozione dei prodotti di scarto cellulare, forniscono nutrienti e fattori di crescita per una migliore differenziazione cellulare e ricreano le condizioni essenziali in vivo. Con l'integrazione delle cellule immunitarie, i sistemi OoC possono imitare la risposta immunitaria umana in vitro2. Ad oggi è stata presentata un'ampia gamma di organi e unità funzionali1. Questi sistemi includono modelli del sistema vascolare3, polmonare4, fegato 2,5 e intestino6 che possono essere facilitati per test farmacologici 5,7 e studi sulle infezioni 6,8.
Qui presentiamo un modello di intestino umano su chip che integra cellule epiteliali umane formando una topografia 3D organotipica di strutture simili a villi e cripte combinate con un rivestimento endoteliale e macrofagi residenti nei tessuti. Il modello viene coltivato in un biochip microfluidicamente perfuso sotto forma di vetrino microscopico. Ogni biochip è costituito da due cavità microfluidiche separate. Ogni cavità è divisa da una membrana porosa in polietilene tereftalato (PET) in una camera superiore e una inferiore. La membrana stessa funge anche da impalcatura per la crescita delle cellule su ciascun lato. I pori della membrana consentono il crosstalk cellulare e la migrazione cellulare tra gli strati cellulari. Ogni camera è accessibile da due porte femmina delle dimensioni di un luer lock. Opzionalmente, un'ulteriore porta delle dimensioni di un mini-luer lock può fornire l'accesso alla camera superiore o inferiore (Figura 1).
La piattaforma OoC offre una serie di letture che possono essere ottenute da un singolo esperimento. L'intestino-on-chip è progettato per combinare la coltura cellulare 3D perfusa, l'analisi degli effluenti e la microscopia a fluorescenza per valutare l'espressione dei marcatori cellulari, i tassi di metabolizzazione, la risposta immunitaria, la colonizzazione e l'infezione microbica e la funzione barriera 3,6,8. Il modello include cellule immunitarie residenti nei tessuti e il contatto diretto di microrganismi viventi con il tessuto ospite, che rappresenta un vantaggio rispetto ad altri modelli pubblicati9. Inoltre, le cellule epiteliali si auto-organizzano in strutture tridimensionali che forniscono un'interfaccia fisiologicamente rilevante per la colonizzazione con un microbiota vivente6.
Protocollo
Questo protocollo richiede l'accesso a ~20 ml di sangue fresco per biochip da donatori sani per isolare i monociti umani primari. Tutti i donatori hanno dato il consenso informato scritto a partecipare a questo studio, che è stato approvato dal comitato etico dell'Ospedale Universitario di Jena (numero di autorizzazione 2018-1052-BO). Per i dettagli sui materiali, fare riferimento alla Tabella dei materiali. Per i dettagli sulla composizione di tutte le soluzioni e i mezzi, fare riferimento alla Tabella 1.
1. Osservazioni generali sulla manipolazione dei biochip
- Separare con cura una striscia di serbatoi e staccare i coperchi utilizzando un coltello riscaldato per ottenere serbatoi e coperchi singoli. Allarga il foro del coperchio in modo che il tubo in silicone si adatti perfettamente.
- Il tubo in silicone ha un diametro interno di 0,5 mm, è asimmetrico ed è separato in lati più lunghi (20 cm) e più corti (12 cm) da due tappi peristaltici della pompa. Assemblare due tubi di ciascuna simmetria per biochip collegando un tubo a un connettore luer lock maschio e il coperchio sul lato opposto del tubo. Assembla anche quattro serbatoi per biochip.
NOTA: Preparare in anticipo i tubi e i serbatoi e sterilizzarli in autoclave prima dell'uso. Poiché il tubo in silicone ha una durata limitata, sostituirlo dopo 3-5 esperimenti. Per alcuni interessi di ricerca, come i test sui farmaci, si consiglia di preparare nuovi tubi per ogni esperimento. Molti passaggi di questo protocollo vengono eseguiti in parallelo. fare riferimento alla figura panoramica, che evidenzia i diversi passaggi eseguiti in un giorno, come mostrato nella Figura 2.
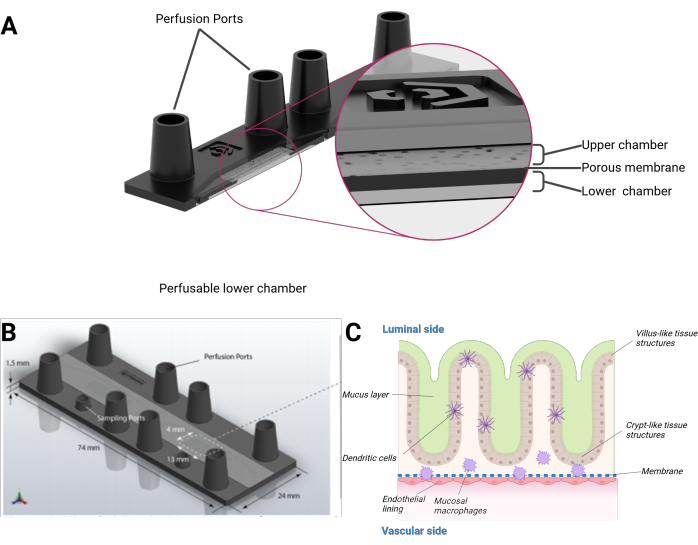
Figura 1: Rappresentazione schematica del modello intestino su chip. (A) Il biochip è presentato in una vista trasversale. (B) È visibile la dimensione dell'intero biochip e della membrana piatta e rimovibile in PET. Il volume totale della camera superiore, comprese le porte femmina di dimensioni luer lock, è rispettivamente di 290 μl e 270 μl per la camera inferiore. (C) Si può vedere una composizione schematica del biochip intestinale, l'epitelio tridimensionale di crescita simile a strutture simili a villi e cripte che includono cellule immunitarie differenziate e uno strato di muco. L'altro lato della membrana in PET è ricoperto da un monostrato endoteliale. Clicca qui per visualizzare una versione più grande di questa figura.
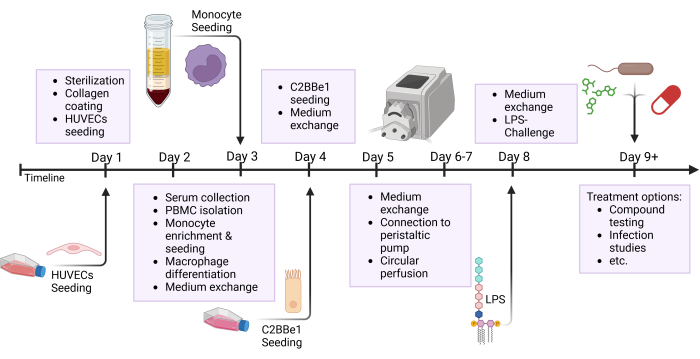
Figura 2: Panoramica schematica della tempistica di costruzione del modello e dell'impostazione sperimentale. Questa figura mostra la panoramica schematica del protocollo presentato. Procedure importanti, come la semina delle cellule e la provocazione epiteliale con LPS, sono indicate da frecce. Abbreviazioni: HUVEC = cellule endoteliali venose ombelicali umane; LPS = lipopolisaccaride. Clicca qui per visualizzare una versione più grande di questa figura.
2. Sterilizzazione con biochip
- Riempire una capsula di Petri di vetro sterile, del diametro di 15 cm, con etanolo non denaturato al 70%. Posizionare il biochip all'interno in modo che tutte le porte del biochip siano completamente coperte dalla soluzione di etanolo.
- Due volte per porta, aspirare 1 mL di etanolo al 70% attraverso tutte le camere del chip. Incubare per 45-60 minuti a temperatura ambiente (RT).
NOTA: Assicurarsi che l'aria non sia intrappolata all'interno del biochip. Da questo punto in poi, l'aria non dovrebbe entrare nel sistema di biochip e le cavità dovrebbero rimanere piene di liquido. - Rimuovere l'etanolo nella capsula di Petri e sostituirlo con acqua sterile a doppia distillazione (ddH2O) fino a coprire completamente tutte le porte. Anche in questo caso, due volte per porta, aspirare 1 mL di ddH2O attraverso la cavità del biochip. Rinfrescare il ddH2O nella piastra di Petri e ripetere la procedura.
- Rimuovere tutto il liquido dalla capsula di Petri. Successivamente, conservare i biochip completamente sterilizzati all'interno della capsula di Petri chiusa ogni volta che si trovano al di fuori di un ambiente sterile. Aggiungere un piccolo serbatoio (ad esempio, il coperchio di una provetta da 50 mL) di 2-5 mL ddH2O alla capsula di Petri per ridurre l'evaporazione del liquido all'interno del biochip.
NOTA: I biochip possono essere sterilizzati fino a 3 giorni prima se conservati in un ambiente sterile fino all'uso. Ciò consente una flessibilità nel carico di lavoro in un solo giorno.
3. Raccolta e semina degli HUVEC
NOTA: Le cellule endoteliali venose ombelicali umane (HUVEC) sono state isolate dai cordoni ombelicali come pubblicato prima di10.
- Prima di seminare gli HUVEC, rivestire la membrana con collagene umano IV. A tal fine, preparare una diluizione 1:100 di una soluzione madre di collagene (Tabella 1) in soluzione salina tamponata con fosfato di Dulbecco contenente magnesio e calcio (PBS +/+). Aggiungere 350 μl della soluzione madre diluita nella rispettiva camera. Incubare per 5 minuti a RT.
NOTA: Se si maneggia il biochip nella cappa sterile, si consiglia di posizionare un fazzoletto sterile sotto di esso per raccogliere il mezzo in eccesso. - Sciacquare due volte tutte le camere con 350 μL di PBS +/+ per eliminare il collagene e l'acido acetico rimanenti. Quindi, aggiungere 350 μl di terreno di crescita delle cellule endoteliali (EC) in ciascuna camera.
NOTA: Da qui, i biochip sono pronti per la semina cellulare e possono essere conservati a 37 °C fino all'uso. - Utilizzare gli HUVEC ai passaggi 1-3 all'80-90% di confluenza cellulare. Coltivare HUVECs in EC-medium contenente una miscela di integratori definita fornita dal produttore. Un'immagine rappresentativa in campo chiaro di una coltura cellulare HUVEC è presentata nella Figura 3A.
NOTA: A seconda del donatore, gli HUVEC dei passaggi superiori possono iniziare a dedifferenziarsi e non possono formare in modo affidabile un monostrato denso e confluente all'interno del biochip. L'uso di antibiotici, ad esempio 100 U/mL di penicillina e 100 μg/mL di streptomicina, è facoltativo ma raccomandato come supplemento al terreno EC per prevenire la contaminazione microbica. - Rimuovere il terreno di coltura cellulare da un pallone di coltura cellulare T25 e lavare delicatamente le cellule con 3-5 mL di soluzione salina tamponata con fosfato di Dulbecco senza magnesio e calcio (PBS -/-). Rimuovere il PBS -/- e aggiungere 1 mL di reagente di dissociazione della tripsina (Tabella 1). Incubare per 5 minuti a 37 °C fino a quando le cellule non si staccano dal pallone di coltura cellulare.
- Trasferire le cellule staccate in una provetta utilizzando 9 mL di siero fetale bovino al 5% (FBS) in PBS -/-. Centrifugare a 350 × g per 5 minuti a RT. Rimuovere il surnatante, risospendere in 1 mL di EC-medium e determinare il numero di cellule. Regolare la concentrazione delle cellule a 0,4 × 106 cellule per 150 μl (semina nella camera inferiore) o per 250 μl (semina nella camera superiore).
- Aggiungere il rispettivo volume di celle alla camera. Se si semina nella camera inferiore, chiudere tutte le porte e posizionare immediatamente il biochip capovolto in modo che le cellule cadano sulla membrana in PET. Incubare i biochip in un incubatore umidificato a 37 °C e 5% di CO2.
- Eseguire una sostituzione del mezzo della camera contenente HUVEC con 350 μL di EC-medium dopo 24 ore. Non è necessario sostituire il fluido nella camera opposta.
4. Raccolta di siero umano e isolamento di monociti derivati da cellule mononucleate del sangue periferico (PBMC)
NOTA: Le PBMC sono state isolate come descritto in Mosig et al.11.
- Prelevare sangue venoso umano da donatori sani. Per ogni biochip, procurarsi un minimo di 10 ml di sangue intero in provette per la raccolta del sangue contenenti silicati per la raccolta del siero. Dopo la coagulazione completa, centrifugare le provette per il prelievo del sangue a 2.500 × g per 10 minuti a RT. Raccogliere il siero, l'aliquota e conservare a -20 °C fino a nuovo utilizzo.
- Procurarsi un minimo di 10 mL di sangue intero dallo stesso donatore in provette per la raccolta del sangue contenenti EDTA per l'isolamento della PBMC. Miscelare delicatamente il sangue non coagulato 1:1 con iso-tampone (Tabella 1) per inversione e sovrapporre lentamente 35 mL di questa miscela sopra 15 mL di un terreno a gradiente di densità con una densità di 1,077 g/mL in una provetta da 50 mL.
- Centrifugare a 800 × g per 20 minuti senza freni a RT. Prelevare con cautela lo strato di cellule immunitarie risultante, che appare sopra il mezzo di gradiente di densità, e trasferirlo in una nuova provetta da 50 mL. Riempire fino a 50 mL con iso-tampone freddo e lavare le cellule mediante inversione delicata.
- Centrifugare a 200 × g per 8 min senza freno a 4 °C. Scartare il surnatante e risospendere in 10 mL di iso-tampone per gradiente di densità. Facoltativo: raggruppare le PBMC di un donatore se vengono eseguiti più gradienti in parallelo.
- Centrifugare a 150 × g per 8 min a 4 °C. Scartare il surnatante e risospendere il pellet in 10 mL di iso-tampone per gradiente di densità. Ripetere il passaggio 4.4 della centrifugazione. Infine, scartare il surnatante e risospendere le cellule in 2 mL di terreno di differenziazione dei monociti (Tabella 1).
NOTA: L'aggiunta di M-CSF e GM-CSF rafforza la differenziazione dei monociti isolati verso macrofagi derivati da monociti e cellule dendritiche derivate da monociti (in combinazione con lipopolisaccaride [LPS], che viene aggiunto in un punto successivo di questo protocollo). L'uso di antibiotici, ad esempio 100 U/mL di penicillina e 100 μg/mL di streptomicina, è facoltativo ma raccomandato come supplemento del terreno per prevenire la contaminazione microbica. - Determinare il numero di cellule e seminare ~10 × 106 cellule per pozzetto di una piastra a 6 pozzetti in 2 mL di terreno di differenziazione dei monociti (Tabella 1). Incubare in un incubatore umidificato a 37 °C per 1 ora per consentire l'adesione dei monociti alla plastica della piastra a 6 pozzetti.
- Eliminare con cura il surnatante e lavare 2 volte con 2 mL di terreno cellulare ematopoietico preriscaldato per rimuovere le cellule non legate. Incubare a 37 °C per altre 24 ore in terreno di differenziazione monocitaria.
- Per raccogliere i monociti, scartare con cura il surnatante e lavare una volta con 2 ml di PBS preriscaldato -/-. Vedere la Figura 3B per un esempio di un'immagine in campo chiaro della coltura dei monociti a questo punto. Quindi, incubare le cellule per 7 minuti in 1 mL di reagente preriscaldato per il distacco dei monociti (Tabella 1) a 37 °C per rafforzare il distacco dei monociti dalla plastica della piastra a 6 pozzetti.
- Trasferire i monociti staccati in una provetta a basso legame. Opzionale: per ottenere una maggiore resa cellulare, lavare accuratamente la piastra a 6 pozzetti più volte con PBS -/-.
- Centrifugare a 300 × g per 8 minuti a RT. Scartare il surnatante e risospendere in mezzo condizionato EC (Tabella 1). Determinare il numero di cellule e regolare la concentrazione cellulare a 0,1 × 106 cellule per 150 μl (semina nella cavità inferiore) o per 250 μl (semina nella cavità superiore).
NOTA: Essere delicati in tutte le fasi dell'isolamento delle cellule immunitarie e ridurre le forze di taglio per prevenire l'attivazione delle cellule immunitarie. Quando si stabilisce questo isolamento, verificare la purezza dei monociti derivati da PBMC (ad esempio, tramite citometria a flusso). Più del 95% di tutte le cellule dovrebbe essere positivo per i tipici marcatori monocitari come il CD14.
5. Semina di monociti
- Eseguire uno scambio di terreno nella camera contenente HUVEC con 350 μl di terreno preriscaldato condizionato EC.
- Aggiungere 150 μl (camera inferiore) o 250 μl (camera superiore) della sospensione di monociti preparata (vedere punto 4.10) nella stessa camera. Se si semina nella camera inferiore, chiudere tutte le porte e posizionare immediatamente il biochip capovolto in modo che le cellule cadano sullo strato di HUVEC. Incubare il biochip in un incubatore umidificato a 37 °C e 5% di CO2.
- Eseguire uno scambio di terreno nella camera contenente monociti HUVEC + con 350 μL di terreno condizionato EC ogni 24 ore.
6. Raccolta e semina di C2BBe1
NOTA: Le cellule 1 (C2BBe1)12 che esprimono il bordo del pennello Caco-2 vengono utilizzate fino al passaggio 35 e vengono prelevate da fiasche con confluenza dell'80-90%. Un'immagine rappresentativa in campo chiaro di una coltura C2BBe1 è presentata nella Figura 3C.
- Coltivare cellule C2BBe1 in C2-medium (Tabella 1).
NOTA: L'uso di antibiotici, ad esempio 20 μg/mL di gentamicina, è facoltativo ma raccomandato come integrazione del terreno C2 per prevenire la contaminazione microbica. - Rimuovere il terreno di coltura cellulare di un pallone per colture cellulari T25 e lavare delicatamente le cellule con 3-5 ml di PBS -/-. Rimuovere il PBS -/- e aggiungere 1 mL di reagente di dissociazione della tripsina (Tabella 1). Incubare per 5 minuti a 37 °C fino a quando le cellule non si staccano dal pallone di coltura cellulare.
- Trasferire le cellule staccate in una provetta utilizzando 9 mL di siero fetale bovino al 5% (FBS) in PBS -/-. Centrifugare a 350 × g per 5 minuti a RT. Rimuovere il surnatante, risospendere in 1 mL di terreno C2 e determinare il numero di cellule. Regolare la concentrazione delle cellule a 0,5 × 106 cellule per 150 μl (semina nella camera inferiore) o per 250 μl (semina nella camera superiore).
- Prima della semina del C2BBe1, lavare delicatamente la rispettiva camera con 350 μL di C2-medium.
- Aggiungere 150 μl (camera inferiore) o 250 μl (camera superiore) della sospensione C2BBe1 preparata (vedere il passaggio 6.3) nella rispettiva camera. Se si semina nella camera inferiore, chiudere tutte le porte e posizionare immediatamente il biochip capovolto in modo che le cellule cadano sulla membrana in PET. Incubare i biochip in un incubatore umidificato a 37 °C e 5% di CO2.

Figura 3: Morfologia cellulare di HUVEC, monociti e C2BBe1 prima della semina nel biochip. Questa figura mostra immagini rappresentative in campo chiaro delle diverse sorgenti cellulari utilizzate nel protocollo. Le immagini sono state scattate con un microscopio a campo chiaro inverso utilizzando un ingrandimento 10x. Tutti i tipi di cellule, (A) HUVEC, (B) monociti e (C) C2BBe1 sono stati coltivati in colture cellulari monostrato 2D come descritto nelle loro sezioni specifiche del protocollo. Barre di scala = 200 μm. Abbreviazioni: HUVEC = cellule endoteliali venose ombelicali umane; PBMCs = cellule mononucleate del sangue periferico. Clicca qui per visualizzare una versione più grande di questa figura.
7. Collegamento alla pompa peristaltica e perfusione circolare
- Preparare un'incubatrice vuota con l'aggiunta di una pompa peristaltica. Pulire accuratamente tutte le aree dell'incubatrice e pompare con disinfettante per fornire un ambiente quasi sterile.
NOTA: Le pompe peristaltiche possono produrre molto calore durante il funzionamento. In incubatori ben isolati o in laboratori scarsamente climatizzati, il numero di pompe utilizzabili per incubatore potrebbe essere limitato poiché gli incubatori tendono a surriscaldarsi. Due pompe peristaltiche per incubatore devono essere adeguate. - Prima di collegare le provette sterilizzate al biochip, lavare ciascuna provetta con 700 μL di PBS +/+ seguiti da 500 μL di terreno C2-R o terreno condizionato EC. Preparare un tubo di ogni simmetria per mezzo (vedere il passaggio 1.2). Utilizzare il tubo con la breve distanza dal luer lock al tappo della pompa peristaltica per la cavità sinistra e il tubo con l'altra simmetria per la cavità destra.
- Recuperare il biochip dall'incubatore ed eseguire uno scambio di terreno con 350 μl per ogni camera. Rimuovere tutte le spine e riempire tutte le porte fino in cima.
- Partendo dalla cavità sinistra, collegare il primo tubo alla porta destra della camera superiore inserendo l'adattatore luer lock nella porta del biochip. Quindi, collegare il secondo tubo alla porta sinistra della camera inferiore. Ripetere questa procedura per la cavità microfluidica destra.
- Prendi un serbatoio e aggiungi una piccola goccia di terreno di coltura cellulare sul fondo del serbatoio. Quindi, inserire il serbatoio sul lato opposto del primo tubo e ripetere per l'altra camera. Una volta che tutte le porte sono collegate a un tubo o a un serbatoio, riempire i serbatoi con 3,5 mL di terreno di coltura cellulare.
- Posizionare il lato libero del tubo, a cui è attaccato il coperchio, sopra il serbatoio per chiudere il sistema microfluidico di ciascuna camera. In questo stato, trasportare il biochip alla pompa peristaltica.
NOTA: A seconda della distanza dall'incubatore e dall'ambiente del laboratorio, è possibile utilizzare una scatola precedentemente pulita e sterilizzata in autoclave per trasferire i chip nell'incubatore. - Utilizzare i tappi peristaltici della pompa per collegare il tubo alla pompa. Collegare ciascun tubo alla pompa peristaltica in modo tale che il fluido fluisca dal serbatoio nella cavità, nel tubo e attraverso la pompa di nuovo nel serbatoio (Figura 4). Il serbatoio funge da trappola per le bolle nella perfusione circolare e impedisce all'aria di rimanere intrappolata nel sistema. Perfondere ogni camera con una portata di 50 μL/min con conseguente sforzo di taglio di 0,013 dyn/cm2 nella camera superiore e 0,006 dyn/cm2 nella camera inferiore8.
NOTA: Se il mezzo delle cavità inferiore e superiore viene spostato in direzioni opposte, è possibile ottenere una maggiore crescita tridimensionale del tessuto intestinale13. Pertanto, i serbatoi delle cavità superiore e inferiore sono posizionati su lati opposti (Figura 4). La perfusione circolare riduce la quantità di terreno di coltura cellulare necessaria, ma potrebbe potenzialmente provocare l'arricchimento di citochine e metaboliti. Se lo si desidera, è possibile anche una perfusione lineare del biochip. - Perfondere il biochip per 72 ore a 37 °C e 5% di CO2.

Figura 4: Biochip collegato alla pompa peristaltica. Viene presentato un esempio di biochip collegato a una pompa peristaltica. Le cellule epiteliali C2BBe1 sono coltivate nella camera inferiore (il C2-medium rosso si trova nei serbatoi anteriori) mentre le HUVEC sono coltivate nella camera superiore (il terreno EC-condizionato giallastro si trova nei serbatoi nella parte posteriore). I diversi terreni di coltura cellulare non si mescolano a causa della funzione barriera del tessuto cresciuto. Il biochip è collegato alla pompa peristaltica in modo tale che il fluido fluisca dal serbatoio nella cavità. Da qui, il fluido rifluisce nel serbatoio attraverso il tubo attraverso la pompa. Clicca qui per visualizzare una versione più grande di questa figura.
8. Condizionamento LPS della barriera epiteliale
- Dopo 72 h di preperfusione, arrestare la pompa peristaltica e rimuovere il coperchio collegato al tubo di ciascun serbatoio. Posizionalo su un fazzoletto sterile accanto alla pompa.
- Rimuovere tutto il terreno e riempire i serbatoi con 2 ml di terreno appena preparato. Per il lato epiteliale contenente le cellule C2BBe1, aggiungere 100 ng/mL di LPS al terreno.
NOTA: L'LPS aumenta la funzione di barriera del tessuto, stimola i macrofagi derivati dai monociti a migrare nel tessuto epiteliale e consente la differenziazione delle cellule dendritiche derivate dai monociti. - Ricollegare il tubo e i coperchi al serbatoio e continuare la perfusione circolare a una portata di 50 μL/min per altre 24 ore.
NOTA: Da questo punto in poi, il modello di chip può essere utilizzato in esperimenti, test di composti o studi sulle infezioni. Si consiglia uno scambio del mezzo di 2 mL per serbatoio ogni 24 ore.
9. Accesso al tessuto per diversi metodi di lettura
- Raccogliere i surnatanti del terreno di coltura cellulare dai serbatoi in ogni momento della perfusione. Aprire il serbatoio e raccogliere il volume desiderato (vedere i passaggi 8.1-8.3). Utilizzare questi surnatanti per il rilevamento di metaboliti, citochine o altre molecole.
- Per accedere al tessuto, utilizzare un bisturi per eseguire un taglio preciso lungo l'esterno della camera superiore e rimuovere la lamina di incollaggio per aprire la cavità microfluidica. Il tessuto del modello di biochip intestinale è ora accessibile. Tagliare con cura lungo l'esterno della camera microfluidica per staccare la membrana dal biochip. Raccogliere la membrana contenente il tessuto usando una pinzetta.
ATTENZIONE: Prestare attenzione al posizionamento delle dita durante questa fase e lavorare attentamente per evitare incidenti. Si consigliano guanti resistenti al taglio. - In alternativa, raccogliere le cellule da strati separati all'interno del biochip utilizzando soluzioni enzimatiche, ad esempio tripsina o cellule lisate utilizzando tamponi contenenti Triton X-100.
10. Valutazione della permeabilità tramite diffusione di FITC-destrano
NOTA: La funzione barriera del tessuto può essere analizzata tramite un test di permeabilità FITC-destrano dopo lo scollegamento della pompa peristaltica. La valutazione della permeabilità FITC-destrano è stata adattata da Deinhardt-Emmer et al.4.
- Preparare una soluzione madre di isotiocianato di fluoresceina (FITC)-destrano (peso molecolare di 3-5 kDa, Tabella 1).
- Svuotare i serbatoi e scollegare il chip dalla perfusione.
- Eseguire uno scambio di fluido nella camera superiore e inferiore con un mezzo privo di rosso fenolo.
NOTA: Questo passaggio non è necessario se durante l'esperimento è già stato utilizzato un terreno privo di rosso fenolo. - Aggiungere 350 μL di soluzione di FITC-destrano da 1 mg/mL nella camera contenente le cellule C2BBe1.
- Chiudere le porte e incubare il chip per 60 minuti a 37 °C con il lato epiteliale rivolto verso l'alto.
- Trascorso il tempo di incubazione, raccogliere separatamente il terreno di coltura da entrambe le camere del chip e conservarlo a 4 °C, al riparo dalla luce fino alla misurazione.
- Per la misurazione, preparare una curva standard in terreno C2 e nel terreno condizionato EC senza rosso fenolo nell'intervallo da 1.000 μg/mL a 0 μg/mL FITC-destrano con 11 diluizioni seriali consecutive 1:2.
- Trasferire 200 μl di ciascun campione in una piastra nera a 96 pozzetti con fondo trasparente. Misurare la fluorescenza con un lettore di micropiastre a una lunghezza d'onda di eccitazione di 495 nm e una lunghezza d'onda di emissione di 517 nm.
- Utilizzare la curva standard per calcolare la concentrazione di destrano FITC dei campioni e, quindi, il coefficiente di permeabilità.
11. Colorazione in immunofluorescenza
NOTA: Il tessuto vivente può essere studiato al microscopio. Per facilitare la manipolazione, si consiglia il distacco del biochip dalla pompa peristaltica e l'uso di obiettivi a lunga distanza su un microscopio invertito. Come analisi degli endpoint, il tessuto può essere fissato all'interno del biochip per procedure come la colorazione in immunofluorescenza.
- Arrestare la pompa peristaltica e aprire i serbatoi di tutte le cavità. Svuotare i serbatoi e scollegare i tubi, così come i serbatoi dal biochip.
- Due volte per camera, lavare le cavità microfluidiche con 500 μL di PBS +/+ freddo. Aggiungere 500 μl di metanolo ghiacciato a tutte le cavità e incubare per 15 minuti a -20 °C. Quindi, due volte per cavità, lavare la camera microfluidica con 500 μL di PBS +/+.
NOTA: Sono adatti anche altri metodi di fissaggio, come il fissaggio con paraformaldeide al 4% o il fissativo di Carnoy. Dopo il fissaggio, i chip possono essere conservati a 4 °C o procedere direttamente alla colorazione con fluorescenza immunitaria. ATTENZIONE: Le sostanze chimiche fissanti come il metanolo o la paraformaldeide sono tossiche. Eseguire le rispettive attività sotto una cappa aspirante e raccogliere i rifiuti di conseguenza. - Aprire il chip come descritto al punto 9.2 per accedere al tessuto. Tagliare la membrana in PET contenente tessuto in un massimo di tre pezzi da colorare in parallelo con diversi pannelli immunologici.
- Trasferire ciascuno dei pezzi della membrana in una piastra separata a 24 pozzetti contenente una soluzione bloccante e permeabilizzante (Tabella 1) utilizzando una pinzetta di precisione. Assicurarsi che lo strato cellulare di interesse sia sempre rivolto verso l'alto durante l'intero processo di colorazione. Incubare i pezzi della membrana per 30 minuti a RT.
NOTA: I migliori risultati di colorazione si ottengono abbinando il siero all'anticorpo secondario. Ad esempio, se gli anticorpi secondari sono ottenuti da specie caprine, si consiglia l'uso di un normale siero di capra. - Trasferire i pezzi della membrana su un vetrino pulito all'interno di una camera umida. Preparare il pannello di anticorpi primari nella soluzione colorante (Tabella 1) e aggiungere 50 μl a ciascun pezzo di membrana. Incubare per una notte a 4 °C.
NOTA: La concentrazione ottimale di anticorpi e l'efficacia della colorazione possono variare tra produttori e cloni. Si consiglia di testare preventivamente i pannelli di colorazione in una coltura cellulare 2D. - Dopo l'incubazione, trasferire i campioni in una piastra a 24 pozzetti e lavare delicatamente le membrane per 3 x 5 minuti con una soluzione di lavaggio (Tabella 1).
- Ancora una volta, trasferisci i pezzi della membrana su un vetrino pulito all'interno di una camera umida. Preparare il pannello di anticorpi secondari in soluzione colorante (Tabella 1) e aggiungere 50 μl a ciascun pezzo di membrana. Se necessario, aggiungere un colorante di contrasto nucleare come il 4',6-diamidino-2-fenilindolo (DAPI) o Hoechst. Incubare per 30 minuti a RT.
NOTA: Durante l'utilizzo dei fluorofori, proteggere i campioni dalla luce per evitare il fotosbiancamento e aumentare la qualità dell'immagine. - Dopo l'incubazione, trasferire i campioni in una piastra a 24 pozzetti e lavare delicatamente le membrane 2 x 5 minuti con una soluzione di lavaggio (Tabella 1). Quindi, lavare una volta con PBS +/+ per 5 minuti.
- Montare i pezzi della membrana su un vetrino di vetro pulito utilizzando un mezzo di montaggio a fluorescenza e un vetro di copertura. Conservare a 4 °C fino all'imaging microscopico.
Risultati
Questi risultati rappresentativi mostrano i distinti strati di tessuto del modello intestino-su-chip. Sono colorati con immunofluorescenza come descritto nella sezione 11 del protocollo. Le immagini sono state scattate con un microscopio a epifluorescenza o a fluorescenza confocale come z-stack ed elaborate in una proiezione ortogonale. Vedere la Tabella dei materiali per i dettagli sulla configurazione microscopica e sul software. La Figura 5 mostra lo strato vascolare, un ...
Discussione
Il protocollo presentato descrive in dettaglio i passaggi necessari per generare un modello di intestino su chip immunocompetente. Sono state descritte tecniche specifiche e possibili metodi di lettura come la microscopia a immunofluorescenza, l'analisi di citochine e metaboliti, la citometria a flusso, l'analisi di proteine e genetiche e la misurazione della permeabilità.
Il modello descritto è costituito da HUVEC primari, macrofagi derivati da monociti e cellule dendritiche derivate da mon...
Divulgazioni
M.R. è CEO di Dynamic42 GmbH e detiene il capitale della società. A.S.M. è consulente scientifico di Dynamic 42 GmbH e detiene il capitale della società.
Riconoscimenti
Il lavoro è stato sostenuto finanziariamente dal Centro di ricerca collaborativa PolyTarget 1278 (numero di progetto 316213987) a V.D.W. e A.S.M. A.F. e A.S.M. riconoscono inoltre il sostegno finanziario del Cluster di Eccellenza "Balance of the Microverse" nell'ambito della Strategia di Eccellenza della Germania - EXC 2051 - Project-ID 690 390713860. Ringraziamo Astrid Tannert e il Jena Biophotonic and Imaging Laboratory (JBIL) per averci fornito l'accesso al loro microscopio a scansione laser confocale ZEISS LSM980. La Figura 1C e la Figura 2 sono state create con Biorender.com.
Materiali
| Name | Company | Catalog Number | Comments |
| 96-well plate black, clear bottom | Thermo Fisher | 10000631 | Consumables |
| Acetic acid | Roth | 3738.4 | Chemicals |
| Alexa Fluor 488 AffiniPure, donkey, anti-mouse IgG (H+L) | Jackson Immuno Research | 715-545-150 | Secondary Antibody Vascular Staining and Epithelial Staining |
| Alexa Fluor 647 AffiniPure, donkey, anti-rabbit IgG (H+L) | Jackson Immuno Research | 711-605-152 | Secondary Antibody Epithelial Staining |
| Alexa Fluor 647, donkey, anti-rabbit IgG (H+L) | Thermo Fisher Scientific, Invitrogen | A31573 | Secondary Antibody Vascular Staining |
| Axiocam ERc5s camera | Zeiss | 426540-9901-000 | Technical equipment |
| Basal Medium MV, phenol red-free | Promocell | C-22225 | Cell culture consumables |
| Biochip | Dynamic 42 | BC002 | Microfluidic consumables |
| BSA fraction V | Gibco | 15260-037 | Cell culture consumables |
| C2BBe1 (clone of Caco-2) | ATCC | CRL-2102 | Epithelial Cell Source |
| Chloroform | Sigma | C2432 | Chemicals |
| CO2 Incubator | Heracell | 150i | Technical equipment |
| Collagen IV from human placenta | Sigma-Aldrich | C5533 | Cell culture consumables |
| Coverslips (24 x 40 mm; #1.5) | Menzel-Gläser | 15747592 | Consumables |
| Cy3 AffiniPure, donkey, anti-goat IgG (H+L) | Jackson Immuno Research | 705-165-147 | Secondary Antibody Vascular Staining |
| Cy3 AffiniPure, donkey, anti-rat IgG (H+L) | Jackson Immuno Research | 712-165-150 | Secondary Antibody Epithelial Staining |
| DAPI (4',6-Diamidin-2-phenylindol, Dilactate) | Thermo Fisher Scientific, Invitrogen | D3571 | Vascular and Epithelial Staining |
| Descosept PUR | Dr.Schuhmacher | 00-323-100 | Cell culture consumables |
| DMEM high glucose | Gibco | 41965-062 | Cell culture consumables |
| DMEM high glucose w/o phenol red | Gibco | 31053028 | Cell culture consumables |
| DPBS (-/-) | Gibco | 14190-169 | Cell culture consumables |
| DPBS (+/+) | Gibco | 14040-133 | Cell culture consumables |
| EDTA solution | Invitrogen | 15575-038 | Cell culture consumables |
| Endothelial Cell Growth Medium | Promocell | C-22020 | Cell culture consumables |
| Endothelial Cell Growth Medium supplement mix | Promocell | C-39225 | Cell culture consumables |
| Ethanol 96%, undenatured | Nordbrand-Nordhausen | 410 | Chemicals |
| Fetal bovine Serum | invitrogen | 10270106 | Cell culture consumables |
| Fluorescein isothiocyanate (FITC)-dextran (3-5 kDa) | Sigma Aldrich | FD4-100MG | Chemicals |
| Fluorescent Mounting Medium | Dako | S3023 | Chemicals |
| Gentamycin (10mg/mL) | Sigma Aldrich | G1272 | Cell culture consumables |
| GlutaMAX Supplement (100x) | Gibco | 35050061 | Cell culture consumables |
| Histopaque | Sigma-Aldrich | 10771 | Cell culture consumables |
| Hoechst (bisBenzimid) H33342 | Sigma-Aldrich | 14533 | Epithelial Staining |
| Holotransferrin (5mg/mL) Transferrin, Holo, Human Plasma | Millipore | 616397 | Cell culture consumables |
| Human recombinant GM-CSF | Peprotech | 300-30 | Cell culture consumables |
| Human recombinant M-CSF | Peprotech | 300-25 | Cell culture consumables |
| Illumination device | Zeiss | HXP 120 C | Fluorescence Microscope Setup |
| Laser Scanning Microscope | Zeiss | CLSM980 | Fluorescence Microscope Setup |
| Lidocain hydrochloride | Sigma-Aldrich | L5647 | Cell culture consumables |
| Lipopolysaccharide (LPS) | Sigma | L2630 | Cell culture consumables |
| Loftex Wipes | Loftex | 1250115 | Consumables |
| Low attachment tubes (PS, 5 mL) | Falcon | 352052 | Consumables |
| Luer adapter for the top cap (M) | Mo Bi Tec | M3003 | Microfluidic consumables |
| Male mini luer plugs, row of four,PP, opaque | Microfluidic chipshop | 09-0556-0336-09 | Microfluidic consumables |
| MEM Non-Essential Amino Acids Solution | Gibco | 11140 | Cell culture consumables |
| Methanol | Roth | 8388.2 | Chemicals |
| Microscope | Zeiss | Axio Observer 5 | Fluorescence Microscope Setup |
| Microscope slides | Menzel | MZ-0002 | Consumables |
| Monoclonal, mouse, anti-human CD68 Antibody (KP1) | Thermo Fisher Scientific, Invitrogen | 14-0688-82 | Primary Antibody Vascular Staining |
| Monoclonal, rat, anti-human E-Cadherin antibody (DECMA-1) | Sigma-Aldrich, Millipore | MABT26 | Primary Antibody Epithelial Staining |
| Multiskan Go plate reader | Thermo Fisher | 51119300 | Technical equipment |
| Normal donkey serum | Biozol | LIN-END9010-10 | Chemicals |
| Optical Sectioning | Zeiss | ApoTome | Fluorescence Microscope Setup |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140-122 | Cell culture consumables |
| Plugs | Cole Parmer | GZ-45555-56 | Microfluidic consumables |
| Polyclonal, goat, anti-human VE-Cadherin Antibody | R&D Systems | AF938 | Primary Antibody Vascular Staining |
| Polyclonal, rabbit, anti-human Von Willebrand Factor Antibody | Dako | A0082 | Primary Antibody Vascular Staining |
| Polyclonal, rabbit, anti-human ZO-1 antibody | Thermo Fisher Scientific, Invitrogen | 61-7300 | Primary Antibody Epithelial Staining |
| Power Supply Microscope | Zeiss | Eplax Vp232 | Fluorescence Microscope Setup |
| Primovert microscope | Zeiss | 415510-1101-000 | Technical equipment |
| Reglo ICC peristaltic pump | Ismatec | ISM4412 | Technical equipment |
| SAHA (Vorinostat) | Sigma Aldrich | SML0061-25MG | Chemicals |
| Saponin | Fluka | 47036 | Chemicals |
| S-Monovette, 7.5 mL Z-Gel | Sarstedt | 01.1602 | Consumables |
| S-Monovette, 9.0 mL K3E | Sarstedt | 02.1066.001 | Consumables |
| Sodium Pyruvate | Gibco | 11360-088 | Cell culture consumables |
| Tank 4.5 mL | ChipShop | 10000079 | Microfluidic consumables |
| Trypane blue stain 0.4% | Invitrogen | T10282 | Cell culture consumables |
| Trypsin | Gibco | 11538876 | Cell culture consumables |
| Tubing | Dynamic 42 | ST001 | Microfluidic consumables |
| Tweezers (Präzisionspinzette DUMONT abgewinkelt Inox08, 5/45, 0,06 mm) | Roth | K343.1 | Consumables |
| Wheat Germ Agglutinin (WGA) | Thermo Fisher Scientific, Invitrogen | W32464 | Epithelial Staining |
| X-VIVO 15 | Lonza | BE02-060F | Cell culture consumables, Hematopoietic cell medium |
| Zellkultur Multiwell Platten, 24 Well, sterile | Greiner Bio-One | 662 160 | Consumables |
| Zellkultur Multiwell Platten, 6 Well, sterile | Greiner Bio-One | 657 160 | Consumables |
| Zen Blue Software | Zeiss | Version 3.7 | Microscopy Software |
Riferimenti
- Alonso-Roman, R., et al. Organ-on-chip models for infectious disease research. Nat Microbiol. 9 (4), 891-904 (2024).
- Fahrner, R., Groger, M., Settmacher, U., Mosig, A. S. Functional integration of natural killer cells in a microfluidically perfused liver on-a-chip model. BMC Res Notes. 16 (1), 285 (2023).
- Raasch, M., et al. Microfluidically supported biochip design for culture of endothelial cell layers with improved perfusion conditions. Biofabrication. 7 (1), 015013 (2015).
- Deinhardt-Emmer, S., et al. Co-infection with Staphylococcus aureus after primary influenza virus infection leads to damage of the endothelium in a human alveolus-on-a-chip model. Biofabrication. 12 (2), 025012 (2020).
- Kaden, T., et al. Generation & characterization of expandable human liver sinusoidal endothelial cells and their application to assess hepatotoxicity in an advanced in vitro liver model. Toxicology. 483, 153374 (2023).
- Maurer, M., et al. A three-dimensional immunocompetent intestine-on-chip model as in vitro platform for functional and microbial interaction studies. Biomaterials. 220, 119396 (2019).
- Hoang, T. N. M., et al. Invasive aspergillosis-on-chip: A quantitative treatment study of human aspergillus fumigatus infection. Biomaterials. 283, 121420 (2022).
- Kaden, T., et al. Modeling of intravenous caspofungin administration using an intestine-on-chip reveals altered Candida albicans microcolonies and pathogenicity. Biomaterials. 307, 122525 (2024).
- Shah, P., et al. A microfluidics-based in vitro model of the gastrointestinal human-microbe interface. Nat Commun. 7, 11535 (2016).
- Jaffe, E. A., Nachman, R. L., Becker, C. G., Minick, C. R. Culture of human endothelial cells derived from umbilical veins. Identification by morphologic and immunologic criteria. J Clin Invest. 52 (11), 2745-2756 (1973).
- Mosig, S., et al. Different functions of monocyte subsets in familial hypercholesterolemia: Potential function of cd14+ cd16+ monocytes in detoxification of oxidized ldl. FASEB J. 23 (3), 866-874 (2009).
- Peterson, M., Mooseker, M. Characterization of the enterocyte-like brush border cytoskeieton of the c2bbe clones of the human intestinal cell line, caco-2. J Cell Sci. 102, 581-600 (1992).
- Shin, W., Hinojosa, C. D., Ingber, D. E., Kim, H. J. Human intestinal morphogenesis controlled by transepithelial morphogen gradient and flow-dependent physical cues in a microengineered gut-on-a-chip. iScience. 15, 391-406 (2019).
- Kim, H. J., Ingber, D. E. Gut-on-a-chip microenvironment induces human intestinal cells to undergo villus differentiation. Integr Biol (Camb). 5 (9), 1130-1140 (2013).
- Kim, H. J., Huh, D., Hamilton, G., Ingber, D. E. Human gut-on-a-chip inhabited by microbial flora that experiences intestinal peristalsis-like motions and flow. Lab Chip. 12 (12), 2165-2174 (2012).
- Karra, N., Fernandes, J., James, J., Swindle, E. J., Morgan, H. The effect of membrane properties on cell growth in an 'airway barrier on a chip'. Organs-on-a-Chip. 5, 10025 (2023).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon