Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Imágenes de transferencia de energía por resonancia de Förster de células vivas de la dinámica de activación de Akt regulada metabólicamente en células HepG2
En este artículo
Resumen
Aquí, presentamos un protocolo para cuantificar la dinámica espacio-temporal de la activación y fosforilación de Akt en células HepG2 vivas. Las imágenes de transferencia de energía por resonancia (FRET) de Förster son una poderosa herramienta que proporciona información valiosa sobre las vías de señalización de la insulina y la regulación metabólica en las células cancerosas.
Resumen
La activación de Akt regulada metabólicamente es un nodo crítico en la cascada de señalización de insulina y proporciona información valiosa sobre la relación entre la diabetes y el cáncer. Para cuantificar con precisión la actividad de Akt en células HepG2, desarrollamos un protocolo robusto y reproducible que utiliza la transferencia de energía por resonancia de Förster (FRET) con biosensores específicos de Akt codificados genéticamente. Este protocolo describe los pasos detallados para el cultivo celular, la preparación de la placa de imágenes y la transfección de células HepG2 para expresar biosensores basados en FRET, junto con pautas específicas para la configuración de hardware y software de microscopio confocal de escaneo láser. Los resultados demostraron patrones únicos de señalización de insulina en las células HepG2, que exhiben un interruptor irreversible caracterizado por la activación constitutiva de Akt con un umbral de encendido definido pero sin umbral de apagado. Por el contrario, los miotubos muestran un interruptor reversible. La activación persistente de Akt en las células HepG2 sugiere mecanismos subyacentes a la resistencia a la insulina y la desregulación metabólica en las células hepáticas, con implicaciones más amplias para comprender la progresión de los trastornos metabólicos y el cáncer. Este protocolo ofrece un marco valioso para explorar las vías de señalización relacionadas con Akt y los comportamientos celulares en diversos contextos de enfermedades.
Introducción
La diabetes mellitus plantea un importante desafío para la salud mundial, caracterizada por resistencia a la insulina y alteración de la homeostasis de la glucosa1. Una comprensión integral de las vías de señalización de la insulina es crucial para dilucidar la fisiopatología de esta enfermedad, ya que la insulina desempeña un papel fundamental en el metabolismo de la glucosa, el crecimiento celulary la supervivencia. Numerosos estudios han demostrado que la señalización de la insulina tiene un impacto significativo en varios tipos de cáncer, relacionando la resistencia a la insulina con la progresión tumoral y los malos resultados de los pacientes 3,4,5,6. Las células HepG2, una línea celular de carcinoma hepatocelular comúnmente utilizada, sirven como un modelo valioso para estudiar la resistencia a la insulina y la interacción entre la desregulación metabólica yel desarrollo del cáncer. Tradicionalmente, los investigadores han visto las respuestas a la insulina como graduadas; Sin embargo, estudios recientes han revelado que las células individuales pueden exhibir respuestas biestables, mostrando transiciones sobresalientes entre la falta de respuesta y la respuesta completa que ocurre en umbrales específicos de concentración de insulina 8,9.
La imagen de transferencia de energía por resonancia de Förster (FRET) es una poderosa herramienta para estudiar la distribución espacio-temporal de biomoléculas en células vivas10. Al extraer información de la dinámica molecular, FRET proporciona información sobre procesos como la activación de Akt en tiempo real, lo que la convierte en una técnica invaluable para el estudio de células vivas11,12. Este método de imagen ha demostrado ser esencial en el estudio de la dinámica celular, particularmente en enfermedades metabólicas y cáncer, donde las interacciones moleculares precisas son cruciales13. FRET también permite la monitorización en tiempo real de las interacciones moleculares, arrojando luz sobre mecanismos como la resistencia a la insulina y la progresión tumoral14,15. Los biosensores FRET son cruciales en la investigación del cáncer para estudiar los microambientes tumorales, la resistencia a los medicamentos y los trastornos metabólicos16. Los métodos de detección de FRET, como la emisión sensibilizada (SE), el blanqueo del aceptor (AB), la microscopía de imágenes de fluorescencia (FLIM) y la espectroscopia, ofrecen ventajas distintas para cuantificar las interacciones moleculares17. SE mide la transferencia de energía entre los fluoróforos donantes y aceptores, lo que resulta en un cambio medible en los espectros de emisión que se correlaciona con la proximidad de las biomoléculas que interactúan18. AB utiliza el fotoblanqueo selectivo del fluoróforo aceptor y rastrea los cambios en la fluorescencia del donante, lo que permite a los investigadores evaluar la cinética de interacción y las distancias19. FLIM evalúa las tasas de decaimiento de fluorescencia del fluoróforo donante, directamente influenciadas por la eficiencia de FRET, para proporcionar mediciones precisas a nanoescala de las interacciones moleculares20.
Utilizando técnicas FRET, recientemente demostramos respuestas de insulina biestable en miotubos derivados de C2C12 8,9,21,22,23,24. Los distintos umbrales de activación y activación de Akt, como descubrimos, sugieren que la dosis-respuesta graduada de insulina en todo el cuerpo oculta la complejidad de la cascada de señalización subcelular a partir del estímulo de la insulina, que culmina en una respuesta de todo o nada a nivel de una sola célula 21,22,23,24. Para probar la presencia de biestabilidad en otros tipos de células, estimulamos las células HepG2 con insulina y registramos su respuesta mediante imágenes FRET de una sola célula. Estimulamos células HepG2 con diferentes concentraciones de insulina y monitoreamos la actividad de Akt a nivel de una sola célula utilizando un biosensor de Akt. El biosensor Akt comprende la proteína fluorescente cian mejorada (ECFP)25 como fluoróforo donante y la variante más brillante de la proteína fluorescente amarilla (YPet)26 como fluoróforo aceptor, unida por un enlazador Eevee que contiene la secuencia peptídica SGRPRTTTFADSCKP. Este péptido actúa como sustrato para el Akt fosforilado (pAkt), optimizado a partir de la glucgenómina sintasa quinasa 3β (GSK3β) humana. En su estado no fosforilado, la separación espacial entre los fluoróforos donantes y aceptores supera el radio de Förster, lo que inhibe la transferencia de energía. Tras la estimulación de la insulina, se produce la fosforilación de Akt y conduce a la fosforilación de SGRPRTTTFADSCKP. Este proceso induce un cambio conformacional que sitúa al donante y al aceptor dentro del radio de Förster, lo que permite FRET27. Como resultado, la intensidad de la señal FRET se correlaciona con la cantidad de moléculas de Akt fosforiladas y permite la cuantificación en tiempo real de las respuestas celulares mediadas por insulina.
Este protocolo, desarrollado inicialmente para estudiar la señalización de la insulina en miotubos derivados de C2C12, se ha aplicado con éxito a las células HepG2 y se ha utilizado en diferentes plataformas de hardware y software, demostrando así su aplicabilidad, adaptabilidad y versatilidad. Las células HepG2 exhiben actividad Akt constitutiva, lo que las convierte en un modelo in vitro ideal para estudiar la señalización de insulina específica del hígado y los procesos metabólicos. Las características clave del protocolo se describen paso a paso en la sección de protocolo.
Protocolo
En la Figura 1 se muestra una descripción general de los pasos experimentales involucrados en la obtención de imágenes de células vivas FRET para monitorear la fosforilación de Akt en células HepG2 individuales.
1. Adquisición, propagación y purificación de plásmidos
NOTA: En esta sección se describen los pasos esenciales para adquirir, amplificar y purificar el plásmido necesario para el análisis FRET de una sola célula.
- Utilice el plásmido pEevee-iAkt-NES-YPet (Figura 2A).
NOTA: El plásmido fue generosamente proporcionado por el Prof. Kazuhiro Aoki en el Instituto Nacional de Biología Básica (NIBB), Japón.Los mapas de plásmidos para los biosensores FRET utilizados para el monitoreo de Akt y sus respectivos controles se muestran en la Figura 2. El pEevee-iAkt-NES-ECFP (donante; Figura 2B) y pEevee-iAkt-NES-Ypet (aceptante; Figura 2C) Los plásmidos se utilizan como controles de calibración durante los experimentos FRET9.
NOTA: La Figura 3 muestra la composición y el mecanismo del biosensor FRET intramolecular. - Para propagar el plásmido, realice la transformación bacteriana utilizando células de E. coli químicamente competentes (consulte la tabla de materiales). Colocar las células transformadas en agar Luria-Bertani (LB) que contenga 100 μg/mL de ampicilina e incubar durante la noche a 37 °C. Al día siguiente, seleccione una colonia resistente a la ampicilina y cultívela en caldo LB suplementado con 100 μg/mL de ampicilina para la amplificación de plásmidos.
- Para purificar el ADN plasmídico, utilice un kit de purificación de ADN plasmídico disponible en el mercado (consulte la Tabla de materiales) para obtener una alta pureza y rendimiento. Evalúe la calidad del ADN midiendo las relaciones de absorbancia (A260/A280 y A260/A230) utilizando un espectrofotómetro (ver Tabla de Materiales). Las relaciones entre 1,8-2,0 y 2,0-2,2 se consideran óptimas. Verifique la integridad del plásmido a través de la electroforesis en gel de agarosa para asegurarse de que es adecuado para la transfección.
2. Procedimiento de cultivo celular
NOTA: Realice todos los procedimientos de cultivo celular dentro de una campana de flujo laminar para mantener un ambiente estéril y evitar la contaminación. El flujo de trabajo de cultivo celular HepG2 se muestra en la Figura 4. Los medios completos para células HepG2 consisten en un medio esencial mínimo (MEM), un 10 % de suero fetal bovino (FBS), un 1 % de aminoácidos no esenciales (NEAA), 1 mM de piruvato de sodio, 2 mM de suplemento de L-glutamina, 100 U/mL de penicilina-estreptomicina y 2,5 μg/mL de solución antibiótica y antimicótica (consulte la Tabla de materiales, Tabla 1).
- Descongele rápidamente las células HepG2 congeladas colocando el vial en una termobatidora a 37 °C o en un baño de agua hasta que se descongele por completo.
- Transfiera las células descongeladas a un tubo cónico de 15 mL que contenga 10 mL de medio de crecimiento completo (consulte la Tabla de materiales).
NOTA: Precaliente todo el medio de crecimiento a 37 °C antes de usarlo para minimizar el choque térmico en las células. - Centrifugar el tubo a 200 x g durante 5 min para pellet las células.
- Aspire cuidadosamente el sobrenadante con una punta de pipeta de diámetro ancho para evitar perturbar el pellet de la célula.
- Vuelva a suspender el pellet en 10 mL de medio de crecimiento completo fresco.
- Transfiera la suspensión celular a un matraz de cultivo de tejidos de 75 cm².
- Incubar el matraz a 37 °C en una atmósfera humidificada con 5% de CO2
NOTA: Observe las células durante las próximas 24-48 h para confirmar la adhesión y evaluar la recuperación. No moleste las celdas durante al menos 4 horas para asegurar una fijación adecuada antes de cambiar el medio. Evite abrir la incubadora con frecuencia durante las primeras 4 horas, ya que esto puede interrumpir la unión de las células. Siga los protocolos institucionales de bioseguridad y utilice el equipo de protección personal (EPP) adecuado para mantener un entorno de trabajo seguro durante todos los procedimientos de cultivo celular. Subcultivo de células HepG2 cuando alcanzan un 70%-80% de confluencia para mantener condiciones óptimas de crecimiento y evitar el hacinamiento, que puede afectar la viabilidad celular y el potencial de crecimiento. - Para el subcultivo, aspire el medio y enjuague las células una vez con 5 mL de solución salina tamponada con fosfato (PBS).
- Añadir 1 mL de tripsina al 0,25% para cubrir la monocapa celular (ver Tabla de Materiales).
NOTA: Asegúrese de que la tripsina esté precalentada a 37 °C para una actividad óptima. No tripsiníes en exceso las células, ya que esto puede reducir la viabilidad. Monitoree las células bajo un microscopio para confirmar el desprendimiento. - Incubar a 37 °C durante unos 5 min.
- Cuando las células se desprendan, agregue 2 mL de medio completo para neutralizar la tripsina y recoja las células mediante pipeteo.
- Pipetea suavemente la suspensión celular para romper los grumos y lograr una suspensión de una sola celda.
- Agregue 3 mL de medio completo a cada matraz nuevo, luego transfiera las células en una proporción de división de 1:2 a cada matraz.
NOTA: Para los primeros pasajes, divida las células en una dilución de 1:2. Después de los pasajes 4-5, se pueden realizar diluciones de 1:4 o 1:5 según corresponda. - Incubar las células a 37 °C en una atmósfera humidificada con 5% deCO2.
NOTA: Revise las celdas después de 24 horas para confirmar la adhesión y evaluar la recuperación. - Para el cultivo de rutina, reemplace el medio de cultivo cada 2-3 días o antes si el indicador de pH cambia de rosa a amarillo, lo que indica acidificación.
NOTA: No permita que el medio se vuelva demasiado ácido, ya que esto puede dañar las células. - Revise la morfología celular regularmente bajo un microscopio para garantizar la salud de las células.
NOTA: Para almacenamiento a corto plazo, congele las células HepG2 a -80 °C; Para almacenamiento a largo plazo, almacene en nitrógeno líquido. La composición del medio de congelación utilizado en este experimento se describe en la (Tabla 2).
3. Recubrimiento de placas de imagen con poli-l-lisina
- Utilice 1 mL de una solución de poli-L-lisina de 0,1 mg/mL por placa de imagen para cubrir toda la superficie (ver Tabla de Materiales).
NOTA: Ajuste la concentración de poli-L-lisina en función de los requisitos específicos del tipo de celda. - Meca el plato suavemente para lograr un recubrimiento uniforme de la superficie de cultivo.
NOTA: Asegure condiciones estériles durante todo el proceso para evitar la contaminación. - Incubar los platos durante la noche a temperatura ambiente (RT).
- Aspire el exceso de solución de Poly-L-Lisina de los platos pipeteando.
- Enjuague la superficie con PBS tres veces, descansando durante 5 minutos cada vez (ver Tabla de Materiales). Retire por completo la poli-L-lisina no unida de la placa de imágenes para evitar la inhibición del crecimiento celular. Enjuague las placas suavemente para evitar raspar o dañar el fondo de vidrio.
- Secar al aire las placas de imagen recubiertas a 37 °C durante al menos 3 h.
- Utilice las placas de imagen recubiertas inmediatamente o guárdelas a 4 °C durante un máximo de 2 semanas.
4. Transfección de células HepG2
NOTA: El método de transfección de HepG2 se ilustra en la Figura 5.
- Siembre las células HepG2 en placas de imagen prerrecubiertas 24-48 h antes de la transfección para garantizar que alcancen una confluencia del 70%-90%.
- Descongele el reactivo de transfección y el plásmido que codifica el biosensor FRET en hielo. Realice un vórtice a fondo y realice un centrifugado corto (por ejemplo, 5.000 x g durante 5 s) antes de usar (consulte la tabla de materiales).
NOTA: Asegúrese de que el reactivo de transfección y el plásmido estén completamente descongelados antes de su uso.
PRECAUCIÓN: Evite los ciclos repetidos de congelación y descongelación, ya que esto puede reducir la eficiencia del reactivo de transfección. - Añadir 8 μg de plásmido que codifica el biosensor FRET con el tampón de reacción hasta un volumen final de 100 μL. Mezclar bien mediante vórtice durante 5 s a alta velocidad (aproximadamente 3.000-5.000 x g).
NOTA: Añada siempre el plásmido al tampón antes de añadir el reactivo de transfección a base de polímeros. Al menos 50 μL de la solución deben consistir en el tampón de reacción (ver Tabla de Materiales). - Añadir 2,4 μL del polímero de transfección al tubo que contiene el ADN plasmídico diluido. Mezclar bien mediante vórtice durante 15 s a alta velocidad (aproximadamente 3.000-5.000 × g).
NOTA: Utilice siempre 0,3 μL del polímero de transfección por 1 μg de ADN.
PRECAUCIÓN: Asegúrese de que el polímero de transfección esté completamente mezclado con el ADN plasmídico para formar complejos de nanopartículas uniformes. - Incubar el biosensor y la mezcla de polímero a 37 °C durante 15 minutos para permitir que se formen complejos de nanopartículas.
NOTA: Evite mantener el polímero de transfección en solución durante más de 30 minutos, ya que esto puede reducir la eficiencia de la transfección.
PRECAUCIÓN: Controle cuidadosamente el tiempo de incubación para evitar la sobreincubación, que puede reducir la eficiencia de la transfección. - Gire el tubo durante 5 s a 5.000 x g para recoger el contenido en la parte inferior, luego agregue los 100 μL completos de solución compleja de nanopartículas gota a gota al medio de cultivo celular. Mueva suavemente el plato hacia adelante y hacia atrás para mezclar.
NOTA: Asegúrese de que la solución compleja de nanopartículas se agregue gota a gota para distribuirla uniformemente por el medio de cultivo celular.
PRECAUCIÓN: Evite el balanceo vigoroso, ya que esto puede desalojar las células o causar una distribución desigual de los complejos. - Incubar la placa a 37 °C durante 4 h o toda la noche.
NOTA: El tiempo de incubación se puede ajustar en función de las necesidades experimentales, pero 4 h suelen ser suficientes para una transfección eficiente.
PRECAUCIÓN: Evite la incubación prolongada (>16 h), ya que esto puede reducir la viabilidad celular. - Retire los complejos de nanopartículas de las células por aspiración, reemplácelos con 2 mL de medio de crecimiento completo fresco y devuelva la placa a la incubadora a 37 °C hasta el momento del análisis. La expresión máxima suele alcanzar las 48 h después de la transfección.
- Analice las células bajo microscopía de fluorescencia.
NOTA: Asegúrese de que el microscopio esté correctamente calibrado para obtener imágenes de fluorescencia para obtener resultados precisos.
PRECAUCIÓN: Minimice la exposición de las células a la luz intensa durante la obtención de imágenes para evitar la fototoxicidad.
5. Inanición de células HepG2
NOTA: Después de completar el paso de transfección, prive de suero a las células antes de la estimulación con insulina y las imágenes FRET. Esto minimiza la activación de la vía Akt debido a la insulina presente en FBS y garantiza niveles basales consistentes de actividad Akt. La composición del medio de inanición utilizado en este experimento se describe en la (Tabla 3). BSA viene en forma de polvo. Para preparar una solución al 0,1% (p/v), reconstituya 0,1 g de BSA en 3 mL de DMEM, mezclando bien. Esterilizar la solución con un filtro de 0,45 μm y ajustar el volumen final a 100 mL añadiendo DMEM.
- Retire el medio de cultivo y enjuague las placas de imágenes con 1x PBS dos veces durante 5 minutos cada una.
NOTA: Los dos lavados de PBS ayudan a eliminar completamente el suero residual y cualquier insulina o factores de crecimiento que puedan interferir con el experimento. - Agregue 2 mL de medio de inanición a las placas de imágenes (ver Tabla de Materiales). Agregue suavemente el medio alrededor del borde del plato para evitar que las células se desprendan del fondo del vidrio. Incubar a 37 °C durante 4 h.
NOTA: La incubación de 4 horas es óptima para sincronizar el metabolismo celular; Sin embargo, la duración puede extenderse dependiendo de las necesidades experimentales.
6. Imágenes de células vivas FRET para células HepG2
NOTA: Esta sección proporciona instrucciones para la obtención de imágenes de células vivas FRET para monitorear la dinámica espacio-temporal de la fosforilación de Akt en células HepG2 individuales. Es esencial optimizar la configuración del microscopio, los procedimientos de manipulación y las condiciones de imagen para las células HepG2 vivas, como se detalla a continuación. La configuración del microscopio es crucial para optimizar las condiciones de imagen para la obtención de imágenes FRET. Siga la configuración paso a paso de la microscopía de escaneo láser PC/confocal (CLSM) de acuerdo con las instrucciones del fabricante para garantizar un funcionamiento estable. La configuración personalizada de CLSM para la adquisición de imágenes FRET se muestra en la (Figura 6).
- Encienda el interruptor remoto para alimentar el microscopio, la computadora, el escáner, el lanzamiento láser, la etapa piezoeléctrica y la fuente de luz LED de epifluorescencia. Asegúrese de que todos los componentes estén conectados correctamente antes de encenderlos para evitar posibles daños.
- Gire la llave a la posición ON en el lanzamiento del láser y presione los botones para activar los dos láseres necesarios para FRET (líneas láser de 457 nm y 514 nm).
NOTA: Asegúrese de que los filtros y ajustes adecuados estén en su lugar para obtener imágenes FRET óptimas. La selección de la línea láser debe basarse en los perfiles de excitación y emisión del biosensor. - Encienda la regleta para alimentar la computadora y el monitor conectados al microscopio.
NOTA: Asegúrese de que todas las conexiones estén seguras antes de encender para evitar daños en el equipo. - Inicie sesión en Windows e inicie el software de microscopía.
- Haga clic en A1 para Adquisición para iniciar la configuración de la imagen. Seleccione las configuraciones ópticas adecuadas en función de los requisitos del experimento FRET.
- Ajuste la potencia del láser y la sensibilidad del detector según sea necesario para obtener condiciones de imagen óptimas. Instale con cuidado la incubadora superior de la etapa del microscopio y asegúrela con tornillos.
NOTA: No apriete demasiado los tornillos para evitar daños en la incubadora o en la platina del microscopio. - Llene el baño de agua interno con agua estéril de doble destilación (ddH2O). Instale el calentador superior de forma segura y encienda la fuente de alimentación para el calentador de etapa, el calentador de baño y el calentador de lentes (Figura 7A).
NOTA: No llene en exceso para evitar derrames en el sistema. Asegúrese de que todos los calentadores funcionen correctamente para mantener condiciones de temperatura constantes para la obtención de imágenes de células vivas. - Utilice la lente de inmersión en aceite de 40x para obtener imágenes (consulte la Tabla de materiales).
NOTA: Consulte el sitio web del fabricante para conocer las especificaciones técnicas. - Limpie la lente del objetivo con papel humedecido con etanol al 95%. Coloque una pequeña gota de aceite de inmersión en la lente del objetivo (Figura 7B). Coloque la placa de imágenes que contiene las células HepG2 en la platina del microscopio y asegúrela con el soporte (Figura 7C).
NOTA: Asegúrese de que la antena parabólica esté correctamente alineada y asegurada para evitar cualquier movimiento durante la toma de imágenes. Evite la presión contra la lente para evitar daños tanto en la lente como en el fondo de vidrio del plato. - Cierre la cámara e incube las células dentro de la cámara de células vivas durante 1-2 h para permitir que se equilibren (Figura 7D).
- Durante la toma de imágenes de lapso de tiempo, haga una pausa a intervalos específicos y retire suavemente el medio (Figura 7E), luego agregue 1 ml de medio recién preparado con la concentración de insulina dada (Figura 7F, Tabla de materiales).
NOTA: Prepare una solución madre de insulina de 1 mg/mL disolviendo 1 mg de insulina en 1 mL de ácido acético de 10 mM. Filtrar la solución a través de un filtro de jeringa estéril de 0,2 μm y almacenar las alícuotas a -20 °C. - Inicie la ventana de adquisición de ND desde el menú Archivo del software de microscopía.
NOTA: La configuración de adquisición ND se muestra en (Figura 8). - Seleccione la pestaña Tiempo para establecer el intervalo, la duración y los bucles deseados para las imágenes de lapso de tiempo. A continuación, haga clic en la pestaña XY y presione el botón + agregar para incluir células HepG2 individuales para la obtención de imágenes.
- Para agregar varias celdas, localice las celdas adecuadas, escanee, ajuste el enfoque y bloquee la función de dispersión de puntos (PSF). Repita este paso para cada nueva celda que se agregue para la obtención de imágenes. Escanee varias celdas en diferentes ubicaciones dentro de las antenas de imágenes para identificar varias celdas objetivo para su inclusión.
- Haga clic en el icono de la X roja para anular la selección de las celdas. Marque la casilla 'Z' para establecer la posición Z. Después de seleccionar todos los parámetros, introduzca el nombre del experimento en el cuadro "Nombre de archivo".
- Haga clic en Examinar para elegir la carpeta de destino y, a continuación, marque la casilla Guardar en archivo. Presione la pestaña Ejecutar ahora para iniciar la adquisición de ND. La ventana de adquisición de ND mostrará el progreso en tiempo real de las imágenes de lapso de tiempo, incluido el tiempo transcurrido y restante.
- Registre las mediciones de referencia durante un máximo de 30 minutos sin estimular las células con insulina.
- Localice las células adecuadas, acerque o aleje el zoom para enfocar una sola célula y seleccione las células con alta intensidad de fluorescencia. Agregue las celdas una por una, hasta un máximo de 6 celdas.
NOTA: Limite el número de celdas para evitar retrasos en la adquisición de imágenes y la congelación de ventanas durante la toma de imágenes. - Establezca la hora y la frecuencia de imagen para las imágenes de lapso de tiempo. Inicie las imágenes de lapso de tiempo.
NOTA: Asegúrese de que todos los parámetros, incluido el tiempo de exposición y la potencia del láser, estén optimizados antes de comenzar la sesión de imágenes. - Haga una pausa en las imágenes a intervalos regulares, retire suavemente el medio del plato con fondo de vidrio y agregue 1 ml de medio complementado con la concentración de insulina adecuada.
- Reanude la adquisición de la imagen. Repita el paso 6.20 según sea necesario.
- Al final del experimento, haga clic en la pestaña Finalizar para cerrar. Realice copias de seguridad y almacene las imágenes adquiridas de forma segura para su análisis.
7. Análisis de datos
- Corrija los datos de imágenes FRET de lapso de tiempo preprocesados de muestras de control para diafonía espectral y autofluorescencia CFP, lo que da como resultado valores FRET corregidos y una eficiencia FRET precisa. Utilizar software de imagen para la adquisición, procesamiento y análisis de imágenes FRET, siguiendo los protocolos descritos en estudios previos 27,28,29,30 (Figura 9).
8. Cálculos de eficiencia FRET
- Para determinar la eficiencia de FRET, adquiera siete imágenes: celda (IDA(D), IDD(D), IDA(A), IAA(A), IDD, IAA y IDA). Calcule la eficiencia de FRET utilizando la siguiente fórmula:
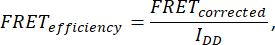
donde FRETCorregido se obtiene de la ecuación:
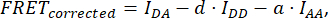
con d y a definidas como:
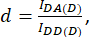
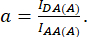
Aquí IDA, IDD y IAA representan imágenes de células transfectadas con el biosensor. IDA(D) e IDD(D)) son imágenes de control solo para donantes, y IDA(A) e IAA(A) son imágenes de control solo para aceptores.
9. Adquisición de imágenes
- IDA : Obtención de la imagen por excitación del donante (434 nm) con emisión de aceptor (530 nm).
- IDD : Obtención de la imagen por excitación del donante (434 nm) con emisión del donante (477 nm).
- IAA : Obtención de la imagen por excitación del aceptor (517 nm) con emisión del aceptor (530 nm).
- IDA(D) y IDA(A) : Adquieren la imagen utilizando la misma excitación y emisión que IDA, pero a partir de células que expresan solo el donante y el aceptor, respectivamente.
- IDD(D) : Adquiere la imagen en las mismas condiciones que IDD para el control solo del donante.
- IAA(A) : Obtiene la imagen utilizando las mismas condiciones que IAA, pero es específica para el control de solo aceptante.
10. Corrección de fondo
- Abra el software de análisis de imágenes en la computadora. Desde la barra de menú superior, acceda al menú Archivo . Seleccione Abrir o Abrir archivo en las opciones desplegables.
- Navegue hasta el directorio que contiene los datos de la imagen de lapso de tiempo. Localice la imagen de lapso de tiempo que requiere análisis (por ejemplo, formato nd2).
- Elija el archivo y haga clic en Abrir o Aceptar para cargarlo en el software para verlo y analizarlo. Defina una región de interés (ROI) dentro de la celda (o utilice toda la celda como ROI) y registre el valor de gris de cada píxel dentro de esta región.
- Elija un área sin celdas como fondo y calcule su valor medio de gris. Cree la imagen corregida restando este promedio de fondo del valor de gris de cada píxel dentro del ROI de la celda.
11. Eliminación de sangrado (diafonía) de FRET
NOTA: La superposición espectral entre la emisión del donante y la excitación del aceptor se muestra en la Figura 3B, que es crítica para la eficiencia de FRET y el proceso de transferencia de energía. El sangrado en las imágenes FRET de lapso de tiempo es un desafío importante que surge de la superposición espectral de los fluoróforos donantes y aceptores, lo que lleva a mediciones inexactas. La diafonía es inherente porque los espectros de los fluoróforos donantes y aceptores se superponen hasta cierto punto (Figura 3C, D). Este problema se ve agravado por factores como las altas concentraciones de fluoróforos y las configuraciones incorrectas de los filtros. Abordar el sangrado es crucial para garantizar la fiabilidad de las mediciones de FRET.
- Consulte un estudio previo para conocer un método para mitigar los efectos de sangrado9.
12. Cuantificación y análisis estadístico
- Realizar análisis estadísticos utilizando software de análisis estadístico.
Resultados
Para investigar la activación de Akt en las células HepG2, las células se sembraron en placas de imagen prerrevestidas y se transfectaron con el biosensor pEevee-iAkt-NES basado en FRET (Figura 2A), diseñado para permitir el monitoreo en tiempo real de la fosforilación de Akt. Después de la transfección, las células se sometieron a inanición sérica durante 4 h en un medio libre de suero para sincronizar su estado metabólico y minimizar la señaliz...
Discusión
El protocolo para la obtención de imágenes FRET de células vivas para monitorizar la fosforilación de Akt en células HepG2 implica varios pasos clave para garantizar resultados fiables y reproducibles. El primer paso crítico es el cultivo celular, que incluye el mantenimiento celular de rutina, el recubrimiento de las placas de imágenes y la siembra de células. El recubrimiento adecuado es esencial para la unión de las células durante los experimentos de imágenes de lapso de t...
Divulgaciones
Los autores declaran no tener intereses contrapuestos.
Agradecimientos
Este trabajo fue apoyado en parte por la Fundación de Ciencias Naturales de Shenzhen (JCYJ20240813113606009), la Zona de Cooperación Shenzhen-Hong Kong para Tecnología e Innovación (HZQB-KCZYB-2020056), la Fundación Nacional de Ciencias Naturales de China (32070681), el Programa Nacional Clave de Investigación y Desarrollo de China (2019YFA0906002) y el Plan del Pavo Real de Shenzhen (KQTD2016053117035204).
Materiales
| Name | Company | Catalog Number | Comments |
| 0.25% trypsin-EDTA | Gibco | Cat#25200-056 | Use ice-cold PBS for cell wash |
| 15 mm glass bottom cell culture dish | NEST | Cat#801001 | |
| 2 mL Nalgene cryogenic vials | Thermo Scientific | Cat#5012-0020 | |
| 5 mL Stripette Serological Pipets | Corning | Cat#4487 | |
| 95% Ethanol | Kermel | Cat#C028005 | |
| A1 HD25/A1R HD25 confocal microscope | Nikon | https://www.nikon.com/ | Magnification: 40×, Numerical Aperture (NA): 1.30, Pixel Dwell Time: 2.4 ms, Pixel Size: 1024 |
| Ampicillin | Sigma-Aldrich | Cat#A9393 | |
| Bovine serum albumin (BSA) | VWR Life Science | Cat#N208-10g | |
| Corning 25 cm2 rectangular culture flasks | Corning | Cat#430639 | |
| Countess 3 automated cell counter | Thermo Scientific | http://www.thermofisher.com/#AMQAX2000 | |
| Countess cell counting chamber slides | Thermo Scientific | http://www.thermofisher.com/#C10228 | |
| Digital vortex mixers | Thermo Scientific | https://www.thermofisher.com/ | |
| Dimethyl sulfoxide | Sigma-Aldrich | Cat#D2650 | |
| Eppendorf Safe-Lock Tubes 1.5 mL | Eppendorf | Cat#022363204 | |
| EZ-PCR mycoplasma detection kit | Biological Industries | Cat# 20-700-20 | |
| Fetal Bovine Serum, qualified, Australia | Gibco | Cat#10099141 | |
| GlutaMAX Supplement | Gibco | Cat#35050061 | |
| GraphPad Prism 9 | GraphPad Software | https://www.graphpad.com/ | |
| HepG2 | National Collection of Authenticated Cell Cultures | #CSTR:19375.09.3101HUMSCSP510 http://www.cellbank.org.cn/ | |
| Immersion Oil Type 37 | Cargille Laboratories | Cat #16237 | |
| Insulin | Sigma-Aldrich | Cat#I5500-50MG | Warm to 37 °C before use |
| LB Broth (1x) | Invitrogen | Cat#10855001 | |
| Minimum Essential Medium (MEM) | Gibco | Cat#11095080 | Warm to 37 °C before use |
| mySPIN 12 mini centrifuge | Thermo Scientific | https://www.thermofisher.com/ | |
| NanoDrop One | Thermo Scientific | https://www.thermofisher.com | |
| Nikon Plan Fluor 40×/1.30 Oil Lens | Nikon | https://www.nikon.com/ | |
| NIS-Elements-AR | Nikon | https://www.nikon.com/ | |
| Non-Essential Amino Acids (NEAA) (100x) | Gibco | Cat#11140050 | |
| One Shot LB Agar Plates | Invitrogen | Cat#A55802 | |
| One Shot Stbl3 chemically competent E. coli | Invitrogen | Cat#C737303 | |
| Parafilm | PARAFILM | Cat#B8R05606 | |
| PBS (phosphate buffered saline) | Gibco | Cat#10010023 | |
| pEevee-iAkt-NES (7,033 bp) | Miura et al31 | https://benchling.com/s/seq-q46zFYCfl0swLAun0t28/edit | |
| Penicillin-streptomycin | Gibco | Cat#15070063 | |
| Plasmocin prophylactic | InvivoGen | Cat#ant-mpp | |
| Poly-L-Lysine Hydrobromide | Sigma-Aldrich | Cat#P4832 | |
| Precision general purpose baths | Thermo Scientific | https://www.thermofisher.com/ | |
| QIAprep spin miniprep kit | QIAGEN | Cat#27106 | |
| SnapGene | SnapGene by Dotmatics | https://www.snapgene.com | |
| Sodium Pyruvate (100 mM) | Gibco | Cat#11360070 | |
| Syringe filter unit, 0.22 μm | Millipore | Cat#SLGP033RS | |
| Tokai Hit stage top incubator | TOKAI HIT | https://www.tokaihit-livecell.com/stagetopincubator | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | Cat#10977015 | |
| Xfect Transfection Reagent | Takara Bio | Cat#631317 |
Referencias
- GBD 2021 Diabetes Collaborators. Global, regional, and national burden of diabetes from 1990 to 2021, with projections of prevalence to 2050: a systematic analysis for the Global Burden of Disease Study 2021. Lancet. 402 (10397), 203-234 (2023).
- Li, M., et al. Trends in insulin resistance: insights into mechanisms and therapeutic strategy. Signal Transduct Target Ther. 7 (1), 216 (2022).
- Poloz, Y., Stambolic, V. Obesity and cancer, a case for insulin signaling. Cell Death Dis. 6 (12), e2037 (2015).
- Arcidiacono, B., et al. Insulin resistance and cancer risk: an overview of the pathogenetic mechanisms. Exp Diabetes Res. 2012, 789174 (2012).
- Godsland, I. F. Insulin resistance and hyperinsulinaemia in the development and progression of cancer. Clin Sci (Lond). 118 (5), 315-332 (2009).
- Tsugane, S., Inoue, M. Insulin resistance and cancer: epidemiological evidence. Cancer Sci. 101 (5), 1073-1079 (2010).
- Yudhani, R. D., et al. In vitro insulin resistance model: A recent update. J Obes. 2023, 1964732 (2023).
- Akhtar, J., et al. Bistable insulin response: The win-win solution for glycemic control. iScience. 25 (12), 105561 (2022).
- Akhtar, J., Imran, M., Wang, G. Protocol for live-cell Forster resonance energy transfer imaging to reveal the bistable insulin response of single C2C12-derived myotubes. STAR Protoc. 5 (2), 103109 (2024).
- Kamino, K., et al. Optimal inference of molecular interaction dynamics in FRET microscopy. Proc Natl Acad Sci U S A. 120 (15), e2211807120 (2023).
- Kraft, A. E., Nikolaev, V. O. FRET microscopy for real-time visualization of second messengers in living cells. Methods Mol Biol. 1563, 85-90 (2017).
- Veeriah, S., et al. High-throughput time-resolved FRET reveals Akt/PKB activation as a poor prognostic marker in breast cancer. Cancer Res. 74 (18), 4983-4995 (2014).
- Conway, J. R. W., et al. Monitoring AKT activity and targeting in live tissue and disease contexts using a real-time Akt-FRET biosensor mouse. Sci Adv. 9 (17), eadf9063 (2023).
- Chandris, P., Giannouli, C. C., Panayotou, G. Imaging approaches for the study of metabolism in real time using genetically encoded reporters. Front Cell Dev Biol. 9, 725114 (2021).
- Yang, J., et al. Longitudinal FRET imaging of glucose and lactate dynamics and response to therapy in breast cancer cells. Mol Imaging Biol. 24 (1), 144-155 (2022).
- Mandrou, E., et al. A reliable system for quantitative G-protein activation imaging in cancer cells. Cells. 13 (13), 1114 (2024).
- Fang, C., Huang, Y., Zhao, Y. Review of FRET biosensing and its application in biomolecular detection. Am J Transl Res. 15 (2), 694-709 (2023).
- Miller, J. N. Fluorescence energy transfer methods in bioanalysis. Analyst. 130 (3), 265-270 (2005).
- Verveer, P. J., Rocks, O., Harpur, A. G., Bastiaens, P. I. Imaging protein interactions by FRET microscopy: FRET measurements by acceptor photobleaching. CSH Protoc. 2006 (6), (2006).
- Vu, C. Q., Arai, S. Quantitative imaging of genetically encoded fluorescence lifetime biosensors. Biosensors (Basel). 13 (10), 939 (2023).
- Wang, G. A more holistic view of the logarithmic dose-response curve offers greater insights into insulin response. J Biol Chem. 301 (1), 108037 (2025).
- Wang, G. Body mass dynamics is determined by the metabolic Ohm's law and adipocyte-autonomous fat mass homeostasis. iScience. 23 (6), 101176 (2020).
- Wang, G. Raison d'être of insulin resistance: the adjustable threshold hypothesis. J R Soc Interface. 11 (101), 20140892 (2014).
- Wang, G. Optimal homeostasis necessitates bistable control. J R Soc Interface. 9, 2723 (2012).
- Heim, R., Tsien, R. Y. Engineering green fluorescent protein for improved brightness, longer wavelengths and fluorescence resonance energy transfer. Curr Biol. 6 (2), 178-182 (1996).
- Nguyen, A. W., Daugherty, P. S. Evolutionary optimization of fluorescent proteins for intracellular FRET. Nat Biotechnol. 23 (3), 355-360 (2005).
- Broussard, J. A., Rappaz, B., Webb, D. J., Brown, C. M. Fluorescence resonance energy transfer microscopy as demonstrated by measuring the activation of the serine/threonine kinase Akt. Nat Protoc. 8 (2), 265-281 (2013).
- Gordon, G. W., Berry, G., Liang, X. H., Levine, B., Herman, B. Quantitative fluorescence resonance energy transfer measurements using fluorescence microscopy. Biophys J. 74 (5), 2702-2713 (1998).
- Tron, L., et al. Flow cytometric measurement of fluorescence resonance energy transfer on cell surfaces. Quantitative evaluation of the transfer efficiency on a cell-by-cell basis. Biophys J. 45 (5), 939-946 (1984).
- Youvan, D. C., et al. Calibration of fluorescence resonance energy transfer in microscopy. , (2002).
- Miura, H., Matsuda, M., Aoki, K. Development of a FRET biosensor with high specificity for Akt. Cell Struct Funct. 39 (1), 9-20 (2014).
- Liberio, M. S., Sadowski, M. C., Soekmadji, C., Davis, R. A., Nelson, C. C. Differential effects of tissue culture coating substrates on prostate cancer cell adherence, morphology and behavior. PLoS One. 9 (11), e112122 (2014).
- Rani, K., Sengupta, S. Multi-stimuli programmable FRET based RGB absorbing antennae towards ratiometric temperature, pH and multiple metal ion sensing. Chem Sci. 12 (47), 15533-15542 (2021).
- Betolngar, D. B., et al. pH sensitivity of FRET reporters based on cyan and yellow fluorescent proteins. Anal Bioanal Chem. 407 (14), 4183-4193 (2015).
- Salonikidis, P. S., et al. An ion-insensitive cAMP biosensor for long term quantitative ratiometric fluorescence resonance energy transfer (FRET) measurements under variable physiological conditions. J Biol Chem. 286 (26), 23419-23431 (2011).
- Youvan, D. C., et al. Calibration of fluorescence resonance energy transfer in microscopy using genetically engineered GFP derivatives on nickel chelating beads. Biotechnol Alia. 3, 1-18 (1997).
- Menaesse, A., et al. Simplified instrument calibration for wide-field fluorescence resonance energy transfer (FRET) measured by the sensitized emission method. Cytometry A. 99 (4), 407-416 (2021).
- Batta, A., Hajdu, T., Nagy, P. Improved estimation of the ratio of detection efficiencies of excited acceptors and donors for FRET measurements. Cytometry A. 103 (7), 563-574 (2023).
- Coullomb, A., et al. QuanTI-FRET: a framework for quantitative FRET measurements in living cells. Sci Rep. 10 (1), 6504 (2020).
- Hoppe, A., Christensen, K., Swanson, J. A. Fluorescence resonance energy transfer-based stoichiometry in living cells. Biophys J. 83 (6), 3652-3664 (2002).
- Zal, T., Gascoigne, N. R. Photobleaching-corrected FRET efficiency imaging of live cells. Biophys J. 86 (6), 3923-3939 (2004).
- Hochreiter, B., Kunze, M., Moser, B., Schmid, J. A. Advanced FRET normalization allows quantitative analysis of protein interactions including stoichiometries and relative affinities in living cells. Sci Rep. 9 (1), 8233 (2019).
- Liu, H., Zhang, F., Mishra, S. K., Zhou, S., Zheng, J. Knowledge-guided fuzzy logic modeling to infer cellular signaling networks from proteomic data. Sci Rep. 6, 35652 (2016).
- Garrido-Rodriguez, M., Zirngibl, K., Ivanova, O., Lobentanzer, S., Saez-Rodriguez, J. Integrating knowledge and omics to decipher mechanisms via large-scale models of signaling networks. Mol Syst Biol. 18 (7), e11036 (2022).
- Greenwald, E. C., Polanowska-Grabowska, R. K., Saucerman, J. J. Integrating fluorescent biosensor data using computational models. Methods Mol Biol. 1071, 227-248 (2014).
- Zhao, Z., Xia, J. Computational Approaches for Modeling Signal Transduction Networks. Encyclopedia of Bioinformatics and Computational Biology. , (2019).
- Wang, G., Krueger, G. R. Computational analysis of mTOR signaling pathway: bifurcation, carcinogenesis, and drug discovery. Anticancer Res. 30 (7), 2683-2688 (2010).
- Wang, G. Singularity analysis of the AKT signaling pathway reveals connections between cancer and metabolic diseases. Phys Biol. 7 (4), 046015 (2010).
- Chedere, A., Hari, K., Kumar, S., Rangarajan, A., Jolly, M. K. Multi-stability and consequent phenotypic plasticity in AMPK-Akt double negative feedback loop in cancer cells. J Clin Med. 10 (3), 472 (2021).
- Mosca, E., et al. Computational modeling of the metabolic States regulated by the kinase akt. Front Physiol. 3, 418 (2012).
- Liao, J., Madahar, V., Dang, R., Jiang, L. Quantitative FRET (qFRET) technology for the determination of protein-protein interaction affinity in solution. Molecules. 26 (21), 6339 (2021).
- Verma, A. K., Noumani, A., Yadav, A. K., Solanki, P. R. FRET based biosensor: Principle applications recent advances and challenges. Diagnostics (Basel). 13 (8), 1375 (2023).
- Mattheisen, J. M., et al. Application of bioluminescence resonance energy transfer to quantitate cell-surface expression of membrane proteins. Anal Biochem. 684, 115361 (2024).
- Lionetti, M. C., La Porta, C. A. M. FLIM-FRET investigation of heterogeneous huntingtin aggregation in HeLa cells. Methods Mol Biol. 2551, 595-604 (2023).
- Petutschnig, E. K., Pierdzig, L., Mittendorf, J., Niebisch, J. M., Lipka, V. A novel fluorescent protein pair facilitates FLIM-FRET analysis of plant immune receptor interaction under native conditions. J Exp Bot. 75 (3), 746-759 (2024).
- Sprenger, J. U., Perera, R. K., Gotz, K. R., Nikolaev, V. O. FRET microscopy for real-time monitoring of signaling events in live cells using unimolecular biosensors. J Vis Exp. 66, e4081 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados