需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
HepG2 细胞中代谢调节的 Akt 活化动力学的活细胞 Förster 共振能量转移成像
摘要
在这里,我们提出了一种方案来量化活 HepG2 细胞中 Akt 激活和磷酸化的时空动力学。Förster 共振能量转移 (FRET) 成像是一种强大的工具,可为癌细胞中的胰岛素信号通路和代谢调节提供有价值的见解。
摘要
代谢调节的 Akt 激活是胰岛素信号转导级联反应的关键节点,为糖尿病与癌症之间的关系提供了有价值的见解。为了精确量化 HepG2 细胞中的 Akt 活性,我们开发了一种稳健、可重复的方案,利用 Förster 共振能量转移 (FRET) 和基因编码的 Akt 特异性生物传感器。该协议概述了细胞培养、成像培养皿制备和 HepG2 细胞转染以表达基于 FRET 的生物传感器的详细步骤,以及激光扫描共聚焦显微镜硬件和软件配置的具体指南。结果显示了 HepG2 细胞中胰岛素信号转导的独特模式,其表现出不可逆的转换,其特征是组成型 Akt 激活,具有明确的开启阈值,但没有关闭阈值。相比之下,肌管显示可逆开关。HepG2 细胞中持续的 Akt 激活表明肝细胞胰岛素抵抗和代谢失调的机制,对理解代谢紊乱和癌症的进展具有更广泛的意义。该协议为探索各种疾病背景下的 Akt 相关信号通路和细胞行为提供了一个有价值的框架。
引言
糖尿病构成了一项重大的全球健康挑战,其特征是胰岛素抵抗和葡萄糖稳态受损1。全面了解胰岛素信号通路对于阐明这种疾病的病理生理学至关重要,因为胰岛素在葡萄糖代谢、细胞生长和存活中起着关键作用2。大量研究表明,胰岛素信号转导对各种癌症有显著影响,将胰岛素抵抗与肿瘤进展和不良患者预后联系起来 3,4,5,6。HepG2 细胞是一种常用的肝细胞癌细胞系,是研究胰岛素抵抗以及代谢失调与癌症发展之间相互作用的宝贵模型7。传统上,研究人员将胰岛素反应视为分级;然而,最近的研究表明,单个细胞可以表现出双稳态反应,在特定胰岛素浓度阈值下表现出无反应和完全反应之间的显着转变 8,9。
Förster 共振能量转移 (FRET) 成像是研究生物分子在活细胞中的时空分布的强大工具10。通过从分子动力学中提取信息,FRET 可以实时了解 Akt 激活等过程,使其成为研究活细胞的宝贵技术11,12。事实证明,这种成像方法在研究细胞动力学方面至关重要,尤其是在代谢疾病和癌症中,其中精确的分子相互作用至关重要13。FRET 还可以实时监测分子相互作用,阐明胰岛素抵抗和肿瘤进展等机制14,15。FRET 生物传感器在癌症研究中对于研究肿瘤微环境、耐药性和代谢紊乱至关重要16。FRET检测方法,如敏化发射(SE)、受体漂白(AB)、荧光寿命成像显微镜(FLIM)和光谱学,在量化分子相互作用方面都具有明显的优势17。SE 测量供体荧光团和受体荧光团之间的能量转移,导致发射光谱发生可测量的变化,这与相互作用的生物分子的接近程度相关18。AB 使用受体荧光团的选择性光漂白并跟踪供体荧光的变化,这使研究人员能够评估相互作用动力学和距离19。FLIM 评估供体荧光团的荧光衰减率,直接受 FRET 效率的影响,以提供分子相互作用的精确纳米级测量20。
使用 FRET 技术,我们最近证明了 C2C12 衍生的肌管中的双稳态胰岛素反应 8,9,21,22,23,24。正如我们发现的那样,Akt 激活的不同开启和关闭阈值表明,分级的全身胰岛素剂量反应掩盖了从胰岛素刺激开始的亚细胞信号级联的复杂性,最终导致单细胞水平的全有或无反应 21,22,23,24.为了测试其他细胞类型中双稳态的存在,我们用胰岛素刺激 HepG2 细胞,并使用单细胞 FRET 成像记录它们的反应。我们用不同的胰岛素浓度刺激 HepG2 细胞,并使用 Akt 生物传感器在单细胞水平监测 Akt 活性。Akt 生物传感器包括作为供体荧光团的增强型青色荧光蛋白 (ECFP)25 和作为受体荧光团的黄色荧光蛋白 (YPet)26 的最亮变体,由包含肽序列 SGRPRTTTFADSCKP 的 Eevee 接头连接。该肽可作为磷酸化 Akt (pAkt) 的底物,由人糖原合成酶激酶 3β (GSK3β) 优化而来。在未磷酸化状态下,供体荧光团和受体荧光团之间的空间分离超过了 Förster 半径,这会抑制能量转移。在胰岛素刺激后,发生 Akt 磷酸化并导致 SGRPRTTTFADSCKP 磷酸化。这个过程诱导构象变化,使供体和受体位于 Förster 半径内,从而启用 FRET27。因此,FRET 信号强度与磷酸化 Akt 分子的量相关,并允许实时定量胰岛素介导的细胞反应。
该方案最初是为了研究 C2C12 衍生的肌管中的胰岛素信号传导而开发的,已成功应用于 HepG2 细胞并在不同的硬件和软件平台上使用,从而证明了其适用性、适应性和多功能性。HepG2 细胞表现出组成型 Akt 活性,这使它们成为研究肝脏特异性胰岛素信号传导和代谢过程的理想 体外 模型。协议的主要功能在 协议 部分。
研究方案
图 1 概述了用于监测单个 HepG2 细胞中 Akt 磷酸化的 FRET 活细胞成像所涉及的实验步骤。
1. 质粒采集、增殖和纯化
注:本节概述了获取、扩增和纯化单细胞 FRET 分析所需质粒的基本步骤。
- 利用 pEevee-iAkt-NES-YPet 质粒(图 2A)。
注意:该质粒由日本国立基础生物学研究所 (NIBB) 的 Kazuhiro Aoki 教授慷慨提供,用于 Akt 监测的 FRET 生物传感器的质粒图谱及其各自的对照如图 2 所示。pEevee-iAkt-NES-ECFP(供体; 图 2B)和 pEevee-iAkt-NES-Ypet(受体; 图 2C)质粒在 FRET 实验中用作校准对照9。
注意: 图 3 显示了分子内 FRET 生物传感器的组成和机制。 - 为了增殖质粒,使用化学感受态大肠杆菌细胞进行细菌转化(参见 材料表)。将转化的细胞接种在含有 100 μg/mL 氨苄青霉素的 Luria-Bertani (LB) 琼脂上,并在 37 °C 下孵育过夜。 第二天,选择一个氨苄青霉素抗性菌落,并在补充有 100 μg/mL 氨苄青霉素的 LB 肉汤中培养,用于质粒扩增。
- 要纯化质粒 DNA,请使用市售的质粒 DNA 纯化试剂盒(参见 材料表)以获得高纯度和产量。通过使用分光光度计测量吸光度比(A260/A280 和 A260/A230)来评估 DNA 质量(参见 材料表)。介于 1.8-2.0 和 2.0-2.2 之间的比率被认为是最佳的。通过琼脂糖凝胶电泳验证质粒完整性,以确保其适合转染。
2. 细胞培养程序
注:在层流罩内执行所有细胞培养程序,以保持无菌环境并防止污染。HepG2 细胞培养工作流程如图 4 所示。HepG2 细胞完全培养基由最低必需培养基 (MEM)、10% 胎牛血清 (FBS)、1% 非必需氨基酸 (NEAA)、1 mM 丙酮酸钠、2 mM L-谷氨酰胺补充剂、100 U/mL 青霉素-链霉素和 2.5 μg/mL 抗生素-抗真菌溶液组成(参见 材料表,表 1)。
- 将冻存管置于 37 °C 热混合器或水浴中,直至完全解冻,快速解冻冷冻的 HepG2 细胞。
- 将解冻的细胞转移到含有 10 mL 完全生长培养基的 15 mL 锥形管中(参见 材料表)。
注:使用前将完全生长培养基预热至 37 °C,以尽量减少对细胞的热冲击。 - 将试管以 200 x g 离心 5 分钟以沉淀细胞。
- 使用大口径移液器吸头小心吸出上清液,以避免干扰细胞沉淀。
- 将沉淀重悬于 10 mL 新鲜完全生长培养基中。
- 将细胞悬液转移到 75 cm² 组织培养瓶中。
- 将培养瓶在 37 °C 下在 5% CO2 的潮湿气氛中孵育
注意:在接下来的 24-48 小时内观察细胞以确认附着并评估恢复情况。更换培养基前至少 4 小时不要干扰细胞,以确保正确附着。避免在前 4 小时内频繁打开培养箱,因为这可能会破坏细胞附着。在所有细胞培养过程中,遵循机构生物安全方案并使用适当的个人防护设备 (PPE) 来维护安全的工作环境。当 HepG2 细胞达到 70%-80% 汇合时,对其进行传代培养,以维持最佳生长条件并防止过度拥挤,过度拥挤会影响细胞活力和生长潜力。 - 对于传代培养,吸出培养基并用 5 mL 磷酸盐缓冲盐水 (PBS) 冲洗细胞一次。
- 加入 1 mL 的 0.25% 胰蛋白酶以覆盖细胞单层(参见 材料表)。
注:确保胰蛋白酶预热至 37 °C 以获得最佳活性。不要过度胰蛋白酶消化细胞,因为这可能会降低细胞活力。在显微镜下监测细胞以确认脱离。 - 在 37 °C 下孵育约 5 分钟。
- 当细胞分离时,加入 2 mL 完全培养基以中和胰蛋白酶,并通过移液收集细胞。
- 轻轻移液细胞悬液以打碎团块并获得单细胞悬液。
- 向每个新培养瓶中加入 3 mL 完全培养基,然后以 1:2 的分流比例将细胞转移到每个培养瓶中。
注:对于早期传代,以 1:2 的稀释度分流细胞。第 4-5 代后,可酌情进行 1:4 或 1:5 的稀释。 - 将细胞在 37 °C 下在 5% CO2 的潮湿环境中孵育。
注意:24 小时后检查细胞以确认附着并评估恢复。 - 对于常规培养,每 2-3 天更换一次培养基,如果 pH 指示剂从粉红色变为黄色,则表示酸化,请尽早更换。
注:不要让培养基变得太酸性,因为这可能会伤害细胞。 - 定期在显微镜下检查细胞形态,以确保细胞健康。
注意:对于短期储存,将 HepG2 细胞在 -80 °C 下冷冻;如需长期储存,请储存在液氮中。本实验中使用的冷冻培养基的成分如(表 2)所示。
3. 用聚-L-赖氨酸涂覆成像皿
- 每个成像皿使用 1 mL 的 0.1 mg/mL 聚-L-赖氨酸溶液覆盖整个表面(参见 材料表)。
注:根据特定的细胞类型要求调整 Poly-L-Lysine 浓度。 - 轻轻摇动培养皿,使培养表面均匀涂覆。
注:确保整个过程的无菌条件,以防止污染。 - 将培养皿在室温 (RT) 下孵育过夜。
- 通过移液从培养皿中吸出多余的 Poly-L-Lysine 溶液。
- 用 PBS 冲洗表面 3 次,每次静置 5 分钟(参见 材料表)。从成像皿中完全去除未结合的 Poly-L-Lysine 以防止细胞生长抑制。轻轻冲洗板,以免刮擦或损坏玻璃底部。
- 将涂层成像皿在 37 °C 下风干至少 3 小时。
- 立即使用涂层成像培养皿或将其在 4 °C 下储存长达 2 周。
4. HepG2 细胞的转染
注意:HepG2 转染方法如图 5 所示。
- 转染前 24-48 小时将 HepG2 细胞接种在预包被的成像培养皿中,以确保它们达到 70%-90% 汇合。
- 在冰上解冻编码 FRET 生物传感器的转染试剂和质粒。使用前彻底涡旋并进行短暂旋转(例如,5,000 x g 持续 5 秒)(参见 材料表)。
注:确保转染试剂和质粒在使用前完全解冻。
注意:避免重复冻融循环,因为这可能会降低转染试剂的效率。 - 将 8 μg 编码 FRET 生物传感器的质粒与反应缓冲液一起加入,最终体积为 100 μL。高速涡旋 5 秒(约 3,000-5,000 x g)充分混合。
注:在添加基于聚合物的转染试剂之前,请务必向缓冲液中加入质粒。至少 50 μL 的溶液必须由反应缓冲液组成(参见 材料表)。 - 将 2.4 μL 转染聚合物添加到含有稀释质粒 DNA 的试管中。通过高速(约 3,000-5,000 × g)涡旋 15 秒充分混合。
注:每 1 μg DNA 始终使用 0.3 μL 转染聚合物。
注意:确保转染聚合物与质粒 DNA 充分混合,以形成均匀的纳米颗粒复合物。 - 将生物传感器和聚合物混合物在 37 °C 下孵育 15 分钟,以形成纳米颗粒复合物。
注:避免将转染聚合物在溶液中放置超过 30 分钟,因为这可能会降低转染效率。
注意:仔细监测孵育时间,以防止过度孵育,否则可能导致转染效率降低。 - 以 5,000 x g 的速度将试管向下旋转 5 秒以收集底部的内容物,然后将整个 100 μL 纳米颗粒复合物溶液滴加到细胞培养基中。轻轻地来回摇动盘子以混合。
注:确保逐滴添加纳米颗粒复合物溶液,以将其均匀分布在细胞培养基中。
注意:避免剧烈摇晃,因为这可能会使细胞脱落或导致复合物分布不均匀。 - 将板在 37 °C 下孵育 4 小时至过夜。
注:孵育时间可根据实验需要进行调整,但 4 小时通常足以实现高效转染。
注意:避免长时间孵育 (>16 小时),因为这可能会降低细胞活力。 - 通过抽吸从细胞中去除纳米颗粒复合物,用 2 mL 新鲜的完全生长培养基替换它们,然后将板放回 37 °C 培养箱中,直到分析时。转染后峰值表达通常达到 48 小时。
- 在荧光显微镜下分析细胞。
注意:确保显微镜已正确校准以进行荧光成像,以获得准确的结果。
注意:在成像过程中尽量减少细胞暴露在强光下,以防止光毒性。
5. HepG2 细胞饥饿
注:完成转染步骤后,在胰岛素刺激和 FRET 成像之前对细胞进行血清饥饿。这最大限度地减少了由于 FBS 中存在的胰岛素而导致的 Akt 通路激活,并确保 Akt 活性的基线水平一致。本实验中使用的饥饿培养基的成分如(表 3)所述。BSA 为粉末状。要制备 0.1% (w/v) 溶液,请在 3 mL DMEM 中配制 0.1 g BSA,充分混合。使用 0.45 μm 过滤器对溶液进行消毒,并通过添加 DMEM 将最终体积调节至 100 mL。
- 取出培养基,用 1x PBS 冲洗成像皿两次,每次 5 分钟。
注:两次 PBS 洗涤有助于完全去除残留的血清和任何可能干扰实验的胰岛素或生长因子。 - 向成像皿中加入 2 mL 饥饿培养基(参见 材料表)。在培养皿边缘周围轻轻添加培养基,以避免细胞从玻璃底部移开。在 37 °C 下孵育 4 小时。
注意:4 小时孵育是同步细胞代谢的最佳选择;但是,可以根据实验需要延长持续时间。
6. HepG2 细胞的 FRET 活细胞成像
注:本节提供了 FRET 活细胞成像的说明,以监测单个 HepG2 细胞中 Akt 磷酸化的时空动力学。优化活 HepG2 细胞的显微镜设置、处理程序和成像条件至关重要,如下所述。显微镜设置对于优化FRET成像的成像条件至关重要。根据制造商的说明逐步遵循 PC/共聚焦激光扫描显微镜 (CLSM) 设置,以确保稳定运行。用于 FRET 成像的定制 CLSM 配置如图 6 所示。
- 打开遥控开关,为显微镜、计算机、扫描仪、激光发射、压电载物台和落射荧光 LED 光源供电。开机前确保所有组件都已正确连接,以避免任何潜在的损坏。
- 将钥匙转到激光发射上的 ON 位置,然后按下按钮以激活 FRET 所需的两个激光器(457 nm 和 514 nm 激光线)。
注意:确保适当的过滤器和设置到位以实现最佳 FRET 成像。激光线的选择应基于生物传感器的激发和发射曲线。 - 打开电源板,为连接到显微镜的计算机和显示器供电。
注意: 开机前请确保所有连接都牢固,以防止任何设备损坏。 - 登录 Windows 并启动显微镜软件。
- 单击 A1 for Acquisition 以启动映像设置。根据 FRET 实验的要求选择合适的光学配置。
- 根据需要调整激光功率和探测器灵敏度,以获得最佳成像条件。小心安装显微镜载物台顶部培养箱并用螺丝固定。
注意:请勿将螺钉拧得过紧,以免损坏培养箱或显微镜载物台。 - 用无菌双蒸水 (ddH2O) 填充内部水浴。牢固地安装顶部加热器并打开舞台加热器、浴缸加热器和镜头加热器的电源(图 7A)。
注意: 请勿装得太满,以免溢出到系统中。确保所有加热器都正常工作,以保持活细胞成像的一致温度条件。 - 使用 40 倍油浸镜头进行成像(参见 材料表)。
注意: 有关技术规格,请参阅制造商的网站。 - 用蘸有 95% 乙醇的镜头纸擦拭物镜。将一小滴浸油放在物镜上(图 7B)。将含有 HepG2 细胞的成像皿放在显微镜载物台上,并用支架固定(图 7C)。
注意:确保成像皿正确对齐并固定,以防止在成像过程中发生任何移动。避免对镜头施加压力,以防止损坏镜头和培养皿的玻璃底部。 - 关闭腔室并将活细胞室内的细胞孵育 1-2 小时以使其平衡(图 7D)。
- 在延时成像期间,以特定的时间间隔暂停并轻轻去除培养基(图 7E),然后加入 1 mL 具有给定胰岛素浓度的新鲜制备的培养基(图 7F, 材料表)。
注:通过将 1 mg 胰岛素溶解在 1 mL 10 mM 乙酸中来制备 1 mg/mL 胰岛素储备液。通过 0.2 μm 无菌注射器过滤器过滤溶液,并将等分试样储存在 −20 °C。 - 从显微镜软件的 File 菜单启动 ND Acquisition 窗口。
注意:ND 采集设置如图 8 所示。 - 选择 Time 选项卡以设置延时成像的间隔、持续时间和所需的循环。接下来,单击 XY 选项卡并按 + 添加 按钮以包含用于成像的单个 HepG2 细胞。
- 要添加多个单元格,请找到合适的单元格、扫描、微调焦点并锁定点扩散函数 (PSF)。对为成像添加的每个新单元格重复此步骤。扫描成像培养皿中不同位置的多个细胞,以识别要包含的各种靶细胞。
- 单击红色的 X 图标以取消选择单元格。选中 'Z' 框以设置 Z 位置。选择所有参数后,在 'File Name' 框中输入实验名称。
- 点击 浏览 选择目标文件夹,然后选中 save to file 复选框。点击 Run now 选项卡以启动 ND 采集。ND 采集窗口将显示延时成像的实时进度,包括经过的时间和剩余时间。
- 记录基线测量值长达 30 分钟,而不用胰岛素刺激细胞。
- 找到合适的细胞,放大/缩小以聚焦单个细胞,并选择具有高荧光强度的细胞。逐个添加单元格,最多 6 个单元格。
注意:限制细胞数量,以防止成像过程中图像采集延迟和窗口冻结。 - 设置延时成像的时间和成像频率。开始延时成像。
注意:在开始成像会话之前,请确保所有参数(包括曝光时间和激光功率)都经过优化。 - 定期暂停成像,轻轻地从玻璃底培养皿中取出培养基,并加入 1 mL 补充有适当胰岛素浓度的培养基。
- 恢复图像采集。根据需要重复步骤 6.20。
- 在实验结束时,单击 Finish 选项卡以关闭。安全地备份和存储采集的图像以供分析。
7. 数据分析
- 校正来自对照样品的预处理延时 FRET 成像数据的光谱串扰和 CFP 自发荧光,从而获得校正的 FRET 值和精确的 FRET 效率。按照先前研究 27,28,29,30 中概述的协议,使用成像软件采集、处理和分析 FRET 图像(图 9)。
8. FRET效率计算
- 为了确定FRET效率,采集七张图像:细胞(IDA(D)、 IDD(D)、 IDA(A)、 IAA(A)、 IDD、 IAA和 IDA)。使用以下公式计算 FRET 效率:
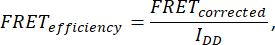
其中,FRET校正由下式获得:
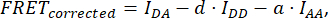
其中 d 和 a 定义为:
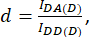
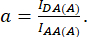
此处 IDA、IDD 和 IAA 表示用生物传感器转染的细胞的图像。IDA(D) 和 IDD(D)) 是仅供体对照图像,IDA(A) 和 IAA(A) 是仅供体对照图像。
9. 图像采集
- IDA : 通过供体激发 (434 nm) 和受体发射 (530 nm) 获得图像。
- IDD : 通过供体激发 (434 nm) 和供体发射 (477 nm) 获得图像。
- IAA : 通过受体激发 (517 nm) 和受体发射 (530 nm) 获得图像。
- IDA(D) 和 IDA(A):使用与 IDA 相同的激发和发射获取图像,但分别来自仅表达供体和受体的细胞。
- IDD(D) :在与仅供体对照的 IDD 相同的条件下获取图像。
- IAA(A) :使用与 IAA 相同的条件获取图像,但特定于仅接受体控件。
10. 背景校正
- 在计算机上打开图像分析软件。从顶部菜单栏中,访问 文件 菜单。选择 可选 or 打开文件 从下拉选项中。
- 导航到包含延时图像数据的目录。找到需要分析的延时图像(例如,nd2 格式)。
- 选择文件并单击 可选 or OK 将其加载到软件中以供查看和分析。在单元格中定义感兴趣区域 (ROI)(或使用整个单元格作为 ROI),并记录此区域内每个像素的灰度值。
- 选择无单元格区域作为背景并计算其平均灰度值。通过从单元格 ROI 中每个像素的灰度值中减去此背景平均值来创建校正后的图像。
11. FRET 渗漏(串扰)消除
注:供体发射和受体激发之间的光谱重叠如图 3B 所示,这对于 FRET 效率和能量转移过程至关重要。延时 FRET 成像中的渗出是一个重大挑战,它是由供体和受体荧光团的光谱重叠引起的,导致测量不准确。串扰是固有的,因为供体和受体荧光团的光谱在一定程度上重叠(图 3C、D)。高荧光团浓度和过滤器配置不当等因素会加剧此问题。解决渗漏问题对于确保 FRET 测量的可靠性至关重要。
- 有关减轻渗漏效应的方法,请参阅以前的研究9。
12. 量化和统计分析
- 使用统计分析软件执行统计分析。
结果
为了研究 HepG2 细胞中的 Akt 活化,将细胞接种到预包被的成像皿上,并用基于 FRET 的生物传感器 pEevee-iAkt-NES(图 2A)转染,旨在实时监测 Akt 磷酸化。转染后,细胞在无血清培养基中进行血清饥饿 4 小时,以同步其代谢状态并最大限度地减少基础胰岛素信号转导。
随后将细胞暴露于不同浓度的胰岛素 (0 pM、300 pM、400 pM、500 pM?...
讨论
用于监测 HepG2 细胞中 Akt 磷酸化的活细胞 FRET 成像方案涉及几个关键步骤,以确保可靠和可重复的结果。第一个关键步骤是细胞培养,包括常规细胞维护、成像培养皿包被和细胞接种。在延时成像实验中,适当的涂层对于细胞粘附至关重要,因为它可以确保稳定的细胞粘附,防止分离,并最大限度地减少漂移,从而导致数据不一致 9,32
披露声明
作者声明没有利益冲突。
致谢
这项工作得到了深圳市自然科学基金 (JCYJ20240813113606009)、深港科技创新合作区 (HZQB-KCZYB-2020056)、国家自然科学基金 (32070681)、国家重点研发计划 (2019YFA0906002) 和深圳孔雀计划 (KQTD2016053117035204) 的部分支持。
材料
| Name | Company | Catalog Number | Comments |
| 0.25% trypsin-EDTA | Gibco | Cat#25200-056 | Use ice-cold PBS for cell wash |
| 15 mm glass bottom cell culture dish | NEST | Cat#801001 | |
| 2 mL Nalgene cryogenic vials | Thermo Scientific | Cat#5012-0020 | |
| 5 mL Stripette Serological Pipets | Corning | Cat#4487 | |
| 95% Ethanol | Kermel | Cat#C028005 | |
| A1 HD25/A1R HD25 confocal microscope | Nikon | https://www.nikon.com/ | Magnification: 40×, Numerical Aperture (NA): 1.30, Pixel Dwell Time: 2.4 ms, Pixel Size: 1024 |
| Ampicillin | Sigma-Aldrich | Cat#A9393 | |
| Bovine serum albumin (BSA) | VWR Life Science | Cat#N208-10g | |
| Corning 25 cm2 rectangular culture flasks | Corning | Cat#430639 | |
| Countess 3 automated cell counter | Thermo Scientific | http://www.thermofisher.com/#AMQAX2000 | |
| Countess cell counting chamber slides | Thermo Scientific | http://www.thermofisher.com/#C10228 | |
| Digital vortex mixers | Thermo Scientific | https://www.thermofisher.com/ | |
| Dimethyl sulfoxide | Sigma-Aldrich | Cat#D2650 | |
| Eppendorf Safe-Lock Tubes 1.5 mL | Eppendorf | Cat#022363204 | |
| EZ-PCR mycoplasma detection kit | Biological Industries | Cat# 20-700-20 | |
| Fetal Bovine Serum, qualified, Australia | Gibco | Cat#10099141 | |
| GlutaMAX Supplement | Gibco | Cat#35050061 | |
| GraphPad Prism 9 | GraphPad Software | https://www.graphpad.com/ | |
| HepG2 | National Collection of Authenticated Cell Cultures | #CSTR:19375.09.3101HUMSCSP510 http://www.cellbank.org.cn/ | |
| Immersion Oil Type 37 | Cargille Laboratories | Cat #16237 | |
| Insulin | Sigma-Aldrich | Cat#I5500-50MG | Warm to 37 °C before use |
| LB Broth (1x) | Invitrogen | Cat#10855001 | |
| Minimum Essential Medium (MEM) | Gibco | Cat#11095080 | Warm to 37 °C before use |
| mySPIN 12 mini centrifuge | Thermo Scientific | https://www.thermofisher.com/ | |
| NanoDrop One | Thermo Scientific | https://www.thermofisher.com | |
| Nikon Plan Fluor 40×/1.30 Oil Lens | Nikon | https://www.nikon.com/ | |
| NIS-Elements-AR | Nikon | https://www.nikon.com/ | |
| Non-Essential Amino Acids (NEAA) (100x) | Gibco | Cat#11140050 | |
| One Shot LB Agar Plates | Invitrogen | Cat#A55802 | |
| One Shot Stbl3 chemically competent E. coli | Invitrogen | Cat#C737303 | |
| Parafilm | PARAFILM | Cat#B8R05606 | |
| PBS (phosphate buffered saline) | Gibco | Cat#10010023 | |
| pEevee-iAkt-NES (7,033 bp) | Miura et al31 | https://benchling.com/s/seq-q46zFYCfl0swLAun0t28/edit | |
| Penicillin-streptomycin | Gibco | Cat#15070063 | |
| Plasmocin prophylactic | InvivoGen | Cat#ant-mpp | |
| Poly-L-Lysine Hydrobromide | Sigma-Aldrich | Cat#P4832 | |
| Precision general purpose baths | Thermo Scientific | https://www.thermofisher.com/ | |
| QIAprep spin miniprep kit | QIAGEN | Cat#27106 | |
| SnapGene | SnapGene by Dotmatics | https://www.snapgene.com | |
| Sodium Pyruvate (100 mM) | Gibco | Cat#11360070 | |
| Syringe filter unit, 0.22 μm | Millipore | Cat#SLGP033RS | |
| Tokai Hit stage top incubator | TOKAI HIT | https://www.tokaihit-livecell.com/stagetopincubator | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | Cat#10977015 | |
| Xfect Transfection Reagent | Takara Bio | Cat#631317 |
参考文献
- GBD 2021 Diabetes Collaborators. Global, regional, and national burden of diabetes from 1990 to 2021, with projections of prevalence to 2050: a systematic analysis for the Global Burden of Disease Study 2021. Lancet. 402 (10397), 203-234 (2023).
- Li, M., et al. Trends in insulin resistance: insights into mechanisms and therapeutic strategy. Signal Transduct Target Ther. 7 (1), 216 (2022).
- Poloz, Y., Stambolic, V. Obesity and cancer, a case for insulin signaling. Cell Death Dis. 6 (12), e2037 (2015).
- Arcidiacono, B., et al. Insulin resistance and cancer risk: an overview of the pathogenetic mechanisms. Exp Diabetes Res. 2012, 789174 (2012).
- Godsland, I. F. Insulin resistance and hyperinsulinaemia in the development and progression of cancer. Clin Sci (Lond). 118 (5), 315-332 (2009).
- Tsugane, S., Inoue, M. Insulin resistance and cancer: epidemiological evidence. Cancer Sci. 101 (5), 1073-1079 (2010).
- Yudhani, R. D., et al. In vitro insulin resistance model: A recent update. J Obes. 2023, 1964732 (2023).
- Akhtar, J., et al. Bistable insulin response: The win-win solution for glycemic control. iScience. 25 (12), 105561 (2022).
- Akhtar, J., Imran, M., Wang, G. Protocol for live-cell Forster resonance energy transfer imaging to reveal the bistable insulin response of single C2C12-derived myotubes. STAR Protoc. 5 (2), 103109 (2024).
- Kamino, K., et al. Optimal inference of molecular interaction dynamics in FRET microscopy. Proc Natl Acad Sci U S A. 120 (15), e2211807120 (2023).
- Kraft, A. E., Nikolaev, V. O. FRET microscopy for real-time visualization of second messengers in living cells. Methods Mol Biol. 1563, 85-90 (2017).
- Veeriah, S., et al. High-throughput time-resolved FRET reveals Akt/PKB activation as a poor prognostic marker in breast cancer. Cancer Res. 74 (18), 4983-4995 (2014).
- Conway, J. R. W., et al. Monitoring AKT activity and targeting in live tissue and disease contexts using a real-time Akt-FRET biosensor mouse. Sci Adv. 9 (17), eadf9063 (2023).
- Chandris, P., Giannouli, C. C., Panayotou, G. Imaging approaches for the study of metabolism in real time using genetically encoded reporters. Front Cell Dev Biol. 9, 725114 (2021).
- Yang, J., et al. Longitudinal FRET imaging of glucose and lactate dynamics and response to therapy in breast cancer cells. Mol Imaging Biol. 24 (1), 144-155 (2022).
- Mandrou, E., et al. A reliable system for quantitative G-protein activation imaging in cancer cells. Cells. 13 (13), 1114 (2024).
- Fang, C., Huang, Y., Zhao, Y. Review of FRET biosensing and its application in biomolecular detection. Am J Transl Res. 15 (2), 694-709 (2023).
- Miller, J. N. Fluorescence energy transfer methods in bioanalysis. Analyst. 130 (3), 265-270 (2005).
- Verveer, P. J., Rocks, O., Harpur, A. G., Bastiaens, P. I. Imaging protein interactions by FRET microscopy: FRET measurements by acceptor photobleaching. CSH Protoc. 2006 (6), (2006).
- Vu, C. Q., Arai, S. Quantitative imaging of genetically encoded fluorescence lifetime biosensors. Biosensors (Basel). 13 (10), 939 (2023).
- Wang, G. A more holistic view of the logarithmic dose-response curve offers greater insights into insulin response. J Biol Chem. 301 (1), 108037 (2025).
- Wang, G. Body mass dynamics is determined by the metabolic Ohm's law and adipocyte-autonomous fat mass homeostasis. iScience. 23 (6), 101176 (2020).
- Wang, G. Raison d'être of insulin resistance: the adjustable threshold hypothesis. J R Soc Interface. 11 (101), 20140892 (2014).
- Wang, G. Optimal homeostasis necessitates bistable control. J R Soc Interface. 9, 2723 (2012).
- Heim, R., Tsien, R. Y. Engineering green fluorescent protein for improved brightness, longer wavelengths and fluorescence resonance energy transfer. Curr Biol. 6 (2), 178-182 (1996).
- Nguyen, A. W., Daugherty, P. S. Evolutionary optimization of fluorescent proteins for intracellular FRET. Nat Biotechnol. 23 (3), 355-360 (2005).
- Broussard, J. A., Rappaz, B., Webb, D. J., Brown, C. M. Fluorescence resonance energy transfer microscopy as demonstrated by measuring the activation of the serine/threonine kinase Akt. Nat Protoc. 8 (2), 265-281 (2013).
- Gordon, G. W., Berry, G., Liang, X. H., Levine, B., Herman, B. Quantitative fluorescence resonance energy transfer measurements using fluorescence microscopy. Biophys J. 74 (5), 2702-2713 (1998).
- Tron, L., et al. Flow cytometric measurement of fluorescence resonance energy transfer on cell surfaces. Quantitative evaluation of the transfer efficiency on a cell-by-cell basis. Biophys J. 45 (5), 939-946 (1984).
- Youvan, D. C., et al. Calibration of fluorescence resonance energy transfer in microscopy. , (2002).
- Miura, H., Matsuda, M., Aoki, K. Development of a FRET biosensor with high specificity for Akt. Cell Struct Funct. 39 (1), 9-20 (2014).
- Liberio, M. S., Sadowski, M. C., Soekmadji, C., Davis, R. A., Nelson, C. C. Differential effects of tissue culture coating substrates on prostate cancer cell adherence, morphology and behavior. PLoS One. 9 (11), e112122 (2014).
- Rani, K., Sengupta, S. Multi-stimuli programmable FRET based RGB absorbing antennae towards ratiometric temperature, pH and multiple metal ion sensing. Chem Sci. 12 (47), 15533-15542 (2021).
- Betolngar, D. B., et al. pH sensitivity of FRET reporters based on cyan and yellow fluorescent proteins. Anal Bioanal Chem. 407 (14), 4183-4193 (2015).
- Salonikidis, P. S., et al. An ion-insensitive cAMP biosensor for long term quantitative ratiometric fluorescence resonance energy transfer (FRET) measurements under variable physiological conditions. J Biol Chem. 286 (26), 23419-23431 (2011).
- Youvan, D. C., et al. Calibration of fluorescence resonance energy transfer in microscopy using genetically engineered GFP derivatives on nickel chelating beads. Biotechnol Alia. 3, 1-18 (1997).
- Menaesse, A., et al. Simplified instrument calibration for wide-field fluorescence resonance energy transfer (FRET) measured by the sensitized emission method. Cytometry A. 99 (4), 407-416 (2021).
- Batta, A., Hajdu, T., Nagy, P. Improved estimation of the ratio of detection efficiencies of excited acceptors and donors for FRET measurements. Cytometry A. 103 (7), 563-574 (2023).
- Coullomb, A., et al. QuanTI-FRET: a framework for quantitative FRET measurements in living cells. Sci Rep. 10 (1), 6504 (2020).
- Hoppe, A., Christensen, K., Swanson, J. A. Fluorescence resonance energy transfer-based stoichiometry in living cells. Biophys J. 83 (6), 3652-3664 (2002).
- Zal, T., Gascoigne, N. R. Photobleaching-corrected FRET efficiency imaging of live cells. Biophys J. 86 (6), 3923-3939 (2004).
- Hochreiter, B., Kunze, M., Moser, B., Schmid, J. A. Advanced FRET normalization allows quantitative analysis of protein interactions including stoichiometries and relative affinities in living cells. Sci Rep. 9 (1), 8233 (2019).
- Liu, H., Zhang, F., Mishra, S. K., Zhou, S., Zheng, J. Knowledge-guided fuzzy logic modeling to infer cellular signaling networks from proteomic data. Sci Rep. 6, 35652 (2016).
- Garrido-Rodriguez, M., Zirngibl, K., Ivanova, O., Lobentanzer, S., Saez-Rodriguez, J. Integrating knowledge and omics to decipher mechanisms via large-scale models of signaling networks. Mol Syst Biol. 18 (7), e11036 (2022).
- Greenwald, E. C., Polanowska-Grabowska, R. K., Saucerman, J. J. Integrating fluorescent biosensor data using computational models. Methods Mol Biol. 1071, 227-248 (2014).
- Zhao, Z., Xia, J. Computational Approaches for Modeling Signal Transduction Networks. Encyclopedia of Bioinformatics and Computational Biology. , (2019).
- Wang, G., Krueger, G. R. Computational analysis of mTOR signaling pathway: bifurcation, carcinogenesis, and drug discovery. Anticancer Res. 30 (7), 2683-2688 (2010).
- Wang, G. Singularity analysis of the AKT signaling pathway reveals connections between cancer and metabolic diseases. Phys Biol. 7 (4), 046015 (2010).
- Chedere, A., Hari, K., Kumar, S., Rangarajan, A., Jolly, M. K. Multi-stability and consequent phenotypic plasticity in AMPK-Akt double negative feedback loop in cancer cells. J Clin Med. 10 (3), 472 (2021).
- Mosca, E., et al. Computational modeling of the metabolic States regulated by the kinase akt. Front Physiol. 3, 418 (2012).
- Liao, J., Madahar, V., Dang, R., Jiang, L. Quantitative FRET (qFRET) technology for the determination of protein-protein interaction affinity in solution. Molecules. 26 (21), 6339 (2021).
- Verma, A. K., Noumani, A., Yadav, A. K., Solanki, P. R. FRET based biosensor: Principle applications recent advances and challenges. Diagnostics (Basel). 13 (8), 1375 (2023).
- Mattheisen, J. M., et al. Application of bioluminescence resonance energy transfer to quantitate cell-surface expression of membrane proteins. Anal Biochem. 684, 115361 (2024).
- Lionetti, M. C., La Porta, C. A. M. FLIM-FRET investigation of heterogeneous huntingtin aggregation in HeLa cells. Methods Mol Biol. 2551, 595-604 (2023).
- Petutschnig, E. K., Pierdzig, L., Mittendorf, J., Niebisch, J. M., Lipka, V. A novel fluorescent protein pair facilitates FLIM-FRET analysis of plant immune receptor interaction under native conditions. J Exp Bot. 75 (3), 746-759 (2024).
- Sprenger, J. U., Perera, R. K., Gotz, K. R., Nikolaev, V. O. FRET microscopy for real-time monitoring of signaling events in live cells using unimolecular biosensors. J Vis Exp. 66, e4081 (2012).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。