Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Lebendzell-Förster-Resonanz-Energietransfer-Bildgebung der metabolisch regulierten Akt-Aktivierungsdynamik in HepG2-Zellen
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll vor, um die räumlich-zeitliche Dynamik der Akt-Aktivierung und Phosphorylierung in lebenden HepG2-Zellen zu quantifizieren. Die Förster-Resonanzenergietransfer-Bildgebung (FRET) ist ein leistungsfähiges Werkzeug, das wertvolle Einblicke in die Insulinsignalwege und die Stoffwechselregulation in Krebszellen liefert.
Zusammenfassung
Die metabolisch regulierte Akt-Aktivierung ist ein kritischer Knotenpunkt in der Insulinsignalkaskade und liefert wertvolle Einblicke in die Beziehung zwischen Diabetes und Krebs. Um die Akt-Aktivität in HepG2-Zellen genau zu quantifizieren, haben wir ein robustes, reproduzierbares Protokoll entwickelt, das den Förster Resonance Energy Transfer (FRET) mit genetisch kodierten Akt-spezifischen Biosensoren verwendet. Dieses Protokoll beschreibt detaillierte Schritte für die Zellkultur, die Vorbereitung der Bildgebungsschale und die Transfektion von HepG2-Zellen zur Expression von FRET-basierten Biosensoren sowie spezifische Richtlinien für die Hardware- und Softwarekonfiguration von konfokalen Laserscanning-Mikroskopen. Die Ergebnisse zeigten einzigartige Muster der Insulinsignalübertragung in HepG2-Zellen, die einen irreversiblen Schalter aufweisen, der durch eine konstitutive Akt-Aktivierung mit einer definierten Einschaltschwelle, aber ohne Abschaltschwelle gekennzeichnet ist. Im Gegensatz dazu weisen Myotuben einen reversiblen Schalter auf. Die anhaltende Akt-Aktivierung in HepG2-Zellen deutet auf Mechanismen hin, die der Insulinresistenz und der metabolischen Dysregulation in Leberzellen zugrunde liegen, mit breiteren Implikationen für das Verständnis des Fortschreitens von Stoffwechselstörungen und Krebs. Dieses Protokoll bietet einen wertvollen Rahmen für die Erforschung von Akt-bezogenen Signalwegen und zellulären Verhaltensweisen in verschiedenen Krankheitskontexten.
Einleitung
Diabetes mellitus stellt eine große globale gesundheitliche Herausforderung dar, die durch Insulinresistenz und eine gestörte Glukosehomöostase gekennzeichnetist 1. Ein umfassendes Verständnis der Insulin-Signalwege ist entscheidend für die Aufklärung der Pathophysiologie dieser Krankheit, da Insulin eine zentrale Rolle für den Glukosestoffwechsel, das Zellwachstum und das Überleben spielt2. Zahlreiche Studien haben gezeigt, dass die Insulinsignalisierung verschiedene Krebsarten signifikant beeinflusst, was die Insulinresistenz mit dem Fortschreiten des Tumors und schlechten Patientenergebnissen in Verbindung bringt 3,4,5,6. HepG2-Zellen, eine häufig verwendete hepatozelluläre Karzinomzelllinie, dienen als wertvolles Modell für die Untersuchung der Insulinresistenz und des Zusammenspiels zwischen metabolischer Dysregulation und Krebsentstehung7. Traditionell haben Forscher die Insulinreaktionen als abgestuft angesehen; Neuere Studien haben jedoch gezeigt, dass einzelne Zellen bistabile Reaktionen aufweisen können, die bei bestimmten Insulinkonzentrationsschwellen deutliche Übergänge zwischen Nichtreaktion und vollständiger Reaktion aufweisen 8,9.
Die Förster-Resonanzenergietransfer-Bildgebung (FRET) ist ein leistungsfähiges Werkzeug zur Untersuchung der räumlich-zeitlichen Verteilung von Biomolekülen in lebenden Zellen10. Durch die Extraktion von Informationen aus der Molekulardynamik bietet FRET Einblicke in Prozesse wie die Akt-Aktivierung in Echtzeit und ist damit eine unschätzbare Technik für die Untersuchung lebender Zellen11,12. Diese bildgebende Methode hat sich bei der Untersuchung der zellulären Dynamik als unverzichtbar erwiesen, insbesondere bei Stoffwechselerkrankungen und Krebs, bei denen präzise molekulare Wechselwirkungen entscheidend sind13. FRET ermöglicht auch die Echtzeitüberwachung molekularer Wechselwirkungen und gibt Aufschluss über Mechanismen wie Insulinresistenz und Tumorprogression14,15. FRET-Biosensoren sind in der Krebsforschung von entscheidender Bedeutung für die Untersuchung von Tumormikroumgebungen, Arzneimittelresistenzen und Stoffwechselstörungen16. FRET-Detektionsmethoden wie sensibilisierte Emission (SE), Akzeptorbleichen (AB), Fluoreszenzlebensdauer-Bildgebungsmikroskopie (FLIM) und Spektroskopie bieten jeweils deutliche Vorteile bei der Quantifizierung molekularer Wechselwirkungen17. SE misst den Energietransfer zwischen Donor- und Akzeptor-Fluorophoren, was zu einer messbaren Verschiebung der Emissionsspektren führt, die mit der Nähe wechselwirkender Biomoleküle korreliert18. AB verwendet selektives Photobleaching des Akzeptorfluorophors und verfolgt Änderungen der Donorfluoreszenz, was es den Forschern ermöglicht, die Interaktionskinetik und die Entfernungen zu beurteilen19. FLIM bewertet die Fluoreszenzzerfallsraten des Donorfluorophors, die direkt von der FRET-Effizienz beeinflusst werden, um präzise nanoskalige Messungen molekularer Wechselwirkungen zu ermöglichen20.
Mit Hilfe von FRET-Techniken haben wir kürzlich bistabile Insulinreaktionen in C2C12-abgeleiteten Myoröhrchen 8,9,21,22,23,24 nachgewiesen. Die unterschiedlichen Ein- und Ausschaltschwellen für die Akt-Aktivierung, wie wir entdeckt haben, deuten darauf hin, dass die abgestufte Ganzkörper-Insulin-Dosis-Wirkungs-Beziehung über die Komplexität der subzellulären Signalkaskade hinwegtäuscht, die mit dem Insulinstimulus beginnt und in einer Alles-oder-Nichts-Reaktion auf Einzelzellebene gipfelt 21,22,23,24. Um das Vorhandensein von Bistabilität in anderen Zelltypen zu testen, stimulierten wir HepG2-Zellen mit Insulin und zeichneten ihre Reaktion mit Hilfe von Einzelzell-FRET-Bildgebung auf. Wir stimulierten HepG2-Zellen mit unterschiedlichen Insulinkonzentrationen und überwachten die Akt-Aktivität auf Einzelzellebene mit einem Akt-Biosensor. Der Akt-Biosensor besteht aus einem verstärkten cyanfluoreszierenden Protein (ECFP)25 als Donorfluorophor und der hellsten Variante des gelb fluoreszierenden Proteins (YPet)26 als Akzeptorfluorophor, verbunden durch einen Evoli-Linker, der die Peptidsequenz SGRPRTTTFADSCKP enthält. Dieses Peptid fungiert als Substrat für phosphoryliertes Akt (pAkt), das aus der humanen Glykogensynthase-Kinase 3β (GSK3β) optimiert ist. Im unphosphorylierten Zustand überschreitet der räumliche Abstand zwischen den Donor- und Akzeptorfluorophoren den Förster-Radius, was den Energietransfer hemmt. Bei Insulinstimulation kommt es zu einer Akt-Phosphorylierung, die zur Phosphorylierung von SGRPRTTTFADSCKP führt. Dieser Prozess induziert eine Konformationsänderung, die Donor und Akzeptor in den Förster-Radius bringt, wodurch FRET27 ermöglicht wird. Infolgedessen korreliert die Intensität des FRET-Signals mit der Menge an phosphorylierten Akt-Molekülen und ermöglicht die Echtzeit-Quantifizierung von insulinvermittelten zellulären Reaktionen.
Dieses Protokoll, das ursprünglich entwickelt wurde, um die Insulinsignalisierung in C2C12-abgeleiteten Myotuben zu untersuchen, wurde erfolgreich auf HepG2-Zellen angewendet und auf verschiedenen Hardware- und Softwareplattformen eingesetzt, was seine Anwendbarkeit, Anpassungsfähigkeit und Vielseitigkeit unter Beweis stellt. HepG2-Zellen weisen eine konstitutive Akt-Aktivität auf, was sie zu einem idealen In-vitro-Modell für die Untersuchung leberspezifischer Insulinsignalwege und Stoffwechselprozesse macht. Die wichtigsten Funktionen des Protokolls werden Schritt für Schritt im Abschnitt Protokoll beschrieben.
Protokoll
Abbildung 1 bietet einen Überblick über die experimentellen Schritte der FRET-Lebendzellbildgebung zur Überwachung der Akt-Phosphorylierung in einzelnen HepG2-Zellen.
1. Plasmidaufnahme, -vermehrung und -reinigung
HINWEIS: In diesem Abschnitt werden die wesentlichen Schritte für die Erfassung, Amplifikation und Reinigung des Plasmids beschrieben, die für die Einzelzell-FRET-Analyse erforderlich sind.
- Verwenden Sie das Plasmid pEevee-iAkt-NES-YPet (Abbildung 2A).
HINWEIS: Das Plasmid wurde freundlicherweise von Prof. Kazuhiro Aoki vom National Institute for Basic Biology (NIBB), Japan, zur Verfügung gestellt.Plasmidkarten für die FRET-Biosensoren, die für die Akt-Überwachung verwendet werden, und ihre jeweiligen Steuerungen sind in Abbildung 2 dargestellt. Der pEevee-iAkt-NES-ECFP (Spender; Abbildung 2B) und pEevee-iAkt-NES-Ypet (Akzeptor; Abbildung 2C) Plasmide werden als Kalibrierungskontrollen bei FRET-Experimenten verwendet9.
HINWEIS: Abbildung 3 zeigt die Zusammensetzung und den Mechanismus des intramolekularen FRET-Biosensors. - Um das Plasmid zu vermehren, führen Sie eine bakterielle Transformation mit chemisch kompetenten E. coli-Zellen durch (siehe Materialtabelle). Die transformierten Zellen werden auf Luria-Bertani (LB)-Agar mit 100 μg/ml Ampicillin aufgetragen und über Nacht bei 37 °C inkubiert. Am nächsten Tag wählen Sie eine Ampicillin-resistente Kolonie aus und kultivieren Sie sie in LB-Bouillon, die mit 100 μg/ml Ampicillin zur Plasmidamplifikation ergänzt wurde.
- Um die Plasmid-DNA aufzureinigen, verwenden Sie ein kommerziell erhältliches Plasmid-DNA-Aufreinigungskit (siehe Materialtabelle), um eine hohe Reinheit und Ausbeute zu erzielen. Beurteilen Sie die DNA-Qualität durch Messung der Absorptionsverhältnisse (A260/A280 und A260/A230) mit einem Spektralphotometer (siehe Materialtabelle). Verhältnisse zwischen 1,8-2,0 und 2,0-2,2 gelten als optimal. Überprüfen Sie die Integrität des Plasmids durch Agarose-Gelelektrophorese, um sicherzustellen, dass es für die Transfektion geeignet ist.
2. Ablauf der Zellkultur
HINWEIS: Führen Sie alle Zellkulturverfahren in einer Laminar-Flow-Haube durch, um eine sterile Umgebung zu gewährleisten und eine Kontamination zu vermeiden. Der Arbeitsablauf der HepG2-Zellkultur ist in Abbildung 4 dargestellt. Das komplette Medium für HepG2-Zellen besteht aus Minimum Essential Medium (MEM), 10 % fötalem Rinderserum (FBS), 1 % nicht-essentiellen Aminosäuren (NEAA), 1 mM Natriumpyruvat, 2 mM L-Glutamin-Ergänzung, 100 U/mL Penicillin-Streptomycin und 2,5 μg/ml Antibiotika-Antimykotika-Lösung (siehe Materialtabelle, Tabelle 1).
- Tauen Sie gefrorene HepG2-Zellen schnell auf, indem Sie das Fläschchen in einen 37 °C Thermomixer oder ein Wasserbad stellen, bis es vollständig aufgetaut ist.
- Übertragen Sie die aufgetauten Zellen in ein konisches 15-ml-Röhrchen mit 10 mL vollständigem Wachstumsmedium (siehe Materialtabelle).
HINWEIS: Erwärmen Sie das gesamte Wachstumsmedium vor der Verwendung auf 37 °C, um einen Temperaturschock für die Zellen zu minimieren. - Zentrifugieren Sie das Röhrchen bei 200 x g für 5 min, um die Zellen zu pelletieren.
- Saugen Sie den Überstand vorsichtig mit einer Pipettenspitze mit breiter Bohrung an, um eine Störung des Zellpellets zu vermeiden.
- Resuspendieren Sie das Pellet in 10 mL frischem Vollwachstumsmedium.
- Die Zellsuspension wird in einen 75 cm² großen Gewebekulturkolben überführt.
- Der Kolben wird bei 37 °C in einer befeuchteten Atmosphäre mit 5 % CO2 inkubiert.
HINWEIS: Beobachten Sie die Zellen in den nächsten 24-48 Stunden, um die Anheftung zu bestätigen und die Erholung zu beurteilen. Stören Sie die Zellen mindestens 4 Stunden lang nicht, um eine korrekte Anheftung zu gewährleisten, bevor Sie das Medium wechseln. Vermeiden Sie es, den Inkubator in den ersten 4 Stunden häufig zu öffnen, da dies die Zelladhäsion stören kann. Befolgen Sie die institutionellen Biosicherheitsprotokolle und verwenden Sie geeignete persönliche Schutzausrüstung (PSA), um während aller Zellkulturverfahren eine sichere Arbeitsumgebung zu gewährleisten. Subkultur von HepG2-Zellen, wenn sie eine Konfluenz von 70 % bis 80 % erreichen, um optimale Wachstumsbedingungen aufrechtzuerhalten und eine Überfüllung zu verhindern, die sich auf die Lebensfähigkeit und das Wachstumspotenzial der Zellen auswirken kann. - Für die Subkultivierung aspirieren Sie das Medium und spülen die Zellen einmal mit 5 ml phosphatgepufferter Kochsalzlösung (PBS).
- Fügen Sie 1 ml 0,25 % Trypsin hinzu, um die Zellmonoschicht zu bedecken (siehe Materialtabelle).
HINWEIS: Stellen Sie sicher, dass das Trypsin für eine optimale Aktivität auf 37 °C vorgewärmt ist. Übertrypsinisieren Sie die Zellen nicht, da dies die Lebensfähigkeit beeinträchtigen kann. Überwachen Sie die Zellen unter einem Mikroskop, um die Ablösung zu bestätigen. - Bei 37 °C ca. 5 min inkubieren.
- Wenn sich die Zellen lösen, fügen Sie 2 ml vollständiges Medium hinzu, um das Trypsin zu neutralisieren, und sammeln Sie die Zellen durch Pipettieren.
- Pipettieren Sie die Zellsuspension vorsichtig, um die Klumpen aufzubrechen und eine Einzelzellsuspension zu erhalten.
- Geben Sie 3 ml vollständiges Medium in jeden neuen Kolben und übertragen Sie die Zellen dann in einem Teilungsverhältnis von 1:2 auf jeden Kolben.
HINWEIS: Für frühe Passagen teilen Sie die Zellen in einer Verdünnung von 1:2. Nach den Passagen 4-5 können je nach Bedarf Verdünnungen von 1:4 oder 1:5 durchgeführt werden. - Inkubieren Sie die Zellen bei 37 °C in einer befeuchteten Atmosphäre mit 5 % CO2.
HINWEIS: Überprüfen Sie die Zellen nach 24 Stunden, um die Anheftung zu bestätigen und die Wiederherstellung zu beurteilen. - Für die Routinekultur wechseln Sie das Nährmedium alle 2-3 Tage oder früher, wenn sich der pH-Indikator von rosa zu gelb ändert, was auf eine Versauerung hinweist.
HINWEIS: Lassen Sie das Medium nicht zu sauer werden, da dies die Zellen schädigen kann. - Überprüfen Sie die Zellmorphologie regelmäßig unter dem Mikroskop, um die Zellgesundheit zu gewährleisten.
HINWEIS: Für die kurzfristige Lagerung hepG2-Zellen bei -80 °C einfrieren; Für die Langzeitlagerung in flüssigem Stickstoff lagern. Die Zusammensetzung des in diesem Versuch verwendeten Gefriermediums ist in Tabelle 2 dargestellt.
3. Beschichten von Bildgebungsschalen mit Poly-L-Lysin
- Verwenden Sie 1 ml einer 0,1 mg/ml-Poly-L-Lysin-Lösung pro Bildgebungsschale, um die gesamte Oberfläche zu bedecken (siehe Materialtabelle).
HINWEIS: Passen Sie die Poly-L-Lysin-Konzentration basierend auf den spezifischen Anforderungen des Zelltyps an. - Schütteln Sie die Schale vorsichtig, um eine gleichmäßige Beschichtung der Kulturoberfläche zu erzielen.
HINWEIS: Stellen Sie während des gesamten Prozesses sterile Bedingungen sicher, um eine Kontamination zu vermeiden. - Inkubieren Sie die Gerichte über Nacht bei Raumtemperatur (RT).
- Überschüssige Poly-L-Lysin-Lösung durch Pipettieren aus den Schalen aspirieren.
- Spülen Sie die Oberfläche dreimal mit PBS ab und ruhen Sie jedes Mal 5 Minuten lang (siehe Materialtabelle). Entfernen Sie das ungebundene Poly-L-Lysin vollständig aus der Bildgebungsschale, um eine Hemmung des Zellwachstums zu verhindern. Spülen Sie die Platten vorsichtig ab, um ein Abkratzen oder Beschädigen des Glasbodens zu vermeiden.
- Trocknen Sie die beschichteten Bildgebungsschalen mindestens 3 h lang bei 37 °C an der Luft.
- Verwenden Sie die beschichteten Bildgebungsschalen sofort oder lagern Sie sie bis zu 2 Wochen bei 4 °C.
4. Transfektion von HepG2-Zellen
HINWEIS: Die HepG2-Transfektionsmethode ist in Abbildung 5 dargestellt.
- Satieren Sie HepG2-Zellen 24-48 Stunden vor der Transfektion in vorbeschichteten Bildgebungsschalen, um sicherzustellen, dass sie eine Konfluenz von 70 % bis 90 % erreichen.
- Tauen Sie das Transfektionsreagenz und das Plasmid, das für den FRET-Biosensor kodiert, auf Eis auf. Gründlich vortexen und vor Gebrauch eine kurze Drehung (z.B. 5.000 x g für 5 s) durchführen (siehe Materialtabelle).
HINWEIS: Stellen Sie sicher, dass das Transfektionsreagenz und das Plasmid vor der Verwendung vollständig aufgetaut sind.
ACHTUNG: Vermeiden Sie wiederholte Gefrier-Auftau-Zyklen, da dies die Wirksamkeit des Transfektionsreagenzes verringern kann. - Geben Sie 8 μg Plasmid, das für den FRET-Biosensor mit dem Reaktionspuffer kodiert, bis zu einem Endvolumen von 100 μl. Mischen Sie gut, indem Sie 5 s lang bei hoher Geschwindigkeit (ca. 3.000-5.000 x g) vortexen.
HINWEIS: Geben Sie immer ein Plasmid in den Puffer, bevor Sie das Transfektionsreagenz auf Polymerbasis hinzufügen. Mindestens 50 μl der Lösung müssen aus dem Reaktionspuffer bestehen (siehe Materialtabelle). - 2,4 μl des Transfektionspolymers werden in das Röhrchen mit der verdünnten Plasmid-DNA gegeben. Gut mischen, indem 15 s bei hoher Geschwindigkeit (ca. 3.000-5.000 × g) vortexen.
HINWEIS: Verwenden Sie immer 0,3 μl des Transfektionspolymers pro 1 μg DNA.
ACHTUNG: Stellen Sie sicher, dass das Transfektionspolymer gründlich mit der Plasmid-DNA vermischt wird, um einheitliche Nanopartikelkomplexe zu bilden. - Inkubieren Sie die Mischung aus Biosensor und Polymer 15 Minuten lang bei 37 °C, damit sich Nanopartikelkomplexe bilden können.
HINWEIS: Vermeiden Sie es, das Transfektionspolymer länger als 30 Minuten in Lösung zu halten, da dies die Transfektionseffizienz verringern kann.
VORSICHT: Überwachen Sie die Inkubationszeit sorgfältig, um eine Überinkubation zu vermeiden, die zu einer verminderten Transfektionseffizienz führen kann. - Drehen Sie das Röhrchen 5 s lang bei 5.000 x g herunter, um den Inhalt am Boden zu sammeln, und geben Sie dann die gesamten 100 μl der Nanopartikel-Komplexlösung tropfenweise in das Zellkulturmedium. Den Teller zum Mischen vorsichtig hin und her wiegen.
HINWEIS: Stellen Sie sicher, dass die Nanopartikel-Komplexlösung tropfenweise hinzugefügt wird, um sie gleichmäßig auf dem Zellkulturmedium zu verteilen.
ACHTUNG: Vermeiden Sie starkes Schaukeln, da dies zu Zellverdrängungen oder zu einer ungleichmäßigen Verteilung der Komplexe führen kann. - Inkubieren Sie die Platte bei 37 °C für 4 h bis über Nacht.
HINWEIS: Die Inkubationszeit kann je nach experimentellem Bedarf angepasst werden, aber 4 h sind in der Regel ausreichend für eine effiziente Transfektion.
ACHTUNG: Vermeiden Sie eine längere Inkubation (>16 h), da dies die Lebensfähigkeit der Zellen verringern kann. - Entfernen Sie die Nanopartikelkomplexe durch Aspiration aus den Zellen, ersetzen Sie sie durch 2 mL frisches vollständiges Wachstumsmedium und stellen Sie die Platte bis zum Zeitpunkt der Analyse wieder in den 37 °C-Inkubator. Die maximale Expression erreicht typischerweise 48 h nach der Transfektion.
- Analysieren Sie die Zellen unter Fluoreszenzmikroskopie.
HINWEIS: Stellen Sie sicher, dass das Mikroskop für die Fluoreszenzbildgebung richtig kalibriert ist, um genaue Ergebnisse zu erhalten.
VORSICHT: Minimieren Sie die Exposition der Zellen gegenüber intensivem Licht während der Bildgebung, um Phototoxizität zu vermeiden.
5. Hunger von HepG2-Zellen
HINWEIS: Nach Abschluss des Transfektionsschritts werden die Zellen vor der Insulinstimulation und der FRET-Bildgebung mit Serum ausgehungert. Dies minimiert die Aktivierung des Akt-Signalwegs aufgrund des im FBS vorhandenen Insulins und gewährleistet ein konsistentes Ausgangsniveau der Akt-Aktivität. Die Zusammensetzung des in diesem Versuch verwendeten Hungermediums ist in (Tabelle 3) beschrieben. BSA kommt in Pulverform. Um eine 0,1%ige (w/v) Lösung herzustellen, rekonstituieren Sie 0,1 g BSA in 3 ml DMEM und mischen Sie gründlich. Sterilisieren Sie die Lösung mit einem 0,45-μm-Filter und stellen Sie das Endvolumen durch Zugabe von DMEM auf 100 mL ein.
- Entfernen Sie das Kulturmedium und spülen Sie die Bildgebungsschalen zweimal für jeweils 5 min mit 1x PBS aus.
HINWEIS: Die beiden PBS-Waschungen helfen, Restserum und Insulin oder Wachstumsfaktoren, die das Experiment beeinträchtigen könnten, vollständig zu entfernen. - Geben Sie 2 ml Hungermedium in die Bildgebungsschalen (siehe Materialtabelle). Geben Sie das Medium vorsichtig um den Rand der Schale herum, um zu vermeiden, dass sich die Zellen vom Glasboden lösen. Inkubieren Sie bei 37 °C für 4 h.
HINWEIS: Die 4-stündige Inkubation ist optimal für die Synchronisierung des Zellstoffwechsels; Die Dauer kann jedoch je nach experimentellem Bedarf verlängert werden.
6. FRET-Lebendzell-Imaging für HepG2-Zellen
HINWEIS: Dieser Abschnitt enthält Anweisungen für die FRET-Lebendzellbildgebung zur Überwachung der räumlich-zeitlichen Dynamik der Akt-Phosphorylierung in einzelnen HepG2-Zellen. Es ist wichtig, den Mikroskopaufbau, die Handhabungsverfahren und die Bildgebungsbedingungen für lebende HepG2-Zellen zu optimieren, wie unten beschrieben. Der Mikroskopaufbau ist entscheidend für die Optimierung der Bildgebungsbedingungen für die FRET-Bildgebung. Befolgen Sie die Einrichtung der PC/konfokalen Laser-Scanning-Mikroskopie (CLSM) schrittweise gemäß den Anweisungen des Herstellers, um einen stabilen Betrieb zu gewährleisten. Die angepasste CLSM-Konfiguration für die FRET-Bildgebung ist in Abbildung 6 dargestellt.
- Schalten Sie den Fernschalter ein, um das Mikroskop, den Computer, den Scanner, den Laserstart, den Piezotisch und die Epifluoreszenz-LED-Lichtquelle mit Strom zu versorgen. Stellen Sie vor dem Einschalten sicher, dass alle Komponenten ordnungsgemäß angeschlossen sind, um mögliche Schäden zu vermeiden.
- Drehen Sie den Schlüssel beim Laserstart auf die ON-Position und drücken Sie die Tasten, um beide für FRET erforderlichen Laser (457 nm und 514 nm Laserlinien) zu aktivieren.
HINWEIS: Stellen Sie sicher, dass die richtigen Filter und Einstellungen für eine optimale FRET-Abbildung vorhanden sind. Die Auswahl der Laserlinie sollte auf den Anregungs- und Emissionsprofilen des Biosensors basieren. - Schalten Sie die Steckdosenleiste ein, um den Computer und den Monitor, die an das Mikroskop angeschlossen sind, mit Strom zu versorgen.
HINWEIS: Stellen Sie vor dem Einschalten sicher, dass alle Anschlüsse fest sind, um Schäden am Gerät zu vermeiden. - Melden Sie sich bei Windows an und starten Sie die Mikroskopie-Software.
- Klicken Sie auf A1 für Aufnahme, um die Bildgebungseinrichtung zu starten. Wählen Sie die geeigneten optischen Konfigurationen basierend auf den Anforderungen des FRET-Experiments.
- Passen Sie die Laserleistung und die Detektorempfindlichkeit nach Bedarf an, um optimale Bildgebungsbedingungen zu erzielen. Setzen Sie den Inkubator für den Mikroskoptisch vorsichtig ein und befestigen Sie ihn mit Schrauben.
HINWEIS: Ziehen Sie die Schrauben nicht zu fest an, um Schäden am Inkubator oder am Mikroskoptisch zu vermeiden. - Füllen Sie das innere Wasserbad mit sterilem doppelt destilliertem Wasser (ddH2O). Installieren Sie die obere Heizung sicher und schalten Sie die Stromversorgung für die Bühnenheizung, die Badheizung und die Linsenheizung ein (Abbildung 7A).
HINWEIS: Nicht überfüllen, um ein Verschütten in das System zu vermeiden. Stellen Sie sicher, dass alle Heizungen ordnungsgemäß funktionieren, um konstante Temperaturbedingungen für die Bildgebung von Lebendzellen aufrechtzuerhalten. - Verwenden Sie die 40-fach Öl-Immersionslinse für die Bildgebung (siehe Materialtabelle).
HINWEIS: Die technischen Spezifikationen finden Sie auf der Website des Herstellers. - Wischen Sie die Objektivlinse mit Linsenpapier ab, das mit 95 % Ethanol angefeuchtet ist. Geben Sie ein kleines Tröpfchen Immersionsöl auf die Objektivlinse (Abbildung 7B). Stellen Sie die Bildgebungsschale mit den HepG2-Zellen auf den Mikroskoptisch und befestigen Sie sie mit der Halterung (Abbildung 7C).
HINWEIS: Stellen Sie sicher, dass die Bildgebungsschüssel richtig ausgerichtet und gesichert ist, um Bewegungen während der Bildgebung zu vermeiden. Vermeiden Sie Druck auf die Linse, um Schäden sowohl an der Linse als auch am Glasboden der Schale zu vermeiden. - Schließen Sie die Kammer und inkubieren Sie die Zellen in der Lebendzellkammer für 1-2 Stunden, damit sie sich äquilibrieren können (Abbildung 7D).
- Halten Sie während der Zeitrafferbildgebung in bestimmten Intervallen eine Pause ein, entfernen Sie das Medium vorsichtig (Abbildung 7E) und fügen Sie dann 1 ml frisch zubereitetes Medium mit der angegebenen Insulinkonzentration hinzu (Abbildung 7F, Materialtabelle).
HINWEIS: Bereiten Sie eine 1 mg/ml-Insulinstammlösung vor, indem Sie 1 mg Insulin in 1 ml 10 mM Essigsäure auflösen. Die Lösung wird durch einen sterilen 0,2-μm-Spritzenvorsatzfilter filtriert und die Aliquote bei -20 °C gelagert. - Starten Sie das ND-Erfassungsfenster über das Menü Datei der Mikroskopiesoftware.
HINWEIS: Die Einrichtung der ND-Erfassung ist in (Abbildung 8) dargestellt. - Wählen Sie die Registerkarte Zeit , um das Intervall, die Dauer und die gewünschten Schleifen für das Zeitraffer-Imaging festzulegen. Klicken Sie anschließend auf die Registerkarte XY und drücken Sie die Schaltfläche + Hinzufügen , um einzelne HepG2-Zellen für die Bildgebung einzubeziehen.
- Wenn Sie mehrere Zellen hinzufügen möchten, suchen Sie geeignete Zellen, scannen Sie, optimieren Sie den Fokus und sperren Sie die Point Spread Function (PSF). Wiederholen Sie diesen Schritt für jede neue Zelle, die für die Bildgebung hinzugefügt wird. Scannen Sie mehrere Zellen an verschiedenen Stellen innerhalb der Bildgebungsschalen, um verschiedene Zielzellen für den Einschluss zu identifizieren.
- Klicken Sie auf das rote X-Symbol , um die Auswahl von Zellen aufzuheben. Aktivieren Sie das Kontrollkästchen "Z", um die Z-Position einzustellen. Nachdem Sie alle Parameter ausgewählt haben, geben Sie den Namen des Experiments in das Feld "Dateiname" ein.
- Klicken Sie auf Durchsuchen , um den Zielordner auszuwählen, und aktivieren Sie dann das Kontrollkästchen In Datei speichern. Klicken Sie auf die Registerkarte Jetzt ausführen , um die ND-Erfassung zu starten. Das ND-Aufnahmefenster zeigt den Fortschritt der Zeitraffer-Bildgebung in Echtzeit an, einschließlich der verstrichenen und verbleibenden Zeit.
- Zeichnen Sie Ausgangswerte bis zu 30 Minuten lang auf, ohne die Zellen mit Insulin zu stimulieren.
- Lokalisieren Sie geeignete Zellen, zoomen Sie hinein/heraus, um eine einzelne Zelle in den Fokus zu bringen, und wählen Sie Zellen mit hoher Fluoreszenzintensität aus. Fügen Sie die Zellen nacheinander hinzu, bis zu einem Maximum von 6 Zellen.
HINWEIS: Begrenzen Sie die Anzahl der Zellen, um Verzögerungen bei der Bildaufnahme und das Einfrieren des Fensters während der Bildgebung zu vermeiden. - Legen Sie die Zeit und die Bildfrequenz für die Zeitrafferaufnahme fest. Starten Sie das Zeitraffer-Imaging.
HINWEIS: Stellen Sie sicher, dass alle Parameter, einschließlich Belichtungszeit und Laserleistung, optimiert sind, bevor Sie mit der Bildgebung beginnen. - Pausieren Sie die Bildgebung in regelmäßigen Abständen, nehmen Sie das Medium vorsichtig aus der Glasbodenschale und fügen Sie 1 ml Medium hinzu, das mit der entsprechenden Insulinkonzentration versetzt ist.
- Setzen Sie die Bildaufnahme fort. Wiederholen Sie Schritt 6.20 bei Bedarf.
- Klicken Sie am Ende des Experiments auf die Registerkarte Fertig stellen, um es zu schließen. Sichern und speichern Sie aufgenommene Bilder sicher für die Analyse.
7. Datenanalyse
- Korrigieren Sie die vorverarbeiteten Zeitraffer-FRET-Bildgebungsdaten von Kontrollproben auf spektrales Übersprechen und CFP-Autofluoreszenz, was zu korrigierten FRET-Werten und präziser FRET-Effizienz führt. Verwenden Sie Bildgebungssoftware für die Erfassung, Verarbeitung und Analyse von FRET-Bildern gemäß den Protokollen, die in früheren Studien beschrieben wurden 27,28,29,30 (Abbildung 9).
8. Berechnungen des FRET-Wirkungsgrads
- Um die FRET-Effizienz zu bestimmen, nehmen Sie sieben Bilder auf: Zelle (IDA(D), IDD(D), IDA(A), IAA(A), IDD, IAA und IDA). Berechnen Sie den FRET-Wirkungsgrad mit der folgenden Formel:
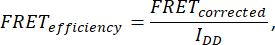
wobeiFRET Corrected aus der Gleichung erhalten wird:
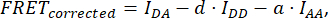
wobei d und a definiert sind als:
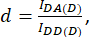
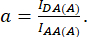
Hier stellen IDA, IDD und IAA Bilder von Zellen dar, die mit dem Biosensor transfiziert wurden. IDA(D) und IDD(D)) SIND REINE SPENDER-KONTROLLBILDER, UND IDA(A) und IAA(A) sind reine Akzeptor-Kontrollbilder.
9. Bilderfassung
- IDA : Das Bild wird durch Donoranregung (434 nm) mit Akzeptoremission (530 nm) erhalten.
- IDD : Das Bild wird durch Donoranregung (434 nm) mit Donoremission (477 nm) erhalten.
- IAA : Das Bild wird durch Akzeptoranregung (517 nm) mit Akzeptoremission (530 nm) erhalten.
- IDA(D) und IDA(A) : Erfassen Sie das Bild mit der gleichen Anregung und Emission wie IDA, jedoch von Zellen, die nur den Donor bzw. den Akzeptor exprimieren.
- IDD(D) : Erfassen Sie das Bild unter den gleichen Bedingungen wie IDD für die Nur-Spender-Kontrolle.
- IAA(A) : Ruft das Bild unter den gleichen Bedingungen wie IAA ab, ist jedoch spezifisch für das Nur-Akzeptor-Steuerelement.
10. Korrektur des Hintergrunds
- Öffnen Sie die Bildanalysesoftware auf dem Computer. Rufen Sie in der oberen Menüleiste das Menü Datei auf. Wählen Sie Öffnen oder Datei öffnen aus den Dropdown-Optionen aus.
- Navigieren Sie zu dem Verzeichnis, das die Zeitraffer-Bilddaten enthält. Suchen Sie das Zeitrafferbild, das analysiert werden muss (z. B. im nd2-Format).
- Wählen Sie die Datei aus und klicken Sie auf Öffnen oder OK , um sie zur Anzeige und Analyse in die Software zu laden. Definieren Sie einen Interessenbereich (ROI) innerhalb der Zelle (oder verwenden Sie die gesamte Zelle als ROI) und notieren Sie den Grauwert jedes Pixels innerhalb dieses Bereichs.
- Wählen Sie einen zellenfreien Bereich als Hintergrund und berechnen Sie dessen durchschnittlichen Grauwert. Erstellen Sie das korrigierte Bild, indem Sie diesen Hintergrunddurchschnitt vom Grauwert jedes Pixels innerhalb der Zellen-ROI subtrahieren.
11. Eliminierung des Durchblutens (Übersprechen) des Fret-Bleed-Throughs (Übersprechen)
HINWEIS: Die spektrale Überlappung zwischen der Donoremission und der Akzeptoranregung ist in Abbildung 3B dargestellt, die für den FRET-Wirkungsgrad und den Energieübertragungsprozess entscheidend ist. Das Durchscheinen in der FRET-Bildgebung im Zeitraffer ist eine große Herausforderung, die sich aus der spektralen Überlappung von Donor- und Akzeptorfluorophoren ergibt, was zu ungenauen Messungen führt. Das Übersprechen ist inhärent, da sich die Spektren sowohl der Donor- als auch der Akzeptorfluorophore bis zu einem gewissen Grad überlappen (Abbildung 3C, D). Dieses Problem wird durch Faktoren wie hohe Fluorophorkonzentrationen und unsachgemäße Filterkonfigurationen noch verschärft. Die Berücksichtigung des Durchblutens ist entscheidend für die Gewährleistung der Zuverlässigkeit von FRET-Messungen.
- In einer früheren Studie finden Sie eine Methode zur Abschwächung von Durchblutungseffekten9.
12. Quantifizierung und statistische Analyse
- Führen Sie statistische Analysen mit statistischer Analysesoftware durch.
Ergebnisse
Um die Akt-Aktivierung in HepG2-Zellen zu untersuchen, wurden die Zellen auf vorbeschichtete Bildgebungsschalen ausgesät und mit dem FRET-basierten Biosensor pEevee-iAkt-NES transfiziert (Abbildung 2A), der eine Echtzeitüberwachung der Akt-Phosphorylierung ermöglicht. Nach der Transfektion wurden die Zellen 4 Stunden lang in einem serumfreien Medium gehungert, um ihren Stoffwechselzustand zu synchronisieren und die Basalinsulinsignalisierung zu minimieren...
Diskussion
Das Protokoll für die FRET-Bildgebung in lebenden Zellen zur Überwachung der Akt-Phosphorylierung in HepG2-Zellen umfasst mehrere wichtige Schritte, um zuverlässige und reproduzierbare Ergebnisse zu gewährleisten. Der erste kritische Schritt ist die Zellkultur, die die routinemäßige Zellpflege, die Beschichtung von Bildgebungsschalen und die Zellaussaat umfasst. Die richtige Beschichtung ist für die Zelladhäsion bei Zeitraffer-Bildgebungsexperimenten unerlässlich, da sie eine st...
Offenlegungen
Die Autoren erklären, dass keine konkurrierenden Interessen bestehen.
Danksagungen
Diese Arbeit wurde teilweise von der Natural Science Foundation of Shenzhen (JCYJ20240813113606009), der Shenzhen-Hong Kong Cooperation Zone for Technology and Innovation (HZQB-KCZYB-2020056), der National Natural Science Foundation of China (32070681), dem National Key R&D Program of China (2019YFA0906002) und dem Shenzhen Peacock Plan (KQTD2016053117035204) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.25% trypsin-EDTA | Gibco | Cat#25200-056 | Use ice-cold PBS for cell wash |
| 15 mm glass bottom cell culture dish | NEST | Cat#801001 | |
| 2 mL Nalgene cryogenic vials | Thermo Scientific | Cat#5012-0020 | |
| 5 mL Stripette Serological Pipets | Corning | Cat#4487 | |
| 95% Ethanol | Kermel | Cat#C028005 | |
| A1 HD25/A1R HD25 confocal microscope | Nikon | https://www.nikon.com/ | Magnification: 40×, Numerical Aperture (NA): 1.30, Pixel Dwell Time: 2.4 ms, Pixel Size: 1024 |
| Ampicillin | Sigma-Aldrich | Cat#A9393 | |
| Bovine serum albumin (BSA) | VWR Life Science | Cat#N208-10g | |
| Corning 25 cm2 rectangular culture flasks | Corning | Cat#430639 | |
| Countess 3 automated cell counter | Thermo Scientific | http://www.thermofisher.com/#AMQAX2000 | |
| Countess cell counting chamber slides | Thermo Scientific | http://www.thermofisher.com/#C10228 | |
| Digital vortex mixers | Thermo Scientific | https://www.thermofisher.com/ | |
| Dimethyl sulfoxide | Sigma-Aldrich | Cat#D2650 | |
| Eppendorf Safe-Lock Tubes 1.5 mL | Eppendorf | Cat#022363204 | |
| EZ-PCR mycoplasma detection kit | Biological Industries | Cat# 20-700-20 | |
| Fetal Bovine Serum, qualified, Australia | Gibco | Cat#10099141 | |
| GlutaMAX Supplement | Gibco | Cat#35050061 | |
| GraphPad Prism 9 | GraphPad Software | https://www.graphpad.com/ | |
| HepG2 | National Collection of Authenticated Cell Cultures | #CSTR:19375.09.3101HUMSCSP510 http://www.cellbank.org.cn/ | |
| Immersion Oil Type 37 | Cargille Laboratories | Cat #16237 | |
| Insulin | Sigma-Aldrich | Cat#I5500-50MG | Warm to 37 °C before use |
| LB Broth (1x) | Invitrogen | Cat#10855001 | |
| Minimum Essential Medium (MEM) | Gibco | Cat#11095080 | Warm to 37 °C before use |
| mySPIN 12 mini centrifuge | Thermo Scientific | https://www.thermofisher.com/ | |
| NanoDrop One | Thermo Scientific | https://www.thermofisher.com | |
| Nikon Plan Fluor 40×/1.30 Oil Lens | Nikon | https://www.nikon.com/ | |
| NIS-Elements-AR | Nikon | https://www.nikon.com/ | |
| Non-Essential Amino Acids (NEAA) (100x) | Gibco | Cat#11140050 | |
| One Shot LB Agar Plates | Invitrogen | Cat#A55802 | |
| One Shot Stbl3 chemically competent E. coli | Invitrogen | Cat#C737303 | |
| Parafilm | PARAFILM | Cat#B8R05606 | |
| PBS (phosphate buffered saline) | Gibco | Cat#10010023 | |
| pEevee-iAkt-NES (7,033 bp) | Miura et al31 | https://benchling.com/s/seq-q46zFYCfl0swLAun0t28/edit | |
| Penicillin-streptomycin | Gibco | Cat#15070063 | |
| Plasmocin prophylactic | InvivoGen | Cat#ant-mpp | |
| Poly-L-Lysine Hydrobromide | Sigma-Aldrich | Cat#P4832 | |
| Precision general purpose baths | Thermo Scientific | https://www.thermofisher.com/ | |
| QIAprep spin miniprep kit | QIAGEN | Cat#27106 | |
| SnapGene | SnapGene by Dotmatics | https://www.snapgene.com | |
| Sodium Pyruvate (100 mM) | Gibco | Cat#11360070 | |
| Syringe filter unit, 0.22 μm | Millipore | Cat#SLGP033RS | |
| Tokai Hit stage top incubator | TOKAI HIT | https://www.tokaihit-livecell.com/stagetopincubator | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | Cat#10977015 | |
| Xfect Transfection Reagent | Takara Bio | Cat#631317 |
Referenzen
- GBD 2021 Diabetes Collaborators. Global, regional, and national burden of diabetes from 1990 to 2021, with projections of prevalence to 2050: a systematic analysis for the Global Burden of Disease Study 2021. Lancet. 402 (10397), 203-234 (2023).
- Li, M., et al. Trends in insulin resistance: insights into mechanisms and therapeutic strategy. Signal Transduct Target Ther. 7 (1), 216 (2022).
- Poloz, Y., Stambolic, V. Obesity and cancer, a case for insulin signaling. Cell Death Dis. 6 (12), e2037 (2015).
- Arcidiacono, B., et al. Insulin resistance and cancer risk: an overview of the pathogenetic mechanisms. Exp Diabetes Res. 2012, 789174 (2012).
- Godsland, I. F. Insulin resistance and hyperinsulinaemia in the development and progression of cancer. Clin Sci (Lond). 118 (5), 315-332 (2009).
- Tsugane, S., Inoue, M. Insulin resistance and cancer: epidemiological evidence. Cancer Sci. 101 (5), 1073-1079 (2010).
- Yudhani, R. D., et al. In vitro insulin resistance model: A recent update. J Obes. 2023, 1964732 (2023).
- Akhtar, J., et al. Bistable insulin response: The win-win solution for glycemic control. iScience. 25 (12), 105561 (2022).
- Akhtar, J., Imran, M., Wang, G. Protocol for live-cell Forster resonance energy transfer imaging to reveal the bistable insulin response of single C2C12-derived myotubes. STAR Protoc. 5 (2), 103109 (2024).
- Kamino, K., et al. Optimal inference of molecular interaction dynamics in FRET microscopy. Proc Natl Acad Sci U S A. 120 (15), e2211807120 (2023).
- Kraft, A. E., Nikolaev, V. O. FRET microscopy for real-time visualization of second messengers in living cells. Methods Mol Biol. 1563, 85-90 (2017).
- Veeriah, S., et al. High-throughput time-resolved FRET reveals Akt/PKB activation as a poor prognostic marker in breast cancer. Cancer Res. 74 (18), 4983-4995 (2014).
- Conway, J. R. W., et al. Monitoring AKT activity and targeting in live tissue and disease contexts using a real-time Akt-FRET biosensor mouse. Sci Adv. 9 (17), eadf9063 (2023).
- Chandris, P., Giannouli, C. C., Panayotou, G. Imaging approaches for the study of metabolism in real time using genetically encoded reporters. Front Cell Dev Biol. 9, 725114 (2021).
- Yang, J., et al. Longitudinal FRET imaging of glucose and lactate dynamics and response to therapy in breast cancer cells. Mol Imaging Biol. 24 (1), 144-155 (2022).
- Mandrou, E., et al. A reliable system for quantitative G-protein activation imaging in cancer cells. Cells. 13 (13), 1114 (2024).
- Fang, C., Huang, Y., Zhao, Y. Review of FRET biosensing and its application in biomolecular detection. Am J Transl Res. 15 (2), 694-709 (2023).
- Miller, J. N. Fluorescence energy transfer methods in bioanalysis. Analyst. 130 (3), 265-270 (2005).
- Verveer, P. J., Rocks, O., Harpur, A. G., Bastiaens, P. I. Imaging protein interactions by FRET microscopy: FRET measurements by acceptor photobleaching. CSH Protoc. 2006 (6), (2006).
- Vu, C. Q., Arai, S. Quantitative imaging of genetically encoded fluorescence lifetime biosensors. Biosensors (Basel). 13 (10), 939 (2023).
- Wang, G. A more holistic view of the logarithmic dose-response curve offers greater insights into insulin response. J Biol Chem. 301 (1), 108037 (2025).
- Wang, G. Body mass dynamics is determined by the metabolic Ohm's law and adipocyte-autonomous fat mass homeostasis. iScience. 23 (6), 101176 (2020).
- Wang, G. Raison d'être of insulin resistance: the adjustable threshold hypothesis. J R Soc Interface. 11 (101), 20140892 (2014).
- Wang, G. Optimal homeostasis necessitates bistable control. J R Soc Interface. 9, 2723 (2012).
- Heim, R., Tsien, R. Y. Engineering green fluorescent protein for improved brightness, longer wavelengths and fluorescence resonance energy transfer. Curr Biol. 6 (2), 178-182 (1996).
- Nguyen, A. W., Daugherty, P. S. Evolutionary optimization of fluorescent proteins for intracellular FRET. Nat Biotechnol. 23 (3), 355-360 (2005).
- Broussard, J. A., Rappaz, B., Webb, D. J., Brown, C. M. Fluorescence resonance energy transfer microscopy as demonstrated by measuring the activation of the serine/threonine kinase Akt. Nat Protoc. 8 (2), 265-281 (2013).
- Gordon, G. W., Berry, G., Liang, X. H., Levine, B., Herman, B. Quantitative fluorescence resonance energy transfer measurements using fluorescence microscopy. Biophys J. 74 (5), 2702-2713 (1998).
- Tron, L., et al. Flow cytometric measurement of fluorescence resonance energy transfer on cell surfaces. Quantitative evaluation of the transfer efficiency on a cell-by-cell basis. Biophys J. 45 (5), 939-946 (1984).
- Youvan, D. C., et al. Calibration of fluorescence resonance energy transfer in microscopy. , (2002).
- Miura, H., Matsuda, M., Aoki, K. Development of a FRET biosensor with high specificity for Akt. Cell Struct Funct. 39 (1), 9-20 (2014).
- Liberio, M. S., Sadowski, M. C., Soekmadji, C., Davis, R. A., Nelson, C. C. Differential effects of tissue culture coating substrates on prostate cancer cell adherence, morphology and behavior. PLoS One. 9 (11), e112122 (2014).
- Rani, K., Sengupta, S. Multi-stimuli programmable FRET based RGB absorbing antennae towards ratiometric temperature, pH and multiple metal ion sensing. Chem Sci. 12 (47), 15533-15542 (2021).
- Betolngar, D. B., et al. pH sensitivity of FRET reporters based on cyan and yellow fluorescent proteins. Anal Bioanal Chem. 407 (14), 4183-4193 (2015).
- Salonikidis, P. S., et al. An ion-insensitive cAMP biosensor for long term quantitative ratiometric fluorescence resonance energy transfer (FRET) measurements under variable physiological conditions. J Biol Chem. 286 (26), 23419-23431 (2011).
- Youvan, D. C., et al. Calibration of fluorescence resonance energy transfer in microscopy using genetically engineered GFP derivatives on nickel chelating beads. Biotechnol Alia. 3, 1-18 (1997).
- Menaesse, A., et al. Simplified instrument calibration for wide-field fluorescence resonance energy transfer (FRET) measured by the sensitized emission method. Cytometry A. 99 (4), 407-416 (2021).
- Batta, A., Hajdu, T., Nagy, P. Improved estimation of the ratio of detection efficiencies of excited acceptors and donors for FRET measurements. Cytometry A. 103 (7), 563-574 (2023).
- Coullomb, A., et al. QuanTI-FRET: a framework for quantitative FRET measurements in living cells. Sci Rep. 10 (1), 6504 (2020).
- Hoppe, A., Christensen, K., Swanson, J. A. Fluorescence resonance energy transfer-based stoichiometry in living cells. Biophys J. 83 (6), 3652-3664 (2002).
- Zal, T., Gascoigne, N. R. Photobleaching-corrected FRET efficiency imaging of live cells. Biophys J. 86 (6), 3923-3939 (2004).
- Hochreiter, B., Kunze, M., Moser, B., Schmid, J. A. Advanced FRET normalization allows quantitative analysis of protein interactions including stoichiometries and relative affinities in living cells. Sci Rep. 9 (1), 8233 (2019).
- Liu, H., Zhang, F., Mishra, S. K., Zhou, S., Zheng, J. Knowledge-guided fuzzy logic modeling to infer cellular signaling networks from proteomic data. Sci Rep. 6, 35652 (2016).
- Garrido-Rodriguez, M., Zirngibl, K., Ivanova, O., Lobentanzer, S., Saez-Rodriguez, J. Integrating knowledge and omics to decipher mechanisms via large-scale models of signaling networks. Mol Syst Biol. 18 (7), e11036 (2022).
- Greenwald, E. C., Polanowska-Grabowska, R. K., Saucerman, J. J. Integrating fluorescent biosensor data using computational models. Methods Mol Biol. 1071, 227-248 (2014).
- Zhao, Z., Xia, J. Computational Approaches for Modeling Signal Transduction Networks. Encyclopedia of Bioinformatics and Computational Biology. , (2019).
- Wang, G., Krueger, G. R. Computational analysis of mTOR signaling pathway: bifurcation, carcinogenesis, and drug discovery. Anticancer Res. 30 (7), 2683-2688 (2010).
- Wang, G. Singularity analysis of the AKT signaling pathway reveals connections between cancer and metabolic diseases. Phys Biol. 7 (4), 046015 (2010).
- Chedere, A., Hari, K., Kumar, S., Rangarajan, A., Jolly, M. K. Multi-stability and consequent phenotypic plasticity in AMPK-Akt double negative feedback loop in cancer cells. J Clin Med. 10 (3), 472 (2021).
- Mosca, E., et al. Computational modeling of the metabolic States regulated by the kinase akt. Front Physiol. 3, 418 (2012).
- Liao, J., Madahar, V., Dang, R., Jiang, L. Quantitative FRET (qFRET) technology for the determination of protein-protein interaction affinity in solution. Molecules. 26 (21), 6339 (2021).
- Verma, A. K., Noumani, A., Yadav, A. K., Solanki, P. R. FRET based biosensor: Principle applications recent advances and challenges. Diagnostics (Basel). 13 (8), 1375 (2023).
- Mattheisen, J. M., et al. Application of bioluminescence resonance energy transfer to quantitate cell-surface expression of membrane proteins. Anal Biochem. 684, 115361 (2024).
- Lionetti, M. C., La Porta, C. A. M. FLIM-FRET investigation of heterogeneous huntingtin aggregation in HeLa cells. Methods Mol Biol. 2551, 595-604 (2023).
- Petutschnig, E. K., Pierdzig, L., Mittendorf, J., Niebisch, J. M., Lipka, V. A novel fluorescent protein pair facilitates FLIM-FRET analysis of plant immune receptor interaction under native conditions. J Exp Bot. 75 (3), 746-759 (2024).
- Sprenger, J. U., Perera, R. K., Gotz, K. R., Nikolaev, V. O. FRET microscopy for real-time monitoring of signaling events in live cells using unimolecular biosensors. J Vis Exp. 66, e4081 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten