É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Imagem de transferência de energia de ressonância de Förster de células vivas da dinâmica de ativação de Akt regulada metabolicamente em células HepG2
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para quantificar a dinâmica espaço-temporal da ativação e fosforilação de Akt em células HepG2 vivas. A imagem de transferência de energia por ressonância de Förster (FRET) é uma ferramenta poderosa que fornece informações valiosas sobre as vias de sinalização da insulina e a regulação metabólica em células cancerígenas.
Resumo
A ativação da Akt regulada metabolicamente é um nó crítico na cascata de sinalização da insulina e fornece informações valiosas sobre a relação entre diabetes e câncer. Para quantificar com precisão a atividade de Akt em células HepG2, desenvolvemos um protocolo robusto e reprodutível utilizando Förster Resonance Energy Transfer (FRET) com biossensores específicos de Akt geneticamente codificados. Este protocolo descreve etapas detalhadas para cultura de células, preparação de placas de imagem e transfecção de células HepG2 para expressar biossensores baseados em FRET, juntamente com diretrizes específicas para configuração de hardware e software de microscópio confocal de varredura a laser. Os resultados demonstraram padrões únicos de sinalização de insulina em células HepG2, que exibem uma mudança irreversível caracterizada pela ativação constitutiva de Akt com um limiar de ativação definido, mas sem limiar de desligamento. Em contraste, os miotubos exibem um interruptor reversível. A ativação persistente de Akt em células HepG2 sugere mecanismos subjacentes à resistência à insulina e desregulação metabólica em células hepáticas, com implicações mais amplas para a compreensão da progressão de distúrbios metabólicos e câncer. Este protocolo oferece uma estrutura valiosa para explorar vias de sinalização relacionadas à Akt e comportamentos celulares em vários contextos de doenças.
Introdução
O diabetes mellitus representa um grande desafio de saúde global, caracterizado por resistência à insulina e comprometimento da homeostase da glicose1. Uma compreensão abrangente das vias de sinalização da insulina é crucial para elucidar a fisiopatologia dessa doença, pois a insulina desempenha um papel fundamental no metabolismo da glicose, no crescimento celular e na sobrevida2. Numerosos estudos demonstraram que a sinalização da insulina afeta significativamente vários tipos de câncer, ligando a resistência à insulina à progressão do tumor e aos maus resultados dos pacientes 3,4,5,6. As células HepG2, uma linhagem celular de carcinoma hepatocelular comumente usada, servem como um modelo valioso para estudar a resistência à insulina e a interação entre a desregulação metabólica e o desenvolvimento do câncer7. Tradicionalmente, os pesquisadores veem as respostas à insulina como graduadas; no entanto, estudos recentes revelaram que células individuais podem exibir respostas biestáveis, exibindo transições salientes entre falta de resposta e resposta completa ocorrendo em limiares específicos de concentração de insulina 8,9.
A imagem de transferência de energia por ressonância de Förster (FRET) é uma ferramenta poderosa para estudar a distribuição espaço-temporal de biomoléculas em células vivas10. Ao extrair informações da dinâmica molecular, o FRET fornece informações sobre processos como a ativação de Akt em tempo real, tornando-se uma técnica inestimável para estudar células vivas11,12. Este método de imagem provou ser essencial no estudo da dinâmica celular, particularmente em doenças metabólicas e câncer, onde interações moleculares precisas são cruciais13. O FRET também permite o monitoramento em tempo real das interações moleculares, lançando luz sobre mecanismos como resistência à insulina e progressão tumoral14,15. Os biossensores FRET são cruciais na pesquisa do câncer para estudar microambientes tumorais, resistência a drogas e distúrbios metabólicos16. Os métodos de detecção de FRET, como emissão sensibilizada (SE), branqueamento aceptor (AB), microscopia de imagem de fluorescência (FLIM) e espectroscopia, oferecem vantagens distintas para quantificar as interações moleculares17. O SE mede a transferência de energia entre os fluoróforos doadores e aceptores, resultando em uma mudança mensurável nos espectros de emissão que se correlaciona com a proximidade das biomoléculas em interação18. O AB usa fotobranqueamento seletivo do fluoróforo aceitador e rastreia as mudanças na fluorescência do doador, o que permite aos pesquisadores avaliar a cinética e as distânciasde interação 19. O FLIM avalia as taxas de decaimento da fluorescência do fluoróforo doador, diretamente influenciado pela eficiência do FRET, para fornecer medições precisas em nanoescala das interações moleculares20.
Usando técnicas de FRET, demonstramos recentemente respostas de insulina biestáveis em miotubos derivados de C2C12 8,9,21,22,23,24. Os distintos limiares de ativação e desativação para ativação de Akt, como descobrimos, sugerem que a dose-resposta gradual de insulina de corpo inteiro desmente a complexidade da cascata de sinalização subcelular a partir do estímulo de insulina, que culmina em uma resposta de tudo ou nada no nível de célula única21 , 22 , 23 , 24. Para testar a presença de biestabilidade em outros tipos de células, estimulamos células HepG2 com insulina e registramos sua resposta usando imagens FRET de célula única. Estimulamos células HepG2 com concentrações variadas de insulina e monitoramos a atividade de Akt no nível de célula única usando um biossensor de Akt. O biossensor Akt compreende a proteína fluorescente ciano aprimorada (ECFP)25 como o fluoróforo doador e a variante mais brilhante da proteína fluorescente amarela (YPet)26 como o fluoróforo aceptor, ligado por um ligante Eevee contendo a sequência peptídica SGRPRTTTFADSCKP. Este peptídeo atua como substrato para Akt fosforilada (pAkt), otimizada a partir da glicogênio sintase quinase humana 3β (GSK3β). Em seu estado não fosforilado, a separação espacial entre os fluoróforos doadores e aceptores excede o raio de Förster, o que inibe a transferência de energia. Após a estimulação da insulina, ocorre a fosforilação de Akt e leva à fosforilação de SGRPRTTTFADSCKP. Este processo induz uma mudança conformacional que traz o doador e o aceitador dentro do raio de Förster, habilitando o FRET27. Como resultado, a intensidade do sinal FRET se correlaciona com a quantidade de moléculas de Akt fosforiladas e permite a quantificação em tempo real das respostas celulares mediadas pela insulina.
Este protocolo, inicialmente desenvolvido para estudar a sinalização de insulina em miotubos derivados de C2C12, foi aplicado com sucesso a células HepG2 e utilizado em diferentes plataformas de hardware e software, demonstrando assim sua aplicabilidade, adaptabilidade e versatilidade. As células HepG2 exibem atividade Akt constitutiva, o que as torna um modelo in vitro ideal para estudar a sinalização de insulina específica do fígado e os processos metabólicos. Os principais recursos do protocolo são descritos passo a passo na seção de protocolo.
Protocolo
Uma visão geral das etapas experimentais envolvidas na imagem de células vivas FRET para monitorar a fosforilação de Akt em células HepG2 únicas é mostrada na Figura 1.
1. Aquisição, propagação e purificação de plasmídeo
NOTA: Esta seção descreve as etapas essenciais para adquirir, amplificar e purificar o plasmídeo necessário para a análise de FRET de célula única.
- Utilize o plasmídeo pEevee-iAkt-NES-YPet (Figura 2A).
NOTA: O plasmídeo foi generosamente fornecido pelo Prof. Kazuhiro Aoki no Instituto Nacional de Biologia Básica (NIBB), Japão.Os mapas de plasmídeo para os biossensores FRET usados para monitoramento de Akt e seus respectivos controles são mostrados na Figura 2. O pEevee-iAkt-NES-ECFP (doador; Figura 2B) e pEevee-iAkt-NES-Ypet (aceitador; Figura 2C) os plasmídeos são usados como controles de calibração durante os experimentos FRET9.
NOTA: A Figura 3 mostra a composição e o mecanismo do biossensor intramolecular FRET. - Para propagar o plasmídeo, execute a transformação bacteriana usando células de E. coli quimicamente competentes (ver Tabela de Materiais). Colocar as células transformadas em ágar Luria-Bertani (LB) contendo 100 μg/ml de ampicilina e incubar durante a noite a 37 °C. No dia seguinte, selecione uma colônia resistente à ampicilina e cultive-a em caldo LB suplementado com 100 μg/mL de ampicilina para amplificação do plasmídeo.
- Para purificar o DNA do plasmídeo, use um kit de purificação de DNA de plasmídeo disponível comercialmente (consulte a Tabela de Materiais) para obter alta pureza e rendimento. Avalie a qualidade do DNA medindo as taxas de absorbância (A260 / A280 e A260 / A230) usando um espectrofotômetro (consulte a Tabela de Materiais). Proporções entre 1,8-2,0 e 2,0-2,2 são consideradas ótimas. Verifique a integridade do plasmídeo por meio de eletroforese em gel de agarose para garantir que seja adequado para transfecção.
2. Procedimento de cultura celular
NOTA: Execute todos os procedimentos de cultura de células dentro de uma capela de fluxo laminar para manter um ambiente estéril e evitar contaminação. O fluxo de trabalho da cultura de células HepG2 é mostrado na Figura 4. O meio completo para células HepG2 consiste em meio essencial mínimo (MEM), 10% de soro bovino fetal (FBS), 1% de aminoácidos não essenciais (NEAA), 1 mM de piruvato de sódio, 2 mM de suplemento de L-glutamina, 100 U / mL de penicilina-estreptomicina e 2,5 μg / mL de solução antibiótica-antimicótica (ver Tabela de Materiais, Tabela 1).
- Descongele rapidamente as células HepG2 congeladas colocando o frasco para injetáveis num termomisturador a 37 °C ou banho-maria até que estejam completamente descongelados.
- Transfira as células descongeladas para um tubo cônico de 15 mL contendo 10 mL de meio de crescimento completo (consulte a Tabela de Materiais).
NOTA: Pré-aqueça o meio de crescimento completo a 37 °C antes de usar para minimizar o choque térmico nas células. - Centrifugue o tubo a 200 x g por 5 min para peletar as células.
- Aspire cuidadosamente o sobrenadante usando uma ponta de pipeta de diâmetro largo para evitar perturbar o pellet celular.
- Ressuspenda o pellet em 10 ml de meio de crescimento completo fresco.
- Transferir a suspensão celular para um balão de cultura de tecidos de 75 cm².
- Incubar o balão a 37 °C numa atmosfera humidificada com 5% de CO2
NOTA: Observe as células nas próximas 24-48 h para confirmar a fixação e avaliar a recuperação. Não perturbe as células por pelo menos 4 h para garantir a fixação adequada antes de trocar o meio. Evite abrir a incubadora com frequência durante as primeiras 4 h, pois isso pode interromper a fixação das células. Siga os protocolos institucionais de biossegurança e use equipamentos de proteção individual (EPI) apropriados para manter um ambiente de trabalho seguro durante todos os procedimentos de cultura de células. Subcultive células HepG2 quando atingirem 70%-80% de confluência para manter as condições ideais de crescimento e prevenir a superlotação, o que pode afetar a viabilidade celular e o potencial de crescimento. - Para subcultura, aspire o meio e enxágue as células uma vez com 5 mL de solução salina tamponada com fosfato (PBS).
- Adicione 1 mL de tripsina a 0,25% para cobrir a monocamada celular (consulte a Tabela de Materiais).
NOTA: Certifique-se de que a tripsina esteja pré-aquecida a 37 ° C para uma atividade ideal. Não tripsinize demais as células, pois isso pode reduzir a viabilidade. Monitore as células ao microscópio para confirmar o descolamento. - Incubar a 37 °C durante cerca de 5 min.
- Quando as células se desprenderem, adicione 2 mL de meio completo para neutralizar a tripsina e coletar as células por pipetagem.
- Pipete suavemente a suspensão celular para quebrar os aglomerados e obter uma suspensão unicelular.
- Adicionar 3 ml de meio completo a cada novo balão e, em seguida, transferir as células com uma proporção dividida de 1:2 para cada balão.
NOTA: Para passagens iniciais, divida as células em uma diluição de 1:2. Após as passagens 4-5, diluições de 1:4 ou 1:5 podem ser realizadas conforme apropriado. - Incubar as células a 37 °C em atmosfera humidificada com 5% de CO2.
NOTA: Verifique as células após 24 h para confirmar a fixação e avaliar a recuperação. - Para cultura de rotina, substitua o meio de cultura a cada 2-3 dias ou antes se o indicador de pH mudar de rosa para amarelo, indicando acidificação.
NOTA: Não deixe o meio ficar muito ácido, pois isso pode prejudicar as células. - Verifique a morfologia celular regularmente sob um microscópio para garantir a saúde celular.
NOTA: Para armazenamento de curto prazo, congele as células HepG2 a -80 °C; Para armazenamento de longo prazo, armazene em nitrogênio líquido. A composição do meio de congelamento usado neste experimento é descrita na (Tabela 2).
3. Revestindo placas de imagem com poli-L-lisina
- Use 1 mL de uma solução de poli-L-lisina 0,1 mg / mL por placa de imagem para cobrir toda a superfície (consulte a Tabela de Materiais).
NOTA: Ajuste a concentração de poli-L-lisina com base nos requisitos específicos do tipo de célula. - Balance o prato suavemente para obter um revestimento uniforme da superfície da cultura.
NOTA: Garanta condições estéreis durante todo o processo para evitar contaminação. - Incube os pratos durante a noite em temperatura ambiente (RT).
- Aspire o excesso de solução de poli-L-lisina da louça pipetando.
- Enxágue a superfície com PBS três vezes, descansando por 5 min de cada vez (consulte a Tabela de Materiais). Remova completamente a poli-L-lisina não ligada da placa de imagem para evitar a inibição do crescimento celular. Enxágue as placas suavemente para evitar raspar ou danificar o fundo do vidro.
- Seque ao ar os pratos de imagem revestidos a 37 °C durante pelo menos 3 h.
- Use as placas de imagem revestidas imediatamente ou armazene-as a 4 ° C por até 2 semanas.
4. Transfecção de células HepG2
NOTA: O método de transfecção HepG2 é ilustrado na Figura 5.
- Semeie células HepG2 em placas de imagem pré-revestidas 24-48 h antes da transfecção para garantir que atinjam 70%-90% de confluência.
- Descongele o reagente de transfecção e o plasmídeo que codifica o biossensor FRET no gelo. Vortex completamente e execute uma rotação curta (por exemplo, 5.000 x g por 5 s) antes de usar (consulte a Tabela de Materiais).
NOTA: Certifique-se de que o reagente de transfecção e o plasmídeo estejam totalmente descongelados antes do uso.
CUIDADO: Evite ciclos repetidos de congelamento e descongelamento, pois isso pode reduzir a eficiência do reagente de transfecção. - Adicione 8 μg de plasmídeo que codifica o biossensor FRET com o tampão de reação a um volume final de 100 μL. Misture bem por vórtice por 5 s em alta velocidade (aproximadamente 3.000-5.000 x g).
NOTA: Sempre adicione plasmídeo ao tampão antes de adicionar o reagente de transfecção à base de polímero. Pelo menos 50 μl da solução devem ser constituídos pelo tampão de reacção (ver quadro de materiais). - Adicione 2,4 μL do polímero de transfecção ao tubo que contém o DNA plasmidial diluído. Misture bem por vórtice por 15 s em alta velocidade (aproximadamente 3.000-5.000 × g).
NOTA: Sempre use 0,3 μL do polímero de transfecção por 1 μg de DNA.
CUIDADO: Certifique-se de que o polímero de transfecção esteja completamente misturado com o DNA do plasmídeo para formar complexos de nanopartículas uniformes. - Incubar o biossensor e a mistura de polímeros a 37 °C por 15 min para permitir a formação de complexos de nanopartículas.
NOTA: Evite manter o polímero de transfecção em solução por mais de 30 min, pois isso pode reduzir a eficiência da transfecção.
CUIDADO: Monitore o tempo de incubação cuidadosamente para evitar a incubação excessiva, o que pode levar à redução da eficiência da transfecção. - Gire o tubo por 5 s a 5.000 x g para coletar o conteúdo na parte inferior e, em seguida, adicione todos os 100 μL de solução complexa de nanopartículas gota a gota ao meio de cultura de células. Balance suavemente o prato para frente e para trás para misturar.
NOTA: Certifique-se de que a solução complexa de nanopartículas seja adicionada gota a gota para distribuí-la uniformemente pelo meio de cultura celular.
CUIDADO: Evite balançar vigorosamente, pois isso pode desalojar as células ou causar distribuição desigual dos complexos. - Incubar a placa a 37 °C durante 4 h a uma noite.
NOTA: O tempo de incubação pode ser ajustado com base nas necessidades experimentais, mas 4 h normalmente são suficientes para uma transfecção eficiente.
CUIDADO: Evite incubação prolongada (>16 h), pois isso pode reduzir a viabilidade celular. - Remover os complexos de nanopartículas das células por aspiração, substituí-los por 2 ml de meio de crescimento completo fresco e voltar a colocar a placa na incubadora a 37 °C até ao momento da análise. O pico de expressão geralmente atinge 48 h após a transfecção.
- Analise as células sob microscopia de fluorescência.
NOTA: Certifique-se de que o microscópio esteja devidamente calibrado para imagens de fluorescência para obter resultados precisos.
CUIDADO: Minimize a exposição das células à luz intensa durante a imagem para evitar fototoxicidade.
5. Fome de células HepG2
NOTA: Depois de concluir a etapa de transfecção, deixe as células sem soro antes da estimulação da insulina e da imagem FRET. Isso minimiza a ativação da via Akt devido à insulina presente na FBS e garante níveis basais consistentes de atividade de Akt. A composição do meio de fome usado neste experimento é descrita na (Tabela 3). A BSA vem em forma de pó. Para preparar uma solução a 0,1% (p / v), reconstitua 0,1 g de BSA em 3 mL de DMEM, misturando bem. Esterilizar a solução com um filtro de 0,45 μm e ajustar o volume final para 100 ml adicionando DMEM.
- Remova o meio de cultura e lave os pratos de imagem com 1x PBS duas vezes por 5 min cada.
NOTA: As duas lavagens de PBS ajudam a remover completamente o soro residual e quaisquer fatores de insulina ou crescimento que possam interferir no experimento. - Adicione 2 mL de meio de inanição às placas de imagem (consulte a Tabela de Materiais). Adicione delicadamente o meio ao redor da borda do prato para evitar desalojar as células do fundo do vidro. Incubar a 37 °C durante 4 h.
NOTA: A incubação de 4 horas é ideal para sincronizar o metabolismo celular; no entanto, a duração pode ser estendida dependendo das necessidades experimentais.
6. Imagem de células vivas FRET para células HepG2
NOTA: Esta seção fornece instruções para imagens de células vivas FRET para monitorar a dinâmica espaço-temporal da fosforilação de Akt em células HepG2 únicas. É essencial otimizar a configuração do microscópio, os procedimentos de manuseio e as condições de imagem para células HepG2 vivas, conforme detalhado abaixo. A configuração do microscópio é crucial para otimizar as condições de imagem para imagens FRET. Siga a configuração de PC/microscopia de varredura a laser confocal (CLSM) passo a passo de acordo com as instruções do fabricante para garantir uma operação estável. A configuração CLSM personalizada para imagens FRET é mostrada na (Figura 6) .
- Ligue o interruptor remoto para alimentar o microscópio, computador, scanner, lançamento a laser, piezo stage e fonte de luz LED de epifluorescência. Certifique-se de que todos os componentes estejam conectados corretamente antes de ligar para evitar possíveis danos.
- Gire a chave para a posição ON no lançamento do laser e pressione os botões para ativar os dois lasers necessários para FRET (linhas de laser de 457 nm e 514 nm).
NOTA: Certifique-se de que os filtros e configurações apropriados estejam em vigor para obter imagens FRET ideais. A seleção da linha de laser deve ser baseada nos perfis de excitação e emissão do biossensor. - Ligue o filtro de linha para alimentar o computador e o monitor conectados ao microscópio.
NOTA: Certifique-se de que todas as conexões estejam seguras antes de ligar para evitar danos ao equipamento. - Faça login no Windows e inicie o software de microscopia.
- Clique em A1 para Aquisição para iniciar a configuração da imagem. Selecione as configurações ópticas apropriadas com base nos requisitos do experimento FRET.
- Ajuste a potência do laser e a sensibilidade do detector conforme necessário para obter as condições ideais de imagem. Instale cuidadosamente a incubadora superior do estágio do microscópio e prenda-a com parafusos.
NOTA: Não aperte demais os parafusos para evitar danos à incubadora ou à platina do microscópio. - Encha o banho-maria interno com água bidestilada estéril (ddH2O). Instale o aquecedor superior com segurança e ligue a fonte de alimentação do stage aquecedor, aquecedor de banho e aquecedor de lente (Figura 7A).
NOTA: Não encha demais para evitar derramamento no sistema. Certifique-se de que todos os aquecedores estejam funcionando corretamente para manter condições de temperatura consistentes para imagens de células vivas. - Use a lente de imersão em óleo de 40x para imagens (consulte a Tabela de Materiais).
NOTA: Consulte o site do fabricante para obter as especificações técnicas. - Limpe a lente objetiva com papel de lente umedecido com etanol a 95%. Coloque uma pequena gota de óleo de imersão na lente objetiva (Figura 7B). Coloque a placa de imagem contendo as células HepG2 no microscópio stage e prenda-o com o suporte (Figura 7C).
NOTA: Certifique-se de que a placa de imagem esteja devidamente alinhada e presa para evitar qualquer movimento durante a imagem. Evite pressão contra a lente para evitar danos à lente e ao fundo de vidro do prato. - Feche a câmara e incube as células dentro da câmara de células vivas por 1-2 h para permitir que elas se equilibrem (Figura 7D).
- Durante a imagem de lapso de tempo, faça uma pausa em intervalos específicos e remova suavemente o meio ( Figura 7E ) e, em seguida, adicione 1 mL de meio recém-preparado com a concentração de insulina fornecida ( Figura 7F , Tabela de Materiais ).
NOTA: Prepare uma solução estoque de insulina de 1 mg / mL dissolvendo 1 mg de insulina em 1 mL de ácido acético 10 mM. Filtrar a solução através de um filtro de seringa estéril de 0,2 μm e armazenar alíquotas a -20 °C. - Inicie a janela ND Acquisition no menu File do software de microscopia.
NOTA: A configuração de aquisição ND é mostrada na (Figura 8). - Selecione a guia Tempo para definir o intervalo, a duração e os loops desejados para imagens de lapso de tempo. Em seguida, clique na guia XY e pressione o botão + adicionar para incluir células HepG2 individuais para geração de imagens.
- Para adicionar várias células, localize células adequadas, digitalize, ajuste o foco e bloqueie a função de propagação de pontos (PSF). Repita esta etapa para cada nova célula adicionada para geração de imagens. Escaneie várias células em diferentes locais dentro das placas de imagem para identificar várias células-alvo para inclusão.
- Clique no ícone X vermelho para desmarcar as células. Marque a caixa 'Z' para definir a posição Z. Depois de selecionar todos os parâmetros, insira o nome do experimento na caixa 'Nome do arquivo' .
- Clique em Procurar para escolher a pasta de destino e marque a caixa salvar no arquivo. Clique na guia Executar agora para iniciar a aquisição do ND. A janela de aquisição ND mostrará o progresso em tempo real da imagem de lapso de tempo, incluindo o tempo decorrido e restante.
- Registre as medições de linha de base por até 30 minutos sem estimular as células com insulina.
- Localize células adequadas, aumente/diminua o zoom para colocar uma única célula em foco e selecione células com alta intensidade de fluorescência. Adicione as células uma a uma, até um máximo de 6 células.
NOTA: Limite o número de células para evitar atrasos na aquisição de imagens e congelamento de janelas durante a geração de imagens. - Defina o tempo e a frequência da imagem para a imagem de lapso de tempo. Inicie a imagem de lapso de tempo.
NOTA: Certifique-se de que todos os parâmetros, incluindo o tempo de exposição e a potência do laser, sejam otimizados antes de iniciar a sessão de imagem. - Pause a imagem em intervalos regulares, remova suavemente o meio do prato com fundo de vidro e adicione 1 mL de meio suplementado com a concentração apropriada de insulina.
- Retome a aquisição da imagem. Repita a etapa 6.20 conforme necessário.
- No final do experimento, clique na guia Concluir para fechar. Faça backup e armazene as imagens adquiridas com segurança para análise.
7. Análise dos dados
- Corrija os dados de imagem FRET de lapso de tempo pré-processados de amostras de controle para diafonia espectral e autofluorescência CFP, o que resulta em valores FRET corrigidos e eficiência FRET precisa. Utilizar software de imagem para aquisição, processamento e análise de imagens FRET, seguindo protocolos descritos em estudos anteriores27,28,29,30 (Figura 9).
8. Cálculos de eficiência FRET
- Para determinar a eficiência do FRET, adquira sete imagens: célula (IDA(D), IDD(D), IDA(A), IAA(A), IDD, IAA e IDA). Calcule a eficiência do FRET usando a seguinte fórmula:
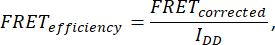
onde FRETcorrigido é obtido a partir da equação:
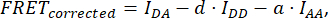
com d e a definido como:
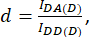
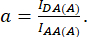
Aqui IDA, IDD e IAA representam imagens de células transfectadas com o biossensor. IDA(D) e IDD(D)) são imagens de controle somente para doadores, e IDA(A) e IAA(A) são imagens de controle somente para aceitadores.
9. Aquisição de imagem
- IDA: Obter a imagem por excitação do doador (434 nm) com emissão do aceptor (530 nm).
- IDD : Obter a imagem por excitação do doador (434 nm) com emissão do doador (477 nm).
- IAA : Obter a imagem por excitação do aceptor (517 nm) com emissão do aceptor (530 nm).
- IDA(D) e IDA(A) : Adquira a imagem usando a mesma excitação e emissão que IDA, mas a partir de células que expressam apenas o doador e o aceitador, respectivamente.
- IDD(D): Adquira a imagem nas mesmas condições que euDD para o controle somente doador.
- IAA(A) : Obtenha a imagem utilizando as mesmas condições que IAA, mas é específica para o controlo apenas para aceitadores.
10. Correção de fundo
- Abra o software de análise de imagem no computador. Na barra de menu superior, acesse o menu Arquivo . Selecione Abrir ou Abrir arquivo nas opções suspensas.
- Navegue até o diretório que contém os dados da imagem de lapso de tempo. Localize a imagem de lapso de tempo que requer análise (por exemplo, formato nd2).
- Escolha o arquivo e clique em Abrir ou OK para carregá-lo no software para visualização e análise. Defina uma região de interesse (ROI) dentro da célula (ou use a célula inteira como ROI) e registre o valor de cinza de cada pixel dentro dessa região.
- Escolha uma área livre de células como plano de fundo e calcule seu valor médio de cinza. Crie a imagem corrigida subtraindo essa média de fundo do valor de cinza de cada pixel dentro do ROI da célula.
11. Eliminação de sangramento (diafonia) FRET
NOTA: A sobreposição espectral entre a emissão do doador e a excitação do aceptor é representada na Figura 3B, que é crítica para a eficiência do FRET e o processo de transferência de energia. O sangramento na imagem FRET de lapso de tempo é um desafio significativo que surge da sobreposição espectral de fluoróforos doadores e aceptores, levando a medições imprecisas. A diafonia é inerente porque os espectros dos fluoróforos doadores e aceitadores se sobrepõem até certo ponto (Figura 3C, D). Esse problema é exacerbado por fatores como altas concentrações de fluoróforos e configurações inadequadas de filtro. Abordar o sangramento é crucial para garantir a confiabilidade das medições de FRET.
- Consulte um estudo anterior para obter um método para mitigar os efeitos de sangramento9.
12. Quantificação e análise estatística
- Realize análises estatísticas usando software de análise estatística.
Resultados
Para investigar a ativação de Akt em células HepG2, as células foram semeadas em placas de imagem pré-revestidas e transfectadas com o biossensor baseado em FREST pEevee-iAkt-NES (Figura 2A), projetado para permitir o monitoramento em tempo real da fosforilação de Akt. Após a transfecção, as células foram submetidas à falta de soro por 4 h em meio livre de soro para sincronizar seu estado metabólico e minimizar a sinalização basal da insulina....
Discussão
O protocolo para imagens FRET de células vivas para monitorar a fosforilação de Akt em células HepG2 envolve várias etapas importantes para garantir resultados confiáveis e reprodutíveis. A primeira etapa crítica é a cultura de células, que inclui manutenção celular de rotina, revestimento de placas de imagem e semeadura celular. O revestimento adequado é essencial para a fixação celular durante experimentos de imagem com lapso de tempo, pois garante uma aderência estáve...
Divulgações
Os autores declaram não haver interesses conflitantes.
Agradecimentos
Este trabalho foi parcialmente apoiado pela Fundação de Ciências Naturais de Shenzhen (JCYJ20240813113606009), pela Zona de Cooperação Shenzhen-Hong Kong para Tecnologia e Inovação (HZQB-KCZYB-2020056), Fundação Nacional de Ciências Naturais da China (32070681), Programa Nacional de P&D da China (2019YFA0906002) e Plano Peacock de Shenzhen (KQTD2016053117035204).
Materiais
| Name | Company | Catalog Number | Comments |
| 0.25% trypsin-EDTA | Gibco | Cat#25200-056 | Use ice-cold PBS for cell wash |
| 15 mm glass bottom cell culture dish | NEST | Cat#801001 | |
| 2 mL Nalgene cryogenic vials | Thermo Scientific | Cat#5012-0020 | |
| 5 mL Stripette Serological Pipets | Corning | Cat#4487 | |
| 95% Ethanol | Kermel | Cat#C028005 | |
| A1 HD25/A1R HD25 confocal microscope | Nikon | https://www.nikon.com/ | Magnification: 40×, Numerical Aperture (NA): 1.30, Pixel Dwell Time: 2.4 ms, Pixel Size: 1024 |
| Ampicillin | Sigma-Aldrich | Cat#A9393 | |
| Bovine serum albumin (BSA) | VWR Life Science | Cat#N208-10g | |
| Corning 25 cm2 rectangular culture flasks | Corning | Cat#430639 | |
| Countess 3 automated cell counter | Thermo Scientific | http://www.thermofisher.com/#AMQAX2000 | |
| Countess cell counting chamber slides | Thermo Scientific | http://www.thermofisher.com/#C10228 | |
| Digital vortex mixers | Thermo Scientific | https://www.thermofisher.com/ | |
| Dimethyl sulfoxide | Sigma-Aldrich | Cat#D2650 | |
| Eppendorf Safe-Lock Tubes 1.5 mL | Eppendorf | Cat#022363204 | |
| EZ-PCR mycoplasma detection kit | Biological Industries | Cat# 20-700-20 | |
| Fetal Bovine Serum, qualified, Australia | Gibco | Cat#10099141 | |
| GlutaMAX Supplement | Gibco | Cat#35050061 | |
| GraphPad Prism 9 | GraphPad Software | https://www.graphpad.com/ | |
| HepG2 | National Collection of Authenticated Cell Cultures | #CSTR:19375.09.3101HUMSCSP510 http://www.cellbank.org.cn/ | |
| Immersion Oil Type 37 | Cargille Laboratories | Cat #16237 | |
| Insulin | Sigma-Aldrich | Cat#I5500-50MG | Warm to 37 °C before use |
| LB Broth (1x) | Invitrogen | Cat#10855001 | |
| Minimum Essential Medium (MEM) | Gibco | Cat#11095080 | Warm to 37 °C before use |
| mySPIN 12 mini centrifuge | Thermo Scientific | https://www.thermofisher.com/ | |
| NanoDrop One | Thermo Scientific | https://www.thermofisher.com | |
| Nikon Plan Fluor 40×/1.30 Oil Lens | Nikon | https://www.nikon.com/ | |
| NIS-Elements-AR | Nikon | https://www.nikon.com/ | |
| Non-Essential Amino Acids (NEAA) (100x) | Gibco | Cat#11140050 | |
| One Shot LB Agar Plates | Invitrogen | Cat#A55802 | |
| One Shot Stbl3 chemically competent E. coli | Invitrogen | Cat#C737303 | |
| Parafilm | PARAFILM | Cat#B8R05606 | |
| PBS (phosphate buffered saline) | Gibco | Cat#10010023 | |
| pEevee-iAkt-NES (7,033 bp) | Miura et al31 | https://benchling.com/s/seq-q46zFYCfl0swLAun0t28/edit | |
| Penicillin-streptomycin | Gibco | Cat#15070063 | |
| Plasmocin prophylactic | InvivoGen | Cat#ant-mpp | |
| Poly-L-Lysine Hydrobromide | Sigma-Aldrich | Cat#P4832 | |
| Precision general purpose baths | Thermo Scientific | https://www.thermofisher.com/ | |
| QIAprep spin miniprep kit | QIAGEN | Cat#27106 | |
| SnapGene | SnapGene by Dotmatics | https://www.snapgene.com | |
| Sodium Pyruvate (100 mM) | Gibco | Cat#11360070 | |
| Syringe filter unit, 0.22 μm | Millipore | Cat#SLGP033RS | |
| Tokai Hit stage top incubator | TOKAI HIT | https://www.tokaihit-livecell.com/stagetopincubator | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | Cat#10977015 | |
| Xfect Transfection Reagent | Takara Bio | Cat#631317 |
Referências
- GBD 2021 Diabetes Collaborators. Global, regional, and national burden of diabetes from 1990 to 2021, with projections of prevalence to 2050: a systematic analysis for the Global Burden of Disease Study 2021. Lancet. 402 (10397), 203-234 (2023).
- Li, M., et al. Trends in insulin resistance: insights into mechanisms and therapeutic strategy. Signal Transduct Target Ther. 7 (1), 216 (2022).
- Poloz, Y., Stambolic, V. Obesity and cancer, a case for insulin signaling. Cell Death Dis. 6 (12), e2037 (2015).
- Arcidiacono, B., et al. Insulin resistance and cancer risk: an overview of the pathogenetic mechanisms. Exp Diabetes Res. 2012, 789174 (2012).
- Godsland, I. F. Insulin resistance and hyperinsulinaemia in the development and progression of cancer. Clin Sci (Lond). 118 (5), 315-332 (2009).
- Tsugane, S., Inoue, M. Insulin resistance and cancer: epidemiological evidence. Cancer Sci. 101 (5), 1073-1079 (2010).
- Yudhani, R. D., et al. In vitro insulin resistance model: A recent update. J Obes. 2023, 1964732 (2023).
- Akhtar, J., et al. Bistable insulin response: The win-win solution for glycemic control. iScience. 25 (12), 105561 (2022).
- Akhtar, J., Imran, M., Wang, G. Protocol for live-cell Forster resonance energy transfer imaging to reveal the bistable insulin response of single C2C12-derived myotubes. STAR Protoc. 5 (2), 103109 (2024).
- Kamino, K., et al. Optimal inference of molecular interaction dynamics in FRET microscopy. Proc Natl Acad Sci U S A. 120 (15), e2211807120 (2023).
- Kraft, A. E., Nikolaev, V. O. FRET microscopy for real-time visualization of second messengers in living cells. Methods Mol Biol. 1563, 85-90 (2017).
- Veeriah, S., et al. High-throughput time-resolved FRET reveals Akt/PKB activation as a poor prognostic marker in breast cancer. Cancer Res. 74 (18), 4983-4995 (2014).
- Conway, J. R. W., et al. Monitoring AKT activity and targeting in live tissue and disease contexts using a real-time Akt-FRET biosensor mouse. Sci Adv. 9 (17), eadf9063 (2023).
- Chandris, P., Giannouli, C. C., Panayotou, G. Imaging approaches for the study of metabolism in real time using genetically encoded reporters. Front Cell Dev Biol. 9, 725114 (2021).
- Yang, J., et al. Longitudinal FRET imaging of glucose and lactate dynamics and response to therapy in breast cancer cells. Mol Imaging Biol. 24 (1), 144-155 (2022).
- Mandrou, E., et al. A reliable system for quantitative G-protein activation imaging in cancer cells. Cells. 13 (13), 1114 (2024).
- Fang, C., Huang, Y., Zhao, Y. Review of FRET biosensing and its application in biomolecular detection. Am J Transl Res. 15 (2), 694-709 (2023).
- Miller, J. N. Fluorescence energy transfer methods in bioanalysis. Analyst. 130 (3), 265-270 (2005).
- Verveer, P. J., Rocks, O., Harpur, A. G., Bastiaens, P. I. Imaging protein interactions by FRET microscopy: FRET measurements by acceptor photobleaching. CSH Protoc. 2006 (6), (2006).
- Vu, C. Q., Arai, S. Quantitative imaging of genetically encoded fluorescence lifetime biosensors. Biosensors (Basel). 13 (10), 939 (2023).
- Wang, G. A more holistic view of the logarithmic dose-response curve offers greater insights into insulin response. J Biol Chem. 301 (1), 108037 (2025).
- Wang, G. Body mass dynamics is determined by the metabolic Ohm's law and adipocyte-autonomous fat mass homeostasis. iScience. 23 (6), 101176 (2020).
- Wang, G. Raison d'être of insulin resistance: the adjustable threshold hypothesis. J R Soc Interface. 11 (101), 20140892 (2014).
- Wang, G. Optimal homeostasis necessitates bistable control. J R Soc Interface. 9, 2723 (2012).
- Heim, R., Tsien, R. Y. Engineering green fluorescent protein for improved brightness, longer wavelengths and fluorescence resonance energy transfer. Curr Biol. 6 (2), 178-182 (1996).
- Nguyen, A. W., Daugherty, P. S. Evolutionary optimization of fluorescent proteins for intracellular FRET. Nat Biotechnol. 23 (3), 355-360 (2005).
- Broussard, J. A., Rappaz, B., Webb, D. J., Brown, C. M. Fluorescence resonance energy transfer microscopy as demonstrated by measuring the activation of the serine/threonine kinase Akt. Nat Protoc. 8 (2), 265-281 (2013).
- Gordon, G. W., Berry, G., Liang, X. H., Levine, B., Herman, B. Quantitative fluorescence resonance energy transfer measurements using fluorescence microscopy. Biophys J. 74 (5), 2702-2713 (1998).
- Tron, L., et al. Flow cytometric measurement of fluorescence resonance energy transfer on cell surfaces. Quantitative evaluation of the transfer efficiency on a cell-by-cell basis. Biophys J. 45 (5), 939-946 (1984).
- Youvan, D. C., et al. Calibration of fluorescence resonance energy transfer in microscopy. , (2002).
- Miura, H., Matsuda, M., Aoki, K. Development of a FRET biosensor with high specificity for Akt. Cell Struct Funct. 39 (1), 9-20 (2014).
- Liberio, M. S., Sadowski, M. C., Soekmadji, C., Davis, R. A., Nelson, C. C. Differential effects of tissue culture coating substrates on prostate cancer cell adherence, morphology and behavior. PLoS One. 9 (11), e112122 (2014).
- Rani, K., Sengupta, S. Multi-stimuli programmable FRET based RGB absorbing antennae towards ratiometric temperature, pH and multiple metal ion sensing. Chem Sci. 12 (47), 15533-15542 (2021).
- Betolngar, D. B., et al. pH sensitivity of FRET reporters based on cyan and yellow fluorescent proteins. Anal Bioanal Chem. 407 (14), 4183-4193 (2015).
- Salonikidis, P. S., et al. An ion-insensitive cAMP biosensor for long term quantitative ratiometric fluorescence resonance energy transfer (FRET) measurements under variable physiological conditions. J Biol Chem. 286 (26), 23419-23431 (2011).
- Youvan, D. C., et al. Calibration of fluorescence resonance energy transfer in microscopy using genetically engineered GFP derivatives on nickel chelating beads. Biotechnol Alia. 3, 1-18 (1997).
- Menaesse, A., et al. Simplified instrument calibration for wide-field fluorescence resonance energy transfer (FRET) measured by the sensitized emission method. Cytometry A. 99 (4), 407-416 (2021).
- Batta, A., Hajdu, T., Nagy, P. Improved estimation of the ratio of detection efficiencies of excited acceptors and donors for FRET measurements. Cytometry A. 103 (7), 563-574 (2023).
- Coullomb, A., et al. QuanTI-FRET: a framework for quantitative FRET measurements in living cells. Sci Rep. 10 (1), 6504 (2020).
- Hoppe, A., Christensen, K., Swanson, J. A. Fluorescence resonance energy transfer-based stoichiometry in living cells. Biophys J. 83 (6), 3652-3664 (2002).
- Zal, T., Gascoigne, N. R. Photobleaching-corrected FRET efficiency imaging of live cells. Biophys J. 86 (6), 3923-3939 (2004).
- Hochreiter, B., Kunze, M., Moser, B., Schmid, J. A. Advanced FRET normalization allows quantitative analysis of protein interactions including stoichiometries and relative affinities in living cells. Sci Rep. 9 (1), 8233 (2019).
- Liu, H., Zhang, F., Mishra, S. K., Zhou, S., Zheng, J. Knowledge-guided fuzzy logic modeling to infer cellular signaling networks from proteomic data. Sci Rep. 6, 35652 (2016).
- Garrido-Rodriguez, M., Zirngibl, K., Ivanova, O., Lobentanzer, S., Saez-Rodriguez, J. Integrating knowledge and omics to decipher mechanisms via large-scale models of signaling networks. Mol Syst Biol. 18 (7), e11036 (2022).
- Greenwald, E. C., Polanowska-Grabowska, R. K., Saucerman, J. J. Integrating fluorescent biosensor data using computational models. Methods Mol Biol. 1071, 227-248 (2014).
- Zhao, Z., Xia, J. Computational Approaches for Modeling Signal Transduction Networks. Encyclopedia of Bioinformatics and Computational Biology. , (2019).
- Wang, G., Krueger, G. R. Computational analysis of mTOR signaling pathway: bifurcation, carcinogenesis, and drug discovery. Anticancer Res. 30 (7), 2683-2688 (2010).
- Wang, G. Singularity analysis of the AKT signaling pathway reveals connections between cancer and metabolic diseases. Phys Biol. 7 (4), 046015 (2010).
- Chedere, A., Hari, K., Kumar, S., Rangarajan, A., Jolly, M. K. Multi-stability and consequent phenotypic plasticity in AMPK-Akt double negative feedback loop in cancer cells. J Clin Med. 10 (3), 472 (2021).
- Mosca, E., et al. Computational modeling of the metabolic States regulated by the kinase akt. Front Physiol. 3, 418 (2012).
- Liao, J., Madahar, V., Dang, R., Jiang, L. Quantitative FRET (qFRET) technology for the determination of protein-protein interaction affinity in solution. Molecules. 26 (21), 6339 (2021).
- Verma, A. K., Noumani, A., Yadav, A. K., Solanki, P. R. FRET based biosensor: Principle applications recent advances and challenges. Diagnostics (Basel). 13 (8), 1375 (2023).
- Mattheisen, J. M., et al. Application of bioluminescence resonance energy transfer to quantitate cell-surface expression of membrane proteins. Anal Biochem. 684, 115361 (2024).
- Lionetti, M. C., La Porta, C. A. M. FLIM-FRET investigation of heterogeneous huntingtin aggregation in HeLa cells. Methods Mol Biol. 2551, 595-604 (2023).
- Petutschnig, E. K., Pierdzig, L., Mittendorf, J., Niebisch, J. M., Lipka, V. A novel fluorescent protein pair facilitates FLIM-FRET analysis of plant immune receptor interaction under native conditions. J Exp Bot. 75 (3), 746-759 (2024).
- Sprenger, J. U., Perera, R. K., Gotz, K. R., Nikolaev, V. O. FRET microscopy for real-time monitoring of signaling events in live cells using unimolecular biosensors. J Vis Exp. 66, e4081 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados