このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
HepG2細胞における代謝制御Akt活性化ダイナミクスの生細胞フェルスター共鳴エネルギー移動イメージング
要約
ここでは、生きたHepG2細胞におけるAkt活性化とリン酸化の時空間ダイナミクスを定量化するためのプロトコルを紹介します。フェルスター共鳴エネルギー移動(FRET)イメージングは、がん細胞におけるインスリンシグナル伝達経路と代謝調節に関する貴重な洞察を提供する強力なツールです。
要約
代謝的に制御されたAkt活性化は、インスリンシグナル伝達カスケードの重要なノードであり、糖尿病とがんの関係について貴重な洞察を提供します。HepG2細胞におけるAkt活性を正確に定量するために、遺伝的にコードされたAkt特異的バイオセンサーを備えたFörster Resonance Energy Transfer(FRET)を利用した堅牢で再現性のあるプロトコルを開発しました。このプロトコルでは、細胞培養、イメージングディッシュ調製、およびHEPG2細胞のトランスフェクションによるFRETベースのバイオセンサーの発現に関する詳細な手順と、レーザー走査型共焦点顕微鏡のハードウェアおよびソフトウェア構成に関する特定のガイドラインを概説しています。その結果、HepG2細胞におけるインスリンシグナル伝達のユニークなパターンが示され、これは、定義されたスイッチオン閾値を持つがスイッチオフ閾値を持たない構成的Akt活性化を特徴とする不可逆的なスイッチを示します。対照的に、筋管はリバーシブルスイッチを表示します。HepG2細胞における持続的なAkt活性化は、肝細胞におけるインスリン抵抗性と代謝調節不全の根底にあるメカニズムを示唆しており、代謝障害とがんの進行を理解するためのより広範な意味を持っています。このプロトコールは、さまざまな疾患状況におけるAkt関連のシグナル伝達経路と細胞の挙動を調査するための貴重なフレームワークを提供します。
概要
糖尿病は、インスリン抵抗性とグルコース恒常性の障害を特徴とする、世界的な健康上の大きな課題となっています1。インスリンはグルコース代謝、細胞増殖、および生存において極めて重要な役割を果たすため、この疾患の病態生理学を解明するためには、インスリンシグナル伝達経路の包括的な理解が重要です2。多くの研究により、インスリンシグナル伝達がさまざまながんに大きく影響し、インスリン抵抗性が腫瘍の進行と患者の転帰不良に結び付けられることが実証されています3,4,5,6。HepG2細胞は、一般的に使用されている肝細胞がん細胞株であり、インスリン抵抗性や代謝調節不全とがん発生との相互作用を研究するための貴重なモデルとして機能します7。従来、研究者はインスリンの反応を段階的に見なしてきました。しかし、最近の研究では、個々の細胞が双安定応答を示すことがあり、特定のインスリン濃度閾値8,9で起こる無反応と完全反応との間の顕著な移行を示すことが明らかになりました。
フェルスター共鳴エネルギー移動(FRET)イメージングは、生細胞内の生体分子の時空間分布を研究するための強力なツールです10。分子動力学から情報を抽出することにより、FRETはAkt活性化などのプロセスに対する洞察をリアルタイムで提供し、生細胞11,12を研究するための非常に貴重な技術となっています。このイメージング法は、細胞ダイナミクスの研究、特に正確な分子相互作用が重要な代謝性疾患や癌の研究に不可欠であることが証明されています13。また、FRETは分子間相互作用のリアルタイムモニタリングを可能にし、インスリン抵抗性や腫瘍の進行などのメカニズムを明らかにします14,15。FRETバイオセンサーは、腫瘍微小環境、薬剤耐性、および代謝障害を研究するための癌研究において重要です16。増感蛍光(SE)、アクセプター漂白(AB)、蛍光寿命イメージング顕微鏡(FLIM)、および分光法などのFRET検出法は、それぞれ分子相互作用を定量化するための明確な利点を提供する17。SEは、ドナー蛍光色素とアクセプター蛍光色素の間のエネルギー移動を測定し、その結果、相互作用する生体分子の近接性と相関する発光スペクトルの測定可能なシフトが生じる18。ABは、アクセプター蛍光色素の選択的光退色を使用し、ドナー蛍光の変化を追跡することで、研究者は相互作用の速度論と距離を評価することができる19。FLIMは、FRET効率に直接影響されるドナー蛍光色素の蛍光崩壊速度を評価し、分子相互作用の正確なナノスケール測定を提供する20。
FRET技術を使用して、最近、C2C12由来の筋管8,9,21,22,23,24における双安定インスリン応答を示しました。我々が発見したように、Akt活性化のスイッチオンとスイッチオフの閾値は、段階的な全身インスリン投与量反応が、インスリン刺激から始まる細胞内シグナル伝達カスケードの複雑さを裏切っていることを示唆しており、これは単一細胞レベルでのオールオアナッシング応答で最高潮に達する21,22,23,24.他の細胞タイプにおける双安定性の存在をテストするために、HepG2細胞をインスリンで刺激し、シングルセルFRETイメージングを使用してそれらの応答を記録しました。さまざまなインスリン濃度のHepG2細胞を刺激し、Aktバイオセンサーを使用して単一細胞レベルでAkt活性を監視しました。Aktバイオセンサーは、ドナー蛍光色素分子として増強シアン蛍光タンパク質(ECFP)25、アクセプター蛍光色素分子として黄色蛍光タンパク質(YPet)26の最も明るい変異体を含み、ペプチド配列SGRPRTTTFADSCKPを含むEeveeリンカーによって連結されています。このペプチドは、ヒトグリコーゲン合成酵素キナーゼ3β(GSK3β)から最適化されたリン酸化Akt(pAkt)の基質として機能します。リン酸化されていない状態では、ドナーとアクセプターのフルオロフォアとの間の空間的分離がフェルスター半径を超え、エネルギー伝達が阻害されます。インスリン刺激を受けると、Aktリン酸化が起こり、SGRPRTTTFADSCKPのリン酸化につながります。このプロセスにより、ドナーとアクセプターがフェルスター半径内に収まるコンフォメーション変化が引き起こされ、FRET27が可能になります。その結果、FRETシグナル強度はリン酸化Akt分子の量と相関し、インスリンを介した細胞応答のリアルタイム定量が可能になります。
このプロトコルは、当初はC2C12由来筋管のインスリンシグナル伝達を研究するために開発されましたが、HepG2細胞に適用され、さまざまなハードウェアおよびソフトウェアプラットフォームで利用されることに成功しており、その適用性、適応性、および汎用性が実証されています。HepG2細胞は構成的なAkt活性を示すため、肝臓特異的なインスリンシグナル伝達および代謝プロセスを研究するための理想的な in vitro モデルとなります。プロトコルの主な特徴については、プロトコルのセクションで順を追って説明します。
プロトコル
図1に、単一HepG2細胞のAktリン酸化をモニターするためのFRET生細胞イメージングに関連する実験ステップの概要を示します。
1. プラスミドの獲得、増殖、精製
注:このセクションでは、シングルセルFRET分析に必要なプラスミドを取得、増幅、および精製するための基本的なステップを概説します。
- pEevee-iAkt-NES-YPetプラスミドを利用します(図2A)。
注:プラスミドは、基礎生物学研究所(基礎生物学研究所)の青木和弘教授から提供されました。Aktモニタリングに使用されたFRETバイオセンサーのプラスミドマップとそれぞれの制御を 図2に示します。pEevee-iAkt-NES-ECFP(ドナー; 図2B)および pEevee-iAkt-NES-Ypet (アクセプター; 図2C)プラスミドは、FRET実験中のキャリブレーションコントロールとして使用されます9。
注: 図3 は、分子内FRETバイオセンサーの構成とメカニズムを示しています。 - プラスミドを増殖させるには、化学的にコンピテントな大腸菌細胞を用いて細菌形質転換を行います( 材料の表を参照)。形質転換した細胞を100 μg/mLのアンピシリンを含むLuria-Bertani(LB)寒天に播種し、37°Cで一晩インキュベートします。 翌日、アンピシリン耐性コロニーを選択し、プラスミド増幅のために 100 μg/mL アンピシリンを添加した LB ブロスで培養します。
- プラスミドDNAを精製するには、市販のプラスミドDNA精製キット( 材料表を参照)を使用して、高純度と高収率を得ます。分光光度計を使用して吸光度比(A260/A280およびA260/A230)を測定することにより、DNAの品質を評価します( 材料の表を参照)。1.8-2.0と2.0-2.2の間の比率が最適と見なされます。アガロースゲル電気泳動によりプラスミドの完全性を検証し、トランスフェクションに適していることを確認します。
2. 細胞培養の手順
注:無菌環境を維持し、汚染を防ぐために、層流フード内ですべての細胞培養手順を実行してください。HepG2細胞培養のワークフローを 図4に示します。HepG2細胞用の完全培地は、Minimum Essential Medium(MEM)、10%ウシ胎児血清(FBS)、1%非必須アミノ酸(NEAA)、1 mMピルビン酸ナトリウム、2 mM L-グルタミンサプリメント、100 U/mLペニシリン-ストレプトマイシン、および2.5 μg/mL抗生物質-抗真菌溶液で構成されています( 材料の表、表1を参照)。
- 凍結したHepG2細胞を37°Cのサーモミキサーまたはウォーターバスに完全に解凍するまで置いて、急速に解凍します。
- 解凍した細胞を、10 mLの完全増殖培地が入った15 mLの円錐形チューブに移します( 「材料の表」を参照)。
注:細胞への熱衝撃を最小限に抑えるために、使用前に増殖培地全体を37°Cに予温してください。 - チューブを200 x g で5分間遠心分離し、細胞をペレット化します。
- 細胞ペレットを乱さないように、広口径ピペットチップを使用して上清を慎重に吸引します。
- ペレットを新鮮な完全増殖培地10 mLに再懸濁します。
- 細胞懸濁液を75cm²の組織培養フラスコに移します。
- フラスコを37°Cで加湿雰囲気下で5%CO2でインキュベートします
注:次の24〜48時間にわたって細胞を観察して、付着を確認し、回復を評価します。培地を交換する前に、適切な付着を確保するために、少なくとも4時間細胞を乱さないでください。最初の4時間は、細胞の接着を乱す可能性があるため、インキュベーターを頻繁に開けることは避けてください。施設のバイオセーフティプロトコルに従い、適切な個人用保護具(PPE)を使用して、すべての細胞培養手順で安全な作業環境を維持してください。HepG2細胞が70%〜80%のコンフルエンスに達したときに継代培養することで、最適な増殖条件を維持し、細胞の生存率と増殖の可能性に影響を与える可能性のある過密状態を防ぎます。 - 継代培養では、培地を吸引し、5 mLのリン酸緩衝生理食塩水(PBS)で細胞を1回すすぎます。
- 0.25%トリプシン1mLを細胞単層を覆うように加えます( 材料の表を参照)。
注意: 最適な活動のために、トリプシンが37°Cに事前に温められていることを確認してください。細胞を過度にトリプシン化すると生存率が低下する可能性があるため、しないでください。顕微鏡で細胞を観察し、剥離を確認します。 - 37°Cで約5分間インキュベートします。
- 細胞が剥離したら、2 mLの完全培地を加えてトリプシンを中和し、ピペッティングで細胞を回収します。
- 細胞懸濁液を静かにピペットで動かして凝集体を分解し、単一細胞懸濁液にします。
- 新しい各フラスコに3 mLの完全培地を加え、各フラスコに1:2の分割比で細胞を移します。
注:早期継代の場合は、細胞を1:2の希釈で分割します。継代4〜5の後、1:4または1:5の希釈を適宜行いてもよい。 - 細胞を37°Cで5%CO2の加湿雰囲気でインキュベートします。
注:24時間後にセルをチェックして、付着を確認し、回復を評価します。 - ルーチン培養では、2〜3日ごとに、またはpHインジケーターがピンクから黄色に変化して酸性化を示している場合は、それより早く培地を交換してください。
注:培地が酸性になりすぎると、細胞に害を及ぼす可能性があるため、避けてください。 - 細胞の形態を顕微鏡で定期的にチェックし、細胞の健康状態を確認します。
注:短期保存の場合は、HepG2細胞を-80°Cで凍結します。長期保存の場合は、液体窒素で保存してください。この実験で用いた凍結媒体の組成を(表2)に示します。
3. イメージング皿のポリ-l-リジンによるコーティング
- イメージングディッシュ1枚につき0.1 mg/mL Poly-L-Lysine溶液1 mLを使用して、表面全体を覆います( 材料の表を参照)。
注:特定の細胞タイプの要件に基づいてPoly-L-Lysine濃度を調整してください。 - 皿を優しく揺らして、培養表面の均一なコーティングを実現します。
注:汚染を防ぐために、プロセス全体で無菌状態を確保してください。 - 皿を室温(RT)で一晩インキュベートします。
- ピペッティングアウトにより、皿から余分なPoly-L-Lysine溶液を吸引します。
- PBSで表面を3回すすぎ、毎回5分間休ませます( 材料の表を参照)。結合していないPoly-L-Lysineをイメージングディッシュから完全に取り除き、細胞増殖阻害を防ぎます。ガラスの底をこすったり損傷したりしないように、プレートをやさしくすすいでください。
- コーティングされたイメージングディッシュを37°Cで少なくとも3時間風乾します。
- コーティングされたイメージングディッシュはすぐに使用するか、4°Cで最大2週間保存してください。
4. HepG2細胞のトランスフェクション
注:HepG2トランスフェクション法を 図5に示します。
- トランスフェクションの24〜48時間前に、コーティング済みのイメージングディッシュにHepG2細胞を播種し、70%〜90%のコンフルエントに達することを確認します。
- トランスフェクション試薬とFRETバイオセンサーをコードするプラスミドを氷上で解凍します。十分にボルテックスし、使用前に短いスピン(例:5,000 x gで5秒)を行います( 材料の表を参照)。
注:使用前に、トランスフェクション試薬とプラスミドが完全に解凍されていることを確認してください。
注意:凍結融解サイクルを繰り返すと、トランスフェクション試薬の効率が低下する可能性があるため、避けてください。 - FRETバイオセンサーをコードするプラスミド8 μgを反応バッファーとともに最終容量100 μLまで加え、高速で5秒間ボルテックスしてよく混合します(約3,000-5,000 x g)。
注:ポリマーベースのトランスフェクション試薬を添加する前に、必ずプラスミドをバッファーに添加してください。溶液の少なくとも50μLは反応バッファーで構成されている必要があります( 材料の表を参照)。 - 2.4 μLのトランスフェクションポリマーを、希釈したプラスミドDNAを含むチューブに加えます。高速で15秒間ボルテックスしてよく混ぜます(約3,000〜5,000 × g)。
注:DNA1 μgあたり0.3 μLのトランスフェクションポリマーを常に使用してください。
注意:トランスフェクションポリマーがプラスミドDNAと完全に混合され、均一なナノ粒子複合体を形成していることを確認してください。 - バイオセンサーとポリマー混合物を37°Cで15分間インキュベートし、ナノ粒子複合体の形成を待ちます。
注:トランスフェクションポリマーを溶液中に30分以上保持すると、トランスフェクション効率が低下する可能性があるため、避けてください。
注意:インキュベーション時間を注意深く監視して、トランスフェクション効率の低下につながる可能性のあるインキュベーションの過剰を防ぎます。 - チューブを5,000 x gで5秒間スピンダウンして内容物を底部に回収し、100 μLのナノ粒子複合体溶液全体を細胞培養培地に滴下します。プレートを前後にそっと揺らして混ぜます。
注:ナノ粒子複合体溶液を滴下して、細胞培養培地全体に均一に分散させてください。
注意:激しい揺れは、細胞が外れたり、複合体の分布が不均一になったりする可能性がありますので、避けてください。 - プレートを37°Cで4時間から一晩インキュベートします。
注:インキュベーション時間は実験のニーズに基づいて調整できますが、効率的なトランスフェクションには通常4時間で十分です。
注意:長時間のインキュベーション(>16時間)は、細胞の生存率を低下させる可能性があるため、避けてください。 - ナノ粒子複合体を吸引して細胞から除去し、2 mLの新鮮な完全増殖培地と交換し、分析時までプレートを37°Cインキュベーターに戻します。ピーク発現は通常、トランスフェクション後48時間に達します。
- 蛍光顕微鏡で細胞を解析します。
注:正確な結果を得るために、顕微鏡が蛍光イメージング用に適切に校正されていることを確認してください。
注意:イメージング中の細胞の強い光への曝露を最小限に抑えて、光毒性を防ぎます。
5. HepG2細胞の飢餓
注:トランスフェクションステップを完了した後、インスリン刺激およびFRETイメージングの前に細胞を血清欠乏させます。これにより、FBSに存在するインスリンによるAkt経路の活性化が最小限に抑えられ、Akt活性の一貫したベースラインレベルが確保されます。この実験で用いた飢餓培地の組成は、(表3)に記載されています。BSAは粉末状で提供されます。0.1%(w/v)溶液を調製するには、0.1 gのBSAを3 mLのDMEMに再構成し、十分に混合します。0.45 μmフィルターを使用して溶液を滅菌し、DMEMを添加して最終容量を100 mLに調整します。
- 培地を取り出し、イメージングディッシュを1x PBSで5分間ずつ2回すすぎます。
注:2つのPBS洗浄は、実験を妨げる可能性のある残留血清とインスリンまたは成長因子を完全に除去するのに役立ちます。 - イメージングディッシュに2 mLの飢餓培地を追加します( 材料の表を参照)。皿の端に培地をそっと加えて、ガラスの底から細胞が外れないようにします。37°Cで4時間インキュベートします。
注:4時間インキュベーションは、細胞代謝の同期に最適です。ただし、実験の必要性に応じて期間を延長することができます。
6. HEPG2細胞のFRET生細胞イメージング
注:このセクションでは、単一のHepG2細胞におけるAktリン酸化の時空間ダイナミクスをモニターするためのFRET生細胞イメージングの手順を説明します。以下に詳述するように、生HepG2細胞の顕微鏡のセットアップ、取り扱い手順、およびイメージング条件を最適化することが不可欠です。顕微鏡のセットアップは、FRETイメージングのイメージング条件を最適化するために重要です。PC/共焦点レーザー走査型顕微鏡(CLSM)のセットアップを、製造元の指示に従って段階的に進め、安定した動作を確保してください。FRETイメージング用にカスタマイズされたCLSM構成を(図6)に示します。
- リモートスイッチをオンにして、顕微鏡、コンピューター、スキャナー、レーザー発射、ピエゾステージ、落射蛍光LED光源に電力を供給します。潜在的な損傷を避けるために、電源を入れる前にすべてのコンポーネントが正しく接続されていることを確認してください。
- レーザー発射時にキーをONの位置に回し、ボタンを押してFRETに必要な両方のレーザー(457nmおよび514nmレーザーライン)をアクティブにします。
注意: 最適なFRETイメージングのために、適切なフィルターと設定が配置されていることを確認してください。レーザーラインの選択は、バイオセンサーの励起プロファイルと発光プロファイルに基づいて行う必要があります。 - 電源タップをオンにして、顕微鏡に接続されたコンピューターとモニターに電力を供給します。
注意: 機器の損傷を防ぐために、電源を入れる前にすべての接続がしっかりと固定されていることを確認してください。 - Windowsにログインし、顕微鏡ソフトウェアを起動します。
- A1 for Acquisitionをクリックして、イメージングセットアップを開始します。FRET実験の要件に基づいて、適切な光学構成を選択します。
- 必要に応じてレーザー出力と検出器の感度を調整し、最適なイメージング条件を実現します。顕微鏡ステージトップインキュベーターを慎重に取り付け、ネジで固定します。
注意: インキュベーターや顕微鏡ステージの損傷を防ぐために、ネジを締めすぎないでください。 - 内部の水浴に滅菌二重蒸留水(ddH2O)を入れます。トップヒーターをしっかりと取り付け、ステージヒーター、バスヒーター、レンズヒーターの電源をオンにします(図7A)。
メモ: システムにこぼれないように、いっぱいにしすぎないでください。すべてのヒーターが適切に機能し、生細胞イメージングの一貫した温度条件を維持していることを確認してください。 - イメージングには40倍油浸レンズを使用します( 材料の表を参照)。
注意: メーカーを参照してください web技術仕様のサイト。 - 95%エタノールで湿らせたレンズペーパーで対物レンズを拭きます。対物レンズに少量の液浸油を注ぎます(図7B)。HepG2細胞が入ったイメージングディッシュを顕微鏡ステージに置き、ホルダーで固定します(図7C)。
注意: イメージングディッシュが適切に位置合わせされ、固定されていることを確認して、イメージング中の動きを防ぎます。レンズと皿のガラス底部の両方の損傷を防ぐために、レンズへの圧力を避けてください。 - チャンバーを閉じ、生細胞チャンバー内で細胞を1〜2時間インキュベートして、平衡化させます(図7D)。
- タイムラプスイメージング中に、一定の間隔で一時停止し、培地を静かに取り出し(図7E)、次に、所定のインスリン濃度で新たに調製した培地1mLを追加します(図7F、 材料表)。
注:1 mgのインスリンを1 mLの10 mM酢酸に溶解して、1 mg / mLのインスリンストック溶液を調製します。.0.2 μmの滅菌シリンジフィルターで溶液をろ過し、アリコートを-20°Cで保存します。 - 顕微鏡ソフトウェアのFileメニューからND Acquisitionウィンドウを起動します。
メモ: ND Acquisition のセットアップは (図 8) に示されています。 - [ 時間 ] タブを選択して、タイムラプス イメージングの間隔、期間、および目的のループを設定します。次に、 XY タブをクリックし、 +追加 ボタンを押して、イメージング用の個々のHepG2細胞を含めます。
- 複数のセルを追加するには、適切なセルを見つけ、スキャンし、フォーカスを微調整し、ポイントスプレッドファンクション(PSF)をロックします。イメージング用に追加された新しいセルごとに、この手順を繰り返します。イメージングディッシュ内の異なる位置で複数の細胞をスキャンし、含めるさまざまな標的細胞を同定します。
- 赤い X アイコンをクリックして、セルの選択を解除します。「Z」ボックスをチェックして、Z位置を設定します。すべてのパラメータを選択したら、[ ファイル名] ボックスに実験名を入力します。
- [ 参照 ]をクリックして宛先フォルダを選択し、[ ファイルに保存]チェックボックスをオンにします。「 Run now 」タブを押して、NDの取得を開始します。ND取得ウィンドウには、経過時間や残り時間など、タイムラプスイメージングのリアルタイムの進行状況が表示されます。
- インスリンで細胞を刺激せずに、最大30分間のベースライン測定値を記録します。
- 適切な細胞を見つけ、ズームイン/ズームアウトして1つの細胞に焦点を合わせ、蛍光強度の高い細胞を選択します。セルを 1 つずつ追加し、最大 6 つのセルまで追加します。
注:画像取得の遅延やイメージング中のウィンドウフリーズを防ぐために、セルの数を制限してください。 - タイムラプス撮影の時間と撮影頻度を設定します。タイムラプス撮影を開始します。
注意: イメージングセッションを開始する前に、露光時間やレーザー出力を含むすべてのパラメータが最適化されていることを確認してください。 - イメージングを一定の間隔で一時停止し、ガラス底皿から培地を静かに取り出し、適切なインスリン濃度を補充した培地1 mLを加えます。
- 画像取得を再開します。必要に応じて、手順6.20を繰り返します。
- テストの最後に、[完了] タブをクリックして閉じます。取得した画像をバックアップして安全に保存して分析します。
7. データ分析
- コントロールサンプルから前処理されたタイムラプスFRETイメージングデータを補正し、スペクトルクロストークとCFP自家蛍光を検出すると、FRET値が補正され、正確なFRET効率が得られます。FRET画像の取得、処理、および解析には、先行研究27,28,29,30(図9)で概説されているプロトコルに従って、イメージングソフトウェアを使用します。
8. FRET効率の計算
- FRET効率を決定するには、セル(IDA(D)、IDD(D)、IDA(A)、IAA(A)、I DD、IAA、IDA)の7つの画像を取得します。次の式を使用してFRET効率を計算します。
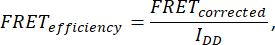
ここで、FRET Correctedは次の式から得られます。
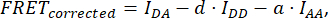
d と a は次のように定義されます。
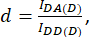
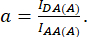
ここで、IDA、IDD、およびIAAは、バイオセンサーでトランスフェクションされた細胞の画像を表しています。IDA(D) およびIDD(D))はドナーのみの制御画像であり、IDA(A)およびIAA(A)はアクセプターのみの制御画像です。
9. 画像取得
- IDA : ドナー励起(434 nm)とアクセプター発光(530 nm)で画像を取得します。
- IDD : ドナー発光(477 nm)を伴うドナー励起(434 nm)により画像を取得します。
- IAA:アクセプター励起(517 nm)とアクセプター発光(530 nm)で画像を取得します。
- IDA(D)およびIDA(A):IDAと同じ励起と発光を使用して、それぞれドナーとアクセプターのみを発現する細胞から画像を取得します。
- IDD(D):ドナーのみのコントロールでIDDと同じ条件で画像を取得します。
- IAA(A):IAAと同じ条件を使用して画像を取得しますが、アクセプターのみのコントロールに固有です。
10.背景補正
- コンピューターで画像解析ソフトウェアを開きます。上部のメニューバーから、[ ファイル ]メニューにアクセスします。ドロップダウンオプションから[ 開く ]または [ファイルを開く ]を選択します。
- タイムラプス画像データが含まれているディレクトリに移動します。分析が必要なタイムラプス画像(nd2形式など)を見つけます。
- ファイルを選択し、[ 開く ]または [OK ]をクリックして、表示と分析のためにソフトウェアにロードします。セル内の関心領域 (ROI) を定義し (または、セル全体を ROI として使用)、この領域内の各ピクセルのグレー値を記録します。
- セルのない領域を背景として選択し、その平均グレー値を計算します。セル ROI 内の各ピクセルのグレー値からこの背景平均を差し引いて、補正された画像を作成します。
11. FRETブリードスルー(クロストーク)の除去
注:ドナー発光とアクセプター励起の間のスペクトルオーバーラップは図 3Bに示されており、これはFRETの効率とエネルギー伝達プロセスにとって重要です。タイムラプスFRETイメージングにおけるブリードスルーは、ドナーとアクセプターの蛍光色素のスペクトルの重なりから生じる大きな課題であり、不正確な測定につながります。クロストークは、ドナーとアクセプターの両方の蛍光色素のスペクトルがある程度重なるため、固有のものです(図3C、D)。この問題は、高濃度の蛍光色素や不適切なフィルター構成などの要因によって悪化します。ブリードスルーへの対処は、FRET測定の信頼性を確保するために重要です。
- ブリードスルー効果を軽減する方法については、先行研究を参照してください9。
12. 定量化と統計分析
- 統計解析ソフトウェアを使用して統計解析を行います。
結果
HepG2細胞におけるAkt活性化を調べるために、細胞をプレコートイメージングディッシュに播種し、Aktリン酸化のリアルタイムモニタリングを可能にするように設計されたFRETベースのバイオセンサーpEevee-iAkt-NES(図2A)でトランスフェクションしました。トランスフェクション後、細胞は無血清培地で4時間血清飢餓状態を経験し、代謝状態を同期さ?...
ディスカッション
HepG2細胞のAktリン酸化をモニタリングするための生細胞FRETイメージングのプロトコールには、信頼性と再現性のある結果を確保するためのいくつかの重要なステップが含まれます。最初の重要なステップは細胞培養であり、これには定期的な細胞のメンテナンス、イメージング皿のコーティング、および細胞の播種が含まれます。タイムラプスイメージング実験にお...
開示事項
著者は、競合する利益を宣言しません。
謝辞
この研究は、深セン自然科学基金会(JCYJ20240813113606009)、深セン・香港技術革新協力区(HZQB-KCZYB-2020056)、中国国家自然科学基金会(32070681)、中国国家重点研究開発プログラム(2019YFA0906002)、深セン孔雀計画(KQTD2016053117035204)の支援を受けた。
資料
| Name | Company | Catalog Number | Comments |
| 0.25% trypsin-EDTA | Gibco | Cat#25200-056 | Use ice-cold PBS for cell wash |
| 15 mm glass bottom cell culture dish | NEST | Cat#801001 | |
| 2 mL Nalgene cryogenic vials | Thermo Scientific | Cat#5012-0020 | |
| 5 mL Stripette Serological Pipets | Corning | Cat#4487 | |
| 95% Ethanol | Kermel | Cat#C028005 | |
| A1 HD25/A1R HD25 confocal microscope | Nikon | https://www.nikon.com/ | Magnification: 40×, Numerical Aperture (NA): 1.30, Pixel Dwell Time: 2.4 ms, Pixel Size: 1024 |
| Ampicillin | Sigma-Aldrich | Cat#A9393 | |
| Bovine serum albumin (BSA) | VWR Life Science | Cat#N208-10g | |
| Corning 25 cm2 rectangular culture flasks | Corning | Cat#430639 | |
| Countess 3 automated cell counter | Thermo Scientific | http://www.thermofisher.com/#AMQAX2000 | |
| Countess cell counting chamber slides | Thermo Scientific | http://www.thermofisher.com/#C10228 | |
| Digital vortex mixers | Thermo Scientific | https://www.thermofisher.com/ | |
| Dimethyl sulfoxide | Sigma-Aldrich | Cat#D2650 | |
| Eppendorf Safe-Lock Tubes 1.5 mL | Eppendorf | Cat#022363204 | |
| EZ-PCR mycoplasma detection kit | Biological Industries | Cat# 20-700-20 | |
| Fetal Bovine Serum, qualified, Australia | Gibco | Cat#10099141 | |
| GlutaMAX Supplement | Gibco | Cat#35050061 | |
| GraphPad Prism 9 | GraphPad Software | https://www.graphpad.com/ | |
| HepG2 | National Collection of Authenticated Cell Cultures | #CSTR:19375.09.3101HUMSCSP510 http://www.cellbank.org.cn/ | |
| Immersion Oil Type 37 | Cargille Laboratories | Cat #16237 | |
| Insulin | Sigma-Aldrich | Cat#I5500-50MG | Warm to 37 °C before use |
| LB Broth (1x) | Invitrogen | Cat#10855001 | |
| Minimum Essential Medium (MEM) | Gibco | Cat#11095080 | Warm to 37 °C before use |
| mySPIN 12 mini centrifuge | Thermo Scientific | https://www.thermofisher.com/ | |
| NanoDrop One | Thermo Scientific | https://www.thermofisher.com | |
| Nikon Plan Fluor 40×/1.30 Oil Lens | Nikon | https://www.nikon.com/ | |
| NIS-Elements-AR | Nikon | https://www.nikon.com/ | |
| Non-Essential Amino Acids (NEAA) (100x) | Gibco | Cat#11140050 | |
| One Shot LB Agar Plates | Invitrogen | Cat#A55802 | |
| One Shot Stbl3 chemically competent E. coli | Invitrogen | Cat#C737303 | |
| Parafilm | PARAFILM | Cat#B8R05606 | |
| PBS (phosphate buffered saline) | Gibco | Cat#10010023 | |
| pEevee-iAkt-NES (7,033 bp) | Miura et al31 | https://benchling.com/s/seq-q46zFYCfl0swLAun0t28/edit | |
| Penicillin-streptomycin | Gibco | Cat#15070063 | |
| Plasmocin prophylactic | InvivoGen | Cat#ant-mpp | |
| Poly-L-Lysine Hydrobromide | Sigma-Aldrich | Cat#P4832 | |
| Precision general purpose baths | Thermo Scientific | https://www.thermofisher.com/ | |
| QIAprep spin miniprep kit | QIAGEN | Cat#27106 | |
| SnapGene | SnapGene by Dotmatics | https://www.snapgene.com | |
| Sodium Pyruvate (100 mM) | Gibco | Cat#11360070 | |
| Syringe filter unit, 0.22 μm | Millipore | Cat#SLGP033RS | |
| Tokai Hit stage top incubator | TOKAI HIT | https://www.tokaihit-livecell.com/stagetopincubator | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | Cat#10977015 | |
| Xfect Transfection Reagent | Takara Bio | Cat#631317 |
参考文献
- GBD 2021 Diabetes Collaborators. Global, regional, and national burden of diabetes from 1990 to 2021, with projections of prevalence to 2050: a systematic analysis for the Global Burden of Disease Study 2021. Lancet. 402 (10397), 203-234 (2023).
- Li, M., et al. Trends in insulin resistance: insights into mechanisms and therapeutic strategy. Signal Transduct Target Ther. 7 (1), 216 (2022).
- Poloz, Y., Stambolic, V. Obesity and cancer, a case for insulin signaling. Cell Death Dis. 6 (12), e2037 (2015).
- Arcidiacono, B., et al. Insulin resistance and cancer risk: an overview of the pathogenetic mechanisms. Exp Diabetes Res. 2012, 789174 (2012).
- Godsland, I. F. Insulin resistance and hyperinsulinaemia in the development and progression of cancer. Clin Sci (Lond). 118 (5), 315-332 (2009).
- Tsugane, S., Inoue, M. Insulin resistance and cancer: epidemiological evidence. Cancer Sci. 101 (5), 1073-1079 (2010).
- Yudhani, R. D., et al. In vitro insulin resistance model: A recent update. J Obes. 2023, 1964732 (2023).
- Akhtar, J., et al. Bistable insulin response: The win-win solution for glycemic control. iScience. 25 (12), 105561 (2022).
- Akhtar, J., Imran, M., Wang, G. Protocol for live-cell Forster resonance energy transfer imaging to reveal the bistable insulin response of single C2C12-derived myotubes. STAR Protoc. 5 (2), 103109 (2024).
- Kamino, K., et al. Optimal inference of molecular interaction dynamics in FRET microscopy. Proc Natl Acad Sci U S A. 120 (15), e2211807120 (2023).
- Kraft, A. E., Nikolaev, V. O. FRET microscopy for real-time visualization of second messengers in living cells. Methods Mol Biol. 1563, 85-90 (2017).
- Veeriah, S., et al. High-throughput time-resolved FRET reveals Akt/PKB activation as a poor prognostic marker in breast cancer. Cancer Res. 74 (18), 4983-4995 (2014).
- Conway, J. R. W., et al. Monitoring AKT activity and targeting in live tissue and disease contexts using a real-time Akt-FRET biosensor mouse. Sci Adv. 9 (17), eadf9063 (2023).
- Chandris, P., Giannouli, C. C., Panayotou, G. Imaging approaches for the study of metabolism in real time using genetically encoded reporters. Front Cell Dev Biol. 9, 725114 (2021).
- Yang, J., et al. Longitudinal FRET imaging of glucose and lactate dynamics and response to therapy in breast cancer cells. Mol Imaging Biol. 24 (1), 144-155 (2022).
- Mandrou, E., et al. A reliable system for quantitative G-protein activation imaging in cancer cells. Cells. 13 (13), 1114 (2024).
- Fang, C., Huang, Y., Zhao, Y. Review of FRET biosensing and its application in biomolecular detection. Am J Transl Res. 15 (2), 694-709 (2023).
- Miller, J. N. Fluorescence energy transfer methods in bioanalysis. Analyst. 130 (3), 265-270 (2005).
- Verveer, P. J., Rocks, O., Harpur, A. G., Bastiaens, P. I. Imaging protein interactions by FRET microscopy: FRET measurements by acceptor photobleaching. CSH Protoc. 2006 (6), (2006).
- Vu, C. Q., Arai, S. Quantitative imaging of genetically encoded fluorescence lifetime biosensors. Biosensors (Basel). 13 (10), 939 (2023).
- Wang, G. A more holistic view of the logarithmic dose-response curve offers greater insights into insulin response. J Biol Chem. 301 (1), 108037 (2025).
- Wang, G. Body mass dynamics is determined by the metabolic Ohm's law and adipocyte-autonomous fat mass homeostasis. iScience. 23 (6), 101176 (2020).
- Wang, G. Raison d'être of insulin resistance: the adjustable threshold hypothesis. J R Soc Interface. 11 (101), 20140892 (2014).
- Wang, G. Optimal homeostasis necessitates bistable control. J R Soc Interface. 9, 2723 (2012).
- Heim, R., Tsien, R. Y. Engineering green fluorescent protein for improved brightness, longer wavelengths and fluorescence resonance energy transfer. Curr Biol. 6 (2), 178-182 (1996).
- Nguyen, A. W., Daugherty, P. S. Evolutionary optimization of fluorescent proteins for intracellular FRET. Nat Biotechnol. 23 (3), 355-360 (2005).
- Broussard, J. A., Rappaz, B., Webb, D. J., Brown, C. M. Fluorescence resonance energy transfer microscopy as demonstrated by measuring the activation of the serine/threonine kinase Akt. Nat Protoc. 8 (2), 265-281 (2013).
- Gordon, G. W., Berry, G., Liang, X. H., Levine, B., Herman, B. Quantitative fluorescence resonance energy transfer measurements using fluorescence microscopy. Biophys J. 74 (5), 2702-2713 (1998).
- Tron, L., et al. Flow cytometric measurement of fluorescence resonance energy transfer on cell surfaces. Quantitative evaluation of the transfer efficiency on a cell-by-cell basis. Biophys J. 45 (5), 939-946 (1984).
- Youvan, D. C., et al. Calibration of fluorescence resonance energy transfer in microscopy. , (2002).
- Miura, H., Matsuda, M., Aoki, K. Development of a FRET biosensor with high specificity for Akt. Cell Struct Funct. 39 (1), 9-20 (2014).
- Liberio, M. S., Sadowski, M. C., Soekmadji, C., Davis, R. A., Nelson, C. C. Differential effects of tissue culture coating substrates on prostate cancer cell adherence, morphology and behavior. PLoS One. 9 (11), e112122 (2014).
- Rani, K., Sengupta, S. Multi-stimuli programmable FRET based RGB absorbing antennae towards ratiometric temperature, pH and multiple metal ion sensing. Chem Sci. 12 (47), 15533-15542 (2021).
- Betolngar, D. B., et al. pH sensitivity of FRET reporters based on cyan and yellow fluorescent proteins. Anal Bioanal Chem. 407 (14), 4183-4193 (2015).
- Salonikidis, P. S., et al. An ion-insensitive cAMP biosensor for long term quantitative ratiometric fluorescence resonance energy transfer (FRET) measurements under variable physiological conditions. J Biol Chem. 286 (26), 23419-23431 (2011).
- Youvan, D. C., et al. Calibration of fluorescence resonance energy transfer in microscopy using genetically engineered GFP derivatives on nickel chelating beads. Biotechnol Alia. 3, 1-18 (1997).
- Menaesse, A., et al. Simplified instrument calibration for wide-field fluorescence resonance energy transfer (FRET) measured by the sensitized emission method. Cytometry A. 99 (4), 407-416 (2021).
- Batta, A., Hajdu, T., Nagy, P. Improved estimation of the ratio of detection efficiencies of excited acceptors and donors for FRET measurements. Cytometry A. 103 (7), 563-574 (2023).
- Coullomb, A., et al. QuanTI-FRET: a framework for quantitative FRET measurements in living cells. Sci Rep. 10 (1), 6504 (2020).
- Hoppe, A., Christensen, K., Swanson, J. A. Fluorescence resonance energy transfer-based stoichiometry in living cells. Biophys J. 83 (6), 3652-3664 (2002).
- Zal, T., Gascoigne, N. R. Photobleaching-corrected FRET efficiency imaging of live cells. Biophys J. 86 (6), 3923-3939 (2004).
- Hochreiter, B., Kunze, M., Moser, B., Schmid, J. A. Advanced FRET normalization allows quantitative analysis of protein interactions including stoichiometries and relative affinities in living cells. Sci Rep. 9 (1), 8233 (2019).
- Liu, H., Zhang, F., Mishra, S. K., Zhou, S., Zheng, J. Knowledge-guided fuzzy logic modeling to infer cellular signaling networks from proteomic data. Sci Rep. 6, 35652 (2016).
- Garrido-Rodriguez, M., Zirngibl, K., Ivanova, O., Lobentanzer, S., Saez-Rodriguez, J. Integrating knowledge and omics to decipher mechanisms via large-scale models of signaling networks. Mol Syst Biol. 18 (7), e11036 (2022).
- Greenwald, E. C., Polanowska-Grabowska, R. K., Saucerman, J. J. Integrating fluorescent biosensor data using computational models. Methods Mol Biol. 1071, 227-248 (2014).
- Zhao, Z., Xia, J. Computational Approaches for Modeling Signal Transduction Networks. Encyclopedia of Bioinformatics and Computational Biology. , (2019).
- Wang, G., Krueger, G. R. Computational analysis of mTOR signaling pathway: bifurcation, carcinogenesis, and drug discovery. Anticancer Res. 30 (7), 2683-2688 (2010).
- Wang, G. Singularity analysis of the AKT signaling pathway reveals connections between cancer and metabolic diseases. Phys Biol. 7 (4), 046015 (2010).
- Chedere, A., Hari, K., Kumar, S., Rangarajan, A., Jolly, M. K. Multi-stability and consequent phenotypic plasticity in AMPK-Akt double negative feedback loop in cancer cells. J Clin Med. 10 (3), 472 (2021).
- Mosca, E., et al. Computational modeling of the metabolic States regulated by the kinase akt. Front Physiol. 3, 418 (2012).
- Liao, J., Madahar, V., Dang, R., Jiang, L. Quantitative FRET (qFRET) technology for the determination of protein-protein interaction affinity in solution. Molecules. 26 (21), 6339 (2021).
- Verma, A. K., Noumani, A., Yadav, A. K., Solanki, P. R. FRET based biosensor: Principle applications recent advances and challenges. Diagnostics (Basel). 13 (8), 1375 (2023).
- Mattheisen, J. M., et al. Application of bioluminescence resonance energy transfer to quantitate cell-surface expression of membrane proteins. Anal Biochem. 684, 115361 (2024).
- Lionetti, M. C., La Porta, C. A. M. FLIM-FRET investigation of heterogeneous huntingtin aggregation in HeLa cells. Methods Mol Biol. 2551, 595-604 (2023).
- Petutschnig, E. K., Pierdzig, L., Mittendorf, J., Niebisch, J. M., Lipka, V. A novel fluorescent protein pair facilitates FLIM-FRET analysis of plant immune receptor interaction under native conditions. J Exp Bot. 75 (3), 746-759 (2024).
- Sprenger, J. U., Perera, R. K., Gotz, K. R., Nikolaev, V. O. FRET microscopy for real-time monitoring of signaling events in live cells using unimolecular biosensors. J Vis Exp. 66, e4081 (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved