Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Imagerie de transfert d’énergie par résonance de Förster en direct de la dynamique d’activation d’Akt régulée métaboliquement dans les cellules HepG2
Dans cet article
Résumé
Ici, nous présentons un protocole pour quantifier la dynamique spatio-temporelle de l’activation et de la phosphorylation d’Akt dans des cellules HepG2 vivantes. L’imagerie par transfert d’énergie par résonance de Förster (FRET) est un outil puissant qui fournit des informations précieuses sur les voies de signalisation de l’insuline et la régulation métabolique dans les cellules cancéreuses.
Résumé
L’activation d’Akt régulée métaboliquement est un nœud critique dans la cascade de signalisation de l’insuline et fournit des informations précieuses sur la relation entre le diabète et le cancer. Pour quantifier précisément l’activité d’Akt dans les cellules HepG2, nous avons développé un protocole robuste et reproductible utilisant le transfert d’énergie par résonance de Förster (FRET) avec des biocapteurs spécifiques à Akt génétiquement codés. Ce protocole décrit en détail les étapes de la culture cellulaire, de la préparation de la boîte d’imagerie et de la transfection des cellules HepG2 pour exprimer des biocapteurs basés sur FRET, ainsi que des directives spécifiques pour la configuration matérielle et logicielle du microscope confocal à balayage laser. Les résultats ont mis en évidence des modèles uniques de signalisation de l’insuline dans les cellules HepG2, qui présentent un interrupteur irréversible caractérisé par une activation constitutive d’Akt avec un seuil d’activation défini mais aucun seuil d’arrêt. En revanche, les myotubes présentent un interrupteur réversible. L’activation persistante d’Akt dans les cellules HepG2 suggère des mécanismes sous-jacents à la résistance à l’insuline et à la dérégulation métabolique dans les cellules hépatiques, avec des implications plus larges pour la compréhension de la progression des troubles métaboliques et du cancer. Ce protocole offre un cadre précieux pour explorer les voies de signalisation et les comportements cellulaires liés à Akt dans divers contextes pathologiques.
Introduction
Le diabète sucré pose un défi majeur pour la santé mondiale, caractérisé par une résistance à l’insuline et une altération de l’homéostasie du glucose1. Une compréhension complète des voies de signalisation de l’insuline est cruciale pour élucider la physiopathologie de cette maladie, car l’insuline joue un rôle central dans le métabolisme du glucose, la croissance cellulaire et la survie2. De nombreuses études ont démontré que la signalisation de l’insuline a un impact significatif sur divers cancers, liant la résistance à l’insuline à la progression tumorale et aux mauvais résultats des patients 3,4,5,6. Les cellules HepG2, une lignée cellulaire de carcinome hépatocellulaire couramment utilisée, servent de modèle précieux pour étudier la résistance à l’insuline et l’interaction entre la dérégulation métabolique et le développement du cancer7. Traditionnellement, les chercheurs considèrent les réponses à l’insuline comme classées ; Cependant, des études récentes ont révélé que les cellules individuelles peuvent présenter des réponses bistables, affichant des transitions saillantes entre l’absence de réponse et la réponse complète se produisant à des seuils spécifiques de concentration d’insuline 8,9.
L’imagerie par transfert d’énergie par résonance de Förster (FRET) est un outil puissant pour étudier la distribution spatio-temporelle des biomolécules dans les cellules vivantes10. En extrayant des informations de la dynamique moléculaire, FRET fournit des informations sur des processus tels que l’activation d’Akt en temps réel, ce qui en fait une technique inestimable pour étudier les cellules vivantes11,12. Cette méthode d’imagerie s’est avérée essentielle dans l’étude de la dynamique cellulaire, en particulier dans les maladies métaboliques et le cancer, où des interactions moléculaires précises sont cruciales13. La FRET permet également de surveiller en temps réel les interactions moléculaires, mettant en lumière des mécanismes tels que la résistance à l’insuline et la progression tumorale14,15. Les biocapteurs FRET sont cruciaux dans la recherche sur le cancer pour étudier les microenvironnements tumoraux, la résistance aux médicaments et les troubles métaboliques16. Les méthodes de détection FRET, telles que l’émission sensibilisée (SE), le blanchiment accepteur (AB), la microscopie d’imagerie à durée de vie de fluorescence (FLIM) et la spectroscopie, offrent chacune des avantages distincts pour quantifier les interactions moléculaires17. L’ES mesure le transfert d’énergie entre les fluorophores donneurs et accepteurs, ce qui entraîne un décalage mesurable des spectres d’émission en corrélation avec la proximité des biomolécules en interaction18. L’AB utilise le photoblanchiment sélectif du fluorophore accepteur et suit les changements de fluorescence du donneur, ce qui permet aux chercheurs d’évaluer la cinétique d’interaction et les distances19. FLIM évalue les taux de décroissance de la fluorescence du fluorophore donneur, directement influencés par l’efficacité du FRET, afin de fournir des mesures précises à l’échelle nanométrique des interactions moléculaires20.
En utilisant des techniques FRET, nous avons récemment démontré des réponses insuliniques bitables dans des myotubes 8,9,21,22,23,24 dérivés de C2C12. Les seuils d’activation et d’arrêt distincts de l’activation d’Akt, comme nous l’avons découvert, suggèrent que la dose-réponse graduelle de l’insuline du corps entier dément la complexité de la cascade de signalisation subcellulaire à partir du stimulus de l’insuline, qui culmine dans une réponse tout ou rien au niveau de la cellule unique 21,22,23,24. Pour tester la présence de la bistabilité dans d’autres types de cellules, nous avons stimulé les cellules HepG2 avec de l’insuline et enregistré leur réponse à l’aide de l’imagerie FRET unicellulaire. Nous avons stimulé des cellules HepG2 avec des concentrations d’insuline variables et surveillé l’activité d’Akt au niveau de la cellule unique à l’aide d’un biocapteur Akt. Le biocapteur Akt comprend la protéine fluorescente cyan améliorée (ECFP)25 en tant que fluorophore donneur et la variante la plus brillante de la protéine fluorescente jaune (YPet)26 en tant que fluorophore accepteur, reliée par un linker Évoli contenant la séquence peptidique SGRPRTTTFADSCKP. Ce peptide agit comme un substrat pour l’Akt phosphorylé (pAkt), optimisé à partir de la glycogène synthase kinase 3β humaine (GSK3β). Dans son état non phosphorylé, la séparation spatiale entre les fluorophores donneur et accepteur dépasse le rayon de Förster, ce qui inhibe le transfert d’énergie. Lors de la stimulation à l’insuline, la phosphorylation d’Akt se produit et conduit à la phosphorylation de SGRPRTTTFADSCKP. Ce processus induit un changement de conformation qui amène le donneur et l’accepteur dans le rayon de Förster, permettant FRET27. En conséquence, l’intensité du signal FRET est corrélée à la quantité de molécules Akt phosphorylées et permet de quantifier en temps réel les réponses cellulaires médiées par l’insuline.
Ce protocole, initialement développé pour étudier la signalisation de l’insuline dans les myotubes dérivés de C2C12, a été appliqué avec succès aux cellules HepG2 et utilisé sur différentes plateformes matérielles et logicielles, démontrant ainsi son applicabilité, son adaptabilité et sa polyvalence. Les cellules HepG2 présentent une activité Akt constitutive, ce qui en fait un modèle in vitro idéal pour étudier la signalisation de l’insuline et les processus métaboliques spécifiques au foie. Les principales caractéristiques du protocole sont décrites étape par étape dans la section protocole.
Protocole
La figure 1 présente un aperçu des étapes expérimentales de l’imagerie de cellules vivantes FRET pour surveiller la phosphorylation d’Akt dans des cellules HepG2 uniques.
1. Acquisition, propagation et purification des plasmides
REMARQUE : Cette section décrit les étapes essentielles pour acquérir, amplifier et purifier le plasmide requis pour l’analyse FRET sur cellule unique.
- Utilisez le plasmide pEevee-iAkt-NES-YPet (Figure 2A).
REMARQUE : Le plasmide a été généreusement fourni par le professeur Kazuhiro Aoki de l’Institut national de biologie fondamentale (NIBB), au Japon.Les cartes plasmidiques des biocapteurs FRET utilisés pour la surveillance d’Akt et leurs contrôles respectifs sont illustrées à la figure 2. Le programme pEevee-iAkt-NES-ECFP (donateur ; Graphique 2B) et pEevee-iAkt-NES-Ypet (accepteur ; Graphique 2C) Les plasmides sont utilisés comme contrôles d’étalonnage lors des expériences FRET9.
REMARQUE : La figure 3 montre la composition et le mécanisme du biocapteur FRET intramoléculaire. - Pour propager le plasmide, effectuez une transformation bactérienne à l’aide de cellules E. coli chimiquement compétentes (voir le tableau des matériaux). Déposer les cellules transformées dans une gélose Luria-Bertani (LB) contenant 100 μg/mL d’ampicilline et incuber toute la nuit à 37 °C. Le lendemain, sélectionnez une colonie résistante à l’ampicilline et cultivez-la dans un bouillon LB complété par 100 μg/mL d’ampicilline pour l’amplification plasmidique.
- Pour purifier l’ADN plasmidique, utilisez un kit de purification d’ADN plasmidique disponible dans le commerce (voir le tableau des matériaux) afin d’obtenir une pureté et un rendement élevés. Évaluer la qualité de l’ADN en mesurant les rapports d’absorbance (A260/A280 et A260/A230) à l’aide d’un spectrophotomètre (voir le tableau des matériaux). Les rapports entre 1,8-2,0 et 2,0-2,2 sont considérés comme optimaux. Vérifiez l’intégrité du plasmide par électrophorèse sur gel d’agarose pour vous assurer qu’il convient à la transfection.
2. Procédure de culture cellulaire
REMARQUE : Effectuez toutes les procédures de culture cellulaire à l’intérieur d’une hotte à flux laminaire pour maintenir un environnement stérile et prévenir la contamination. Le flux de travail de culture cellulaire HepG2 est illustré à la figure 4. Le milieu complet pour les cellules HepG2 comprend un milieu essentiel minimal (MEM), 10 % de sérum de veau fœtal (FBS), 1 % d’acides aminés non essentiels (NEAA), 1 mM de pyruvate de sodium, 2 mM de supplément de L-glutamine, 100 U/mL de pénicilline-streptomycine et 2,5 μg/mL de solution d’antibiotique-antimycosique (voir le tableau des matières, tableau 1).
- Décongelez rapidement les cellules HepG2 congelées en plaçant le flacon dans un thermomélangeur à 37 °C ou un bain-marie jusqu’à ce qu’il soit complètement décongelé.
- Transférez les cellules décongelées dans un tube conique de 15 mL contenant 10 mL de milieu de croissance complet (voir le tableau des matériaux).
REMARQUE : Préchauffez l’ensemble du milieu de croissance à 37 °C avant utilisation afin de minimiser le choc thermique des cellules. - Centrifugez le tube à 200 x g pendant 5 min pour granuler les cellules.
- Aspirez soigneusement le surnageant à l’aide d’une pointe de pipette à large calibre pour éviter de perturber la pastille cellulaire.
- Remettre la pastille en suspension dans 10 mL de milieu de croissance complet frais.
- Transvaser la suspension cellulaire dans une fiole de culture tissulaire de 75 cm².
- Incuber le ballon à 37 °C dans une atmosphère humidifiée avec 5 % de CO2
REMARQUE : Observez les cellules au cours des prochaines 24 à 48 heures pour confirmer l’attachement et évaluer la récupération. Ne dérangez pas les cellules pendant au moins 4 h pour assurer une bonne fixation avant de changer de milieu. Évitez d’ouvrir l’incubateur fréquemment pendant les 4 premières heures, car cela pourrait perturber l’attachement des cellules. Suivre les protocoles de biosécurité de l’établissement et utiliser l’équipement de protection individuelle (EPI) approprié pour maintenir un environnement de travail sûr pendant toutes les procédures de culture cellulaire. Sous-culture de cellules HepG2 lorsqu’elles atteignent une confluence de 70 % à 80 % pour maintenir des conditions de croissance optimales et prévenir le surpeuplement, ce qui peut avoir une incidence sur la viabilité cellulaire et le potentiel de croissance. - Pour la sous-culture, aspirez le milieu et rincez les cellules une fois avec 5 ml de solution saline tamponnée au phosphate (PBS).
- Ajouter 1 mL de trypsine à 0,25 % pour couvrir la monocouche cellulaire (voir le tableau des matériaux).
REMARQUE : Assurez-vous que la trypsine est préchauffée à 37 °C pour une activité optimale. Ne pas trop trypsiniser les cellules, car cela pourrait réduire la viabilité. Surveillez les cellules au microscope pour confirmer le détachement. - Incuber à 37 °C pendant environ 5 min.
- Lorsque les cellules se détachent, ajoutez 2 ml de milieu complet pour neutraliser la trypsine et prélevez les cellules par pipetage.
- Pipetez doucement la suspension cellulaire pour briser les amas et obtenir une suspension unicellulaire.
- Ajouter 3 mL de milieu complet dans chaque nouvelle fiole, puis transférer les cellules dans un rapport de 1:2 dans chaque fiole.
REMARQUE : Pour les passages précoces, divisez les cellules à une dilution de 1:2. Après les passages 4-5, des dilutions de 1:4 ou 1:5 peuvent être effectuées selon le cas. - Incuber les cellules à 37 °C dans une atmosphère humidifiée avec 5 % de CO2.
REMARQUE : Vérifiez les cellules après 24 h pour confirmer la fixation et évaluer la récupération. - Pour une culture de routine, remplacez le milieu de culture tous les 2-3 jours ou plus tôt si l’indicateur de pH passe du rose au jaune, indiquant une acidification.
REMARQUE : Ne laissez pas le milieu devenir trop acide, car cela pourrait endommager les cellules. - Vérifiez régulièrement la morphologie cellulaire au microscope pour vous assurer de la santé des cellules.
REMARQUE : Pour un entreposage à court terme, congelez les cellules HepG2 à -80 °C ; Pour un stockage à long terme, stocker dans de l’azote liquide. La composition du milieu de congélation utilisé dans cette expérience est décrite au tableau 2.
3. Revêtement des boîtes d’imagerie avec de la poly-l-lysine
- Utiliser 1 mL d’une solution de poly-L-L-Lysine à 0,1 mg/mL par parabole d’imagerie pour couvrir toute la surface (voir le Tableau des matériaux).
REMARQUE : Ajustez la concentration de poly-L-lysine en fonction des exigences spécifiques du type de cellule. - Secouez doucement le plat pour obtenir un revêtement uniforme de la surface de la culture.
REMARQUE : Assurez-vous que les conditions sont stériles tout au long du processus pour éviter toute contamination. - Incuber les plats pendant la nuit à température ambiante (RT).
- Aspirer l’excès de solution de poly-L-lysine de la boîte par pipetage.
- Rincez la surface avec du PBS trois fois, en laissant reposer 5 min à chaque fois (voir le tableau des matériaux). Retirez complètement la poly-L-lysine non liée de la boîte d’imagerie pour empêcher l’inhibition de la croissance cellulaire. Rincez doucement les plaques pour éviter de gratter ou d’endommager le fond du verre.
- Faites sécher à l’air libre les boîtes d’imagerie revêtues à 37 °C pendant au moins 3 h.
- Utilisez immédiatement les boîtes d’imagerie revêtues ou rangez-les à 4 °C jusqu’à 2 semaines.
4. Transfection des cellules HepG2
REMARQUE : La méthode de transfection de l’hépatite G2 est illustrée à la figure 5.
- Ensemencez les cellules HepG2 dans des boîtes d’imagerie pré-enrobées 24 à 48 h avant la transfection pour vous assurer qu’elles atteignent une confluence de 70 % à 90 %.
- Décongeler le réactif de transfection et le plasmide codant pour le biocapteur FRET sur de la glace. Tourbillonnez soigneusement et effectuez une courte rotation (par exemple, 5 000 x g pendant 5 s) avant utilisation (voir le tableau des matériaux).
REMARQUE : Assurez-vous que le réactif de transfection et le plasmide sont complètement décongelés avant utilisation.
ATTENTION : Évitez les cycles répétés de congélation-décongélation, car cela pourrait réduire l’efficacité du réactif de transfection. - Ajouter 8 μg de plasmide codant pour le biocapteur FRET avec le tampon de réaction jusqu’à un volume final de 100 μL. Bien mélanger en tourbillonnant pendant 5 s à grande vitesse (environ 3 000 à 5 000 x g).
REMARQUE : Ajoutez toujours le plasmide au tampon avant d’ajouter le réactif de transfection à base de polymère. Au moins 50 μL de la solution doivent être constitués du tampon de réaction (voir Tableau des matériaux). - Ajoutez 2,4 μL du polymère de transfection dans le tube contenant l’ADN plasmidique dilué. Bien mélanger en tourbillonnant pendant 15 s à grande vitesse (environ 3 000 à 5 000 × g).
REMARQUE : Utilisez toujours 0,3 μL du polymère de transfection pour 1 μg d’ADN.
ATTENTION : Assurez-vous que le polymère de transfection est bien mélangé avec l’ADN plasmidique pour former des complexes de nanoparticules uniformes. - Incuber le mélange de biocapteur et de polymère à 37 °C pendant 15 min pour permettre la formation de complexes de nanoparticules.
REMARQUE : Évitez de garder le polymère de transfection en solution pendant plus de 30 min, car cela pourrait réduire l’efficacité de la transfection.
ATTENTION : Surveillez attentivement le temps d’incubation pour éviter une incubation excessive, qui peut entraîner une réduction de l’efficacité de la transfection. - Faites pivoter le tube pendant 5 s à 5 000 x g pour recueillir le contenu au fond, puis ajoutez la totalité des 100 μL de solution complexe de nanoparticules goutte à goutte dans le milieu de culture cellulaire. Balancez doucement l’assiette d’avant en arrière pour mélanger.
REMARQUE : Assurez-vous que la solution complexe de nanoparticules est ajoutée goutte à goutte pour la répartir uniformément sur le milieu de culture cellulaire.
ATTENTION : Évitez de balancer vigoureusement, car cela pourrait déloger les cellules ou provoquer une distribution inégale des complexes. - Incuber la plaque à 37 °C pendant 4 h à toute la nuit.
REMARQUE : Le temps d’incubation peut être ajusté en fonction des besoins expérimentaux, mais 4 h sont généralement suffisants pour une transfection efficace.
ATTENTION : Évitez l’incubation prolongée (>16 h), car cela pourrait réduire la viabilité cellulaire. - Retirer les complexes de nanoparticules des cellules par aspiration, les remplacer par 2 mL de milieu de croissance complet frais et remettre la plaque dans l’incubateur à 37 °C jusqu’au moment de l’analyse. Le pic d’expression atteint généralement 48 heures après la transfection.
- Analysez les cellules sous microscopie à fluorescence.
REMARQUE : Assurez-vous que le microscope est correctement calibré pour l’imagerie de fluorescence afin d’obtenir des résultats précis.
ATTENTION : Minimisez l’exposition des cellules à une lumière intense pendant l’imagerie pour éviter la phototoxicité.
5. Famine des cellules HepG2
REMARQUE : Après avoir terminé l’étape de transfection, privez les cellules de sérum avant la stimulation à l’insuline et l’imagerie FRET. Cela minimise l’activation de la voie Akt due à l’insuline présente dans le FBS et garantit des niveaux de base cohérents de l’activité d’Akt. La composition du milieu de famine utilisé dans cette expérience est décrite dans le tableau 3. Le BSA se présente sous forme de poudre. Pour préparer une solution à 0,1 % (p/v), reconstituer 0,1 g de BSA dans 3 mL de DMEM, en mélangeant soigneusement. Stérilisez la solution à l’aide d’un filtre de 0,45 μm et ajustez le volume final à 100 mL en ajoutant du DMEM.
- Retirez le milieu de culture et rincez les boîtes d’imagerie avec 1x PBS deux fois pendant 5 min chacune.
REMARQUE : Les deux lavages PBS aident à éliminer complètement le sérum résiduel et toute insuline ou facteurs de croissance qui pourraient interférer avec l’expérience. - Ajouter 2 ml de milieu de famine dans les boîtes d’imagerie (voir le tableau des matériaux). Ajoutez délicatement le milieu sur le pourtour du plat pour éviter de déloger les cellules du fond du verre. Incuber à 37 °C pendant 4 h.
REMARQUE : L’incubation de 4 heures est optimale pour synchroniser le métabolisme cellulaire ; Cependant, la durée peut être prolongée en fonction des besoins expérimentaux.
6. Imagerie FRET de cellules vivantes pour les cellules HepG2
REMARQUE : Cette section fournit des instructions pour l’imagerie de cellules vivantes FRET afin de surveiller la dynamique spatio-temporelle de la phosphorylation d’Akt dans des cellules HepG2 uniques. Il est essentiel d’optimiser la configuration du microscope, les procédures de manipulation et les conditions d’imagerie des cellules HepG2 vivantes, comme détaillé ci-dessous. La configuration du microscope est cruciale pour optimiser les conditions d’imagerie pour l’imagerie FRET. Suivez la configuration de la microscopie à balayage laser confocale (CLSM) PC/CLSM par étapes selon les instructions du fabricant pour assurer un fonctionnement stable. La configuration CLSM personnalisée pour l’imagerie FRET est illustrée à la (Figure 6) .
- Allumez la télécommande pour alimenter le microscope, l’ordinateur, le scanner, le lancement laser, la platine piézoélectrique et la source lumineuse LED d’épifluorescence. Assurez-vous que tous les composants sont correctement connectés avant de mettre sous tension pour éviter tout dommage potentiel.
- Tournez la clé sur la position ON lors du lancement du laser et appuyez sur les boutons pour activer les deux lasers requis pour FRET (lignes laser 457 nm et 514 nm).
REMARQUE : Assurez-vous que les filtres et les paramètres appropriés sont en place pour une imagerie FRET optimale. La sélection de la ligne laser doit être basée sur les profils d’excitation et d’émission du biocapteur. - Allumez la multiprise pour alimenter l’ordinateur et le moniteur connectés au microscope.
REMARQUE : Assurez-vous que toutes les connexions sont sécurisées avant de mettre sous tension pour éviter tout dommage matériel. - Connectez-vous à Windows et lancez le logiciel de microscopie.
- Cliquez sur A1 pour l’acquisition afin de lancer la configuration de l’imagerie. Sélectionnez les configurations optiques appropriées en fonction des exigences de l’expérience FRET.
- Ajustez la puissance du laser et la sensibilité du détecteur selon vos besoins pour des conditions d’imagerie optimales. Installez avec précaution l’incubateur supérieur de la platine du microscope et fixez-le à l’aide de vis.
REMARQUE : Ne serrez pas trop les vis pour éviter d’endommager l’incubateur ou la platine du microscope. - Remplissez le bain-marie interne avec de l’eau stérile doublement distillée (jjH2O). Installez solidement le radiateur supérieur et allumez l’alimentation électrique du radiateur de scène, du chauffage du bain et du chauffage de lentille (Figure 7A).
REMARQUE : Ne remplissez pas trop pour éviter de déverser dans le système. Assurez-vous que tous les appareils de chauffage fonctionnent correctement pour maintenir des conditions de température constantes pour l’imagerie des cellules vivantes. - Utilisez l’objectif à immersion dans l’huile 40x pour l’imagerie (voir le tableau des matériaux).
REMARQUE : Reportez-vous au site Web du fabricant pour les spécifications techniques. - Essuyez l’objectif avec du papier pour objectif imbibé d’éthanol à 95 %. Placez une petite gouttelette d’huile d’immersion sur la lentille de l’objectif (Figure 7B). Placez la boîte d’imagerie contenant les cellules HepG2 sur la platine du microscope et fixez-la avec le support (Figure 7C).
REMARQUE : Assurez-vous que la parabole d’imagerie est correctement alignée et fixée pour éviter tout mouvement pendant l’imagerie. Évitez d’exercer une pression contre l’objectif pour éviter d’endommager l’objectif et le fond en verre du plat. - Fermez la chambre et incubez les cellules à l’intérieur de la chambre des cellules vivantes pendant 1 à 2 h pour leur permettre de s’équilibrer (Figure 7D).
- Pendant l’imagerie en accéléré, faites une pause à des intervalles précis et retirez délicatement le milieu (Figure 7E), puis ajoutez 1 mL de milieu fraîchement préparé avec la concentration d’insuline indiquée (Figure 7F, Tableau des matières).
REMARQUE : Préparez une solution mère d’insuline de 1 mg/mL en dissolvant 1 mg d’insuline dans 1 mL d’acide acétique 10 mM. Filtrer la solution à travers un filtre à seringue stérile de 0,2 μm et stocker les aliquotes à −20 °C. - Lancez la fenêtre d’acquisition ND à partir du menu Fichier du logiciel de microscopie.
REMARQUE : la configuration de l’acquisition ND est illustrée à la Figure 8. - Sélectionnez l’onglet Temps pour définir l’intervalle, la durée et les boucles souhaitées pour l’imagerie en accéléré. Ensuite, cliquez sur l’onglet XY et appuyez sur le bouton + ajouter pour inclure des cellules HepG2 individuelles pour l’imagerie.
- Pour ajouter plusieurs cellules, localisez les cellules appropriées, balayez, affinez la mise au point et verrouillez la fonction d’étalement de points (PSF). Répétez cette étape pour chaque nouvelle cellule ajoutée pour l’imagerie. Scannez plusieurs cellules à différents endroits dans les boîtes d’imagerie pour identifier diverses cellules cibles à inclure.
- Cliquez sur l’icône X rouge pour désélectionner les cellules. Cochez la case « Z » pour définir la position Z. Après avoir sélectionné tous les paramètres, entrez le nom de l’expérience dans la case « Nom du fichier ».
- Cliquez sur Parcourir pour choisir le dossier de destination, puis cochez la case Enregistrer dans un fichier. Appuyez sur l’onglet Exécuter maintenant pour lancer l’acquisition de ND. La fenêtre d’acquisition ND affichera la progression en temps réel de l’imagerie en accéléré, y compris le temps écoulé et le temps restant.
- Enregistrez les mesures de référence jusqu’à 30 minutes sans stimuler les cellules avec de l’insuline.
- Localisez les cellules appropriées, effectuez un zoom avant/arrière pour faire la mise au point sur une seule cellule et sélectionnez des cellules à haute intensité de fluorescence. Ajoutez les cellules une par une, jusqu’à un maximum de 6 cellules.
REMARQUE : Limitez le nombre de cellules pour éviter les retards dans l’acquisition de l’image et le gel de la fenêtre pendant l’imagerie. - Définissez l’heure et la fréquence d’imagerie pour l’imagerie en accéléré. Démarrez l’imagerie en accéléré.
REMARQUE : Assurez-vous que tous les paramètres, y compris le temps d’exposition et la puissance laser, sont optimisés avant de commencer la session d’imagerie. - Interrompez l’imagerie à intervalles réguliers, retirez délicatement le milieu de la boîte à fond de verre et ajoutez 1 ml de milieu complété par la concentration d’insuline appropriée.
- Reprenez l’acquisition de l’image. Répétez l’étape 6.20 si nécessaire.
- À la fin de l’expérience, cliquez sur l’onglet Terminer pour fermer. Sauvegardez et stockez les images acquises en toute sécurité à des fins d’analyse.
7. Analyse des données
- Corrigez les données d’imagerie FRET time-lapse prétraitées à partir d’échantillons de contrôle pour la diaphonie spectrale et l’autofluorescence CFP, ce qui permet d’obtenir des valeurs FRET corrigées et une efficacité FRET précise. Utiliser un logiciel d’imagerie pour l’acquisition, le traitement et l’analyse des images FRET, en suivant les protocoles décrits dans les études précédentes 27,28,29,30 (Figure 9).
8. Calculs d’efficacité FRET
- Pour déterminer l’efficacité du FRET, acquérez sept images : cellule (IDA(D), IDD(D), IDA(A), IAA(A), IDD, IAA et IDA). Calculez l’efficacité FRET à l’aide de la formule suivante :
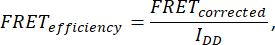
où FRETcorrigé est obtenu à partir de l’équation :
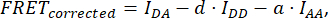
avec d et a définis comme suit :
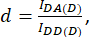
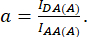
Ici, jeDA, IDD et IAA représentent des images de cellules transfectées avec le biocapteur. IDA(D) et IDD(D)) sont des images de contrôle donneur uniquement, et IDA(A) et IAA(A) sont des images de contrôle accepteur uniquement.
9. Acquisition d’images
- IDA : Obtenir l’image par excitation du donneur (434 nm) avec émission accepteur (530 nm).
- IDD : Obtenir l’image par excitation donneuse (434 nm) avec émission donneur (477 nm).
- IAA : Obtenir l’image par excitation de l’accepteur (517 nm) avec émission de l’accepteur (530 nm).
- IDA(D) et IDA(A) : Acquérir l’image en utilisant la même excitation et émission que IDA mais à partir de cellules exprimant uniquement le donneur et l’accepteur, respectivement.
- IDD(D) : Acquérir l’image dans les mêmes conditions que IDD pour le contrôle donneur uniquement.
- IAA(A) : Obtenir l’image en utilisant les mêmes conditions que IAA mais est spécifique au contrôle accepteur uniquement.
10. Correction de l’arrière-plan
- Ouvrez le logiciel d’analyse d’images sur l’ordinateur. Dans la barre de menu supérieure, accédez au menu Fichier . Sélectionnez Ouvrir ou Ouvrir un fichier dans les options déroulantes.
- Accédez au répertoire contenant les données de l’image time-lapse. Localisez l’image time-lapse qui nécessite une analyse (par exemple, au format nd2).
- Choisissez le fichier et cliquez sur Ouvrir ou sur OK pour le charger dans le logiciel à des fins de visualisation et d’analyse. Définissez une région d’intérêt (ROI) dans la cellule (ou utilisez la cellule entière comme ROI) et enregistrez la valeur de gris de chaque pixel dans cette région.
- Choisissez une zone sans cellule comme arrière-plan et calculez sa valeur de gris moyenne. Créez l’image corrigée en soustrayant cette moyenne d’arrière-plan de la valeur de gris de chaque pixel dans la zone d’intérêt de la cellule.
11. Élimination du saignement des FRET (diaphonie)
REMARQUE : Le chevauchement spectral entre l’émission du donneur et l’excitation de l’accepteur est illustré à la figure 3B, ce qui est essentiel pour l’efficacité FRET et le processus de transfert d’énergie. Le saignement dans l’imagerie FRET time-lapse est un défi important qui découle du chevauchement spectral des fluorophores donneurs et accepteurs, conduisant à des mesures imprécises. La diaphonie est inhérente au fait que les spectres des fluorophores donneurs et accepteurs se chevauchent dans une certaine mesure (figures 3C, D). Ce problème est exacerbé par des facteurs tels que des concentrations élevées de fluorophores et des configurations de filtre inappropriées. La prise en compte du saignement est cruciale pour garantir la fiabilité des mesures FRET.
- Reportez-vous à une étude antérieure pour une méthode permettant d’atténuer les effets de saignement9.
12. Quantification et analyse statistique
- Effectuer des analyses statistiques à l’aide d’un logiciel d’analyse statistique.
Résultats
Pour étudier l’activation d’Akt dans les cellules HepG2, les cellules ont été ensemencées sur des boîtes d’imagerie pré-enrobées et transfectées avec le biocapteur basé sur FRET pEevee-iAkt-NES (Figure 2A), conçu pour permettre la surveillance en temps réel de la phosphorylation d’Akt. Après la transfection, les cellules ont subi une famine sérique pendant 4 heures dans un milieu sans sérum afin de synchroniser leur état métabolique e...
Discussion
Le protocole d’imagerie FRET de cellules vivantes pour surveiller la phosphorylation d’Akt dans les cellules HepG2 implique plusieurs étapes clés pour garantir des résultats fiables et reproductibles. La première étape critique est la culture cellulaire, qui comprend l’entretien régulier des cellules, l’enrobage des boîtes d’imagerie et l’ensemencement cellulaire. Un revêtement approprié est essentiel pour la fixation des cellules pendant les expériences d’imageri...
Déclarations de divulgation
Les auteurs ne déclarent aucun intérêt concurrent.
Remerciements
Ce travail a été en partie soutenu par la Fondation des sciences naturelles de Shenzhen (JCYJ20240813113606009), la Zone de coopération Shenzhen-Hong Kong pour la technologie et l’innovation (HZQB-KCZYB-2020056), la Fondation nationale des sciences naturelles de Chine (32070681), le Programme national de R&D clé de Chine (2019YFA0906002) et le Plan Peacock de Shenzhen (KQTD2016053117035204).
matériels
| Name | Company | Catalog Number | Comments |
| 0.25% trypsin-EDTA | Gibco | Cat#25200-056 | Use ice-cold PBS for cell wash |
| 15 mm glass bottom cell culture dish | NEST | Cat#801001 | |
| 2 mL Nalgene cryogenic vials | Thermo Scientific | Cat#5012-0020 | |
| 5 mL Stripette Serological Pipets | Corning | Cat#4487 | |
| 95% Ethanol | Kermel | Cat#C028005 | |
| A1 HD25/A1R HD25 confocal microscope | Nikon | https://www.nikon.com/ | Magnification: 40×, Numerical Aperture (NA): 1.30, Pixel Dwell Time: 2.4 ms, Pixel Size: 1024 |
| Ampicillin | Sigma-Aldrich | Cat#A9393 | |
| Bovine serum albumin (BSA) | VWR Life Science | Cat#N208-10g | |
| Corning 25 cm2 rectangular culture flasks | Corning | Cat#430639 | |
| Countess 3 automated cell counter | Thermo Scientific | http://www.thermofisher.com/#AMQAX2000 | |
| Countess cell counting chamber slides | Thermo Scientific | http://www.thermofisher.com/#C10228 | |
| Digital vortex mixers | Thermo Scientific | https://www.thermofisher.com/ | |
| Dimethyl sulfoxide | Sigma-Aldrich | Cat#D2650 | |
| Eppendorf Safe-Lock Tubes 1.5 mL | Eppendorf | Cat#022363204 | |
| EZ-PCR mycoplasma detection kit | Biological Industries | Cat# 20-700-20 | |
| Fetal Bovine Serum, qualified, Australia | Gibco | Cat#10099141 | |
| GlutaMAX Supplement | Gibco | Cat#35050061 | |
| GraphPad Prism 9 | GraphPad Software | https://www.graphpad.com/ | |
| HepG2 | National Collection of Authenticated Cell Cultures | #CSTR:19375.09.3101HUMSCSP510 http://www.cellbank.org.cn/ | |
| Immersion Oil Type 37 | Cargille Laboratories | Cat #16237 | |
| Insulin | Sigma-Aldrich | Cat#I5500-50MG | Warm to 37 °C before use |
| LB Broth (1x) | Invitrogen | Cat#10855001 | |
| Minimum Essential Medium (MEM) | Gibco | Cat#11095080 | Warm to 37 °C before use |
| mySPIN 12 mini centrifuge | Thermo Scientific | https://www.thermofisher.com/ | |
| NanoDrop One | Thermo Scientific | https://www.thermofisher.com | |
| Nikon Plan Fluor 40×/1.30 Oil Lens | Nikon | https://www.nikon.com/ | |
| NIS-Elements-AR | Nikon | https://www.nikon.com/ | |
| Non-Essential Amino Acids (NEAA) (100x) | Gibco | Cat#11140050 | |
| One Shot LB Agar Plates | Invitrogen | Cat#A55802 | |
| One Shot Stbl3 chemically competent E. coli | Invitrogen | Cat#C737303 | |
| Parafilm | PARAFILM | Cat#B8R05606 | |
| PBS (phosphate buffered saline) | Gibco | Cat#10010023 | |
| pEevee-iAkt-NES (7,033 bp) | Miura et al31 | https://benchling.com/s/seq-q46zFYCfl0swLAun0t28/edit | |
| Penicillin-streptomycin | Gibco | Cat#15070063 | |
| Plasmocin prophylactic | InvivoGen | Cat#ant-mpp | |
| Poly-L-Lysine Hydrobromide | Sigma-Aldrich | Cat#P4832 | |
| Precision general purpose baths | Thermo Scientific | https://www.thermofisher.com/ | |
| QIAprep spin miniprep kit | QIAGEN | Cat#27106 | |
| SnapGene | SnapGene by Dotmatics | https://www.snapgene.com | |
| Sodium Pyruvate (100 mM) | Gibco | Cat#11360070 | |
| Syringe filter unit, 0.22 μm | Millipore | Cat#SLGP033RS | |
| Tokai Hit stage top incubator | TOKAI HIT | https://www.tokaihit-livecell.com/stagetopincubator | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | Cat#10977015 | |
| Xfect Transfection Reagent | Takara Bio | Cat#631317 |
Références
- GBD 2021 Diabetes Collaborators. Global, regional, and national burden of diabetes from 1990 to 2021, with projections of prevalence to 2050: a systematic analysis for the Global Burden of Disease Study 2021. Lancet. 402 (10397), 203-234 (2023).
- Li, M., et al. Trends in insulin resistance: insights into mechanisms and therapeutic strategy. Signal Transduct Target Ther. 7 (1), 216 (2022).
- Poloz, Y., Stambolic, V. Obesity and cancer, a case for insulin signaling. Cell Death Dis. 6 (12), e2037 (2015).
- Arcidiacono, B., et al. Insulin resistance and cancer risk: an overview of the pathogenetic mechanisms. Exp Diabetes Res. 2012, 789174 (2012).
- Godsland, I. F. Insulin resistance and hyperinsulinaemia in the development and progression of cancer. Clin Sci (Lond). 118 (5), 315-332 (2009).
- Tsugane, S., Inoue, M. Insulin resistance and cancer: epidemiological evidence. Cancer Sci. 101 (5), 1073-1079 (2010).
- Yudhani, R. D., et al. In vitro insulin resistance model: A recent update. J Obes. 2023, 1964732 (2023).
- Akhtar, J., et al. Bistable insulin response: The win-win solution for glycemic control. iScience. 25 (12), 105561 (2022).
- Akhtar, J., Imran, M., Wang, G. Protocol for live-cell Forster resonance energy transfer imaging to reveal the bistable insulin response of single C2C12-derived myotubes. STAR Protoc. 5 (2), 103109 (2024).
- Kamino, K., et al. Optimal inference of molecular interaction dynamics in FRET microscopy. Proc Natl Acad Sci U S A. 120 (15), e2211807120 (2023).
- Kraft, A. E., Nikolaev, V. O. FRET microscopy for real-time visualization of second messengers in living cells. Methods Mol Biol. 1563, 85-90 (2017).
- Veeriah, S., et al. High-throughput time-resolved FRET reveals Akt/PKB activation as a poor prognostic marker in breast cancer. Cancer Res. 74 (18), 4983-4995 (2014).
- Conway, J. R. W., et al. Monitoring AKT activity and targeting in live tissue and disease contexts using a real-time Akt-FRET biosensor mouse. Sci Adv. 9 (17), eadf9063 (2023).
- Chandris, P., Giannouli, C. C., Panayotou, G. Imaging approaches for the study of metabolism in real time using genetically encoded reporters. Front Cell Dev Biol. 9, 725114 (2021).
- Yang, J., et al. Longitudinal FRET imaging of glucose and lactate dynamics and response to therapy in breast cancer cells. Mol Imaging Biol. 24 (1), 144-155 (2022).
- Mandrou, E., et al. A reliable system for quantitative G-protein activation imaging in cancer cells. Cells. 13 (13), 1114 (2024).
- Fang, C., Huang, Y., Zhao, Y. Review of FRET biosensing and its application in biomolecular detection. Am J Transl Res. 15 (2), 694-709 (2023).
- Miller, J. N. Fluorescence energy transfer methods in bioanalysis. Analyst. 130 (3), 265-270 (2005).
- Verveer, P. J., Rocks, O., Harpur, A. G., Bastiaens, P. I. Imaging protein interactions by FRET microscopy: FRET measurements by acceptor photobleaching. CSH Protoc. 2006 (6), (2006).
- Vu, C. Q., Arai, S. Quantitative imaging of genetically encoded fluorescence lifetime biosensors. Biosensors (Basel). 13 (10), 939 (2023).
- Wang, G. A more holistic view of the logarithmic dose-response curve offers greater insights into insulin response. J Biol Chem. 301 (1), 108037 (2025).
- Wang, G. Body mass dynamics is determined by the metabolic Ohm's law and adipocyte-autonomous fat mass homeostasis. iScience. 23 (6), 101176 (2020).
- Wang, G. Raison d'être of insulin resistance: the adjustable threshold hypothesis. J R Soc Interface. 11 (101), 20140892 (2014).
- Wang, G. Optimal homeostasis necessitates bistable control. J R Soc Interface. 9, 2723 (2012).
- Heim, R., Tsien, R. Y. Engineering green fluorescent protein for improved brightness, longer wavelengths and fluorescence resonance energy transfer. Curr Biol. 6 (2), 178-182 (1996).
- Nguyen, A. W., Daugherty, P. S. Evolutionary optimization of fluorescent proteins for intracellular FRET. Nat Biotechnol. 23 (3), 355-360 (2005).
- Broussard, J. A., Rappaz, B., Webb, D. J., Brown, C. M. Fluorescence resonance energy transfer microscopy as demonstrated by measuring the activation of the serine/threonine kinase Akt. Nat Protoc. 8 (2), 265-281 (2013).
- Gordon, G. W., Berry, G., Liang, X. H., Levine, B., Herman, B. Quantitative fluorescence resonance energy transfer measurements using fluorescence microscopy. Biophys J. 74 (5), 2702-2713 (1998).
- Tron, L., et al. Flow cytometric measurement of fluorescence resonance energy transfer on cell surfaces. Quantitative evaluation of the transfer efficiency on a cell-by-cell basis. Biophys J. 45 (5), 939-946 (1984).
- Youvan, D. C., et al. Calibration of fluorescence resonance energy transfer in microscopy. , (2002).
- Miura, H., Matsuda, M., Aoki, K. Development of a FRET biosensor with high specificity for Akt. Cell Struct Funct. 39 (1), 9-20 (2014).
- Liberio, M. S., Sadowski, M. C., Soekmadji, C., Davis, R. A., Nelson, C. C. Differential effects of tissue culture coating substrates on prostate cancer cell adherence, morphology and behavior. PLoS One. 9 (11), e112122 (2014).
- Rani, K., Sengupta, S. Multi-stimuli programmable FRET based RGB absorbing antennae towards ratiometric temperature, pH and multiple metal ion sensing. Chem Sci. 12 (47), 15533-15542 (2021).
- Betolngar, D. B., et al. pH sensitivity of FRET reporters based on cyan and yellow fluorescent proteins. Anal Bioanal Chem. 407 (14), 4183-4193 (2015).
- Salonikidis, P. S., et al. An ion-insensitive cAMP biosensor for long term quantitative ratiometric fluorescence resonance energy transfer (FRET) measurements under variable physiological conditions. J Biol Chem. 286 (26), 23419-23431 (2011).
- Youvan, D. C., et al. Calibration of fluorescence resonance energy transfer in microscopy using genetically engineered GFP derivatives on nickel chelating beads. Biotechnol Alia. 3, 1-18 (1997).
- Menaesse, A., et al. Simplified instrument calibration for wide-field fluorescence resonance energy transfer (FRET) measured by the sensitized emission method. Cytometry A. 99 (4), 407-416 (2021).
- Batta, A., Hajdu, T., Nagy, P. Improved estimation of the ratio of detection efficiencies of excited acceptors and donors for FRET measurements. Cytometry A. 103 (7), 563-574 (2023).
- Coullomb, A., et al. QuanTI-FRET: a framework for quantitative FRET measurements in living cells. Sci Rep. 10 (1), 6504 (2020).
- Hoppe, A., Christensen, K., Swanson, J. A. Fluorescence resonance energy transfer-based stoichiometry in living cells. Biophys J. 83 (6), 3652-3664 (2002).
- Zal, T., Gascoigne, N. R. Photobleaching-corrected FRET efficiency imaging of live cells. Biophys J. 86 (6), 3923-3939 (2004).
- Hochreiter, B., Kunze, M., Moser, B., Schmid, J. A. Advanced FRET normalization allows quantitative analysis of protein interactions including stoichiometries and relative affinities in living cells. Sci Rep. 9 (1), 8233 (2019).
- Liu, H., Zhang, F., Mishra, S. K., Zhou, S., Zheng, J. Knowledge-guided fuzzy logic modeling to infer cellular signaling networks from proteomic data. Sci Rep. 6, 35652 (2016).
- Garrido-Rodriguez, M., Zirngibl, K., Ivanova, O., Lobentanzer, S., Saez-Rodriguez, J. Integrating knowledge and omics to decipher mechanisms via large-scale models of signaling networks. Mol Syst Biol. 18 (7), e11036 (2022).
- Greenwald, E. C., Polanowska-Grabowska, R. K., Saucerman, J. J. Integrating fluorescent biosensor data using computational models. Methods Mol Biol. 1071, 227-248 (2014).
- Zhao, Z., Xia, J. Computational Approaches for Modeling Signal Transduction Networks. Encyclopedia of Bioinformatics and Computational Biology. , (2019).
- Wang, G., Krueger, G. R. Computational analysis of mTOR signaling pathway: bifurcation, carcinogenesis, and drug discovery. Anticancer Res. 30 (7), 2683-2688 (2010).
- Wang, G. Singularity analysis of the AKT signaling pathway reveals connections between cancer and metabolic diseases. Phys Biol. 7 (4), 046015 (2010).
- Chedere, A., Hari, K., Kumar, S., Rangarajan, A., Jolly, M. K. Multi-stability and consequent phenotypic plasticity in AMPK-Akt double negative feedback loop in cancer cells. J Clin Med. 10 (3), 472 (2021).
- Mosca, E., et al. Computational modeling of the metabolic States regulated by the kinase akt. Front Physiol. 3, 418 (2012).
- Liao, J., Madahar, V., Dang, R., Jiang, L. Quantitative FRET (qFRET) technology for the determination of protein-protein interaction affinity in solution. Molecules. 26 (21), 6339 (2021).
- Verma, A. K., Noumani, A., Yadav, A. K., Solanki, P. R. FRET based biosensor: Principle applications recent advances and challenges. Diagnostics (Basel). 13 (8), 1375 (2023).
- Mattheisen, J. M., et al. Application of bioluminescence resonance energy transfer to quantitate cell-surface expression of membrane proteins. Anal Biochem. 684, 115361 (2024).
- Lionetti, M. C., La Porta, C. A. M. FLIM-FRET investigation of heterogeneous huntingtin aggregation in HeLa cells. Methods Mol Biol. 2551, 595-604 (2023).
- Petutschnig, E. K., Pierdzig, L., Mittendorf, J., Niebisch, J. M., Lipka, V. A novel fluorescent protein pair facilitates FLIM-FRET analysis of plant immune receptor interaction under native conditions. J Exp Bot. 75 (3), 746-759 (2024).
- Sprenger, J. U., Perera, R. K., Gotz, K. R., Nikolaev, V. O. FRET microscopy for real-time monitoring of signaling events in live cells using unimolecular biosensors. J Vis Exp. 66, e4081 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon