È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Imaging del trasferimento di energia della risonanza di Förster su cellule vive delle dinamiche di attivazione di Akt metabolicamente regolate nelle cellule HepG2
In questo articolo
Riepilogo
Qui, presentiamo un protocollo per quantificare le dinamiche spazio-temporali dell'attivazione e della fosforilazione di Akt in cellule HepG2 vive. L'imaging a trasferimento di energia per risonanza di Förster (FRET) è uno strumento potente che fornisce preziose informazioni sulle vie di segnalazione dell'insulina e sulla regolazione metabolica nelle cellule tumorali.
Abstract
L'attivazione metabolicamente regolata di Akt è un nodo critico nella cascata di segnalazione dell'insulina e fornisce preziose informazioni sulla relazione tra diabete e cancro. Per quantificare con precisione l'attività di Akt nelle cellule HepG2, abbiamo sviluppato un protocollo robusto e riproducibile che utilizza il trasferimento di energia di risonanza Förster (FRET) con biosensori specifici per Akt geneticamente codificati. Questo protocollo delinea i passaggi dettagliati per la coltura cellulare, la preparazione della piastra di imaging e la trasfezione di cellule HepG2 per esprimere biosensori basati su FRET, insieme a linee guida specifiche per la configurazione hardware e software del microscopio confocale a scansione laser. I risultati hanno dimostrato modelli unici di segnalazione dell'insulina nelle cellule HepG2, che mostrano un interruttore irreversibile caratterizzato dall'attivazione costitutiva di Akt con una soglia di accensione definita ma nessuna soglia di spegnimento. Al contrario, i miotubi mostrano un interruttore reversibile. L'attivazione persistente di Akt nelle cellule HepG2 suggerisce meccanismi alla base della resistenza all'insulina e della disregolazione metabolica nelle cellule epatiche, con implicazioni più ampie per la comprensione della progressione dei disturbi metabolici e del cancro. Questo protocollo offre un quadro prezioso per esplorare le vie di segnalazione correlate ad Akt e i comportamenti cellulari in vari contesti patologici.
Introduzione
Il diabete mellito rappresenta una delle principali sfide per la salute globale, caratterizzato da insulino-resistenza e alterata omeostasi del glucosio1. Una comprensione completa delle vie di segnalazione dell'insulina è fondamentale per chiarire la fisiopatologia di questa malattia, poiché l'insulina svolge un ruolo fondamentale nel metabolismo del glucosio, nella crescita cellulare e nella sopravvivenza2. Numerosi studi hanno dimostrato che la segnalazione dell'insulina ha un impatto significativo su vari tipi di cancro, collegando l'insulino-resistenza alla progressione del tumore e agli scarsi risultati dei pazienti 3,4,5,6. Le cellule HepG2, una linea cellulare di carcinoma epatocellulare comunemente usata, fungono da modello prezioso per studiare l'insulino-resistenza e l'interazione tra disregolazione metabolica e sviluppo del cancro7. Tradizionalmente, i ricercatori hanno visto le risposte all'insulina come classificate; Tuttavia, studi recenti hanno rivelato che le singole cellule possono mostrare risposte bistabili, mostrando transizioni salienti tra la mancata risposta e la risposta completa che si verificano a specifiche soglie di concentrazione di insulina 8,9.
L'imaging FRET (Förster Resonance Energy Transfer) è un potente strumento per studiare la distribuzione spazio-temporale delle biomolecole nelle cellule viventi10. Estraendo informazioni dalla dinamica molecolare, FRET fornisce informazioni su processi come l'attivazione di Akt in tempo reale, rendendola una tecnica inestimabile per lo studio delle cellule viventi11,12. Questo metodo di imaging si è dimostrato essenziale nello studio delle dinamiche cellulari, in particolare nelle malattie metaboliche e nel cancro, dove le interazioni molecolari precise sono cruciali13. FRET consente inoltre il monitoraggio in tempo reale delle interazioni molecolari, facendo luce su meccanismi come l'insulino-resistenza e la progressione tumorale14,15. I biosensori FRET sono fondamentali nella ricerca sul cancro per lo studio dei microambienti tumorali, della resistenza ai farmaci e dei disturbi metabolici16. I metodi di rilevamento FRET, come l'emissione sensibilizzata (SE), lo sbiancamento accettore (AB), la microscopia per imaging a fluorescenza (FLIM) e la spettroscopia, offrono vantaggi distinti per quantificare le interazioni molecolari17. SE misura il trasferimento di energia tra i fluorofori donatore e accettore, determinando uno spostamento misurabile negli spettri di emissione che è correlato alla vicinanza di biomolecole interagenti18. AB utilizza il fotosbiancamento selettivo del fluoroforo accettore e tiene traccia dei cambiamenti nella fluorescenza del donatore, il che consente ai ricercatori di valutare la cinetica e le distanze di interazione19. FLIM valuta i tassi di decadimento della fluorescenza del fluoroforo donatore, direttamente influenzati dall'efficienza FRET, per fornire misurazioni precise su scala nanometrica delle interazioni molecolari20.
Utilizzando le tecniche FRET, abbiamo recentemente dimostrato risposte insuliniche bistabili in miotubiderivati da C2C12 8,9,21,22,23,24. Le distinte soglie di accensione e spegnimento per l'attivazione di Akt, come abbiamo scoperto, suggeriscono che la dose-risposta graduata all'insulina in tutto il corpo smentisce la complessità della cascata di segnalazione subcellulare a partire dallo stimolo insulinico, che culmina in una risposta tutto o niente a livello di singola cellula 21,22,23,24 . Per testare la presenza di bistabilità in altri tipi di cellule, abbiamo stimolato le cellule HepG2 con insulina e registrato la loro risposta utilizzando l'imaging FRET a cellula singola. Abbiamo stimolato le cellule HepG2 con concentrazioni variabili di insulina e monitorato l'attività di Akt a livello di singola cellula utilizzando un biosensore Akt. Il biosensore Akt comprende la proteina fluorescente ciano potenziata (ECFP)25 come fluoroforo donatore e la variante più brillante della proteina fluorescente gialla (YPet)26 come fluoroforo accettore, collegato da un linker Eevee contenente la sequenza peptidica SGRPRTTTFADSCKP. Questo peptide funge da substrato per Akt fosforilato (pAkt), ottimizzato dalla glicogeno sintasi chinasi 3β umana (GSK3β). Nel suo stato non fosforilato, la separazione spaziale tra i fluorofori donatori e accettori supera il raggio di Förster, che inibisce il trasferimento di energia. Dopo la stimolazione dell'insulina, si verifica la fosforilazione di Akt che porta alla fosforilazione di SGRPRTTTFADSCKP. Questo processo induce un cambiamento conformazionale che porta il donatore e l'accettore all'interno del raggio di Förster, consentendo FRET27. Di conseguenza, l'intensità del segnale FRET è correlata alla quantità di molecole di Akt fosforilate e consente la quantificazione in tempo reale delle risposte cellulari mediate dall'insulina.
Questo protocollo, inizialmente sviluppato per studiare la segnalazione dell'insulina in miotubi derivati da C2C12, è stato applicato con successo alle cellule HepG2 e utilizzato su diverse piattaforme hardware e software, dimostrando così la sua applicabilità, adattabilità e versatilità. Le cellule HepG2 mostrano un'attività costitutiva di Akt, che le rende un modello in vitro ideale per studiare la segnalazione dell'insulina specifica del fegato e i processi metabolici. Le caratteristiche principali del protocollo sono descritte passo dopo passo nella sezione relativa al protocollo.
Protocollo
Una panoramica delle fasi sperimentali coinvolte nell'imaging di cellule vive FRET per monitorare la fosforilazione di Akt in singole cellule HepG2 è mostrata nella Figura 1.
1. Acquisizione, propagazione e purificazione dei plasmidi
NOTA: Questa sezione descrive i passaggi essenziali per l'acquisizione, l'amplificazione e la purificazione del plasmide necessari per l'analisi FRET su singola cellula.
- Utilizzare il plasmide pEevee-iAkt-NES-YPet (Figura 2A).
NOTA: Il plasmide è stato generosamente fornito dal Prof. Kazuhiro Aoki presso l'Istituto Nazionale di Biologia di Base (NIBB), Giappone.Le mappe plasmidi per i biosensori FRET utilizzati per il monitoraggio di Akt e i rispettivi controlli sono illustrati nella Figura 2. Il pEevee-iAkt-NES-ECFP (donatore; Figura 2B) e pEevee-iAkt-NES-Ypet (accettore; Figura 2C) i plasmidi sono utilizzati come controlli di calibrazione durante gli esperimenti FRET9.
NOTA: La Figura 3 mostra la composizione e il meccanismo del biosensore FRET intramolecolare. - Per propagare il plasmide, eseguire la trasformazione batterica utilizzando cellule di E. coli chimicamente competenti (vedi Tabella dei materiali). Placcare le cellule trasformate su agar Luria-Bertani (LB) contenente 100 μg/mL di ampicillina e incubare per una notte a 37 °C. Il giorno successivo, selezionare una colonia resistente all'ampicillina e coltivarla in brodo LB integrato con 100 μg/mL di ampicillina per l'amplificazione plasmidica.
- Per purificare il DNA plasmidico, utilizzare un kit di purificazione del DNA plasmidico disponibile in commercio (vedi Tabella dei materiali) per ottenere un'elevata purezza e resa. Valutare la qualità del DNA misurando i rapporti di assorbanza (A260/A280 e A260/A230) utilizzando uno spettrofotometro (vedi Tabella dei materiali). I rapporti compresi tra 1,8-2,0 e 2,0-2,2 sono considerati ottimali. Verificare l'integrità del plasmide attraverso l'elettroforesi su gel di agarosio per assicurarsi che sia adatto alla trasfezione.
2. Procedura di coltura cellulare
NOTA: Eseguire tutte le procedure di coltura cellulare all'interno di una cappa a flusso laminare per mantenere un ambiente sterile e prevenire la contaminazione. Il flusso di lavoro della coltura cellulare HepG2 è mostrato nella Figura 4. Il terreno completo per le cellule HepG2 è costituito da un terreno essenziale minimo (MEM), 10% di siero fetale bovino (FBS), 1% di aminoacidi non essenziali (NEAA), 1 mM di piruvato di sodio, 2 mM di integratore di L-glutammina, 100 U/mL di penicillina-streptomicina e 2,5 μg/mL di soluzione antibiotico-antimicotica (vedi Tabella dei materiali, Tabella 1).
- Scongelare rapidamente le cellule HepG2 congelate ponendo la fiala in un termomiscelatore a 37 °C o in un bagnomaria fino a completo scongelamento.
- Trasferire le cellule scongelate in una provetta conica da 15 mL contenente 10 mL di terreno di crescita completo (vedere la Tabella dei Materiali).
NOTA: Preriscaldare l'intero terreno di coltura a 37 °C prima dell'uso per ridurre al minimo lo shock termico delle cellule. - Centrifugare la provetta a 200 x g per 5 minuti per pellettare le cellule.
- Aspirare con cura il surnatante utilizzando una punta per pipetta a foro largo per evitare di disturbare il pellet cellulare.
- Risospendere il pellet in 10 mL di terreno di coltura completo fresco.
- Trasferire la sospensione cellulare in un pallone per coltura tissutale da 75 cm².
- Incubare il matraccio a 37 °C in atmosfera umidificata con CO2 al 5%
NOTA: Osservare le cellule nelle successive 24-48 ore per confermare l'attaccamento e valutare il recupero. Non disturbare le celle per almeno 4 ore per garantire un corretto fissaggio prima di cambiare il fluido. Evitare di aprire frequentemente l'incubatrice durante le prime 4 ore, poiché ciò potrebbe interrompere l'attacco delle cellule. Seguire i protocolli istituzionali di biosicurezza e utilizzare dispositivi di protezione individuale (DPI) appropriati per mantenere un ambiente di lavoro sicuro durante tutte le procedure di coltura cellulare. Subcoltura delle cellule HepG2 quando raggiungono la confluenza del 70%-80% per mantenere condizioni di crescita ottimali e prevenire il sovraffollamento, che può influire sulla vitalità cellulare e sul potenziale di crescita. - Per la subcoltura, aspirare il terreno e sciacquare le cellule una volta con 5 mL di soluzione salina tamponata con fosfato (PBS).
- Aggiungere 1 mL di tripsina allo 0,25% per coprire il monostrato cellulare (vedere la Tabella dei materiali).
NOTA: Assicurarsi che la tripsina sia preriscaldata a 37 °C per un'attività ottimale. Non tripsinizzare eccessivamente le cellule, poiché ciò potrebbe ridurre la vitalità. Monitorare le cellule al microscopio per confermare il distacco. - Incubare a 37 °C per circa 5 min.
- Quando le cellule si staccano, aggiungere 2 mL di terreno completo per neutralizzare la tripsina e raccogliere le cellule mediante pipettaggio.
- Pipettare delicatamente la sospensione cellulare per rompere i grumi e ottenere una sospensione a cellula singola.
- Aggiungere 3 mL di terreno completo a ciascun nuovo pallone, quindi trasferire le cellule in ciascun pallone con un rapporto di divisione di 1:2.
NOTA: Per i primi passaggi, dividere le celle a una diluizione di 1:2. Dopo i passaggi 4-5, possono essere eseguite diluizioni di 1:4 o 1:5, a seconda dei casi. - Incubare le cellule a 37 °C in atmosfera umidificata con CO2 al 5%.
NOTA: Controllare le celle dopo 24 ore per confermare l'attaccamento e valutare il recupero. - Per la coltura di routine, sostituire il terreno di coltura ogni 2-3 giorni o prima se l'indicatore di pH cambia da rosa a giallo, indicando acidificazione.
NOTA: Non lasciare che il terreno diventi troppo acido, poiché ciò potrebbe danneggiare le cellule. - Controllare regolarmente la morfologia cellulare al microscopio per garantire la salute delle cellule.
NOTA: Per la conservazione a breve termine, congelare le celle HepG2 a -80 °C; Per la conservazione a lungo termine, conservare in azoto liquido. La composizione del mezzo di congelamento utilizzato in questo esperimento è descritta nella (Tabella 2).
3. Rivestimento di piastre di imaging con poli-l-lisina
- Utilizzare 1 mL di una soluzione di poli-L-lisina 0,1 mg/mL per piastra di imaging per coprire l'intera superficie (vedere la Tabella dei materiali).
NOTA: Regolare la concentrazione di poli-L-lisina in base ai requisiti specifici del tipo di cella. - Scuotere delicatamente il piatto per ottenere un rivestimento uniforme della superficie di coltura.
NOTA: Garantire condizioni sterili durante tutto il processo per evitare contaminazioni. - Incubare le piastre per una notte a temperatura ambiente (RT).
- Aspirare la soluzione di poli-L-lisina in eccesso dalle piastre pipettandola.
- Risciacquare la superficie con PBS tre volte, riposando ogni volta per 5 minuti (vedi Tabella dei Materiali). Rimuovere completamente la poli-L-lisina non legata dalla piastra di imaging per prevenire l'inibizione della crescita cellulare. Sciacquare delicatamente le piastre per evitare di raschiare o danneggiare il fondo del vetro.
- Asciugare all'aria le piastre di imaging rivestite a 37 °C per almeno 3 ore.
- Utilizzare immediatamente le piastre per immagini rivestite o conservarle a 4 °C per un massimo di 2 settimane.
4. Trasfezione di cellule HepG2
NOTA: Il metodo di trasfezione HepG2 è illustrato nella Figura 5.
- Seminare le cellule HepG2 in piastre di imaging pre-rivestite 24-48 ore prima della trasfezione per assicurarsi che raggiungano il 70%-90% di confluenza.
- Scongelare il reagente di trasfezione e il plasmide che codifica il biosensore FRET su ghiaccio. Agitare accuratamente ed eseguire una breve centrifuga (ad es. 5.000 x g per 5 s) prima dell'uso (vedi Tabella dei materiali).
NOTA: Assicurarsi che il reagente di trasfezione e il plasmide siano completamente scongelati prima dell'uso.
ATTENZIONE: Evitare cicli ripetuti di congelamento-scongelamento, in quanto ciò potrebbe ridurre l'efficienza del reagente di trasfezione. - Aggiungere 8 μg di plasmide codificante il biosensore FRET con il tampone di reazione fino a un volume finale di 100 μL. Mescolare bene agitando per 5 s ad alta velocità (circa 3.000-5.000 x g).
NOTA: Aggiungere sempre il plasmide al tampone prima di aggiungere il reagente di trasfezione a base di polimero. Almeno 50 μl della soluzione devono essere costituiti dal tampone di reazione (vedi Tabella dei materiali). - Aggiungere 2,4 μl del polimero di trasfezione alla provetta contenente il DNA plasmidico diluito. Mescolare bene agitando per 15 s ad alta velocità (circa 3.000-5.000 × g).
NOTA: Utilizzare sempre 0,3 μl di polimero di trasfezione per 1 μg di DNA.
ATTENZIONE: Assicurarsi che il polimero di trasfezione sia accuratamente miscelato con il DNA plasmidico per formare complessi di nanoparticelle uniformi. - Incubare la miscela di biosensore e polimero a 37 °C per 15 minuti per consentire la formazione di complessi di nanoparticelle.
NOTA: Evitare di tenere il polimero di trasfezione in soluzione per più di 30 minuti, poiché ciò potrebbe ridurre l'efficienza di trasfezione.
ATTENZIONE: Monitorare attentamente il tempo di incubazione per evitare un'incubazione eccessiva, che può portare a una riduzione dell'efficienza di trasfezione. - Centrifugare la provetta per 5 s a 5.000 x g per raccogliere il contenuto sul fondo, quindi aggiungere tutti i 100 μl di soluzione di complesso di nanoparticelle goccia a goccia al terreno di coltura cellulare. Scuotere delicatamente il piatto avanti e indietro per mescolare.
NOTA: Assicurarsi che la soluzione del complesso di nanoparticelle venga aggiunta goccia a goccia per distribuirla uniformemente sul terreno di coltura cellulare.
ATTENZIONE: Evitare un forte dondolio, in quanto ciò potrebbe spostare le cellule o causare una distribuzione non uniforme dei complessi. - Incubare la piastra a 37 °C per 4 ore o tutta la notte.
NOTA: Il tempo di incubazione può essere regolato in base alle esigenze sperimentali, ma 4 ore sono in genere sufficienti per una trasfezione efficiente.
ATTENZIONE: Evitare l'incubazione prolungata (>16 ore), poiché ciò potrebbe ridurre la vitalità cellulare. - Rimuovere i complessi di nanoparticelle dalle cellule mediante aspirazione, sostituirli con 2 mL di terreno di coltura completo fresco e rimettere la piastra nell'incubatore a 37 °C fino al momento dell'analisi. Il picco di espressione raggiunge tipicamente le 48 ore dopo la trasfezione.
- Analizzare le cellule al microscopio a fluorescenza.
NOTA: Assicurarsi che il microscopio sia calibrato correttamente per l'imaging a fluorescenza per ottenere risultati accurati.
ATTENZIONE: Ridurre al minimo l'esposizione delle cellule alla luce intensa durante l'imaging per prevenire la fototossicità.
5. Fame delle cellule HepG2
NOTA: Dopo aver completato la fase di trasfezione, affamare le cellule prima della stimolazione con insulina e dell'imaging FRET. Ciò riduce al minimo l'attivazione della via Akt dovuta all'insulina presente nell'FBS e garantisce livelli basali costanti di attività Akt. La composizione del mezzo di innesto utilizzato in questo esperimento è descritta in (Tabella 3). BSA si presenta in polvere. Per preparare una soluzione allo 0,1% (p/v), ricostituire 0,1 g di BSA in 3 mL di DMEM, mescolando accuratamente. Sterilizzare la soluzione utilizzando un filtro da 0,45 μm e regolare il volume finale a 100 mL aggiungendo DMEM.
- Rimuovere il terreno di coltura e sciacquare le piastre di imaging con 1 PBS due volte per 5 minuti ciascuna.
NOTA: I due lavaggi PBS aiutano a rimuovere completamente il siero residuo e qualsiasi insulina o fattore di crescita che possa interferire con l'esperimento. - Aggiungere 2 mL di terreno di prova alle piastre di imaging (vedere Tabella dei materiali). Aggiungere delicatamente il terreno attorno al bordo del piatto per evitare di staccare le celle dal fondo del bicchiere. Incubare a 37 °C per 4 ore.
NOTA: L'incubazione di 4 ore è ottimale per sincronizzare il metabolismo cellulare; Tuttavia, la durata può essere estesa a seconda delle esigenze sperimentali.
6. Imaging FRET su cellule vive per cellule HepG2
NOTA: Questa sezione fornisce istruzioni per l'imaging FRET su cellule vive per monitorare le dinamiche spazio-temporali della fosforilazione di Akt in singole cellule HepG2. È essenziale ottimizzare la configurazione del microscopio, le procedure di manipolazione e le condizioni di imaging per le cellule HepG2 vive, come descritto di seguito. La configurazione del microscopio è fondamentale per ottimizzare le condizioni di imaging per l'imaging FRET. Seguire la configurazione della microscopia a scansione laser confocale (CLSM) del PC/passo dopo passo secondo le istruzioni del produttore per garantire un funzionamento stabile. La configurazione CLSM personalizzata per l'imaging FRET è mostrata in (Figura 6).
- Accendere l'interruttore remoto per alimentare il microscopio, il computer, lo scanner, il lancio laser, il piezoelettrico e la sorgente luminosa a LED per epifluorescenza. Assicurarsi che tutti i componenti siano collegati correttamente prima dell'accensione per evitare potenziali danni.
- Ruotare la chiave in posizione ON sul lancio laser e premere i pulsanti per attivare entrambi i laser necessari per FRET (linee laser a 457 nm e 514 nm).
NOTA: Assicurarsi che siano presenti i filtri e le impostazioni appropriati per un'imaging FRET ottimale. La selezione della linea laser deve essere basata sui profili di eccitazione ed emissione del biosensore. - Accendere la ciabatta per alimentare il computer e il monitor collegati al microscopio.
NOTA: Assicurarsi che tutti i collegamenti siano sicuri prima dell'accensione per evitare danni alle apparecchiature. - Accedi a Windows e avvia il software di microscopia.
- Fare clic su A1 per l'acquisizione per avviare la configurazione dell'imaging. Selezionare le configurazioni ottiche appropriate in base ai requisiti dell'esperimento FRET.
- Regolare la potenza del laser e la sensibilità del rivelatore secondo necessità per ottenere condizioni di imaging ottimali. Installare con cura l'incubatore superiore del tavolino del microscopio e fissarlo con le viti.
NOTA: Non serrare eccessivamente le viti per evitare danni all'incubatrice o al tavolino del microscopio. - Riempire il bagnomaria interno con acqua sterile bidistillata (ddH2O). Installare saldamente il riscaldatore superiore e accendere l'alimentazione per il stage riscaldatore, riscaldatore per vasca e riscaldatore per lenti (Figura 7A).
NOTA: Non riempire eccessivamente per evitare fuoriuscite nel sistema. Assicurarsi che tutti i riscaldatori funzionino correttamente per mantenere condizioni di temperatura costanti per l'imaging di cellule vive. - Utilizzare la lente a immersione in olio 40x per l'imaging (vedere la Tabella dei materiali).
NOTA: Fare riferimento al sito Web del produttore per le specifiche tecniche. - Pulire la lente dell'obiettivo con carta per lenti inumidita con etanolo al 95%. Posizionare una piccola goccia di olio da immersione sulla lente dell'obiettivo (Figura 7B). Posizionare la capsula di imaging contenente le cellule HepG2 sul tavolino del microscopio e fissarla con il supporto (Figura 7C).
NOTA: Assicurarsi che la piastra di imaging sia correttamente allineata e fissata per evitare qualsiasi movimento durante l'imaging. Evitare la pressione contro l'obiettivo per evitare danni sia all'obiettivo che al fondo di vetro del piatto. - Chiudere la camera e incubare le cellule all'interno della camera delle cellule vive per 1-2 ore per consentire loro di equilibrarsi (Figura 7D).
- Durante l'imaging time-lapse, fare una pausa a intervalli specifici e rimuovere delicatamente il terreno (Figura 7E), quindi aggiungere 1 ml di terreno appena preparato con la concentrazione di insulina data (Figura 7F, Tabella dei materiali).
NOTA: Preparare una soluzione madre di insulina da 1 mg/mL sciogliendo 1 mg di insulina in 1 mL di acido acetico da 10 mM. Filtrare la soluzione attraverso un filtro per siringa sterile da 0,2 μm e conservare le aliquote a -20 °C. - Avviare la finestra Acquisizione ND dal menu File del software di microscopia.
NOTA: La configurazione dell'acquisizione ND è mostrata in (Figura 8). - Selezionare la scheda Tempo per impostare l'intervallo, la durata e i loop desiderati per l'imaging time-lapse. Quindi, fai clic sulla scheda XY e premi il pulsante + aggiungi per includere le singole cellule HepG2 per l'imaging.
- Per aggiungere più celle, individuare le celle adatte, eseguire la scansione, regolare la messa a fuoco e bloccare la funzione di diffusione dei punti (PSF). Ripetere questo passaggio per ogni nuova cella aggiunta per l'imaging. Scansiona più cellule in posizioni diverse all'interno delle piastre di imaging per identificare le varie cellule bersaglio da includere.
- Fare clic sull'icona X rossa per deselezionare le celle. Seleziona la casella "Z" per impostare la posizione Z. Dopo aver selezionato tutti i parametri, inserisci il nome dell'esperimento nella casella "Nome file".
- Fare clic su Sfoglia per scegliere la cartella di destinazione, quindi selezionare la casella Salva su file. Premi la scheda Esegui ora per avviare l'acquisizione ND. La finestra di acquisizione ND mostrerà l'avanzamento in tempo reale dell'imaging time-lapse, incluso il tempo trascorso e rimanente.
- Registra le misurazioni di base per un massimo di 30 minuti senza stimolare le cellule con l'insulina.
- Individua le celle adatte, ingrandisci/rimpicciolisci per mettere a fuoco una singola cellula e seleziona le celle con un'elevata intensità di fluorescenza. Aggiungere le celle una alla volta, fino a un massimo di 6 celle.
NOTA: Limitare il numero di celle per evitare ritardi nell'acquisizione dell'immagine e il congelamento della finestra durante l'imaging. - Impostare l'ora e la frequenza di imaging per l'imaging time-lapse. Avviare l'imaging time-lapse.
NOTA: Assicurarsi che tutti i parametri, inclusi il tempo di esposizione e la potenza del laser, siano ottimizzati prima di iniziare la sessione di imaging. - Mettere in pausa l'imaging a intervalli regolari, rimuovere delicatamente il terreno dal piatto con fondo di vetro e aggiungere 1 ml di terreno integrato con la concentrazione di insulina appropriata.
- Riprendere l'acquisizione dell'immagine. Ripetere il passaggio 6.20 secondo necessità.
- Al termine dell'esperimento, fare clic sulla scheda Fine per chiudere. Esegui il backup e archivia le immagini acquisite in modo sicuro per l'analisi.
7. Analisi dei dati
- Correggere i dati di imaging FRET time-lapse pre-elaborati da campioni di controllo per la diafonia spettrale e l'autofluorescenza CFP, il che si traduce in valori FRET corretti e un'efficienza FRET precisa. Utilizzare il software di imaging per l'acquisizione, l'elaborazione e l'analisi delle immagini FRET, seguendo i protocolli delineati negli studi precedenti 27,28,29,30 (Figura 9).
8. Calcoli dell'efficienza FRET
- Per determinare l'efficienza FRET, acquisire sette immagini: cella (IDA(D), IDD(D), IDA(A), IAA(A), I DD, IAA e IDA). Calcola l'efficienza FRET utilizzando la seguente formula:
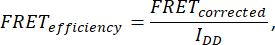
dove FRETCorrected si ottiene dall'equazione:
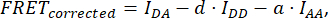
dove d e a sono definiti come:
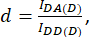
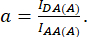
Qui IDA, IDD e IAA rappresentano immagini di cellule trasfettate con il biosensore. IDA(D) e IDD(D)) sono immagini di controllo solo donatore, e IDA(A) e IAA(A) sono immagini di controllo solo accettore.
9. Acquisizione delle immagini
- IDA : Ottenere l'immagine mediante eccitazione del donatore (434 nm) con emissione accettore (530 nm).
- IDD : Ottenere l'immagine mediante eccitazione del donatore (434 nm) con emissione del donatore (477 nm).
- IAA : Ottenere l'immagine mediante eccitazione dell'accettore (517 nm) con emissione dell'accettore (530 nm).
- IDA(D) e IDA(A) : Acquisiscono l'immagine utilizzando la stessa eccitazione ed emissione di IDA ma da cellule che esprimono rispettivamente solo il donatore e l'accettore.
- IDD(D) : Acquisire l'immagine alle stesse condizioni di IDD per il controllo solo donatore.
- IAA(A) : Ottiene l'immagine utilizzando le stesse condizioni di IAA ma è specifica per il controllo solo accettore.
10. Correzione dello sfondo
- Aprire il software di analisi delle immagini sul computer. Dalla barra dei menu in alto, accedi al menu File . Seleziona Apri o Apri file dalle opzioni a discesa.
- Passare alla directory contenente i dati dell'immagine time-lapse. Individua l'immagine time-lapse che richiede l'analisi (ad esempio, il formato nd2).
- Scegli il file e fai clic su Apri o OK per caricarlo nel software per la visualizzazione e l'analisi. Definite un'area di interesse (ROI) all'interno della cella (o utilizzate l'intera cella come ROI) e registrate il valore di grigio di ogni pixel all'interno di questa regione.
- Scegli un'area priva di celle come sfondo e calcola il suo valore medio di grigio. Crea l'immagine corretta sottraendo questa media di sfondo dal valore di grigio di ciascun pixel all'interno del ROI della cella.
11. Eliminazione del cross-through (diafonia) dei FRET
NOTA: La sovrapposizione spettrale tra l'emissione del donatore e l'eccitazione dell'accettore è illustrata nella Figura 3B, che è fondamentale per l'efficienza FRET e il processo di trasferimento dell'energia. Il bleed-through nell'imaging FRET time-lapse è una sfida significativa che deriva dalla sovrapposizione spettrale dei fluorofori donatore e accettore, che porta a misurazioni imprecise. La diafonia è intrinseca perché gli spettri dei fluorofori donatori e accettori si sovrappongono in una certa misura (Figura 3C, D). Questo problema è aggravato da fattori quali alte concentrazioni di fluorofori e configurazioni improprie dei filtri. Affrontare il problema del bleed-through è fondamentale per garantire l'affidabilità delle misure FRET.
- Fare riferimento a uno studio precedente per un metodo per mitigare gli effetti di sanguinamento9.
12. Quantificazione e analisi statistica
- Esegui analisi statistiche utilizzando un software di analisi statistica.
Risultati
Per studiare l'attivazione di Akt nelle cellule HepG2, le cellule sono state seminate su piastre di imaging pre-rivestite e trasfettate con il biosensore pEevee-iAkt-NES basato su FRET (Figura 2A), progettato per consentire il monitoraggio in tempo reale della fosforilazione di Akt. Dopo la trasfezione, le cellule sono state sottoposte a fame sierica per 4 ore in un terreno privo di siero per sincronizzare il loro stato metabolico e ridurre al minimo la segn...
Discussione
Il protocollo per l'imaging FRET su cellule vive per monitorare la fosforilazione di Akt nelle cellule HepG2 prevede diversi passaggi chiave per garantire risultati affidabili e riproducibili. La prima fase critica è la coltura cellulare, che include la manutenzione ordinaria delle cellule, il rivestimento delle piastre di imaging e la semina cellulare. Un rivestimento adeguato è essenziale per l'adesione delle cellule durante gli esperimenti di imaging time-lapse, in quanto garantisce...
Divulgazioni
Gli autori dichiarano di non avere interessi concorrenti.
Riconoscimenti
Questo lavoro è stato in parte sostenuto dalla Natural Science Foundation di Shenzhen (JCYJ20240813113606009), dalla Shenzhen-Hong Kong Cooperation Zone for Technology and Innovation (HZQB-KCZYB-2020056), dalla National Natural Science Foundation of China (32070681), dal National Key R&D Program of China (2019YFA0906002) e dallo Shenzhen Peacock Plan (KQTD2016053117035204).
Materiali
| Name | Company | Catalog Number | Comments |
| 0.25% trypsin-EDTA | Gibco | Cat#25200-056 | Use ice-cold PBS for cell wash |
| 15 mm glass bottom cell culture dish | NEST | Cat#801001 | |
| 2 mL Nalgene cryogenic vials | Thermo Scientific | Cat#5012-0020 | |
| 5 mL Stripette Serological Pipets | Corning | Cat#4487 | |
| 95% Ethanol | Kermel | Cat#C028005 | |
| A1 HD25/A1R HD25 confocal microscope | Nikon | https://www.nikon.com/ | Magnification: 40×, Numerical Aperture (NA): 1.30, Pixel Dwell Time: 2.4 ms, Pixel Size: 1024 |
| Ampicillin | Sigma-Aldrich | Cat#A9393 | |
| Bovine serum albumin (BSA) | VWR Life Science | Cat#N208-10g | |
| Corning 25 cm2 rectangular culture flasks | Corning | Cat#430639 | |
| Countess 3 automated cell counter | Thermo Scientific | http://www.thermofisher.com/#AMQAX2000 | |
| Countess cell counting chamber slides | Thermo Scientific | http://www.thermofisher.com/#C10228 | |
| Digital vortex mixers | Thermo Scientific | https://www.thermofisher.com/ | |
| Dimethyl sulfoxide | Sigma-Aldrich | Cat#D2650 | |
| Eppendorf Safe-Lock Tubes 1.5 mL | Eppendorf | Cat#022363204 | |
| EZ-PCR mycoplasma detection kit | Biological Industries | Cat# 20-700-20 | |
| Fetal Bovine Serum, qualified, Australia | Gibco | Cat#10099141 | |
| GlutaMAX Supplement | Gibco | Cat#35050061 | |
| GraphPad Prism 9 | GraphPad Software | https://www.graphpad.com/ | |
| HepG2 | National Collection of Authenticated Cell Cultures | #CSTR:19375.09.3101HUMSCSP510 http://www.cellbank.org.cn/ | |
| Immersion Oil Type 37 | Cargille Laboratories | Cat #16237 | |
| Insulin | Sigma-Aldrich | Cat#I5500-50MG | Warm to 37 °C before use |
| LB Broth (1x) | Invitrogen | Cat#10855001 | |
| Minimum Essential Medium (MEM) | Gibco | Cat#11095080 | Warm to 37 °C before use |
| mySPIN 12 mini centrifuge | Thermo Scientific | https://www.thermofisher.com/ | |
| NanoDrop One | Thermo Scientific | https://www.thermofisher.com | |
| Nikon Plan Fluor 40×/1.30 Oil Lens | Nikon | https://www.nikon.com/ | |
| NIS-Elements-AR | Nikon | https://www.nikon.com/ | |
| Non-Essential Amino Acids (NEAA) (100x) | Gibco | Cat#11140050 | |
| One Shot LB Agar Plates | Invitrogen | Cat#A55802 | |
| One Shot Stbl3 chemically competent E. coli | Invitrogen | Cat#C737303 | |
| Parafilm | PARAFILM | Cat#B8R05606 | |
| PBS (phosphate buffered saline) | Gibco | Cat#10010023 | |
| pEevee-iAkt-NES (7,033 bp) | Miura et al31 | https://benchling.com/s/seq-q46zFYCfl0swLAun0t28/edit | |
| Penicillin-streptomycin | Gibco | Cat#15070063 | |
| Plasmocin prophylactic | InvivoGen | Cat#ant-mpp | |
| Poly-L-Lysine Hydrobromide | Sigma-Aldrich | Cat#P4832 | |
| Precision general purpose baths | Thermo Scientific | https://www.thermofisher.com/ | |
| QIAprep spin miniprep kit | QIAGEN | Cat#27106 | |
| SnapGene | SnapGene by Dotmatics | https://www.snapgene.com | |
| Sodium Pyruvate (100 mM) | Gibco | Cat#11360070 | |
| Syringe filter unit, 0.22 μm | Millipore | Cat#SLGP033RS | |
| Tokai Hit stage top incubator | TOKAI HIT | https://www.tokaihit-livecell.com/stagetopincubator | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | Cat#10977015 | |
| Xfect Transfection Reagent | Takara Bio | Cat#631317 |
Riferimenti
- GBD 2021 Diabetes Collaborators. Global, regional, and national burden of diabetes from 1990 to 2021, with projections of prevalence to 2050: a systematic analysis for the Global Burden of Disease Study 2021. Lancet. 402 (10397), 203-234 (2023).
- Li, M., et al. Trends in insulin resistance: insights into mechanisms and therapeutic strategy. Signal Transduct Target Ther. 7 (1), 216 (2022).
- Poloz, Y., Stambolic, V. Obesity and cancer, a case for insulin signaling. Cell Death Dis. 6 (12), e2037 (2015).
- Arcidiacono, B., et al. Insulin resistance and cancer risk: an overview of the pathogenetic mechanisms. Exp Diabetes Res. 2012, 789174 (2012).
- Godsland, I. F. Insulin resistance and hyperinsulinaemia in the development and progression of cancer. Clin Sci (Lond). 118 (5), 315-332 (2009).
- Tsugane, S., Inoue, M. Insulin resistance and cancer: epidemiological evidence. Cancer Sci. 101 (5), 1073-1079 (2010).
- Yudhani, R. D., et al. In vitro insulin resistance model: A recent update. J Obes. 2023, 1964732 (2023).
- Akhtar, J., et al. Bistable insulin response: The win-win solution for glycemic control. iScience. 25 (12), 105561 (2022).
- Akhtar, J., Imran, M., Wang, G. Protocol for live-cell Forster resonance energy transfer imaging to reveal the bistable insulin response of single C2C12-derived myotubes. STAR Protoc. 5 (2), 103109 (2024).
- Kamino, K., et al. Optimal inference of molecular interaction dynamics in FRET microscopy. Proc Natl Acad Sci U S A. 120 (15), e2211807120 (2023).
- Kraft, A. E., Nikolaev, V. O. FRET microscopy for real-time visualization of second messengers in living cells. Methods Mol Biol. 1563, 85-90 (2017).
- Veeriah, S., et al. High-throughput time-resolved FRET reveals Akt/PKB activation as a poor prognostic marker in breast cancer. Cancer Res. 74 (18), 4983-4995 (2014).
- Conway, J. R. W., et al. Monitoring AKT activity and targeting in live tissue and disease contexts using a real-time Akt-FRET biosensor mouse. Sci Adv. 9 (17), eadf9063 (2023).
- Chandris, P., Giannouli, C. C., Panayotou, G. Imaging approaches for the study of metabolism in real time using genetically encoded reporters. Front Cell Dev Biol. 9, 725114 (2021).
- Yang, J., et al. Longitudinal FRET imaging of glucose and lactate dynamics and response to therapy in breast cancer cells. Mol Imaging Biol. 24 (1), 144-155 (2022).
- Mandrou, E., et al. A reliable system for quantitative G-protein activation imaging in cancer cells. Cells. 13 (13), 1114 (2024).
- Fang, C., Huang, Y., Zhao, Y. Review of FRET biosensing and its application in biomolecular detection. Am J Transl Res. 15 (2), 694-709 (2023).
- Miller, J. N. Fluorescence energy transfer methods in bioanalysis. Analyst. 130 (3), 265-270 (2005).
- Verveer, P. J., Rocks, O., Harpur, A. G., Bastiaens, P. I. Imaging protein interactions by FRET microscopy: FRET measurements by acceptor photobleaching. CSH Protoc. 2006 (6), (2006).
- Vu, C. Q., Arai, S. Quantitative imaging of genetically encoded fluorescence lifetime biosensors. Biosensors (Basel). 13 (10), 939 (2023).
- Wang, G. A more holistic view of the logarithmic dose-response curve offers greater insights into insulin response. J Biol Chem. 301 (1), 108037 (2025).
- Wang, G. Body mass dynamics is determined by the metabolic Ohm's law and adipocyte-autonomous fat mass homeostasis. iScience. 23 (6), 101176 (2020).
- Wang, G. Raison d'être of insulin resistance: the adjustable threshold hypothesis. J R Soc Interface. 11 (101), 20140892 (2014).
- Wang, G. Optimal homeostasis necessitates bistable control. J R Soc Interface. 9, 2723 (2012).
- Heim, R., Tsien, R. Y. Engineering green fluorescent protein for improved brightness, longer wavelengths and fluorescence resonance energy transfer. Curr Biol. 6 (2), 178-182 (1996).
- Nguyen, A. W., Daugherty, P. S. Evolutionary optimization of fluorescent proteins for intracellular FRET. Nat Biotechnol. 23 (3), 355-360 (2005).
- Broussard, J. A., Rappaz, B., Webb, D. J., Brown, C. M. Fluorescence resonance energy transfer microscopy as demonstrated by measuring the activation of the serine/threonine kinase Akt. Nat Protoc. 8 (2), 265-281 (2013).
- Gordon, G. W., Berry, G., Liang, X. H., Levine, B., Herman, B. Quantitative fluorescence resonance energy transfer measurements using fluorescence microscopy. Biophys J. 74 (5), 2702-2713 (1998).
- Tron, L., et al. Flow cytometric measurement of fluorescence resonance energy transfer on cell surfaces. Quantitative evaluation of the transfer efficiency on a cell-by-cell basis. Biophys J. 45 (5), 939-946 (1984).
- Youvan, D. C., et al. Calibration of fluorescence resonance energy transfer in microscopy. , (2002).
- Miura, H., Matsuda, M., Aoki, K. Development of a FRET biosensor with high specificity for Akt. Cell Struct Funct. 39 (1), 9-20 (2014).
- Liberio, M. S., Sadowski, M. C., Soekmadji, C., Davis, R. A., Nelson, C. C. Differential effects of tissue culture coating substrates on prostate cancer cell adherence, morphology and behavior. PLoS One. 9 (11), e112122 (2014).
- Rani, K., Sengupta, S. Multi-stimuli programmable FRET based RGB absorbing antennae towards ratiometric temperature, pH and multiple metal ion sensing. Chem Sci. 12 (47), 15533-15542 (2021).
- Betolngar, D. B., et al. pH sensitivity of FRET reporters based on cyan and yellow fluorescent proteins. Anal Bioanal Chem. 407 (14), 4183-4193 (2015).
- Salonikidis, P. S., et al. An ion-insensitive cAMP biosensor for long term quantitative ratiometric fluorescence resonance energy transfer (FRET) measurements under variable physiological conditions. J Biol Chem. 286 (26), 23419-23431 (2011).
- Youvan, D. C., et al. Calibration of fluorescence resonance energy transfer in microscopy using genetically engineered GFP derivatives on nickel chelating beads. Biotechnol Alia. 3, 1-18 (1997).
- Menaesse, A., et al. Simplified instrument calibration for wide-field fluorescence resonance energy transfer (FRET) measured by the sensitized emission method. Cytometry A. 99 (4), 407-416 (2021).
- Batta, A., Hajdu, T., Nagy, P. Improved estimation of the ratio of detection efficiencies of excited acceptors and donors for FRET measurements. Cytometry A. 103 (7), 563-574 (2023).
- Coullomb, A., et al. QuanTI-FRET: a framework for quantitative FRET measurements in living cells. Sci Rep. 10 (1), 6504 (2020).
- Hoppe, A., Christensen, K., Swanson, J. A. Fluorescence resonance energy transfer-based stoichiometry in living cells. Biophys J. 83 (6), 3652-3664 (2002).
- Zal, T., Gascoigne, N. R. Photobleaching-corrected FRET efficiency imaging of live cells. Biophys J. 86 (6), 3923-3939 (2004).
- Hochreiter, B., Kunze, M., Moser, B., Schmid, J. A. Advanced FRET normalization allows quantitative analysis of protein interactions including stoichiometries and relative affinities in living cells. Sci Rep. 9 (1), 8233 (2019).
- Liu, H., Zhang, F., Mishra, S. K., Zhou, S., Zheng, J. Knowledge-guided fuzzy logic modeling to infer cellular signaling networks from proteomic data. Sci Rep. 6, 35652 (2016).
- Garrido-Rodriguez, M., Zirngibl, K., Ivanova, O., Lobentanzer, S., Saez-Rodriguez, J. Integrating knowledge and omics to decipher mechanisms via large-scale models of signaling networks. Mol Syst Biol. 18 (7), e11036 (2022).
- Greenwald, E. C., Polanowska-Grabowska, R. K., Saucerman, J. J. Integrating fluorescent biosensor data using computational models. Methods Mol Biol. 1071, 227-248 (2014).
- Zhao, Z., Xia, J. Computational Approaches for Modeling Signal Transduction Networks. Encyclopedia of Bioinformatics and Computational Biology. , (2019).
- Wang, G., Krueger, G. R. Computational analysis of mTOR signaling pathway: bifurcation, carcinogenesis, and drug discovery. Anticancer Res. 30 (7), 2683-2688 (2010).
- Wang, G. Singularity analysis of the AKT signaling pathway reveals connections between cancer and metabolic diseases. Phys Biol. 7 (4), 046015 (2010).
- Chedere, A., Hari, K., Kumar, S., Rangarajan, A., Jolly, M. K. Multi-stability and consequent phenotypic plasticity in AMPK-Akt double negative feedback loop in cancer cells. J Clin Med. 10 (3), 472 (2021).
- Mosca, E., et al. Computational modeling of the metabolic States regulated by the kinase akt. Front Physiol. 3, 418 (2012).
- Liao, J., Madahar, V., Dang, R., Jiang, L. Quantitative FRET (qFRET) technology for the determination of protein-protein interaction affinity in solution. Molecules. 26 (21), 6339 (2021).
- Verma, A. K., Noumani, A., Yadav, A. K., Solanki, P. R. FRET based biosensor: Principle applications recent advances and challenges. Diagnostics (Basel). 13 (8), 1375 (2023).
- Mattheisen, J. M., et al. Application of bioluminescence resonance energy transfer to quantitate cell-surface expression of membrane proteins. Anal Biochem. 684, 115361 (2024).
- Lionetti, M. C., La Porta, C. A. M. FLIM-FRET investigation of heterogeneous huntingtin aggregation in HeLa cells. Methods Mol Biol. 2551, 595-604 (2023).
- Petutschnig, E. K., Pierdzig, L., Mittendorf, J., Niebisch, J. M., Lipka, V. A novel fluorescent protein pair facilitates FLIM-FRET analysis of plant immune receptor interaction under native conditions. J Exp Bot. 75 (3), 746-759 (2024).
- Sprenger, J. U., Perera, R. K., Gotz, K. R., Nikolaev, V. O. FRET microscopy for real-time monitoring of signaling events in live cells using unimolecular biosensors. J Vis Exp. 66, e4081 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon