JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
HepG2 세포에서 대사적으로 조절되는 Akt 활성화 역학의 Live-Cell Förster Resonance Energy Transfer Imaging
요약
여기에서는 살아있는 HepG2 세포에서 Akt 활성화 및 인산화의 시공간 역학을 정량화하기 위한 프로토콜을 제시합니다. Förster 공명 에너지 전달(FRET) 이미징은 암세포의 인슐린 신호 전달 경로 및 대사 조절에 대한 귀중한 통찰력을 제공하는 강력한 도구입니다.
초록
대사적으로 조절되는 Akt 활성화는 인슐린 신호 전달 캐스케이드의 중요한 노드이며 당뇨병과 암의 관계에 대한 귀중한 통찰력을 제공합니다. HepG2 세포에서 Akt 활성을 정확하게 정량화하기 위해 유전적으로 암호화된 Akt 특이적 바이오센서와 함께 Förster Resonance Energy Transfer(FRET)를 활용하여 강력하고 재현 가능한 프로토콜을 개발했습니다. 이 프로토콜은 FRET 기반 바이오센서를 발현하기 위한 HepG2 세포의 세포 배양, 이미징 접시 준비 및 transfection에 대한 자세한 단계를 설명하고 레이저 스캐닝 컨포칼 현미경 하드웨어 및 소프트웨어 구성에 대한 구체적인 지침을 제공합니다. 그 결과, HepG2 세포에서 인슐린 신호전달의 독특한 패턴을 보여주었으며, 이는 정의된 스위치 온 임계값을 가지고 있지만 스위치 오프 임계값은 없는 구성적 Akt 활성화를 특징으로 하는 비가역적 스위치를 나타냅니다. 대조적으로, myotubes는 가역 스위치를 표시합니다. HepG2 세포의 지속적인 Akt 활성화는 간 세포의 인슐린 저항성 및 대사 조절 장애의 기저에 있는 메커니즘을 시사하며, 대사 장애 및 암의 진행을 이해하는 데 더 넓은 의미를 갖습니다. 이 프로토콜은 다양한 질병 맥락에서 Akt 관련 신호 전달 경로 및 세포 행동을 탐색하기 위한 귀중한 프레임워크를 제공합니다.
서문
진성 당뇨병은 인슐린 저항성과 포도당 항상성 손상1을 특징으로 하는 전 세계적으로 주요 건강 문제를 제기합니다. 인슐린은 포도당 대사, 세포 성장 및 생존에 중추적인 역할을 하기 때문에 인슐린 신호 경로에 대한 포괄적인 이해는 이 질병의 병태 생리학을 밝히는 데 매우 중요합니다2. 수많은 연구에서 인슐린 신호전달이 다양한 암에 상당한 영향을 미치며, 인슐린 저항성이 종양 진행 및 환자의 나쁜 예후와 관련이 있음을 입증했습니다 3,4,5,6. 일반적으로 사용되는 간세포암 세포주인 HepG2 세포는 인슐린 저항성 및 대사 조절 장애와 암 발병 간의 상호 작용을 연구하는 데 유용한 모델 역할을 합니다7. 전통적으로 연구자들은 인슐린 반응을 등급이 매겨진 것으로 간주해 왔습니다. 그러나 최근 연구에 따르면 개별 세포는 쌍안정 반응을 보일 수 있으며, 특정 인슐린 농도 역치에서 발생하는 무반응과 완전 반응 사이의 두드러진 전이를 보일 수 있습니다 8,9.
Förster 공명 에너지 전달(FRET) 이미징은 살아있는 세포에서 생체 분자의 시공간 분포를 연구하기 위한 강력한 도구입니다10. FRET는 분자 역학에서 정보를 추출하여 Akt 활성화와 같은 과정에 대한 통찰력을 실시간으로 제공하여 살아있는 세포를 연구하는 데 매우 유용한 기술입니다11,12. 이 이미징 방법은 세포 역학을 연구하는 데 필수적인 것으로 입증되었으며, 특히 정확한 분자 상호 작용이 중요한 대사 질환 및 암에서 필수적입니다13. FRET는 또한 분자 상호 작용의 실시간 모니터링을 가능하게 하여 인슐린 저항성 및 종양 진행과 같은 메커니즘을 밝힐 수 있습니다14,15. FRET 바이오센서는 종양 미세환경, 약물 내성 및 대사 장애를 연구하기 위한 암 연구에서 매우 중요하다16. 민감 방출(SE), 수용체 표백(AB), 형광 수명 이미징 현미경(FLIM) 및 분광학과 같은 FRET 검출 방법은 각각 분자 상호 작용을 정량화하는 데 뚜렷한 이점을 제공합니다17. SE는 공여체와 수용체 형광단 사이의 에너지 전달을 측정하여 상호 작용하는 생체 분자의 근접성과 상관관계가 있는 방출 스펙트럼의 측정 가능한 변화를 초래합니다18. AB는 수용체 형광단의 선택적 광표백을 사용하고 공여체 형광의 변화를 추적하여 연구자들이 상호 작용 역학 및 거리를 평가할 수 있도록 합니다19. FLIM은 FRET 효율의 직접적인 영향을 받는 공여 형광단의 형광 감쇠율을 평가하여 분자 상호 작용의 정확한 나노 단위 측정을 제공합니다20.
FRET 기술을 사용하여 최근 C2C12 유래 근관에서 쌍안정 인슐린 반응을 입증했습니다 8,9,21,22,23,24. 우리가 발견한 바와 같이 Akt 활성화에 대한 뚜렷한 스위치 켜기 및 끄기 임계값은 등급이 매겨진 전신 인슐린 용량 반응이 인슐린 자극에서 시작하는 세포 내 신호 캐스케이드의 복잡성을 무시하는 것으로 시사하며, 이는 단일 세포 수준에서 전부 또는 전무 반응으로 절정에 이릅니다 21,22,23,24. 다른 세포 유형에서 쌍안정성의 존재를 테스트하기 위해 인슐린으로 HepG2 세포를 자극하고 단일 세포 FRET 이미징을 사용하여 반응을 기록했습니다. 다양한 인슐린 농도로 HepG2 세포를 자극하고 Akt 바이오센서를 사용하여 단일 세포 수준에서 Akt 활성을 모니터링했습니다. Akt 바이오센서는 공여 형광단으로 강화된 청록색 형광 단백질(ECFP)25과 수용체 형광단으로 황색 형광 단백질(YPet)26의 가장 밝은 변종으로 구성되며, 펩타이드 서열 SGRPRTTFADSCKP를 포함하는 Eevee 링커로 연결됩니다. 이 펩타이드는 인간 글리코겐 합성효소 키나아제 3β(GSK3β)에서 최적화된 인산화된 Akt(pAkt)의 기질로 작용합니다. 인산화되지 않은 상태에서 공여체와 수용체 형광단 사이의 공간적 분리는 Förster 반경을 초과하여 에너지 전달을 억제합니다. 인슐린 자극 시, Akt 인산화가 일어나 SGRPRTTTFADSCKP의 인산화로 이어집니다. 이 과정은 공여체와 수용체를 푀르스터 반경 내로 가져오는 구조적 변화를 유도하여 FRET27을 가능하게 한다. 결과적으로, FRET 신호 강도는 인산화된 Akt 분자의 양과 상관관계가 있으며 인슐린 매개 세포 반응의 실시간 정량화를 가능하게 합니다.
C2C12 유래 근관에서 인슐린 신호전달을 연구하기 위해 처음 개발된 이 프로토콜은 HepG2 세포에 성공적으로 적용되었으며 다양한 하드웨어 및 소프트웨어 플랫폼에서 활용되어 적용 가능성, 적응성 및 다양성을 입증했습니다. HepG2 세포는 구성적 Akt 활성을 나타내므로 간 특이적 인슐린 신호 전달 및 대사 과정을 연구하기 위한 이상적인 체외 모델입니다. 프로토콜의 주요 기능은 프로토콜 섹션에서 단계별로 설명합니다.
프로토콜
단일 HepG2 세포에서 Akt 인산화를 모니터링하기 위한 FRET 라이브 셀 이미징과 관련된 실험 단계의 개요가 그림 1에 나와 있습니다.
1. 플라스미드 획득, 증식 및 정제
참고: 이 섹션에서는 단일 세포 FRET 분석에 필요한 플라스미드를 획득, 증폭 및 정제하기 위한 필수 단계를 간략하게 설명합니다.
- pEevee-iAkt-NES-YPet 플라스미드를 활용합니다(그림 2A).
참고: 플라스미드는 일본 NIBB(National Institute for Basic Biology)의 Kazuhiro Aoki 교수가 아낌없이 제공했으며, Akt 모니터링에 사용되는 FRET 바이오센서와 해당 대조군에 대한 플라스미드 맵은 그림 2에 나와 있습니다. pEevee-iAkt-NES-ECFP (공여체; 그림 2B) 및 pEevee-iAkt-NES-Ypet (수용체; 그림 2C) 플라스미드는 FRET 실험9 동안 보정 대조군으로 사용됩니다.
참고: 그림 3 은 분자내 FRET 바이오센서의 구성과 메커니즘을 보여줍니다. - 플라스미드를 증식시키려면 화학적으로 적극적인 대장균 세포를 사용하여 박테리아 형질전환을 수행합니다( Table of Materials(재료표 참조). 형질전환된 세포를 100μg/mL 암피실린이 함유된 Luria-Bertani(LB) 한천에 플레이트화하고 37°C에서 하룻밤 동안 배양합니다. 다음날, 암피실린 내성 콜로니를 선택하고 플라스미드 증폭을 위해 100μg/mL 암피실린이 보충된 LB 배지에서 배양합니다.
- 플라스미드 DNA를 정제하려면 시판되는 플라스미드 DNA 정제 키트( 재료 표 참조)를 사용하여 높은 순도와 수율을 얻을 수 있습니다. 분광 광도계를 사용하여 흡광도 비율(A260/A280 및 A260/A230)을 측정하여 DNA 품질을 평가합니다( 재료 표 참조). 1.8-2.0 및 2.0-2.2 사이의 비율이 최적으로 간주됩니다. 아가로스 겔 전기영동을 통해 플라스미드 무결성을 검증하여 transfection에 적합한지 확인합니다.
2. 세포 배양 절차
참고: 멸균 환경을 유지하고 오염을 방지하기 위해 층류 후드 내에서 모든 세포 배양 절차를 수행하십시오. HepG2 세포 배양 워크플로우는 그림 4에 나와 있습니다. HepG2 세포를 위한 완전한 배지는 최소 필수 배지(MEM), 10% 소 태아 혈청(FBS), 1% 비필수 아미노산(NEAA), 1mM 피루브산나트륨, 2mM L-글루타민 보충제, 100U/mL 페니실린-스트렙토마이신 및 2.5μg/mL 항생제-항진균 용액으로 구성됩니다( 표 1 재료 표 참조).
- 완전히 해동될 때까지 바이알을 37°C 써모믹서 또는 수조에 넣어 동결된 HepG2 세포를 빠르게 해동합니다.
- 해동된 세포를 10mL의 완전 성장 배지가 들어 있는 15mL 원추형 튜브로 옮깁니다( 재료 표 참조).
알림: 세포에 대한 열 충격을 최소화하기 위해 사용하기 전에 전체 성장 배지를 37°C로 예열하십시오. - 200 x g 에서 튜브를 5분 동안 원심분리하여 세포를 펠릿화합니다.
- 세포 펠릿을 방해하지 않도록 넓은 구멍의 피펫 팁을 사용하여 상등액을 조심스럽게 흡입합니다.
- 펠릿을 10mL의 새로운 완전 성장 배지에 재현탁시킵니다.
- 세포 현탁액을 75cm² 조직 배양 플라스크로 옮깁니다.
- 37 ° C의 가습 된 분위기에서 5 % CO2로 플라스크를 배양합니다.
참고: 다음 24-48시간 동안 세포를 관찰하여 부착을 확인하고 회복을 평가합니다. 배지를 교체하기 전에 적절한 부착을 보장하기 위해 최소 4시간 동안 세포를 방해하지 마십시오. 처음 4시간 동안은 세포 부착을 방해할 수 있으므로 인큐베이터를 자주 열지 마십시오. 제도적인 생물 안전 프로토콜을 따르고 적절한 개인 보호 장비(PPE)를 사용하여 모든 세포 배양 절차 중에 안전한 작업 환경을 유지하십시오. 최적의 성장 조건을 유지하고 세포 생존력과 성장 잠재력에 영향을 줄 수 있는 과밀을 방지하기 위해 70%-80% 합류점에 도달하는 HepG2 세포를 하위 배양합니다. - 계대배양을 위해 배지를 흡인하고 5mL의 인산염 완충 식염수(PBS)로 세포를 한 번 헹굽니다.
- 세포 단층을 덮기 위해 0.25% 트립신 1mL를 추가합니다( 재료 표 참조).
알림: 최적의 활동을 위해 트립신이 37°C로 예열되었는지 확인하십시오. 세포를 과도하게 트립신화하면 생존력이 감소할 수 있으므로 사용하지 마십시오. 현미경으로 세포를 모니터링하여 박리를 확인합니다. - 37°C에서 약 5분 동안 배양합니다.
- 세포가 분리되면 트립신을 중화하고 피펫팅으로 세포를 수집하기 위해 2mL의 완전한 배지를 추가합니다.
- 세포 현탁액을 부드럽게 피펫팅하여 덩어리를 분해하고 단일 세포 현탁액을 달성합니다.
- 각각의 새 플라스크에 3mL의 완전한 배지를 추가한 다음 각 플라스크에 1:2의 분할 비율로 세포를 옮깁니다.
참고: 초기 계대의 경우 1:2의 희석으로 세포를 분할합니다. 4-5절 이후에는 1:4 또는 1:5의 희석을 적절하게 수행할 수 있습니다. - 37 ° C에서 5 % CO2로 가습 된 분위기에서 세포를 배양합니다.
참고: 24시간 후에 세포를 확인하여 부착을 확인하고 회복을 평가합니다. - 일상적인 배양의 경우 2-3일마다 또는 pH 지시약이 분홍색에서 노란색으로 변경되어 산성화를 나타내는 경우 더 빨리 배양 배지를 교체하십시오.
알림: 배지가 너무 산성화되면 세포에 해를 끼칠 수 있으므로 허용하지 마십시오. - 세포 건강을 보장하기 위해 현미경으로 정기적으로 세포 형태를 확인하십시오.
참고: 단기 보관의 경우 HepG2 세포를 -80°C에서 동결합니다. 장기 보관을 위해 액체 질소에 보관하십시오. 이 실험에 사용된 동결 매체의 조성은 (표 2)에 요약되어 있습니다.
3. poly-l-lysine를 가진 코팅 화상 진찰 접시
- 이미징 접시당 1mL의 0.1mg/mL Poly-L-Lysine 용액을 사용하여 전체 표면을 덮습니다( 재료 표 참조).
참고: 특정 세포 유형 요구 사항에 따라 Poly-L-Lysine 농도를 조정합니다. - 접시를 부드럽게 흔들어 배양 표면의 균일한 코팅을 얻습니다.
참고: 오염을 방지하기 위해 공정 전반에 걸쳐 멸균 상태를 확인하십시오. - 접시를 실온(RT)에서 밤새 배양합니다.
- 피펫팅을 통해 접시에서 과도한 Poly-L-Lysine 용액을 흡입합니다.
- PBS로 표면을 세 번 헹구고 매번 5분 동안 그대로 둡니다( 재료 표 참조). 세포 성장 억제를 방지하기 위해 이미징 접시에서 결합되지 않은 Poly-L-Lysine을 완전히 제거합니다. 유리 바닥이 긁히거나 손상되지 않도록 접시를 부드럽게 헹굽니다.
- 코팅된 이미징 접시를 37°C에서 최소 3시간 동안 자연 건조합니다.
- 코팅된 이미징 접시를 즉시 사용하거나 4°C에서 최대 2주 동안 보관하십시오.
4. HepG2 세포의 형질주입(Transfection)
참고: HepG2 transfection 방법은 그림 5에 설명되어 있습니다.
- 70%-90% confluence에 도달하도록 transfection 24-48시간 전에 사전 코팅된 이미징 접시에 HepG2 세포를 시드합니다.
- 얼음 위에서 FRET 바이오센서를 인코딩하는 transfection 시약과 플라스미드를 해동합니다. 사용하기 전에 철저히 소용돌이치고 짧은 회전(예: 5,000초 동안 5 x g)을 수행합니다( 재료 표 참조).
참고: 사용하기 전에 transfection 시약과 플라스미드가 완전히 해동되었는지 확인하십시오.
주의: 동결-해동 주기가 반복되면 transfection 시약의 효율성이 감소할 수 있으므로 피하십시오. - 반응 완충액과 함께 FRET 바이오센서를 인코딩하는 8μg의 플라스미드를 100μL의 최종 부피에 추가합니다. 고속(약 3,000-5,000 x g)으로 5초 동안 볼텍싱하여 잘 혼합합니다.
참고: 폴리머 기반 transfection 시약을 추가하기 전에 항상 buffer에 plasmid를 추가하십시오. 용액의 최소 50μL는 반응 완충액으로 구성되어야 합니다( 재료 표 참조). - 2.4 μL의 transfection polymer를 희석된 plasmid DNA가 들어 있는 튜브에 추가합니다. 고속으로 15초 동안 볼텍싱하여 잘 섞습니다(약 3,000-5,000 × g).
참고: 항상 DNA 1μg당 0.3μL의 트랜스펙션 폴리머를 사용하십시오.
주의: 형질 주입 중합체가 플라스미드 DNA와 완전히 혼합되어 균일한 나노 입자 복합체를 형성하는지 확인합니다. - 바이오센서와 폴리머 혼합물을 37°C에서 15분 동안 배양하여 나노 입자 복합체가 형성되도록 합니다.
참고: transfection 폴리머를 용액에 30분 이상 보관하면 transfection 효율이 저하될 수 있으므로 사용하지 마십시오.
주의: 인큐베이션 시간을 주의 깊게 모니터링하여 transfection 효율 감소로 이어질 수 있는 과도한 인큐베이션을 방지하십시오. - 튜브를 5,000 x g에서 5초 동안 회전시켜 바닥에 내용물을 수집한 다음 전체 100μL의 나노 입자 복합 용액을 세포 배양 배지에 적가합니다. 접시를 앞뒤로 부드럽게 흔들어 섞습니다.
참고: 나노 입자 복합 용액을 적가적으로 첨가하여 세포 배양 배지 전체에 고르게 분포시킵니다.
주의 : 격렬한 흔들림은 세포를 밀어내거나 복합체의 고르지 않은 분포를 유발할 수 있으므로 피하십시오. - 플레이트를 37 ° C에서 4 시간 동안 하룻밤 동안 배양합니다.
참고: 배양 시간은 실험 요구 사항에 따라 조정할 수 있지만 효율적인 transfection을 위해서는 일반적으로 4시간이 충분합니다.
주의: 장시간의 배양(>16시간)은 세포 생존력을 감소시킬 수 있으므로 피하십시오. - 흡인에 의해 세포에서 나노 입자 복합체를 제거하고, 2mL의 새로운 완전 성장 배지로 교체하고, 분석 시점까지 플레이트를 37°C 인큐베이터에 다시 넣습니다. 피크 발현은 일반적으로 transfection 후 48시간에 이릅니다.
- 형광 현미경 검사로 세포를 분석합니다.
알림: 정확한 결과를 얻으려면 형광 이미징을 위해 현미경이 적절하게 보정되었는지 확인하십시오.
주의: 광독성을 방지하기 위해 이미징 중 세포가 강렬한 빛에 노출되는 것을 최소화하십시오.
5. HepG2 세포의 결핍
참고: transfection 단계를 완료한 후 인슐린 자극 및 FRET 이미징 전에 세포를 혈청 결핍시킵니다. 이는 FBS에 존재하는 인슐린으로 인한 Akt 경로 활성화를 최소화하고 Akt 활동의 일관된 기준선 수준을 보장합니다. 본 실험에 사용된 기아 매체의 조성은 (표 3)에 기재되어 있다. BSA는 분말 형태로 제공됩니다. 0.1%(w/v) 용액을 준비하려면 0.1g의 BSA를 DMEM 3mL에 재구성하고 완전히 혼합합니다. 0.45μm 필터를 사용하여 용액을 멸균하고 DMEM을 추가하여 최종 부피를 100mL로 조정합니다.
- 배양 배지를 제거하고 이미징 접시를 1x PBS로 각각 5분씩 두 번 헹굽니다.
참고: 두 번의 PBS 세척은 실험을 방해할 수 있는 잔류 혈청과 인슐린 또는 성장 인자를 완전히 제거하는 데 도움이 됩니다. - 이미징 접시에 2mL의 기아 배지를 추가합니다( 재료 표 참조). 유리 바닥에서 세포가 빠지지 않도록 접시 가장자리 주위에 매체를 부드럽게 추가하십시오. 37 ° C에서 4 시간 동안 배양하십시오.
참고: 4-h 인큐베이션은 세포 대사를 동기화하는 데 최적입니다. 그러나 실험 요구 사항에 따라 기간을 연장할 수 있습니다.
6. HepG2 세포에 대한 FRET 라이브 셀 이미징
참고: 이 섹션에서는 단일 HepG2 세포에서 Akt 인산화의 시공간 역학을 모니터링하기 위한 FRET 라이브 셀 이미징에 대한 지침을 제공합니다. 아래에 자세히 설명된 바와 같이 살아있는 HepG2 세포에 대한 현미경 설정, 취급 절차 및 이미징 조건을 최적화하는 것이 필수적입니다. 현미경 설정은 FRET 이미징을 위한 이미징 조건을 최적화하는 데 매우 중요합니다. 안정적인 작동을 위해 제조업체의 지침에 따라 PC/컨포칼 레이저 스캐닝 현미경(CLSM) 설정을 단계적으로 따르십시오. FRET 이미징을 위한 맞춤형 CLSM 구성은 (그림 6)에 나와 있습니다.
- 원격 스위치를 켜서 현미경, 컴퓨터, 스캐너, 레이저 발사, 피에조 스테이지 및 에피 형광 LED 광원에 전원을 공급합니다. 잠재적인 손상을 방지하기 위해 전원을 켜기 전에 모든 구성 요소가 제대로 연결되어 있는지 확인하십시오.
- 레이저 발사의 키를 ON 위치로 돌리고 버튼을 눌러 FRET(457nm 및 514nm 레이저 라인)에 필요한 두 레이저를 모두 활성화합니다.
알림: 최적의 FRET 이미징을 위해 적절한 필터와 설정이 있는지 확인하십시오. 레이저 라인 선택은 바이오센서의 여기 및 방출 프로파일을 기반으로 해야 합니다. - 멀티탭을 켜서 현미경에 연결된 컴퓨터와 모니터에 전원을 공급합니다.
알림: 장비 손상을 방지하기 위해 전원을 켜기 전에 모든 연결이 안전한지 확인하십시오. - Windows에 로그인하고 현미경 소프트웨어를 실행합니다.
- Acquisition을 위해 A1 을 클릭하여 이미징 설정을 시작합니다. FRET 실험의 요구 사항에 따라 적절한 광학 구성을 선택합니다.
- 최적의 이미징 조건을 위해 필요에 따라 레이저 출력과 검출기 감도를 조정합니다. 현미경 스테이지 상단 인큐베이터를 조심스럽게 설치하고 나사로 고정합니다.
알림: 인큐베이터 또는 현미경 s의 손상을 방지하기 위해 나사를 과도하게 조이지 마십시오.tage. - 내부 수조에 멸균 이중 증류수(ddH2O)를 채웁니다. 상부 히터를 단단히 설치하고 s의 전원을 켭니다.tage 히터, 목욕 히터 및 렌즈 히터(그림 7A).
알림: 시스템에 엎질러지지 않도록 과도하게 채우지 마십시오. 라이브 셀 이미징을 위해 일관된 온도 조건을 유지하기 위해 모든 히터가 제대로 작동하는지 확인하십시오. - 이미징을 위해 40x oil immersion lens를 사용합니다( 재료 표 참조).
알림: 제조업체의 web기술 사양 사이트. - 95% 에탄올을 적신 렌즈 종이로 대물 렌즈를 닦습니다. 대물 렌즈에 작은 이멀젼 오일 방울을 놓습니다(그림 7B). HepG2 세포가 들어 있는 이미징 접시를 현미경 스테이지에 놓고 홀더로 고정합니다(그림 7C).
알림: 이미징 중 움직임을 방지하기 위해 이미징 접시가 올바르게 정렬되고 고정되어 있는지 확인하십시오. 렌즈와 접시의 유리 바닥이 모두 손상되지 않도록 렌즈에 가해지는 압력을 피하십시오. - 챔버를 닫고 살아있는 세포 챔버 내의 세포를 1-2시간 동안 배양하여 평형을 이룰 수 있도록 합니다(그림 7D).
- 타임 랩스 이미징 중에는 특정 간격으로 일시 중지하고 매체를 부드럽게 제거한 다음(그림 7E) 주어진 인슐린 농도로 새로 준비된 배지 1mL를 추가합니다(그림 7F, 재료 표).
참고: 10mM 아세트산 1mL에 인슐린 1mg을 용해하여 1mg/mL 인슐린 스톡 용액을 준비합니다. 0.2μm 멸균 주사기 필터를 통해 용액을 여과하고 -20°C에서 부분 표본을 보관합니다. - 현미경 소프트웨어의 파일 메뉴에서 ND 수집 창을 시작합니다.
참고: ND 획득 설정은 (그림 8)에 나와 있습니다. - 시간 탭을 선택하여 타임랩스 이미징을 위한 간격, 지속 시간 및 원하는 루프를 설정합니다. 그런 다음 XY 탭을 클릭하고 + 추가 버튼을 눌러 이미징을 위한 개별 HepG2 세포를 포함합니다.
- 여러 셀을 추가하려면 적합한 셀을 찾고, 스캔하고, 초점을 미세 조정하고, PSF(Point Spread Function)를 잠급니다. 이미징을 위해 추가된 각 새 세포에 대해 이 단계를 반복합니다. 이미징 접시 내의 서로 다른 위치에 있는 여러 세포를 스캔하여 포함할 다양한 표적 세포를 식별합니다.
- 빨간색 X 아이콘을 클릭하여 셀을 선택 취소합니다. 'Z' 상자를 선택하여 Z 위치를 설정합니다. 모든 매개변수를 선택한 후 '파일 이름' 상자에 실험 이름을 입력합니다.
- Browse(찾아보기)를 클릭하여 대상 폴더를 선택한 다음 파일에 저장 상자를 선택합니다. 지금 실행 탭을 눌러 ND 획득을 시작합니다. ND 획득 창에는 경과 시간 및 남은 시간을 포함하여 타임랩스 이미징의 실시간 진행 상황이 표시됩니다.
- 인슐린으로 세포를 자극하지 않고 최대 30분 동안 기준선 측정값을 기록합니다.
- 적합한 세포를 찾고, 확대/축소하여 단일 세포에 초점을 맞추고, 형광 강도가 높은 세포를 선택합니다. 최대 6개의 셀까지 셀을 하나씩 추가합니다.
참고: 이미징 중 이미지 획득 지연 및 창 동결을 방지하기 위해 세포 수를 제한하십시오. - 타임랩스 이미징의 시간과 이미징 빈도를 설정합니다. 타임랩스 이미징을 시작합니다.
참고: 이미징 세션을 시작하기 전에 노출 시간 및 레이저 출력을 포함한 모든 매개변수가 최적화되었는지 확인하십시오. - 일정한 간격으로 이미징을 일시 중지하고, 유리 바닥 접시에서 배지를 부드럽게 제거하고, 적절한 인슐린 농도가 보충된 배지 1mL를 추가합니다.
- 이미지 획득을 다시 시작합니다. 필요에 따라 6.20단계를 반복합니다.
- 실험이 끝나면 마침 탭을 클릭하여 닫습니다. 분석을 위해 획득한 이미지를 안전하게 백업하고 저장할 수 있습니다.
7. 데이터 분석
- 스펙트럼 누화 및 CFP 자가형광에 대해 대조군 샘플에서 사전 처리된 타임 랩스 FRET 이미징 데이터를 수정하여 FRET 값을 수정하고 FRET 효율성을 정밀하게 높일 수 있습니다. 이전 연구27,28,29,30(그림 9)에 설명된 프로토콜에 따라 FRET 이미지의 획득, 처리 및 분석을 위해 이미징 소프트웨어를 사용하십시오.
8. FRET 효율 계산
- FRET 효율성을 결정하기 위해 셀(IDA(D)), IDD(D), IDA(A), IAA(A), I DD, IAA 및 IDA)의 7개 이미지를 획득합니다. 다음 공식을 사용하여 FRET 효율성을 계산합니다.
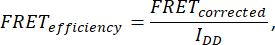
여기서 FRETCorrected는 방정식에서 얻습니다.
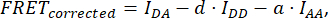
d 와 a 는 다음과 같이 정의됩니다.
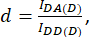
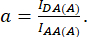
여기서 IDA, I DD 및 IAA는 바이오센서로 형질주입된 세포의 이미지를 나타냅니다. IDA(D) 및 IDD(D))는 공여체 단독 대조군 이미지이고 IDA(A) 및 IAA(A)는 수용체 단독 대조군 이미지입니다.
9. 이미지 획득
- IDA : 수용체 방출(530nm)과 함께 donor excitation(434nm)으로 이미지를 얻습니다.
- IDD : 공여체 방출 (477 nm)과 함께 공여자 여기 (434 nm)에 의해 이미지를 획득합니다.
- IAA : 수용체 방출(530nm)과 함께 수용체 여기(517nm)에 의해 이미지를 획득합니다.
- IDA(D) 및 IDA(A) : IDA와 동일한 excitation 및 emission을 사용하지만 각각 donor와 acceptor만 발현하는 세포에서 이미지를 획득합니다.
- IDD(D) : DONOR-ONLY CONTROL에 대해 IDD와 동일한 조건에서 이미지를 획득합니다.
- IAA(A) : IAA와 동일한 조건을 사용하여 이미지를 얻지만 acceptor-only control에만 해당됩니다.
10. 배경 보정
- 컴퓨터에서 이미지 분석 소프트웨어를 엽니다. 상단 메뉴 모음에서 File 메뉴에 액세스합니다. 드롭다운 옵션에서 열기 또는 파일 열기 를 선택합니다.
- 타임랩스 이미지 데이터가 포함된 디렉토리로 이동합니다. 분석이 필요한 타임랩스 이미지(예: nd2 형식)를 찾습니다.
- 파일을 선택하고 열기 또는 확인을 클릭하여 보고 분석할 수 있도록 소프트웨어에 로드합니다. 셀 내에서 관심 영역(ROI)을 정의하고(또는 전체 셀을 ROI로 사용) 이 영역 내 각 픽셀의 회색 값을 기록합니다.
- 셀이 없는 영역을 배경으로 선택하고 평균 회색 값을 계산합니다. 셀 ROI 내에서 각 픽셀의 회색 값에서 이 배경 평균을 빼서 수정된 이미지를 만듭니다.
11. FRET bleed-through(누화) 제거
참고: 도너 방출과 수용체 여기 사이의 스펙트럼 중첩은 그림 3B에 나와 있으며, 이는 FRET 효율 및 에너지 전달 프로세스에 매우 중요합니다. 타임 랩스 FRET 이미징의 블리드 스루(bleed-through)는 공여체와 수용체 형광단의 스펙트럼 중복으로 인해 발생하는 중요한 문제이며, 이로 인해 부정확한 측정으로 이어집니다. 누화는 공여체 형광단과 수용체 형광단의 스펙트럼이 어느 정도 겹치기 때문에 내재되어 있습니다(그림 3C, D). 이 문제는 높은 형광단 농도 및 부적절한 필터 구성과 같은 요인으로 인해 악화됩니다. 블리드 스루(bleed-through) 문제는 FRET 측정의 신뢰성을 보장하는 데 매우 중요합니다.
- 블리드스루 효과를 완화하는 방법에 대해서는 이전 연구를 참조한다9.
12. 정량화 및 통계 분석
- 통계 분석 소프트웨어를 사용하여 통계 분석을 수행합니다.
결과
HepG2 세포의 Akt 활성화를 조사하기 위해 세포를 사전 코팅된 이미징 접시에 파종하고 Akt 인산화를 실시간으로 모니터링할 수 있도록 설계된 FRET 기반 바이오센서 pEevee-iAkt-NES(그림 2A)로 형질 주입했습니다. 형질주입 후, 세포는 대사 상태를 동기화하고 기저 인슐린 신호 전달을 최소화하기 위해 무혈청 배지에서 4시간 동안 혈청 결핍을 겪었습니다.
토론
HepG2 세포에서 Akt 인산화를 모니터링하기 위한 라이브 셀 FRET 이미징 프로토콜에는 신뢰할 수 있고 재현성 있는 결과를 보장하기 위한 몇 가지 주요 단계가 포함됩니다. 첫 번째 중요한 단계는 세포 배양이며, 여기에는 일상적인 세포 유지 관리, 이미징 접시 코팅 및 세포 파종이 포함됩니다. 타임 랩스 이미징 실험 중 세포 부착을 위해서는 적절한 코팅이 필수적인데, 이?...
공개
저자는 경쟁 이익이 없음을 선언합니다.
감사의 말
이 연구는 선전시 자연과학재단(JCYJ20240813113606009), 선전-홍콩 기술 및 혁신 협력구(HZQB-KCZYB-2020056), 중국 국립자연과학재단(32070681), 중국 국가 핵심 R&D 프로그램(2019YFA0906002), 선전 공작 계획(KQTD2016053117035204)의 일부 지원을 받았다.
자료
| Name | Company | Catalog Number | Comments |
| 0.25% trypsin-EDTA | Gibco | Cat#25200-056 | Use ice-cold PBS for cell wash |
| 15 mm glass bottom cell culture dish | NEST | Cat#801001 | |
| 2 mL Nalgene cryogenic vials | Thermo Scientific | Cat#5012-0020 | |
| 5 mL Stripette Serological Pipets | Corning | Cat#4487 | |
| 95% Ethanol | Kermel | Cat#C028005 | |
| A1 HD25/A1R HD25 confocal microscope | Nikon | https://www.nikon.com/ | Magnification: 40×, Numerical Aperture (NA): 1.30, Pixel Dwell Time: 2.4 ms, Pixel Size: 1024 |
| Ampicillin | Sigma-Aldrich | Cat#A9393 | |
| Bovine serum albumin (BSA) | VWR Life Science | Cat#N208-10g | |
| Corning 25 cm2 rectangular culture flasks | Corning | Cat#430639 | |
| Countess 3 automated cell counter | Thermo Scientific | http://www.thermofisher.com/#AMQAX2000 | |
| Countess cell counting chamber slides | Thermo Scientific | http://www.thermofisher.com/#C10228 | |
| Digital vortex mixers | Thermo Scientific | https://www.thermofisher.com/ | |
| Dimethyl sulfoxide | Sigma-Aldrich | Cat#D2650 | |
| Eppendorf Safe-Lock Tubes 1.5 mL | Eppendorf | Cat#022363204 | |
| EZ-PCR mycoplasma detection kit | Biological Industries | Cat# 20-700-20 | |
| Fetal Bovine Serum, qualified, Australia | Gibco | Cat#10099141 | |
| GlutaMAX Supplement | Gibco | Cat#35050061 | |
| GraphPad Prism 9 | GraphPad Software | https://www.graphpad.com/ | |
| HepG2 | National Collection of Authenticated Cell Cultures | #CSTR:19375.09.3101HUMSCSP510 http://www.cellbank.org.cn/ | |
| Immersion Oil Type 37 | Cargille Laboratories | Cat #16237 | |
| Insulin | Sigma-Aldrich | Cat#I5500-50MG | Warm to 37 °C before use |
| LB Broth (1x) | Invitrogen | Cat#10855001 | |
| Minimum Essential Medium (MEM) | Gibco | Cat#11095080 | Warm to 37 °C before use |
| mySPIN 12 mini centrifuge | Thermo Scientific | https://www.thermofisher.com/ | |
| NanoDrop One | Thermo Scientific | https://www.thermofisher.com | |
| Nikon Plan Fluor 40×/1.30 Oil Lens | Nikon | https://www.nikon.com/ | |
| NIS-Elements-AR | Nikon | https://www.nikon.com/ | |
| Non-Essential Amino Acids (NEAA) (100x) | Gibco | Cat#11140050 | |
| One Shot LB Agar Plates | Invitrogen | Cat#A55802 | |
| One Shot Stbl3 chemically competent E. coli | Invitrogen | Cat#C737303 | |
| Parafilm | PARAFILM | Cat#B8R05606 | |
| PBS (phosphate buffered saline) | Gibco | Cat#10010023 | |
| pEevee-iAkt-NES (7,033 bp) | Miura et al31 | https://benchling.com/s/seq-q46zFYCfl0swLAun0t28/edit | |
| Penicillin-streptomycin | Gibco | Cat#15070063 | |
| Plasmocin prophylactic | InvivoGen | Cat#ant-mpp | |
| Poly-L-Lysine Hydrobromide | Sigma-Aldrich | Cat#P4832 | |
| Precision general purpose baths | Thermo Scientific | https://www.thermofisher.com/ | |
| QIAprep spin miniprep kit | QIAGEN | Cat#27106 | |
| SnapGene | SnapGene by Dotmatics | https://www.snapgene.com | |
| Sodium Pyruvate (100 mM) | Gibco | Cat#11360070 | |
| Syringe filter unit, 0.22 μm | Millipore | Cat#SLGP033RS | |
| Tokai Hit stage top incubator | TOKAI HIT | https://www.tokaihit-livecell.com/stagetopincubator | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | Cat#10977015 | |
| Xfect Transfection Reagent | Takara Bio | Cat#631317 |
참고문헌
- GBD 2021 Diabetes Collaborators. Global, regional, and national burden of diabetes from 1990 to 2021, with projections of prevalence to 2050: a systematic analysis for the Global Burden of Disease Study 2021. Lancet. 402 (10397), 203-234 (2023).
- Li, M., et al. Trends in insulin resistance: insights into mechanisms and therapeutic strategy. Signal Transduct Target Ther. 7 (1), 216 (2022).
- Poloz, Y., Stambolic, V. Obesity and cancer, a case for insulin signaling. Cell Death Dis. 6 (12), e2037 (2015).
- Arcidiacono, B., et al. Insulin resistance and cancer risk: an overview of the pathogenetic mechanisms. Exp Diabetes Res. 2012, 789174 (2012).
- Godsland, I. F. Insulin resistance and hyperinsulinaemia in the development and progression of cancer. Clin Sci (Lond). 118 (5), 315-332 (2009).
- Tsugane, S., Inoue, M. Insulin resistance and cancer: epidemiological evidence. Cancer Sci. 101 (5), 1073-1079 (2010).
- Yudhani, R. D., et al. In vitro insulin resistance model: A recent update. J Obes. 2023, 1964732 (2023).
- Akhtar, J., et al. Bistable insulin response: The win-win solution for glycemic control. iScience. 25 (12), 105561 (2022).
- Akhtar, J., Imran, M., Wang, G. Protocol for live-cell Forster resonance energy transfer imaging to reveal the bistable insulin response of single C2C12-derived myotubes. STAR Protoc. 5 (2), 103109 (2024).
- Kamino, K., et al. Optimal inference of molecular interaction dynamics in FRET microscopy. Proc Natl Acad Sci U S A. 120 (15), e2211807120 (2023).
- Kraft, A. E., Nikolaev, V. O. FRET microscopy for real-time visualization of second messengers in living cells. Methods Mol Biol. 1563, 85-90 (2017).
- Veeriah, S., et al. High-throughput time-resolved FRET reveals Akt/PKB activation as a poor prognostic marker in breast cancer. Cancer Res. 74 (18), 4983-4995 (2014).
- Conway, J. R. W., et al. Monitoring AKT activity and targeting in live tissue and disease contexts using a real-time Akt-FRET biosensor mouse. Sci Adv. 9 (17), eadf9063 (2023).
- Chandris, P., Giannouli, C. C., Panayotou, G. Imaging approaches for the study of metabolism in real time using genetically encoded reporters. Front Cell Dev Biol. 9, 725114 (2021).
- Yang, J., et al. Longitudinal FRET imaging of glucose and lactate dynamics and response to therapy in breast cancer cells. Mol Imaging Biol. 24 (1), 144-155 (2022).
- Mandrou, E., et al. A reliable system for quantitative G-protein activation imaging in cancer cells. Cells. 13 (13), 1114 (2024).
- Fang, C., Huang, Y., Zhao, Y. Review of FRET biosensing and its application in biomolecular detection. Am J Transl Res. 15 (2), 694-709 (2023).
- Miller, J. N. Fluorescence energy transfer methods in bioanalysis. Analyst. 130 (3), 265-270 (2005).
- Verveer, P. J., Rocks, O., Harpur, A. G., Bastiaens, P. I. Imaging protein interactions by FRET microscopy: FRET measurements by acceptor photobleaching. CSH Protoc. 2006 (6), (2006).
- Vu, C. Q., Arai, S. Quantitative imaging of genetically encoded fluorescence lifetime biosensors. Biosensors (Basel). 13 (10), 939 (2023).
- Wang, G. A more holistic view of the logarithmic dose-response curve offers greater insights into insulin response. J Biol Chem. 301 (1), 108037 (2025).
- Wang, G. Body mass dynamics is determined by the metabolic Ohm's law and adipocyte-autonomous fat mass homeostasis. iScience. 23 (6), 101176 (2020).
- Wang, G. Raison d'être of insulin resistance: the adjustable threshold hypothesis. J R Soc Interface. 11 (101), 20140892 (2014).
- Wang, G. Optimal homeostasis necessitates bistable control. J R Soc Interface. 9, 2723 (2012).
- Heim, R., Tsien, R. Y. Engineering green fluorescent protein for improved brightness, longer wavelengths and fluorescence resonance energy transfer. Curr Biol. 6 (2), 178-182 (1996).
- Nguyen, A. W., Daugherty, P. S. Evolutionary optimization of fluorescent proteins for intracellular FRET. Nat Biotechnol. 23 (3), 355-360 (2005).
- Broussard, J. A., Rappaz, B., Webb, D. J., Brown, C. M. Fluorescence resonance energy transfer microscopy as demonstrated by measuring the activation of the serine/threonine kinase Akt. Nat Protoc. 8 (2), 265-281 (2013).
- Gordon, G. W., Berry, G., Liang, X. H., Levine, B., Herman, B. Quantitative fluorescence resonance energy transfer measurements using fluorescence microscopy. Biophys J. 74 (5), 2702-2713 (1998).
- Tron, L., et al. Flow cytometric measurement of fluorescence resonance energy transfer on cell surfaces. Quantitative evaluation of the transfer efficiency on a cell-by-cell basis. Biophys J. 45 (5), 939-946 (1984).
- Youvan, D. C., et al. Calibration of fluorescence resonance energy transfer in microscopy. , (2002).
- Miura, H., Matsuda, M., Aoki, K. Development of a FRET biosensor with high specificity for Akt. Cell Struct Funct. 39 (1), 9-20 (2014).
- Liberio, M. S., Sadowski, M. C., Soekmadji, C., Davis, R. A., Nelson, C. C. Differential effects of tissue culture coating substrates on prostate cancer cell adherence, morphology and behavior. PLoS One. 9 (11), e112122 (2014).
- Rani, K., Sengupta, S. Multi-stimuli programmable FRET based RGB absorbing antennae towards ratiometric temperature, pH and multiple metal ion sensing. Chem Sci. 12 (47), 15533-15542 (2021).
- Betolngar, D. B., et al. pH sensitivity of FRET reporters based on cyan and yellow fluorescent proteins. Anal Bioanal Chem. 407 (14), 4183-4193 (2015).
- Salonikidis, P. S., et al. An ion-insensitive cAMP biosensor for long term quantitative ratiometric fluorescence resonance energy transfer (FRET) measurements under variable physiological conditions. J Biol Chem. 286 (26), 23419-23431 (2011).
- Youvan, D. C., et al. Calibration of fluorescence resonance energy transfer in microscopy using genetically engineered GFP derivatives on nickel chelating beads. Biotechnol Alia. 3, 1-18 (1997).
- Menaesse, A., et al. Simplified instrument calibration for wide-field fluorescence resonance energy transfer (FRET) measured by the sensitized emission method. Cytometry A. 99 (4), 407-416 (2021).
- Batta, A., Hajdu, T., Nagy, P. Improved estimation of the ratio of detection efficiencies of excited acceptors and donors for FRET measurements. Cytometry A. 103 (7), 563-574 (2023).
- Coullomb, A., et al. QuanTI-FRET: a framework for quantitative FRET measurements in living cells. Sci Rep. 10 (1), 6504 (2020).
- Hoppe, A., Christensen, K., Swanson, J. A. Fluorescence resonance energy transfer-based stoichiometry in living cells. Biophys J. 83 (6), 3652-3664 (2002).
- Zal, T., Gascoigne, N. R. Photobleaching-corrected FRET efficiency imaging of live cells. Biophys J. 86 (6), 3923-3939 (2004).
- Hochreiter, B., Kunze, M., Moser, B., Schmid, J. A. Advanced FRET normalization allows quantitative analysis of protein interactions including stoichiometries and relative affinities in living cells. Sci Rep. 9 (1), 8233 (2019).
- Liu, H., Zhang, F., Mishra, S. K., Zhou, S., Zheng, J. Knowledge-guided fuzzy logic modeling to infer cellular signaling networks from proteomic data. Sci Rep. 6, 35652 (2016).
- Garrido-Rodriguez, M., Zirngibl, K., Ivanova, O., Lobentanzer, S., Saez-Rodriguez, J. Integrating knowledge and omics to decipher mechanisms via large-scale models of signaling networks. Mol Syst Biol. 18 (7), e11036 (2022).
- Greenwald, E. C., Polanowska-Grabowska, R. K., Saucerman, J. J. Integrating fluorescent biosensor data using computational models. Methods Mol Biol. 1071, 227-248 (2014).
- Zhao, Z., Xia, J. Computational Approaches for Modeling Signal Transduction Networks. Encyclopedia of Bioinformatics and Computational Biology. , (2019).
- Wang, G., Krueger, G. R. Computational analysis of mTOR signaling pathway: bifurcation, carcinogenesis, and drug discovery. Anticancer Res. 30 (7), 2683-2688 (2010).
- Wang, G. Singularity analysis of the AKT signaling pathway reveals connections between cancer and metabolic diseases. Phys Biol. 7 (4), 046015 (2010).
- Chedere, A., Hari, K., Kumar, S., Rangarajan, A., Jolly, M. K. Multi-stability and consequent phenotypic plasticity in AMPK-Akt double negative feedback loop in cancer cells. J Clin Med. 10 (3), 472 (2021).
- Mosca, E., et al. Computational modeling of the metabolic States regulated by the kinase akt. Front Physiol. 3, 418 (2012).
- Liao, J., Madahar, V., Dang, R., Jiang, L. Quantitative FRET (qFRET) technology for the determination of protein-protein interaction affinity in solution. Molecules. 26 (21), 6339 (2021).
- Verma, A. K., Noumani, A., Yadav, A. K., Solanki, P. R. FRET based biosensor: Principle applications recent advances and challenges. Diagnostics (Basel). 13 (8), 1375 (2023).
- Mattheisen, J. M., et al. Application of bioluminescence resonance energy transfer to quantitate cell-surface expression of membrane proteins. Anal Biochem. 684, 115361 (2024).
- Lionetti, M. C., La Porta, C. A. M. FLIM-FRET investigation of heterogeneous huntingtin aggregation in HeLa cells. Methods Mol Biol. 2551, 595-604 (2023).
- Petutschnig, E. K., Pierdzig, L., Mittendorf, J., Niebisch, J. M., Lipka, V. A novel fluorescent protein pair facilitates FLIM-FRET analysis of plant immune receptor interaction under native conditions. J Exp Bot. 75 (3), 746-759 (2024).
- Sprenger, J. U., Perera, R. K., Gotz, K. R., Nikolaev, V. O. FRET microscopy for real-time monitoring of signaling events in live cells using unimolecular biosensors. J Vis Exp. 66, e4081 (2012).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유