Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Quantification automatisée de la fluorescence dans Synaptic
Dans cet article
Résumé
L'abondance des récepteurs de neurotransmetteurs dans les synapses en cluster influe fortement sur la force synaptique. Cette méthode permet de quantifier les récepteurs des neurotransmetteurs marquage fluorescent en trois dimensions avec une seule synapse résolution C. elegans, Permettant à des centaines de synapses d'être rapidement caractérisé au sein d'un échantillon unique sans distorsions introduites par z-plan de projection.
Résumé
La force Synapse se réfère à l'amplitude des réponses postsynaptiques à la libération de neurotransmetteurs présynaptiques des événements, et a un impact majeur sur la fonction circuit global de neurones. Synapse force dépend de façon critique sur l'abondance des récepteurs de neurotransmetteurs en cluster sur les sites synaptiques sur la membrane postsynaptique. Les niveaux de récepteurs sont établis développemental, et peut être modifié par le trafic des récepteurs entre les pools de surface localisée, subsynaptic, et intracellulaires, ce qui représente d'importants mécanismes de la plasticité synaptique et la neuromodulation. Des méthodes rigoureuses pour quantifier synapse-localisée abondance des récepteurs de neurotransmetteurs sont indispensables pour étudier le développement et la plasticité synaptique. La microscopie à fluorescence est une approche optimale, car elle préserve l'information spatiale, en distinguant synaptique de la non-synaptiques piscines, et la discrimination entre les populations de récepteurs localisés à différents types de synapses. La génétique organisme modèle Caenorhabditis elegans est particulièrement bien adapté pour ces études en raison de la petite taille et leur relative simplicité de son système nerveux, sa transparence, et la disponibilité de puissantes techniques génétiques, permettant l'examen des synapses indigènes chez les animaux intacts.
Nous présentons ici une méthode pour quantifier marquage fluorescent récepteurs de neurotransmetteurs synaptiques dans C. elegans. Sa principale caractéristique est l'identification automatique et l'analyse des synapses individuelles en trois dimensions dans plusieurs fichiers de sortie d'avion confocaux microscope, la position tabulation, volume, intensité de la fluorescence et de fluorescence totale pour chaque synapse. Cette approche présente deux avantages principaux sur une analyse manuelle de z-plan des projections de données confocale. Tout d'abord, parce que chaque plan de l'ensemble des données est inclus confocale, pas de données sont perdues à z-plan de projection, généralement basées sur les moyennes d'intensité de pixels ou maxima. Deuxièmement, l'identification des synapses est automatisé, mais peut être inspecté par l'expérimétrique comme les produits d'analyse de données, permettant l'extraction rapide et précise des données provenant de grands nombres de synapses. Des centaines de milliers de synapses par l'échantillon peut être facilement obtenue, la production de grands ensembles de données afin de maximiser la puissance statistique. Considérations pour la préparation de C. elegans pour l'analyse et l'exécution de l'imagerie confocale pour minimiser la variabilité entre les animaux au sein des groupes de traitement sont également discutés. Bien que développé pour analyser C. récepteurs post-synaptiques elegans, cette méthode est généralement utile pour tout type de synapse-localisée protéines, ou même, tout signal de fluorescence qui est localisée aux pôles discrets, puncta, ou les organites.
La procédure est réalisée en trois étapes: 1) la préparation d'échantillons, 2) l'imagerie confocale, et analyse d'image 3). Les étapes 1 et 2 sont spécifiques à C. elegans, tandis que l'étape 3 est généralement applicable à tout signal de fluorescence dans ponctuée micrographies confocales.
Protocole
1. Préparation de Worms pour l'imagerie
Ce segment du protocole est basé sur publia C. techniques de culture elegans 1,2, et est présenté dans la figure 1.
- Cultiver des vers sur des plaques de gélose peptone haute NGM (10 cm) ensemencées avec NA22 E. La bactérie E. jusqu'à près de mourir de faim. Si immunocoloration, une plaque sera fiable de produire assez de vers pour 1 personne tache, compte tenu des pertes le long du chemin, et les critères stricts utilisés pour sélectionner des vers pour l'imagerie (voir partie 2).
- Vers la récolte en versant quelques millilitres de ddH 2 0 sur la plaque, en agitant brièvement, et verser le liquide dans un tube de 15 ml conique (répéter si nécessaire de transférer la plupart des vers dans le tube). Pellet en centrifugeuse clinique pendant 3 min, g 1000x. Laver à granulés 1-3x avec ddH 2 O jusqu'à ce surnageant est clair pour éliminer les bactéries résiduelles. N'utiliser que des pipettes de transfert en polypropylène pour supprimer surnageants, que les vers s'en tenir à verre(À partir de ce étape, il est essentiel pour minimiser le temps entre les étapes pour éviter les pertes dans la viabilité des œufs).
- Après le dernier lavage, le ver de remettre en suspension le culot dans 5 ml solution d'hypochlorite de alcaline pour lyser les vers et les œufs de libération. Bercer doucement, et d'examiner les tubes sous un microscope à dissection environ une fois par minute. Lorsque environ 50% des vers sont lysées (ils apparaissent tordues et brisées ouvert), mettre fin à la lyse par le remplissage du tube vers le haut avec le tampon d'oeufs, retournant plusieurs fois, et la granulation pendant 3 min, g 1000x. Temps de lyse ne doit pas dépasser 5 minutes.
- Enlever le surnageant avec une pipette de transfert, et se laver à granulés 3x avec le tampon d'oeufs.
- Pour séparer les œufs de débris, les faire flotter sur un coussin de saccharose à 30% dans le trou DDH 2 O: après le lavage final de l'étape 1.4, retirez avec précaution le surnageant et remettre en suspension le culot dans 5 ml ddH 2 0. Ajouter 5 ml de saccharose à 60% stérile dans le trou DDH 2 O, et bien mélanger. Spin dans la centrifugeuse clinique 6 min, g 1000x. Oeufs permettra de recueillir au niveau du ménisque (ils have un aspect trouble), tandis que les débris de granules.
- En utilisant une pipette de transfert en plastique, aspirer les oeufs dans un volume minimal et transfert à des boîtes de gélose non ensemencés NGM. Les oeufs qui adhèrent à côté du tube peut être rincé doucement et transférés ainsi. Une plaque de 10 cm ensemencés par la souche analysée est suffisante.
- Incuber les plaques non ensemencés avec les oeufs nuit (~ 16 h) à 20 ° C. Purger la plaque pour les premières heures de la sécher en laissant le couvercle légèrement hors de quelques heures, mais être sûr de couvrir toute la nuit.
- Après l'éclosion, les larves L1 transférer le à un tube de 15 ml conique basale S (ajouter à la plaque basale quelques ml S, tourbillon, versez dans le tube). Pellet en centrifugeuse clinique (3 min, 1000x g), remettre en suspension dans un volume minimal de baso-S, et le transfert de plaques de 10 cm NGM gélose ensemencée avec NA22 bactéries. Utilisez 2 plaques ici pour chaque plaque d'origine de l'étape 1.1. Incuber jusque vers atteignent l'âge de votre choix.
- Surveiller les cultures une ou deux fois par jour. Si ils sont en danger of faim, de transférer à de nouvelles plaques NA22. Pour les vers de type sauvage N2, la famine est précédée par la formation d'une ligne ou une vague de vers qui se déplace à travers la plaque, comme les animaux migrent en masse loin de la nourriture appauvries zones. Une fois cette forme, la nourriture sera complètement épuisé en quelques heures.
- Worms sont maintenant prêts pour immunomarquage ou imagerie en temps réel. Fixation et coloration des vers en suspension est optimale car un grand nombre de vers intacts peuvent être visualisés facilement. Procédures de coloration sur la base du protocole de Finney et Ruvkun 3-6 sont préférable de congeler du crack procédures car un grand nombre de vers sont nécessaires pour l'imagerie.
2. Imagerie confocale
- Monter vers sur des lames pour l'imagerie confocale. Réglage de la densité de vers à quelques centaines par microlitre. Ajouter un tampon de 2% d'agarose à S-basale entourée par un anneau mince de vaseline pour empêcher l'évaporation au cours d'imagerie (appliqué par l'intermédiaire d'une seringue de 3 ml équipé d'un seuil de 25 aiguille de calibre). Aide d'une pipettemicrolitres de la suspension quelques ver sur une lamelle (utiliser l'anesthésie si des vers vivants d'imagerie), et d'abaisser le tampon d'agarose sur la lamelle, la propagation des vers uniformément sur le pavé.
- Sélectionnez les vers destinés à l'imagerie. Choisir vers lequel les synapses d'intérêt sont orientés vers la lentille d'objectif, avec un déplacement radial de pas plus de ± 45 ° (où 0 ° signifie orientée directement vers l'objectif). Effectuez cette évaluation rapide (quelques secondes) pour éviter le photoblanchiment. L'échelle des cultures initiales est optimisé pour assurer des vers suffisantes seront satisfaire à ces critères géométriques.
- Remplissez l'imagerie confocale de l'échantillon. Échantillons relativement plats qui peuvent être envisagées dans environ 14 0,4 um d'épaisseur sections produire les meilleurs résultats. Haute vitesse, des images à faible résolution (512 x 512 pixels) sont suffisants pour la collecte de données rapide précise. Éviter de saturer le détecteur avec les forces de signal excessives, car cela entraînerait une sous-estimation de la fluorescencesignaux.
3. D'identification automatique et de l'analyse des clusters individuels Synaptic
- Ouvrir les fichiers de sortie multi-TIF de la microscopie confocale en utilisant Volocity 4.0 (ou supérieur) logiciel (PerkinElmer).
- Rogner les zones de l'image qui ne contient pas la structure d'intérêt (Image -> Focus étendue -> Découper la sélection> Choisir zone d'intérêt en utilisant «à main levée ROI outil - -> Actions; la figure 2A). Remarque, Focus étendue produit une projection z-plan sur l'écran de l'ordinateur à l'aide de votre analyse, mais n'affecte pas les données sous-jacentes; la même chose est vraie pour les réglages de luminosité et de contraste, le cas échéant).
- Mesurer la longueur de la zone analysée à l'aide de l'outil Ligne. La longueur de la ligne seront calculés et ajoutés à la table de données (figure 2B).
- Identifier les objets en utilisant les 'objets par l'intensité »de filtre (mode Mesures). Spécifiez un seuil tel que les grappes sont synaptiquesmis en évidence et non-synaptique de fond n'est pas (figure 2C). Inclusion d'un marqueur indépendant synaptique marqué avec une autre couleur peut aider à identifier sans ambiguïté les synapses. Synapses typiques vont de quelques-uns à quelques centaines de voxels. Régler le seuil de fois en utilisant un échantillon de contrôle, et l'utiliser pour tous les échantillons subséquents. Seuillage chaque échantillon est séparément inacceptable, car elle introduit la possibilité de biais expérimentateur. Les objets sont sélectionnés et totalisées automatiquement (figure 2D).
- Organiser et exporter les données. Éliminer les objets de 2 ou moins voxels, puisque ceux-ci sont généralement des taches de fond. Ceux-ci peuvent être éliminés par filtrage d'autres, ou lors de l'analyse ultérieure. Afin de faciliter leurs aval de déménagement, re-trier le fichier de données par la 'taille' objet donc ils veulent tout regrouper. Exportez les données au format CSV, qui peut être ouvert par Microsoft Excel ou autres tableurs. Chaque image produit un fichier de sortie.
- Si douce VolocityWare n'est pas disponible, d'autres méthodes pour analyser les Z-plan des projections de données confocale peut être utilisé (par exemple réf. 7, ou l'analyse manuelle à l'aide ImageJ), même si en 3 dimensions l'information est perdue (voir Discussion).
4. Les résultats représentatifs
La méthode de quantification présenté doit être en mesure de faire la distinction entre les populations du cluster de luminosité différente et des volumes différents. Des images représentatives et correspondants les données quantitatives présentées dans la figure 3 montrent des exemples de différenciation sur la base de ces paramètres. En règle générale, les résultats doivent être conformes à ce qui est évident à l'œil. Dans le cas de l'UNC-49 récepteur GABA immunocoloration la corde nerveuse ventrale de C. elegans, tous les animaux en général est apparu très similaire en taille et en maturité, et le total des valeurs de fluorescence pour synaptiques un groupe de cinq vers (normalisé à la longueur de la moelle épinière analysés) a montré l'erreur type des valeurs d'environ 10% de la moyenne 4. Une mutation génétique et d'autres traitements expérimentaux (l'exposition au médicament, par exemple) peuvent modifier non seulement le volume et les valeurs d'intensité et de distributions de fréquence des grappes synaptiques, mais peut-être le développement du temps bien sûr et de la synchronie des vers, conduisant à une plus grande variabilité. Toutefois, les résultats quantitatifs doivent toujours refléter ce qui peut être apprécié visuellement et si elle n'est pas, à l'inspection des images et des objets identifiés par Volocity devrait révéler l'endroit où l'erreur s'est produite et de proposer des mesures correctives telles que re-seuillage ou la suppression d'objets artefactuelles.

Figure 1. Préparation de synchrone C. cultures elegans. cultures synchrones sont obtenus à partir de cette procédure parce que C. arrestations de développement elegans et le stade larvaire L1 en l'absence de nourriture, et reprend lorsque la nourriture est introduite.
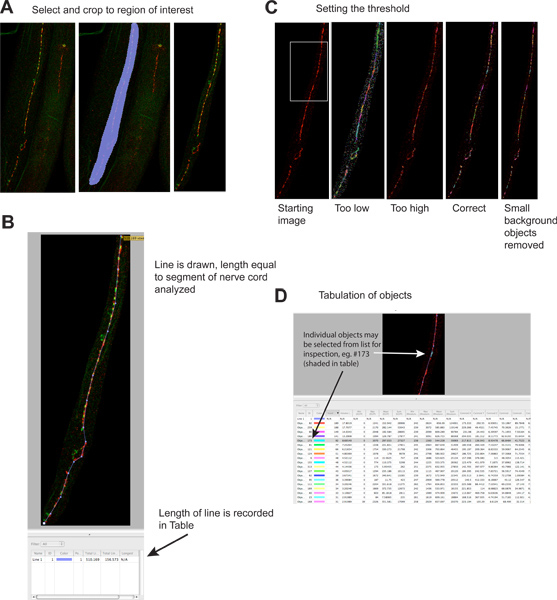
Figure 2. La quantification de puncta synaptique.
- Régions d'origine étrangère de l'image sont rognés pour réduire la taille des fichiers et d'accroître la spécificité. Panneau de gauche montre l'image de départ, le panneau du milieu montre la région sélectionnée, panneau de droite montre l'image après le recadrage. Signal rouge est UNC-49 immunofluorescence récepteur GABA, le signal vert est synaptobrévine-GFP (un marqueur présynaptique) exprimée en présynaptiques des neurones GABA 4. Cliquez ici pour agrandir la figure .
- Une ligne est tracée la longueur du segment moelle épinière à analyser, et sa longueur est automatiquement enregistré dans le tableau des résultats. Cette information est nécessaire pour normaliser les données synaptiques dues à la longueur de la moelle épinière analysable, qui peut varier de plusieurs fois si les vers se fragmenter pendant la coloration.www.jove.com/files/ftp_upload/4090/4090fig2blarge.jpg "target =" _blank "> Cliquez ici pour agrandir la figure.
- Un seuil est appliqué à identifier individuellement les clusters synaptiques. Le panneau de gauche montre l'image avant de seuillage; des exemples de niveaux de seuillage différents sont présentés dans des panneaux séparés (signal vert omis). Chaque objet identifié est représenté sur l'image en tant que région colorée. Notez la correspondance entre les groupes visuellement évidentes synaptiques dans le panneau le plus à gauche et les régions de couleur sur le panneau le plus à droite. Cliquez ici pour agrandir la figure .
- Chaque objet identifié est répertorié séparément dans le tableau des résultats. Objets individuels peuvent être sélectionnés, et sont mises en évidence sur l'image pour l'inspection, si désiré. Notez que la table contient des informations pour le rouge ("rhodamine") et vert ('EGFP ») des canaux; les informations du canal vert est potentiellement trompeuse, car les objets sontidentifiés sur la base strictement sur la fluorescence rouge. Cette analyse peut également être exécutées sur les images d'une seule couleur. Cliquez ici pour agrandir la figure .
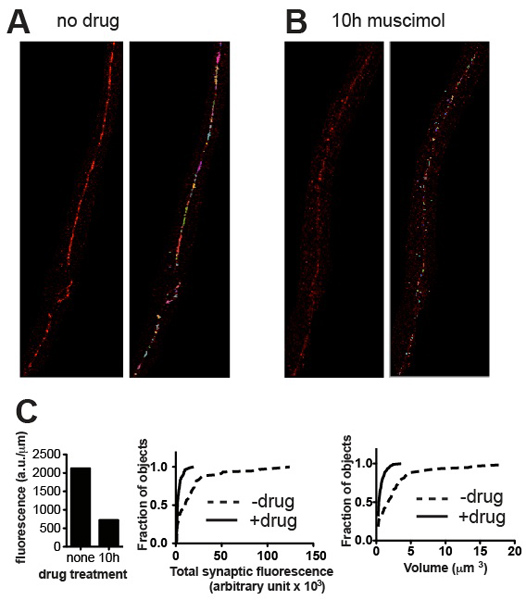
Figure 3. Des résultats représentatifs. Micrographies représentatives de type sauvage C. elegans colorés pour UNC-49 récepteurs GABA avant (A) et après (B) le traitement par le muscimol, un agoniste des récepteurs GABA qui provoque récepteurs deviennent contrerégulés après une longue exposition. Panneaux montrer des images recadrées avant (à gauche) et après (à droite) de seuillage et l'enlèvement des taches de fond de petite taille. (C) Parcelles de paramètres quantitatifs synaptiques pour les spécimens présentés dans (A) et (B): fluorescence totale normalisé à la longueur du cordon nerveux (à gauche), et les histogrammes probabilité cumulative de contenu synapse fluorescence individuelle (au milieu) et le volume des synapses (right), démontrant une réduction statistiquement significative de la teneur et le volume synaptique induite par l'exposition agoniste (n = 60 pour les synapses non traités, n = 115 synapses pour muscimol-traité, p <0,001, test de Kolmogorov-Smirnov http://www.physics.csbsju .edu / stats / KS-test.html ).
Discussion
La méthode présentée ici est conçu pour extraire quantitatives multi-paramètres de données pour de grandes populations de synapses dans C. elegans, tout en maximisant la cohérence au sein des groupes de traitement. Trois caractéristiques contribuent à ces objectifs. Tout d'abord, immunomarquage est effectué sur les populations de vers synchrones afin de s'assurer que tous les animaux ont le même âge. Cette étape est cruciale, car la régulation du développement des niveaux d'expression...
Déclarations de divulgation
Pas de conflits d'intérêt déclarés.
Remerciements
Les auteurs tiennent à remercier A. Benham pour aider à l'élaboration du protocole. Ce travail a été financé par le NIH NS06747 subvention au BAB
matériels
| Name | Company | Catalog Number | Comments | ||||||
| Nom du logiciel | Entreprise | Commentaires (optionnel) | |||||||
| Volocity v4.0 ou supérieur | PerkinElmer / Improvision | Vérifiez votre installation locale de base d'imagerie pour l'accès à ce logiciel. Logiciel de démonstration est disponible sur le site Web de PerkinElmer. Cette méthode ne requiert que le module de quantification de Volocity. | |||||||
| Tableau 2. Réactifs et équipements spécifiques. | |||||||||
| |||||||||
| Tableau 1. Solutions. |
Références
- Christensen, M. A primary culture system for functional analysis of C. elegans neurons and muscle cells. Neuron. 33, 503-514 (2002).
- Lewis, J. A., Fleming, J. T. Basic culture methods. Methods Cell Biol. 48, 3-29 (1995).
- Bettinger, J. C., Lee, K., Rougvie, A. E. Stage-specific accumulation of the terminal differentiation factor LIN-29 during Caenorhabditis elegans development. Development. 122, 2517-2527 (1996).
- Davis, K. M. Regulated lysosomal trafficking as a mechanism for regulating GABAA receptor abundance at synapses in Caenorhabditis elegans. Mol. Cell Neurosci. 44, 307-317 (2010).
- Finney, M., Ruvkun, G. The unc-86 gene product couples cell lineage and cell identity in C. elegans. Cell. 63, 895-905 (1990).
- Rowland, A. M. Presynaptic terminals independently regulate synaptic clustering and autophagy of GABAA receptors in Caenorhabditis elegans. J. Neurosci. 26, 1711-1720 (2006).
- Burbea, M. Ubiquitin and AP180 regulate the abundance of GLR-1 glutamate receptors at postsynaptic elements in C. elegans. Neuron. 35, 107-120 (2002).
- Miesenbock, G., De Angelis, D. A., Rothman, J. E. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins. Nature. 394, 192-195 (1998).
- Oda, S., Tomioka, M., Iino, Y. Neuronal plasticity regulated by the insulin-like signaling pathway underlies salt chemotaxis learning in Caenorhabditis elegans. J. Neurophysiol. 106, 301-308 (2011).
- Sankaranarayanan, S. The use of pHluorins for optical measurements of presynaptic activity. Biophys. J. 79, 2199-2208 (2000).
- McDonald, N. A. Generation and functional characterization of fluorescent, N-terminally tagged CB1 receptor chimeras for live-cell imaging. Mol. Cell Neurosci. 35, 237-248 (2007).
- Shakiryanova, D. Synaptic neuropeptide release induced by octopamine without Ca2+ entry into the nerve terminal. Proc. Natl. Acad. Sci. U. S. A. 108, 4477-4481 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon