È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Quantificazione automatica di fluorescenza in Synaptic
In questo articolo
Riepilogo
L'abbondanza dei recettori dei neurotrasmettitori a livello delle sinapsi cluster influenza fortemente la forza sinaptica. Questo metodo quantifica recettori dei neurotrasmettitori marcato in fluorescenza in tre dimensioni con un singolo sinapsi risoluzione C. elegans, Consentendo a centinaia di sinapsi di essere rapidamente caratterizzato all'interno di un campione unico senza distorsioni introdotte dal z-piano di proiezione.
Abstract
Forza Synapse si riferisce alla ampiezza delle risposte post-sinaptici a eventi presinaptici di rilascio dei neurotrasmettitori, e ha un impatto significativo sulla funzione complessiva del circuito neurale. Synapse forza dipende in modo critico l'abbondanza di recettori neurotrasmettitori raggruppati in siti sinaptici sulla membrana postsinaptica. Livelli di recettori sono stabiliti evolutivamente, e può essere alterato da traffico tra i diversi centri del recettore di superficie localizzato, subsynaptic e intracellulare, che rappresentano importanti meccanismi di plasticità sinaptica e neuromodulazione. Metodi rigorosi per quantificare sinapticamente-localizzato abbondanza recettore del neurotrasmettitore sono essenziali per studiare lo sviluppo e la plasticità sinaptica. Microscopia a fluorescenza è un approccio ottimale perché conserva l'informazione territoriale, distinguendo da synaptic non-sinaptici piscine e discriminando tra le popolazioni dei recettori localizzati a diversi tipi di sinapsi. Il modello genetico dell'organismo Caenorhabditis elegans è particolarmente adatto per questi studi a causa della piccola dimensione e la relativa semplicità del suo sistema nervoso, la trasparenza, e la disponibilità di potenti tecniche genetiche, consentendo l'esame di sinapsi nativi in animali intatti.
Qui vi presentiamo un metodo per quantificare marcato in fluorescenza recettori dei neurotrasmettitori sinaptici in C. elegans. La sua caratteristica fondamentale è l'identificazione automatica e l'analisi delle singole sinapsi in tre dimensioni in multi-piano i file di output microscopio confocale, la posizione tabulazione, volume, intensità di fluorescenza e fluorescenza totale per ogni sinapsi. Questo approccio ha due vantaggi principali rispetto analisi manuale di Z-Plane proiezioni di dati confocale. In primo luogo, perché ogni piano del set di dati confocale è incluso, nessun dato viene perso attraverso z-piano di proiezione, in genere basato sulle medie intensità pixel o massimi. In secondo luogo, l'identificazione delle sinapsi è automatizzato, ma può essere ispezionati da exsperimentatore, come procede l'analisi dei dati, che consente l'estrazione rapida e precisa di dati provenienti da un gran numero di sinapsi. Centinaia di migliaia di sinapsi per campione può essere facilmente ottenuto, producendo grandi quantità di dati per massimizzare la potenza statistica. Considerazioni per la preparazione di C. elegans per l'analisi e l'esecuzione confocale a minimizzare la variabilità tra gli animali all'interno dei gruppi di trattamento vengono anche discussi. Nonostante sia stato sviluppato per analizzare C. recettori postsinaptici elegans, questo metodo è generalmente utile per qualsiasi tipo di sinapticamente-localizzata proteina, o addirittura, qualsiasi segnale di fluorescenza che è localizzato a cluster discreti, puncta o organelli.
La procedura viene eseguita in tre fasi: 1) preparazione dei campioni, 2) confocale, e 3) analisi delle immagini. I passi 1 e 2 sono specifici per C. elegans, mentre il punto 3 è generalmente applicabile a qualsiasi segnale di fluorescenza puntata al microscopio confocale.
Protocollo
1. Preparazione di Worms per l'imaging
Questo segmento del protocollo si basa su Published C. tecniche di coltura elegans 1,2, e viene illustrato in Figura 1.
- Crescere vermi su alti peptoni piastre di agar NGM (10 cm) con teste di serie NA22 E. coli fino quasi morire di fame. Se immunostaining, una piastra in modo affidabile a sufficienza per produrre i vermi 1 macchia individuale, tenendo conto delle perdite lungo la strada, ei rigidi criteri utilizzati per selezionare i vermi per l'imaging (vedi Parte 2).
- Harvest di vermi versando alcuni ml di ddh 2 0 sulla piastra, agitando brevemente, e versando liquido in un tubo da 15 ml (ripetere se necessario per trasferire la maggior parte dei vermi al tubo). Pellet in centrifuga clinica per 3 min, g 1000x. Lavare pellet 1-3x con ddh 2 O fino surnatante è chiaro a rimuovere i batteri residui. Usare pipette in polipropilene per rimuovere surnatanti, come i vermi attaccano al vetro(Da questo punto in poi è fondamentale ridurre al minimo il tempo tra i passaggi per evitare perdite di efficienza uovo).
- Dopo l'ultimo lavaggio, risospendere verme pellet in 5 ml di soluzione di ipoclorito alcalino per lisare vermi e uova di rilascio. Rock delicatamente, ed esaminare i tubi sotto un microscopio da dissezione circa una volta al minuto. Quando circa il 50% di vermi vengono lisati (appariranno piegato e rotto aperto), terminare la lisi compilando il tubo verso l'alto con il tampone Egg, invertendo più volte, e cubettatura per 3 min, g 1000x. Tempo di lisi non deve superare i 5 minuti.
- Rimuovere il surnatante con la pipetta di trasferimento, pellet e lavare 3 volte con il tampone di Egg.
- Per separare le uova dai detriti, li galleggiano su un cuscino del 30% di saccarosio in DDH 2 O: dopo il lavaggio finale dal punto 1.4, rimuovere con cautela il surnatante e risospendere pellet in 5 ml ddh 2 0. Aggiungere 5 ml di saccarosio sterile al 60% in DDH 2 O, e mescolare bene. Spin in centrifuga clinica 6 min, g 1000x. Le uova si raccoglieranno nella parte menisco (saranno sciutaVE aspetto torbido), mentre detriti pellet.
- Utilizzando una pipetta di trasferimento in plastica, aspirare le uova in un volume minimo e trasferire alle piastre di agar non teste di serie NGM. Le uova aderiscono al lato del tubo può essere risciacquato con delicatezza e trasferite. Una piastra di 10 cm non teste di serie per ceppo analizzato è sufficiente.
- Incubare le piastre non seminati con le uova durante la notte (~ 16 ore) a 20 ° C. Ventilare la piastra per le prime ore di asciugare lasciando il coperchio un po 'fuori per qualche ora, ma essere sicuri di coprire tutta la notte.
- Dopo la schiusa, trasferire le larve L1 ad un tubo di 15 ml conica in S basale (aggiungere alcuni ml S basale alla piastra, turbine, versare nel tubo). Pellet in centrifuga clinica (3 min, 1000x g), risospendere in un volume minimo di Basal S, e il trasferimento di 10 cm NGM piastre di agar seminate con NA22 batteri. Utilizzare 2 piastre qui per ciascun piatto originale dal punto 1.1. Incubare fino al raggiungimento dell'età worm desiderato.
- Monitorare le culture una o due volte al giorno. Se sono in pericolo of fame, trasferimento a fresco NA22 piatti. Per la wild-type worm N2, la fame è preceduta dalla formazione di una linea o ondata di worm che si muove attraverso la piastra come animali migrano in massa lontano da alimenti impoverito zone. Una volta queste forme, il cibo sarà completamente esaurito in poche ore.
- Worms sono ora pronti per immunostaining o immagini dal vivo. Fissaggio e colorazione vermi in sospensione è ottimale perché un gran numero di vermi intatti può essere ripreso con facilità. Procedure di colorazione in base al protocollo di Finney e Ruvkun 3-6 sono preferibili a congelare-rompere le procedure perché un gran numero di vermi sono necessari per l'imaging.
2. Confocale Imaging
- Montare i vermi sulle diapositive per l'imaging confocale. Regolare la densità di vermi a qualche centinaio per microlitro. Effettuare una pad di agarosio al 2% in S-basale circondato da un sottile anello di vaselina per impedire l'evaporazione durante l'imaging (applicato tramite una siringa 3 ml dotato di un cut-off 25 gauge). Pipettare unpochi microlitri della sospensione verme su un vetrino (uso anestetico se immagini vermi vivi), e abbassare il pad agarosio sul vetrino, diffondendo i vermi in modo uniforme sul pad.
- Selezionare vermi per l'imaging. Scegliere vermi dove le sinapsi di interesse sono orientate verso la lente obiettivo, con spostamento radiale di non più di ± 45 ° (dove 0 ° mezzo orientato direttamente verso l'obiettivo). Eseguire questa valutazione rapida (pochi secondi) per evitare photobleaching. La scala delle colture iniziali è ottimizzato per garantire sufficienti worm in grado di soddisfare questi criteri geometrici.
- Completare la confocale del campione. Campioni relativamente piatta che può essere compreso all'interno di circa 14 micron di spessore 0,4 sezioni produrre i migliori risultati. Alta velocità, immagini a bassa risoluzione (512 x 512 pixel) sono sufficienti per una rapida raccolta accurata dei dati. Evitare di saturare il rivelatore con intensità di segnale eccessivi, ciò causerebbe sottostima della fluorescenzasegnali.
3. Identificazione automatica e analisi dei singoli cluster Synaptic
- Aprire file TIF multi-uscita dal microscopio confocale utilizzando Volocity 4.0 (o superiore) software (PerkinElmer).
- Crop via le aree dell'immagine che non contengono la struttura di interesse (Image -> Focus estesa -> Seleziona area di interesse utilizzando 'Freehand ROI' Strumenti -> Actions -> Ritaglia alla selezione; Figura 2A). Nota, messa a fuoco estesa produce una z-piano di proiezione sullo schermo del computer per aiutare la vostra analisi, ma non influisce sui dati sottostanti, lo stesso vale per le regolazioni di luminosità e contrasto, se necessario).
- Misurare la lunghezza della zona analizzata utilizzando lo strumento Linea. La lunghezza della linea sarà calcolato e aggiunto alla tabella di dati (Figura 2B).
- Identificare gli oggetti utilizzando i 'oggetti per Intensity' filtro (modalità Misure). Specificare una soglia in modo tale che i cluster sono sinapticheevidenziato e non-sinaptico sfondo non è (Figura 2C). Inclusione di un marcatore indipendente synaptic etichettato con un altro colore può aiutare a identificare in maniera univoca sinapsi. Sinapsi Tipiche variano da pochi a qualche centinaio di voxel. Impostare la soglia di una volta utilizzando un campione di controllo, e utilizzarlo per tutti i campioni successivi. Thresholding ogni campione separatamente è inaccettabile perché introduce la possibilità di bias sperimentatore. Gli oggetti vengono selezionati e tabulati automaticamente (Figura 2D).
- Organizzare ed esportare i dati. Eliminare gli oggetti di 2 o meno voxel, poiché questi sono di solito granelli di sfondo. Questi possono essere eliminati per filtrazione ulteriori, o durante la successiva analisi. Per facilitare la loro valle di trasloco, riordinare i file di dati 'size oggetto' in modo che si raggruppano tutti. Esportare i dati in formato CSV, che possono essere aperti da Microsoft Excel o altri fogli di calcolo. Ogni immagine produce un file di output.
- Se Volocity morbidaware non è disponibile, i metodi alternativi per l'analisi Z-plane proiezioni di dati confocale possono essere utilizzati (per esempio rif. 7, o l'analisi manuale, con ImageJ), anche se a 3 dimensioni la perdita di informazioni (vedi Discussione).
4. Risultati rappresentativi
Il metodo di quantificazione presentate dovrebbero essere in grado di distinguere tra le popolazioni di cluster di differente luminosità e volumi diversi. Immagini rappresentative e corrispondenti dati quantitativi presentati in Figura 3 mostrano esempi di differenziazione sulla base di questi parametri. Come regola generale, i risultati devono essere conformi a quanto è evidente a occhio. Nel caso di UNC-49 del recettore GABA immunocolorazione il cavo ventrale nervo di C. elegans, tutti gli animali in genere è apparso molto simile in dimensioni e grado, e il totale dei valori sinaptici fluorescenza per un gruppo di cinque vermi (normalizzata alla lunghezza del cordone nervoso analizzato) ha mostrato errore standard valori di circa il 10% della media 4. Mutazione genetica e di altri trattamenti sperimentali (farmaco esposizione, ad esempio) possono alterare non solo il volume ed i valori di intensità e di distribuzioni di frequenza di cluster sinaptici, ma forse il tempo di sviluppo portate e la sincronia dei vermi, che porta ad una maggiore variabilità. Tuttavia, i risultati quantitativi deve sempre rispecchiare ciò che si può apprezzare visivamente e in caso contrario, l'ispezione delle immagini e gli oggetti individuati dalla Volocity dovrebbe rivelare dove è verificato l'errore e suggerire azioni correttive, come re-soglia o la rimozione di oggetti artefattuali.

Figura 1. Preparazione di sincrona C. culture elegans. culture sincroni sono ottenuti da questa procedura perché C. arresti di sviluppo elegans e la fase larvale L1 in assenza di cibo, e riprende quando il cibo viene introdotto.
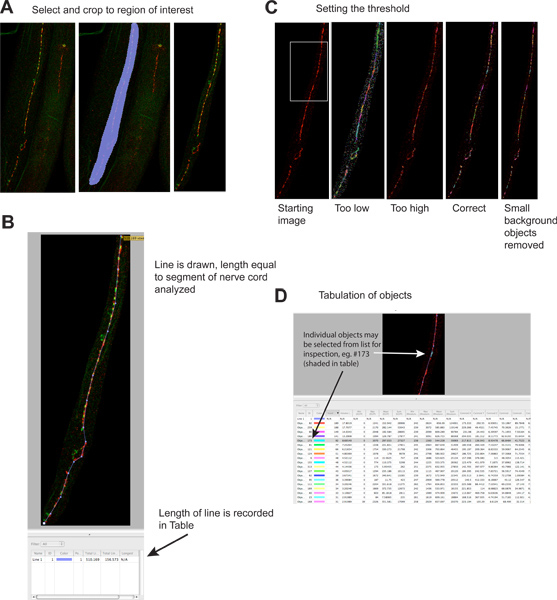
Figura 2. La quantificazione del puncta sinaptica.
- Regioni estranee dell'immagine vengono tagliate per ridurre le dimensioni dei file e aumentare la specificità. Pannello a sinistra mostra l'immagine di partenza, pannello centrale mostra regione selezionata, pannello di destra mostra l'immagine dopo il ritaglio. Il rosso è il segnale UNC-49 immunofluorescenza del recettore GABA, il segnale verde è sinaptobrevina-GFP (un marcatore presinaptico), espresso in neuroni presinaptici GABA 4. Clicca qui per ingrandire la figura .
- Una linea è disegnata la lunghezza del segmento cordone nervoso da analizzare, e la sua lunghezza è automaticamente registrato nella tabella dei risultati. Questa informazione è necessaria per normalizzare i dati sinaptici a causa della lunghezza del cordone nervoso analizzabile, che può variare diverse volte, se i vermi diventano frammentati durante la colorazione.www.jove.com/files/ftp_upload/4090/4090fig2blarge.jpg "target =" _blank "> Clicca qui per ingrandire la figura.
- Una soglia viene applicato per identificare singoli gruppi sinaptici. Pannello a sinistra mostra l'immagine prima della soglia, esempi di diversi livelli di thresholding sono mostrati in pannelli separati (segnale verde omesso). Ogni oggetto identificato è raffigurato sull'immagine come una regione colorata. Si noti la corrispondenza tra i cluster visivamente evidenti sinaptiche in più a sinistra del pannello e le regioni colorati sul più a destra del pannello. Clicca qui per ingrandire la figura .
- Ogni oggetto è identificato elencati separatamente nella tabella dei risultati. Singoli oggetti possono essere selezionati e sono evidenziate sull'immagine per l'ispezione se lo si desidera. Si noti che la tabella contiene le informazioni per il rosso ('rodamina') e verde ('EGFP') canali, le informazioni del canale verde è potenzialmente fuorviante dato che gli oggetti eranoidentificato basata strettamente sul fluorescenza rossa. Questa analisi può essere eseguita anche su immagini a colori singoli. Clicca qui per ingrandire la figura .
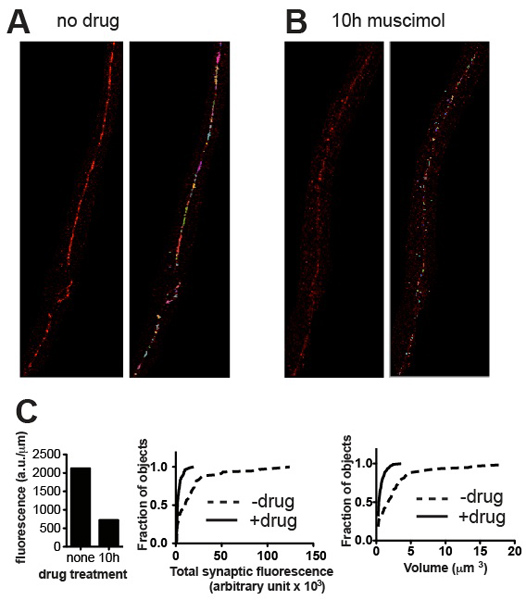
Figura 3. Risultati rappresentativi. Micrografie rappresentative provenienti da wild-type C. elegans colorati per UNC 49 recettori GABA prima (A) e dopo (B) trattamento con muscimolo, un agonista del recettore GABA che provoca recettori per diventare downregulated dopo lunga esposizione. I pannelli mostrano immagini ritagliate prima (a sinistra) e dopo (a destra) della soglia e la rimozione di macchie di fondo di piccole dimensioni. (C) Terreni di parametri quantitativi sinaptici per i modelli di cui (A) e (B): fluorescenza normalizzata alla lunghezza totale del nervo cavo (a sinistra), e istogrammi probabilità cumulativa di singoli contenuti fluorescenza sinapsi (al centro) e il volume sinapsi (right), che evidenzia una riduzione statisticamente significativa del contenuto sinaptica e il volume indotta dall'esposizione agonisti (n = 60 sinapsi per non trattate, n = 115 sinapsi per muscimolo trattati, P <0,001, test di Kolmogorov-Smirnov http://www.physics.csbsju .edu / stats / KS-test.html ).
Discussione
Il metodo qui presentato è stato progettato per estrarre quantitativi multi-parametri dati per grandi popolazioni di sinapsi in C. elegans, massimizzando al contempo la coerenza all'interno dei gruppi di trattamento. Tre caratteristiche contribuiscono a questi obiettivi. In primo luogo, immunostaining viene eseguita su popolazioni sincroni a vite senza fine di garantire che tutti gli animali sono la stessa età. Questo passaggio è fondamentale dato che il regolamento per lo sviluppo dei livelli di espress...
Divulgazioni
Non ci sono conflitti di interesse dichiarati.
Riconoscimenti
Gli autori desiderano ringraziare A. Benham per l'assistenza allo sviluppo del protocollo. Questo lavoro è stato finanziato dal NIH concessione NS06747 al BAB
Materiali
| Name | Company | Catalog Number | Comments | ||||||
| Nome del software | Azienda | Commenti (opzionale) | |||||||
| Volocity v4.0 o superiore | PerkinElmer / Improvision | Controlla il tuo impianto locale nucleo di imaging per l'accesso a questo software. Il software demo è disponibile sul sito PerkinElmer. Questo metodo richiede solo il modulo La determinazione quantitativa di Volocity. | |||||||
| Tabella 2. Reagenti e attrezzature specifiche. | |||||||||
| |||||||||
| Tabella 1. Solutions. |
Riferimenti
- Christensen, M. A primary culture system for functional analysis of C. elegans neurons and muscle cells. Neuron. 33, 503-514 (2002).
- Lewis, J. A., Fleming, J. T. Basic culture methods. Methods Cell Biol. 48, 3-29 (1995).
- Bettinger, J. C., Lee, K., Rougvie, A. E. Stage-specific accumulation of the terminal differentiation factor LIN-29 during Caenorhabditis elegans development. Development. 122, 2517-2527 (1996).
- Davis, K. M. Regulated lysosomal trafficking as a mechanism for regulating GABAA receptor abundance at synapses in Caenorhabditis elegans. Mol. Cell Neurosci. 44, 307-317 (2010).
- Finney, M., Ruvkun, G. The unc-86 gene product couples cell lineage and cell identity in C. elegans. Cell. 63, 895-905 (1990).
- Rowland, A. M. Presynaptic terminals independently regulate synaptic clustering and autophagy of GABAA receptors in Caenorhabditis elegans. J. Neurosci. 26, 1711-1720 (2006).
- Burbea, M. Ubiquitin and AP180 regulate the abundance of GLR-1 glutamate receptors at postsynaptic elements in C. elegans. Neuron. 35, 107-120 (2002).
- Miesenbock, G., De Angelis, D. A., Rothman, J. E. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins. Nature. 394, 192-195 (1998).
- Oda, S., Tomioka, M., Iino, Y. Neuronal plasticity regulated by the insulin-like signaling pathway underlies salt chemotaxis learning in Caenorhabditis elegans. J. Neurophysiol. 106, 301-308 (2011).
- Sankaranarayanan, S. The use of pHluorins for optical measurements of presynaptic activity. Biophys. J. 79, 2199-2208 (2000).
- McDonald, N. A. Generation and functional characterization of fluorescent, N-terminally tagged CB1 receptor chimeras for live-cell imaging. Mol. Cell Neurosci. 35, 237-248 (2007).
- Shakiryanova, D. Synaptic neuropeptide release induced by octopamine without Ca2+ entry into the nerve terminal. Proc. Natl. Acad. Sci. U. S. A. 108, 4477-4481 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon