Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Induction de murin inflammation intestinale par transfert adoptif de effecteur CD4
Dans cet article
Résumé
Here, we present a protocol to induce colonic inflammation in mice by adoptive transfer of syngeneic CD4+CD45RBhigh T cells into T and B cell deficient recipients. Clinical and histopathological features mimic human inflammatory bowel diseases. This method allows the study of the initiation of colonic inflammation and progression of disease.
Résumé
Il ya beaucoup de différents modèles animaux disponibles pour étudier la pathogenèse des maladies humaines inflammatoires de l'intestin (MICI), chacun avec ses propres avantages et inconvénients. Nous décrivons ici un modèle de colite expérimentale qui est initié par transfert adoptif de cellules de haute T CD4 + spléniques syngéniques CD45RB dans les souris receveuses déficientes cellules T et B. La haute T population de cellules CD4 + CD45RB qui se compose essentiellement de cellules effectrices naïfs est capable d'induire une inflammation intestinale chronique, ressemblant étroitement à des aspects clés de l'IBD humaine. Cette méthode peut être manipulé pour étudier les aspects apparition et la progression de la maladie. En outre, il peut être utilisé pour étudier la fonction de innée et adaptative, et les populations immunitaires de régulation cellulaire, et le rôle des expositions environnementales, ce est à dire, le microbiote, dans l'inflammation intestinale. Dans cet article, nous illustrons la méthode pour induire une colite avec un protocole étape par étape. Ce incLudes une vidéo de démonstration des aspects techniques clés nécessaires pour développer avec succès ce modèle murin de colite expérimentale à des fins de recherche.
Introduction
The inflammatory bowel diseases (IBD) Crohn’s disease and ulcerative colitis result from an incompletely defined and complex interaction between host immune responses, genetic susceptibility, environmental factors, and the enteric luminal contents1. Recent genome-wide association studies report associations between immune cell regulatory genes and IBD susceptibility2,3. Both innate and adaptive immune cell intrinsic genes are represented in these studies, indicating a central role for these cell populations in IBD pathogenesis.
There currently exist more than 50 animal models of human IBD. While no one model perfectly phenocopies human IBD, many are useful for studying various aspects of human disease, including disease onset and progression and the wound-healing response. In the method described here, intestinal inflammation is initiated with syngeneic splenic CD4+CD45RBhigh T cell adoptive transfer into T and B cell deficient recipient mice4. The CD4+CD45RBhigh T cell population contains mainly naïve T cells primed for activation that are capable of inducing chronic small bowel and colonic inflammation. This method allows the researcher to modify key experimental variables, including both innate and adaptive immune cell populations, to answer biologically relevant questions relating to disease pathogenesis. Additionally, this method provides precise initiation of disease onset and a well-characterized experimental time course. This permits the kinetic study of clinical features of disease progression in mice. Intestinal inflammation induced by this method shares many features with human IBD, including chronic large and small bowel transmural inflammation, pathogenesis driven by cytokines such as TNF and IL-12, and systemic symptoms such as wasting5. Thus, it is an ideal model system for studying the pathogenesis of human IBD.
The method here describes in detail the protocol for inducing experimental colitis by adoptive transfer of CD4+CD45RBhigh T cells into Rag1-/- mice. We discuss key technical steps, expected results, optimization, and trouble-shooting. We address the required elements for the successful development of this murine model of intestinal inflammation for research purposes.
Access restricted. Please log in or start a trial to view this content.
Protocole
REMARQUE: Assurez-vous que tous les protocoles d'animaux sont approuvés par et en conformité avec institutionnel de protection des animaux et l'utilisation du Comité (IACUC) règlements et le Guide du Conseil national de recherches pour le soin et l'utilisation des animaux de laboratoire. Les souris donneuses peuvent être soit mâle ou femelle, mais les souris receveuses doivent être de sexe masculin. Si les bénéficiaires de sexe féminin doivent être utilisés, les souris les donateurs doivent être des femmes 5. Maintenir colonies utilisant régulière, literie non stérile et l'eau non acidifié, car ils peuvent affecter la microflore intestinale, et, par conséquent, le phénotype de la colite de la souris 5,6.
1. Préparation expérimentale
- Utiliser les médias et les tampons glacées. Gardez cellules sur la glace pendant toute l'expérience.
- Réaliser l'expérience dans le capot de risque biologique stérile en utilisant une technique stérile.
2. Isolement des cellules T spléniques
- Euthanasier souris donneuse / souris de CO 2 chambre suivies par dislocatio col de l'utérusn. Pulvériser abdomen avec 70% d'éthanol.
- Faire une incision horizontale dans l'abdomen et décoller la peau pour exposer péritoine. Tenez péritoine loin des organes internes avec la pince et faire une incision dans le péritoine abdominal gauche pour exposer et exciser la rate.
- Placez la rate dans 10 ml de médias complète dans une boîte de Petri. Retirez et jetez l'excès de tissu de la rate.
- Utilisez deux lames de verre stérilisés pour écraser et de démêler la rate en suspension à cellule unique. suspension de cellules de filtre à travers un tamis de 70 um dans un tube conique de 50 ml et rincer filtre avec 5 ml de milieu complet. Placez jusqu'à 5 rates dans un tube conique de 50 ml.
- Centrifuger les cellules à 450 g pendant 7 min. Rejeter le surnageant en versant à pied ou par aspiration sous vide dans un conteneur à déchets.
- Resuspendre doucement les cellules dans 5 ml par rate de tampon de lyse pour lyser les globules rouges pendant 10 minutes à température ambiante. Ajouter un volume égal de milieu complet (5 ml par rate) au tube.
- centrifuger les cellules à 450 g pendant 7 min. Rejeter le surnageant en versant à pied ou par aspiration sous vide dans un conteneur à déchets.
- Resuspendre doucement les cellules dans 10 ml de Labeling Buffer.
- Compter les cellules par exclusion au bleu trypan.
- Retirer 20 ul de suspension cellulaire et ajouter 180 ul bleu trypan à un tube de centrifugeuse et bien mélanger. Après 5 min, ajouter 10 ul cellules hémocytomètre et compter les cellules non-bleu sous le microscope étiquetés. Déterminer le nombre total de cellules viables. Jeter cellules / trypan bleu mix.
- Centrifuger les cellules dans 10 ml de Labeling Buffer à 450 g pendant 7 min. Rejeter le surnageant en versant à pied ou par aspiration sous vide dans un conteneur à déchets.
3. Enrichissement de cellules T CD4 +
REMARQUE: Suivez les instructions du fabricant pour des produits spécifiques utilisés dans cette section.
- Resuspendre doucement les cellules à une suspension à cellule unique de 20 x 10 6 cellules / ml dans du froid ÉtiquetteBuffer ing.
- Ajouter 5 ul par 1 x 10 6 cellules d'anticorps d'enrichissement de cellules T CD4 biotinylé. Incuber les cellules sur de la glace pendant 15 min.
- Ajouter 10x volume de Labeling Buffer. Centrifuger les cellules à 450 g pendant 7 min. Aspirer soigneusement tout le surnageant par aspiration sous vide dans un conteneur à déchets.
- Bien agiter au vortex particules magnétiques de streptavidine conjugué. Ajouter 5 ul de particules par 1 x 10 6 cellules.
- Mélanger soigneusement mais doucement. Gardez mélange à 6-12 ° C pendant 30 min.
- Ajouter étiquetage tampon à une concentration de 20 à 80 x 10 6 cellules / ml. Transfert jusqu'à 1,0 ml des cellules marquées par 12 x 75 mm tube à essai à fond rond (dénommé "tube-positif fraction»).
- Placez chaque tube positif fraction sur l'aimant pour les 6-8 min.
- Avec les tubes positif fraction encore sur l'aimant, transférer soigneusement surnageant à partir du tube positif fraction avec pipette Pasteur en verre à une nouvelle stériles 50 ml tube conique (referred pour que la "fraction enrichie»). Cette fraction enrichie contient des cellules T CD4 +. Veillez à ne pas perturber les cellules marquées attirés par l'aimant.
- Resuspendre les cellules laissées dans les tubes positif fraction dans le même volume de Labeling Buffer comme dans l'étape 3.6 par aspiration et vigoureusement vers le bas. Placez le tube positif fraction de retour sur aimant pour les 6-8 min.
- Avec tubes positif fraction encore sur l'aimant, transférer soigneusement surnageant (fraction enrichie, CD4 +) du tube positif fraction de verre Pasteur pipette pour stérile de 50 ml tube conique de l'étape 3.8 sans perturber les cellules attachées à l'aimant.
- Répéter les étapes 3.9 à 3.10 pour augmenter le rendement en cellules T CD4 + obtenues. Continuer protocole utilisant fraction enrichie (CD4 + cellules).
4. L'étiquetage et le tri des cellules 7
- Centrifugeuse enrichi cellules à 450 g pendant 7 min. Rejeter le surnageant en versant hors tension ou sous vide SUCTions dans un conteneur à déchets.
- Resuspendre les cellules dans 1 ml de Labeling Buffer. Retirer aliquote de cellules pour compter et pour évaluer la viabilité des cellules par exclusion au bleu trypan comme à l'étape 2.9.
- Ajouter un volume de Labeling Buffer à 10 x 10 6 cellules / ml; si les cellules sont déjà <10 x 10 6, centrifuger à 450 g pendant 7 min, éliminer le surnageant en versant off ou d'aspiration sous vide dans un conteneur à déchets, et d'ajouter volume de Labeling Buffer à 10 x 10 6 cellules / ml.
- Mettre en place des portions séparées d'environ 5 à 10 x 10 5 cellules chacune des cellules de contrôle non colorées, isotype teinté et simples teinté dans des microtubes.
- Ajouter 5 ug / ml de CD4-FITC et 1 ug / ml de CD45RB-PE aux cellules. Ajouter taches de contrôle isotypique et les taches simples à la même concentration se approprier aliquotes dans des microtubes. Mélangez bien mais doucement et incuber sur de la glace abri de la lumière pendant 30 min.
- Ajouter 10x volume de Labeling Buffer aux cellules et le contrôles. Centrifugeuse 450 xg pendant 7 min. Rejeter le surnageant par aspiration sous vide dans un conteneur à déchets.
- Remettre en suspension dans Labeling Buffer au volume à l'étape 4.5. Centrifugeuse 450 xg pendant 7 min. Rejeter le surnageant par aspiration sous vide dans un conteneur à déchets.
- Remettre en suspension dans Labeling Buffer à 10 x 10 6 cellules / ml. Passez cellules à 70 um passoire dans FACS tube. Garder sur la glace abri de la lumière jusqu'au moment de FACS.
- Mettre en place et déterminer la rémunération appropriée sur le trieur de cellules avec des cellules non colorées et des contrôles simples teinté.
- Exclure les cellules non viables avec déclenchement prospectifs et dispersion latérale (figure 1A). Mettre en place gating pour CD4 + et + CD45RB cellules avec contrôles isotypiques teinté. Cellules porte sur la population CD4 +.
- Trier cellules CD4 + dans CD45RB élevé et CD45RB faibles populations en utilisant un histogramme simple pour les cellules de PE teinté dans des tubes avec 2 ml de milieu complet. Le CD45RB haute population représente le plus haut de 40% des CD4 + CD45RB + cellules (CD45RB haute), et de la faible densité de population est la plus faible CD45RB 20% des cellules CD4 + CD45RB + (CD45RB bas, figure 1B).
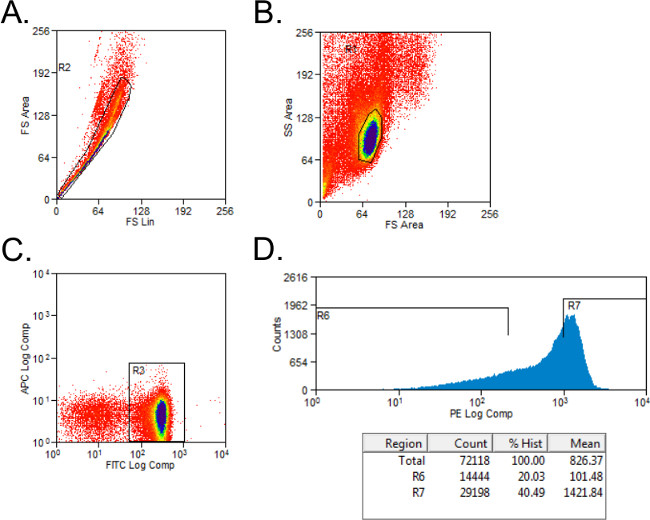
Figure 1: flux Représentant cytométrie parcelles de populations de cellules CD4 + CD45RB T lors de l'analyse FACS (A - C) FITC CD4 et PE CD45RB-taché splénocytes de donneurs C57BL / 6 ont été triées par FACS en CD4 + CD45RB élevé et CD4 +. populations de cellules T CD45RB bas. Événements (A) Doublet ont été exclus sur le nuage de points avant. (B) Les lymphocytes ont été bloqués dans le complot de l'avant et la diffusion latérale. (C) CD4 + cellules T ont été bloqués, et les cellules (D) CD4 + CD45RB + T ont été reportées sur un PE par rapport Events histogramme. Cellules CD4 + CD45RB faibles ont été considérés comme la plus faible de 20% des cellules CD45RB +. Cellules CD4 + CD45RB élevés ont été définis comme la plus haute de 40% des cellules CD45RB +.
- Exécuter une aliquote de chaque population de cellules sur la machine FACS pour évaluer la pureté des populations.
- Centrifugeuse triée cellules à 450 g pendant 7 min. Remettre en suspension dans 1 ml de PBS. Retirer aliquote de cellules pour compter et pour évaluer la viabilité des cellules par exclusion au bleu trypan comme à l'étape 2.9.
5. injection de cellules dans des receveurs
- Remettre en suspension les cellules triées à 4 x 10 6 cellules / ml (CD45RB haut) ou 2 x 10 6 cellules / ml (CD45RB bas) dans du PBS.
- Transférer 100 ul de cellules de haute CD45RB par bénéficiaire dans un nouveau tube stérile. Ajouter 100 ul de PBS pardestinataire de ce tube. Ainsi, le volume total d'injection par animal est de 200 pi, et le montant total de cellules par bénéficiaire est de 4 cellules T naïves élevés x 10 5 CD4 + CD45RB.
- Si groupe expérimental recevant cellules régulatrices T est souhaitée, transférer 100 pi de cellules élevées CD45RB par bénéficiaire dans un nouveau tube stérile. Ajouter 100 pi de cellules par CD45RB faible destinataire dans le même tube. Volume d'injection total par animal est de 200 pi; rapport de CD45RB haute: CD45RB cellules faibles est de 2: 1.
- Injecter 100 ul de CD45RB élevé ou CD45RB high / low CD45RB cellules CD4 + par voie intrapéritonéale dans le droit et le côté gauche de l'abdomen (total de 200 pi) de chaque destinataire.
6. Suivi de progression de la maladie chez les animaux receveurs
- Pour évaluer l'état clinique des animaux receveurs, attribuer des notes cliniques globales pour la paramete suivanters 8 le jour de l'injection, toutes les semaines par la suite, et au moment du sacrifice:
- Déterminer perdre en mesurant la perte de poids: 0 - aucune perte de poids; Perte de poids corporel initial de 0,1 à 10% de - 1; 2 - La perte de plus de 10% du poids corporel initial (figure 2A).

Figure 2: signes pathologiques cliniques et bruts de l'inflammation se produisent après le transfert de cellules de haute T CD4 + CD45RB de type sauvage dans Rag1 - / - et NRDKO souris receveuses 11 (A) bénéficiaires NRDKO perdu en moyenne 10% de leur poids corporel initial de 5. semaines après le transfert, alors que Rag1- / - Les destinataires ne ont pas présenté de signes cliniques de la maladie à ce moment. Chaque point représente le pourcentage moyen de poids corporel initial pour la cohorte ± SEM. **, P <0,005. (B) Certaines souris ont développé une inflammation intestinale sévère, comme en témoigne la présence d'un prolapsus rectal. Ce est une image représentative de prolapsus rectal chez un receveur souris NRDKO. (C) Grossièrement, deux points à la fois Rag1 - / - et les souris receveuses NRDKO sont épaissies et raccourci par rapport à deux points de Rag1 - / - et souris sans NRDKO cellules T transfert adoptif. Colons de souris receveuses NRDKO montrent une inflammation sévère et le poids du côlon augmentation (données non présentées). Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.
- Déterminer la qualité des selles en plaçant l'animal dans un récipient propre jusqu'à ce qu'il ait passéselles: 0 - aucun; 1 - selles molles; 2 - selles liquides et / ou sanglante.
- Déterminer la santé générale de l'animal par la présence des signes de maladie suivants: 0 - pas une posture voûtée, poils de fourrure, la peau ou des lésions; 1 - l'un quelconque des présents suivante: posture voûtée, fourrure à poils, ou des lésions de la peau.
- Déterminer la présence d'un prolapsus rectal: 0 - absent; 1 - présente (figure 2B).
- Sacrifier des animaux par inhalation de CO 2 suivie par dislocation cervicale quand ils ont perdu 20% de leur poids corporel de départ ou au point de temps désiré, selon la première éventualité. Maladie clinique est habituellement apparente à partir de 5 semaines post-réplétion.
- Évaluer la gravité de la maladie, comme décrit précédemment 5,7.
- Attribuer des notes cliniques que dans l'étape 6.1 8.
- Mesurer la longueur du côlon et du poids (figure 2C).
- Effectuer l'analyse histologique de l'inflammation5.
- Déterminer l'expression de cytokine spontanée dans les cultures d'explants intestinaux de tissu 9, 10 nœuds lymphatiques mésentériques, et / ou le sérum 11.
- En bref, pour les cultures d'explants neuf, retirer deux points après le sacrifice, ouverte longitudinalement, et propre avec du PBS. Incuber deux points sur un agitateur orbital réglé à 250 rpm dans un milieu complet pendant 30 min à température ambiante.
- Hacher le tissu en petits morceaux et incubées à 37 ° C dans un milieu complet pendant 24 heures. Recueillir le surnageant et utiliser pour la quantification de cytokines par 100 mg de tissu par ELISA.
- Effectuer la caractérisation ex vivo des phénotypes et / ou de la fonction 10 cellules T.
Access restricted. Please log in or start a trial to view this content.
Résultats
Environ 10 x 10 6 cellules CD4 + CD45RB de 10 rates de souris donneuses adultes C57BL / 6 de haute T sont fiable isolé. Ce nombre varie en fonction de l'âge et de la souche de la souris entre les donateurs et les compétences du chercheur. Lorsque 4 x 10 5 C57BL / 6 cellules CD4 + CD45RB haute T sont transférés dans C57BL / 6 Rag1 - / - souris receveuses, des signes cliniques de la maladie apparaissent autour de la semaine 5 po...
Access restricted. Please log in or start a trial to view this content.
Discussion
Nous décrivons ici un protocole étape par étape induisant une inflammation du côlon chez la souris par transfert adoptif de cellules T CD4 + CD45RB + T dans des souris immunodéficientes. Nous avons utilisé des rates C57BL / 6 donateurs et Rag1 syngénique - / - souris receveuses, bien que d'autres souches (par exemple, BALB / c, 129S6 / SvEv, diabétiques non obèses (NOD)) modèles et génétiques de l'immunodéficience (par exemple, SCID, Ra...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
The authors declare that they have no competing financial interests.
Remerciements
Ce travail a été soutenu par American Gastroenterological Association (AGA) Scholars Research Award et la maladie de Crohn et la colite Foundation of America (CCFA) Award développement de carrière (à SPR), NIH NIDDK F30 DK089692 (ECS), et l'Université de Centre de la Caroline du Nord pour la biologie gastro- et les maladies Grant P30 DK34987 (histologie). Le Fonds pour l'UNC cytométrie en flux de base est financé en partie par une subvention de soutien de base NCI Center (P30CA016086) à l'UNC Lineberger Comprehensive Cancer Center. Nous remercions Luc B. Borst de North Carolina State University College de médecine vétérinaire pour son aide à l'analyse histopathologique et immunohistochimie.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Name of Reagent/ Equipment | Company | Catalog Number | Comments/Description |
| 10x PBS | Gibco | 14200075 | |
| 12 mm x 75 mm round-bottom tube | Falcon | 352052 | |
| 15 ml conical | Corning | 430790 | |
| 26 G x 3/8 Needle | BD Biosciences | 305110 | |
| 50 ml conical | Corning | 430828 | |
| 70 μm Cell Strainer | Fisherbrand | 22363548 | |
| BD IMagnet | BD Biosciences | 552311 | |
| β-mercaptoethanol | Thermo Scientific | 35602 | |
| CD4-FITC IgG2b | eBioscience | 11-0041 | |
| CD45RB-PE IgG2a | BD Pharminogen | 553101 | |
| Complete Media | RPMI-1640, 1% Pen/Strep, 10% FBS, 0.0004% β-ME | ||
| FACS tube + strainer | BD Falcon | 352235 | |
| Glass Microscope Slides | Fisherbrand | 12550A3 | |
| Heat-inactivated FBS | Gemini | 100-106 | |
| Labeling Buffer | 1x PBS, 0.5% BSA, 2 mM EDTA | ||
| Lysis Buffer | 0.08% NH4Cl, 0.1% KHCO3, 1 mM EDTA | ||
| MoFlo XDP | Beckman Coulter | ||
| Mouse CD4 T lymphocyte Enrichment Set - DM | BD Biosciences | 558131 | |
| Mouse IgG2a-PE | BD Pharminogen | 553457 | |
| Mouse IgG2b-FITC | eBioscience | 11-4732 | |
| Pasteur pipet | Fisherbrand | 13-678-20D | |
| Penicillin-Streptomycin Solution, 100X | Corning Cellgro | 30-002-CI | |
| [header] | |||
| Petri Dish | Fisherbrand | 875713 | |
| Pure Ethanol 200 Proof | Decon Labs | 2705-HC | |
| RPMI-1640 | Gibco | 11-875-093 | |
| Syringe | BD Biosciences | 309597 | |
| Trypan blue | Corning Cellgro | 25-900-CI | |
| Wash Media | RPMI-1640, 1% Pen/Strep, 0.0004% β-ME | ||
Références
- Xavier, R. J., Podolsky, D. K. Unravelling the pathogenesis of inflammatory bowel disease. Nature. 448 (7152), 427-434 (2007).
- Cho, J. H., Brant, S. R. Recent insights into the genetics of inflammatory bowel disease. Gastroenterology. 140 (6), 1704-1712 (2011).
- Jostins, L., et al. Host-microbe interactions have shaped the genetic architecture of inflammatory bowel disease. Nature. 491 (7422), 119-124 (2012).
- Powrie, F., Leach, M. W., Mauze, S., Caddle, L. B., Coffman, R. L. Phenotypically distinct subsets of CD4+ T cells induce or protect from chronic intestinal inflammation in C. B-17 scid mice. Int Immunol. 5 (11), 1461-1471 (1993).
- Ostanin, D. V., et al. T cell transfer model of chronic colitis: concepts, considerations, and tricks of the trade. Am J Physiol Gastrointest Liver Physiol. 296 (2), 135-146 (2009).
- Ma, B. W., et al. Routine habitat change: a source of unrecognized transient alteration of intestinal microbiota in laboratory mice. PLoS One. 7 (10), e47416(2012).
- Read, S., Powrie, F. Induction of inflammatory bowel disease in immunodeficient mice by depletion of regulatory T cells. Curr Protoc Immunol. Chapter 15 (Unit 15 13), (1999).
- Maillard, M. H., et al. The Wiskott-Aldrich syndrome protein is required for the function of CD4(+)CD25(+)Foxp3(+) regulatory T cells. J Exp Med. 204 (2), 381-391 (2007).
- Hegazi, R. A., et al. Carbon monoxide ameliorates chronic murine colitis through a heme oxygenase 1-dependent pathway. J Exp Med. 202 (12), 1703-1713 (2005).
- Kole, A., et al. Type I IFNs regulate effector and regulatory T cell accumulation and anti-inflammatory cytokine production during T cell-mediated colitis. J Immunol. 191 (5), 2771-2779 (2013).
- Kobayashi, T., et al. NFIL3-deficient mice develop microbiota-dependent, IL-12/23-driven spontaneous colitis. J Immunol. 192 (4), 1918-1927 (2014).
- Steinbach, E. C., et al. Innate PI3K p110delta Regulates Th1/Th17 Development and Microbiota-Dependent Colitis. J Immunol. 192 (8), 3958-3968 (2014).
- Kobayashi, T., et al. NFIL3 is a regulator of IL-12 p40 in macrophages and mucosal immunity. J Immunol. 186 (8), 4649-4655 (2011).
- Leach, M. W., Bean, A. G., Mauze, S., Coffman, R. L., Powrie, F. Inflammatory bowel disease in C.B-17 scid mice reconstituted with the CD45RBhigh subset of CD4+ T cells. Am J Pathol. 148 (5), 1503-1515 (1996).
- Powrie, F., et al. Inhibition of Th1 responses prevents inflammatory bowel disease in scid mice reconstituted with CD45RBhi CD4. T cells. Immunity. 1 (7), 553-562 (1994).
- Read, S., Malmstrom, V., Powrie, F. Cytotoxic T lymphocyte-associated antigen 4 plays an essential role in the function of CD25(+)CD4(+) regulatory cells that control intestinal inflammation. J Exp Med. 192 (2), 295-302 (2000).
- Rogers, G. B., et al. Functional divergence in gastrointestinal microbiota in physically-separated genetically identical mice. Sci Rep. 4, 5437(2014).
- Fukata, M., et al. The myeloid differentiation factor 88 (MyD88) is required for CD4+ T cell effector function in a murine model of inflammatory bowel disease. J Immunol. 180 (3), 1886-1894 (2008).
- Kurtz, C. C., et al. Extracellular adenosine regulates colitis through effects on lymphoid and nonlymphoid cells. Am J Physiol Gastrointest Liver Physiol. 307 (3), 338-346 (2014).
- Naganuma, M., et al. Cutting edge: Critical role for A2A adenosine receptors in the T cell-mediated regulation of colitis. J Immunol. 177 (5), 2765-2769 (2006).
- Ranatunga, D. C., et al. A protective role for human IL-10-expressing CD4+ T cells in colitis. J Immunol. 189 (3), 1243-1252 (2012).
- Srikrishna, G., et al. Carboxylated glycans mediate colitis through activation of NF-kappa. B. J Immunol. 175 (8), 5412-5422 (2005).
- Wang, F., et al. IFN-gamma-induced TNFR2 expression is required for TNF-dependent intestinal epithelial barrier dysfunction. Gastroenterology. 131 (4), 1153-1163 (2006).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon