É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Indução de Murino inflamação intestinal por transferência adotiva de Effector CD4
Neste Artigo
Resumo
Here, we present a protocol to induce colonic inflammation in mice by adoptive transfer of syngeneic CD4+CD45RBhigh T cells into T and B cell deficient recipients. Clinical and histopathological features mimic human inflammatory bowel diseases. This method allows the study of the initiation of colonic inflammation and progression of disease.
Resumo
Existem muitos modelos diferentes de animais disponíveis para estudar a patogênese de doenças humanas inflamatórias intestinais (DII), cada um com suas próprias vantagens e desvantagens. Descrevemos aqui um modelo de colite experimental que é iniciada por transferência adoptiva de células de baço singeneicas CD4 + CD45RB high T em ratinhos deficientes em receptores de células T e B. A população de células T CD4 + elevada CD45RB que consiste em grande parte de células efectoras naive é capaz de induzir a inflamação intestinal crónica, assemelhando-se intimamente os aspectos chave da DII humana. Este método pode ser manipulado para estudar os aspectos do aparecimento e progressão da doença. Além disso, ele pode ser utilizado para estudar a função de inata e adaptativa, e as populações de células imunitárias reguladoras, e o papel da exposição ambiental, ou seja, a microbiota, na inflamação intestinal. Neste artigo, ilustram a metodologia para a indução de colite com um protocolo passo-a-passo. Este includes um vídeo de demonstração de aspectos técnicos essenciais necessários para desenvolver com sucesso este modelo murino de colite experimental para fins de pesquisa.
Introdução
The inflammatory bowel diseases (IBD) Crohn’s disease and ulcerative colitis result from an incompletely defined and complex interaction between host immune responses, genetic susceptibility, environmental factors, and the enteric luminal contents1. Recent genome-wide association studies report associations between immune cell regulatory genes and IBD susceptibility2,3. Both innate and adaptive immune cell intrinsic genes are represented in these studies, indicating a central role for these cell populations in IBD pathogenesis.
There currently exist more than 50 animal models of human IBD. While no one model perfectly phenocopies human IBD, many are useful for studying various aspects of human disease, including disease onset and progression and the wound-healing response. In the method described here, intestinal inflammation is initiated with syngeneic splenic CD4+CD45RBhigh T cell adoptive transfer into T and B cell deficient recipient mice4. The CD4+CD45RBhigh T cell population contains mainly naïve T cells primed for activation that are capable of inducing chronic small bowel and colonic inflammation. This method allows the researcher to modify key experimental variables, including both innate and adaptive immune cell populations, to answer biologically relevant questions relating to disease pathogenesis. Additionally, this method provides precise initiation of disease onset and a well-characterized experimental time course. This permits the kinetic study of clinical features of disease progression in mice. Intestinal inflammation induced by this method shares many features with human IBD, including chronic large and small bowel transmural inflammation, pathogenesis driven by cytokines such as TNF and IL-12, and systemic symptoms such as wasting5. Thus, it is an ideal model system for studying the pathogenesis of human IBD.
The method here describes in detail the protocol for inducing experimental colitis by adoptive transfer of CD4+CD45RBhigh T cells into Rag1-/- mice. We discuss key technical steps, expected results, optimization, and trouble-shooting. We address the required elements for the successful development of this murine model of intestinal inflammation for research purposes.
Access restricted. Please log in or start a trial to view this content.
Protocolo
NOTA: Certifique-se que todos os protocolos de animais são aprovados por e em conformidade com Institutional Animal Care e do Comitê Use regulamentos (IACUC) e no Guia do Conselho Nacional de Pesquisa para a Conservação e Uso de Animais de Laboratório. Camundongos doadores podem ter sexo masculino ou feminino, mas ratos destinatário devem ser do sexo masculino. Se os destinatários do sexo feminino devem ser utilizados, os camundongos doadores têm que ser fêmeas 5. Manter colónias usando, roupa de cama não estéril normal e água não acidificado, uma vez que estes podem causar impacto na microbiota intestinal, e, portanto, o fenótipo dos ratinhos colite 5,6.
1. Preparação Experimental
- Use mídia e buffers geladas. Mantenha as células no gelo durante todo o experimento.
- Realizar o experimento na capa de risco biológico estéril usando técnica estéril.
2. Isolamento de células T do baço
- Sacrificá camundongo doador / mice em CO 2 câmara seguido por dislocatio cervicaln. Spray de abdômen com etanol 70%.
- Faça uma incisão horizontal no abdômen e descascar a pele para expor peritônio. Segure peritônio longe dos órgãos internos com a pinça e fazer uma incisão no peritônio abdominal esquerda para expor e extirpar o baço.
- Inserir o baço em 10 ml de Meios completa em uma placa de Petri. Retire e elimine o excesso de tecido do baço.
- Use duas lâminas de vidro esterilizados para esmagar e provocar uma separação baço em suspensão de uma única célula. Suspensão de células Filtrar através de um filtro de 70 mm em um tubo de 50 ml e lavar filtro com 5 ml de Meio Completo. Coloque até 5 baços em um tubo de 50 ml cônico.
- Centrifugar células a 450 xg por 7 min. Elimine o sobrenadante por vazamento off ou por sucção a vácuo em um recipiente de resíduos.
- Suavemente ressuspender as células em 5 ml por baço de um tampão de lise, para lisar as células vermelhas do sangue durante 10 min à temperatura ambiente. Adicionar um volume igual de meio completo (5 ml por baço) para o tubo.
- células centrifugar a 450 xg por 7 min. Elimine o sobrenadante por vazamento off ou por sucção a vácuo em um recipiente de resíduos.
- Volte a suspender suavemente as células em 10 ml de Etiquetagem Buffer.
- Contagem de células por exclusão de azul de tripano.
- Retirar 20 l de suspensão de células e adicionar 180 mL de azul tripano para um tubo de microcentrífuga e misture bem. Após 5 min, adicionar 10 ul de células e contagem em hemocitómetro células não-azul sob o microscópio rotulados. Determinar o número total de células viáveis. Descarte de células / trypan azul mix.
- Centrifugar células em 10 ml de Etiquetagem tampão a 450 xg por 7 min. Elimine o sobrenadante por vazamento off ou por sucção a vácuo em um recipiente de resíduos.
3. O enriquecimento de células T CD4 +
NOTA: Siga as instruções do fabricante para produtos específicos utilizados nesta seção.
- Suavemente ressuspender as células a uma suspensão de uma única célula de 20 x 10 6 células / ml em Etiqueta frioing Buffer.
- Adicionar 5 mL por 1 x 10 6 células de anticorpos de enriquecimento de células T CD4 bioestanhados. Incubar as células em gelo durante 15 min.
- Adicionar 10x volume de Etiquetagem Buffer. Centrifugar células a 450 × g por 7 min. Aspirar cuidadosamente todo o sobrenadante usando sucção a vácuo em um recipiente de resíduos.
- Absolutamente vortex partículas magnéticas de estreptavidina conjugada. Adiciona-se 5 ul de partículas por 1 x 10 6 células.
- Misture bem, mas com cuidado. Mantenha a mistura a 6-12 ° C durante 30 min.
- Adicionar Etiquetagem Tampão a uma concentração de 20-80 x 10 6 células / ml. Transferir-se para 1,0 ml de células marcadas por 12 x 75 mm Tubo de ensaio de fundo redondo (referido como o "tubo-fracção positiva").
- Coloque cada tubo fração positivo no ímã para 6-8 min.
- Com os tubos de fração positiva ainda no ímã, transferir cuidadosamente sobrenadante do tubo-fração positiva com Pasteur de vidro pipeta para uma nova estéreis tubo de 50 ml (referred como a "fracção enriquecida"). Esta fração enriquecida contém células T CD4 +. Tenha cuidado para não perturbar as células marcadas atraídos pelo ímã.
- Suspenda as células deixaram nos tubos-fração positiva no mesmo volume de Etiquetagem tampão como no passo 3,6 por pipetagem cima e para baixo vigorosamente. Coloque tube-fração positiva para trás em ímã para 6-8 min.
- Com tubos de fração positiva ainda no ímã, transferir cuidadosamente sobrenadante (fração enriquecida, CD4 +) a partir de tubo-fração positiva com vidro Pasteur pipeta para estéril tubo de 50 ml a partir do passo 3.8, sem perturbar as células unidas a ímã.
- Repita os passos 3,9-3,10 para aumentar o rendimento de células CD4 + T obtidas. Continuar protocolo utilizando fracção enriquecida (células CD4 +).
4. Rotulagem e Classificando Cells 7
- Centrífuga enriquecido células a 450 xg por 7 min. Elimine o sobrenadante por vazamento off ou suct vácuoíon no recipiente de resíduos.
- Ressuspender as células em 1 ml de Etiquetagem buffer. Remover alíquota de células para contar, para avaliar a viabilidade celular por exclusão com azul de tripano como no passo 2.9.
- Adicionar um volume de Etiquetagem Buffer para 10 x 10 6 células / ml; se as células já estão <10 x 10 6, centrifugar a 450 g durante 7 min, elimine o sobrenadante por vazamento off ou de sucção a vácuo em um recipiente de resíduos, e adicionar volume de Etiquetagem Buffer para 10 x 10 6 células / ml.
- Configure alíquotas separadas de aproximadamente 5-10 × 10 5 células cada para células de controlo não coradas, manchada de isótipo, e single-coradas em tubos de microcentrífuga.
- Adicionam-se 5 ug / ml de CD4-FITC e 1 ug / ml a CD45RB-PE para as células. Adicionar manchas de controlo de isotipo e as manchas individuais com a mesma concentração de alíquotas em tubos de microcentrífuga apropriados. Misture bem, mas com cuidado e incubar em gelo protegida da luz durante 30 min.
- Adicionar 10x volume de Etiquetagem Buffer para células e controles. Centrífuga 450 g durante 7 min. Elimine o sobrenadante por sucção a vácuo em um recipiente de resíduos.
- Ressuspender em Labeling Buffer para o volume na etapa 4.5. Centrífuga 450 g durante 7 min. Elimine o sobrenadante por sucção a vácuo em um recipiente de resíduos.
- Ressuspender em Labeling Buffer para 10 x 10 6 células / ml. Passa através de células 70 um filtro em tubo FACS. Mantenha no gelo protegido da luz até que esteja pronto para FACS.
- Configure e determinar a compensação apropriada no separador de células com células não coradas e controles individuais manchada.
- Excluir células não viáveis com forwarder e side-dispersão gating (Figura 1A). Configure gating para células CD45RB + CD4 + e com controles manchada de isótipo. Células portão à população CD4 +.
- Ordenar CD4 + células em CD45RB alta e CD45RB populações de baixa usando um histograma simples para as células marcadas com PE em tubos com 2 ml de Meio Completo. O CD45RB alta população representa o mais alto de 40% das células T CD4 + CD45RB + células (CD45RB alta), e a população de baixa CD45RB é o mais baixo de 20% das células CD4 + CD45RB + (CD45RB baixo; Figura 1B).
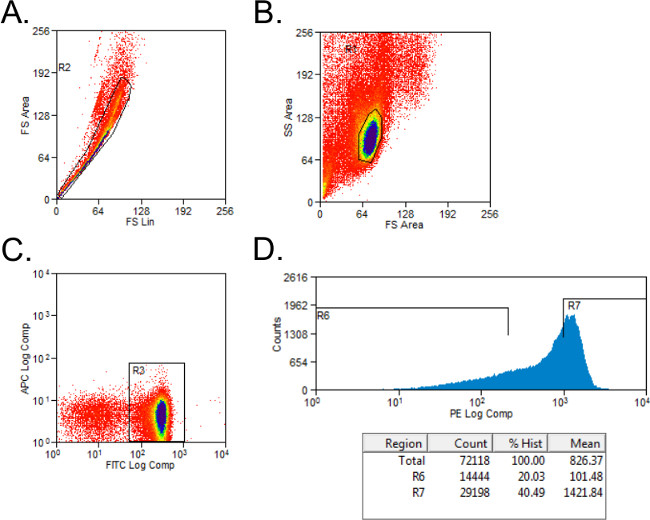
Figura 1: citometria de fluxo representativas parcelas de populações de células T CD4 + CD45RB durante a análise FACS (A - C) com FITC e CD4-PE esplenócitos CD45RB-corados de dadores murganhos C57BL / 6 foram classificadas por FACS em células CD4 + CD45RB high e CD4 +. populações de células T CD45RB baixo. Eventos (A) Doublet foram excluídos do gráfico de dispersão para a frente. (B) Os linfócitos foram fechado na trama dispersão frontal e lateral. (C) CD4 + células T foram fechado, e células (D) CD4 + CD45RB + T foram plotados em um PE contra Eventos histograma. As células T CD4 + CD45RB baixas foram consideradas ser a mais baixa de 20% de células + CD45RB. As células T CD4 + CD45RB elevados foram definidos como a mais alta de 40% de células + CD45RB.
- Executar uma alíquota de cada população de células sobre a máquina de FACS para avaliar a pureza das populações.
- Centrífuga classificadas células a 450 xg por 7 min. Ressuspender em 1 ml de PBS. Remover alíquota de células para contar, para avaliar a viabilidade celular por exclusão com azul de tripano como no passo 2.9.
5. injeção de células em Destinatários
- Ressuspender classificados células a 4 x 10 6 células / ml (alta CD45RB) ou 2 x 10 6 células / ml (CD45RB baixo) em PBS.
- Transferir 100 ml de células de alta CD45RB por beneficiário para novo tubo estéril. Adicionar 100 ul de PBS pordestinatário a este tubo. Assim, o volume total de injecção por animal é de 200 l, e valor total de células por beneficiário é de 4 x 10 5 CD4 + CD45RB células T naive elevados.
- Se o grupo experimental receber células T reguladoras é desejada, transferir 100 ml de células de alta CD45RB por beneficiário para novo tubo estéril. Adicionar 100 ul de células de baixa CD45RB por recipiente para o mesmo tubo. O volume total de injecção por animal é de 200 uL; proporção de CD45RB alta: células de baixa CD45RB é 2: 1.
- Injectar 100 ul de CD45RB alta ou CD45RB alta CD45RB baixo CD4 + células / intraperitoneal em o lado direito e esquerdo do abdômen (total de 200 ul) de cada destinatário.
6. Monitoramento da progressão da doença em animais receptores
- Para avaliar o estado clínico dos animais receptores, atribuir escores clínicos agregados para o seguinte parameters 8 no dia da injecção, decorrida uma semana, e no momento do sacrifício:
- Determine desperdiçando medindo a perda de peso: 0 - sem perda de peso; Perda de 0,1-10% do peso corporal inicial - 1; 2 - A perda de mais de 10% do peso corporal inicial (Figura 2A).

Figura 2: sinais patológicos clínicos e brutas de inflamação ocorrem após a transferência de tipo selvagem CD4 + CD45RB células T em alta RAG1 - / - e ratos destinatário NRDKO 11 (a) destinatários NRDKO perderam, em média, 10% do seu peso corporal inicial por 5. semanas após a transferência, enquanto RAG1- / - Os beneficiários não apresentaram sinais clínicos da doença neste momento. Cada ponto representa a média percentual do peso corporal inicial para a coorte ± SEM. **, P <0,005. (B) Alguns ratos desenvolveram inflamação intestinal grave, como demonstrado pela presença de prolapso retal. Esta é uma imagem representativa de prolapso retal em um mouse destinatário NRDKO. (C) Grosseiramente, a partir de dois pontos, tanto RAG1 - / - e ratinhos receptores NRDKO são espessadas e menor do que os dois pontos de RAG1 - / - e ratinhos NRDKO sem transferência adoptiva de células T. Colons de ratos receptores NRDKO mostrar inflamação grave e aumentou o peso dos dois pontos (dados não mostrados). Por favor, clique aqui para ver uma versão maior desta figura.
- Determinar a qualidade das fezes dos animais, colocando em recipiente limpo até que ele passoubanquinho: 0 - none; 1 - fezes moles; 2 - fezes aquosas e / ou com sangue.
- Determinar a saúde geral do animal através presença dos seguintes sinais da doença: 0 - nenhuma postura arqueada, eriçou pele, ou pele lesões; 1 - qualquer um de presente o seguinte: debruçado postura, eriçou pele, ou lesões de pele.
- Determinar a presença de prolapso retal: 0 - ausente; 1 - presente (Figura 2B).
- Sacrificar animais por inalação de CO2 seguido por deslocamento cervical, quando eles perderam 20% do seu peso corporal inicial ou no ponto de tempo desejado, o que ocorrer primeiro. A doença clínica é tipicamente evidente a partir de 5 semanas de pós-repletion.
- Avaliar para a gravidade da doença, como descrito anteriormente 5,7.
- Atribuir escores clínicos como no passo 6.1 8.
- Medir o comprimento do cólon e do peso (Figura 2C).
- Realizar análise histológica de inflamação5.
- Determine a expressão das citocinas espontânea em intestinais culturas de explantes tecido 9, linfa mesentéricos 10, e / ou soro 11.
- Resumidamente, para culturas de explantes 9, remover dois pontos após o sacrifício, aberta longitudinalmente, e limpo, com PBS. Incubar dois pontos, num agitador orbital a 250 rpm, em Meio Completo, durante 30 min à temperatura ambiente.
- Picar o tecido em pequenos pedaços e incubados a 37 ° C em Meio Completo, durante 24 h. Recolhe-se o sobrenadante e utilizar para a quantificação de citocinas por 100 mg de tecido por ELISA.
- Realizar ex vivo caracterização de fenótipos de células T e / ou função 10.
Access restricted. Please log in or start a trial to view this content.
Resultados
Aproximadamente 10 x 10 6 de células T CD4 + CD45RB high T de baços de 10 adultos C57BL / 6 murganhos dadores são seguramente isolado. Este número variará, dependendo da idade e estirpe do rato dador e a proficiência do investigador. Quando 4 x 10 5 C57BL / 6 células CD4 + T CD45RB alta são transferidos para C57BL / 6 RAG1 - / - ratinhos receptores, sinais clínicos da doença surgir em torno semana 5 pós-repletion ou antes, s...
Access restricted. Please log in or start a trial to view this content.
Discussão
Aqui nós descrevemos um protocolo passo-a-passo induzindo a inflamação do cólon em camundongos por transferência adotiva de células CD4 + T + CD45RB em ratos imunodeficientes. Usamos baços C57BL / 6 doadores e RAG1 singeneico - / - ratinhos receptores, embora outras estirpes (por exemplo, BALB / c, 129S6 / SvEv, não-obesos diabéticos (NOD)) modelos e genéticos da imunodeficiência (por exemplo, SCID, RAG2 - / -) também pode ser ut...
Access restricted. Please log in or start a trial to view this content.
Divulgações
The authors declare that they have no competing financial interests.
Agradecimentos
Este trabalho foi financiado pela Associação Americana de Gastroenterologia (AGA) Research Award Scholars e Doença de Crohn e Colite Foundation of America (CCFA) Desenvolvimento de Carreira Award (a ENS), NIH NIDDK F30 DK089692 (a ECS) e University of North Carolina Centro de Biologia Gastrointestinal e Doença Grant P30 DK34987 (Histologia Core). O Mecanismo UNC Citometria de Fluxo Core é apoiado em parte por uma NCI Centro de Suporte Grant Núcleo (P30CA016086) à Comprehensive Cancer Center UNC Lineberger. Agradecemos Luke B. Borst da North Carolina State University College de Medicina Veterinária por sua ajuda com a análise histopatológica e imuno-histoquímica.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Name of Reagent/ Equipment | Company | Catalog Number | Comments/Description |
| 10x PBS | Gibco | 14200075 | |
| 12 mm x 75 mm round-bottom tube | Falcon | 352052 | |
| 15 ml conical | Corning | 430790 | |
| 26 G x 3/8 Needle | BD Biosciences | 305110 | |
| 50 ml conical | Corning | 430828 | |
| 70 μm Cell Strainer | Fisherbrand | 22363548 | |
| BD IMagnet | BD Biosciences | 552311 | |
| β-mercaptoethanol | Thermo Scientific | 35602 | |
| CD4-FITC IgG2b | eBioscience | 11-0041 | |
| CD45RB-PE IgG2a | BD Pharminogen | 553101 | |
| Complete Media | RPMI-1640, 1% Pen/Strep, 10% FBS, 0.0004% β-ME | ||
| FACS tube + strainer | BD Falcon | 352235 | |
| Glass Microscope Slides | Fisherbrand | 12550A3 | |
| Heat-inactivated FBS | Gemini | 100-106 | |
| Labeling Buffer | 1x PBS, 0.5% BSA, 2 mM EDTA | ||
| Lysis Buffer | 0.08% NH4Cl, 0.1% KHCO3, 1 mM EDTA | ||
| MoFlo XDP | Beckman Coulter | ||
| Mouse CD4 T lymphocyte Enrichment Set - DM | BD Biosciences | 558131 | |
| Mouse IgG2a-PE | BD Pharminogen | 553457 | |
| Mouse IgG2b-FITC | eBioscience | 11-4732 | |
| Pasteur pipet | Fisherbrand | 13-678-20D | |
| Penicillin-Streptomycin Solution, 100X | Corning Cellgro | 30-002-CI | |
| [header] | |||
| Petri Dish | Fisherbrand | 875713 | |
| Pure Ethanol 200 Proof | Decon Labs | 2705-HC | |
| RPMI-1640 | Gibco | 11-875-093 | |
| Syringe | BD Biosciences | 309597 | |
| Trypan blue | Corning Cellgro | 25-900-CI | |
| Wash Media | RPMI-1640, 1% Pen/Strep, 0.0004% β-ME | ||
Referências
- Xavier, R. J., Podolsky, D. K. Unravelling the pathogenesis of inflammatory bowel disease. Nature. 448 (7152), 427-434 (2007).
- Cho, J. H., Brant, S. R. Recent insights into the genetics of inflammatory bowel disease. Gastroenterology. 140 (6), 1704-1712 (2011).
- Jostins, L., et al. Host-microbe interactions have shaped the genetic architecture of inflammatory bowel disease. Nature. 491 (7422), 119-124 (2012).
- Powrie, F., Leach, M. W., Mauze, S., Caddle, L. B., Coffman, R. L. Phenotypically distinct subsets of CD4+ T cells induce or protect from chronic intestinal inflammation in C. B-17 scid mice. Int Immunol. 5 (11), 1461-1471 (1993).
- Ostanin, D. V., et al. T cell transfer model of chronic colitis: concepts, considerations, and tricks of the trade. Am J Physiol Gastrointest Liver Physiol. 296 (2), 135-146 (2009).
- Ma, B. W., et al. Routine habitat change: a source of unrecognized transient alteration of intestinal microbiota in laboratory mice. PLoS One. 7 (10), e47416(2012).
- Read, S., Powrie, F. Induction of inflammatory bowel disease in immunodeficient mice by depletion of regulatory T cells. Curr Protoc Immunol. Chapter 15 (Unit 15 13), (1999).
- Maillard, M. H., et al. The Wiskott-Aldrich syndrome protein is required for the function of CD4(+)CD25(+)Foxp3(+) regulatory T cells. J Exp Med. 204 (2), 381-391 (2007).
- Hegazi, R. A., et al. Carbon monoxide ameliorates chronic murine colitis through a heme oxygenase 1-dependent pathway. J Exp Med. 202 (12), 1703-1713 (2005).
- Kole, A., et al. Type I IFNs regulate effector and regulatory T cell accumulation and anti-inflammatory cytokine production during T cell-mediated colitis. J Immunol. 191 (5), 2771-2779 (2013).
- Kobayashi, T., et al. NFIL3-deficient mice develop microbiota-dependent, IL-12/23-driven spontaneous colitis. J Immunol. 192 (4), 1918-1927 (2014).
- Steinbach, E. C., et al. Innate PI3K p110delta Regulates Th1/Th17 Development and Microbiota-Dependent Colitis. J Immunol. 192 (8), 3958-3968 (2014).
- Kobayashi, T., et al. NFIL3 is a regulator of IL-12 p40 in macrophages and mucosal immunity. J Immunol. 186 (8), 4649-4655 (2011).
- Leach, M. W., Bean, A. G., Mauze, S., Coffman, R. L., Powrie, F. Inflammatory bowel disease in C.B-17 scid mice reconstituted with the CD45RBhigh subset of CD4+ T cells. Am J Pathol. 148 (5), 1503-1515 (1996).
- Powrie, F., et al. Inhibition of Th1 responses prevents inflammatory bowel disease in scid mice reconstituted with CD45RBhi CD4. T cells. Immunity. 1 (7), 553-562 (1994).
- Read, S., Malmstrom, V., Powrie, F. Cytotoxic T lymphocyte-associated antigen 4 plays an essential role in the function of CD25(+)CD4(+) regulatory cells that control intestinal inflammation. J Exp Med. 192 (2), 295-302 (2000).
- Rogers, G. B., et al. Functional divergence in gastrointestinal microbiota in physically-separated genetically identical mice. Sci Rep. 4, 5437(2014).
- Fukata, M., et al. The myeloid differentiation factor 88 (MyD88) is required for CD4+ T cell effector function in a murine model of inflammatory bowel disease. J Immunol. 180 (3), 1886-1894 (2008).
- Kurtz, C. C., et al. Extracellular adenosine regulates colitis through effects on lymphoid and nonlymphoid cells. Am J Physiol Gastrointest Liver Physiol. 307 (3), 338-346 (2014).
- Naganuma, M., et al. Cutting edge: Critical role for A2A adenosine receptors in the T cell-mediated regulation of colitis. J Immunol. 177 (5), 2765-2769 (2006).
- Ranatunga, D. C., et al. A protective role for human IL-10-expressing CD4+ T cells in colitis. J Immunol. 189 (3), 1243-1252 (2012).
- Srikrishna, G., et al. Carboxylated glycans mediate colitis through activation of NF-kappa. B. J Immunol. 175 (8), 5412-5422 (2005).
- Wang, F., et al. IFN-gamma-induced TNFR2 expression is required for TNF-dependent intestinal epithelial barrier dysfunction. Gastroenterology. 131 (4), 1153-1163 (2006).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados