È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Induzione di Murine infiammazione intestinale per trasferimento adottivo di Effector CD4
In questo articolo
Riepilogo
Here, we present a protocol to induce colonic inflammation in mice by adoptive transfer of syngeneic CD4+CD45RBhigh T cells into T and B cell deficient recipients. Clinical and histopathological features mimic human inflammatory bowel diseases. This method allows the study of the initiation of colonic inflammation and progression of disease.
Abstract
Ci sono molti modelli animali differenti disponibili per lo studio della patogenesi delle malattie infiammatorie croniche intestinali umane (IBD), ognuno con i suoi vantaggi e svantaggi. Descriviamo qui un modello di colite sperimentale che è iniziata da trasferimento adottivo di cellule della milza alta T CD4 + CD45RB singenici in T e cellule B topi riceventi deficienti. La popolazione di cellule T CD4 + alta CD45RB che consiste in gran parte di cellule effettrici naïve è in grado di indurre infiammazione intestinale cronica, molto simili aspetti chiave di IBD umana. Questo metodo può essere manipolato per studiare gli aspetti di insorgenza della malattia e la progressione. Inoltre può essere utilizzata per studiare la funzione di innata, adattabile e popolazioni regolamentazione cellule immunitarie, e il ruolo delle esposizioni ambientali, cioè, il microbiota, infiammazione intestinale. In questo articolo si illustra il metodo per indurre la colite con un protocollo passo-passo. Questo incLudes un video dimostrativo di aspetti tecnici chiave necessari per sviluppare con successo questo modello murino di colite sperimentale per scopi di ricerca.
Introduzione
The inflammatory bowel diseases (IBD) Crohn’s disease and ulcerative colitis result from an incompletely defined and complex interaction between host immune responses, genetic susceptibility, environmental factors, and the enteric luminal contents1. Recent genome-wide association studies report associations between immune cell regulatory genes and IBD susceptibility2,3. Both innate and adaptive immune cell intrinsic genes are represented in these studies, indicating a central role for these cell populations in IBD pathogenesis.
There currently exist more than 50 animal models of human IBD. While no one model perfectly phenocopies human IBD, many are useful for studying various aspects of human disease, including disease onset and progression and the wound-healing response. In the method described here, intestinal inflammation is initiated with syngeneic splenic CD4+CD45RBhigh T cell adoptive transfer into T and B cell deficient recipient mice4. The CD4+CD45RBhigh T cell population contains mainly naïve T cells primed for activation that are capable of inducing chronic small bowel and colonic inflammation. This method allows the researcher to modify key experimental variables, including both innate and adaptive immune cell populations, to answer biologically relevant questions relating to disease pathogenesis. Additionally, this method provides precise initiation of disease onset and a well-characterized experimental time course. This permits the kinetic study of clinical features of disease progression in mice. Intestinal inflammation induced by this method shares many features with human IBD, including chronic large and small bowel transmural inflammation, pathogenesis driven by cytokines such as TNF and IL-12, and systemic symptoms such as wasting5. Thus, it is an ideal model system for studying the pathogenesis of human IBD.
The method here describes in detail the protocol for inducing experimental colitis by adoptive transfer of CD4+CD45RBhigh T cells into Rag1-/- mice. We discuss key technical steps, expected results, optimization, and trouble-shooting. We address the required elements for the successful development of this murine model of intestinal inflammation for research purposes.
Protocollo
NOTA: Assicurarsi che tutti i protocolli animali sono approvati dal e nel rispetto Istituzionale Animal Care and Use Committee (IACUC) regolamenti e guida del Consiglio Nazionale delle Ricerche per la cura e l'uso di animali da laboratorio. Topo donatore può essere maschio o femmina, ma topi riceventi devono essere di sesso maschile. Se i destinatari femmina devono essere utilizzati, i topi donatori devono essere femmina 5. Mantenere le colonie con regolare, biancheria da letto non sterili e acqua non acidificato, in quanto potrebbero avere un impatto del microbiota intestinale, e, quindi, il fenotipo colite dei topi 5,6.
Preparazione 1. Sperimentale
- Utilizzare supporti e tamponi ghiacciate. Mantenere cellule in ghiaccio durante l'esperimento.
- Eseguire l'esperimento in cappa di rischio biologico sterile con tecnica sterile.
2. Isolamento delle cellule della milza T
- Euthanize topo donatore / mice in CO 2 da camera seguiti da dislocatio cervicalen. Spray addome con etanolo al 70%.
- Fare un incisione orizzontale nell'addome e staccarsi la pelle per esporre il peritoneo. Tenere peritoneo lontano dagli organi interni con le pinze e fare un'incisione nel peritoneo addominale sinistro per esporre e asportare la milza.
- Posizionare la milza in 10 ml di completo media in una capsula di Petri. Rimuovere e gettare il tessuto in eccesso dalla milza.
- Utilizzare 2 vetrini sterilizzati per schiacciare e prendere in giro a parte milza in cella singola sospensione. Sospensione cellulare Filtrare attraverso un filtro 70 micron in una provetta conica da 50 ml e risciacquare il filtro con 5 ml di completo media. Luogo fino a 5 milze in un tubo da 50 ml conica.
- Cellule centrifugare a 450 xg per 7 minuti. Gettare il surnatante versando off o per aspirazione di vuoto nel contenitore dei rifiuti.
- Risospendere delicatamente le cellule in 5 ml per milza di tampone di lisi per lisare i globuli rossi per 10 min a temperatura ambiente. Aggiungere un volume uguale di Complete mezzi (5 ml per milza) al tubo.
- cellule centrifugare a 450 xg per 7 minuti. Gettare il surnatante versando off o per aspirazione di vuoto nel contenitore dei rifiuti.
- Risospendere delicatamente le cellule in 10 ml di Labeling Buffer.
- Contare le celle di trypan esclusione blu.
- Rimuovere 20 ml di sospensione cellulare e aggiungere 180 microlitri blu trypan ad una provetta per microcentrifuga e mescolare accuratamente. Dopo 5 minuti, aggiungere 10 ml di cellule emocitometro e contare le cellule non-blu al microscopio etichettati. Determinare il numero totale di cellule vitali. Eliminare cell / trypan blue mix.
- Centrifugare le cellule in 10 ml di Labeling Buffer a 450 xg per 7 minuti. Gettare il surnatante versando off o per aspirazione di vuoto nel contenitore dei rifiuti.
3. Arricchimento di cellule CD4 + T
NOTA: Seguire le istruzioni del produttore per i prodotti specifici utilizzati in questa sezione.
- Risospendere delicatamente le cellule a una cella singola sospensione di 20 x 10 6 cellule / ml in freddo LabelBuffer ing.
- Aggiungere 5 ml per 1 x 10 6 cellule di anticorpi arricchimento delle cellule T CD4 biotinilati. Incubare le cellule in ghiaccio per 15 min.
- Aggiungere 10x volume di Labeling Buffer. Cellule centrifugare a 450 g per 7 min. Aspirare accuratamente tutto il surnatante con aspirazione di vuoto nel contenitore dei rifiuti.
- Vortice accuratamente particelle magnetiche streptavidina coniugato. Aggiungere 5 ml di particelle per 1 x 10 6 cellule.
- Mescolare accuratamente, ma delicatamente. Tenere mix a 6-12 ° C per 30 min.
- Aggiungere Labeling Buffer ad una concentrazione di 20-80 x 10 6 cellule / ml. Trasferimento fino a 1,0 ml cellule marcate per 12 x 75 mm provetta a fondo tondo (denominato "tubo positivo frazione").
- Collocare ogni provetta positiva frazione sulla calamita per 6-8 min.
- Con i tubi positivo frazione ancora sul magnete, trasferire accuratamente surnatante dal tubo positiva frazione con Pasteur vetro pipetta ad un nuovo tubo conico sterili 50 ml (referred come la "frazione arricchita"). Questa frazione arricchita contiene cellule CD4 + T. Fare attenzione a non distruggere le cellule marcate attratti al magnete.
- Risospendere le cellule lasciati nei tubi positivo frazione nello stesso volume di Labeling Buffer come al punto 3.6 pipettando su e giù vigorosamente. Posizionare il tubo positivo frazione indietro calamita per 6-8 min.
- Con i tubi positiva frazione ancora sul magnete, trasferire attentamente surnatante (arricchito frazione, CD4 +) dal tubo di positivo-frazione con vetro Pasteur pipetta per sterile tubo da 50 ml da punto 3.8 senza interrompere le cellule attaccate a magnete.
- Ripetere i passaggi 3,9-3,10 per aumentare la resa delle cellule CD4 + T ottenuti. Continuare protocollo utilizzando frazione arricchita (cellule CD4 +).
4. L'etichettatura e Ordinamento Cells 7
- Centrifugare arricchito cellule a 450 xg per 7 minuti. Gettare il surnatante versando off o SUCT vuotoione nel contenitore dei rifiuti.
- Risospendere le cellule in 1 ml di Labeling Buffer. Rimuovere aliquota di cellule per contare e valutare per la vitalità cellulare del trypan blu come al punto 2.9.
- Aggiungere un volume di Labeling Buffer a 10 x 10 6 cellule / ml; se le cellule sono già <10 x 10 6, centrifugare a 450 xg per 7 minuti, scartare il surnatante versando off o di aspirazione di vuoto nel contenitore dei rifiuti, e aggiungere il volume di Labeling Buffer a 10 x 10 6 cellule / ml.
- Impostare aliquote separate di circa 5-10 × 10 5 celle ciascuno per le cellule di controllo non colorate, isotipo macchiati, e single-macchiato in tubi microcentrifuga.
- Aggiungere 5 ug / ml CD4-FITC e 1 mg / ml CD45RB-PE alle cellule. Aggiungere macchie di controllo isotipico e macchie singoli alla stessa concentrazione di appropriarsi aliquote in tubi microcentrifuga. Mescolare bene ma delicatamente e incubare su ghiaccio al riparo dalla luce per 30 min.
- Aggiungere 10x volume di Labeling Buffer alle cellule e di controllos. Centrifuga 450 xg per 7 minuti. Gettare il surnatante mediante aspirazione di vuoto nel contenitore dei rifiuti.
- Risospendere in Labeling Buffer di volume in fase 4.5. Centrifuga 450 xg per 7 minuti. Gettare il surnatante mediante aspirazione di vuoto nel contenitore dei rifiuti.
- Risospendere in Labeling Buffer a 10 x 10 6 cellule / ml. Passare cellule attraverso 70 micron filtro nel tubo FACS. Tenere su ghiaccio al riparo dalla luce fino al momento FACS.
- Impostare e stabilire un'adeguata compensazione sul cell sorter con le cellule non colorate e controlli singoli macchiati.
- Escludi cellule non vitali con forward e side-scatter gating (Figure 1A). Impostare gating per CD4 + e + cellule CD45RB con controlli isotipici macchiato. Cellule CD4 + cancello sulla popolazione.
- Ordina CD4 + cellule in CD45RB alta e CD45RB basse popolazioni utilizzando una semplice istogramma per le cellule PE macchiate in tubi con 2 ml di completo media. Il CD45RB alta popolazione rappresenta la più alta del 40% di CD4 + CD45RB + cellule (CD45RB alta), e la popolazione bassa CD45RB è il più basso del 20% + CD45RB cellule CD4 + (CD45RB basso; Figura 1B).
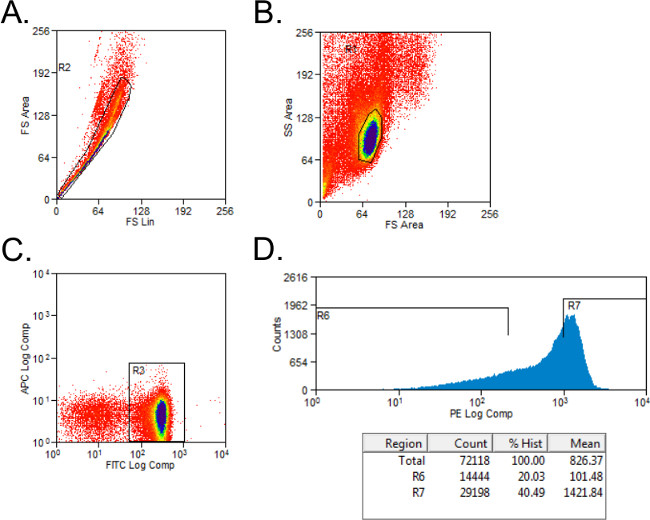
Figura 1: flusso rappresentante citometria appezzamenti di popolazioni di cellule CD4 + T CD45RB durante l'analisi FACS (A - C) FITC CD4- e splenociti PE CD45RB-macchiato da donatori C57BL / 6 topi sono stati ordinati per FACS in CD4 + CD45RB alta e CD4 +. CD45RB popolazioni di cellule T basso. Eventi (A) Doppietti stati esclusi nella rappresentazione della dispersione in avanti. (B) I linfociti sono stati gated nella trama in avanti e scatter laterale. (C) CD4 + cellule T sono stati gated, e cellule (D) CD4 + CD45RB + T sono state tracciate su un PE contro Eventi istogramma. Cellule CD4 + bassi CD45RB sono stati considerati i più bassi del 20% delle cellule + CD45RB. Cellule di alta CD4 + CD45RB sono stati definiti come la più alta del 40% delle cellule + CD45RB.
- Eseguire un'aliquota di ciascuna popolazione cellulare sulla macchina FACS per valutare la purezza delle popolazioni.
- Centrifugare allineati cellule a 450 xg per 7 minuti. Risospendere in 1 ml di PBS. Rimuovere aliquota di cellule per contare e valutare per la vitalità cellulare del trypan blu come al punto 2.9.
5. iniezione di cellule in Destinatari
- Risospendere allineati alle cellule di 4 x 10 6 cellule / ml (CD45RB alta) o 2 x 10 6 cellule / ml (CD45RB basso) in PBS.
- Trasferire 100 ml di cellule di alta CD45RB per destinatario di nuova provetta sterile. Aggiungere 100 ml di PBS perdestinatario di questo tubo. Così, il volume di iniezione totale per animali è di 200 ml, e l'importo totale delle cellule per destinatario è di 4 alti cellule T naive x 10 5 CD4 + CD45RB.
- Se si desidera gruppo sperimentale che riceve le cellule T regolatorie, trasferire 100 ml di cellule di alta CD45RB per destinatario di nuova provetta sterile. Aggiungere 100 ml di CD45RB bassi cellule per destinatario lo stesso tubo. Volume di iniezione totale per animali è di 200 ml; Rapporto di alto CD45RB: CD45RB cellule bassi è di 2: 1.
- Iniettare 100 ml di CD45RB alta o CD45RB alto / basso CD45RB CD4 + cellule per via intraperitoneale nel lato destro e sinistro dell'addome (totale di 200 ml) di ciascun destinatario.
6. Monitoraggio della progressione della malattia in animali destinatari
- Per valutare lo stato clinico degli animali beneficiari, assegnare punteggi clinici aggregati per le seguenti parameters 8 il giorno dell'iniezione, poi settimanalmente e al momento del sacrificio:
- Determinare sprecare misurando la perdita di peso: 0 - nessuna perdita di peso; Perdita 0,1-10% del peso corporeo iniziale - 1; 2 - perdita più del 10% del peso corporeo iniziale (Figura 2A).

Figura 2: segni patologici clinici e lordi di infiammazione si verificano dopo il trasferimento di tipo selvaggio cellule T CD4 + alta CD45RB in Rag1 - / - e NRDKO topi riceventi 11 (A) i destinatari NRDKO perso in media il 10% del loro peso corporeo iniziale del 5. settimane dopo il trasferimento, mentre Rag1- / - I beneficiari non hanno mostrato segni clinici di malattia in questo momento. Ogni punto rappresenta la percentuale media del peso corporeo iniziale per la coorte ± SEM. **, P <0.005. (B) Alcuni topi hanno sviluppato una grave infiammazione intestinale, come dimostrato dalla presenza di prolasso rettale. Questo è un quadro rappresentativo di prolasso rettale in un topo ricevente NRDKO. (C) fortemente, i due punti sia da Rag1 - / - e NRDKO topi riceventi sono ispessite e accorciati rispetto a due punti dal Rag1 - / - mice e NRDKO senza T cell trasferimento adottivo. I due punti di NRDKO topi riceventi mostrano grave infiammazione e aumento del peso del colon (dati non riportati). Cliccate qui per vedere una versione più grande di questa figura.
- Determinare la qualità delle feci mettendo animali in recipiente pulito fino a quando non è passatosgabello: 0 - nessuno; 1 - feci molli; 2 - feci acquose e / o sanguinosa.
- Determinare la salute generale di animale presenza dei seguenti segni di malattia: 0 - no postura ingobbita, setole pelliccia, pelle o lesioni; 1 - uno qualsiasi dei seguenti presente: curvo postura, setole di pelliccia, o lesioni cutanee.
- Determinare la presenza di prolasso rettale: 0 - assente; 1 - presente (Figura 2B).
- Sacrifica animali da CO 2 inalazione seguita da dislocazione cervicale quando hanno perso il 20% del loro peso corporeo iniziale o nel punto desiderato momento, quello che viene prima. Malattia clinica è di solito evidente a partire da settimana 5 post-sazietà.
- Valutare per la gravità della malattia, come descritto in precedenza 5,7.
- Assegnare punteggi clinici come al punto 6.1 8.
- Misurare la lunghezza del colon e del peso (Figura 2C).
- Eseguire l'analisi istologica di infiammazione5.
- Determinare citochine spontanea in intestinali culture espianto dei tessuti 9, la linfa mesenterica nodi 10, e / o siero 11.
- In breve, per le culture espianto 9, rimuovere i due punti dopo il sacrificio, aperta longitudinalmente, e pulire con PBS. Incubare due punti in un agitatore orbitale a 250 rpm impostato in completa media per 30 minuti a temperatura ambiente.
- Tritare il tessuto in piccoli pezzi e incubate a 37 ° C in media completa per 24 ore. Raccogliere il surnatante e utilizzare per la quantificazione di citochine per 100 mg di tessuto di ELISA.
- Eseguire caratterizzazione ex vivo di fenotipi di cellule T e / o la funzione 10.
Risultati
Circa 10 x 10 6 CD4 + CD45RB cellule di alta T da 10 milza di adulti C57BL / 6 topi donatori sono affidabile isolati. Questo numero varia a seconda dell'età e la tensione del mouse donatore e la competenza del ricercatore. Quando 4 x 10 5 C57BL / 6 celle ad alta T CD4 + CD45RB vengono trasferiti in C57BL / 6 Rag1 - / - topi riceventi, i segni clinici della malattia emergono intorno settimana 5 post-sazietà o prima, se i topi sono...
Discussione
Qui si descrive un protocollo step-by-step indurre infiammazione del colon nei topi mediante trasferimento adottivo di cellule CD4 + T + CD45RB in topi immunodeficienti. Abbiamo usato milza C57BL / 6 donatori e Rag1 singenico - / - topi riceventi, anche se altri ceppi (ad esempio, BALB / c, 129S6 / SvEv, non-obesi diabetici (NOD)) Modelli e genetici di immunodeficienza (ad esempio, SCID, Rag2 - / -) può essere utilizzato anche 4,14-16....
Divulgazioni
The authors declare that they have no competing financial interests.
Riconoscimenti
Questo lavoro è stato sostenuto da Gastroenterologiche American Association (AGA) Research Scholars Award e morbo di Crohn e Colite Foundation of America (CCFA) Career Development Award (a SOI), NIH NIDDK F30 DK089692 (ECS), e University of North Carolina Centro di Biologia gastrointestinale e la malattia di Grant P30 DK34987 (Istologia Core). Il Fondo UNC Citometria a flusso Core è sostenuto in parte da un nucleo di supporto NSC Centro Grant (P30CA016086) alla UNC Lineberger Comprehensive Cancer Center. Ringraziamo Luca B. Borst da North Carolina State University College di Medicina Veterinaria per il suo aiuto con analisi istopatologica e immunoistochimica.
Materiali
| Name | Company | Catalog Number | Comments |
| Name of Reagent/ Equipment | Company | Catalog Number | Comments/Description |
| 10x PBS | Gibco | 14200075 | |
| 12 mm x 75 mm round-bottom tube | Falcon | 352052 | |
| 15 ml conical | Corning | 430790 | |
| 26 G x 3/8 Needle | BD Biosciences | 305110 | |
| 50 ml conical | Corning | 430828 | |
| 70 μm Cell Strainer | Fisherbrand | 22363548 | |
| BD IMagnet | BD Biosciences | 552311 | |
| β-mercaptoethanol | Thermo Scientific | 35602 | |
| CD4-FITC IgG2b | eBioscience | 11-0041 | |
| CD45RB-PE IgG2a | BD Pharminogen | 553101 | |
| Complete Media | RPMI-1640, 1% Pen/Strep, 10% FBS, 0.0004% β-ME | ||
| FACS tube + strainer | BD Falcon | 352235 | |
| Glass Microscope Slides | Fisherbrand | 12550A3 | |
| Heat-inactivated FBS | Gemini | 100-106 | |
| Labeling Buffer | 1x PBS, 0.5% BSA, 2 mM EDTA | ||
| Lysis Buffer | 0.08% NH4Cl, 0.1% KHCO3, 1 mM EDTA | ||
| MoFlo XDP | Beckman Coulter | ||
| Mouse CD4 T lymphocyte Enrichment Set - DM | BD Biosciences | 558131 | |
| Mouse IgG2a-PE | BD Pharminogen | 553457 | |
| Mouse IgG2b-FITC | eBioscience | 11-4732 | |
| Pasteur pipet | Fisherbrand | 13-678-20D | |
| Penicillin-Streptomycin Solution, 100X | Corning Cellgro | 30-002-CI | |
| [header] | |||
| Petri Dish | Fisherbrand | 875713 | |
| Pure Ethanol 200 Proof | Decon Labs | 2705-HC | |
| RPMI-1640 | Gibco | 11-875-093 | |
| Syringe | BD Biosciences | 309597 | |
| Trypan blue | Corning Cellgro | 25-900-CI | |
| Wash Media | RPMI-1640, 1% Pen/Strep, 0.0004% β-ME | ||
Riferimenti
- Xavier, R. J., Podolsky, D. K. Unravelling the pathogenesis of inflammatory bowel disease. Nature. 448 (7152), 427-434 (2007).
- Cho, J. H., Brant, S. R. Recent insights into the genetics of inflammatory bowel disease. Gastroenterology. 140 (6), 1704-1712 (2011).
- Jostins, L., et al. Host-microbe interactions have shaped the genetic architecture of inflammatory bowel disease. Nature. 491 (7422), 119-124 (2012).
- Powrie, F., Leach, M. W., Mauze, S., Caddle, L. B., Coffman, R. L. Phenotypically distinct subsets of CD4+ T cells induce or protect from chronic intestinal inflammation in C. B-17 scid mice. Int Immunol. 5 (11), 1461-1471 (1993).
- Ostanin, D. V., et al. T cell transfer model of chronic colitis: concepts, considerations, and tricks of the trade. Am J Physiol Gastrointest Liver Physiol. 296 (2), 135-146 (2009).
- Ma, B. W., et al. Routine habitat change: a source of unrecognized transient alteration of intestinal microbiota in laboratory mice. PLoS One. 7 (10), e47416 (2012).
- Read, S., Powrie, F. Induction of inflammatory bowel disease in immunodeficient mice by depletion of regulatory T cells. Curr Protoc Immunol. Chapter 15 (Unit 15 13), (1999).
- Maillard, M. H., et al. The Wiskott-Aldrich syndrome protein is required for the function of CD4(+)CD25(+)Foxp3(+) regulatory T cells. J Exp Med. 204 (2), 381-391 (2007).
- Hegazi, R. A., et al. Carbon monoxide ameliorates chronic murine colitis through a heme oxygenase 1-dependent pathway. J Exp Med. 202 (12), 1703-1713 (2005).
- Kole, A., et al. Type I IFNs regulate effector and regulatory T cell accumulation and anti-inflammatory cytokine production during T cell-mediated colitis. J Immunol. 191 (5), 2771-2779 (2013).
- Kobayashi, T., et al. NFIL3-deficient mice develop microbiota-dependent, IL-12/23-driven spontaneous colitis. J Immunol. 192 (4), 1918-1927 (2014).
- Steinbach, E. C., et al. Innate PI3K p110delta Regulates Th1/Th17 Development and Microbiota-Dependent Colitis. J Immunol. 192 (8), 3958-3968 (2014).
- Kobayashi, T., et al. NFIL3 is a regulator of IL-12 p40 in macrophages and mucosal immunity. J Immunol. 186 (8), 4649-4655 (2011).
- Leach, M. W., Bean, A. G., Mauze, S., Coffman, R. L., Powrie, F. Inflammatory bowel disease in C.B-17 scid mice reconstituted with the CD45RBhigh subset of CD4+ T cells. Am J Pathol. 148 (5), 1503-1515 (1996).
- Powrie, F., et al. Inhibition of Th1 responses prevents inflammatory bowel disease in scid mice reconstituted with CD45RBhi CD4. T cells. Immunity. 1 (7), 553-562 (1994).
- Read, S., Malmstrom, V., Powrie, F. Cytotoxic T lymphocyte-associated antigen 4 plays an essential role in the function of CD25(+)CD4(+) regulatory cells that control intestinal inflammation. J Exp Med. 192 (2), 295-302 (2000).
- Rogers, G. B., et al. Functional divergence in gastrointestinal microbiota in physically-separated genetically identical mice. Sci Rep. 4, 5437 (2014).
- Fukata, M., et al. The myeloid differentiation factor 88 (MyD88) is required for CD4+ T cell effector function in a murine model of inflammatory bowel disease. J Immunol. 180 (3), 1886-1894 (2008).
- Kurtz, C. C., et al. Extracellular adenosine regulates colitis through effects on lymphoid and nonlymphoid cells. Am J Physiol Gastrointest Liver Physiol. 307 (3), 338-346 (2014).
- Naganuma, M., et al. Cutting edge: Critical role for A2A adenosine receptors in the T cell-mediated regulation of colitis. J Immunol. 177 (5), 2765-2769 (2006).
- Ranatunga, D. C., et al. A protective role for human IL-10-expressing CD4+ T cells in colitis. J Immunol. 189 (3), 1243-1252 (2012).
- Srikrishna, G., et al. Carboxylated glycans mediate colitis through activation of NF-kappa. B. J Immunol. 175 (8), 5412-5422 (2005).
- Wang, F., et al. IFN-gamma-induced TNFR2 expression is required for TNF-dependent intestinal epithelial barrier dysfunction. Gastroenterology. 131 (4), 1153-1163 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon