Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Lucifer Yellow - Un marqueur de perméabilité paracellulaire robuste dans un modèle cellulaire de la barrière de sang-cerveau humain
Dans cet article
Résumé
Nous présentons un test de fluorescence pour démontrer que Lucifer Yellow (LY) est un marqueur robuste pour déterminer la perméabilité paracellulaire apparente des monocouches de cellules hCMEC/D3, un modèle in vitro de la barrière hémato-encéphalique humaine. Nous avons utilisé cet exemple pour déterminer la cinétique d'une formation de monocouches confluentes dans les cellules cultivées de hCMEC/D3.
Résumé
La barrière hémato-encéphalique BBB se compose de cellules endothéliales qui forment une barrière entre la circulation systémique et le cerveau pour empêcher l'échange d'ions non essentiels et de substances toxiques. Les jonctions serrées (TJ) scellent efficacement l'espace paracellulaire dans les monocouches, ce qui donne lieu à une barrière intacte. Cette étude décrit un analyse de fluorescence basée sur LY qui peut êtreutilisé pour déterminer son coefficient de perméabilité apparente (application P ) et à son tour peut être utilisé pour déterminer la cinétique de la formation de monocouches confluents et la jonction serrée résultante l'intégrité de la barrière dans les monocouches hCMEC/D3. Nous démontrons en outre une utilité additionnelle de cet analyse pour déterminer l'intégrité fonctionnelle de TJ dans les cellules transfected. Nos données de l'analyse de l'application LY P montrent que les cellules hCMEC/D3 ensemant dans une configuration de transwell limitent efficacement le transport paracellulaire LY 7 jours-post culture. En tant qu'utilité supplémentaire de l'analyse présentée, nous démontrons également que la transfection de nanoparticules d'ADN ne modifie pas le transport paracellulaire de LY dans les monocouches de hCMEC/D3.
Introduction
La barrière hémato-encéphalique (BBB) est la barrière protectrice limitant l'afflux de composants plasmatiques dans le tissu cérébral et se compose de cellules endothéliales du cerveau ainsi que de cellules de soutien telles que les péritéytes. Le rôle principal de BBB est de servir de barrière qui scelle l'espace entre le sang périphérique et le système nerveux central (SNC) pour maintenir l'hémostase du microenvironnement neuronal1,2. Les cellules endothéliales capillaires du cerveau scellent efficacement la voie paracellulaire par la formation de jonctions intercellulaires serrées (TJs)1. Cette barrière protectrice permet au glucose et aux nutriments sélectionnés d'entrer dans le cerveau alors qu'elle empêche la majorité des ions, des substances toxiques et des médicaments de passer à travers cette barrière serrée. Outre son rôle protecteur, la fonction de barrière naturelle du BBB pose un défi grave dans le développement de systèmes d'administration de médicaments ciblant le SNC.
Les modèles de culture cellulaire in vitro du BBB sont des outils utiles pour étudier sa biologie et comprendre les effets du traitement médicamenteux sur l'intégrité de la barrière tJ. Nous avons utilisé la lignée cellulaire endothéliale microvasculaire humaine (hCMEC/D3) comme modèle in vitro puisqu'il s'agit d'un modèle accepté d'endothélium du cerveau humain3 et récapitule de nombreuses fonctions du BBB humain. hCMEC/D3is l'une des lignées cellulaires les plus couramment utilisées pour modéliser le BBB in vitro4,5,6,7,8,9. Malgré ses valeurs relativement faibles de résistance électrique transendothéliale (TEER), une mesure de l'étanchéité de barrière, cette ligne cellulaire conserve la plupart des propriétés morphologiques et fonctionnelles des cellules endothéliales de cerveau, même comme monoculture en l'absence de cellules gliales cocultivées6,7. La lignée cellulaire hCMEC/D3 exprime plusieurs marqueurs BBB, y compris les transporteurs actifs et les récepteurs jusqu'à environ passage 35 sans subir de dédifférenciation aux phénotypes instables6,7,9 ,10,11. La caractéristique la plus frappante de la lignée cellulaire hCMEC/D3 en tant que modèle BBB in vitro est sa capacité à former des TJs5,9,11,12. Il convient de noter que bien que les modèles BBB dérivés de cellules souches aient montré une perméabilité plus élevée dans de nombreuses études par rapport à la lignée cellulaire hCMEC/D3 et qu'ils expriment certains marqueurs BBB, ils n'ont pas encore évolué en tant que modèle de cellules BBB le plus commun13. Fait important, les modèles BBB dérivés de cellules souches restent à caractériser en ce qui concerne les nombres de passage maximaux qui permettent aux cellules de maintenir des phénotypes BBB stables14.
Trois méthodes primaires sont couramment utilisées pour déterminer l'intégrité de la barrière TJ, ycompris la mesure de TEER, la mesure du coefficient de perméabilité apparente (application P ) de petites molécules de traceur hydrophile telles que le saccharose, l'inuline, Lucifer Yellow, etc. et l'immunostaining des marqueurs moléculaires connus des TJs tels que claudin-5, ZO-1, occludin, etc.5. TEER est une méthode relativement simple et quantitative qui mesure la résistance électrique à travers les monocouches cellulaires cultivées sur un substrat de membrane poreux5. Cependant, les valeurs TEER peuvent être influencées par des variables expérimentales telles que la composition du milieu de culture et le type d'instrument de mesure. Une combinaison probable de ces facteurs conduit à une large distribution des valeurs TEER allant de 2 à 1150 cm2 dans la lignée cellulaire hCMEC/D3 cultivée pendant 2-21 jours13. Immunostaining est une méthode visuelle pour déterminer la présence de protéines TJ en étiquetant la protéine ciblée à l'aide d'anticorps. Cependant, l'immunostaining implique une série d'étapes expérimentales, y compris la nécessité de fixer/perméabilize des cellules qui peuvent avoir comme conséquence des artefacts expérimentaux et les signaux fluorescents peuvent s'estomper avec le temps. Les facteurs ci-dessus peuvent entraîner des erreurs subjectives affectant la qualité des données.
L'objectif principal de ce travail est de présenter un assay apparent de perméabilité basé sur LY déterminer la cinétique d'une formation de monocouche confluente dans les cellules cultivées de hCMEC/D3. Bien que d'autres systèmes BBB in vitro avancés, tels que les systèmes de co-culture, les systèmes microfluidiques, sont physiologiquement plus pertinents imite avec la fonction de barrière sensiblement améliorée15,16,17, le hCMEC/D3 la configuration de transwell est un modèle simple et fiable pour estimer les cinétiques de la formation de TJ et examiner rapidement l'effet de différentes formulations de drogue sur la fonction de barrière. En général, les valeurs de l'application P sont cohérentes pour divers solutés hydrophiles dans les monocouches hCMEC/D3. Par exemple, les valeurs de l'application P signalées pour divers solutés de masse moléculaire faible (comme le saccharose, le mannitol, le LY, etc.) dans différents modèles BBB in vitro sont de l'ordre de 10-4 cm/min5,18,19 , 20. Dans notre configuration expérimentale, les cellules endothéliales de cerveau sont ensemies sur une membrane micropore collagène-enduite pour l'attachement cellulaire et la formation de monocouche pour imiter la barrière in vivo. Le LY ajouté dans le côté apical devrait traverser les jonctions intercellulaires serrées et s'accumuler dans le côté basolatéral. De plus grandes concentrations de LY dans le côté basolatéral indiquent une barrière immature et non entièrement fonctionnelle tandis que des concentrations plus faibles reflètent le transport restreint dû à la présence de TJs fonctionnels ayant pour résultat une barrière mûre.
LY est un colorant hydrophile avec des pics d'excitation/émission distincts et évite la nécessité de radiolabel molécules traceurs telles que le saccharose, le mannitol ou l'inuline. Ainsi, les valeurs de fluorescence de LY peuvent être utilisées pour calculer directement sa perméabilité paracellulaire à travers les monocouches BBB. En outre, par rapport à de nombreux colorants disponibles dans le commerce utilisés dans les domaines biomédicaux qui souffrent de petits changements Stokes tels que la fluorescéine21, le changement Stokes de LY est d'environ 108 nm avec une séparation spectrale suffisante, permettant ainsi des données de fluorescence LY comme un lecture robuste pour déterminer la perméabilité paracellulaire. Nous avons utilisé le ballonnement occidental comme technique orthogonale pour démontrer des changements dans l'expression de la protéine de marqueur de jonction serrée, ZO-1, au cours du temps de culture. L'expression ZO-1 détectée par le biais de l'adhérence occidentale est utilisée pour compléter les données de l'application LY P et, en combinaison, ces données suggèrent que les changements observés dans les valeurs de l'application LY P reflètent la formation d'un monocouche avec une augmentation graduelle l'expression du marqueur de jonction serré, ZO-1.
Comme nous l'avons souligné précédemment, l'objectif central de ce travail est de démontrer un test LY comme une technique simple pour surveiller la formation d'une monocouche confluente avec des jonctions fonctionnelles serrées. Cependant, pour démontrer une utilité supplémentaire de l'analyse développée, nous avons mesuré l'application LY P dans des monocouches hCMEC/D3 transfées d'ADN. Les acides nucléiques peuvent être condensés en nanoparticules de polyélectrolyte d'un diamètre de 100-200 nm par l'intermédiaire d'une interaction électrostatique entre les groupes de polymères chargés positivement et les groupes de phosphate chargés négativement d'acides nucléiques22, 23. Nous nous référons à ces complexes sous le compte des nanoparticules d'ADN (NP d'ADN) dans nos travaux. Bien que notre intention soit de transfect cellules et d'exprimer la protéine désirée, nous devons nous assurer que les propriétés de barrière des monocouches hCMEC/D3 ne sont pas compromises. Nos données suggèrent qu'un régime standard de transfection de gène de luciferase de 4 h ne change pas mesurablement la perméabilité de LY démontrant l'utilité de l'analyse d'application de LY P pour déterminer des changements dans l'intégrité de barrière de TJ.
Protocole
1.Général hCMEC/D3 culture cellulaire
- Réanimation de cellules congelées
REMARQUE : Tous les entretiens et expériences de culture cellulaire ont été exécutés à l'intérieur d'une hotte stérile de biosécurité. Les supports culturels, les suppléments et les réactifs ont été achetés comme produits stériles ou stérilisés par filtration à l'aide d'un filtre à membrane de 0,22 m pour prévenir la contamination microbienne.- Ajouter 8,5 ml de solution de collagène (0,15 mg/ml) dans un flacon de culture tissulaire (75 cm2 zone de croissance; désormais appelé T75) et le placer dans un incubateur (37 oC, 5 % CO2) pendant 1 h.
- Retirez la solution de collagène et lavez doucement le flacon à l'intérieur d'une solution saline stérilisée tamponnée par le phosphate (PBS). Ajouter 15 ml de milieu de croissance complet au flacon et laisser dans l'incubateur CO2 pendant 15 min.
REMARQUE : Le milieu complet (concentration finale) contenait endothélial Cell Growth Basal Medium-2 (500 mL) complété par un sérum bovin fœtal (5 %), pénicilline-streptomycine (1 %), hydrocortisone (1,4 M), acide ascorbique (5 g/mL), lipide chimiquement défini concentré (1/100), HEPES (10 mM) et facteur de croissance de base du fibroblaste (1 ng/mL). - Déplacer un cryovial de cellules hCMEC/D3 congelées du réservoir d'azote liquide et décongeler rapidement le flacon dans un bain d'eau de 37 oC (1 min).
- Une fois qu'un petit flocon de glace est visible, aspirez rapidement et transférez les cellules dans le flacon contenant un milieu préréchauffé. Secouez doucement le flacon pour permettre le mélange des cellules avec le milieu de croissance.
- Placer le flacon dans l'incubateur (37 oC, 5 % de CO2)et observer les cellules sous un microscope léger après 2 h pour s'assurer que les cellules sont attachées.
- Une fois que les cellules se fixent au fond du flacon, retirez l'ancien milieu de croissance et ajoutez 10 ml de milieu de croissance frais pré-chauffé pour remplacer le diméthylsulfoxide dans l'ancien milieu de croissance24.
- Après 24 h, vérifiez sous un microscope léger pour observer les cellules en forme de fuseau et remplacez l'ancien milieu de croissance par un milieu de croissance frais pré-chauffé.
- Entretien de la culture cellulaire
- Réapprovisionnez le milieu de croissance tous les deux jours jusqu'à la confluence à 100 %. Vérifier les cellules sous le microscope avant d'enlever l'ancien milieu de croissance et aussi après l'ajout de milieu de croissance frais. Sortez le flacon de l'incubateur et examinez les cellules hCMEC/D3 sous microscope de contraste de phase pour s'assurer qu'elles semblent saines.
REMARQUE: La majorité des cellules doivent être fixées au fond du flacon, ont une morphologie en forme de fuseau et souvent, la lumière réfractant autour de leurs membranes est également vu. Le milieu de croissance doit être transparent (non nuageux) et de couleur rose-orange. - Retirer le vieux milieu de croissance du flacon et transférer 10 ml de milieu frais pré-chauffé dans le flacon.
REMARQUE : Le milieu doit être ajouté au côté supérieur du flacon et non directement sur la surface des cellules pour éviter d'affecter l'attachement cellulaire. - Retournez le flacon en position horizontale et balancez-le doucement plusieurs fois et vérifiez les cellules hCMEC/D3 au microscope avant de retourner le flacon à l'incubateur (37 oC, 5 % CO2).
- Observez les cellules sous un microscope à lumière inversée à chaque fois avant et après la manipulation des cellules, à la fois pendant les travaux de culture réguliers et pendant les expériences. Enregistrez tout changement notable dans le nombre de cellules ou la morphologie dans le cahier de laboratoire.
- Réapprovisionnez le milieu de croissance tous les deux jours jusqu'à la confluence à 100 %. Vérifier les cellules sous le microscope avant d'enlever l'ancien milieu de croissance et aussi après l'ajout de milieu de croissance frais. Sortez le flacon de l'incubateur et examinez les cellules hCMEC/D3 sous microscope de contraste de phase pour s'assurer qu'elles semblent saines.
- Passaging cellulaire
- Incuber un nouveau flacon T75 avec 8,5 ml de solution de collagène pour 1 h dans l'incubateur (37 oC, 5 % CO2).
- Retirez la solution de collagène et lavez doucement le flacon avec du PBS stérilisé. Ajouter 10 ml de milieu hCMEC/D3 préréchauffé au nouveau flacon et placer le flacon dans l'incubateur (37 oC, 5 % CO2).
- Sortez le flacon de l'incubateur et examinez les cellules hCMEC/D3 sous un microscope de contraste de phase pour vérifier si les cellules sont 100% confluentes.
- Enlever le milieu cellulaire hCMEC/D3 du flacon contenant des cellules et laver les cellules hCMEC/D3 avec 10 ml de PBS.
REMARQUE : Le FBS ajouté au milieu de croissance contient des inhibiteurs de la protéase tels que l'antitrypsine 1 et la macroglobuline 2. Ceux-ci inhibent le processus de trypsinisation. Ainsi, il est essentiel de laver les cellules avec PBS pour enlever les traces de FBS pour empêcher l'inhibition du processus de trypsinisation. - Ajouter 1 ml de solution trypsine de 0,25 % contenant 0,02 % d'EDTA et trypsiniser pendant 2 à 5 min dans l'incubateur (37 oC, 5 % CO2) (appuyez doucement sur le flacon sur les côtés pour aider au détachement).
REMARQUE : Ne quittez jamais les cellules sous trypsine/EDTA pendant plus de 6 min. - Ajouter 10 ml de milieu hCMEC/D3 préréchauffé pour arrêter le processus de trypsinisation et suspendre la cellule hCMEC/D3 en faisant monter et descendre plusieurs fois. Ensuite, retirez toute la suspension cellulaire du flacon dans un tube de 15 ml.
- Transférer 1 ml de la suspension cellulaire du tube de 15 ml vers le nouveau flacon avec un milieu frais pré-chauffé (cellules de fractionnement 1:10) et retourner le nouveau flacon à l'incubateur.
REMARQUE : Avant de transférer sur le nouveau flacon, pipette la suspension de la cellule de haut en bas plusieurs fois pour minimiser les gradients de concentration cellulaire.
2. Placage cellulaire
- Placez les inserts de culture tissulaire avec des membranes microporeuses (taille des pores : 0,4 m, matériau : polyéthylène téphtalate (PET)) dans une plaque de culture de 24 puits.
- Ajouter 400 l de collagène de type I (0,15 mg/mL) dans chaque insertion de culture tissulaire et incuber pendant 1 h dans l'incubateur co2 (37 oC, 5 % CO2). Basculer doucement la plaque de 24 puits pour permettre même la diffusion de la solution de collagène sur la membrane microporeuse dans les inserts de culture tissulaire.
- Retirez la solution de collagène et lavez doucement la membrane microporeuse avec 0,4 ml de tampon 1x PBS.
- Cellules hCMEC/D3 de plaque avec la densité de 50.000 cellules/cm2 dans les inserts de cellules (15.000 cellules dans 500 'L de milieu).
REMARQUE : Afin de minimiser les différences dans le nombre de cellules dans chaque insert de culture de tissu, la suspension de cellules a été suspendue avec une pipette de 10 ml avant d'ajouter des cellules aux inserts. - Placez la plaque de 24 puits avec la configuration de la culture tissulaire dans un incubateur (37 oC, 5 % DE CO2) pour permettre l'attachement cellulaire et la prolifération.
- Incuber la plaque pendant 7 jours pour permettre aux cellules d'atteindre 100% de confluence. Retirez le milieu de croissance tous les deux jours et transférez 0,5 ml de milieux frais préréchauffés dans des inserts de culture tissulaire.
- Répétez la procédure de placage (étapes 2.2-2.6) sur une plaque de 12 puits, une plaque de 48 puits et une plaque de 96 puits. Utilisez la plaque de 12 puits pour le ballonnement occidental pour déterminer les changements dans l'expression ZO-1. Utilisez la plaque de 48 puits pour la transfection de l'ADN NP. Utilisez la plaque de 96 puits pour l'assiduité de l'ATP afin de déterminer la viabilité cellulaire dans les cellules transfectées.
3. Cinétique de la croissance cellulaire.
- Ensemencer les cellules à une densité de 50.000 cellules/cm2 dans une plaque de culture de tissu de 24 puits recouverte de collagène.
- Chaque jour de l'expérience, retirer le milieu de croissance et laver doucement les cellules deux fois avec 500 OL de 1x PBS. Ensuite, ajouter 30 l de la solution trypsine de 0,25 % contenant 0,02 % d'EDTA et laisser la plaque pendant environ 2 à 5 min dans un incubateur (37 oC, 5 % CO2).
REMARQUE : La formation graduelle d'une monocouche confluente peut affecter l'étendue du détachement cellulaire et il est nécessaire d'augmenter les volumes de trypsine/EDTA comme indiqué ici : 30 'L pendant 1-5 jours après l'ensemencement, 60 'L pendant 6-7 jours après l'ensemencement et 100 'L pendant 8-10 jours après l'ensemencement . - Ajouter soit 470 L, 440 'L ou 400 'L de support de croissance basé sur le volume de la solution trypsine/EDTA ajoutée à l'étape 3.2 pour préparer 500 'L de suspension cellulaire dans chaque puits.
- Suspendre les cellules en pipetting de haut en bas dans chaque puits plusieurs fois et observer les cellules sous un microscope pour s'assurer que toutes les cellules sont suspendues dans le milieu de croissance. Si certaines cellules sont encore attachées au fond de la plaque après le tuyauterie à plusieurs reprises, gratter délicatement les cellules à l'aide d'un grattoir à cellules en plastique pour faciliter le détachement cellulaire.
- Retirez 0,1 ml de suspension cellulaire de la suspension cellulaire de 500 l à l'étape 3.3 et ajoutez-le à un tube de 1,5 ml. Ensuite, ajouter 0,1 ml de 0,4% solution trypan bleu à la suspension cellulaire et bien mélanger.
- Nettoyer un hémacytomètre avec 70% d'alcool isopropyl. Ajouter 20 l de mélange à partir de l'étape 3.5 de chaque côté dans le V-groove et localiser les 16 carrés sous le microscope. Les 16 carrés sont considérés comme une grille. Localisez deux grilles aléatoires de chaque côté de l'hémacytomètre et comptez toutes les cellules vivantes non bleues.
REMARQUE : Cellules qui sont apparues de couleur bleue de exclus du comptage, lt;1% des cellules souillées en bleu à tous les points de temps. - Calculer la densité cellulaire (cellules/cm2) en fonction des formules suivantes.
Moyenne des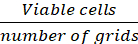 cellules/grilles (Eq.1)
cellules/grilles (Eq.1)
Facteur de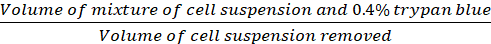 dilutionMD (Eq.2)
dilutionMD (Eq.2)
Densité cellulaire (cellules viables/cm2)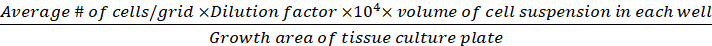
(Eq.3)
Équations 1-3. Les cellules viables sont le nombre de cellules comptées dans chaque grille, le nombre de grilles correspond au nombre de grilles situées sous le microscope, le volume du mélange de suspension cellulaire et 0,4% Trypan bleu est le volume préparé à l'étape 3.5, le volume de suspension cellulaire enlevé est le volume retiré de la suspension cellulaire de 500 l à l'étape 3.5, le volume de suspension cellulaire dans chaque puits est la suspension de cellules de 500 ll de l'étape 3.3, la zone de croissance de la plaque de culture de tissu est la zone de croissance du puits simple dans la plaque de 24 puits.
4. Lucifer jaune perméabilité apparente (ly P app ) assay
- Pour déterminer l'application LY P chaque jour après l'ensemencement, suivez les étapes à partir de 4.3. Pour déterminer l'application P dans les cellules hCMEC/D3 transfectées, ajouter 8,3 L de la formulation de transfection (Figure 1) mélangée à 50 oL de milieu de croissance complet et incubée pendant 4 h. Les formulations de transfection sont décrites à la section 5.
- Après la transfection de 4 h, lavez doucement le côté apical deux fois à l'aide d'un tampon 1x PBS stérile pour éliminer tout mélange de transfection résiduel. Des volumes variables de tampon PBS laissés après l'enlèvement peuvent affecter la concentration de LY dans le côté apaïque. Veillez à ce que le tampon PBS résiduel dans les inserts de culture tissulaire soit complètement enlevé. Aspirez soigneusement les réactifs résiduels et le milieu pour minimiser le détachement cellulaire.
REMARQUE : Cette étape est ignorée lors de la mesurede la perméabilité apparente quotidienne (application P ) des cellules hCMEC/D3. - Retirez le milieu de croissance et ajoutez 1,5 ml de tampon de transport pré-chauffé (37 oC) (25 mM HEPES, 145 mM NaCl, 3 mM KCl, 1 mM CaCl2, 0,5 mM MgCl2, 1 mN NaH2PO4, 5 mM de glucose, pH 7,4) au côté basolatéral.
REMARQUE : Le volume de tampon de transport dans tous les compartiments basolatéraux doit être égal pour assurer l'exactitude du calcul du coefficient de perméabilité. - Ajouter une solution de 58,3 L de 20 M LY sur le côté apical de chaque insert transwell. Économisez 50 L de la solution LY de 20 M pour les mesures de fluorescence. Après avoir enlevé complètement le tampon PBS résiduel du côté apical, ajoutez la solution LY aussi rapidement que possible pour éviter de sécher les cellules hCMEC/D3. Assurer des volumes précis de solution LY dans le côté apaïque.
REMARQUE : Pour minimiser la carie de l'intensité de la fluorescence LY, l'exposition à la lumière devrait être limitée. Une fois la poudre LY reconstituée, la solution doit être stockée à 4 oC, à l'abri de la lumière. - Incuber dans un shaker rotatif (37 oC, 100 tr/min) pendant 60 min. Ensuite, retirez 30 L de l'échantillon LY de chaque compartiment apaïque. Transférer ensuite la solution LY de 20 M et les échantillons latéraux apical dans des tubes pré-étiquetés et diluer l'échantillon par 10 à l'aide d'un tampon de transport.
REMARQUE : Il est nécessaire de diluer la solution de stock de 20 M LY et les échantillons latéraux apical parce que l'intensité élevée de fluorescence de ces échantillons peut potentiellement surcharger et endommager le détecteur de fluorescence du lecteur de microplaque de fluorescence. - Retirer 500 l de chaque compartiment basolatéral et transférer l'échantillon dans des tubes pré-étiquetés.
REMARQUE : Les échantillons sont prélevés à partir de transwell distincts aux points de temps indiqués. Les points de temps sont chaque jour après l'ensemencement à partir du jour 1 jusqu'au jour 10. - Préparer une série de normes LY pour la courbe standard (39,00 nM, 78,13 nM, 156,25 nM, 312 nM, 625 nM, 1250 nM, 2500 nM).
- Ajouter 100 ll de chaque type (en double), apical et basolatéral échantillon à chaque puits dans une plaque noire de 96 puits (Figure 1).
REMARQUE : Les plaques noires absorbent la lumière et réduisent le croisement de fond et de fluorescence entre les puits. - Utilisez un lecteur de microplaque de fluorescence (points de jeu : excitation 428 nm, émission 536 nm) pour mesurer l'intensité de fluorescence LY pour calculer l'application P. Un lecteur de plaque de fluorescence est utilisé pour cette étude.
- Calculez les valeurs de récupération de l'application P et %LY telles que décrites dans le texte manuscrit.
Équation 4. Formule pour calculer les valeursde l'application P .
Calculez le coefficientde perméabilité apparente (application P) et la récupération %LY en utilisant les équations suivantes. Il convient de noter que les valeurs de l'application P peuvent être calculées soit en fonction de la masse25 ou de la concentration de LY.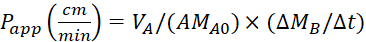 ou
ou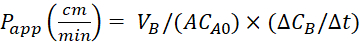
VA - volume dans le compartiment apaïque
VB - volume dans le compartiment basolatéral
A - la surface de la membrane d'insertion transwell (0,3 cm2)
MA0 - la masse initiale dans le compartiment apaïque
Lechangement de masse au fil du temps dans le compartiment basolatéral
CA0 - la concentration initiale dans le compartiment apaïque
CB/ T - le changement de concentration au fil du temps dans le compartiment basolatéral.
Équation 5. Formule pour calculer % DE récupération LY.
Récupération (%)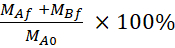
MAf est la masse dans le compartiment apaïque au point de fin, MBf est la masse dans le compartiment basolatéral au point de fin, MA0 est la masse initiale dans le compartiment apaïque. 26 Note : La masse initiale est calculée en fonction du volume de la solution LY de 20 M dans l'étape 4.5. Cette expérience a toujours été faite à l'aide de cellules sous le passage numéro 35 et a été menée quatre fois de façon indépendante.
5. Épuisement du calcium
- Retirer le milieu de croissance des inserts transwell et de la plaque de 12 puits et laver doucement le côté apical et la plaque de 12 puits à l'aide d'un milieu mi-chauffé sans calcium (minimum Essential Medium Eagle Spinner (S-MEM) moyen) pour enlever les ions de calcium des inserts transwell et une assiette de 12 puits.
- Ajouter 500 l ou 2 ml de S-MEM 1xmedium préréchauffé aux inserts ou à la plaque de 12 puits et couver pendant 24 h dans l'incubateur (37 oC, 5 % CO2). Après l'incubation, retirer le milieu S-MEM 1x et laver les cellules une fois à l'aide d'un tampon 1x PBS préréchauffé.
- Suivez les étapes 4.4-4.10 décrites dans la section 4 pour le traitement DEL et les étapes suivantes. Suivre les étapes 8.1.2-8.2.4 décrites à la section 8 pour les ballonnements occidentaux et les étapes subséquentes.
6. Transfection
- Préparation des nanoparticules d'ADN (NP d'ADN).
- Diluer la solution de stock de plasmide gWIZ-Luc dans un tampon d'acétate de sodium (NaAc) de 10 mM (PH 5,0) et permettre à la solution d'ADN de se tenir debout pendant 10 min à température ambiante (RT).
REMARQUE : La NP d'ADN contenant principalement des molécules d'ADN uniques peut être préparée à des concentrations d'ADN de 20 à 40 g/mL23. Ainsi, la solution de stock de plasmide de gWIZ-Luc doit être diluée. Le stock de plasmides gWIZ-Luc congelé doit être complètement décongelé sur la glace afin de minimiser le stress de température. Vortex doucement la solution de stock d'ADN dilué pour 30 s sur un vortexer standard de banc réglé à la position de bouton 3-5. - Calculez les ratios N/P souhaités, utilisés ici comme paramètre numérique pour refléter la composition nP.

Équation 6. Calcul du ratio N/P : rapport des grains de beauté des groupes aminés de polymères cationiques à ceux des groupes de phosphate de l'ADN. La masse des polymères cationic signifie la quantité totale pesée de polymère cationic ; (Masse/Charge) les polymères cationic se réfèrent au poids moléculaire du polymère cationic (poly (éthylèneglycol)5k-bloc-polyaspartamide avec 48 chaînes latérales de diethylènetriamine (PEG-DET)) normalisées au nombre d'amines primaires chargées (48) par polymère cationic ( mol/mol), cette valeur pour notre polymère est 306 Da; La masse d'ADN signifie la quantité totale d'ADN utilisée dans la formulation obtenue en multipliant le volume et la concentration en mg/mL; (Masse/Charge) L'ADN se réfère au poids moléculaire de l'ADN normalisé au nombre de groupes de phosphate par ADN à double brin (325 Da par nucléobase).
REMARQUE : Le tableau de préparation de l'ADN NP (tableau 1) contient la recette de formulation des différents échantillons testés dans nos expériences. DNA NP ont été préparés en utilisant une technique de titration rapide. La solution de polymère PEG-DET a été ajoutée le long des parois du tube tout en maintenant le tube en position horizontale. Ensuite, le tube a été commuté à une position verticale, suivie d'un vortex rapide à la vitesse maximale pendant 10s. L'ADN NP ont été autorisés à se tenir pendant 30 min à RT avant d'utiliser. La règle de base pour le dosage de nP d'ADN est 0.5 'g D'ADN pour environ 1 cm2 zone de croissance. Ainsi, pour chaque insertion de transwell/chaque puits dans une plaque de 48 puits/chaque puits dans une plaque de 96 puits, préparez l'ADN NP à N/P 10 contenant 0.157/0.5/0.195 g/puits d'ADN de gWIZ-Luc. - Pour les échantillons contenant une concentration indiquée de Poloxamer P84 (P84), ajouter le P84 à l'ADN NP et le vortex pendant 5 s. La concentration finale de P84 dans chaque échantillon est soit de 0,01% soit de 0,03% wt.
- Diluer la solution de stock de plasmide gWIZ-Luc dans un tampon d'acétate de sodium (NaAc) de 10 mM (PH 5,0) et permettre à la solution d'ADN de se tenir debout pendant 10 min à température ambiante (RT).
- Transfection de NP d'ADN dans une configuration de plaque de 48 puits
- Ensemencer les cellules avec la densité de 50.000 cellules/cm2 dans une plaque de puits 48 et se développer jusqu'à la confluence dans l'incubateur (37 oC, 5% CO2).
- Pour chaque groupe de traitement, mélanger 25 ll de l'échantillon indiqué (formulation de transfection) et 150 oL de milieu de croissance complet et 175 l de ce mélange à chaque puits.
- Observez les cellules hCMEC/D3 au microscope pour s'assurer que les cellules semblent saines et sont 100% confluentes au moment de l'expérience.
- Retirez le milieu de croissance des puits et du mélange de transfection de 175 l à chaque puits. Ensuite, incuber la plaque pendant 4 h dans l'incubateur (37 oC, 5 % CO2).
- Après 4 h, retirer le mélange de transfection et laver les cellules hCMEC/D3 avec un tampon stérile 1x PBS préréchauffé.
REMARQUE : Pour réduire au minimum le détachement accidentel des cellules hCMEC/D3 de la surface de plaque/insert pendant les étapes de lavage, soigneusement pipette assez stérile PBS le long des murs des puits et enlever toutes les nanoparticules dans le support de culture résiduel. - Basculer doucement la plaque à quelques reprises et aspirer soigneusement et jeter le lavage PBS et ajouter 500 L de hCMEC/D3 milieu de culture pré-chauffé.
- Examiner au microscope les cellules et enregistrer les observations sur la morphologie cellulaire et les effets possibles de la transfection.
- Incuber pendant 24 h dans l'incubateur de culture cellulaire de 37 oC pour permettre la production de luciferase. Après 24 h, retirer complètement le milieu de croissance et laver les cellules une fois avec 1x PBS pré-chauffé.
- Lyse les cellules transfectées en ajoutant 100 L de lyse de culture de cellules de luciferase glacée 1x réagent par puits.
- Pour la mesure de la teneur en protéines de la luciferase, ajouter 20 l de lysate cellulaire et 100 l de tampon d'analyse de luciferase (20 mM de glycylglycine (pH 8), 1 mMg MgCl2, 0,1 mM EDTA, 3,5 mM DTT, 0,5 mM ATP, 0,27 mM coenzyme A) dans un tube de 1,5 mL.
- Lisez la luminescence de l'échantillon décrit à l'étape 6.2.10 sur un Luminomètre avec un auto-injecteur unique.
REMARQUE : La luminescence doit être intégrée au-dessus de 10 s avant la lecture. - Mesurez la quantité totale de protéines cellulaires dans le lysate à l'aide d'un kit d'assay d'acide bicinchoninique (BCA) en suivant le protocole du fabricant.
- Calculer et exprimer l'expression du gène de la luciferase en tant qu'unités de lumière relative (RLU) par protéine cellulaire totale.
7. Luminescent ATP d'assay
- Ensemencer les cellules avec la densité de 50.000 cellules/cm2 dans une plaque de puits 96 et se développer jusqu'à la confluence dans l'incubateur (37 oC, 5% CO2).
- Transfect les cellules avec 9,7 L de la formulation de transfection (les détails de préparation sont dans la section 6) et 58,4 L de milieu de croissance complet pendant 4 h.
- Retirez le mélange de transfection et lavez doucement les cellules avec un tampon PBS 1x préréchauffé deux fois pour enlever complètement les réactifs de traitement.
REMARQUE : Différents volumes de mémoire tampon résiduel pourraient diluer les réactifs d'analyse ATP dans une mesure différente et peuvent potentiellement affecter les données. - Mélanger 75 'L de milieu frais pré-chauffé et le réactif d'essai ATP dans une dilution 1:1 à l'aide d'une pipette multicanal. Assurez-vous que le niveau de liquide dans tous les conseils de pipette multicanal est le même.
- Placer l'assiette sur un shaker de noix pendant 15 min à température ambiante. Après 15 min d'ajout des réactifs, transférer 60 l de chaque échantillon dans une plaque blanche de 96 puits.
REMARQUE : Les plaques blanches sont mieux pour refléter la lumière de sortie que les plaques claires ou noires. - Pop toutes les bulles d'air à l'aide d'une aiguille avant de lire la plaque. Lisez la plaque sur un luminomètre avec un temps d'intégration de 1 s. Lisez la plaque dans les 20 minutes après l'ajout de réactifs d'assay ATP. Le moment est critique pour la comparaison entre les différentes plaques, parce que le signal de luminescence est transitoire avec un taux de décomposition rapide.
- Calculer le pourcentage (%) viabilité cellulaire à l'aide de cette formule : (luminescence des cellules transfectées/luminescence du contrôle, cellules non traitées) x 100.
8. Le ballonnement occidental pour la mesure de la protéine de jonction serrée ZO-1
-
Lyse cellulaire et extraction de protéines
REMARQUE : Toutes les étapes pour l'extraction des protéines à partir de cellules doivent être effectuées à 2-8 oC.- Ensemencer les cellules à une densité de 50.000 cellules/cm2 dans une plaque de culture de tissu de 12 puits recouverte de collagène.
- Le jour 3, jour 5, jour 7, jour 10 après l'ensemencement et le jour 7 (cellules pré-incubées avec le milieu sans calcium), enlevez le milieu de croissance et lavez doucement les cellules deux fois avec 2 ml de 1x PBS glacé. Ensuite, ajoutez un mélange de 300 l de tampon de lyse RIPA contenant 3 aprotinins de 3 g/mL dans chaque puits.
REMARQUE : Le mélange doit être fraîchement préparé et conservé sur la glace. Aprotinin est utilisé pour empêcher les protéases présentes dans les lysates de dégrader la protéine d'intérêt. - Après deux cycles de gel-dégel (-80 oC), gratter les cellules à l'aide d'un grattoir à cellules en plastique froid. Recueillir les lysates cellulaires dans des tubes de microfuge. Ensuite, centrifuger les tubes à 200 x g pendant 30 min à 4 oC.
- Recueillir le supernatant dans des tubes propres et les placer sur la glace. Mesurez la quantité totale de protéines cellulaires dans les lysates à l'aide du kit d'assi du BCA en suivant le protocole du fabricant.
-
Ballonnement s'amonce pour la détection de la protéine de jonction serrée ZO-1
- Dénaturer les aliquots des homogénéités totales contenant 40 g de protéines totales avec 1 x tampon Laemmli à 95 oC, 5 min et soumis à l'électrophoresis dans une réduction de 6-7,5% de sodium dodecyl sulfate gel polyacrylamide (SDS-PAGE) (90 V, 10 min par gel d'empilage, 120 V à V gels de résolution).
REMARQUE : Lorsque vous chargez les échantillons ou les normes, n'oubliez pas de charger lentement et soigneusement dans chaque voie, en faisant attention de ne pas briser le puits dans le processus. - Transférer les protéines séparées sur une membrane de nitrocellulose avec un tampon de transfert de pH 8,5 qui contient 192 mM de glycine, 25 mM de base Tris, 10 % de méthanol et 0,1 % de SDS (75 V, 110 min à température ambiante).
REMARQUE : Ne touchez pas la membrane. Utilisez 70% de forceps en plastique lavés à l'isopropanol pour manipuler la membrane. Afin de transférer avec succès la protéine ZO-1 (MW 200 kDa) à la membrane, le pH devrait être d'environ 8,3-8,5. Si le tampon de transfert est plus acide que cela, le transfert ne se produirait pas. Si les bandes d'échelle sont visibles encore sur le gel, il serait utile d'augmenter le temps de transfert et la concentration de SDS. - Après avoir lavé la membrane en utilisant la solution saline tamponnée Tris contenant 0,1 %Tween 20 (T-TBS), utilisez la solution de blocage (1:1 LiCOR-Odyssey Block: 1x Tris tamponné saline) pour bloquer les membranes pendant 60 min.
- Couper soigneusement la membrane en deux bandes. Incuber avec deux anticorps primaires (anticorps monoclonaux ZO-1, dilution, 1: 900, et glycéraldéhyde 3-phosphate déshydrogénase (GAPDH) anticorps, dilution, 1: 10.000) nuit à 4 oC. Ensuite, incuber les membranes ZO-1 et GAPDH avec l'âne anti-souris IgG (dilution, 1:50,000). Après avoir lavé les membranes à l'aide de T-TBS, imagez les membranes du canal 700 sur un imageur 16 bits.
- Dénaturer les aliquots des homogénéités totales contenant 40 g de protéines totales avec 1 x tampon Laemmli à 95 oC, 5 min et soumis à l'électrophoresis dans une réduction de 6-7,5% de sodium dodecyl sulfate gel polyacrylamide (SDS-PAGE) (90 V, 10 min par gel d'empilage, 120 V à V gels de résolution).
Résultats
Tout d'abord, nous avons déterminé l'effet du temps de culture sur la perméabilité de LY pour déterminer les cinétique apparentes de la formation de TJ. Les valeurs moyennes de l'application LY P du jour 1 à 10 après l'ensemencement sont indiquées dans la figure 2a. Le jour 1, l'application P moyenne était de 4,25 x 10-4 cm/min et a légèrement chuté à 3,32 x 10-4 cm/min le jour 2. La valeur ...
Discussion
Un rôle clé du BBB est d'empêcher l'échange d'ions non essentiels et de substances toxiques entre la circulation systémique et le cerveau pour maintenir l'hémostase du microenvironnement neural. Une des caractéristiques du BBB est la capacité des cellules endothéliales capillaires à former des jonctions serrées (TJs) qui scellent efficacement la voie paracellulaire du transport. Nous avons démontré un essai d'application de LY P comme méthode quantitative pour déterminer la cinétique apparente ...
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Les auteurs sont reconnaissants du soutien financier du New Investigator Award 2017 de l'American Association of Pharmacy, d'un prix Hunkele Dreaded Disease de l'Université Duquesne et des fonds de démarrage de l'École de pharmacie pour le laboratoire Manickam. Nous tenons à remercier le laboratoire Leak (Université Duquesne) pour l'aide à l'ouest de l'adjonction et l'utilisation de leur imageur Odyssey 16 bits. Nous aimerions également inclure une note spéciale d'appréciation pour Kandarp Dave (laboratoire Manickam) pour l'aide avec le ballonnement occidental.
matériels
| Name | Company | Catalog Number | Comments |
| hCMEC/D3 cell line | Cedarlane Laboratories | 102114.3C-P25 | human cerebral microvascular endothelial cell line |
| gWizLuc | Aldevron | 5000-5001 | Plasmid DNA encoding luciferase gene |
| lucifer yellow CH dilithium salt | Invitrogen | 155267 | |
| Transwell inserts with polyethylene terephthalate (PET) track-etched membranes | Falcon | 353095 | |
| Tissue culture flask | Olympus Plastics | 25-207 | |
| 24-well Flat Bottom | Olympus Plastics | 25-107 | |
| Black 96-Well Immuno Plates | Thermo Scientific | 437111 | |
| S-MEM 1X | Gibco | 1951695 | Spinner-minimum essential medium (S-MEM) |
| EBM-2 | Clonetics | CC-3156 | Endothelial cell basal medium-2(EBM-2) |
| phosphate-buffered saline 1X | HyClone | SH3025601 | |
| Collagen Type I | Discovery Labware, Inc. | 354236 | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23227 | |
| Cell Culture Lysis 5X Reagent | Promega | E1531 | |
| Beetle Luciferin, Potassium Salt | Promega | E1601 | |
| SpectraMax i3 | Molecular Devices | Fluorescence Plate Reader | |
| Trypan Blue Solution, 0.4% | Gibco | 15250061 | |
| ZO-1 Polyclonal Antibody | ThermoFisher | 61-7300 | |
| anti-GAPDH antibody | abcam | ab8245 | |
| Alexa Fluor680-conjugated AffiniPure Donkey Anti-Mouse LgG(H+L) | Jackson ImmunoResearch Inc | 128817 | |
| 12-well, Flat Bottom | Olympus Plastics | 25-106 | |
| RIPA buffer (5X) | Alfa Aesar | J62524 | |
| Aprotinin | Fisher BioReagents | BP2503-10 | |
| Odyssey CLx imager | LI-COR Biosciences | for scanning western blot membranes |
Références
- Abbott, N. J., Patabendige, A. A., Dolman, D. E., Yusof, S. R., Begley, D. J. Structure and function of the blood-brain barrier. Neurobiology Of Disease. 37 (1), 13-25 (2010).
- Griep, L. M., et al. BBB on chip: microfluidic platform to mechanically and biochemically modulate blood-brain barrier function. Biomedical Microdevices. 15 (1), 145-150 (2013).
- Camos, S., Mallolas, J. Experimental models for assaying microvascular endothelial cell pathophysiology in stroke. Molecules. 15 (12), 9104-9134 (2010).
- Cucullo, L., et al. Immortalized human brain endothelial cells and flow-based vascular modeling: a marriage of convenience for rational neurovascular studies. Journal of Cerebral Blood Flow & Metabolism. 28 (2), 312-328 (2008).
- Wolff, A., Antfolk, M., Brodin, B., Tenje, M. In vitro Blood-Brain Barrier Models-An Overview of Established Models and New Microfluidic Approaches. Journal of Pharmaceutical Sciences. 104 (9), 2727-2746 (2015).
- Weksler, B., Romero, I. A., Couraud, P. O. The hCMEC/D3 cell line as a model of the human blood brain barrier. Fluids and Barriers of the CNS. 10 (1), 16 (2013).
- Ohtsuki, S., et al. Quantitative targeted absolute proteomic analysis of transporters, receptors and junction proteins for validation of human cerebral microvascular endothelial cell line hCMEC/D3 as a human blood-brain barrier model. Molecular Pharmaceutics. 10 (1), 289-296 (2013).
- Tornabene, E., Brodin, B. Stroke and Drug Delivery--In vitro Models of the Ischemic Blood-Brain Barrier. Journal of Pharmaceutical Sciences. 105 (2), 398-405 (2016).
- Weksler, B., Romero, I. A., Couraud, P. O. The hCMEC/D3 cell line as a model of the human blood brain barrier. Fluids and Barriers of the CNS. 10 (1), 16 (2013).
- Llombart, V., et al. Characterization of secretomes from a human blood brain barrier endothelial cells in-vitro model after ischemia by stable isotope labeling with aminoacids in cell culture (SILAC). Journal of Proteomics. 133, 100-112 (2016).
- Weksler, B. B., et al. Blood-brain barrier-specific properties of a human adult brain endothelial cell line. The FASEB Journal. 19 (13), 1872-1874 (2005).
- Avdeef, A. How well can in vitro brain microcapillary endothelial cell models predict rodent in vivo blood-brain barrier permeability?. European Journal of Pharmaceutical Sciences. 43 (3), 109-124 (2011).
- Rahman, N. A., et al. Immortalized endothelial cell lines for in vitro blood-brain barrier models: A systematic review. Brain Research. 1642, 532-545 (2016).
- Cecchelli, R., et al. A stable and reproducible human blood-brain barrier model derived from hematopoietic stem cells. PLoS One. 9 (6), e99733 (2014).
- Shao, X., et al. Development of a blood-brain barrier model in a membrane-based microchip for characterization of drug permeability and cytotoxicity for drug screening. Analytica Chimica Acta. 934, 186-193 (2016).
- Walter, F. R., et al. A versatile lab-on-a-chip tool for modeling biological barriers. Sensors and Actuators B: Chemical. 222, 1209-1219 (2016).
- Cecchelli, R., et al. In vitro model for evaluating drug transport across the blood–brain barrier. Advanced Drug Delivery Reviews. 36, (1999).
- Cecchelli, R., et al. Modelling of the blood–brain barrier in drug discovery and development. Nature reviews Drug discovery. 6 (8), 650 (2007).
- Reichel, A., Begley, D. J., Abbott, N. J. . The Blood-Brain Barrier. , 307-324 (2003).
- Deli, M. A., Ábrahám, C. S., Kataoka, Y., Niwa, M. Permeability Studies on In vitro Blood–Brain Barrier Models: Physiology, Pathology, and Pharmacology. Cellular and Molecular Neurobiology. 25 (1), 59-127 (2005).
- Ren, T. B., et al. A General Method To Increase Stokes Shift by Introducing Alternating Vibronic Structures. Journal of the American Chemical Society. 140 (24), 7716-7722 (2018).
- Pack, D. W., Hoffman, A. S., Pun, S., Stayton, P. S. Design and development of polymers for gene delivery. Nature Reviews Drug Discovery. 4 (7), 581-593 (2005).
- Oupický, D., Konák, C., Ulbrich, K., Wolfert, M. A., Seymour, L. W. DNA delivery systems based on complexes of DNA with synthetic polycations and their copolymers. Journal of Controlled Release. 65, (2000).
- Couraud, P. O. . The hCMEC/D3 CELL LINE: IMMORTALIZED HUMAN CEREBRAL MICROVASCULAR ENDOTHELIAL CELLS As a model of human Blood-Brain Barrier. , (2012).
- Youdim, K. u. r. e. s. h. A., A, A. A. a. N. J. In vitro trans-monolayer permeability calculations: often forgotten assumptions. research focus reviews. 8, (2003).
- Eigenmann, D. E., Xue, G., Kim, K. S., Moses, A. V., Hamburger, M., Oufir, M. Comparative study of four immortalized human brain capillary endothelial cell lines, hCMEC/D3, hBMEC, TY10, and BB19, and optimization of culture conditions, for an in vitro blood-brain barrier model for drug permeability studies. Fluid and Barriers of the CNS. 10 (33), (2013).
- Hubatsch, I., Ragnarsson, E. G. E., Artursson, P. Determination of drug permeability and prediction of drug absorption in Caco-2 monolayers. Nature Protocols. 2 (9), 2111-2119 (2007).
- Balda, M. S., Anderson, J. M. Two classes of tight junctions are revealed by ZO-1 isoforms. The American Physiological Society. , (1992).
- Brown, R. C., Davis, T. P. Calcium Modulation of Adherens and Tight Junction Function: A Potential Mechanism for Blood-Brain Barrier Disruption After Stroke. Stroke. 33 (6), 1706-1711 (2002).
- Gorodeski, G., Jin, W., Hopfer, U. Extracellular Ca2+ directly regulates tight junctional permeability in the human cervical cell line CaSki. American Journal of Physiology-Cell Physiology. 272 (2), C511-C524 (1997).
- Stuart, R. O., Sun, A., Panichas, M., Hebert, S. C., Brenner, B. M., Nigam, S. K. Critical Role for lntracellular Calcium in Tight Junction Biogenesis. Journal of Cellular Physiology. 159, (1994).
- Tobey, N. A. Calcium-switch technique and junctional permeability in native rabbit esophageal epithelium. American Journal of Physiology-Gastrointestinal and Liver Physiology. , (2004).
- Tobey, N. A., Argote, C. M., Hosseini, S. S., Orlando, R. C. Calcium-switch technique and junctional permeability in native rabbit esophageal epithelium. American Journal of Physiology-Gastrointestinal and Liver Physiology. 286, (2004).
- Posimo, J. M., et al. Viability assays for cells in culture. Journal of visualized experiments : JoVE. (83), e50645 (2014).
- Cipolla, M. J., Crete, R., Vitullo, L., Rix, R. D. Transcellular transport as a mechanism of blood-brain barrier disruption during stroke. Frontiers in Bioscience. 9 (3), 777-785 (2004).
- Kreuter, J. Influence of the surface properties on nanoparticle-mediated transport of drugs to the brain. Journal of nanoscience and nanotechnology. 4 (5), 484-488 (2004).
- Markoutsa, E., et al. Uptake and permeability studies of BBB-targeting immunoliposomes using the hCMEC/D3 cell line. European Journal of Pharmaceutics and Biopharmaceutics. 77 (2), 265-274 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon