È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Lucifero Giallo - Un Robusto Indicatore di Permeabilità Paracellulare in un Modello Cellulare della Barriera Sangue-Cervello Umano
In questo articolo
Riepilogo
Presentiamo un saggio di fluorescenza per dimostrare che Lucifero Giallo (LY) è un marcatore robusto per determinare l'apparente permeabilità paracellulare dei monostrati cellulari hCMEC/ D3, un modello in vitro della barriera emato-encefalica umana. Abbiamo usato questo saggio per determinare la cinetica di una formazione di monostrato confluente nelle cellule hCMEC/D3 coltivate.
Abstract
La barriera emato-encefalica BBB è costituita da cellule endoteliali che formano una barriera tra la circolazione sistemica e il cervello per impedire lo scambio di ioni non essenziali e sostanze tossiche. Giunzioni strette (TJ) sigillano efficacemente lo spazio paracellulare nei monostrati, causando una barriera intatta. Questo studio descrive un saggio di fluorescenza basato su LY che può essereutilizzato per determinare il suo coefficiente di permeabilità apparente (P app) e a sua volta può essere utilizzato per determinare la cinetica della formazione di monostrati confluenti e la risultante giunzione stretta risultante l'integrità della barriera nei monostrati hCMEC/D3. Dimostriamo inoltre un'utilità aggiuntiva di questo saggio per determinare l'integrità funzionale di TJ nelle cellule trasfette. I nostri dati dal saggiodell'app LY P mostrano che le cellule hCMEC/D3 seminato in una configurazione transwell limitano efficacemente il trasporto paracellulare LY 7 giorni dopo la coltura. Come utilità aggiuntiva del saggio presentato, dimostriamo anche che la trasfezione delle nanoparticelle di DNA non altera il trasporto paracellulare LY nei monostrati hCMEC/D3.
Introduzione
La barriera emato-encefalica (BBB) è la barriera protettiva che limita l'afflusso di componenti plasmatici nel tessuto cerebrale ed è costituita da cellule endoteliali cerebrali insieme a cellule di supporto come i periciti. Il ruolo principale di BBB è quello di fungere da barriera che sigilla lo spazio tra il sangue periferico e il sistema nervoso centrale (SNC) per mantenere l'emostasi del microambiente neurale1,2. Le cellule endoteliali capillari del cervello sigillano efficacemente la via paracellulare attraverso la formazione di giunzioni strette intercellulari (TJ)1. Questa barriera protettiva consente al glucosio e ai nutrienti selezionati di entrare nel cervello mentre impedisce alla maggior parte degli ioni, sostanze tossiche e farmaci di passare attraverso questa barriera stretta. Oltre al suo ruolo protettivo, la funzione di barriera naturale della BBB rappresenta una grave sfida nello sviluppo di sistemi di somministrazione di farmaci rivolti al SNC.
I modelli di coltura cellulare in vitro del BBB sono strumenti utili per studiare la sua biologia e per comprendere gli effetti del trattamento farmacologico sull'integrità delle barriere TJ. Abbiamo usato la linea cellulare endoteliale cerebrale cerebrale (hCMEC/D3) come modello in vitro poiché è un modello accettato di endotelio cerebrale umano3 e riassume molte funzioni del BBB umano. hCMEC/D3 è una delle linee cellulari più comunemente utilizzate per la modellazione del BBB in vitro4,5,6,7,8,9. Nonostante i suoi valori relativamente bassi di resistenza elettrica transeteliale (TEER), una misura di tenuta della barriera, questa linea cellulare mantiene la maggior parte delle proprietà morfologiche e funzionali delle cellule endoteliali cerebrali, anche come monocoltura in assenza di cellule gliali coculture6,7. La linea cellulare hCMEC/D3 esprime più marcatori BBB, compresi trasportatori attivi e recettori fino al passaggio 35 circa senza subire la dedifferenziazione ai fenotipi instabili6,7,9 ,10,11. La caratteristica più sorprendente della linea cellulare hCMEC/D3 come modello BBB in vitro è la sua capacità di formare TJ5,9,11,12. Va notato che, sebbene i modelli BBB derivati da cellule staminali abbiano mostrato una maggiore permeabilità in molti studi rispetto alla linea cellulare hCMEC/D3 e esprimano alcuni marcatori BBB, devono ancora evolversi come il modello cellulare BBB più comune13. È importante sottolineare che i modelli BBB derivati dalle cellule staminali devono essere caratterizzati rispetto ai numeri massimi di passaggio che consentono alle cellule di mantenere i fenotipi BBB stabili14.
Tre metodi primari sono comunemente utilizzati per determinare l'integrità della barriera TJ,compresa la misurazione del TEER, la misurazione del coefficiente di permeabilità apparente (P app) di piccole molecole di tracciante idrofilo come il saccarosio, l'inulina, il giallo di Lucifero, ecc. e immunostaining di marcatori molecolari noti di TJ come claudin-5, zo-1, occludina, ecc5. TEER è un metodo relativamente semplice e quantitativo che misura la resistenza elettrica attraverso i monostrati cellulari coltivati su un substrato di membrana porosa5 . Tuttavia, i valori TEER possono essere influenzati da variabili sperimentali come la composizione del mezzo di coltura e il tipo di strumento di misurazione. Una probabile combinazione di questi fattori porta ad un'ampia distribuzione dei valori di TEER che vanno da 2 a 1150 cm2 nella linea cellulare hCMEC /D3 coltivata per 2-21 giorni13. L'immunostaining è un metodo visivo per determinare la presenza di proteine TJ etichettando la proteina mirata utilizzando anticorpi. Tuttavia, l'immunostaining comporta una serie di passaggi sperimentali, tra cui la necessità di fissare/permeabilizzare le cellule che possono provocare artefatti sperimentali e i segnali fluorescenti possono svanire nel tempo. I fattori di cui sopra possono portare a errori soggettivi che influiscono sulla qualità dei dati.
L'obiettivo principale di questo lavoro è quello di presentare un saggio di permeabilità apparente basata su LY determinare la cinetica di una formazione di monostrato confluente in cellule hCMEC/D3 coltivate. Sebbene altri sistemi BBB in vitro avanzati, come i sistemi di cocoltura, i sistemi microfluidici, siano imitazioni fisiologicamente più rilevanti con una funzione di barriera significativamente migliorata15,16,17, l'hCMEC/D3 l'installazione del transwell è un modello semplice e affidabile per stimare la cinetica della formazione di TJ e selezionare rapidamente l'effetto di diverse formulazioni farmacologiche sulla funzione di barriera. In generale, i valoridell'app P sono coerenti per vari soluti idrofili nei monostrati hCMEC/D3. Ad esempio, i valori diApp P segnalati per vari soluti di massa molecolare bassa (come saccarosio, mannitolo, LY, ecc.) in diversi modelli BBB in vitro sono nell'ordine di 10-4 cm/min5,18,19 , 20.Nella nostra configurazione sperimentale, le cellule endoteliali cerebrali vengono semiate su una membrana micropororosa rivestita di collagene per l'attaccamento cellulare e la formazione di monostrato per imitare la barriera in vivo. Il LY aggiunto nel lato apicale dovrebbe attraversare le giunzioni strette intercellulari e accumularsi nel lato basolaterale. Concentrazioni maggiori di LY nel lato basolaterale indicano una barriera immatura e non completamente funzionale, mentre concentrazioni più basse riflettono il trasporto limitato a causa della presenza di TJ funzionali con conseguente barriera matura.
LY è un tintura idrofilo con picchi di eccitazione/emissione distinti ed evita la necessità di radioetichettare molecole traccianti come saccarosio, mannitolo o inulina. Così, i valori di fluorescenza di LY possono essere utilizzati per calcolare direttamente la sua permeabilità paracellulare attraverso i monostrati BBB. Inoltre, rispetto a molti coloranti disponibili in commercio utilizzati in campi biomedici che soffrono di piccoli spostamenti Di Stokes come la fluoresceina21, lo spostamento di Stokes di LY è di circa 108 nm con sufficiente separazione spettrale, consentendo così i dati di fluorescenza LY come robusta lettura per determinare la permeabilità paracellulare. Abbiamo usato il gonfiore occidentale come tecnica ortogonale per dimostrare i cambiamenti nell'espressione della proteina del marcatore di giunzione stretta, o-1, nel tempo della coltura. L'espressione di O-1 rilevata tramite il gonfiore occidentale viene utilizzata per integrare i datidell'app LY P e, in combinazione, questi dati suggeriscono che i cambiamenti osservati nei valoridell'app LY P riflettono la formazione di un monostrato con un graduale aumento espressione del marcatore di giunzione stretto, .
Come sottolineato in precedenza, l'obiettivo centrale di questo lavoro è quello di dimostrare un saggio LY come una semplice tecnica per monitorare la formazione di un monostrato confluente con giunzioni strette funzionali. Tuttavia, per dimostrare un'ulteriore utilità del saggio sviluppato, abbiamo misuratol'app LY P in monostrati hCMEC/D3 trasaffetti da nanoparticelle di DNA. Gli acidi nucleici possono essere condensati in nanoparticelle polielettrolitiche con un diametro di 100-200 nm attraverso l'interazione elettrostatica tra i gruppi di polimeri caricati positivamente e i gruppi di fosfati caricati negativamente di acidi nucleici22, 23. Ci riferiamo a questi complessi come nanoparticelle di DNA (DNA NP) nel nostro lavoro. Mentre la nostra intenzione è quella di trasfecare le cellule ed esprimere la proteina desiderata, dobbiamo garantire che le proprietà della barriera dei monostrati hCMEC/D3 non siano compromesse. I nostri dati suggeriscono che un regime di trasfezione genica luciferasi standard di 4 h non cambia misurabilmente la permeabilità LY dimostrando l'utilità del saggiodell'app LY P per determinare i cambiamenti nell'integrità della barriera TJ.
Protocollo
1.Coltura cellulare generale hCMEC/D3
- Rianimazione delle cellule congelate
NOTA: tutti i lavori di manutenzione e di manutenzione della coltura cellulare sono stati eseguiti all'interno di una cappa sterile di biosicurezza. I mezzi di coltura, gli integratori e i reagenti sono stati acquistati come prodotti sterili o sterilizzati tramite filtrazione utilizzando un filtro di membrana di 0,22 m per prevenire la contaminazione microbica.- Aggiungere 8,5 mL di soluzione di collagene (0,15mg/mL) in un flacone di coltura tissutale (75 cm2 area di crescita; indicato d'ora in poi come T75) e posizionarlo in un'incubatrice (37 , 5% CO2)per 1 h.
- Rimuovere la soluzione di collagene e lavare delicatamente il pallone con salina sterilizzata con buffer di fosfato (PBS). Aggiungere 15 mL di mezzo di crescita completa al pallone e lasciare nell'incubatrice CO2 per 15 min.
NOTA: Il mezzo completo (concentrazione finale) contiene Endothelial Cell Growth Basal Medium-2 (500 mL) integrato con siero bovino fetale (5%), penicillina-streptomicina (1%), idrocortisone (1,4 m), acido asbiccor (5 g/mL), lipidi chimico definito (1/100), HEPES (10 mM) e fattore di crescita fibroblasto di base (1 ng/mL). - Spostare una crioviale di cellule hCMEC/D3 congelate dal serbatoio di azoto liquido e scongelare rapidamente la fiala in un bagno d'acqua a 37 gradi centigradi (< 1 min).
- Una volta che è visibile solo un piccolo fiocco di ghiaccio, aspirare rapidamente e trasferire le cellule al flacone contenente mezzo preriscaldato. Scuotere delicatamente il pallone per consentire la miscelazione delle cellule con il mezzo di crescita.
- Posizionare il pallone nell'incubatrice (37 gradi centigradi, 5% CO2) e osservare le cellule al microscopio leggero dopo 2 h per assicurarsi che le cellule siano attaccate.
- Una volta che le cellule si attaccano al fondo del pallone, rimuovere il vecchio mezzo di crescita e aggiungere 10 mL di fresco mezzo di crescita preriscaldato per sostituire dimetilsulfoxide nel vecchio mezzo di crescita24.
- Dopo 24 h, controllare al microscopio leggero per osservare le cellule a forma di mandrino e sostituire il vecchio mezzo di crescita con un mezzo di crescita fresco preriscaldato.
- Manutenzione della coltura cellulare
- Rifornire il mezzo di crescita a giorni alterni fino alla confluenza al 100%. Controllare le cellule al microscopio prima di rimuovere il vecchio mezzo di crescita e anche dopo l'aggiunta di un nuovo mezzo di crescita. Estrarre il pallone dall'incubatrice ed esaminare le cellule hCMEC/D3 al microscopio a contrasto di fase per assicurarsi che appaiano sane.
NOTA: La maggior parte delle cellule deve essere attaccata alla parte inferiore del pallone, avere una morfologia a forma di mandrino e spesso si vede anche una luce che si rifraga intorno alle loro membrane. Il mezzo di crescita deve essere trasparente (non nuvoloso) e di colore rosato-arancione. - Rimuovere il vecchio mezzo di crescita dal pallone e trasferire 10 mL di mezzo fresco preriscaldato nel pallone.
NOTA: Il mezzo deve essere aggiunto al lato superiore del pallone e non direttamente sulla superficie delle celle per evitare di influenzare l'attaccamento cellulare. - Riportare il pallone in posizione orizzontale e rocciarlo delicatamente più volte e controllare le cellule hCMEC/D3 al microscopio prima di restituire il pallone all'incubatrice (37 gradi centigradi, 5% CO2).
- Osservare le cellule al microscopio luminoso invertito ogni volta prima e dopo la manipolazione delle cellule, sia durante i normali lavori di coltura che durante gli esperimenti. Registrare eventuali cambiamenti evidenti nel numero di cellulare o morfologia nel taccuino di laboratorio.
- Rifornire il mezzo di crescita a giorni alterni fino alla confluenza al 100%. Controllare le cellule al microscopio prima di rimuovere il vecchio mezzo di crescita e anche dopo l'aggiunta di un nuovo mezzo di crescita. Estrarre il pallone dall'incubatrice ed esaminare le cellule hCMEC/D3 al microscopio a contrasto di fase per assicurarsi che appaiano sane.
- Passaggio cellulare
- Incubare un nuovo flacone T75 con 8,5 mL di soluzione di collagene per 1 h nell'incubatrice (37 , 5% CO2).
- Rimuovere la soluzione di collagene e lavare delicatamente il pallone con PBS sterilizzato. Aggiungere 10 mL di mezzo hCMEC/D3 preriscaldato al nuovo pallone e posizionare il pallone nell'incubatrice (37 gradi centigradi, 5% CO2).
- Estrarre il pallone dall'incubatrice ed esaminare le cellule hCMEC/D3 al microscopio a contrasto di fase per verificare se le cellule sono confluenti al 100%.
- Rimuovere il mezzo cellahCMEC/D3 dal flacone contenente cellule e lavare le cellule hCMEC/D3 con 10 mL di PBS.
NOTA: FBS aggiunto al mezzo di crescita contiene inibitori proteasi come l'antitrypsin e la 2-macroglobulina. Questi inibiscono il processo di ripsinizzazione. Pertanto, è essenziale lavare le cellule con PBS per rimuovere le tracce di FBS per prevenire l'inibizione del processo di trypsinization. - Aggiungere 1 mL di 0,25% soluzione di prova contenente 0.02% EDTA e provare per 2-5 min nell'incubatrice (37 , 5% CO2) (toccare il flacone delicatamente sui lati per aiutare il distacco).
NOTA: Non lasciare mai le celle su trypsin/EDTA per più di 6 min. - Aggiungere 10 mL di mezzo hCMEC/D3 preriscaldato per arrestare il processo di trippsinizzazione e risospendere la cella hCMEC/D3 pipettando su e giù più volte. Quindi, rimuovere l'intera sospensione cellulare dal flacone in un tubo da 15 mL.
- Trasferire 1 mL della sospensione cellulare dal tubo da 15 mL al nuovo flacone con un mezzo fresco preriscaldato (celle di divisione 1:10) e riportare il nuovo pallone all'incubatrice.
NOTA: Prima di passare al nuovo pallone, eseguire la deformazione cellulare verso l'alto e verso il basso più volte per ridurre al minimo i gradienti di concentrazione cellulare.
2. Placcatura cellulare
- Posizionare gli inserti di coltura tissutale con membrane micropororose (dimensione del poro: 0,4 m, materiale: terephthalato di polietilene (PET)) in una piastra di coltura a 24 pozze.
- Aggiungere 400 l di collagene di tipo I (0,15 mg/mL) in ogni inserto di coltura tissutale e incubare per 1 h nell'incubatrice di CO2 (37 , 5% CO2). Rocciare delicatamente la piastra di 24 pozze per consentire anche la diffusione della soluzione di collagene sulla membrana microporosa negli inserti di coltura dei tessuti.
- Rimuovere la soluzione di collagene e lavare delicatamente la membrana microporosa con 0,4 mL di 1x tamponi PBS.
- Piastra hCMEC/D3 cellule con la densità di 50.000 celle/cm2 negli inserti della cella (15.000 celle in 500 : L di media).
NOTA: Al fine di ridurre al minimo le differenze nel numero di cellule in ogni inserto di coltura del tessuto, la sospensione cellulare è stata risospesa con una pipetta da 10 mL prima di aggiungere celle agli inserti. - Collocare la piastra di 24 pozze con coltura tissutalein un'incubatrice (37 gradi centigradi, 5% di CO 2) per consentire l'attaccamento e la proliferazione delle cellule.
- Incubare la piastra per 7 giorni per consentire alle cellule di raggiungere il 100% di confluenza. Rimuovere il mezzo di crescita ogni due giorni e trasferire 0,5 mL di supporti freschi preriscaldati in inserti di coltura tissutale.
- Ripetere la procedura di placcatura (passaggi 2.2-2.6) su una piastra da 12 pozze, piastra 48-po e 96-bene piastra. Utilizzare la piastra di 12 pozzetti per il gonfiore occidentale per determinare i cambiamenti nell'espressione di .O-1. Utilizzare la piastra di 48 pozzetti per la trasfezione NP del DNA. Utilizzare la piastra di 96 pozzetto per il saggio ATP per determinare la vitalità cellulare nelle cellule trasfette.
3. Cinetica della crescita cellulare.
- Semina le cellule ad una densità di 50.000 cellule/cm2 in un piatto di coltura dei tessuti rivestiti di collagene.
- In ogni giorno dell'esperimento, rimuovere il mezzo di crescita e lavare delicatamente le cellule due volte con 500 -L di 1x PBS. Quindi, aggiungere 30 - L di 0,25% soluzione di prova contenente 0.02% EDTA e lasciare la piastra per circa 2-5 min in un'incubatrice (37 gradi centigradi, 5% CO2).
NOTA: La formazione graduale di un mostrato confluente può influenzare l'estensione del distacco cellulare ed è necessario aumentare i volumi di orpina/EDTA come indicato qui: 30 l per 1-5 giorni dopo la semina, 60 l per 6-7 giorni dopo il seeding e 100 L per 8-10 giorni dopo la semina . - Aggiungete la durata di 470 l, 440 l o 400 l di mezzo di crescita in base al volume della soluzione trypsin/EDTA aggiunta nel passaggio 3.2 per preparare 500 l. di sospensione cellulare in ogni pozzo.
- Sospendere le cellule pipetting su e giù in ogni bene più volte e osservare le cellule al microscopio per assicurarsi che tutte le cellule sono sospese nel mezzo di crescita. Se alcune cellule sono ancora attaccate al fondo della piastra dopo la pipista più volte, raschiare delicatamente le cellule utilizzando un raschietto di cellule di plastica per facilitare il distacco delle cellule.
- Rimuovere 0,1 mL di sospensione cellulare da 500 s sospensione cellulare nel passaggio 3.3 e aggiungere a un tubo da 1,5 mL. Quindi, aggiungere 0,1 mL di 0,4% Soluzione blu Trypan alla sospensione cellulare e mescolare bene.
- Pulire un emacytometro con 70% di alcool isopropile. Aggiungere 20 - L di miscela dal passo 3.5 su ogni lato nella scanalatura a V e individuare i 16 quadrati al microscopio. I 16 quadrati sono considerati come una griglia. Individuare due griglie casuali su ogni lato dell'emacytometro e contare tutte le cellule viventi non blu.
NOTA: celle che sono apparse di colore blu dal conteggio escluso, <1% delle celle colorate di blu in tutti i punti temporali. - Calcolare la densità dellecelle (celle/cm 2) in base alle seguenti formule.
Numero medio di celle/griglia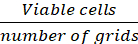 (Eq.1)
(Eq.1)
Fattore di diluizione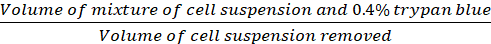 (Eq.2)
(Eq.2)
Densità di cellule (cellule vitali/cm2)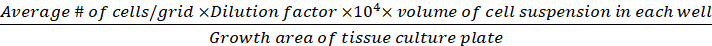
(Eq.3)
Equazioni 1-3. Le cellule vitali sono il numero di cellule contate in ogni griglia, il numero di griglie corrisponde al numero di griglie situate al microscopio, Volume di miscela di sospensione cellulare e 0,4% Trypan blu è il volume preparato al passo 3.5, il volume delle sospensioni cellulari rimosse è il volume rimosso da una sospensione cellulare da 500, nel passaggio 3,5, il volume della sospensione cellulare in ogni pozzo è la sospensione cellulare 500, dal passo 3.3, l'area di crescita della piastra di coltura dei tessuti è l'area di crescita del singolo pozzo in 24 pozze.
4. Lucifero giallo apparente permeabilità (LY P app ) saggio
- Per determinarel'app LY P in ogni giorno successivo al seeding, segui i passaggi a partire dalla 4.3. Per determinarel'app P nelle cellule hCMEC/D3 trasfette, aggiungere 8,3 l_l della formulazione di trasfezione (Figura 1) mescolata con 50 - L di mezzo di crescita completa e incubazione per 4 h. Le formulazioni di trasfezione sono descritte nella sezione 5.
- Dopo la trasfezione di 4 h, lavare delicatamente il lato apicale due volte utilizzando il buffer sterile 1x PBS per rimuovere qualsiasi miscela di trasfezione residua. La variazione dei volumi di tampone PBS lasciato dopo la rimozione può influire sulla concentrazione di LY sul lato apicale. Fare attenzione a garantire che il buffer PBS residuo negli inserti di coltura dei tessuti sia completamente rimosso. Aspirare con attenzione reagenti residui e mezzi per ridurre al minimo il distacco cellulare.
NOTA: questo passaggio viene ignorato quando si misurala permeabilità apparente (P app) di celle hCMEC/D3. - Togliere il mezzo di crescita e aggiungere 1,5 mL di buffer di trasporto preriscaldato (37 mM) (25 mM HEPES, 145 mM NaCl, 3 mM KCl, 1 mM CaCl2, 0,5 mM MgCl2, 1 mM NaH2PO4, 5 mM di glucosio, pH 7,4) al lato baso laterale.
NOTA: il volume del buffer di trasporto in tutti i compartimenti basolaterali deve essere uguale per garantire l'accuratezza del calcolo del coefficiente di permeabilità. - Aggiungete una soluzione di 20 GRADI al lato apicale di ogni inserto transwell. Risparmiare 50 gradi della soluzione LY da 20 m per le misurazioni della fluorescenza. Dopo aver rimosso completamente il buffer PBS residuo dal lato apicale, aggiungere la soluzione LY il più rapidamente possibile per evitare l'essiccazione delle cellule hCMEC/D3. Garantire volumi accurati di soluzione LY sul lato apicale.
NOTA: Per ridurre al minimo il decadimento dell'intensità della fluorescenza LY, l'esposizione alla luce deve essere limitata. Una volta ricostituita la polvere LY, la soluzione deve essere conservata a 4 gradi centigradi, protetta dalla luce. - Incubare in uno shaker rotante (37 o 100 giri/m) per 60 min. Quindi, rimuovere 30 l del campione LY da ogni vano apicale. Trasferire quindi la soluzione LY da 20 M e i campioni lato apicale in tubi pre-etichettati e diluire il campione di 10 volte utilizzando il buffer di trasporto.
NOTA: È necessario diluire la soluzione di 20 -M LY perché l'elevata intensità di fluorescenza di questi campioni può potenzialmente sovraccaricare e danneggiare il rilevatore di fluorescenza del lettore di microplacca a fluorescenza. - Togliere 500 l da ogni scompartimento basolaterale e trasferire il campione su tubi pre-etichettati.
NOTA: i campioni vengono rimossi dal transwell separato nei punti temporali indicati. I punti temporali sono ogni giorno successivo al seeding a partire dal giorno 1 fino al giorno 10. - Preparare una serie di standard LY per la curva standard (39,00 nM, 78,13 nM, 156,25 nM, 312 nM, 625 nM, 1250 nM, 2500 nM).
- Aggiungete 100 l di ogni campione standard (in duplicato), apicale e basolaterale in ogni pozzo in una piastra nera di 96 pozze (Figura1).
NOTA: Le placche nere assorbono la luce e riducono il crossover di sfondo e fluorescenza tra i pozzi. - Utilizzare un lettore di microplassiche a fluorescenza (set point: excitation 428 nm, emissione 536 nm) per misurare l'intensità della fluorescenza LY per calcolarel'appP. Per questo studio viene utilizzato un lettore di lastre di fluorescenza.
- Calcolarel'app P e i valori di recupero %LY come descritto nel testo del manoscritto.
Equazione 4. Formula per il calcolo dei valoridell'app P.
Calcolare il coefficiente di permeabilità apparente (Papp) e il recupero %LY utilizzando le equazioni seguenti. Va notato che i valoridell'app P possono essere calcolati in base alla massa25 o alla concentrazione di LY.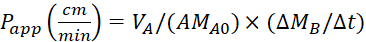 o
o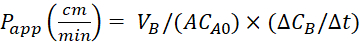
VA - volume nel compartimento apicale
VB - volume nel compartimento basolaterale
A - la superficie della membrana di inserto transwell (0,3 cm2)
MA0 - la massa iniziale nel compartimento apicale
-la variazione di massa nel tempo nel compartimento basolaterale
CA0 - la concentrazione iniziale nel compartimento apicale
-la variazione di concentrazione nel tempo nel compartimento basolaterale.
Equazione 5. Formule per il calcolo del recupero % LY.
Recupero (%)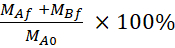
MAf è la massa nel compartimento apicale nel punto finale, MBf è la massa nel compartimento basolaterale nel punto finale, MA0 è la massa iniziale nel compartimento apicale. 26 Nota: La massa iniziale viene calcolata in base al volume della soluzione LY da 20 m nel passaggio 4.5. Questo esperimento è stato sempre fatto utilizzando cellule sotto il passaggio numero 35 ed è stato condotto quattro volte indipendenti.
5. Esaurimento del calcio
- Rimuovere il mezzo di crescita dagli inserti transwell e dalla piastra da 12 pozze e lavare delicatamente il lato apicale e la piastra da 12 pozze utilizzando un mezzo preriscaldato senza calcio (mezzo minimo essenziale medio aquila (S-MEM) per rimuovere gli ioni di calcio dagli inserti transwell e piastra da 12 pozzi.
- Aggiungere 500 l o 2 mL di S-MEM preriscaldato 1xmedium agli inserti o alla piastra 12 e incubare per 24 h nell'incubatrice (37 gradi centigradi, 5% CO2). Dopo l'incubazione, rimuovere il mezzo S-MEM 1x e lavare le cellule una volta utilizzando il buffer 1x PBS preriscaldato.
- Seguire i passaggi 4.4-4.10 descritti nella sezione 4 per il trattamento LY e i passaggi successivi. Seguire i passaggi 8.1.2-8.2.4 descritti nella sezione 8 per il gonfiore occidentale e i passaggi successivi.
6. Trasfezione
- Preparazione di nanoparticelle di DNA (NP DEL DNA).
- Diluire la soluzione di stock del plasmide gWI-Luc in tampone di acetato di sodio da 10 mM (NaAc) (pH 5.0) e consentire alla soluzione di DNA di stare per 10 min a temperatura ambiente (RT).
NOTA: Il DNA NP contenente prevalentemente molecole di DNA singolo può essere preparato a concentrazioni di DNA 20-40 g/mL23. Pertanto, è necessario diluire la soluzione di stock gWI-Luc.-Luc. Lo stock di plasmid gWI-Luc congelati deve essere scongelato completamente sul ghiaccio per ridurre al minimo lo stress da temperatura. Vorticare delicatamente la soluzione dismessa del DNA per 30 s su un vortice standard da banco impostato in posizione manopola 3-5. - Calcolare i rapporti N/P desiderati, utilizzati qui come parametro numerico per riflettere la composizione NP.

Equazione 6. Calcolo del rapporto N/P: il rapporto tra le talpe dei gruppi di ammina dei polimeri cationici e quelle dei gruppi fosfati del DNA. La massa di polimeri cationici significa quantità totale pesata di polimero cationico; (Massa/Carica) I polimeri cationici si riferiscono al peso molecolare del polimero cationico (poli (etilenegliglycol)5k-block-polyaspartamide con 48 catene laterali di etilenetriamina (PEG-DET)) normalizzate al numero di ammine primarie cariche (48) per polimero cationico ( mol/mol), questo valore per il nostro polimero è 306 Da; Massa di DNA significa quantità totale di DNA utilizzato nella formulazione ottenuta moltiplicando il volume e la concentrazione in mg/mL; (Massa/Carica) Il DNA si riferisce al peso molecolare del DNA normalizzato al numero di gruppo di fosfati per DNA a doppio filamento (325 Da per nucleobase).
NOTA: La tabella di preparazione del DNA NP (Tabella 1) contiene la ricetta di formulazione per i diversi campioni testati nei nostri esperimenti. Il DNA NP è stato preparato utilizzando una tecnica di titolazione rapida. La soluzione polimerica PEG-DET è stata aggiunta lungo le pareti del tubo tenendo il tubo in posizione orizzontale. Poi il tubo è stato commutato in posizione verticale, seguita da rapida vortice alla massima velocità per 10s. Il DNA NP è stato permesso di stare per 30 minuti a RT prima dell'uso. La regola del dosso DNA NP è di 0,5 g di DNA per l'area di crescita di ca. 1 cm 2. Quindi, per ogni inserto/bene transwell in una piastra/ogni pozzo di 48 pozze in una piastra di 96 pozze, preparate il DNA NP a N/P 10 contenente 0,157/0,5/0,195 g/g/bene di DNA gWI-Luc. - Per i campioni contenenti la concentrazione indicata di Poloxamer P84 (P84), aggiungere P84 al DNA NP e vortice per 5 s. La concentrazione finale di P84 in ogni campione è dello 0,01% o dello 0,03% wt.
- Diluire la soluzione di stock del plasmide gWI-Luc in tampone di acetato di sodio da 10 mM (NaAc) (pH 5.0) e consentire alla soluzione di DNA di stare per 10 min a temperatura ambiente (RT).
- Trasfezione DNA NP in una configurazione piastra 48 pozzetto
- Semina le cellule con una densità di 50.000 cellule/cm2 in una piastra di 48 pozze e crescono fino alla confluenza nell'incubatrice (37 , 5% di CO2).
- Per ogni gruppo di trattamento, mescolare 25 : L del campione indicato (formulazione della trasfezione) e 150 L di mezzo di crescita completo e 175 - L di questa miscela ad ogni pozzo.
- Osservare le cellule hCMEC/D3 al microscopio per assicurarsi che le cellule appaiano sane e siano confluenti al 100% al momento dell'esperimento.
- Rimuovere il mezzo di crescita dai pozzi e la miscela di trasfezione di 175 gradi in ogni pozzo. Quindi, incubare la piastra per 4 h nell'incubatrice (37 gradi centigradi, 5% CO2).
- Dopo 4 h, rimuovere la miscela di trasfezione e lavare le cellule hCMEC/D3 con buffer sterile 1x PBS preriscaldato.
NOTA: Per ridurre al minimo il distacco accidentale delle cellule hCMEC/D3 dalla superficie della piastra/inserimento durante le fasi di lavaggio, pipette con attenzione abbastanza sterile PBS lungo le pareti dei pozzi e rimuovere eventuali nanoparticelle nel supporto di coltura residua. - Appiattiamo delicatamente la piastra un paio di volte e aspirate con cura e scartate il lavaggio PBS e aggiungete 500 gradi di hCMEC/D3 preriscaldato nel mezzo di coltura.
- Esaminando microscopicamente le cellule e registrate le osservazioni sulla morfologia cellulare e sui possibili effetti della trasparenza.
- Incubare per 24 h nell'incubatore a coltura cellulare 37 c per consentire la produzione di luciferasi. Dopo 24 h, rimuovere completamente il mezzo di crescita e lavare le cellule una volta con 1x PBS preriscaldato.
- Lyse le cellule trasfette aggiungendo 100 l di ghiaccio-freddo luciferasi coltura cellulare lisi 1x reagente per pozzo.
- Per la misurazione del contenuto di proteine luciferate, aggiungere 20 m L di lisate cellulare e 100 L di macchia di luciferasi (20 mM di glicilglycine (pH 8), 1 mM MgCl2, 1,1 mM EDTA, 3,5 mM DTT, 0,5 mM ATP, 0,27 m).
- Leggere la luminescenza del campione descritto nel passaggio 6.2.10 su un Luminometer con un singolo iniettore automatico.
NOTA: La luminescenza deve essere integrata su 10 s prima di leggere. - Misurare la quantità totale di proteine cellulari nel lisato utilizzando un kit di analisi dell'acido bicinchoninico (BCA assay) seguendo il protocollo del produttore.
- Calcolare ed esprimere l'espressione genica della luciferasi come unità di luce relativa (RLU) per ogni proteina cellulare totale.
7. Saggio ATP luminescente
- Semina le cellule con una densità di 50.000 cellule/cm2 in una piastra di 96 pozze e crescono fino alla confluenza nell'incubatrice (37 , 5% di CO2).
- Trasfecare le cellule con 9,7 litri della formulazione della trasfezione (i dettagli di preparazione sono nella sezione 6) e 58,4 l di mezzo di crescita completa per 4 h.
- Rimuovere la miscela di trasfezione e lavare delicatamente le cellule con il tampone PREriscaldato PBS 1x due volte per rimuovere completamente i reagenti di trattamento.
NOTA: volumi diversi di buffer residuo potrebbero diluire il file atP analisi reagenti in misura diversa e possono potenzialmente influenzare i dati. - Mescolare 75 -L di fresco preriscaldato medio e ATP saggio reagente in una diluizione 1:1 utilizzando una pipetta multicanale. Assicurarsi che il livello del liquido in tutte le punte pipette multicanale sia lo stesso.
- Posizionare la piastra su uno shaker nutating per 15 min a temperatura ambiente. Dopo 15 min di aggiunta dei reagenti, trasferire 60 l di ogni campione in una piastra bianca di 96 pozze.
NOTA: Le piastre bianche sono meglio riflettere la luce di uscita rispetto alle piastre chiare o nere. - Pop eventuali bolle d'aria utilizzando un ago prima di leggere la piastra. Leggere la piastra su un luminometro con un tempo di integrazione 1 s. Leggere la piastra entro 20 min dopo l'aggiunta di REagenti di test ATP. La tempistica è fondamentale per il confronto tra diverse piastre, perché il segnale di luminescenza è transitorio con un tasso di decadimento veloce.
- Calcolare la percentuale (%) viabilità cellulare con questa formula: (luminescenza delle cellule trafette / luminescenza del controllo, cellule non trattate) x 100.
8. Gonfiore occidentale per la misurazione della proteina giunzione stretta-
-
La lisi cellulare e l'estrazione di proteine
NOTA: Tutti i passaggi per l'estrazione di proteine dalle cellule devono essere effettuati a 2-8 gradi centigradi.- Semina le cellule ad una densità di 50.000 cellule/cm2 in un piatto di coltura dei tessuti a 12 pozze di collagene.
- Il giorno 3, giorno 5, giorno 7, giorno 10 post-semina e il giorno 7 (cellule pre-incubate con mezzo libero di calcio), rimuovere il mezzo di crescita e lavare delicatamente le cellule due volte con 2 mL di 1x PBS ghiacciato. Quindi, aggiungere 300 ll di ghiaccio freddo 1x getto di lisi RIPA contenente 3 aprotinina di g/mL in ogni pozzo.
NOTA: Il composto deve essere appena fatto e tenuto sul ghiaccio. L'aprotinina è usata per inibire le proteasi presenti nei lisati dal degradare la proteina di interesse. - Dopo due cicli di congelamento-scongelamento (-80 gradi centigradi), raschiare le cellule utilizzando un raschietto a cellule di plastica fredda. Raccogliere i lismi cellulari in tubi di microfuge. Quindi, centrifugare i tubi a 200 x g per 30 min a 4 gradi centigradi.
- Raccogliere il supernatante in tubi puliti e metterli sul ghiaccio. Misurare la quantità totale di proteine cellulari nei lisati utilizzando il kit di analisi BCA seguendo il protocollo del produttore.
-
Gonfiore occidentale per rilevare la proteina giunzione stretta
- Denature di ottenni totali contenenti 40 g di proteine totali con 1x laemmli tamponamento a 95 , 5 min e soggetto a elettroforesi in un 6-7,5% di gel di poliacrita di solfato di solfato di solfato di sodio (SDS-PAGE) (90 V, 10 min attraverso l'impilamento del gel, 120 V risoluzione dei gel).
NOTA: Quando si caricano i campioni o gli standard, ricordarsi di caricare lentamente e con attenzione in ogni corsia, facendo attenzione a non rompere il pozzo nel processo. - Trasferire le proteine separate su una membrana di nitrocellulosa con tampone di trasferimento pH 8,5 che contiene 192 mM Di cicina, 25 mM Tris Base, 10% metanolo e 0,1% SDS (75 V, 110 min a temperatura ambiente).
NOTA: Non toccare la membrana. Utilizzare il 70% di pinze in plastica lavate con isopropanolo per maneggiare la membrana. Al fine di trasferire con successo la proteina o-1 (MW 200 kDa) alla membrana, il pH dovrebbe essere di circa 8,3-8,5. Se il buffer di trasferimento è più acido di quello, il trasferimento non sarebbe accaduto. Se le bande della scala sono visibili ancora sul gel, sarebbe utile aumentare il tempo di trasferimento e la concentrazione di SDS. - Dopo aver lavato la membrana utilizzando la salina Tamponata Tris contenente lo 0,1%Tween 20 (T-TBS), utilizzare la soluzione di blocco (1:1 LiCOR-Odyssey Block: 1x Tris buffered salina) per bloccare le membrane per 60 min.
- Tagliare con cura la membrana in due strisce. Incubare con due anticorpi primari (anticorpi monoclonali zo-1, diluizione, 1: 900, e dehydrogenasi di gliceraldeide 3-fosfato (GAPDH), diluizione, 1: 10.000) durante la notte a 4 gradi centigradi. Quindi, incubare le membrane di z-1 e GAPDH con i gFoci antito-tono antiinchiave (diluizione, 1:50.000). Dopo aver lavato le membrane utilizzando T-TBS, immagini le membrane nel canale 700 su un imager a 16 bit.
- Denature di ottenni totali contenenti 40 g di proteine totali con 1x laemmli tamponamento a 95 , 5 min e soggetto a elettroforesi in un 6-7,5% di gel di poliacrita di solfato di solfato di solfato di sodio (SDS-PAGE) (90 V, 10 min attraverso l'impilamento del gel, 120 V risoluzione dei gel).
Risultati
In primo luogo, abbiamo determinato l'effetto del tempo di coltura sulla permeabilità LY per determinare l'apparente cinetica della formazione di TJ. I valori medidell'app LY P dal giorno 1 al seeding di 10 post sono illustrati nella Figura 2a. Il giorno 1,l'app P media era 4,25 x 10-4 cm/min e leggermente sceso a 3,32 x 10-4 cm/min il giorno 2. Il valore mediodell'app P è leggermente aumentato a 3,93 x...
Discussione
Un ruolo chiave della BBB è quello di impedire lo scambio di ioni non essenziali e sostanze tossiche tra la circolazione sistemica e il cervello per mantenere l'emostasi del microambiente neurale. Una delle caratteristiche del BBB è la capacità delle cellule endoteliane capillari di formare giunzioni strette (TJ) che sigillano efficacemente il percorso paracellulare di trasporto. Abbiamo dimostrato un saggiodell'app LY P come metodo quantitativo per determinare l'apparente cinetica della formazione della ba...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori sono grati per il sostegno finanziario dal 2017 New Investigator Award dall'American Association of Pharmacy, un hunkele Dreaded Disease award dalla Duquesne University e i fondi di start-up School of Pharmacy per il laboratorio Manickam. Ringraziamo il laboratorio Leak (Duquesne University) per l'assistenza occidentale del blotting e per aver permesso l'uso del loro imager A 16 bit Odyssey. Vorremmo anche includere una nota speciale di apprezzamento per Kandarp Dave (laboratorio Manickam) per l'aiuto con il gonfiore occidentale.
Materiali
| Name | Company | Catalog Number | Comments |
| hCMEC/D3 cell line | Cedarlane Laboratories | 102114.3C-P25 | human cerebral microvascular endothelial cell line |
| gWizLuc | Aldevron | 5000-5001 | Plasmid DNA encoding luciferase gene |
| lucifer yellow CH dilithium salt | Invitrogen | 155267 | |
| Transwell inserts with polyethylene terephthalate (PET) track-etched membranes | Falcon | 353095 | |
| Tissue culture flask | Olympus Plastics | 25-207 | |
| 24-well Flat Bottom | Olympus Plastics | 25-107 | |
| Black 96-Well Immuno Plates | Thermo Scientific | 437111 | |
| S-MEM 1X | Gibco | 1951695 | Spinner-minimum essential medium (S-MEM) |
| EBM-2 | Clonetics | CC-3156 | Endothelial cell basal medium-2(EBM-2) |
| phosphate-buffered saline 1X | HyClone | SH3025601 | |
| Collagen Type I | Discovery Labware, Inc. | 354236 | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23227 | |
| Cell Culture Lysis 5X Reagent | Promega | E1531 | |
| Beetle Luciferin, Potassium Salt | Promega | E1601 | |
| SpectraMax i3 | Molecular Devices | Fluorescence Plate Reader | |
| Trypan Blue Solution, 0.4% | Gibco | 15250061 | |
| ZO-1 Polyclonal Antibody | ThermoFisher | 61-7300 | |
| anti-GAPDH antibody | abcam | ab8245 | |
| Alexa Fluor680-conjugated AffiniPure Donkey Anti-Mouse LgG(H+L) | Jackson ImmunoResearch Inc | 128817 | |
| 12-well, Flat Bottom | Olympus Plastics | 25-106 | |
| RIPA buffer (5X) | Alfa Aesar | J62524 | |
| Aprotinin | Fisher BioReagents | BP2503-10 | |
| Odyssey CLx imager | LI-COR Biosciences | for scanning western blot membranes |
Riferimenti
- Abbott, N. J., Patabendige, A. A., Dolman, D. E., Yusof, S. R., Begley, D. J. Structure and function of the blood-brain barrier. Neurobiology Of Disease. 37 (1), 13-25 (2010).
- Griep, L. M., et al. BBB on chip: microfluidic platform to mechanically and biochemically modulate blood-brain barrier function. Biomedical Microdevices. 15 (1), 145-150 (2013).
- Camos, S., Mallolas, J. Experimental models for assaying microvascular endothelial cell pathophysiology in stroke. Molecules. 15 (12), 9104-9134 (2010).
- Cucullo, L., et al. Immortalized human brain endothelial cells and flow-based vascular modeling: a marriage of convenience for rational neurovascular studies. Journal of Cerebral Blood Flow & Metabolism. 28 (2), 312-328 (2008).
- Wolff, A., Antfolk, M., Brodin, B., Tenje, M. In vitro Blood-Brain Barrier Models-An Overview of Established Models and New Microfluidic Approaches. Journal of Pharmaceutical Sciences. 104 (9), 2727-2746 (2015).
- Weksler, B., Romero, I. A., Couraud, P. O. The hCMEC/D3 cell line as a model of the human blood brain barrier. Fluids and Barriers of the CNS. 10 (1), 16 (2013).
- Ohtsuki, S., et al. Quantitative targeted absolute proteomic analysis of transporters, receptors and junction proteins for validation of human cerebral microvascular endothelial cell line hCMEC/D3 as a human blood-brain barrier model. Molecular Pharmaceutics. 10 (1), 289-296 (2013).
- Tornabene, E., Brodin, B. Stroke and Drug Delivery--In vitro Models of the Ischemic Blood-Brain Barrier. Journal of Pharmaceutical Sciences. 105 (2), 398-405 (2016).
- Weksler, B., Romero, I. A., Couraud, P. O. The hCMEC/D3 cell line as a model of the human blood brain barrier. Fluids and Barriers of the CNS. 10 (1), 16 (2013).
- Llombart, V., et al. Characterization of secretomes from a human blood brain barrier endothelial cells in-vitro model after ischemia by stable isotope labeling with aminoacids in cell culture (SILAC). Journal of Proteomics. 133, 100-112 (2016).
- Weksler, B. B., et al. Blood-brain barrier-specific properties of a human adult brain endothelial cell line. The FASEB Journal. 19 (13), 1872-1874 (2005).
- Avdeef, A. How well can in vitro brain microcapillary endothelial cell models predict rodent in vivo blood-brain barrier permeability?. European Journal of Pharmaceutical Sciences. 43 (3), 109-124 (2011).
- Rahman, N. A., et al. Immortalized endothelial cell lines for in vitro blood-brain barrier models: A systematic review. Brain Research. 1642, 532-545 (2016).
- Cecchelli, R., et al. A stable and reproducible human blood-brain barrier model derived from hematopoietic stem cells. PLoS One. 9 (6), e99733 (2014).
- Shao, X., et al. Development of a blood-brain barrier model in a membrane-based microchip for characterization of drug permeability and cytotoxicity for drug screening. Analytica Chimica Acta. 934, 186-193 (2016).
- Walter, F. R., et al. A versatile lab-on-a-chip tool for modeling biological barriers. Sensors and Actuators B: Chemical. 222, 1209-1219 (2016).
- Cecchelli, R., et al. In vitro model for evaluating drug transport across the blood–brain barrier. Advanced Drug Delivery Reviews. 36, (1999).
- Cecchelli, R., et al. Modelling of the blood–brain barrier in drug discovery and development. Nature reviews Drug discovery. 6 (8), 650 (2007).
- Reichel, A., Begley, D. J., Abbott, N. J. . The Blood-Brain Barrier. , 307-324 (2003).
- Deli, M. A., Ábrahám, C. S., Kataoka, Y., Niwa, M. Permeability Studies on In vitro Blood–Brain Barrier Models: Physiology, Pathology, and Pharmacology. Cellular and Molecular Neurobiology. 25 (1), 59-127 (2005).
- Ren, T. B., et al. A General Method To Increase Stokes Shift by Introducing Alternating Vibronic Structures. Journal of the American Chemical Society. 140 (24), 7716-7722 (2018).
- Pack, D. W., Hoffman, A. S., Pun, S., Stayton, P. S. Design and development of polymers for gene delivery. Nature Reviews Drug Discovery. 4 (7), 581-593 (2005).
- Oupický, D., Konák, C., Ulbrich, K., Wolfert, M. A., Seymour, L. W. DNA delivery systems based on complexes of DNA with synthetic polycations and their copolymers. Journal of Controlled Release. 65, (2000).
- Couraud, P. O. . The hCMEC/D3 CELL LINE: IMMORTALIZED HUMAN CEREBRAL MICROVASCULAR ENDOTHELIAL CELLS As a model of human Blood-Brain Barrier. , (2012).
- Youdim, K. u. r. e. s. h. A., A, A. A. a. N. J. In vitro trans-monolayer permeability calculations: often forgotten assumptions. research focus reviews. 8, (2003).
- Eigenmann, D. E., Xue, G., Kim, K. S., Moses, A. V., Hamburger, M., Oufir, M. Comparative study of four immortalized human brain capillary endothelial cell lines, hCMEC/D3, hBMEC, TY10, and BB19, and optimization of culture conditions, for an in vitro blood-brain barrier model for drug permeability studies. Fluid and Barriers of the CNS. 10 (33), (2013).
- Hubatsch, I., Ragnarsson, E. G. E., Artursson, P. Determination of drug permeability and prediction of drug absorption in Caco-2 monolayers. Nature Protocols. 2 (9), 2111-2119 (2007).
- Balda, M. S., Anderson, J. M. Two classes of tight junctions are revealed by ZO-1 isoforms. The American Physiological Society. , (1992).
- Brown, R. C., Davis, T. P. Calcium Modulation of Adherens and Tight Junction Function: A Potential Mechanism for Blood-Brain Barrier Disruption After Stroke. Stroke. 33 (6), 1706-1711 (2002).
- Gorodeski, G., Jin, W., Hopfer, U. Extracellular Ca2+ directly regulates tight junctional permeability in the human cervical cell line CaSki. American Journal of Physiology-Cell Physiology. 272 (2), C511-C524 (1997).
- Stuart, R. O., Sun, A., Panichas, M., Hebert, S. C., Brenner, B. M., Nigam, S. K. Critical Role for lntracellular Calcium in Tight Junction Biogenesis. Journal of Cellular Physiology. 159, (1994).
- Tobey, N. A. Calcium-switch technique and junctional permeability in native rabbit esophageal epithelium. American Journal of Physiology-Gastrointestinal and Liver Physiology. , (2004).
- Tobey, N. A., Argote, C. M., Hosseini, S. S., Orlando, R. C. Calcium-switch technique and junctional permeability in native rabbit esophageal epithelium. American Journal of Physiology-Gastrointestinal and Liver Physiology. 286, (2004).
- Posimo, J. M., et al. Viability assays for cells in culture. Journal of visualized experiments : JoVE. (83), e50645 (2014).
- Cipolla, M. J., Crete, R., Vitullo, L., Rix, R. D. Transcellular transport as a mechanism of blood-brain barrier disruption during stroke. Frontiers in Bioscience. 9 (3), 777-785 (2004).
- Kreuter, J. Influence of the surface properties on nanoparticle-mediated transport of drugs to the brain. Journal of nanoscience and nanotechnology. 4 (5), 484-488 (2004).
- Markoutsa, E., et al. Uptake and permeability studies of BBB-targeting immunoliposomes using the hCMEC/D3 cell line. European Journal of Pharmaceutics and Biopharmaceutics. 77 (2), 265-274 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon