Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Synthèse et caractérisation de nanoparticules poly(bêta-aminoesters) chargées d’ARNm à des fins de vaccination
Dans cet article
Résumé
Ici, un protocole simple est présenté pour produire des nanoparticules d’ARNm à base de polymères poly(bêta aminoesster), faciles à adapter en changeant l’ARNm encapsulé. Le flux de travail de synthèse des polymères, des nanoparticules et de leur caractérisation essentielle in vitro est également décrit. Une preuve de concept concernant la vaccination est également ajoutée.
Résumé
La vaccination a été l’un des succès majeurs de la société moderne et est indispensable pour contrôler et prévenir les maladies. Les vaccins traditionnels étaient composés de tout ou de fractions de l’agent infectieux. Cependant, des défis demeurent et les nouvelles technologies vaccinales sont obligatoires. Dans ce contexte, l’utilisation de l’ARNm à des fins d’immunisation a montré une performance accrue, comme en témoigne l’approbation rapide de deux vaccins à ARNm prévenant l’infection par le SRAS-CoV-2. Au-delà du succès dans la prévention des infections virales, les vaccins à ARNm peuvent également être utilisés pour des applications thérapeutiques contre le cancer.
Néanmoins, l’instabilité de l’ARNm et sa clairance rapide du corps en raison de la présence de nucléases rendent son administration nue impossible. Dans ce contexte, les nanomédicaments, et en particulier les nanoparticules polymères, sont des systèmes critiques d’administration d’ARNm. Ainsi, le but de cet article est de décrire le protocole de formulation et de test d’un candidat vaccin à ARNm basé sur les nanoparticules polymères propriétaires. La synthèse et la caractérisation chimique des polymères poly(bêta-aminoesters) utilisés, leur complexation avec l’ARNm pour former des nanoparticules et leur méthodologie de lyophilisation seront discutés ici. Il s’agit d’une étape cruciale pour réduire les coûts de stockage et de distribution. Enfin, les tests requis pour démontrer leur capacité à transfecter in vitro et à modéliser des cellules dendritiques matures seront indiqués. Ce protocole profitera à la communauté scientifique travaillant sur la vaccination en raison de sa grande polyvalence qui permet à ces vaccins de prévenir ou de guérir une grande variété de maladies.
Introduction
Les maladies infectieuses ont représenté une grave menace pour des millions d’êtres humains dans le monde et restent l’une des principales causes de décès dans certains pays en développement. La vaccination prophylactique a été l’une des interventions les plus efficaces de la société moderne pour prévenir et contrôler les maladies infectieuses1,2. Ces jalons critiques de la science dans la pertinencedu 20esiècle ont été remarqués par la récente pandémie mondiale de Covid-19 causée par le virus SARS-CoV-23. Reconnaissant l’importance de disposer de vaccins efficaces pour réduire la dissémination de la maladie, les efforts de coopération de toutes les communautés biomédicales ont abouti à la mise sur le marché de nombreux vaccins prophylactiques en moins d’un an4.
Traditionnellement, les vaccins étaient composés de virus atténués (vivants, à virulence réduite) ou inactivés (particules de mort). Cependant, pour certaines maladies sans marge d’erreurs de sécurité, les particules virales ne sont pas possibles et des sous-unités protéiques sont utilisées à la place. Néanmoins, les sous-unités ne permettent généralement pas la combinaison de plus d’un épitope / antigène, et des adjuvants sont nécessaires pour améliorer la puissance vaccinale5,6. Par conséquent, le besoin de nouveaux types de vaccins est évident.
Comme démontré pendant la pandémie actuelle, de nouveaux candidats vaccins à base d’acides nucléiques peuvent être avantageux pour éviter de longs processus de développement et offrir une grande polyvalence tout en produisant, en même temps, une immunisation vitale pour les patients. C’est le cas des vaccins à ARNm, qui ont été initialement conçus comme des vaccins expérimentaux contre le cancer. Grâce à leur capacité naturelle à produire des réponses de lymphocytes T spécifiques à l’antigène3,5,6,7. Étant l’ARNm la molécule qui code pour la protéine antigénique, ne changeant que la même chose, le vaccin peut être rapidement adapté pour immuniser d’autres variantes du même micro-organisme, différentes souches, d’autres micro-organismes infectieux, ou même devenir un traitement immunothérapeutique du cancer. De plus, ils sont avantageux en termes de coûts de production à grande échelle. Cependant, l’ARNm a un obstacle important qui entrave leur administration nue: sa stabilité et son intégrité sont compromises dans des milieux physiologiques, pleins de nucléases. Pour cette raison, l’utilisation d’un support nanométrique qui le protège et vectorise l’ARNm vers les cellules présentatrices d’antigène est nécessaire2,8.
Dans ce contexte, les poly(bêta-aminoesters) (pBAE) sont une classe de polymères biocompatibles et biodégradables qui ont démontré une capacité remarquable à complexer l’ARNm dans des particules nanométriques, grâce à leurs charges cationiques 9,10,11. Ces polymères sont composés de liaisons esters, ce qui facilite leur dégradation par les estérases dans des conditions physiologiques. Parmi les candidats de la bibliothèque pBAE, ceux fonctionnalisés avec des oligopeptides cationiques finaux ont montré une plus grande capacité à former de petites nanoparticules pour pénétrer efficacement dans les cellules par endocytose et transfecter le matériel génétique encapsulé. De plus, grâce à leur capacité tampon, l’acidification du compartiment endosomique permet une fuite endosomale12,13. À savoir, un type spécifique de pBAE, y compris des fractions hydrophobes sur leur épine dorsale (le soi-disant C6 pBAE) pour améliorer leur stabilité et la combinaison d’oligopeptides finaux (60% de polymère modifié avec une tri-lysine et 40% du polymère avec une tri-histidine) qui transfecte sélectivement les cellules présentatrices d’antigènes après administration parentérale et produit la présentation de l’antigène codé par ARNm suivie de l’immunisation des souris a été récemment publié14 . En outre, il a également été démontré que ces formulations pouvaient contourner l’une des principales étapes goulots d’étranglement des formulations de nanomédecine: la possibilité de les lyophiliser sans perdre leur fonctionnalité, ce qui permet une stabilité à long terme dans des environnements mous et secs15.
Dans ce contexte, l’objectif du protocole actuel est de mettre la procédure de formation des nanoparticules d’ARNm à la disposition de la communauté scientifique en donnant une description des étapes critiques du protocole et en permettant la production de vaccins efficaces pour la prévention des maladies infectieuses et les applications de traitement des tumeurs.
Le protocole suivant décrit l’entraînement complet pour synthétiser des polymères poly(aminoesters bêta) modifiés par l’extrémité d’oligopeptides - OM-pBAE qui seront ensuite utilisés pour la synthèse de nanoparticules. Dans le protocole, la formulation de nanoparticules est également incluse. En outre, des étapes critiques pour le succès de la procédure et des résultats représentatifs sont également fournis pour s’assurer que les formulations résultantes remplissent les caractéristiques de caractérisation de contrôle de la qualité requises pour définir un résultat positif ou négatif. Ce protocole est résumé à la figure 1.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Synthèse du polymère pBAE avec des oligopeptides finaux (OM-pBAE)
- Polymérisation du C6-pBAE
- Ajouter le 5-amino-1-pentanol (38 mmol; MW = 103,16 Da) 1-hexylamine (38 mmol; MW = 101,19 Da) dans une fiole en verre à fond rond (100 mL). Ensuite, ajouter le diacrylate de 1,4-butanediol (82 mmol; MW = 198,22 Da).
- Préchauffez le bain d’huile de silicone à 90 °C, placez la fiole à fond rond dans le bain d’huile et remuez le mélange à l’aide d’une barre d’agitation magnétique pendant la nuit (~18 h). Ensuite, retirez le produit de la fiole à fond rond et placez-le au congélateur à -20 °C.
REMARQUE: Le produit se présente sous la forme d’une poudre collante et est sorti de la fiole à l’aide d’une spatule. Il est essentiel de valider la structure du polymère obtenu par 1H-RMN. Les spectres RMN ont été enregistrés dans un instrument de 400 MHz (voir tableau des matériaux)en utilisant le chloroforme-d et D2O comme solvants. Environ 10 mg de chaque ester poly(β-amino) ont été prélevés et dissous dans 1 mL de solvant deutéré.
- Réaction avec des peptides pour obtenir OM-pBAE
- Ajouter 25 mL de 0,1 M HCl à des peptides sélectionnés, par exemple le peptide Cys-His-His-His avec de l’acide trifluoroacétique (TFA, 200 mg), dans un tube centrifuge de 50 mL précédemment pesé qui peut supporter la lyophilisation et remuer manuellement pendant la nuit pour obtenir une solution claire.
- Congeler la solution à -80 °C pendant 1 h et lyophiliser le chlorhydrate peptidique obtenu.
REMARQUE: Vérifiez si le poids final correspond à la valeur théorique. - Faire une solution de C6-pBAE (0,031 mmol) dans du sulfoxyde de diméthyle. Faites également une solution du chlorhydrate peptidique (0,078 mmol) dans le sulfoxyde de diméthyle.
- Mélangez les deux solutions dans un tube à bouchon à vis et vissez le bouchon. Remuer la solution du mélange au bain-marie à température contrôlée de 25 °C pendant 20 h à l’aide d’une barre d’agitation magnétique.
- Ajouter le mélange à 7:3 (v/v) éther diéthylique/acétone. Centrifuger la suspension résultante à 25 000 x g à 4 °C pour éliminer le solvant. Ensuite, lavez le solide avec 7:3 (v / v) éther diéthylique / acétone deux fois. Ensuite, séchez le produit sous vide (<0,2 atm).
- Faire une solution de 100 mg/mL du produit dans du sulfoxyde de diméthyle. Le produit résultant est nommé C6-peptide-pBAE. Il est essentiel de valider la structure du polymère obtenu par 1H-RMN pour confirmer la disparition des signaux d’oléfines associés aux acrylates terminaux. S’il n’est pas utilisé, le polymère peut être congelé à -20 °C.
2. Formation de polyplexes
REMARQUE: Toutes les procédures doivent être effectuées à l’intérieur d’une pièce conditionnée pour maintenir une température constante.
- Décongeler les polymères C6-peptide-pBAE et vortex la solution.
- Pipeter le mélange de polymère de haut en bas et préparer une solution de 12,5 mM(V1)dans de l’acétate de sodium (NaAc). Ensuite, vortex le mélange et attendez 10 min.
- Préparer l’ARNm à 0,5 mg/mL et mélanger par pipetage (V2).
REMARQUE: Il est crucial d’éviter de vortexer l’ARNm. - Vortex le mélange de polymères à la concentration finale pour obtenir une solution homogène entre la souche de polymère dans le DMSO et le tampon d’acétate.
REMARQUE: La concentration finale de polymère dépend du rapport N/P (groupes azote/phosphate) sélectionné. Le rapport N/P dépend de chaque ARNm spécifique à utiliser. Pour l’encapsulation eGFP, par exemple, un rapport de 25:1 a été utilisé, comme indiqué précédemment14. - Mélanger la solution de matériel génétique et la solution de C6-peptidepBAE (25x de la concentration d’ARNm) dans un rapport de 1:1 (Vi = V1 + V2).
REMARQUE: C6-peptide-pBAE est chargé dans un tube de microcentrifugation où l’ARNm est ajouté par pipetage de haut en bas pour le mélange. Une fois préparés, les concentrations de polyplex, d’acide nucléique et de C6-peptide-pBAE sont à moitié diluées. - Incuber à 25 °C pendant 30 min dans un thermobloc. Précipiter avec de l’eau sans RNase 1:2 en ajoutant l’échantillon à un tube de microcentrifugation préchargé avec de l’eau.
- Inclure les excipients. Ajouter le même volume que le mélange d’ARNm et de pBAE (Vi)dans HEPES 20 mM et le saccharose 4% en pipetant de haut en bas. À ce stade, l’échantillon a été dilué 3x.
3. Lyophilisation des polyplexes
- Congeler instantanément au congélateur à -80 °C de la solution polyplex précédente pendant 1 h.
- Effectuer le séchage primaire en suivant les étapes suivantes: (1) 1 h à -60 °C et 0,001 hPa; (2) 1 h à -40 °C et 0,0001 hPa; 3° 4 h à -20 °C et 0,0001 hPa; (4) 12 h à 5 °C et 0,0001 hPa.
- Conserver à -20 °C immédiatement pour éviter la réhydratation jusqu’à l’utilisation.
4. Remise en suspension Polyplex
NOTE: Ce protocole décrit le processus utilisé pour reconstruire les nanoparticules de C6-peptide-pBAE lyophilisées pour leur utilisation ultérieure, que ce soit pour la caractérisation, l’analyse in vitro ou in vivo.
- Prenez les nanoparticules lyophilisées à partir de -20 °C juste au moment de l’utilisation et ajoutez rapidement la quantité correspondante d’eau dépyrogénée (DEPC) pour redisperser le solide afin d’atteindre la concentration souhaitée.
NOTE: Le volume sera le même que le volume initial des nanoparticules si la même concentration est envisagée, mais il sera indiqué pour chaque expérience. - Pipette doucement jusqu’à remise en suspension totale, en accompagnant le liquide avec la pipette.
REMARQUE: Assurez-vous qu’aucun matériau ne reste sur la paroi du flacon. - Une fois dissous, pipettez vigoureusement de haut en bas, en évitant les bulles.
REMARQUE: L’échantillon doit avoir un aspect transparent à translucide, ce qui est plus évident à des concentrations plus élevées - Conserver les échantillons sur de la glace ou à 4 °C pendant une période maximale de 24 h une fois reconstitués. Évitez de geler.
5. Caractérisation polyplex
- Diffusion dynamique de la lumière
REMARQUE: Le diamètre hydrodynamique (nm), l’indice de polydispersité (PDI) et la charge de surface des nanoparticules ont été mesurés à 25 ° C, longueur d’onde laser de 633 nm et détecteur de signal à 173 °, à l’aide d’un analyseur de potentiel Zeta (voir tableau des matériaux).- Diamètre hydrodynamique (nm)
- Préparer soigneusement les nanoparticules mélangeant l’ARNm et le pBAE en pipetant le matériel génétique à la fraction polymère jusqu’à une concentration finale d’ARNm de 0,25 mg/mL comme décrit précédemment ci-dessus (étape 2).
- Pré-rincer une microcuvette avec un milieu de dilution filtré pour la nettoyer complètement des impuretés. Ensuite, remplissez la cuvette avec au moins 50 μL de la solution d’échantillon et recouvrez-la. Ensuite, introduisez l’échantillon à l’intérieur de l’instrument DLS, assurez-vous que la cellule est correctement insérée.
REMARQUE: Le milieu de dilution est filtré à l’aide d’un filtre à seringue de 0,22 μm. - Ouvrez le fichier SOP (Standard Operation Procedure) créé et introduisez le nom d’exemplesouhaité. Sélectionnez Mesure de taille.
REMARQUE : Tous les paramètres permettant de créer une POS pour les mesures de taille sont fournis dans le tableau 1. - Exécutez la mesure de la taille des particules à l’aide de DLS en cliquant sur Lecture.
REMARQUE: Après le triple bip sonore, l’analyse est terminée. - Sélectionnez les résultats correspondant à l’échantillon sur la feuille de mesure pour obtenir la taille moyenne des particules, le PDI moyen, l’écart-type et les graphiques. Une fois cela fait, retirez la cellule de l’équipement DLS.
- Retirez l’échantillon, conservez-le pour l’analyse du potentiel zêta, puis nettoyez et rincez la cellule avec de l’eau désionisée. Enfin, séchez la cuvette nettoyée sous un flux de gaz d’air comprimé.
- Charge de surface (mV)
- Préparer soigneusement les nanoparticules mélangeant l’ARNm et le pBAE en pipetant le matériel génétique à la fraction polymère jusqu’à une concentration finale d’ARNm de 0,25 mg/mL comme décrit précédemment ci-dessus (étape 2, formation de Polyplex). Ensuite, congelez et séchez la solution.
REMARQUE: Dans ce cas, pour la mesure de la charge de surface, la lyophilisation de l’échantillon est fortement recommandée avant d’effectuer les mesures visant à redisperger l’échantillon dans le tampon approprié, en simulant les conditions corporelles de pH et de concentration d’électrolytes. - Remettre en suspension l’échantillon lyophilisé à l’eau. Diluer l’échantillon 1/10 dans de l’eau (concentration finale 0,025 mg/mL).
- Pré-rincer une cellule capillaire pliée jetable (cuvette zeta potential) avec un milieu de dilution filtré pour la nettoyer complètement des impuretés. Ensuite, remplissez la cuvette avec des nanoparticules diluées, à l’aide d’une seringue de 1 mL, et boucher les deux côtés des charges.
REMARQUE: La dispersion de nanoparticules doit remplir le volume disponible dans la cuvette (~ 1 mL), en accordant une attention particulière à la formation de la bulle, ce qui pourrait perturber les mesures. - Introduisez l’exemple dans DLS ; assurez-vous que la cellule est correctement insérée.
- Ouvrez le fichier SOP créé pour l’analyse du potentiel zêta et introduisez le nom d’échantillonsouhaité. Sélectionnez Mesure du potentiel zêta.
REMARQUE: Les paramètres permettant de créer une POS pour les mesures du potentiel zêta sont donnés dans le tableau 2. - Exécutez la mesure du potentiel zêta à l’aide de DLS en cliquant sur Play.
REMARQUE: Après le triple bip sonore, l’analyse est terminée. - Sélectionnez les résultats correspondant à l’échantillon sur la feuille de mesure pour obtenir le potentiel zêta moyen, l’écart type et les graphiques. Une fois cela fait, retirez la cellule de l’équipement DLS.
- Retirez l’échantillon et conservez-le si nécessaire. Ensuite, nettoyez et rincez la cellule de la cuvette avec de l’eau désionisée, suivie d’éthanol et d’eau à nouveau. Enfin, séchez la cuvette nettoyée sous un flux de gaz d’air comprimé.
REMARQUE: Pour analyser les données, utilisez le logiciel recommandé.
- Préparer soigneusement les nanoparticules mélangeant l’ARNm et le pBAE en pipetant le matériel génétique à la fraction polymère jusqu’à une concentration finale d’ARNm de 0,25 mg/mL comme décrit précédemment ci-dessus (étape 2, formation de Polyplex). Ensuite, congelez et séchez la solution.
- Diamètre hydrodynamique (nm)
- Analyse de suivi des nanoparticules (NTA)
REMARQUE: Le diamètre hydrodynamique (nm) et la concentration de nanoparticules ont été mesurés à 25 ° C, longueur d’onde laser de 488 nm à l’aide d’un analyseur de suivi des nanoparticules (voir Tableau des matériaux).- NTA et préparation des échantillons
- Allumez l’ordinateur et l’équipement. Tout d’abord, vérifiez si tous les composants sont correctement branchés. Ensuite, ouvrez le logiciel recommandé. Le logiciel vérifiera si tous les accessoires sont correctement connectés.
- Connectez la plaque supérieure, ici, la cellule du joint torique, au module laser.
REMARQUE : Ne pas dévisser cette pièce. - Chargez d’abord la chambre avec de l’eau tampon et/ou désionisée avec une seringue de 1 mL. Répétez la procédure au moins deux fois. Évitez d’introduire des bulles dans la chambre.
- Préparer l’échantillon en diluant 1/1000 dans de l’eau désionisée à partir de la concentration utilisée dans la mesure de la taille du DLS. Préparez au moins 1 mL et chargez-le dans une seringue de 1 mL, en évitant d’introduire des bulles.
- Chargez les échantillons dans la chambre à l’aide d’une seringue. Ensuite, branchez le module laser dans la grande chambre NTA.
REMARQUE: Grande chambre désigne l’espace où la chambre est placée pour la mesure.
- Optimisation de l’image
- Tout d’abord, dans le matériel, vérifiez si la caméra et le laser approprié sont sélectionnés.
REMARQUE: Ici, il n’y a qu’une seule caméra et le Blue Laser 488 nm. - Commencez avec le niveau de la caméra à 0 et appuyez sur Démarrer la caméra dans la fenêtre Capture.
REMARQUE: À ce stade, ajustez les paramètres suivants. Position du faisceau (il peut être déplacé de haut en bas sur l’écran). Niveau caméra: évitez les pixels sursaturés. Mise au point (roue latérale): essayez de vous concentrer le mieux possible. Il est préférable d’avoir des particules avec un halo au lieu de particules non focalisées. Concentration : Ajustez la concentration pour avoir entre 10 et 100 particules par champ.
- Tout d’abord, dans le matériel, vérifiez si la caméra et le laser approprié sont sélectionnés.
- Enregistrement vidéo
- Dans le logiciel, accédez à la fenêtre Sélection de mesure- SOP (en bas à gauche) et sélectionnez Mesure standard.
REMARQUE: Programme pour effectuer trois mesures de 30 s chacune (pour permettre au logiciel d’effectuer des calculs de moyenne et d’écart type). Fournissez un nom et un chemin d’accès au dossier (sur Nom de fichier de base) pour enregistrer les enregistrements de l’exemple. Lorsque les paramètres SOP sont correctement configurés, appuyez sur Create and Run Script. Une fois la première réplication mesurée, le programme demandera d’ajouter un nouvel échantillon. Ensuite, une autre fraction de l’échantillon doit être introduite dans la chambre en poussant le piston de la seringue. Enfin, répétez une troisième fois pour analyser la troisième réplique.
- Dans le logiciel, accédez à la fenêtre Sélection de mesure- SOP (en bas à gauche) et sélectionnez Mesure standard.
- Traitement vidéo
- Réajustez le gain d’écran et le seuil de détection une fois les trois mesures terminées pour analyser les particules mesurées. À ce stade, environ 100 particules devraient apparaître dans chaque image pour effectuer l’analyse (des croix rouges élevées et des croix bleues basses sont attendues).
- Exportez les résultats dans un fichier pdf une fois l’analyse terminée. En outre, il est possible d’exporter sous forme de vidéos et de fichiers Excel.
- Nettoyage de la NTA
REMARQUE: Fermez toutes les mesures ouvertes avant d’étudier l’échantillon suivant; étant donné que les fichiers générés sont énormes et dépendent de l’ordinateur, il n’est pas facile de maintenir plus d’un ouvert.- Nettoyer la chambre NTA en rinçant l’eau à plusieurs reprises avant d’effectuer la mesure ultérieure jusqu’à ce qu’aucune particule ne soit observée; par la suite, vider le tampon utilisé (PBS) pour poursuivre les mesures.
- Rincez l’air à l’intérieur de la chambre et séchez-le avec du papier de qualité microscopique une fois la dernière mesure de la journée terminée.
- Caractériser la taille et la forme par microscopie électronique à transmission (TEM). Préparez les échantillons. 30 μL du volume final suffisent pour la caractérisation TEM.
REMARQUE: Les nanoparticules fraîches et lyophilisées - après remise en suspension - peuvent être mesurées. - Déposer 10 μL d’échantillon sur une grille de cuivre recouverte de carbone. Laissez sécher pendant 10 min. Retirez l’excès de liquide, si nécessaire, en tapotant doucement sur du papier filtre.
- Laisser tomber 10 μL de solution d’acétate d’uranyle (2 % p/v) pour une coloration négative. Laissez sécher pendant 1 min. Retirez l’excès de liquide, si nécessaire, en tapotant doucement sur le papier filtre.
- Introduire l’échantillon au microscope et le scanner (fonctionnement en tension 80 kV).
REMARQUE: Un logiciel approprié peut être utilisé pour une analyse plus approfondie des images.
- NTA et préparation des échantillons
- Efficacité d’encapsulation
- Préparation des échantillons
- Préparez les nanoparticules à la concentration souhaitée et congelez-les et séchez-les. Ensuite, remettez en suspension les nanoparticules et diluez-les à une concentration finale de 6 μg/mL.
- Préparez 1x tampon TE à partir de la solution mère 20x.
NOTE: Vt (Volume total) = (nombre d’échantillons x 100 μL x 4) (nombre d’échantillons x 290 μL) + 2 mL. - Préparer le tampon TE avec 3 μg/μL d’héparine à partir du stock de 100 μg/μL.
REMARQUE: Vt = Nombre d’échantillons x 50 μL x 2. - Préparer les normes
- Préparer un étalon d’ARN allant de 0,2 μg/mL à 0,025 μg/mL dans 1x tampon Tris-EDTA (TE)(Tableau S1A).
REMARQUE: Utilisez l’ARNm dans l’étalon d’ARN dans le cas où cela diffère de l’ARN ribosomique. - Préparer l’étalon ARN:pBAE avec l’héparine (Tableau S1B). Préparer l’étalon d’héparine (Tableau S1C).
- Préparez une assiette de 96 puits comme suit.
- Chargez 295 μL de tampon TE dans la première voie d’une plaque de 96 puits (autant de puits que d’échantillons à analyser). Ensuite, chargez 50 μL de tampon 1x TE dans les voies B et C (doublons pour chaque échantillon).
- Chargez 50 μL de tampon TBE 1x avec 3 μg/μL d’héparine dans les voies D et E (doublons pour chaque échantillon). Ensuite, chargez 100 μL de chaque étalon dans les voies F, G et H (doublons pour chaque étalon).
- Chargez 5 μL de chaque échantillon dans la voie A (la concentration dans chaque puits est de 0,1 μg/mL). Mélanger correctement par pipetage et charger 50 μL de chaque échantillon sur les quatre puits ci-dessous (deux d’entre eux contenant 50 μL de tampon 1x TE et deux autres contenant 1x tampon TE avec 3 μg/μL d’héparine).
- Incuber l’échantillon pendant 30 min à 37 °C. Préparez le réactif RiboGreen selon le protocole du fabricant (voir Tableau des matériaux)dans un tampon 1x TE.
REMARQUE: Diluer 1:200 et vortex. Protéger de la lumière. Vt = Nombre de puits complets x 100 μL. - Chargez 100 μL de solution RiboGreen dans chaque puits. Détectez la fluorescence à l’aide du lecteur de microplaques avec une longueur d’onde d’excitation de 500 nm et une longueur d’onde d’émission de 525 nm.
- Analyse des résultats
- Préparer les trois courbes d’étalonnage
REMARQUE: Tout d’abord, l’ARN ribosomique standard. Interpolez dans cette courbe d’étalonnage les échantillons qui ne contiennent pas d’héparine. Deuxièmement, aRNm: pBAE avec héparine. Interpolez dans cette courbe d’étalonnage l’échantillon contenant de l’héparine. Troisièmement, l’héparine. Utilisé pour s’assurer que la concentration de travail est dans la gamme linéaire. - Calculez l’efficacité d’encapsulation (EE%) comme suit :
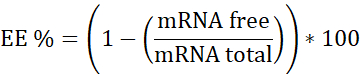
- Préparer les trois courbes d’étalonnage
- Préparation des échantillons
6. Caractérisation in vitro
- Microscopie à fluorescence pour l’évaluation qualitative
- Placez la plaque de 96 puits sur le microscope après 24 heures de transfection. Commencez à visualiser les cellules avec l’objectif 10x.
REMARQUE: Les cellules HeLa sont utilisées dans ce cas. Les cellules sont localisées à l’aide du mode de transmission (champ lumineux) et la mise au point doit être ajustée à ce moment-là.- Tout d’abord, appliquez une balance des blancs pour référencer le logiciel sur l’arrière-plan des échantillons. Ensuite, acquérez une image pour la superposer à l’image de fluorescence pendant l’analyse.
- Passez le mode microscope en mode réflexion (fluorescence) et déplacez la roue filtrante vers le laser bleu (488 nm) pour visualiser eGFP.
REMARQUE: À ce stade, le temps d’exposition et le gain doivent être ajustés. Le gain doit être ajusté dans des valeurs comprises entre 3 et 5 pour éviter les artefacts et l’excès sur le signal d’arrière-plan. Le réglage du temps d’exposition dépend de l’efficacité de transfection (de ms à 1 s). - Acquérir des images pour toutes les conditions ou puits en utilisant le même temps d’exposition pour comparer tous les échantillons.
- Placez la plaque de 96 puits sur le microscope après 24 heures de transfection. Commencez à visualiser les cellules avec l’objectif 10x.
- Cytométrie en flux (FC) pour l’évaluation quantitative
- Utilisez une pipette multicanal pour préparer la plaque de 96 puits pour l’expérience de cytométrie en flux.
REMARQUE: Lorsque vous travaillez avec des cellules semi-adhérentes, comme dans la lignée cellulaire JAWSII, récupérez tout le volume du support et stockez-le dans une autre plaque de 96 puits. N’aspirez pas à ce milieu car il pourrait contenir un nombre important de cellules transfectées. - Nettoyez les cellules (les cellules HeLa sont utilisées ici) avec 100 μL / puits de 1x PBS et aspirez-y. Ensuite, ajoutez 25 μL/puits de trypsine et incubez-la à 37 °C pendant 5 min.
- Une fois les cellules détachées, ajoutez un milieu préalablement récupéré pour arrêter l’action de la trypsine et fixez les cellules, en ajoutant 31,25 μL / puits de formol à 10% pendant 20 min (concentration finale 2,5%).
REMARQUE: À ce stade, il est crucial de visualiser l’état des cellules par microscopie pour assurer le détachement des cellules. Il est fortement recommandé de pipeter de haut en bas plusieurs fois pour aider les cellules à se détacher et éviter leur agglomération. Cela produira un résultat fiable. - Allumez le cytomètre en flux et le logiciel. Dans le logiciel, configurez les conditions appropriées pour l’expérience (type de plaque, volume de l’échantillon et autres paramètres tels que l’agitation, le rinçage entre les échantillons.
REMARQUE: Pour l’étape 6.2.5, le débit doit être de 120 μL / min. Mélangez un cycle toutes les 1 min et rincez bien un cycle tous les jours. Le mélange est essentiel pour maintenir les cellules dispersées dans le milieu et permettre une meilleure analyse des cellules individuelles. De plus, un rinçage est nécessaire pour nettoyer la microfluidique du cytomètre entre les échantillons. Enfin, vérifiez s’il y a suffisamment de tampons et si tous les composants sont correctement connectés. - Définissez les paramètres appropriés pour quantifier le pourcentage de cellules transfectées positivement.
- Tout d’abord, visualisez les données de flux sur le diagramme de dispersion FSC (lumière diffusée vers l’avant) par rapport au SCC (lumière diffusée latérale) pour distinguer les cellules des débris. Ensuite, un autre nuage de points comparant l’amplitude (FSC-A) par rapport à la hauteur (FSC-H) est tracé pour griller et discriminer les cellules individuelles.
NOTE: La population non traitée permet de griller les populations correspondant à des cellules positivement transfectées. Ensuite, la quantification des cellules positivement transfectées est effectuée sur le canal approprié sur un graphique d’histogramme. Par conséquent, les cellules positivement transfectées doivent être représentées comme un continuum d’événements sur l’histogramme.
- Tout d’abord, visualisez les données de flux sur le diagramme de dispersion FSC (lumière diffusée vers l’avant) par rapport au SCC (lumière diffusée latérale) pour distinguer les cellules des débris. Ensuite, un autre nuage de points comparant l’amplitude (FSC-A) par rapport à la hauteur (FSC-H) est tracé pour griller et discriminer les cellules individuelles.
- Utilisez une pipette multicanal pour préparer la plaque de 96 puits pour l’expérience de cytométrie en flux.
7. Tests de fonctionnalité in vitro : capacité d’activer des cellules immunitaires modèles en utilisant l’ovalbumine (OVA) comme ARNm modèle antigénique
- Plaque 10 000 cellules/puits (les cellules JAWSII sont utilisées ici) dans une plaque de 96 puits la veille de la transfection.
REMARQUE: Plaquez autant de puits que nécessaire pour avoir des triples pour chaque condition. Laissez les cellules s’attacher à la plaque au moins pendant 12 h (une incubation de nuit est recommandée). - Préparer les NP pBAE comme décrit dans la NOTE ci-dessous pour transfecter 0,6 μg d’ARNm par puits.
REMARQUE: En tant que témoin positif, transfecter les cellules à l’aide d’un réactif de transfection. Ici, nous avons utilisé 0,1 μg d’ARNm par puits et 0,25 μL de réactif de transfection par puits. La codification de l’ARNm pour l’OVA a été achetée (voir tableau des matériaux). Il est polyadénylé, modifié avec de la 5-méthoxyuridine, optimisé pour les systèmes mammifères, et est protégé par un bouchon final avec une structure Cap1, une méthode exclusive du fournisseur. - Incuber la plaque de 96 puits pendant 24 h dans un incubateur à air sec à 37 °C et 5% de CO2.
REMARQUE: Passé ce délai, n’aspirez pas le média car il peut contenir un certain nombre de cellules. Récupérez les cellules et conservez-les dans un autre puits ou une autre assiette. - Lavez les cellules restantes sur le puits avec 25 μL de 1x PBS. Ensuite, aspirez-le et ajoutez 25 μL de trypsine et incubez la plaque pendant 5 min à 37 °C pour détacher les cellules. Arrêtez la réaction de trypsine en ajoutant le média précédemment récupéré sur le puits correspondant.
- Centrifuger la plaque à 400 x g pendant 5 min à 4 °C. Aspirez les médias. Ajouter 50 μL/puits d’un PBS 1x et 2,5 % de formol. Incuber à 4 °C pendant 20 min pour fixer les cellules.
- Répétez les étapes 7.5 et 7.5.1. Ensuite, ajoutez 50 μL/puits de 1x PBS et 3% de BSA (tampon bloquant) et incubez pendant 30 min à 4 °C. Encore une fois, répétez les étapes 7.5 et 7.5.1, puis ajoutez 50 μL/puits de l’anticorps primaire (souris α-OVA) dans 1x PBS et 3% de BSA et incubez pendant 30 min à 4 °C.
- Répétez les étapes 7.5 et 7.5.1, puis lavez les cellules avec 50 μL/puits de 1x PBS. Aspirer un milieu, puis ajouter une solution d’anticorps secondaires (α-souris-AlexaFluor488/PerCP et Cy5.5-CD11b/APC-CD86) dans 1x PBS et 3% BSA. Incubez-le pendant 1 h à 4 °C.
- Centrifuger la plaque à 400 x g pendant 5 min. Ensuite, lavez les cellules avec 50 μL / puits de 1x PBS. Ensuite, centrifugez (400 x g pendant 5 min), aspirez, puis remettez en suspension les cellules dans 100 μL / puits de 1x PBS et 2,5% de formol.
- Analysez-le par cytométrie en flux, comme décrit ci-dessus (étape 6.2).
Access restricted. Please log in or start a trial to view this content.
Résultats
Synthèse et caractérisation des polymères
La procédure de synthèse OM-pBAE est donnée à la figure 2. Comme le montre la figure 2A, la première étape pour obtenir l’OM-pBAE consiste à synthétiser le C6-pBAE en ajoutant les amines (1-hexylamine et 5-amino-1-pentanol, rapport 1:1) au diacrylate (diacrylate de 1,4-butanediol). Cette réaction est réalisée à 90 °C pendant 20 h et sous agitation constante. Ensuite, une solution d?...
Access restricted. Please log in or start a trial to view this content.
Discussion
Après l’apparition de la pandémie de Covid-19 l’année dernière, l’importance des vaccins en termes de contrôle des maladies infectieuses s’est manifestée comme une composante critique8. Les efforts des scientifiques du monde entier ont permis la mise sur le marché de nombreux vaccins. Pour la première fois dans l’histoire, les vaccins à ARNm ont démontré leur succès supposé auparavant, grâce à leur conception rapide en raison de leur capacité à s’adapter à tout nouvel...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer ni aucun conflit d’intérêts.
Remerciements
Le soutien financier de MINECO/FEDER (subventions SAF2015-64927-C2-2-R, RTI2018-094734-B-C22 et COV20/01100) est reconnu. CGF a reconnu sa bourse de doctorat IQS.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 1,4-butanediol diacrylate | Sigma Aldrich | 123048 | |
| 1-hexylamine | Sigma Aldrich | 219703 | |
| 5-amino-1-pentanol | Sigma Aldrich | 411744 | |
| Acetone | Panreac | 141007 | |
| CD11b antibody | BD | 550993 | |
| CD86 antibody | Bioligend | 105007 | |
| Chlor hydroxhyde | Panreac | 181023 | |
| Chloroform-d | Sigma Aldrich | 151823 | |
| Cys-His-His-His peptide | Ontores | Custom | |
| Cys-Lys-Lys-Lys peptide | Ontores | Custom | |
| D2O | Sigma Aldrich | 151882 | |
| DEPC reagent for Rnase free water | Sigma Aldrich | D5758 | This reagent is important to treat MilliQ water to remove any RNases of the buffers |
| Diethyl eter | Panreac | 212770 | |
| dimethyl sulfoxide | Sigma Aldrich | 276855 | |
| HEPES | Sigma Aldrich | H3375 | |
| mRNA EGFP | TriLink Technologies | L-7601 | |
| mRNA OVA | TriLink Technologies | L-7610 | |
| RiboGreen kit | ThermoFisher | R11490 | |
| sodium acetate | Sigma Aldrich | 71196 | |
| sucrose | Sigma Aldrich | S0389 | |
| Trifluoroacetic acid | Sigma Aldrich | 302031 | |
| Trypsin-EDTA | Fisher Scientific | 11570626 | |
| α-mouse AlexaFluor488 antibody | Abcam | Ab450105 | |
| Equipment | |||
| Nanoparticle Tracking Analyzer | Malvern Panalytical | NanoSight NS300 | |
| Nuclear Magnetic Ressonance Spectrometer | Varian | 400 MHz | |
| ZetaSizer | Malvern Panalytical | Nano ZS | For zeta potential and hydrodynamic size determination |
| Software | |||
| NanoSight NTA software | Malvern Panalytical | MAN0515-02-EN-00 | |
| NovoExpress Software | Agilent | Not specified | |
| ZetaSizer software | Malvern Panalytical | DTS Application | To analyze surface charge and hydrodynamic sizes |
Références
- Chumakov, K., Benn, C. S., Aaby, P., Kottilil, S., Gallo, R. Can existing live vaccines prevent COVID-19. Science. 368 (6496), 1187-1188 (2020).
- Zhang, C., Maruggi, G., Shan, H., Li, J. Advances in mRNA vaccines for infectious diseases. Frontiers in Immunology. 10, 1-13 (2019).
- Wherry, E. J., Jaffee, E. M., Warren, N., D'Souza, G., Ribas, A. How did we get a COVID-19 vaccine in less than 1 year. Clinical Cancer Research. 27 (8), 2136-2138 (2021).
- Folegatti, P. M., et al. Safety and immunogenicity of the ChAdOx1 nCoV-19 vaccine against SARS-CoV-2: a preliminary report of a phase 1/2, single-blind, randomised controlled trial. The Lancet. 396 (10249), 467-478 (2020).
- Geall, A. J., Mandl, C. W., Ulmer, J. B. RNA: The new revolution in nucleic acid vaccines. Seminars in Immunology. 25 (2), 152-159 (2013).
- Ulmer, J. B., Geall, A. J. Recent innovations in mRNA vaccines. Current Opinion in Immunology. 41, 18-22 (2016).
- Kranz, L. M., et al. Systemic RNA delivery to dendritic cells exploits antiviral defence for cancer immunotherapy. Nature. 534 (7607), 396-401 (2016).
- Milane, L., Amiji, M. Clinical approval of nanotechnology-based SARS-CoV-2 mRNA vaccines: impact on translational nanomedicine. Drug Delivery and Translational Research. 1 (4), 3(2020).
- Green, J. J., Langer, R., Anderson, D. G. A combinatorial polymer library approach yields insight into nonviral gene delivery. Accounts of Chemical Research. 41 (6), 749-759 (2008).
- Guerrero-Cázares, H., et al. Biodegradable polymeric nanoparticles show high efficacy and specificity at DNA delivery to human glioblastoma in vitro and in vivo. ACS Nano. 8 (5), 5141-5153 (2014).
- Kozielski, K. L., Tzeng, S. Y., Hurtado De Mendoza, B. A., Green, J. J. Bioreducible cationic polymer-based nanoparticles for efficient and environmentally triggered cytoplasmic siRNA delivery to primary human brain cancer cells. ACS Nano. 8 (4), 3232-3241 (2014).
- Segovia, N., Dosta, P., Cascante, A., Ramos, V., Borrós, S. Oligopeptide-terminated poly(β-amino ester)s for highly efficient gene delivery and intracellular localization. Acta Biomaterialia. 10 (5), 2147-2158 (2014).
- Dosta, P., Segovia, N., Cascante, A., Ramos, V., Borrós, S. Surface charge tunability as a powerful strategy to control electrostatic interaction for high efficiency silencing, using tailored oligopeptide- modified poly (beta-amino ester)s (PBAEs). Acta Biomaterialia. 20, 82-93 (2015).
- Fornaguera, C., et al. mRNA delivery system for targeting antigen-presenting cells in vivo. Advanced Healthcare Materials. 7 (17), 180033(2018).
- Fornaguera, C., Castells-Sala, C., Lázaro, M. Á, Cascante, A., Borrós, S. Development of an optimized freeze-drying protocol for OM-PBAE nucleic acid polyplexes. International Journal Pharmaceutics. 569, (2019).
- Fornaguera, C., Solans, C. Analytical methods to characterize and purify polymeric nanoparticles. International Journal of Polymer Science. , (2018).
- Fornaguera, C., Solans, C. Characterization of polymeric nanoparticle dispersions for biomedical applications: size, surface charge and stability. Pharmaceutical Nanotechnology. 6 (3), 147-164 (2018).
- Sahin, U., Karikó, K., Türeci, Ö MRNA-based therapeutics-developing a new class of drugs. Nature Reviews Drug Discovery. 13 (10), 759-780 (2014).
- Fan, Y. N., et al. Cationic lipid-assisted nanoparticles for delivery of mRNA cancer vaccine. Biomaterials Science. 6 (11), 3009-3018 (2018).
- Le Moignic, A., et al. Preclinical evaluation of mRNA trimannosylated lipopolyplexes as therapeutic cancer vaccines targeting dendritic cells. Journal of Controlled Release. 278, 110-121 (2018).
- Banerji, A., et al. mRNA vaccines to prevent COVID-19 disease and reported allergic reactions: Current evidence and approach. Journal of Allergy and Clinical Immunology: In Practice. 9 (4), 1423-1437 (2021).
- Kaczmarek, J. C., Kowalski, P. S., Anderson, D. G. Advances in the delivery of RNA therapeutics: from concept to clinical reality. Genome Medicine. 9 (1), 1-16 (2017).
- Dosta, P., et al. Delivery of anti-microRNA-712 to inflamed endothelial cells using poly(β-amino ester) nanoparticles conjugated with VCAM-1 targeting peptide. Advanced Healthcare Materials. , 1-11 (2021).
- Segovia, N., et al. Hydrogel doped with nanoparticles for local sustained release of siRNA in breast cancer. Advanced Healthcare Materials. 4 (2), 271-280 (2015).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon