JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
mRNA로 로드된 폴리(베타 아미노에스) 나노입자의 합성 및 특성화
요약
여기서, 폴리(베타 아미노에스) 폴리머를 기반으로 mRNA 나노입자를 제조하기 위한 간단한 프로토콜이 제시되고, 캡슐화된 mRNA를 변경하여 맞춤화되기 쉽다. 중합체, 나노입자 및 체외 필수 특성화를 합성하기 위한 워크플로우도 설명되어 있습니다. 예방 접종에 관한 개념 증명도 추가됩니다.
초록
예방 접종은 현대 사회의 주요 성공 중 하나이며 질병을 통제하고 예방하는 데 필수적입니다. 전통적인 백신은 전염하는 에이전트의 전체 또는 분수로 구성되었다. 그러나 과제는 남아 있으며 새로운 백신 기술은 필수입니다. 이러한 맥락에서, 예방 접종 목적을 위한 mRNA의 사용은 SARS-CoV-2 감염을 방지하는 2개의 mRNA 백신의 신속한 승인에 의해 입증된 바와 같이 향상된 성능을 보여주었습니다. 바이러스 감염 예방에 성공 하는 것 외에도 mRNA 백신 치료 암 응용 프로그램에 사용할 수 있습니다.
그럼에도 불구 하 고, mRNA의 불안정과 핵의 존재로 인해 신체에서 그것의 빠른 간격은 그것의 알몸 배달 불가능. 이러한 맥락에서, 나노 의약품 및 특히 중합체 나노 입자는 중요한 mRNA 전달 시스템입니다. 따라서, 본 문서의 목적은 독점적 중합체 나노입자에 기초하여 mRNA 백신 후보의 제형 및 시험을 위한 프로토콜을 기술하는 것이다. 폴리(베타 아미노에스) 폴리머의 합성 및 화학적 특성화, 나노입자를 형성하기 위한 mRNA와의 복합성, 및 이들의 구종화 방법론이 여기에서 논의될 것이다. 이는 스토리지 및 유통 비용을 줄이는 중요한 단계입니다. 마지막으로, 시험관 내 및 성숙한 모델 수지상 세포에 대한 능력을 입증하는 데 필요한 검사가 표시됩니다. 이 프로토콜은 이러한 백신이 다양한 질병을 예방하거나 치료할 수 있도록 하는 높은 다기능성 때문에 예방 접종에 종사하는 과학 계에 도움이 될 것입니다.
서문
전염병은 전 세계 수백만 명의 인간에게 심각한 위협을 나타내고 있으며 여전히 일부 개발 도상국에서 주요 사망 원인 중 하나입니다. 예방 접종은 전염병을 예방하고 통제하기 위해 현대 사회의 가장 효과적인 개입 중 하나였습니다1,2. 20세기관련성에 있는 과학의 이 중요한 이정표는 SARS-CoV-2 바이러스3에기인한 최근 세계적인 Covid-19 전염병에 의해 언급되었습니다. 질병의 보급을 줄이기 위해 효율적인 백신을 갖는 것의 중요성을 인식, 모든 생물 의학 지역 사회에서 협력 노력은 성공적으로 1 년 이내에 시장에서 많은 예방 백신을 초래했다4.
전통적으로, 백신은 감쇠 (살아있는, 감소된 독성) 또는 불활성화 (죽음 입자) 바이러스로 구성되었다. 그러나, 안전 오류에 대한 여백이없는 일부 질병의 경우, 바이러스 성 입자는 불가능하고, 단백질 하위 단위는 대신 사용된다. 그럼에도 불구하고, 서브유닛은 일반적으로 하나 이상의 에피토프/항원의 조합을 활성화하지 않으며, 유족은 백신 접종 효능5,6을향상시켜야 한다. 따라서 새로운 백신 유형에 대한 필요성은 분명합니다.
현재 전염병 동안 입증된 바와 같이, 핵산을 기반으로 한 새로운 백신 후보는 긴 발달 과정을 피하고 동시에 중요한 환자 예방 접종을 생산하는 동안 높은 다기능성을 제공하는 측면에서 유리할 수 있다. 이것은 처음에 실험적인 암 백신으로 디자인된 mRNA 백신의 케이스입니다. 항원 특이적 T세포 반응3,5,6,7을생성하는 자연적인 능력 덕분. mRNA는 항원 단백질을 인코딩하는 분자로서, 단지 동일하게 변화하는, 백신은 동일한 미생물, 다른 균주, 다른 전염성 미생물의 다른 변이체를 예방 접종하기 위해 신속하게 조정될 수 있거나, 심지어 암 면역 치료 치료법이 될 수 있다. 또한 대규모 생산 비용 면에서 유리합니다. 그러나, mRNA는 그들의 벌거 벗은 행정을 방해하는 중요한 장애물이 있습니다: 그것의 안정성 및 무결성은 생리적인 매체에서 손상됩니다, 핵의 가득 차있는. 이러한 이유로, 이를 보호하고 mRNA를 항원 제시 세포로 벡터화하는 나노메트릭 담체의 사용은2,8이필요하다.
이러한 맥락에서, 폴리(베타 아미노에스)(pBAE)는 생체적합성 및 생분해성 폴리머의 일종으로, 양이온화9,10,11덕분에나노메트릭 입자에서 mRNA를 복합화하는 놀라운 능력을 입증하였다. 이 중합체는 생리적인 조건에서 에스테라아에 의해 그들의 분해를 쉽게 만드는 에스테르 결합으로 이루어집니다. pBAE 라이브러리 후보 중, 종말 양이온 올리고펩타이드로 기능화된 사람들은 내분비증을 통해 세포를 효율적으로 침투시키고 캡슐화된 유전자 물질을 이식하기 위해 작은 나노입자를 형성하는 더 높은 용량을 보였다. 또한, 버퍼링 용량 덕분에 내성 구획의 산성화는 내도 탈출12,13을허용한다. 즉, pBAE의 특정 종류, 그들의 백본에 소수성 모니티를 포함 (소위 C6 pBAE) 그들의 안정성을 향상 시키고 말 올리고펩티드 조합 (3-lysine로 변형 된 폴리머의 60%와 삼부스티딘을 가진 폴리머의 40%)는 비경구 투여 후 항원 제시 세포를 선택적으로 트랜스펙트 및 mRNA 코드 내포 항원 발표1 . 또한, 이러한 제형은 나노의학 제제의 주요 병목 현상 단계 중 하나를 우회할 수 있다는 것을 입증하였다: 그들의 기능을 잃지 않고 동결 건조할 수 있는 가능성, 이는 연약한 건조한 환경에서 장기적인 안정성을 가능하게한다(15).
이러한 맥락에서, 현재 프로토콜의 목적은 프로토콜의 중요한 단계에 대한 설명을 제공하고 전염병 예방 및 종양 치료 응용을 위한 효율적인 백신의 생산을 가능하게 함으로써 과학 계에 사용할 수 있는 mRNA 나노입자의 형성을 위한 절차를 만드는 것이다.
다음 프로토콜은 올리고펩티드 최종 변형 폴리(베타 아미노에스)를 합성하기 위한 완전한 운동을 설명합니다- 나노입자 합성에 더 사용될 OM-pBAE 폴리머. 프로토콜에서, 나노입자 제형도 포함되어 있다. 또한, 절차 및 대표 결과의 성공을 위한 중요한 단계는 또한 결과 제형이 양수 또는 음수 결과를 정의하는 데 필요한 품질 관리 특성화 기능을 달성할 수 있도록 제공된다. 이 프로토콜은 그림 1로요약되어 있습니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
1. 끝 올리고펩타이드를 함유한 pBAE 폴리머합성(OM-pBAE)
- C6-pBAE의 중합
- 5-아미노-1-펜타놀을 추가 (38 mmol; MW = 103.16 다) 1-헥실라민 (38 mmol; MW = 101.19 Da)를 둥근 바닥 유리 플라스크(100 mL)로 넣습니다. 그런 다음 1,4-부탄디올 디아크릴레이트(82mmol)를 추가합니다. MW = 198.22 Da).
- 실리콘 오일 목욕을 90°C에서 예열하고, 둥근 바닥 플라스크를 오일 목욕에 넣고 하룻밤 동안 자기 교반 바(~18h)의 도움으로 혼합물을 저어줍니다. 그런 다음, 둥근 바닥 플라스크에서 제품을 가지고 -20 °C에서 냉동실에 놓습니다.
참고 : 제품은 끈적 끈적한 분말의 형태로 주걱의 도움으로 플라스크에서 꺼낸다. 1H-NMR을 통해 얻어진 폴리머의 구조를 검증하는 것이 필수적이다. NMR 스펙트럼은 클로로폼-d 및 D2O를 용매로 사용하여 400MHz 기기(재료 표참조)에 기록되었습니다. 각 폴리(β-amino ester)의 약 10 mg을 중음용매의 1mL에 취하고 용해하였다.
- OM-pBAE를 얻기 위한 펩티드와의 반응
- 25mL의 0.1 M HCl을 선택한 펩티드에 추가하십시오( 예: 펩타이드 Cys-His-His-His-His-With Trifluoroacetic acid (TFA, 200 mg), 이전에 무게가 있는 50mL 원심분리기 튜브에 동결 건조를 서 고 하룻밤 수동으로 저어 맑은 용액을 얻을 수 있습니다.
- 용액을 -80°C에서 1h로 동결하고 생성된 펩타이드 염산염을 동결건조시한다.
참고: 최종 가중치가 이론값에 해당하는지 확인합니다. - 디메틸 설프리산화물에서 C6-pBAE(0.031 mmol)의 용액을 만드십시오. 또한, 디메틸 설산화물에서 펩타이드 염산염(0.078 mmol)의 용액을 만든다.
- 두 가지 솔루션을 나사 캡 튜브에 섞고 캡에 나사를 넣습니다. 마그네틱 스터드 바와 함께 25°C의 온도가 25°C인 수조에서 혼합물 용액을 저어줍니다.
- 혼합물을 7:3 (v/v) 디틸 에테르/아세톤에 넣습니다. 4°C에서 25,000 x g의 결과 현탁액을 원심분리하여 용매를 제거합니다. 다음으로 7:3 (v/v) 디틸 에테르/아세톤으로 솔리드를 두 번 씻으세요. 그런 다음 진공(<0.2 atm)에서 제품을 건조시합니다.
- 디메틸 설프리산화물로 제품의 100 mg/mL의 용액을 만드십시오. 결과 제품은 C6-펩티드-pBAE라고 명명된다. 1 H-NMR을 통해 얻어진 중합체의 구조를 검증하여 말단 아크릴산과 관련된 올레핀 신호의 실종을 확인하는 것이 필수적이다. 사용하지 않으면 폴리머는 -20 °C에서 동결 될 수 있습니다.
2. 폴리플렉스 형성
참고: 일정한 온도를 유지하기 위해 조건부 실 내에서 모든 절차를 수행해야 합니다.
- 폴리머 C6-펩티드-pBAE를 해동하고 용액을 소용돌이시다.
- 파이펫 폴리머는 위아래로 혼합하고 아세테이트 나트륨(NaAc)에서 12.5mM(V1)의 용액을 준비한다. 그런 다음 혼합물을 소용돌이쳐 10 분 동안 기다립니다.
- mRNA를 0.5 mg/mL로 준비하고 파이펫팅(V2)으로섞는다.
참고: mRNA를 피하는 것이 중요합니다. - 최종 농도에서 중합체 혼합물을 소용돌이시키고 DMSO및 아세테이트 버퍼의 중합체 스톡 사이의 균일한 용액을 달성한다.
참고: 최종 폴리머 농도는 선택된 N/P(질소 대 인산염 군) 비율에 따라 달라집니다. N/P 비율은 사용할 각 특정 mRNA에 따라 달라집니다. eGFP 캡슐화의 경우, 예를 들어 이전에 보고된14와같이 25:1 비율이 사용되었습니다. - 1:1(Vi =V1 + V2)의비율로 유전 물질 용액 및 C6-펩티드프BAE 용액(mRNA 농도의 25배)을 혼합한다.
참고: C6-펩티드-pBAE는 마이크로센트심분리기 튜브에 적재되어 있으며, 여기서 mRNA는 혼합을 위해 위아래로 파이프팅하여 첨가됩니다. 일단 준비되면, 폴리플렉스, 핵산 및 C6-펩티드-pBAE 농도가 반 희석된다. - 온도 블록에서 30 분 동안 25 °C에서 배양하십시오. 1:2 RNase 자유 물로 침전하여 미리 장재된 미세 원심 분리기 튜브에 물을 첨가합니다.
- 부형제를 포함합니다. HEPES 20 mM에서 mRNA 및 pBAE(Vi)의혼합물과 동일한 부피를 추가하고 위아래로 피펫팅하여 4%를 자당합니다. 이 시점에서 샘플은 3배 희석되었습니다.
3. 폴리플렉스 라이오필화
- 즉각적으로, 이전 폴리플렉스 용액의 -80°C 냉동고에서 1h로 동결한다.
- 단계 수행: (1) -60°C및 0.001 hPa에서 1h; (2) -40°C 및 0.0001 hPa에서 1h; (3) -20°C 및 0.0001 hPa에서 4h; (4) 5 °C및 0.0001 hPa에서 12 h.
- 사용 전까지 수분을 보충하지 않도록 -20 °C에 즉시 보관하십시오.
4. 폴리플렉스 리서스펜션
참고: 이 프로토콜은 특성화, 시험관 내 또는 생체 내 분석을 위해 추가 용도로 사용하기 위해 lyophilized C6-펩티드-pBAE 나노입자를 재구성하는 데 사용되는 프로세스를 설명합니다.
- 사용 시 -20°C에서 lyophilized 나노 입자를 취하고 원하는 농도를 달성하기 위해 고체를 재분산시키기 위해 상응하는 양의 depyrogenated (DEPC) 물을 신속하게 첨가하십시오.
참고: 동일한 농도가 예상되는 경우 부피는 나노입자의 초기 부피와 동일하지만 각 실험에 대해 표시됩니다. - 파이펫과 액체를 동반, 총 재중단 까지 부드럽게 파이펫.
참고: 유리병 벽에 는 물질이 남아 있지 않도록 하십시오. - 일단 용해되면, 피펫은 거품을 피하면서 격렬하게 위아래로 내려.
참고: 샘플은 반투명 측면에 투명해야 하며, 이는 더 높은 농도에서 더 분명합니다. - 샘플을 얼음 또는 4°C에 저장하여 재구성한 경우 최대 24시간 동안 보관합니다. 동결을 피하십시오.
5. 폴리플렉스 특성화
- 동적 조명 산란
참고: 유체역학 직경(nm), 다분산지수(PDI), 나노입자의 표면 전하는 제타 전위 분석기를 사용하여 25°C, 633nm 레이저 파장 및 173° 신호 검출기로 측정하였다(재료표참조).- 유체 역학 직경 (nm)
- 앞서 설명한 바와 같이 mRNA와 pBAE를 완전히 혼합하는 나노입자를 0.25 mg/mL의 mRNA의 최종 농도로 폴리머 분획으로 배관하여(2단계)를 준비한다.
- 여과 된 희석 매체로 마이크로 큐벳을 미리 헹구어 불순물에서 완전히 청소하십시오. 그런 다음, 큐벳을 샘플 용액의 최소 50 μL로 채우고 캡을 씌웁니다. 그런 다음 DLS 기기 내부에 샘플을 도입하여 셀이 올바르게 삽입되었는지 확인합니다.
참고: 희석 매체는 0.22 μm 주사기 필터를 사용하여 필터링됩니다. - 만든 표준 작업 절차(SOP) 파일을 열고 원하는 샘플 이름을소개합니다. 크기 측정을 선택합니다.
참고: 크기 측정을 위한 SOP를 만드는 모든 매개 변수는 표 1에제공됩니다. - 재생을 클릭하여 DLS를 사용하여 파티클 크기 측정을 실행합니다.
참고: 트리플 비프음 소리가 나면 분석이 완료됩니다. - 측정 시트의 샘플에 해당하는 결과를 선택하여 평균 입자 크기, 평균 PDI, 표준 편차 및 그래픽을 가져옵니다. 완료되면 DLS 장비에서 셀을 제거합니다.
- 샘플을 제거하고 제타 전위 분석을 위해 보관하고 탈이온화된 물로 세포를 깨끗하게 헹구십시오. 마지막으로, 압축 공기 가스 스트림에서 청소 된 큐벳을 건조.
- 표면 충전(mV)
- 앞서 설명한 바와 같이 mRNA와 pBAE를 완전히 혼합하는 나노입자를 0.25 mg/mL의 mRNA의 최종 농도로 폴리머 분획으로 배관하여(2단계, 폴리플렉스 형성)을 준비한다. 다음으로 용액을 동결하고 건조시십시오.
참고: 이 경우, 표면 전하의 측정을 위해, 시료의 동결 건조는 pH 및 전해질 농도의 체조건을 시뮬레이션하여 적절한 버퍼로 시료를 재분산시키는 조치를 수행하기 전에 강력하게 권장된다. - 물을 사용하여 lyophilized 샘플을 다시 중단합니다. 물에서 1/10 샘플을 희석 (최종 농도 0.025 mg/mL).
- 일회용 접힌 모세관 세포(제타 잠재적 큐벳)를 여과 희석 매체로 미리 헹구어 불순물에서 완전히 청소합니다. 그런 다음 큐벳을 희석 된 나노 입자로 채우고 1 mL 주사기를 사용하여 필러의 양쪽을 캡하십시오.
참고: 나노 입자 분산은 커벳(~1mL)에서 사용할 수 있는 부피를 채우며, 거품의 형성에 특별한 주의를 기울여야 하며, 이는 대책을 왜곡할 수 있습니다. - DLS 내부의 샘플을 소개합니다. 셀이 올바르게 삽입되었는지 확인합니다.
- 제타 잠재 분석을 위해 만든 SOP 파일을 열고 원하는 샘플 이름을 소개합니다. 제타 전위 측정을선택합니다.
참고: 제타 전위 측정을 위한 SOP를 만드는 매개 변수는 표 2에서제공됩니다. - 재생을 클릭하여 DLS를 사용하여 제타 전위 측정을 실행합니다.
참고: 트리플 비프음 소리가 나면 분석이 완료됩니다. - 측정 시트의 샘플에 해당하는 결과를 선택하여 평균 제타 전위, 표준 편차 및 그래픽을 가져옵니다. 완료되면 DLS 장비에서 셀을 제거합니다.
- 샘플을 제거하고 필요한 경우 보관하십시오. 다음으로, 큐벳 세포를 탈온화된 물로 헹구고 에탄올과 물을 다시 헹구십시오. 마지막으로, 압축 공기 가스 스트림에서 청소 된 큐벳을 건조.
참고: 데이터를 분석하려면 권장 소프트웨어를 사용합니다.
- 앞서 설명한 바와 같이 mRNA와 pBAE를 완전히 혼합하는 나노입자를 0.25 mg/mL의 mRNA의 최종 농도로 폴리머 분획으로 배관하여(2단계, 폴리플렉스 형성)을 준비한다. 다음으로 용액을 동결하고 건조시십시오.
- 유체 역학 직경 (nm)
- 나노 입자 추적 분석 (NTA)
참고: 유체역학 직경(nm) 및 나노입자의 농도는 나노입자 추적 분석기를 사용하여 25°C, 488nm 레이저 파장에서 측정하였다(참조) 재료 테이블).- NTA 및 샘플 준비
- 컴퓨터와 장비를 켭다. 먼저 모든 구성 요소가 올바르게 연결되어 있는지 확인합니다. 다음으로 권장 소프트웨어를 엽니다. 소프트웨어는 모든 액세서리가 올바르게 연결되어 있는지 여부를 확인합니다.
- 상단 플레이트, 여기, O 링 셀을 레이저 모듈에 연결합니다.
참고: 이 부분을 과도하게 절개하지 마십시오. - 먼저 1mL 주사기로 완충 및/또는 탈이온된 물로 챔버를 로드합니다. 절차를 두 번 이상 반복합니다. 챔버에 거품을 도입하지 마십시오.
- DLS 크기 측정에 사용되는 농도로부터 1/1000을 탈화된 물에 희석하여 샘플을 준비합니다. 적어도 1mL를 준비하고 거품을 방출하지 않도록 1mL 주사기에 적재하십시오.
- 주사기를 사용하여 샘플을 챔버에 로드합니다. 그런 다음 레이저 모듈을 NTA 큰 챔버에 연결합니다.
참고: 큰 챔버는 측정을 위해 챔버가 배치되는 공간을 나타냅니다.
- 이미지 최적화
- 첫째, 하드웨어에서 카메라와 적절한 레이저가 선택되었는지 확인합니다.
참고 : 여기에, 하나의 카메라와 블루 레이저 488 nm가있다. - 카메라 레벨0으로 시작하여 캡처 창에서 카메라 시작을 누릅니다.
참고: 이 시점에서 다음 매개 변수를 조정합니다. 빔 위치(화면에서 위아래로 이동할 수 있음). 카메라 레벨: 과포화 픽셀을 피하십시오. 초점 (측면 휠): 가능한 한 더 나은 초점을 하려고합니다. 초점을 맞추지 않은 파티클 대신 후광이 있는 파티클을 갖는 것이 좋습니다. 농도: 필드당 10~100개의 입자가 있는 농도를 조정합니다.
- 첫째, 하드웨어에서 카메라와 적절한 레이저가 선택되었는지 확인합니다.
- 비디오 녹화
- 소프트웨어에서 측정 선택- SOP 창(왼쪽 아래)으로 이동하여 표준 측정을 선택합니다.
참고: 프로그램 (소프트웨어가 평균 및 표준 편차 계산을 수행 할 수 있도록) 각각 30 의 세 가지 측정을 수행할 수 있습니다. 샘플의 레코드를 저장하는 이름과 폴더 경로(기본 파일 이름에)를 제공합니다. SOP 매개 변수가 올바르게 설정되면 스크립트 만들기 및 실행을누릅니다. 첫 번째 복제를 측정하면 프로그램은 새 샘플을 추가하도록 요청합니다. 그런 다음 주사기플런저를 밀어 챔버에 시료의 또 다른 부분을 도입해야 합니다. 마지막으로 세 번째 복제를 분석하려면 세 번째 복제를 반복합니다.
- 소프트웨어에서 측정 선택- SOP 창(왼쪽 아래)으로 이동하여 표준 측정을 선택합니다.
- 비디오 처리
- 측정된 입자를 분석하기 위해 세 가지 측정값이 완료되면 화면 게인 및 감지 임계값을 재조정합니다. 이 시점에서 분석을 수행하기 위해 각 프레임에 약 100개의 파티클이 나타나야 합니다(높은 적색 십자가와 낮은 파란색 십자가가 예상됩니다).
- 분석이 완료된 후 결과를 PDF 파일로 내보냅니다. 또한, 비디오 및 엑셀 파일로 내보낼 수 있습니다.
- NTA 청소
참고: 다음 샘플을 연구하기 전에 열린 모든 측정값을 닫습니다. 생성된 파일은 엄청나고 컴퓨터에 따라 둘 이상의 열려 있는 것은 쉽지 않습니다.- NTA 챔버를 반복적으로 세척하여 물을 세척한 후 입자가 관찰되지 때까지 후속 측정을 수행합니다. 이어서, PBS(사용된 버퍼)를 플러시하여 측정값을 계속한다.
- 챔버 내부의 공기를 플러시하고 하루의 마지막 측정이 완료되면 미세한 학년 종이로 건조.
- 전송 전자 현미경 (TEM)에 의해 크기와 모양을 특성화. 샘플을 준비합니다. 최종 부피의 30 μL은 TEM 특성화에 충분합니다.
참고: 신선하고 lyophilized 둘 다 - 정지 후에- 나노 입자는 측정될 수 있습니다. - 탄소 코팅 구리 그리드에 10 μL의 샘플을 떨어뜨립니다. 10 분 동안 건조하십시오. 필터 용지를 부드럽게 눌러 필요한 경우 과도한 액체를 제거합니다.
- 음염색을 위해 우란 아세테이트 10 μL(2%w/v) 용액을 떨어뜨립니다. 1 분 동안 건조하십시오. 필터 용지를 부드럽게 눌러 필요한 경우 액체 과잉을 제거합니다.
- 현미경 및 스캔(전압 조작 80 kV)에 샘플을 소개합니다.
참고: 이미지의 추가 분석에 적합한 소프트웨어를 사용할 수 있습니다.
- NTA 및 샘플 준비
- 캡슐화 효율
- 샘플 준비
- 원하는 농도로 나노 입자를 준비하고 동결하고 건조시킵니다. 그런 다음 나노 입자를 다시 중단하고 6 μg /mL의 최종 농도로 희석하십시오.
- 20배 스톡 솔루션에서 1x TE 버퍼를 준비합니다.
참고: Vt (총 부피) = (샘플 수 x 100 μL x 4) (샘플 수 x 290 μL) + 2 mL. - 100 μg/μL의 주식에서 헤파린의 3 μg/μL로 TE 버퍼를 준비합니다.
참고 : Vt = 샘플 수 x 50 μL x 2. - 표준 준비
- 1x Tris-EDTA(TE)버퍼(표 S1A)에서0.2 μg/mL에서 0.025 μg/mL에 이르는 RNA 표준을 준비한다.
참고: 리보소말 RNA와 다른 경우 RNA 표준에 mRNA를 사용합니다. - Heparin(표 S1B)와RNA: pBAE 표준을 준비합니다. 헤파린표준(표 S1C)을준비합니다.
- 다음과 같이 96웰 플레이트를 준비합니다.
- 96웰 플레이트의 첫 번째 차선에 TE 버퍼 295 μL을 적재합니다(분석할 샘플만큼 많은 우물). 그런 다음 1x TE 버퍼의 50 μL을 차선 B 및 C(각 샘플에 대한 복제)에 로드합니다.
- 헤파린의 3μg/μL을 1x TBE 버퍼의 50 μL을 차선 D 및 E(각 샘플에 대한 복제)에 적재합니다. 다음으로 각 표준의 100 μL을 차선 F, G 및 H(각 표준에 대한 복제)에 로드합니다.
- 차선 A에 각 샘플의 5 μL을로드 (각 우물의 농도는 0.1 μg / mL입니다). 아래 4개의 우물에 각 샘플의 50 μL을 파이프팅하고 적재하여 적절히 섞는다(버퍼 1x TE 50 μL, 1x TE 버퍼 2개, 헤파린의 3μg/μL을 함유한 1x TE 버퍼 2개 포함).
- 37°C에서 30분 동안 시료를 배양한다. 제조업체의 프로토콜에 따라 RiboGreen 시약을 1x TE 버퍼로 준비합니다(재료 표참조).
참고: 희석 1:200 및 소용돌이. 빛으로부터 보호하십시오. Vt = 전체 우물 수 x 100 μL. - 리보그린 용액의 100 μL을 각 우물에 적재합니다. 500 nm의 난초 파장과 525 nm의 방출 파장을 가진 마이크로 플레이트 판독기를 사용하여 형광을 감지합니다.
- 결과 분석
- 세 가지 교정 곡선 준비
참고 : 첫째, 리보소말 RNA 표준. 이 보정 곡선에서 헤파린을 포함하지 않는 샘플을 보간합니다. 둘째, 헤파린과 mRNA:pBAE. 이 보정 곡선에서 헤파린이 있는 샘플을 보간합니다. 셋째, 헤파린. 작업 농도가 선형 범위에 있는지 확인하는 데 사용됩니다. - 다음과 같이 캡슐화 효율(EE%)을 계산합니다.
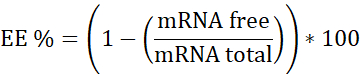
- 세 가지 교정 곡선 준비
- 샘플 준비
6. 시험관 내 특성화
- 질적 평가를 위한 형광 현미경 검사법
- 96웰 플레이트를 24시간 의 형질 전환 후 현미경에 놓습니다. 10배 의 목표로 셀 시각화를 시작합니다.
참고: HeLa 세포는 이 경우에 사용됩니다. 세포는 전송 모드(밝은 필드)를 사용하여 국소화되며, 이 때 초점을 조정해야 합니다.- 먼저 화이트 밸런스를 적용하여 샘플의 배경에 대한 소프트웨어를 참조합니다. 그런 다음 분석 중에 형광 영상과 오버레이하는 이미지를 습득한다.
- 현미경 모드를 반사 모드(형광)로 변경하고 필터 휠을 파란색 레이저(488 nm)로 이동하여 eGFP를 시각화합니다.
참고: 이 시점에서 노출 시간과 게인을 조정해야 합니다. 아티팩트와 배경 신호의 과잉을 피하기 위해 게인을 3에서 5 사이의 값으로 조정해야 합니다. 노출 시간의 조정은 (MS에서 1 s로) 형질 변환 효율에 따라 달라집니다. - 모든 샘플을 비교하기 위해 동일한 노출 시간을 사용하는 모든 조건 이나 우물에 대 한 이미지를 수집 합니다.
- 96웰 플레이트를 24시간 의 형질 전환 후 현미경에 놓습니다. 10배 의 목표로 셀 시각화를 시작합니다.
- 정량적 평가를 위한 유동 세포측정(FC)
- 멀티채널 파이펫을 사용하여 유동 세포 측정 실험을 위해 96웰 플레이트를 준비합니다.
참고: JAWSII 세포주에서와 같은 반 부착 셀로 작업할 때 모든 미디어 볼륨을 복구하고 다른 96웰 플레이트에 저장합니다. 그것은 전염 된 세포의 상당수를 포함 할 수 있기 때문에이 미디어에 열망 하지 마십시오. - 100 μL/웰 1x PBS로 세포를 청소하고 열망합니다. 다음으로, 트립신의 25 μL / 웰을 추가하고 5 분 동안 37 °C에서 배양.
- 세포가 분리되면 이전에 회수된 미디어를 추가하여 트립신 작용을 중지하고 세포를 수정하여 20분 동안 31.25 μL/웰의 포르말린 10%를 추가합니다(최종 농도 2.5%).
참고: 이 시점에서 세포의 분리를 보장하기 위해 현미경 검사법에 의해 세포의 상태를 시각화하는 것이 중요합니다. 세포가 분리되고 응집을 피하기 위해 여러 번 피펫을 위아래로 피펫하는 것이 좋습니다. 이렇게 하면 신뢰할 수 있는 결과가 생성됩니다. - 흐름 사이토미터와 소프트웨어를 켭니다. 소프트웨어에서 실험에 적합한 조건(플레이트 유형, 샘플 볼륨 및 흔들림, 샘플 간 헹구기 와 같은 기타 매개 변수)을 설정합니다.
참고: 6.2.5 단계의 경우 유량은 120 μL/min이어야 합니다. 1분마다 한 주기를 섞고 한 주기를 잘 헹구습니다. 혼합은 미디어에 분산된 세포를 유지하고 개별 세포의 더 나은 분석을 허용하는 데 필수적입니다. 또한, 헹구는 것은 시료 사이의 세포계의 미세유체계를 청소하는 데 필요합니다. 마지막으로 버퍼가 충분한지 여부와 모든 구성 요소가 올바르게 연결되어 있는지 확인합니다. - 양분형 세포의 백분율을 정량화하기 위해 적절한 매개변수를 설정합니다.
- 먼저, FSC 대 SCC(측면 산란광) 분산도에 대한 유동 데이터를 보고 세포를 파편으로부터 구별한다. 이어서, 진폭(FSC-A)과 높이(FSC-H)를 비교하는 또 다른 분산 플롯은 개별 세포를 게이트및 차별하도록 플롯된다.
참고: 처리되지 않은 인구는 긍정적으로 전감염된 세포에 대응하는 인구를 게이트할 수 있습니다. 이어서, 양해전환된 세포의 정량화는 히스토그램 플롯상에서 적절한 채널상에서 수행된다. 따라서, 양립형 세포는 히스토그램에 있는 사건의 연속체로 표현되어야 한다.
- 먼저, FSC 대 SCC(측면 산란광) 분산도에 대한 유동 데이터를 보고 세포를 파편으로부터 구별한다. 이어서, 진폭(FSC-A)과 높이(FSC-H)를 비교하는 또 다른 분산 플롯은 개별 세포를 게이트및 차별하도록 플롯된다.
- 멀티채널 파이펫을 사용하여 유동 세포 측정 실험을 위해 96웰 플레이트를 준비합니다.
7. 체외 기능 성 테스트 : 항원 모델 mRNA로 ovalbumin (OVA)를 사용하여 모델 면역 세포를 활성화 할 수있는 능력
- 플레이트 10,000 세포/우물 (JAWSII 세포는 여기에서 사용됩니다) 96 웰 플레이트에서 전날 형질 전환 하기 전에.
참고 : 각 조건에 대한 삼중 을 갖는 데 필요한 만큼 많은 우물을 접시. 세포가 적어도 12 시간 동안 접시에 부착하게하십시오 (하룻밤 잠복이 권장됩니다). - 아래 NOTE에 설명된 바와 같이 pBAEs NP를 준비하여 우물당 0.6 μg mRNA를 변형한다.
참고: 양수 조절으로서, 경질 시약을 사용하여 세포를 트랜스펙트한다. 여기서, 우리는 우물 당 0.1 μg mRNA를 사용하고 잘 당 형질 전환 시약의 0.25 μL을 사용했다. OVA용 mRNA 명문화(재료표참조)를 구입하였다. 5-메톡시우리딘으로 변형되고 포유류 시스템에 최적화되어 있으며, 공급업체의 독점 방법인 Cap1 구조로 엔드 캡핑을 통해 보호됩니다. - 37°C 및 5% CO2에서건조한 공기 인큐베이터에서 24시간 동안 96웰 플레이트를 배양한다.
참고: 이 시간 이후에는 특정 수의 셀이 포함될 수 있으므로 미디어를 갈망하지 마십시오. 세포를 복구하고 다른 우물이나 접시에 저장합니다. - 1x PBS의 25 μL로 우물에 남아 있는 세포를 씻으시다. 다음으로, 그것을 갈망하고 트립신의 25 μL을 추가하고 세포를 분리하기 위해 37 °C에서 5 분 동안 플레이트를 배양한다. 이전에 회수된 미디어를 특파원에게 잘 추가하여 트립신 반응을 중지하십시오.
- 4°C에서 5분 동안 400 x g의 플레이트원심분리기. 미디어를 갈망합니다. 1x PBS의 50 μL/웰과 포르말린의 2.5%를 추가합니다. 세포를 고치기 위해 20 분 동안 4 °C에서 배양하십시오.
- 7.5 및 7.5.1 단계를 반복합니다. 그런 다음 1x PBS의 50 μL/웰과 3% BSA(블로킹 버퍼)를 추가하고 4°C에서 30분 동안 배양합니다. 다시, 7.5 및 7.5.1 단계를 반복한 다음, 1차 항체(마우스 α-OVA)의 50 μL/웰을 1x PBS및 3% BSA에 넣고 4°C에서 30분 동안 배양한다.
- 7.5 및 7.5.1 단계를 반복한 다음 50 μL/웰1x PBS로 셀을 세척합니다. 1x PBS 및 3% BSA에 이차 항체 용액(α-마우스-AlexaFluor488/PerCP 및 Cy5.5-CD11b/APC-CD86)을 추가합니다. 4 °C에서 1 시간 동안 인큐베이션하십시오.
- 5 분 동안 400 x g에서 접시를 원심 분리합니다. 그런 다음, 1x PBS의 50 μL /웰로 세포를 씻는다. 다음으로, 원심분리기(5분 동안 400 x g), 갈망한 다음, 100 μL/웰100μL/웰의 1x PBS 및 2.5%의 서퍼민으로 세포를 다시 분리한다.
- 전술한 바와 같이 유동 세포측정에 의해 분석한다(6.2단계).
Access restricted. Please log in or start a trial to view this content.
결과
중합체 합성 및 특성화
OM-pBAE 합성 절차는 도 2에서주어진다. 도 2A에서 알 수 있듯이 OM-pBAE를 획득하는 첫 번째 단계는 아민(1-헥실라민 및 5-아미노-1-펜타놀, 비율 1:1)을 디아크릴레이트(1,4부티디올 디아크릴레이트)에 추가하여 C6-pBAE를 합성하는 것이다. 이 반응은 20 시간 동안 90 °C에서 수행되며 일정한 교반으로 수행됩니다. 그 후, 올?...
Access restricted. Please log in or start a trial to view this content.
토론
작년에 Covid-19 전염병이 발병한 후, 전염병 통제 측면에서 백신의 중요성은 중요한 구성 요소8로나타났다. 전 세계 과학자들의 노력으로 많은 백신 시장이 출시되었습니다. 역사상 처음으로 mRNA 백신은 몇달내에 새로운 항원5,6,21에적응할 수 있는 능력 때문에 빠른 설계 덕분에 이전에 가설적인 성공을 입증했...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 공개할 것이 없고 이해 상충도 없습니다.
감사의 말
마인코/FEDER의 재정 지원(SAF2015-64927-C2-2-R, RTI2018-094734-B-C22, COV20/01100)의 재정 지원이 인정됩니다. CGF는 그녀의 IQS 박사 학위 펠로우십을 인정했다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| 1,4-butanediol diacrylate | Sigma Aldrich | 123048 | |
| 1-hexylamine | Sigma Aldrich | 219703 | |
| 5-amino-1-pentanol | Sigma Aldrich | 411744 | |
| Acetone | Panreac | 141007 | |
| CD11b antibody | BD | 550993 | |
| CD86 antibody | Bioligend | 105007 | |
| Chlor hydroxhyde | Panreac | 181023 | |
| Chloroform-d | Sigma Aldrich | 151823 | |
| Cys-His-His-His peptide | Ontores | Custom | |
| Cys-Lys-Lys-Lys peptide | Ontores | Custom | |
| D2O | Sigma Aldrich | 151882 | |
| DEPC reagent for Rnase free water | Sigma Aldrich | D5758 | This reagent is important to treat MilliQ water to remove any RNases of the buffers |
| Diethyl eter | Panreac | 212770 | |
| dimethyl sulfoxide | Sigma Aldrich | 276855 | |
| HEPES | Sigma Aldrich | H3375 | |
| mRNA EGFP | TriLink Technologies | L-7601 | |
| mRNA OVA | TriLink Technologies | L-7610 | |
| RiboGreen kit | ThermoFisher | R11490 | |
| sodium acetate | Sigma Aldrich | 71196 | |
| sucrose | Sigma Aldrich | S0389 | |
| Trifluoroacetic acid | Sigma Aldrich | 302031 | |
| Trypsin-EDTA | Fisher Scientific | 11570626 | |
| α-mouse AlexaFluor488 antibody | Abcam | Ab450105 | |
| Equipment | |||
| Nanoparticle Tracking Analyzer | Malvern Panalytical | NanoSight NS300 | |
| Nuclear Magnetic Ressonance Spectrometer | Varian | 400 MHz | |
| ZetaSizer | Malvern Panalytical | Nano ZS | For zeta potential and hydrodynamic size determination |
| Software | |||
| NanoSight NTA software | Malvern Panalytical | MAN0515-02-EN-00 | |
| NovoExpress Software | Agilent | Not specified | |
| ZetaSizer software | Malvern Panalytical | DTS Application | To analyze surface charge and hydrodynamic sizes |
참고문헌
- Chumakov, K., Benn, C. S., Aaby, P., Kottilil, S., Gallo, R. Can existing live vaccines prevent COVID-19. Science. 368 (6496), 1187-1188 (2020).
- Zhang, C., Maruggi, G., Shan, H., Li, J. Advances in mRNA vaccines for infectious diseases. Frontiers in Immunology. 10, 1-13 (2019).
- Wherry, E. J., Jaffee, E. M., Warren, N., D'Souza, G., Ribas, A. How did we get a COVID-19 vaccine in less than 1 year. Clinical Cancer Research. 27 (8), 2136-2138 (2021).
- Folegatti, P. M., et al. Safety and immunogenicity of the ChAdOx1 nCoV-19 vaccine against SARS-CoV-2: a preliminary report of a phase 1/2, single-blind, randomised controlled trial. The Lancet. 396 (10249), 467-478 (2020).
- Geall, A. J., Mandl, C. W., Ulmer, J. B. RNA: The new revolution in nucleic acid vaccines. Seminars in Immunology. 25 (2), 152-159 (2013).
- Ulmer, J. B., Geall, A. J. Recent innovations in mRNA vaccines. Current Opinion in Immunology. 41, 18-22 (2016).
- Kranz, L. M., et al. Systemic RNA delivery to dendritic cells exploits antiviral defence for cancer immunotherapy. Nature. 534 (7607), 396-401 (2016).
- Milane, L., Amiji, M. Clinical approval of nanotechnology-based SARS-CoV-2 mRNA vaccines: impact on translational nanomedicine. Drug Delivery and Translational Research. 1 (4), 3(2020).
- Green, J. J., Langer, R., Anderson, D. G. A combinatorial polymer library approach yields insight into nonviral gene delivery. Accounts of Chemical Research. 41 (6), 749-759 (2008).
- Guerrero-Cázares, H., et al. Biodegradable polymeric nanoparticles show high efficacy and specificity at DNA delivery to human glioblastoma in vitro and in vivo. ACS Nano. 8 (5), 5141-5153 (2014).
- Kozielski, K. L., Tzeng, S. Y., Hurtado De Mendoza, B. A., Green, J. J. Bioreducible cationic polymer-based nanoparticles for efficient and environmentally triggered cytoplasmic siRNA delivery to primary human brain cancer cells. ACS Nano. 8 (4), 3232-3241 (2014).
- Segovia, N., Dosta, P., Cascante, A., Ramos, V., Borrós, S. Oligopeptide-terminated poly(β-amino ester)s for highly efficient gene delivery and intracellular localization. Acta Biomaterialia. 10 (5), 2147-2158 (2014).
- Dosta, P., Segovia, N., Cascante, A., Ramos, V., Borrós, S. Surface charge tunability as a powerful strategy to control electrostatic interaction for high efficiency silencing, using tailored oligopeptide- modified poly (beta-amino ester)s (PBAEs). Acta Biomaterialia. 20, 82-93 (2015).
- Fornaguera, C., et al. mRNA delivery system for targeting antigen-presenting cells in vivo. Advanced Healthcare Materials. 7 (17), 180033(2018).
- Fornaguera, C., Castells-Sala, C., Lázaro, M. Á, Cascante, A., Borrós, S. Development of an optimized freeze-drying protocol for OM-PBAE nucleic acid polyplexes. International Journal Pharmaceutics. 569, (2019).
- Fornaguera, C., Solans, C. Analytical methods to characterize and purify polymeric nanoparticles. International Journal of Polymer Science. , (2018).
- Fornaguera, C., Solans, C. Characterization of polymeric nanoparticle dispersions for biomedical applications: size, surface charge and stability. Pharmaceutical Nanotechnology. 6 (3), 147-164 (2018).
- Sahin, U., Karikó, K., Türeci, Ö MRNA-based therapeutics-developing a new class of drugs. Nature Reviews Drug Discovery. 13 (10), 759-780 (2014).
- Fan, Y. N., et al. Cationic lipid-assisted nanoparticles for delivery of mRNA cancer vaccine. Biomaterials Science. 6 (11), 3009-3018 (2018).
- Le Moignic, A., et al. Preclinical evaluation of mRNA trimannosylated lipopolyplexes as therapeutic cancer vaccines targeting dendritic cells. Journal of Controlled Release. 278, 110-121 (2018).
- Banerji, A., et al. mRNA vaccines to prevent COVID-19 disease and reported allergic reactions: Current evidence and approach. Journal of Allergy and Clinical Immunology: In Practice. 9 (4), 1423-1437 (2021).
- Kaczmarek, J. C., Kowalski, P. S., Anderson, D. G. Advances in the delivery of RNA therapeutics: from concept to clinical reality. Genome Medicine. 9 (1), 1-16 (2017).
- Dosta, P., et al. Delivery of anti-microRNA-712 to inflamed endothelial cells using poly(β-amino ester) nanoparticles conjugated with VCAM-1 targeting peptide. Advanced Healthcare Materials. , 1-11 (2021).
- Segovia, N., et al. Hydrogel doped with nanoparticles for local sustained release of siRNA in breast cancer. Advanced Healthcare Materials. 4 (2), 271-280 (2015).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유