Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Synthese und Charakterisierung von mRNA-beladenen Poly(beta-Aminoestern) Nanopartikeln für Impfzwecke
In diesem Artikel
Zusammenfassung
Hier wird ein einfaches Protokoll zur Herstellung von mRNA-Nanopartikeln auf Basis von Poly(beta-Aminoester)-Polymeren vorgestellt, das einfach durch Änderung der verkapselten mRNA angepasst werden kann. Der Workflow für die Synthese der Polymere, der Nanopartikel und deren in vitro essentielle Charakterisierung werden ebenfalls beschrieben. Ein Proof-of-Concept zur Immunisierung wird ebenfalls hinzugefügt.
Zusammenfassung
Die Impfung ist einer der größten Erfolge der modernen Gesellschaft und unverzichtbar bei der Kontrolle und Vorbeugung von Krankheiten. Traditionelle Impfstoffe bestanden aus ganz oder aus Bruchteilen des Infektionserregers. Es bleiben jedoch Herausforderungen bestehen, und neue Impfstofftechnologien sind obligatorisch. In diesem Zusammenhang hat die Verwendung von mRNA zu Immunisierungszwecken eine verbesserte Leistung gezeigt, wie die schnelle Zulassung von zwei mRNA-Impfstoffen zeigt, die eine SARS-CoV-2-Infektion verhindern. Neben dem Erfolg bei der Prävention von Virusinfektionen können mRNA-Impfstoffe auch für therapeutische Krebsanwendungen eingesetzt werden.
Dennoch macht die Instabilität der mRNA und ihre schnelle Clearance aus dem Körper aufgrund der Anwesenheit von Nukleasen ihre nackte Abgabe nicht möglich. In diesem Zusammenhang sind Nanoarzneimittel und insbesondere polymere Nanopartikel kritische mRNA-Abgabesysteme. Ziel dieses Artikels ist es daher, das Protokoll für die Formulierung und den Test eines mRNA-Impfstoffkandidaten auf Basis der proprietären polymeren Nanopartikel zu beschreiben. Die Synthese und chemische Charakterisierung der verwendeten Poly(beta-aminoester)-Polymere, ihre Komplexierung mit mRNA zu Nanopartikeln und ihre Lyophilisierungsmethodik werden hier diskutiert. Dies ist ein entscheidender Schritt zur Senkung der Lager- und Verteilungskosten. Schließlich werden die erforderlichen Tests zum Nachweis ihrer Fähigkeit, dendritische Modellzellen in vitro zu transfizieren und zu reifen, angezeigt. Dieses Protokoll wird der wissenschaftlichen Gemeinschaft, die an der Impfung arbeitet, aufgrund seiner hohen Vielseitigkeit zugute kommen, die es diesen Impfstoffen ermöglicht, eine Vielzahl von Krankheiten zu verhindern oder zu heilen.
Einleitung
Infektionskrankheiten stellen eine ernsthafte Bedrohung für Millionen von Menschen auf der ganzen Welt dar und sind in einigen Entwicklungsländern immer noch eine der häufigsten Todesursachen. Die prophylaktische Impfung war eine der wirksamsten Interventionen der modernen Gesellschaft zur Vorbeugung und Bekämpfung von Infektionskrankheiten1,2. Diese entscheidenden Meilensteine der Wissenschaft in der Relevanz des20.Jahrhunderts wurden durch die jüngste weltweite Covid-19-Pandemie, die durch das SARS-CoV-2-Virus verursacht wurde, hervorgehoben3. In Anerkennung der Bedeutung effizienter Impfstoffe, um die Verbreitung der Krankheit einzudämmen, haben die Kooperationsbemühungen aller biomedizinischen Gemeinschaften in weniger als einem Jahr erfolgreich zu vielen prophylaktischen Impfstoffen auf dem Markt geführt4.
Traditionell bestanden Impfstoffe aus abgeschwächten (lebend, reduzierte Virulenz) oder inaktivierten (Todespartikel) Viren. Für einige Krankheiten ohne Spielraum für Sicherheitsfehler sind virale Partikel jedoch nicht möglich, und stattdessen werden Proteinuntereinheiten verwendet. Dennoch ermöglichen Untereinheiten in der Regel nicht die Kombination von mehr als einem Epitop / Antigen, und Adjuvantien sind erforderlich, um die Impfkraft zu erhöhen5,6. Daher ist der Bedarf an neuartigen Impfstofftypen klar.
Wie während der aktuellen Pandemie gezeigt wurde, können neuartige Impfstoffkandidaten auf basis von Nukleinsäuren vorteilhaft sein, um lange Entwicklungsprozesse zu vermeiden und eine hohe Vielseitigkeit zu bieten und gleichzeitig eine lebenswichtige Immunisierung des Patienten zu erzeugen. Dies ist der Fall bei mRNA-Impfstoffen, die ursprünglich als experimentelle Krebsimpfstoffe konzipiert wurden. Dank ihrer natürlichen Fähigkeit, antigenspezifische T-Zell-Antworten zu produzieren3,5,6,7. Da mRNA das Molekül ist, das für das antigene Protein kodiert und sich nur gleich verändert, kann der Impfstoff schnell angepasst werden, um andere Varianten desselben Mikroorganismus, verschiedene Stämme, andere infektiöse Mikroorganismen zu immunisieren oder sogar eine Krebsimmuntherapie zu werden. Darüber hinaus sind sie vorteilhaft in Bezug auf die Produktionskosten im großen Maßstab. mRNA hat jedoch eine erhebliche Hürde, die ihre nackte Verabreichung behindert: Ihre Stabilität und Integrität sind in physiologischen Medien voller Nukleasen beeinträchtigt. Aus diesem Grund ist die Verwendung eines nanometrischen Trägers erforderlich, der sie schützt und mRNA zu den Antigen-präsentierenden Zellenvektorisiert 2,8.
In diesem Zusammenhang sind Poly(beta-Aminoester) (pBAE) eine Klasse von biokompatiblen und biologisch abbaubaren Polymeren, die dank ihrer kationischen Ladungen eine bemerkenswerte Fähigkeit gezeigt haben, mRNA in nanometrischen Partikeln zu komplexieren9,10,11. Diese Polymere bestehen aus Esterbindungen, was ihren Abbau durch Esterasen unter physiologischen Bedingungen erleichtert. Unter den pBAE-Bibliothekskandidaten zeigten diejenigen, die mit endkationischen Oligopeptiden funktionalisiert wurden, eine höhere Fähigkeit, kleine Nanopartikel zu bilden, um durch Endozytose effizient in Zellen einzudringen und das verkapselte Genmaterial zu transfizieren. Darüber hinaus ermöglicht die Versauerung des Endosomenkompartiments dank ihrer Pufferkapazität ein endosomales Entweichen12,13. Nämlich eine spezifische Art von pBAE, einschließlich hydrophober Einheiten auf ihrem Rückgrat (die sogenannte c6 pBAE), um ihre Stabilität zu verbessern, und eine End-Oligopeptid-Kombination (60% des polymeren modifizierten Polymers mit einem Tri-Lysin und 40% des Polymers mit einem Tri-Histidin), die selektiv Antigen-präsentierende Zellen nach parenteraler Verabreichung transfiziert und die mRNA-kodierte Antigenpräsentation gefolgt von einer Mäuse-Immunisierung erzeugt, wurde kürzlich veröffentlicht14 . Darüber hinaus wurde auch gezeigt, dass diese Formulierungen einen der Hauptengpässe von Nanomedizinformulierungen umgehen könnten: die Möglichkeit, sie zu gefriertrocknen, ohne ihre Funktionalität zu verlieren, was eine Langzeitstabilität in weichen, trockenen Umgebungen ermöglicht15.
In diesem Zusammenhang besteht das Ziel des aktuellen Protokolls darin, das Verfahren zur Bildung der mRNA-Nanopartikel der wissenschaftlichen Gemeinschaft zur Verfügung zu stellen, indem eine Beschreibung der kritischen Schritte im Protokoll gegeben und die Herstellung effizienter Impfstoffe für die Prävention von Infektionskrankheiten und Tumorbehandlungsanwendungen ermöglicht wird.
Das folgende Protokoll beschreibt das komplette Training zur Synthese von Oligopeptid-endmodifizierten Poly(beta-Aminoestern) - OM-pBAE-Polymeren, die weiterhin für die Nanopartikelsynthese verwendet werden. Im Protokoll ist auch die Formulierung von Nanopartikeln enthalten. Darüber hinaus werden kritische Schritte für den Erfolg des Verfahrens und repräsentative Ergebnisse bereitgestellt, um sicherzustellen, dass die resultierenden Formulierungen die erforderlichen Qualitätskontrollcharakterisierungsmerkmale erfüllen, um ein positives oder negatives Ergebnis zu definieren. Dieses Protokoll ist in Abbildung 1zusammengefasst.
Protokoll
1. Synthese von pBAE-Polymer mit Endoligopeptiden (OM-pBAE)
- Polymerisation von C6-pBAE
- 5-Amino-1-pentanol (38 mmol; MW = 103,16 Da) 1-Hexylamin (38 mmol; MW = 101,19 Da) in einen Glaskolben mit rundem Boden (100 ml). Dann fügen Sie 1,4-Butandioldiacrylat (82 mmol; MW = 198,22 Da).
- Das Silikonölbad bei 90 °C vorheizen, den Rundkolben in das Ölbad stellen und die Mischung mit Hilfe eines Magnetrührbalkens über Nacht (~18 h) umrühren. Nehmen Sie dann das Produkt aus dem Rundkolben und legen Sie es bei -20 °C in den Gefrierschrank.
HINWEIS: Das Produkt liegt in Form eines klebrigen Pulvers vor und wird mit Hilfe eines Spatels aus dem Kolben entnommen. Es ist wichtig, die Struktur des erhaltenen Polymers durch 1H-NMR zu validieren. NMR-Spektren wurden in einem 400-MHz-Instrument (siehe Materialtabelle)unter Verwendung von Chloroform-d undD2Oals Lösungsmittel aufgezeichnet. Etwa 10 mg jedes Poly(β-Aminoesters) wurden entnommen und in 1 ml des deuterierten Lösungsmittels gelöst.
- Reaktion mit Peptiden zur Gewinnung von OM-pBAE
- Geben Sie 25 ml 0,1 M HCl zu ausgewählten Peptiden, z. B. dem Peptid Cys-His-His-His mit Trifluoressigsäure (TFA, 200 mg), in ein zuvor gewogenes 50-ml-Zentrifugenröhrchen, das der Gefriertrocknung standhält und über Nacht manuell umgerührt wird, um eine klare Lösung zu erhalten.
- Die Lösung bei -80 °C für 1 h einfrieren und das resultierende Peptidhydrochlorid gefriertrocknen.
HINWEIS: Prüfen Sie, ob das Endgewicht dem theoretischen Wert entspricht. - Eine Lösung von C6-pBAE (0,031 mmol) in Dimethylsulfoxid herstellen. Machen Sie auch eine Lösung des Peptidhydrochlorids (0,078 mmol) in Dimethylsulfoxid.
- Mischen Sie die beiden Lösungen in einem Schraubverschlussrohr und schrauben Sie den Deckel auf. Rühren Sie die Mischungslösung in einem Wasserbad mit einer kontrollierten Temperatur von 25 °C für 20 h mit einem magnetischen Rührstab.
- Das Gemisch wird zu 7:3 (v/v) Diethylether/Aceton gegeben. Die resultierende Suspension wird bei 25.000 x g bei 4 °C zentrifugiert, um das Lösungsmittel zu entfernen. Als nächstes waschen Sie den Feststoff zweimal mit 7:3 (v/v) Diethylether/Aceton. Trocknen Sie das Produkt dann unter Vakuum (<0,2 atm).
- Machen Sie eine Lösung von 100 mg / ml des Produkts in Dimethylsulfoxid. Das resultierende Produkt heißt C6-peptide-pBAE. Es ist wichtig, die Struktur des erhaltenen Polymers durch 1H-NMR zu validieren, um das Verschwinden der Olefinsignale zu bestätigen, die mit terminalen Acrylaten assoziiert sind. Bei Nichtgebrauch kann das Polymer bei -20 °C eingefroren werden.
2. Bildung von Polyplexen
HINWEIS: Alle Verfahren sollten in einem klimatisierten Raum durchgeführt werden, um eine konstante Temperatur aufrechtzuerhalten.
- Tauen Sie die Polymere C6-Peptid-pBAE auf und wirbeln Sie die Lösung.
- Das Polymergemisch wird nach oben und unten pipettiert und eine Lösung von 12,5 mM(V1)in Natriumacetat (NaAc) hergestellt. Dann wirbeln Sie die Mischung und warten Sie 10 Minuten.
- Die mRNA bei 0,5 mg/ml herstellen und durch Pipettieren mischen(V2).
HINWEIS: Es ist wichtig, ein Wirbeln der mRNA zu vermeiden. - Wirbeln Sie die Polymermischung in der Endkonzentration, um eine homogene Lösung zwischen dem Polymermaterial in DMSO und dem Acetatpuffer zu erhalten.
HINWEIS: Die endgültige Polymerkonzentration hängt vom gewählten N/P-Verhältnis (Stickstoff-Phosphat-Gruppen) ab. Das N/P-Verhältnis hängt von jeder spezifischen zu verwendenden mRNA ab. Für die eGFP-Kapselung wurde beispielsweise ein Verhältnis von 25:1 verwendet, wie zuvor berichtet14. - Mischen Sie die genetische Materiallösung und die C6-PeptidpBAE-Lösung (25x der mRNA-Konzentration) im Verhältnis 1:1 (Vi =V1 +V2).
HINWEIS: C6-Peptid-pBAE wird in ein Mikrozentrifugenröhrchen geladen, in dem die mRNA durch Pipettieren zum Mischen nach oben und unten hinzugefügt wird. Nach der Herstellung werden der Polyplex, die Nukleinsäure und die C6-Peptid-pBAE-Konzentrationen halb verdünnt. - Bei 25 °C für 30 min in einem Thermoblock inkubieren. Mit 1:2 RNase-freiem Wasser ausfällen, indem die Probe in ein vorbespanntes Mikrozentrifugenröhrchen mit Wasser gegeben wird.
- Schließen Sie die Hilfsstoffe ein. Fügen Sie das gleiche Volumen wie die Mischung aus mRNA und pBAE (Vi) in HEPES 20 mM und Saccharose 4% durch Pipettieren auf und ab hinzu. Zu diesem Zeitpunkt wurde die Probe 3x verdünnt.
3. Polyplexe Lyophilisation
- Sofort bei -80 °C Gefrierschrank der vorherigen Polyplexlösung für 1 h einfrieren.
- Führen Sie die Primärtrocknung durch, indem Sie die folgenden Schritte ausführen: (1) 1 h bei -60 °C und 0,001 hPa; (2) 1 h bei -40 °C und 0,0001 hPa; (3) 4 h bei -20 °C und 0,0001 hPa; (4) 12 h bei 5 °C und 0,0001 hPa.
- Sofort bei -20 °C lagern, um eine Rehydrierung bis zur Anwendung zu vermeiden.
4. Polyplex-Resuspension
HINWEIS: Dieses Protokoll beschreibt den Prozess, der zur Rekonstruktion der lyophilisierten C6-Peptid-pBAE-Nanopartikel für ihre weitere Verwendung entweder zur Charakterisierung, in vitro oder zur In-vivo-Analyse verwendet wird.
- Nehmen Sie die lyophilisierten Nanopartikel ab -20 °C gerade zum Zeitpunkt der Verwendung und fügen Sie schnell die entsprechende Menge depyrogeniertes (DEPC) Wasser hinzu, um den Feststoff erneut zu dispergieren, um die gewünschte Konzentration zu erreichen.
HINWEIS: Das Volumen entspricht dem Anfangsvolumen der Nanopartikel, wenn die gleiche Konzentration vorgesehen ist, wird jedoch für jedes Experiment angegeben. - Sanft pipettieren, bis die Flüssigkeit vollständig resuspensioniert ist, wobei die Flüssigkeit mit der Pipette einhergeht.
HINWEIS: Stellen Sie sicher, dass kein Material an der Wand der Durchstechflasche verbleibt. - Einmal aufgelöst, pipettieren Sie kräftig auf und ab, um Blasen zu vermeiden.
HINWEIS: Die Probe sollte einen transparenten bis transluzenten Aspekt aufweisen, der bei höheren Konzentrationen deutlicher ist. - Lagern Sie die Proben auf Eis oder bei 4 °C für einen Zeitraum von maximal 24 h nach der Rekonstitution. Vermeiden Sie das Einfrieren.
5. Polyplex-Charakterisierung
- Dynamische Lichtstreuung
HINWEIS: Der hydrodynamische Durchmesser (nm), der Polydispersitätsindex (PDI) und die Oberflächenladung von Nanopartikeln wurden bei 25 °C, 633 nm Laserwellenlänge und 173° Signaldetektor mit einem Zeta-Potentialanalysator gemessen (siehe Materialtabelle).- Hydrodynamischer Durchmesser (nm)
- Bereiten Sie die Nanopartikel, die mRNA und pBAE mischen, gründlich vor, indem Sie das Genmaterial in die Polymerfraktion auf eine endgültige mRNA-Konzentration von 0,25 mg/ml pipettieren, wie oben beschrieben (Schritt 2).
- Spülen Sie eine Mikrocuvette mit gefilterten Verdünnungsmedien vor, um sie vollständig von Verunreinigungen zu reinigen. Füllen Sie dann die Küvette mit mindestens 50 μL der Probenlösung und verschließen Sie sie. Als nächstes führen Sie die Probe in das DLS-Instrument ein und stellen Sie sicher, dass die Zelle korrekt eingefügt ist.
HINWEIS: Das Verdünnungsmedium wird mit einem 0,22 μm Spritzenfilter filtriert. - Öffnen Sie die erstellte SOP-Datei (Standard Operation Procedure), und geben Sie den gewünschten Beispielnamen ein. Wählen Sie Größenmessungaus.
HINWEIS: Alle Parameter zum Erstellen einer SOP für Größenmessungen sind in Tabelle 1 aufgeführt. - Führen Sie die Partikelgrößenmessung mit DLS durch, indem Sie auf Wiedergabeklicken.
HINWEIS: Nachdem der dreifache Signalton ertönt, ist die Analyse abgeschlossen. - Wählen Sie die Ergebnisse aus, die der Probe auf dem Messblatt entsprechen, um die durchschnittliche Partikelgröße, den durchschnittlichen PDI, die Standardabweichung und die Grafiken zu erhalten. Sobald Sie fertig sind, entfernen Sie die Zelle aus dlS-Geräten.
- Entfernen Sie die Probe, bewahren Sie sie für die Zetapotenzialanalyse auf und reinigen und spülen Sie die Zelle mit entionisiertem Wasser ab. Trocknen Sie schließlich die gereinigte Küvette unter einem Druckluftgasstrom.
- Oberflächenladung (mV)
- Bereiten Sie die Nanopartikel, die mRNA und pBAE mischen, gründlich vor, indem Sie das Genmaterial auf die Polymerfraktion pipettieren, bis zu einer endgültigen mRNA-Konzentration von 0,25 mg/ml, wie oben beschrieben (Schritt 2, Polyplexbildung). Als nächstes die Lösung einfrieren und trocknen.
ANMERKUNG: In diesem Fall wird für die Messung der Oberflächenladung dringend empfohlen, die Probe gefriertrocknen zu lassen, bevor die Maßnahmen zur erneuten Reduktion der Probe im geeigneten Puffer durchgeführt werden, wobei die Körperbedingungen von pH-Wert und Elektrolytkonzentration simuliert werden. - Resuspendieren Sie die lyophilisierte Probe mit Wasser. Verdünnen Sie die Probe 1/10 in Wasser (Endkonzentration 0,025 mg/ml).
- Spülen Sie eine gefaltete Einweg-Kapillarzelle (Zetapotentialküvette) mit gefilterten Verdünnungsmedien vor, um sie vollständig von Verunreinigungen zu reinigen. Füllen Sie dann die Küvette mit verdünnten Nanopartikeln mit einer 1-ml-Spritze und verschließen Sie beide Seiten der Füllstoffe.
HINWEIS: Die Nanopartikeldispersion muss das in der Küvette verfügbare Volumen (~ 1 ml) füllen und dabei besonders auf die Bildung der Blase achten, die die Messungen stören könnte. - Führen Sie das Beispiel in DLS ein. Stellen Sie sicher, dass die Zelle korrekt eingefügt ist.
- Öffnen Sie die für die Zetapotenzialanalyse erstellte SOP-Datei und geben Sie den gewünschten Beispielnamen ein. Wählen Sie Zeta-Potentialmessung.
ANMERKUNG: Die Parameter zum Erstellen einer SOP für Zetapotentialmessungen sind in Tabelle 2 aufgeführt. - Führen Sie die Zetapotenzialmessung mit DLS durch, indem Sie auf Wiedergabeklicken.
HINWEIS: Nach dem dreifachen Signalton ist die Analyse abgeschlossen. - Wählen Sie die Ergebnisse aus, die der Probe auf dem Messblatt entsprechen, um das durchschnittliche Zetapotential, die Standardabweichung und die Grafiken zu erhalten. Wenn Sie fertig sind, entfernen Sie die Zelle aus dem DLS-Gerät.
- Entfernen Sie die Probe und bewahren Sie sie bei Bedarf auf. Als nächstes reinigen und spülen Sie die Küvettenzelle mit entionisiertem Wasser, gefolgt von Ethanol und erneutem Wasser. Trocknen Sie schließlich die gereinigte Küvette unter einem Druckluftgasstrom.
HINWEIS: Um die Daten zu analysieren, verwenden Sie die empfohlene Software.
- Bereiten Sie die Nanopartikel, die mRNA und pBAE mischen, gründlich vor, indem Sie das Genmaterial auf die Polymerfraktion pipettieren, bis zu einer endgültigen mRNA-Konzentration von 0,25 mg/ml, wie oben beschrieben (Schritt 2, Polyplexbildung). Als nächstes die Lösung einfrieren und trocknen.
- Hydrodynamischer Durchmesser (nm)
- Nanopartikel-Tracking-Analyse (NTA)
HINWEIS: Der hydrodynamische Durchmesser (nm) und die Konzentration von Nanopartikeln wurden bei 25 °C, 488 nm Laserwellenlänge mit einem Nanopartikel-Tracking-Analysator gemessen (siehe Tabelle der Materialien).- NTA und Probenvorbereitung
- Schalten Sie den Computer und die Ausrüstung ein. Überprüfen Sie zunächst, ob alle Komponenten korrekt angeschlossen sind. Öffnen Sie als Nächstes die empfohlene Software. Die Software prüft, ob das gesamte Zubehör korrekt angeschlossen ist.
- Verbinden Sie die obere Platte, hier die O-Ring-Zelle, mit dem Lasermodul.
HINWEIS: Schrauben Sie dieses Teil nicht über. - Beladen Sie die Kammer zunächst mit Puffer und/oder entionisiertem Wasser mit einer 1 ml Spritze. Wiederholen Sie den Vorgang mindestens zweimal. Vermeiden Sie das Einbringen von Blasen in die Kammer.
- Bereiten Sie die Probe vor, indem Sie 1/1000 in deionisiertem Wasser aus der bei der DLS-Größenmessung verwendeten Konzentration verdünnen. Bereiten Sie mindestens 1 ml vor und laden Sie es in eine 1 ml Spritze, um das Einbringen von Blasen zu vermeiden.
- Laden Sie die Proben mit einer Spritze in die Kammer. Stecken Sie dann das Lasermodul in die große NTA-Kammer.
HINWEIS: Große Kammer bezeichnet den Raum, in dem die Kammer für die Messung platziert ist.
- Bildoptimierung
- Überprüfen Sie zunächst in der Hardware,ob die Kamera und der richtige Laser ausgewählt sind.
HINWEIS: Hier gibt es nur eine Kamera und den Blue Laser 488 nm. - Beginnen Sie mit der Kameraebene bei 0 und drücken Sie im Aufnahmefenster auf Kamera starten.
HINWEIS: Passen Sie an dieser Stelle die folgenden Parameter an. Strahlposition (es kann auf dem Bildschirm auf und ab bewegt werden). Kamera-Level:Vermeiden Sie übersättigte Pixel. Fokus (seitliches Rad):Versuchen Sie, so besser wie möglich zu fokussieren. Es ist besser, Partikel mit einem Halo anstelle von unfokussierten Partikeln zu haben. Konzentration: Stellen Sie die Konzentration auf 10 bis 100 Partikel pro Feld ein.
- Überprüfen Sie zunächst in der Hardware,ob die Kamera und der richtige Laser ausgewählt sind.
- Videoaufzeichnung
- Gehen Sie in der Software zum Fenster Messauswahl- SOP (unten links) und wählen Sie Standardmessung.
HINWEIS: Programm zur Durchführung von drei Messungen von jeweils 30 s (damit die Software Mittelwert- und Standardabweichungsberechnungen durchführen kann). Geben Sie einen Namen und einen Ordnerpfad (unter Basisdateiname) an, um die Datensätze des Beispiels zu speichern. Wenn die SOP-Parameter korrekt eingerichtet sind, drücken Sie Skript erstellen und ausführen. Sobald die erste Replikation gemessen wurde, fordert das Programm Sie auf, eine neue Probe hinzuzufügen. Dann muss ein weiterer Bruchteil der Probe in die Kammer eingeführt werden, indem der Kolben der Spritze gedrückt wird. Wiederholen Sie schließlich ein drittes Mal, um das dritte Replikat zu analysieren.
- Gehen Sie in der Software zum Fenster Messauswahl- SOP (unten links) und wählen Sie Standardmessung.
- Videoverarbeitung
- Passen Sie die Bildschirmverstärkung und die Erkennungsschwelle neu an, sobald die drei Messungen abgeschlossen sind, um die gemessenen Partikel zu analysieren. In diesem Moment sollten etwa 100 Partikel in jedem Frame erscheinen, um die Analyse durchzuführen (hohe rote Kreuze und niedrige blaue Kreuze werden erwartet).
- Exportieren Sie die Ergebnisse in eine PDF-Datei, nachdem die Analyse abgeschlossen ist. Darüber hinaus ist es möglich, als Videos und Excel-Dateien zu exportieren.
- Reinigung der NTA
HINWEIS: Schließen Sie alle geöffneten Messungen, bevor Sie die folgende Probe untersuchen. Da die generierten Dateien enorm sind und je nach Computer ist es nicht einfach, mehr als eine offen zu halten.- Reinigen Sie die NTA-Kammer durch wiederholtes Spülen von Wasser, bevor Sie die anschließende Messung durchführen, bis keine Partikel mehr beobachtet werden. Anschließend spülen Sie den verwendeten Puffer (PBS), um mit den Maßnahmen fortzufahren.
- Spülen Sie Luft in die Kammer und trocknen Sie sie mit mikroskopisch kleinem Papier, sobald die letzte Messung des Tages abgeschlossen ist.
- Charakterisieren Sie die Größe und Form durch Transmission Electronic Microscopy (TEM). Bereiten Sie die Proben vor. 30 μL des Endvolumens reichen für die TEM-Charakterisierung aus.
HINWEIS: Sowohl frische als auch lyophilisierte - nach Resuspension - nanopartikel können gemessen werden. - 10 μL Probe auf ein kohlenstoffbeschichtetes Kupfergitter fallen lassen. 10 Min. trocknen lassen. Entfernen Sie die überschüssige Flüssigkeit, falls erforderlich, indem Sie sanft auf Filterpapier klopfen.
- Tropfen Sie 10 μL Uranylacetat (2% w/v) Lösung für negative Färbung. 1 Min. trocknen lassen. Entfernen Sie den Flüssigkeitsüberschuss gegebenenfalls durch sanftes Klopfen auf Filterpapier.
- Die Probe in das Mikroskop einführen und scannen (Spannungsbetrieb 80 kV).
HINWEIS: Für die weitere Analyse der Bilder kann eine entsprechende Software verwendet werden.
- NTA und Probenvorbereitung
- Verkapselungseffizienz
- Probenvorbereitung
- Bereiten Sie die Nanopartikel in der gewünschten Konzentration vor und frieren und trocknen Sie sie ein. Anschließend werden die Nanopartikel resuspend und in einer Endkonzentration von 6 μg/ml verdünnt.
- Bereiten Sie 1x TE-Puffer aus der 20x-Standardlösung vor.
ANMERKUNG: Vt (Gesamtvolumen) = (Anzahl der Proben x 100 μL x 4) (Anzahl der Proben x 290 μL) + 2 mL. - Te-Puffer mit 3 μg/μL Heparin aus dem Vorrat von 100 μg/μL herstellen.
ANMERKUNG: Vt = Anzahl der Proben x 50 μL x 2. - Vorbereiten der Standards
- Stellen Sie einen RNA-Standard im Bereich von 0,2 μg/ml bis 0,025 μg/ml in 1x Tris-EDTA (TE)-Puffer her (Tabelle S1A).
HINWEIS: Verwenden Sie mRNA im RNA-Standard, falls dies von der ribosomalen RNA abweicht. - RNA:pBAE-Standard mit Heparin herstellen (Tabelle S1B). Heparin-Standard vorbereiten (Tabelle S1C).
- Bereiten Sie einen 96-Well-Teller wie folgt vor.
- Laden Sie 295 μL TE-Puffer in die erste Spur einer 96-Well-Platte (so viele Wells wie zu analysierende Proben). Laden Sie dann 50 μL 1x TE-Puffer in die Bahnen B und C (Duplikate für jede Probe).
- Laden Sie 50 μL 1x FSME-Puffer mit 3 μg/μL Heparin in die Bahnen D und E (Duplikate für jede Probe). Als nächstes laden Sie 100 μL jedes Standards in die Lanes F, G und H (Duplikate für jeden Standard).
- Belastung von 5 μL jeder Probe in Bahn A (Konzentration in jeder Vertiefung beträgt 0,1 μg/ml). Mischen Sie richtig durch Pipettieren und laden Sie 50 μL jeder Probe auf die vier Vertiefungen unten (zwei von ihnen enthalten 50 μL Puffer 1x TE und zwei weitere enthalten 1x TE-Puffer mit 3 μg / μL Heparin).
- Die Probe wird 30 min bei 37 °C inkubiert. Bereiten Sie das RiboGreen-Reagenz gemäß dem Protokoll des Herstellers (siehe Materialtabelle)in 1x TE-Puffer vor.
HINWEIS: Verdünnen Sie 1:200 und Wirbel. Vor Licht schützen. Vt = Anzahl der Vollbohrungen x 100 μL. - Laden Sie 100 μL RiboGreen-Lösung in jede Vertiefung. Detektieren Sie Fluoreszenz mit dem Mikroplattenleser mit einer Anregungswellenlänge von 500 nm und einer Emissionswellenlänge von 525 nm.
- Ergebnisanalyse
- Vorbereiten der drei Kalibrierkurven
HINWEIS: Erstens, ribosomaler RNA-Standard. Interpolieren Sie in dieser Kalibrierkurve die Proben, die kein Heparin enthalten. Zweitens, mRNA:pBAE mit Heparin. Interpolieren Sie in dieser Kalibrierkurve die Probe, die Heparin enthält. Drittens, Heparin. Wird verwendet, um sicherzustellen, dass die Arbeitskonzentration im linearen Bereich liegt. - Berechnen Sie die Verkapselungseffizienz (EE%) wie folgt:
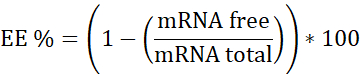
- Vorbereiten der drei Kalibrierkurven
- Probenvorbereitung
6. In-vitro-Charakterisierung
- Fluoreszenzmikroskopie zur qualitativen Beurteilung
- Legen Sie die 96-Well-Platte nach 24 h Transfektion auf das Mikroskop. Beginnen Sie mit der Visualisierung von Zellen mit dem 10-fachen Ziel.
HINWEIS: In diesem Fall werden HeLa-Zellen verwendet. Die Zellen werden über den Übertragungsmodus (Helles Feld) lokalisiert, und der Fokus muss zu diesem Zeitpunkt angepasst werden.- Wenden Sie zunächst einen Weißabgleich an, um die Software über den Hintergrund der Beispiele zu referenzieren. Erfassen Sie dann ein Bild, um es während der Analyse mit dem Fluoreszenzbild zu überlagern.
- Schalten Sie den Mikroskopmodus in den Reflexionsmodus (Fluoreszenz) und bewegen Sie das Filterrad zum blauen Laser (488 nm), um eGFP zu visualisieren.
HINWEIS: An dieser Stelle müssen die Belichtungszeit und die Verstärkung angepasst werden. Die Verstärkung muss in Werten zwischen 3 und 5 eingestellt werden, um Artefakte und Überschuss am Hintergrundsignal zu vermeiden. Die Einstellung der Belichtungszeit hängt von der Transfektionseffizienz (von ms bis 1 s) ab. - Erfassen Sie Bilder für alle Bedingungen oder Vertiefungen mit der gleichen Belichtungszeit, um alle Proben zu vergleichen.
- Legen Sie die 96-Well-Platte nach 24 h Transfektion auf das Mikroskop. Beginnen Sie mit der Visualisierung von Zellen mit dem 10-fachen Ziel.
- Durchflusszytometrie (FC) zur quantitativen Beurteilung
- Verwenden Sie eine Mehrkanalpipette, um die 96-Well-Platte für das Durchflusszytometrie-Experiment vorzubereiten.
HINWEIS: Wenn Sie mit semi-adhärenten Zellen arbeiten, z. B. in der JAWSII-Zelllinie, stellen Sie das gesamte Medienvolumen wieder her und speichern Sie es in einer anderen 96-Well-Platte. Streben Sie dieses Medium nicht an, da es eine beträchtliche Anzahl von transfizierten Zellen enthalten kann. - Reinigen Sie die Zellen (Hier werden HeLa-Zellen verwendet) mit 100 μL/Well 1x PBS und streben Sie danach. Als nächstes 25 μL/Well Trypsin hinzufügen und bei 37 °C für 5 min inkubieren.
- Sobald die Zellen abgelöst sind, fügen Sie zuvor wiederhergestellte Medien hinzu, um die Trypsinwirkung zu stoppen und die Zellen zu fixieren, indem Sie 31,25 μL / Well Formalin 10% für 20 min hinzufügen (Endkonzentration 2,5%).
HINWEIS: An dieser Stelle ist es wichtig, den Zustand der Zellen durch Mikroskopie sichtbar zu machen, um die Ablösung der Zellen sicherzustellen. Es wird dringend empfohlen, mehrmals auf und ab zu pipettieren, um den Zellen zu helfen, sich zu lösen und ihre Agglomeration zu vermeiden. Dies führt zu einem zuverlässigen Ergebnis. - Schalten Sie das Durchflusszytometer und die Software ein. Richten Sie in der Software die richtigen Bedingungen für das Experiment ein (Art der Platte, Probenvolumen und andere Parameter wie Schütteln, Spülen zwischen den Proben).
HINWEIS: Für Schritt 6.2.5 sollte der Durchfluss 120 μL/min betragen. Mischen Sie alle 1 Minute einen Zyklus und spülen Sie einen Zyklus gut ab. Das Mischen ist unerlässlich, um die im Medium dispergierten Zellen zu erhalten und eine bessere Analyse einzelner Zellen zu ermöglichen. Darüber hinaus ist eine Spülung erforderlich, um die Mikrofluidik des Zytometers zwischen den Proben zu reinigen. Prüfen Sie abschließend, ob genügend Puffer vorhanden sind und ob alle Komponenten korrekt angeschlossen sind. - Richten Sie die entsprechenden Parameter ein, um den Prozentsatz der positiv transfizierten Zellen zu quantifizieren.
- Zeigen Sie zunächst die Strömungsdaten auf (Forward Scattered Light) FSC versus SCC (Side Scattered Light) Streudiagramm an, um die Zellen von den Trümmern zu unterscheiden. Dann wird ein weiteres Streudiagramm, das die Amplitude (FSC-A) mit der Höhe (FSC-H) vergleicht, gezeichnet, um einzelne Zellen zu gateen und zu unterscheiden.
HINWEIS: Die unbehandelte Population ermöglicht es, die Populationen entsprechend positiv transfizierten Zellen zu schützen. Dann wird die Quantifizierung der positiv transfizierten Zellen auf dem richtigen Kanal auf einem Histogrammdiagramm durchgeführt. Daher müssen die positiv transfizierten Zellen als Kontinuum von Ereignissen auf dem Histogramm dargestellt werden.
- Zeigen Sie zunächst die Strömungsdaten auf (Forward Scattered Light) FSC versus SCC (Side Scattered Light) Streudiagramm an, um die Zellen von den Trümmern zu unterscheiden. Dann wird ein weiteres Streudiagramm, das die Amplitude (FSC-A) mit der Höhe (FSC-H) vergleicht, gezeichnet, um einzelne Zellen zu gateen und zu unterscheiden.
- Verwenden Sie eine Mehrkanalpipette, um die 96-Well-Platte für das Durchflusszytometrie-Experiment vorzubereiten.
7. In-vitro-Funktionstests: Fähigkeit, Modellimmunzellen durch Verwendung von Ovalbumin (OVA) als antigene Modell-mRNA zu aktivieren
- Platte 10.000 Zellen / Well (JAWSII-Zellen werden hier verwendet) in einer 96-Well-Platte am Tag vor der Transfektion.
HINWEIS: Platten Sie so viele Vertiefungen, wie für jede Bedingung erforderlich sind, um Triplikate zu haben. Lassen Sie die Zellen mindestens 12 h an der Platte haften (eine Inkubation über Nacht wird empfohlen). - Bereiten Sie pBAEs-NPs wie in der anmerkung unten beschrieben vor, um 0,6 μg mRNA pro Vertiefung zu transfizieren.
HINWEIS: Als Positivkontrolle transfizieren Sie die Zellen mit einem Transfektionsreagenz. Hier verwendeten wir 0,1 μg mRNA pro Vertiefung und 0,25 μL des Transfektionsreagenzes pro Vertiefung. mRNA-Kodifizierung für OVA wurde gekauft (siehe Tabelle der Materialien). Es ist polyadenyliert, mit 5-Methoxyuridin modifiziert und für Säugetiersysteme optimiert und wird durch eine Endkappe mit Cap1-Struktur, einer proprietären Methode des Lieferanten, geschützt. - Inkubieren Sie die 96-Well-Platte für 24 h in einem Trockenluftinkubator bei 37 °C und 5%CO2.
HINWEIS: Streben Sie nach dieser Zeit nicht nach den Medien, da sie eine bestimmte Anzahl von Zellen enthalten können. Stellen Sie die Zellen wieder her und speichern Sie sie in einem anderen Brunnen oder einer anderen Platte. - Waschen Sie die auf dem Bohrloch verbleibenden Zellen mit 25 μL 1x PBS. Als nächstes streben Sie es an und fügen Sie 25 μL Trypsin hinzu und inkubieren Sie die Platte für 5 min bei 37 ° C, um die Zellen zu lösen. Stoppen Sie die Trypsin-Reaktion, indem Sie die zuvor wiederhergestellten Medien in den Korrespondentenbrunnen geben.
- Zentrifugieren Sie die Platte bei 400 x g für 5 min bei 4 °C. Streben Sie nach den Medien. Fügen Sie 50 μL / Well eines 1x PBS und 2,5% Formalin hinzu. Inkubieren Sie es bei 4 °C für 20 min, um die Zellen zu fixieren.
- Wiederholen Sie die Schritte 7.5 und 7.5.1. Dann 50 μL/Well 1x PBS und 3% BSA (Blocking Buffer) zugeben und 30 min bei 4 °C inkubieren. Wiederholen Sie erneut die Schritte 7.5 und 7.5.1 und fügen Sie dann 50 μL/Well des primären Antikörpers (Maus-α-OVA) in 1x PBS und 3% BSA hinzu und inkubieren Sie für 30 min bei 4 °C.
- Wiederholen Sie die Schritte 7.5 und 7.5.1 und waschen Sie dann die Zellen mit 50 μL/Well 1x PBS. Aspire-Medien und fügen Sie dann sekundäre Antikörperlösung (α-Maus-AlexaFluor488 / PerCP und Cy5.5-CD11b / APC-CD86) in 1x PBS und 3% BSA hinzu. Inkubieren Sie es für 1 h bei 4 °C.
- Zentrifugieren Sie die Platte bei 400 x g für 5 min. Dann waschen Sie die Zellen mit 50 μL / Well von 1x PBS. Als nächstes zentrifugieren (400 x g für 5 min), streben sie an und resuspendieren Sie dann die Zellen in 100 μL / Well von 1x PBS und 2,5% Formalin.
- Analysieren Sie es mittels Durchflusszytometrie, wie oben beschrieben (Schritt 6.2).
Ergebnisse
Polymersynthese und -charakterisierung
Das OM-pBAE-Syntheseverfahren ist in Abbildung 2 dargestellt. Wie Abbildung 2A zeigt, besteht der erste Schritt zur Gewinnung des OM-pBAE darin, das C6-pBAE durch Zugabe der Amine (1-Hexylamin und 5-Amino-1-pentanol, Verhältnis 1:1) zum Diakrylat (1,4-Butandioldiacrylat) zu synthetisieren. Diese Reaktion wird bei 90 °C für 20 h und unter ständigem Rühren durchgeführt. Danach wird eine Lösung von O...
Diskussion
Nach dem Ausbruch der Covid-19-Pandemie im vergangenen Jahr hat sich die Bedeutung von Impfstoffen für die Bekämpfung von Infektionskrankheiten als kritische Komponente manifestiert8. Bemühungen von Wissenschaftlern weltweit haben die Markteinführung vieler Impfstoffe ermöglicht. Zum ersten Mal in der Geschichte haben mRNA-Impfstoffe ihren bisher hypothetischen Erfolg gezeigt, dank ihres schnellen Designs aufgrund ihrer Fähigkeit, sich innerhalb einiger Monate an jedes neue Antigen anzupasse...
Offenlegungen
Autoren haben weder preiszugeben noch Interessenkonflikte.
Danksagungen
Finanzielle Unterstützung von MINECO/FEDER (Zuschüsse SAF2015-64927-C2-2-R, RTI2018-094734-B-C22 und COV20/01100) wird anerkannt. CGF erkannte ihr IQS PhD Fellowship an.
Materialien
| Name | Company | Catalog Number | Comments |
| 1,4-butanediol diacrylate | Sigma Aldrich | 123048 | |
| 1-hexylamine | Sigma Aldrich | 219703 | |
| 5-amino-1-pentanol | Sigma Aldrich | 411744 | |
| Acetone | Panreac | 141007 | |
| CD11b antibody | BD | 550993 | |
| CD86 antibody | Bioligend | 105007 | |
| Chlor hydroxhyde | Panreac | 181023 | |
| Chloroform-d | Sigma Aldrich | 151823 | |
| Cys-His-His-His peptide | Ontores | Custom | |
| Cys-Lys-Lys-Lys peptide | Ontores | Custom | |
| D2O | Sigma Aldrich | 151882 | |
| DEPC reagent for Rnase free water | Sigma Aldrich | D5758 | This reagent is important to treat MilliQ water to remove any RNases of the buffers |
| Diethyl eter | Panreac | 212770 | |
| dimethyl sulfoxide | Sigma Aldrich | 276855 | |
| HEPES | Sigma Aldrich | H3375 | |
| mRNA EGFP | TriLink Technologies | L-7601 | |
| mRNA OVA | TriLink Technologies | L-7610 | |
| RiboGreen kit | ThermoFisher | R11490 | |
| sodium acetate | Sigma Aldrich | 71196 | |
| sucrose | Sigma Aldrich | S0389 | |
| Trifluoroacetic acid | Sigma Aldrich | 302031 | |
| Trypsin-EDTA | Fisher Scientific | 11570626 | |
| α-mouse AlexaFluor488 antibody | Abcam | Ab450105 | |
| Equipment | |||
| Nanoparticle Tracking Analyzer | Malvern Panalytical | NanoSight NS300 | |
| Nuclear Magnetic Ressonance Spectrometer | Varian | 400 MHz | |
| ZetaSizer | Malvern Panalytical | Nano ZS | For zeta potential and hydrodynamic size determination |
| Software | |||
| NanoSight NTA software | Malvern Panalytical | MAN0515-02-EN-00 | |
| NovoExpress Software | Agilent | Not specified | |
| ZetaSizer software | Malvern Panalytical | DTS Application | To analyze surface charge and hydrodynamic sizes |
Referenzen
- Chumakov, K., Benn, C. S., Aaby, P., Kottilil, S., Gallo, R. Can existing live vaccines prevent COVID-19. Science. 368 (6496), 1187-1188 (2020).
- Zhang, C., Maruggi, G., Shan, H., Li, J. Advances in mRNA vaccines for infectious diseases. Frontiers in Immunology. 10, 1-13 (2019).
- Wherry, E. J., Jaffee, E. M., Warren, N., D'Souza, G., Ribas, A. How did we get a COVID-19 vaccine in less than 1 year. Clinical Cancer Research. 27 (8), 2136-2138 (2021).
- Folegatti, P. M., et al. Safety and immunogenicity of the ChAdOx1 nCoV-19 vaccine against SARS-CoV-2: a preliminary report of a phase 1/2, single-blind, randomised controlled trial. The Lancet. 396 (10249), 467-478 (2020).
- Geall, A. J., Mandl, C. W., Ulmer, J. B. RNA: The new revolution in nucleic acid vaccines. Seminars in Immunology. 25 (2), 152-159 (2013).
- Ulmer, J. B., Geall, A. J. Recent innovations in mRNA vaccines. Current Opinion in Immunology. 41, 18-22 (2016).
- Kranz, L. M., et al. Systemic RNA delivery to dendritic cells exploits antiviral defence for cancer immunotherapy. Nature. 534 (7607), 396-401 (2016).
- Milane, L., Amiji, M. Clinical approval of nanotechnology-based SARS-CoV-2 mRNA vaccines: impact on translational nanomedicine. Drug Delivery and Translational Research. 1 (4), 3 (2020).
- Green, J. J., Langer, R., Anderson, D. G. A combinatorial polymer library approach yields insight into nonviral gene delivery. Accounts of Chemical Research. 41 (6), 749-759 (2008).
- Guerrero-Cázares, H., et al. Biodegradable polymeric nanoparticles show high efficacy and specificity at DNA delivery to human glioblastoma in vitro and in vivo. ACS Nano. 8 (5), 5141-5153 (2014).
- Kozielski, K. L., Tzeng, S. Y., Hurtado De Mendoza, B. A., Green, J. J. Bioreducible cationic polymer-based nanoparticles for efficient and environmentally triggered cytoplasmic siRNA delivery to primary human brain cancer cells. ACS Nano. 8 (4), 3232-3241 (2014).
- Segovia, N., Dosta, P., Cascante, A., Ramos, V., Borrós, S. Oligopeptide-terminated poly(β-amino ester)s for highly efficient gene delivery and intracellular localization. Acta Biomaterialia. 10 (5), 2147-2158 (2014).
- Dosta, P., Segovia, N., Cascante, A., Ramos, V., Borrós, S. Surface charge tunability as a powerful strategy to control electrostatic interaction for high efficiency silencing, using tailored oligopeptide- modified poly (beta-amino ester)s (PBAEs). Acta Biomaterialia. 20, 82-93 (2015).
- Fornaguera, C., et al. mRNA delivery system for targeting antigen-presenting cells in vivo. Advanced Healthcare Materials. 7 (17), 180033 (2018).
- Fornaguera, C., Castells-Sala, C., Lázaro, M. &. #. 1. 9. 3. ;., Cascante, A., Borrós, S. Development of an optimized freeze-drying protocol for OM-PBAE nucleic acid polyplexes. International Journal Pharmaceutics. 569, (2019).
- Fornaguera, C., Solans, C. Analytical methods to characterize and purify polymeric nanoparticles. International Journal of Polymer Science. , (2018).
- Fornaguera, C., Solans, C. Characterization of polymeric nanoparticle dispersions for biomedical applications: size, surface charge and stability. Pharmaceutical Nanotechnology. 6 (3), 147-164 (2018).
- Sahin, U., Karikó, K., Türeci, &. #. 2. 1. 4. ;. MRNA-based therapeutics-developing a new class of drugs. Nature Reviews Drug Discovery. 13 (10), 759-780 (2014).
- Fan, Y. N., et al. Cationic lipid-assisted nanoparticles for delivery of mRNA cancer vaccine. Biomaterials Science. 6 (11), 3009-3018 (2018).
- Le Moignic, A., et al. Preclinical evaluation of mRNA trimannosylated lipopolyplexes as therapeutic cancer vaccines targeting dendritic cells. Journal of Controlled Release. 278, 110-121 (2018).
- Banerji, A., et al. mRNA vaccines to prevent COVID-19 disease and reported allergic reactions: Current evidence and approach. Journal of Allergy and Clinical Immunology: In Practice. 9 (4), 1423-1437 (2021).
- Kaczmarek, J. C., Kowalski, P. S., Anderson, D. G. Advances in the delivery of RNA therapeutics: from concept to clinical reality. Genome Medicine. 9 (1), 1-16 (2017).
- Dosta, P., et al. Delivery of anti-microRNA-712 to inflamed endothelial cells using poly(β-amino ester) nanoparticles conjugated with VCAM-1 targeting peptide. Advanced Healthcare Materials. , 1-11 (2021).
- Segovia, N., et al. Hydrogel doped with nanoparticles for local sustained release of siRNA in breast cancer. Advanced Healthcare Materials. 4 (2), 271-280 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten