Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un test de chimiotaxie amélioré pour l’identification rapide des chimioattractifs rhizobactériens dans les exsudats racinaires
Dans cet article
Résumé
Ici, nous présentons un protocole de test de chimiotaxie amélioré. L’objectif de ce protocole est de réduire les étapes et les coûts des méthodes traditionnelles de chimiotaxie bactérienne et de servir de ressource précieuse pour comprendre les interactions plantes-microbes.
Résumé
L’identification de la chimiotaxie est très importante pour la recherche et l’application des bactéries favorisant la croissance de la rhizosphère. Nous avons établi une méthode simple pour identifier rapidement les chimioattractifs qui pourraient induire le mouvement chimiotactique des bactéries favorisant la croissance de la rhizosphère sur des lames de verre stériles par des étapes simples. Une solution bactérienne (OD600 = 0,5) et une solution aqueuse chimioattractante stérile ont été ajoutées goutte à goutte sur la lame de verre à un intervalle de 1 cm. Une boucle d’inoculation a été utilisée pour connecter la solution aqueuse chimioattractante à la solution bactérienne. La glissière a été maintenue à température ambiante pendant 20 minutes sur le banc propre. Enfin, la solution aqueuse chimioattractante a été collectée pour le comptage bactérien et l’observation microscopique. Dans cette étude, grâce à de multiples comparaisons de résultats expérimentaux, la méthode a surmonté de multiples lacunes des méthodes traditionnelles de chimiotaxie bactérienne. La méthode a réduit l’erreur de comptage des plaques et raccourci le cycle expérimental. Pour l’identification des substances chimioattractantes, cette nouvelle méthode permet d’économiser 2-3 jours par rapport à la méthode traditionnelle. De plus, cette méthode permet à tout chercheur de compléter systématiquement une expérience de chimiotaxie bactérienne en 1-2 jours. Le protocole peut être considéré comme une ressource précieuse pour comprendre les interactions plantes-microbes.
Introduction
La chimiotaxie est importante pour la colonisation des rhizobactéries favorisant la croissance des plantes (PGPR) sur les racines et pour comprendre les interactions plantes-microbes1. Une classe de composés de faible poids moléculaire (chimioattractifs) dans les exsudats racinaires des plantes induit le mouvement chimiotactique du PGPR vers la rhizosphère2. L’acide malique, l’acide citrique et d’autres composants des exsudats racinaires stimulent la chimiotaxie des souches de Bacillus3. Par exemple, le glucose, l’acide citrique et l’acide fumarique dans les exsudats de racines de maïs recrutent des bactéries à la surface des racines4. Le D-galactose, qui est dérivé des exsudats racinaires, induit la chimiotaxie de Bacillus velezensis SQR95. Les acides organiques, y compris le fumarate, l’acide malique et le succinate, influencent la chimiotaxie et la colonisation de divers PGPR dans le système de culture intercalaire Cajanus cajan - Zea mays6. L’acide oléanolique dans les exsudats de racine de riz agit comme chimioattractif pour la souche FP357. D’autres exsudats végétaux (y compris l’histidine, l’arginine et l’aspartate) peuvent jouer un rôle crucial dans la réponse chimiotactique des bactéries8. Les exsudats végétaux fonctionnent comme un signal pour diriger le mouvement des bactéries, ce qui est la première étape de la colonisation de la rhizosphère. La colonisation des plantes par le PGPR est un processus d’une énorme pertinence, car les PGPR sont bénéfiques pour l’hôte de la plante.
De nombreuses méthodes ont été utilisées pour analyser la chimiotaxie bactérienne. La méthode de la plaque de natation est l’une des méthodes décrites précédemment9. Dans cette méthode, les plaques ont été fabriquées avec un milieu semi-solide. Un tampon chimiotactique contenant de la gélose (1,0 %, p/v) a été ajouté à la plaque. Le tampon est chauffé, puis mélangé avec le chimioattractif. Ensuite, 8 μL de suspension bactérienne ont été ajoutés goutte à goutte au milieu de la plaque et la plaque a été placée dans un incubateur à 28 ° C. La plaque était régulièrement observée et photographiée. Cependant, le cycle expérimental de la méthode de la plaque de natation a été très long. Dans la méthode capillaire10, une pointe de pipette sert de chambre pour contenir 100 μL de suspension bactérienne. Une aiguille de seringue de 1 mL a été utilisée comme capillaire. Une aiguille de seringue contenant des chimioattractifs avec différents gradients de concentration a été insérée dans l’embout de la pipette de 100 μL. Après incubation à température ambiante pendant 3 h, l’aiguille de la seringue a été retirée, le contenu a été dilué et plaqué sur le milieu. L’accumulation bactérienne dans la seringue était représentée par des unités formant des colonies (UFC) dans les plaques. Cependant, l’erreur expérimentale dans les répétitions pour la méthode capillaire était importante. Une autre méthode utilisait un dispositif microfluidique SlipChip11. En bref, la solution d’albumine sérique bovine (BSA) a été injectée dans tous les canaux et éliminée sous vide. Les solutions contenant différents chimioattractifs (concentration de 1 mM pour la détection qualitative uniquement), des cellules bactériennes en suspension dans une solution saline tamponnée au phosphate et un tampon salin tamponné au phosphate (contrôle négatif) ont été ajoutées aux micropuits supérieur, moyen et inférieur, respectivement. L’incubation a ensuite été réalisée dans un environnement sombre à température ambiante pendant 30 min. Les cellules bactériennes ont ensuite été détectées dans les micropuits. Le dispositif microfluidique SlipChip, cependant, était coûteux. Par conséquent, chacune des méthodes décrites ci-dessus présentait des avantages et des inconvénients.
Nous avons établi un test de chimiotaxie amélioré pour l’identification rapide des chimioattractifs rhizobactériens dans les exsudats racinaires à l’aide de lames de verre stériles sans étapes compliquées. Dans cette étude, grâce à de multiples comparaisons de résultats expérimentaux, la méthode a surmonté de multiples lacunes des méthodes traditionnelles de chimiotaxie bactérienne. La méthode a réduit l’erreur de comptage des plaques et raccourci le cycle expérimental. Par conséquent, si elle est utilisée pour identifier une substance chimioattractante, cette nouvelle méthode peut économiser 2-3 jours et réduire le coût des matériaux expérimentaux.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Matériaux et équipements
NOTE: Bacillus altitudinis LZP02 (CP075052) a été isolé de la rhizosphère du riz dans le nord-est de la Chine12,13 pour cette étude.
- Culture B. altitudinis LZP02 dans le milieu Luria-Bertani (LB) (peptone, 10 g de L-1; NaCl, 8 g L-1 et extrait de levure, 5 g L-1) pendant 10 h. Prélever les cellules par centrifugation à 9 569 x g pendant 2 min à 4 °C. et les conserver avec 15 % de glycérol à -80 °C.
NOTE: Pour cette expérience, des graines de riz (Oryza sativa Longgeng 46) ont été fournies par l’Institut de recherche sur le riz de l’Académie des sciences agricoles du Heilongjiang.
2. Collecte des exsudats racinaires
- Distribuer au hasard les graines de riz dans une chambre de croissance.
REMARQUE: Les graines de riz ont été stérilisées avec 30% de H2O2 pendant 30 minutes et trempées dans l’eau pendant la nuit. Les conditions étaient les suivantes : la lumière contrôlée (cycle lumière/obscurité de 16/8 h), la température (22 ± 2 °C) et l’humidité relative (͂70 %). - Cultivez des graines de riz pendant une semaine et ajoutez de l’eau stérile deux fois.
- Sélectionner des plants de riz de taille et de plantation similaires dans 50 mL de milieu liquide Murashige et Skoog (MS). Incuber pendant 48 h à 22 °C dans des conditions aseptiques.
REMARQUE: Les exsudats de racine de riz seront libérés dans le milieu MS14,15,16.
3. Chromatographie liquide-spectrométrie de masse analyse des exsudats racinaires
- Prélever 100 μL de l’échantillon (milieu MS contenant des exsudats racinaires) dans un tube centrifuge de 1,5 mL. Ajouter 20 μL du solvant d’extraction (acétonitrile-méthanol-eau, 2:2:1, y compris l’étalon interne). Homogénéiser l’échantillon à 45 Hz pendant 4 min, suivi d’une échographie sur glace pendant 5 min au bain-marie.
- Répétez le cycle d’homogénéisation et d’ultrasons trois fois. Incuber l’échantillon à -20 °C pendant 1 h, suivi d’une centrifugation à 133 778 x g et à 4 °C pendant 15 min.
- Transférer le surnageant résultant dans des flacons LC-MS et conserver à -80 °C jusqu’à l’analyse UHPLC-QE. Préparer les échantillons de contrôle de la qualité (CQ) en mélangeant des portions égales du surnageant de tous les échantillons.
REMARQUE: Chaque volume d’échantillon était de 600 μL (six répétitions par expérience) dans l’expérience présentée. - Effectuer une analyse LC-MS/MS à l’aide du système UHPLC, de la colonne UPLC HSS T3 (2,1 mm x 100 mm, 1,8 μm) et de Q Exactive12.
- Utiliser une solution aqueuse d’acide formique à 0,1 % et une solution aqueuse d’acétate d’ammonium à 5 mmol/L en phase mobile A et en acétonitrile en phase B mobile. Utiliser l’acide formique et l’acétate d’ammonium comme modes ioniques positifs et négatifs, respectivement.
- Réglez le gradient d’élution comme suit : 0 min, 1 % B ; 1 min, 1% B; 8 min, 99% B. 10 min, 99% B. 10,1 min, 1% B; 12 min, 1 % B. Réglez le débit et le volume d’injection à 0,5 mL/min et 2 μL, respectivement.
REMARQUE : La macromolécule (> 1 000 Daltons) ne peut pas être détectée.
4. Dosage de la chimiotaxie
- Préparer la solution aqueuse chimioattractante. Assurez-vous qu’il est stérile. Filtrer la solution chimioattractante avec un filtre bactérien de 0,22 μm.
NOTE: La solution aqueuse chimioattractante était la substance unique obtenue à partir des études LC-MS dissoute dans l’eau. La concentration et le volume peuvent être ajustés de manière appropriée en fonction de différentes études. La solution d’acide citrique a été utilisée comme exemple. Toutes les actions doivent être effectuées sur le côté d’une lampe. - Marquez la position médiane de la glissière de verre à un intervalle de 1 cm. Assurez-vous que la lame de verre est stérilisée plusieurs fois sur la flamme.
- Ajouter les 30 μL de solution chimioattractante sur la gauche de la lame de verre. S’assurer que les bactéries ont été cultivées au stade logarithmique (2 x 108 UFC/mL) dans un milieu LB. Ajouter 30 μL de la solution bactérienne à droite de la lame de verre.
REMARQUE: Préparez un groupe témoin négatif avec un volume égal d’eau stérile. La solution saline (NaCI à 0,9%) a été utilisée comme témoin positif afin d’éliminer les changements causés par la force intermoléculaire sur l’expérience. - Stériliser une boucle d’inoculation plusieurs fois sur la flamme. Utilisez la boucle d’inoculation pour connecter la solution aqueuse chimioattractante à la solution bactérienne et maintenez-la à température ambiante pendant 20 minutes sur un banc propre.
REMARQUE: L’expérience doit être réalisée dans un environnement sans vent. Ajustez l’heure avant de déconnecter la ligne de connexion pour les souches de différentes capacités athlétiques de manière appropriée. - Après 20 min, débranchez la ligne de connexion avec du papier filtre.
- Assurez-vous que le tube de centrifugeuse de 1,5 mL est stérile. Recueillir la solution aqueuse chimioattractante à gauche de la lame de verre. Transférer la solution dans le tube de centrifugeuse stérile de 1,5 mL.
- Ajouter le volume approprié de safranine dans le tube de centrifugeuse. Après 2 min, prélever les liquides miscibles pour compter les bactéries et l’observation microscopique avec une chambre de comptage sanguin.
5. Analyse des résultats
- Déterminez le nombre de micro-organismes viables attirés à l’aide de l’équation suivante :
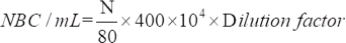
où NBC: Nombre total de cellules bactériennes; N : Nombre de bactéries dans 80 grilles.
REMARQUE : Un logiciel d’analyse statistique a été utilisé pour l’analyse des données. L’erreur était basée sur trois valeurs expérimentales répliquées différentes et a été calculée à l’aide d’une ANOVA unidirectionnelle suivie de l’analyse post-hoc de la Turquie. P ≤ 0,05 a été jugé significatif.
Access restricted. Please log in or start a trial to view this content.
Résultats
Au total, 584 et 937 métabolites connus ont été détectés dans les indices d’ions positifs et négatifs, respectivement. Des études antérieures ont montré que les chimioattractifs sont généralement des acides organiques, des acides aminés et des glucides17,18.
Dans cette étude, 16 types de chimioattractifs provenant des études LC-MS dans les exsudats de la rhizosphère du riz ont été sélectionnés pour des expériences...
Access restricted. Please log in or start a trial to view this content.
Discussion
De plus en plus de recherches indiquent que les interactions plantes-bactéries se produisent principalement dans la rhizosphère et sont influencées par les exsudats racinaires20,21,22,23,24. Les exsudats de racines végétales comprennent un large éventail de métabolites primaires, y compris des acides phénoliques, des acides organiques et des acides ami...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu par la Fondation nationale des sciences naturelles de Chine (n° 31870493), les principaux projets de recherche et de développement du Heilongjiang, en Chine (GA21B007), et les frais de recherche fondamentale des universités de la province du Heilongjiang, en Chine (n° 135409103).
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 2,5-dihydroxybenzoic acid | Beijing InnoChem Science & Technology C.,Ltd. | 490-79-9 | |
| Acetonitrile | CNW Technologies | 75-05-8 | |
| Ammonium acetate | CNW Technologies | 631-61-8 | |
| Caffeic acid | Beijing InnoChem Science & Technology C.,Ltd. | 331-39-5 | |
| Centrifuge | Thermo Fisher Scientific | Heraeus Fresco17 | |
| Citric acid | Beijing InnoChem Science & Technology C.,Ltd. | 77-92-9 | |
| Clean bench | Shanghai Boxun Industrial Co., Ltd. | BJ-CD | |
| Ferulic acid | Beijing InnoChem Science & Technology C.,Ltd. | 1135-24-6 | |
| Formic acid | CNW Technologies | 64-18-6 | |
| Fructose | Beijing InnoChem Science & Technology C.,Ltd. | 57-48-7 | |
| Galactose | Beijing InnoChem Science & Technology C.,Ltd. | 59-23-4 | |
| Glycine | Beijing InnoChem Science & Technology C.,Ltd. | 56-40-6 | |
| Grinding Mill | Shanghai Jingxin Industrial Development Co., Ltd. | JXFSTPRP-24 | |
| Histidine | Beijing InnoChem Science & Technology C.,Ltd. | 71-00-1 | |
| Internal standard: 2-Chloro-L-phenylalanine | Shanghai Hengbai Biotech C.,Ltd. | 103616-89-3 | |
| Leucine | Beijing InnoChem Science & Technology C.,Ltd. | 61-90-5 | |
| Malic acid | Beijing InnoChem Science & Technology C.,Ltd. | 6915-15-7 | |
| Mannose | Beijing InnoChem Science & Technology C.,Ltd. | 3458-28-4 | |
| Mass Spectrometer | Thermo Fisher Scientific | Q Exactive Focus | |
| Methanol | CNW Technologies | 67-56-1 | |
| Optical Microscope | Olympus | BX43 | |
| Phenylalanine | Beijing InnoChem Science & Technology C.,Ltd. | 63-91-2 | |
| Proline | Beijing InnoChem Science & Technology C.,Ltd. | 147-85-3 | |
| Scales | Sartorius | BSA124S-CW | |
| Serine | Beijing InnoChem Science & Technology C.,Ltd. | 56-45-1 | |
| Threonine | Beijing InnoChem Science & Technology C.,Ltd. | 72-19-5 | |
| UHPLC | Agilent | 1290 UHPLC | |
| Ultrasound Instrument | Shenzhen Leidebang Electronics Co., Ltd. | PS-60AL | |
| Valine | Beijing InnoChem Science & Technology C.,Ltd. | 7004-03-7 |
Références
- Belas, R. Biofilms, flagella, and mechanosensing of surfaces by bacteria. Trends in Microbiology. 22 (9), 517-527 (2014).
- Haichar, Z., Santaella, C., Heulin, T., Achouak, W. Root exudates mediated interactions belowground. Soil Biology and Biochemistry. 77 (7), 69-80 (2014).
- Zhang, N., et al. Effects of different plant root exudates and their organic acid components on chemotaxis, biofilm formation and colonization by beneficial rhizosphere-associated bacterial strains. Plant and Soil. 374 (1-2), 689-700 (2014).
- Zhang, N., et al. Whole transcriptomic analysis of the plant-beneficial rhizobacterium Bacillus amyloliquefaciens SQR9 during enhanced biofilm formation regulated by maize root exudates. BMC Genomics. 16 (1), 685(2015).
- Lui, Y., et al. Induced root-secreted D-galactose functions as a chemoattractant and enhances the biofilm formation of Bacillus velezensis SQR9 in an mcpa-dependent manner. Applied Microbiology and Biotechnology. 104 (17), 785-797 (2020).
- Vora, S. M., Joshi, P., Belwalkar, M., Archana, G. Root exudates influence chemotaxis and colonization of diverse plant growth-promoting rhizobacteria in the Cajanus cajan - Zea mays intercropping system. Rhizosphere. 18 (12), 100331(2021).
- Sampedro, I., et al. Effects of halophyte root exudates and their components on chemotaxis, biofilm formation and colonization of the halophilic bacterium halomonas anticariensis FP35T. Microorganisms. 8 (4), 575(2020).
- Liu, X. L., Raza, W., Ma, J. H., Huang, Q. W., Shen, Q. R. A dual role amino acid from sesbania rostrata seed exudates in the chemotaxis response of Azorhizobium caulinodans ORS571. Molecular Plant-Microbe Interactions. 32 (9), 1134-1147 (2019).
- Ling, N., Raza, W., Ma, J. H., Huang, Q. W., Shen, Q. R. Identification and role of organic acids in watermelon root exudates for recruiting Paenibacillus polymyxa SQR-21 in the rhizosphere. European Journal of Soil Biology. 47 (6), 374-379 (2011).
- Rudrappa, T., Czymmek, K. J., Pare, P. W., Bais, H. P. Root-secreted malic acid recruits beneficial soil bacteria. Plant Physiology. 148 (3), 1547-1556 (2008).
- Shen, C., et al. Bacterial chemotaxis on Slipchip. Lab on a Chip. 14 (16), 3074-3080 (2014).
- Liu, H., et al. Bacillus pumilus LZP02 promotes rice root growth by improving carbohydrate metabolism and phenylpropanoid biosynthesis. Molecular Plant-Microbe Interactions. 33 (10), 1222-1231 (2020).
- Goswami, M., Deka, S. Isolation of a novel rhizobacteria having multiple plant growth promoting traits and antifungal activity against certain phytopathogens. Microbiological Research. 240, 126516(2020).
- Kaiira, M., Chemining'Wa, G., Ayuke, F., Baguma, Y., Nganga, F. Profiles of compounds in root exudates of rice, cymbopogon, desmodium, mucuna and maize. Journal of Agricultural Sciences Belgrade. 64 (4), 399-412 (2019).
- Shi, Y., et al. Effect of rice root exudates and strain combination on biofilm formation of Paenibacillus polymyxa and Paenibacillus macerans. African Journal of Microbiology Research. 6 (13), 3343-3347 (2012).
- Lee, H. W., Ghimire, S. R., Shin, D. H., Lee, I. J., Kim, K. U. Allelopathic effect of the root exudates of K21, a potent allelopathic rice. Weed Biology and Management. 8 (2), 85-90 (2008).
- Belimov, A. A., et al. Rhizobacteria that produce auxins and contain 1-amino-cyclopropane-1-carboxylic acid deaminase decrease amino acid concentrations in the rhizosphere and improve growth and yield of well-watered and water-limited potato (Solanum tuberosum). Annals of Applied Biology. 167 (1), 11-25 (2015).
- Ankati, S., Podile, A. R. Metabolites in the root exudates of groundnut change during interaction with plant growth promoting rhizobacteria in a strain-specific manner. Journal of Plant Physiology. 243, 153057(2019).
- Gordillo, F., Chavez, F., Jerez, C. A. Motility and chemotaxis of Pseudomonas sp. B4 towards polychlorobiphenyls and chlorobenzoates. FEMS Microbiology Ecology. 60 (2), 322-328 (2007).
- Bais, H. P., Weir, T. L., Perry, L. G., Gilroy, S., Vivanco, J. M. The role of root exudates in rhizosphere interactions with plants and other organisms. Annual Review of Plant Biology. 57, 233-266 (2006).
- Badri, D. V., Vivanco, J. M. Regulation and function of root exudates. Plant Cell Environment. 32 (6), 666-681 (2009).
- Badri, D. V., Weir, T. L., Lelie, D., Vivanco, J. M. Rhizosphere chemical dialogues: plant-microbe interactions. Curr Opin Biotech. Current Opinion in Biotechnology. 20 (6), 642-650 (2009).
- Kamilova, F., Kravchenko, L. V., Shaposhnikov, A. I., Makarova, N., Lugtenberg, B. Effects of the tomato pathogen Fusarium oxysporum f. sp. radicis-lycopersici and of the biocontrol bacterium Pseudomonas fluorescens WCS365. Molecular Plant-Microbe Interactions. 19 (10), 1121-1126 (2006).
- Kamilova, F., et al. Organic acids, sugars, and L-tryptophane in exudates of vegetables growing on stonewool and their effects on activities of rhizosphere bacteria. Molecular Plant-Microbe Interactions. 19 (3), 250-256 (2006).
- Badri, D. V., Vivanco, J. M. Regulation and function of root exudates. Plant Cell Environment. 32 (6), 666-681 (2009).
- Hao, W. Y., Ren, L. X., Ran, W., Shen, Q. R. Allelopathic effects of root exudates from watermelon and rice plants on Fusarium oxysporum f.sp. Niveum. Plant and Soil. 336 (1-2), 485-497 (2010).
- Hao, Z. P., Wang, Q., Christie, P., Li, X. L. Allelopathic potential of watermelon tissues and root exudates. Scientia Horticulturae. 112 (3), 315-320 (2007).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon