Method Article
In vitro Modèle de vaisseau humain fœtal sur puce pour étudier la mécanobiologie du développement
Dans cet article
Résumé
Il s’agit d’un flux de travail simple permettant de différencier les cellules endothéliales des cellules souches pluripotentes humaines, suivi d’un protocole détaillé pour leur stimulation mécanique. Cela permet d’étudier la mécanobiologie du développement des cellules endothéliales. Cette approche est compatible avec les dosages en aval de cellules vivantes prélevées sur la puce de culture après stimulation mécanique.
Résumé
Le cœur est le premier organe à être établi fonctionnellement au cours du développement, initiant ainsi la circulation sanguine très tôt dans la gestation. En plus de transporter l’oxygène et les nutriments pour assurer la croissance du fœtus, la circulation fœtale contrôle de nombreux événements de développement cruciaux qui se déroulent dans la couche endothéliale par le biais de signaux mécaniques. Les signaux biomécaniques induisent des changements structurels des vaisseaux sanguins, établissent la spécification artérioveineuse et contrôlent le développement des cellules souches hématopoïétiques. L’inaccessibilité des tissus en développement limite la compréhension du rôle de la circulation dans le développement humain précoce ; Par conséquent, les modèles in vitro sont des outils essentiels pour l’étude de la mécanobiologie des vaisseaux. Cet article décrit un protocole permettant de différencier les cellules endothéliales des cellules souches pluripotentes induites humaines et leur ensemencement ultérieur dans un dispositif fluidique afin d’étudier leur réponse aux signaux mécaniques. Cette approche permet une culture à long terme de cellules endothéliales sous stimulation mécanique, suivie d’une récupération des cellules endothéliales pour une caractérisation phénotypique et fonctionnelle. Le modèle in vitro établi ici sera déterminant pour élucider les mécanismes moléculaires intracellulaires qui transduisent la signalisation médiée par des signaux mécaniques, qui orchestrent finalement le développement des vaisseaux au cours de la vie fœtale humaine.
Introduction
Au cours du développement embryonnaire, le cœur est le premier organe à établir une fonctionnalité1, avec des contractions détectables dès le stade le plus précoce de la formation du tube endocardique2. La circulation, ainsi que les signaux mécaniques médiés par le flux sanguin dans le vaisseau, contrôlent de nombreux aspects cruciaux du développement précoce. Avant l’établissement de la circulation fœtale, le système vasculaire est organisé en un plexus capillaire primaire ; Lors du fonctionnement cardiaque, ce plexus se réorganise en vascularisation veineuse et artérielle3. Le rôle des signaux mécaniques dans la spécification artérioveineuse est reflété par l’expression pan-endothéliale des marqueurs artériels et veineux avant l’initiation du flux sanguin4.
Les forces hémodynamiques contrôlent non seulement le développement du système vasculaire lui-même, mais jouent également un rôle fondamental dans le contrôle de la formation des cellules sanguines. Les cellules souches et progénitrices hématopoïétiques (HSPC) émergent de cellules endothéliales spécialisées appelées endothélium hémogène 5,6,7,8, présentes dans différentes régions anatomiques de l’embryon exclusivement au stade précoce du développement. Des modèles déficients cardiaques, ainsi que des modèles in vitro, ont démontré que les signaux mécaniques instruisent et augmentent la production de HSPC à partir de l’endothélium hémogène 9,10,11,12,13,14.
Il a été démontré que différents types de dynamique d’écoulement contrôlent de manière différentielle le cycle cellulaire15, connu pour être important à la fois dans l’endothélium hémogène 16,17 et dans la spécification des cellules artérielles18. Dans l’ensemble, les signaux mécaniques sont des déterminants essentiels de l’identité et de la fonction cellulaires au cours du développement. De nouveaux dispositifs fluidiques in vitro nous permettent de surmonter les limites liées à l’étude de la mécanobiologie du développement au cours du développement du sang humain in vivo.
L’objectif général du protocole de ce manuscrit est de décrire, étape par étape, le pipeline expérimental pour étudier l’effet de la contrainte de cisaillement sur les cellules endothéliales humaines dérivées in vitro de cellules souches pluripotentes induites humaines (hiPSC). Ce protocole contient des instructions détaillées sur la différenciation des hiPSC en cellules endothéliales et leur ensemencement ultérieur dans des puces fluidiques pour le protocole de stimulation. Grâce à cela, différentes cellules endothéliales dérivées in vitro peuvent être testées pour leur capacité à détecter la contrainte de cisaillement en analysant leur orientation en réponse à l’écoulement. Cela permettra à d’autres laboratoires de répondre à des questions sur la réponse au stress de cisaillement et ses conséquences fonctionnelles sur différentes identités de cellules endothéliales.
Protocole
REMARQUE : Toutes les techniques de culture cellulaire doivent être effectuées dans des conditions stériles dans une hotte à flux laminaire et les cellules doivent être incubées à 37 °C dans une atmosphère humifiée avec 5 % de CO2. Les instructions pour toutes les préparations de cytokines à la fois pour l’entretien (rhbFGF) et pour le protocole de différenciation (rhBMP4, rhVEGF, rhbFGF, rhIL6, rhFLT3L, rhIGF1, rhIL11, rhSCF, rhEPO, rhTPO, rhIL3) se trouvent dans le tableau supplémentaire S1.
1. Culture des hiPSC - décongélation, entretien et congélation des cellules
- Préparation du milieu d’entretien, des facteurs de croissance et d’autres réactifs
- Préparer le milieu de culture en ajoutant le supplément de milieu sans sérum hESCs entier, 36 mL d’albumine sérique bovine (BSA) à 25 % et 1 mL de 55 mM de β-mercaptoéthanol au milieu basal Eagle Medium/F12 (DMEM/F-12) de Dulbecco (voir le tableau des matériaux).
- Remettre en suspension 1 mg d’inhibiteur de la Rho kinase (iRock) dans 1 mL de DMSO, faire des aliquotes de 50 μL et les conserver à -20 °C.
NOTE : Ces aliquotes peuvent être conservées à -20 °C pendant 1 an. Une fois décongelés, ils peuvent être conservés à 4 °C pendant 1 semaine. - Décongeler la solution de vitronectine (VTN-N) sur de la glace et aliquote 60 μL par flacon avant de la conserver à -80 °C. Juste avant d’enrober les plaques, diluer le bouillon de 60 μL dans 6 mL de solution saline tamponnée au phosphate (DPBS) de Dulbecco ; la concentration finale de 5 μg/mL.
- Décongélation de lignées cellulaires hiPSC
NOTE : La lignée de cellules souches pluripotentes humaines SFCi55 a déjà été dérivée en interne et largement utilisée pour la différenciation en divers types cellulaires et différentes lignées embryonnaires 19,20,21,22.- Enduire un puits d’une plaque à 6 puits avec 1 mL de solution de VTN-N pendant 1 h à 37 °C.
REMARQUE : Après incubation, les plaques revêtues peuvent être conservées à 4 °C jusqu’à 1 semaine. - Aspirer la solution de VTN-N à l’aide d’une pipette d’aspiration et ajouter 1 mL de milieu de culture préchauffé complété par 20 ng/mL de rhbFGF (tableau supplémentaire S1).
- Décongeler rapidement le flacon contenant les hiPSC au bain-marie et transférer la cellule dans 5 mL de milieu de culture préchauffé.
- Faites tourner les cellules pendant 3 min à 300 × g à température ambiante.
- Remettre en suspension la pastille cellulaire dans 0,5 mL de milieu de culture complété par 20 ng/mL de rhbFGF.
- Transférer les cellules dans un puits enrobé contenant déjà 1 mL de milieu.
- Ajouter 5 μL d’iRock dans les puits contenant les cellules dans un total de 1,5 mL de milieu.
- Cultivez les cellules dans l’incubateur, changez le milieu tous les jours pendant la semaine et nourrissez deux fois les cellules, en ajoutant deux fois le volume normal de milieu aux cellules pour assurer l’alimentation pendant le week-end.
REMARQUE : Les cellules sont cultivées en présence d’iRock pendant 24 h seulement.
- Enduire un puits d’une plaque à 6 puits avec 1 mL de solution de VTN-N pendant 1 h à 37 °C.
- Maintenance et transmission des hiPSC
- Changez le milieu tous les jours avec un milieu de culture frais préchauffé complété par 20 ng/mL de rhbFGF.
- Passage des cellules lorsqu’elles atteignent environ 80 % de confluence, généralement deux fois par semaine.
- Pour faire passer les cellules, enduisez une plaque de VTN-N comme décrit précédemment aux étapes 1.2.1 et 1.2.2.
- Aspirez le milieu des puits avec des cellules et lavez-les avec du DPBS.
- Aspirer le DPBS et ajouter 1 mL de réactif de dissociation (voir tableau des matériaux) et incuber pendant 1 min.
- Aspirer le réactif de dissociation et incuber pendant 3 minutes supplémentaires. Tapotez fermement l’assiette 10 fois de chaque côté.
REMARQUE : L’étape de dissociation peut nécessiter une optimisation spécifique au type de cellule dans le temps d’incubation et la procédure de taraudage. - Ajouter 1 mL de milieu de culture dans les cellules et, à l’aide d’une pipette Pasteur, laver une fois avec le milieu pour s’assurer que la plupart des colonies sont recueillies.
- Ajouter 150 μL de la suspension cellulaire à chaque puits pour obtenir un rapport de passage de 1 puits dans 6.
REMARQUE : Immédiatement après la décongélation d’un nouveau flacon, il est préférable de faire passer les cellules à un rapport plus faible tel que 1 :1 ou 1 :2 pour un ou deux passages afin de leur permettre d’atteindre une phase de croissance régulière avant de passer à un rapport de 1 :6. - Cultivez les cellules dans l’incubateur, changez le milieu tous les jours pendant la semaine et nourrissez les cellules une fois pendant le week-end.
- Congélation de lignées cellulaires hiPSCs
REMARQUE : Congeler les cellules dans leurs deux premiers passages après la décongélation pour s’assurer de maintenir un lot constant de flacons congelés à faible passage pour commencer la culture. Congelez les cellules lorsqu’elles atteignent une confluence d’environ 80 %.- Remplacer le milieu par un milieu de culture frais préchauffé complété par 20 ng/mL de rhbFGF et 5 μL d’iRock et incuber pendant au moins 1 h.
- Détachez les cellules comme décrit aux étapes 1.3.2.2-1.3.2.5.
- Prélever les cellules détachées dans un tube à centrifuger de 15 mL contenant 5 mL de milieu de culture.
- Centrifuger pendant 3 min à 300 × g à température ambiante.
- Aspirer le surnageant et ajouter 1 mL de solution de cryoconservation (voir Tableau des matériaux).
- À l’aide d’une pipette Pasteur, pipetez doucement les cellules de haut en bas pour les mélanger à la solution de cryoconservation.
REMARQUE : Évitez le pipetage excessif, qui pourrait entraîner la dissociation des grappes de cellules. - Divisez la suspension cellulaire dans deux flacons de cryoconservation de 0,5 ml chacun.
- Transvaser les flacons de cryoconservation dans un récipient de cryoconservation prérefroidi à 4 °C.
- Transférez le récipient contenant les cellules dans un congélateur à -80 °C pendant 24 h avant de transférer les flacons dans de l’azote liquide pour un stockage à long terme.
2. Différenciation des hiPSC en cellules endothéliales
- Préparation du milieu de différenciation, des cytokines et des facteurs de croissance
- Préparer le milieu de différenciation sans sérum (SFD) conformément au tableau 1. Utilisez ce support du jour 0 au jour 5 de différenciation.
- Préparer un milieu sans sérum pour les cellules CD34+ (SFM-34) en ajoutant 34 suppléments nutritifs et 5 mL de L-glutamine au milieu de base 34 SFM (voir le tableau des matériaux). Utilisez ce support à partir du 6e jour de différenciation.
- Remettre en suspension 1 mg de CHIR99021 dans 716 μL de DMSO pour obtenir une solution de 3 mM. Incuber à température ambiante jusqu’à ce qu’il soit complètement remis en suspension ; si nécessaire, réchauffez-vous rapidement à 37 °C. Fabriquez des aliquotes de 20 μL et conservez-les à -20 °C jusqu’à 6 mois. Utiliser immédiatement après la décongélation et ne pas congeler à nouveau ou stocker.
- Remettre les cytokines en suspension conformément aux instructions du tableau supplémentaire S1. Conserver toutes les aliquotes des cytokines à -80 °C.
- Différenciation des cellules endothéliales
REMARQUE : Pour chaque jour de différenciation, préparer 18 mL (3 mL milieu/puits) de milieu SFD préchauffé, selon les mélanges de cytokines décrits dans la section Tableau 2.- Jour 0 - formation des corps embryoïdes (EB)
- Préparer 18 mL de milieu SFD avec Mélanger 1 cytokine selon le tableau 2, pour chaque plaque de 6 puits (3 mL/puits).
- Ajouter 2 mL de milieu SFD préchauffé avec Mélanger 1 cytokine dans chaque puits d’une plaque à 6 puits répulsif pour cellules (voir le tableau des matériaux).
- Pour former des EB, suivez les étapes décrites aux étapes 1.3.2.2 à 1.3.2.4.
REMARQUE : Assurez-vous que les hiPSC sont confluents à 70-80% pour commencer la différenciation. - Ajouter 1 mL de milieu SFD préchauffé avec des cytokines Mix 1 dans chaque puits d’amas de cellules détachées.
- À l’aide d’une pipette de Pasteur, transférez délicatement les amas de cellules dans un seul puits de répulsif cellulaire pour la formation d’EB dans un rapport de 1 :1.
- Après avoir placé la plaque dans l’incubateur, déplacez-la vers l’avant et vers l’arrière, à droite et à gauche, pour disperser les EB uniformément dans le puits.
- Jour 1 - Modification moyenne des EB
REMARQUE : Cette étape n’est nécessaire que si, au jour 1 de la différenciation, il y a beaucoup de cellules individuelles en suspension à côté des EB.- Préparer 18 mL de milieu SFD avec Mélanger 1 cytokine selon le tableau 2, pour chaque plaque de 6 puits (3 mL/puits).
- Faites tourner la plaque avec les EB pour les déplacer vers le centre et collectez-les à l’aide d’une pipette Pasteur dans un tube à centrifuger de 15 ml.
REMARQUE : Si les EB semblent agglomérés comme dans les cordes, séparez-les en les pipetant de haut en bas avec un P1000 avant de les recueillir dans le tube à centrifuger de 15 ml. - Attendez 5 à 10 minutes pour que les EB se déposent au fond du tube.
REMARQUE : Si les EB sont trop petits, centrifugez-les pendant 5 min à 100 × g pour les aider à se déposer. - Lavez les plaques anticellulaires avec de l’eau stérile ou du DPBS pour éliminer les cellules ou les débris.
- Aspirez soigneusement et lentement le surnageant des EB sans les déloger.
- Ajouter 2 mL de SFD avec des cytokines Mix 1 dans chaque puits des plaques répulsives cellulaires.
- Remettre les EB en suspension à l’aide de 1 mL de milieu SFD avec Mix 1 cytokines pour chaque puits de départ - pour une plaque à 6 puits, ajouter 6 mL de milieu.
- Transférez les EB dans les plaques répulsives cellulaires dans un volume de 1 mL par puits, qui contient déjà 2 mL de milieu SFD.
- Après avoir placé la plaque dans l’incubateur, déplacez-la vers l’avant et vers l’arrière, à droite et à gauche, pour disperser les EB uniformément dans le puits.
- Jour 2 - ajout de CHIR99021
- Faites tourbillonner les EB au centre de la plaque et ajoutez CHIR99021 selon le tableau 2 sur le côté du puits pour éviter tout contact direct avec les cellules.
REMARQUE : Si le milieu n’a pas été changé au jour 1, remplacez le milieu entier au lieu d’ajouter CHIR seul. Préparer 18 mL de milieu SFD avec le mélange 2 selon le tableau 2, pour chaque plaque de 6 puits (3 mL/puits). - Après avoir placé la plaque dans l’incubateur, déplacez-la vers l’avant et vers l’arrière, à droite et à gauche, pour disperser les EB uniformément dans le puits.
- Faites tourbillonner les EB au centre de la plaque et ajoutez CHIR99021 selon le tableau 2 sur le côté du puits pour éviter tout contact direct avec les cellules.
- Jour 3 - modification moyenne des EB et ajout de cytokines du mélange 3
- Préparer 18 mL de milieu SFD avec Mélanger 3 cytokines selon le tableau 2, pour chaque plaque de 6 puits (3 mL/puits).
- Collectez les EB comme décrit aux étapes 2.2.2.2 à 2.2.2.4.
- Ajouter 2 mL de milieu SFD préchauffé avec les cytokines Mix 3 dans les plaques répulsives cellulaires.
- Aspirer soigneusement le surnageant des EB. Ajouter 1 mL/puits de SFD avec Mix 3 cytokines.
- Répartissez les EB entre les puits comme décrit aux étapes 2.2.2.8 à 2.2.2.9.
- Jour 6 - changement de milieu pour SFM-34 et ajout de cytokines du mélange 4
- Préparer 18 mL de milieu SFD avec Mélanger 4 cytokines selon le tableau 2, pour chaque plaque de 6 puits (3 mL/puits).
- Collectez les EB comme décrit aux étapes 2.2.2.2 à 2.2.2.4.
- Ajouter 2 mL de milieu SFM-34 préchauffé avec les cytokines Mix 4 dans les plaques répulsives cellulaires.
- Aspirer soigneusement le surnageant des EB. Ajouter 1 mL/puits de SFM-34 avec le mélange de 4 cytokines.
- Répartissez les EB entre les puits comme décrit aux étapes 2.2.2.8 à 2.2.2.9.
- Jour 0 - formation des corps embryoïdes (EB)
3. Isolement et ensemencement des cellules CD34+ dans la puce
REMARQUE : Les cellules CD34+ sont isolées par une approche d’isolement positif à l’aide d’un kit de microbilles CD34 (voir le tableau des matériaux), qui contient des microbilles CD34 conjuguées à des anticorps monoclonaux de souris, à des anticorps anti-CD34 humains et à un réactif bloquant FcR (IgG humaines). Il est important de valider l’efficacité de l’isolement de la colonne en colorant les cellules avant et après l’isolement pour l’analyse par cytométrie en flux, ci-dessous il est indiqué quand les cellules doivent être prélevées pour cette analyse.
- Préparer les matériaux et les réactifs.
- Préparer le tampon de lavage en ajoutant 5 mL de solution BSA à 5 % et 200 μL d’EDTA 0,5 M à 45 mL de DPBS pour obtenir PBS + 0,5 % BSA + 2 mM EDTA. Préparez-les frais pour chaque isolement, filtrez-stérilisez et conservez-les au réfrigérateur jusqu’à utilisation.
- Enrober les copeaux fluidiques d’une solution de laminine préparée en diluant la rhLaminin 521 1 :50 dans du DPBS Ca 2+ Mg2+. Enrober chaque copeau avec le volume approprié pour la puce utilisée et incuber dans l’incubateur pendant 2 h avant le semis.
REMARQUE : D’autres matrices peuvent être utilisées pour le revêtement et doivent être testées pour le type de cellule/expérience spécifique. - Préparez le milieu Mix 4 SFM-34 en complétant 18 mL de milieu SFM-34 avec les cytokines Mix 4 selon le tableau 2 et placez le mélange dans un tube de 50 mL dans l’incubateur avec le couvercle légèrement dévissé pour faciliter les échanges gazeux.
- Placez les kits de perfusion sélectionnés et tout autre tube à utiliser dans l’incubateur pour dégazer.
- Jour 8 - dissociation des EB et isolement de CD34+
- Collectez les EB comme décrit aux étapes 2.2.2.2 à 2.2.2.5.
- Ajouter 1 mL de réactif de dissociation cellulaire par puits de départ d’EB prélevés (si 6 puits ont été collectés, ajouter 6 mL).
- Retransférer 1 mL de la suspension EB dans le réactif de dissociation cellulaire dans chaque puits de la plaque répulsive cellulaire.
- Incuber pendant 10 min dans l’incubateur.
- Pipeter doucement les EB de haut en bas contre le puits avec un P1000, pas plus de 10 fois.
- Répétez les étapes 3.2.4 à 3.2.5 pour un total de 3x.
REMARQUE : Si les EB sont difficiles à dissocier, répétez les étapes ci-dessus 4 fois au total. - Ajouter 5 mL de tampon de lavage pour chaque puits d’EB dissociés.
- Recueillir les cellules dans un tube à centrifuger de 50 ml en les faisant passer à travers une crépine de 40 μm. Prélever 10 μL de la suspension cellulaire pour compter les cellules.
REMARQUE : Pour tester l’efficacité de l’isolement, transférer 105 cellules/tube dans deux tubes différents (échantillon témoin non coloré et échantillon d’essai prétrié) qui seront utilisés ultérieurement pour la cytométrie en flux (comme décrit aux étapes 4.3.9 à 4.3.13). Pour une plaque à 6 puits, ~10cellules de 6 doivent être collectées après filtration. - Faites tourner les cellules pendant 10 min à 300 × g.
- Remettre les cellules en suspension dans 300 μL de tampon de lavage, en pipetant doucement plusieurs fois pour s’assurer qu’il n’y a pas de grumeaux. Continuez à suivre le protocole du fabricant (voir le tableau des matériaux).
- Ensemencement de cellules CD34+ dans des puces fluidiques
NOTA : La puce fluidique utilisée dans le protocole a une hauteur de canal de 0,6 mm et une longueur de 50 mm, pour une surface de croissance totale de 2,5cm2 (figure supplémentaire S1). Ce type de puce est ensemencé avec un volume total de suspension cellulaire de 150 μL. Différentes puces peuvent être utilisées, et le volume d’ensemencement et la densité cellulaire doivent être adaptés en fonction de la zone de croissance. Une optimisation supplémentaire peut être nécessaire en fonction de la lignée cellulaire utilisée et de sa croissance.- Remettre en suspension les cellules CD34+ isolées dans 300 μL de milieu SFM-34 préchauffé avec des cytokines Mix 4.
- Prélevez 10 μL de la suspension cellulaire et comptez les cellules.
- Remettre en suspension 2,5 × 105 cellules dans un volume final de 150 μL complété par SFM-34 ; ajouter 0,5 μL d’iRock.
REMARQUE : Pour tester l’efficacité de l’isolement, transférer 105 cellules/tube dans un tube (échantillon d’essai post-trié) qui sera utilisé plus tard pour la cytométrie en flux (comme décrit aux étapes 4.3.9-4.3.13). - Aspirez lentement la laminine des copeaux fluidiques en plaçant la pointe d’un P200 à l’intérieur du réservoir sur le bord du canal.
REMARQUE : Si le liquide est difficile à collecter, soulevez lentement un côté de la puce pour aider le liquide à se déplacer vers le réservoir opposé. - Ajoutez la suspension cellulaire régulièrement dans le canal pour vous assurer qu’aucune bulle ne se forme.
REMARQUE : Effectuez les étapes 3.3.4 à 3.3.5 rapidement mais doucement pour éviter que la laminine ne se dessèche et ne forme des bulles dans les canaux de la puce. Si des bulles se forment, soulevez un côté de la puce et tapotez doucement la lame pour mobiliser les bulles ; Lorsqu’ils atteignent le réservoir, ils s’élèvent jusqu’à l’interface aérienne et ne devraient pas être en mesure de louer le canal. - Transférez la puce dans l’incubateur et laissez-les toute la nuit pour que les cellules soient complètement attachées au canal et aient l’air allongées.
- Lorsque les cellules sont complètement attachées, aspirez le milieu comme à l’étape 3.3.4 et remplacez-le par 200 μL de SFM-34 supplémenté en cytokines.
- À partir de maintenant, remplacez le milieu quotidiennement jusqu’à ce que les cellules aient atteint 90%-100% de confluence.
4. Application d’un flux continu aux cellules endothéliales - Aorte sur puce
- Préparer les matériaux et les réactifs.
- Préparez le milieu Mix 4 SFM-34 en complétant 18 mL de milieu SFM-34 avec les cytokines Mix 4 selon le tableau 2 et placez-le dans un tube de 50 mL dans l’incubateur avec le couvercle légèrement dévissé pour faciliter les échanges gazeux.
- Placez les ensembles de perfusion sélectionnés et tout tuyau à utiliser pour la configuration fluidique dans l’incubateur pour dégazer.
- Assemblage d’un système fluidique
- Installez le set de perfusion dans l’appareil conformément au protocole du fabricant.
REMARQUE : N’oubliez pas d’utiliser des pinces dans le système. Si des pinces coulissantes sont utilisées pour cette étape, elles doivent être glissées sur le tube avant de se connecter à la puce. - Fixez une nouvelle puce fluidique et ajoutez le milieu SFM-34 supplémenté en cytokines, en veillant à remplir les deux réservoirs dans des conditions stériles dans la hotte.
- Effectuez le programme d’élimination des bulles et l’étape d’étalonnage.
- Retirez l’unité fluidique avec l’ensemble connecté de l’incubateur et transférez-la dans la hotte ; Prenez également les puces contenant les cellules de l’incubateur.
- Fixez le tube des deux côtés de la puce de test.
- Retirez le tube de la puce de test.
REMARQUE : Vérifiez qu’aucune bulle n’est présente dans le connecteur Luer à l’extrémité du tube. Si des bulles sont visibles, aspirez-les soigneusement à l’aide d’une pipette P200 et, si nécessaire, ajoutez plus de milieu pour vous assurer que le connecteur est rempli de milieu. - Connectez la puce contenant les cellules avec le tube.
- Retirez ou ouvrez les pinces.
- Transférez le système dans l’incubateur et connectez la pompe à air à l’unité fluidique.
- Démarrez le programme présélectionné à l’aide du logiciel dédié à la pompe (figure supplémentaire S2) avec l’augmentation progressive de la contrainte de cisaillement décrite dans le tableau 3.
REMARQUE : Selon l’expérience spécifique nécessaire, le programme de stimulation peut avoir besoin d’être optimisé. Il s’agit ici d’une augmentation progressive de la contrainte de cisaillement conduisant à la valeur finale de 5 dyn/cm2, qui imite la contrainte de cisaillement calculée pour être subie par la paroi de l’aorte dorsale au début de la circulation fœtale 9. Indépendamment de la contrainte de cisaillement finale qui sera utilisée, il est nécessaire d’augmenter progressivement au fil du temps pour permettre aux cellules de s’adapter à la force sans provoquer le détachement des cellules de la puce. Si le protocole de stimulation sélectionné dure plus de 3 jours, les cytokines doivent être complétées dans le système en ajoutant 1 mL de SFM-34 contenant les cytokines Mix 4 qui seraient normalement ajoutées dans 18 mL. Pour ce faire, le programme de pompe est rapidement mis en pause et 500 μL de milieu supplémenté sont ajoutés à chacune des deux seringues du kit fluidique.
- Installez le set de perfusion dans l’appareil conformément au protocole du fabricant.
- Prélèvement de cellules pour analyse
- Préchauffer le tampon de dissociation au bain-marie.
- Retirez l’unité fluidique de l’incubateur et déplacez-la dans la hotte.
- Fixez le tube flanquant le copeau des deux côtés et retirez le tube des réservoirs du chip.
- Retirez délicatement le milieu de la puce et remplacez-le par du DPBS Ca2 + Mg2+ pour laver les cellules.
REMARQUE : Cette étape de lavage avec PBS peut être sautée si les cellules commencent à se détacher. - Ajouter délicatement 150 μL de tampon de dissociation et incuber pendant 3 min à 37 °C.
REMARQUE : Vérifiez au microscope si les cellules sont simples et détachées ; sinon, incuber pendant 2 minutes supplémentaires. Il est essentiel que les cellules soient complètement détachées du canal avant d’aspirer le milieu, car la puce ne permet pas de faciliter le détachement des cellules par pipetage. D’autres solutions peuvent être utilisées pour détacher les cellules, telles que la trypsine ou les tampons à base d’EDTA. - Récupérez le tampon de dissociation contenant les cellules d’un réservoir et transférez-le dans un tube à centrifuger de 15 ml et lavez le canal une fois avec du DPBS pour recueillir toutes les cellules détachées.
- Ajouter 1 mL de tampon de lavage dans le tube de 15 mL avec les cellules et prendre 10 μL pour compter les cellules.
- Divisez la suspension cellulaire dans des tubes de cytométrie en flux pour avoir 10 à5 cellules par tube à essai.
- Essorez les tubes pendant 5 min à 300 × g.
- Préparez la solution de coloration pour qu’elle contienne 50 μL pour chaque tube à essai à colorer. Ajouter le CD34 PerCP-efluor710 ou le CD34-PE à une dilution de 1 :100 et 1 :200, respectivement.
- Remettre les cellules en suspension dans 50 μL de solution colorante et incuber pendant 30 min à 4 °C.
- Lavez les cellules en ajoutant 2 mL de tampon de lavage et essorez pendant 5 min à 300 × g.
- Remettre les pastilles en suspension dans 100 μL de solution colorante et acquérir les données à l’aide d’un cytomètre en flux.
REMARQUE : Les cellules peuvent également être lysées directement dans la puce pour l’extraction de l’ARN à l’aide de 150 μL de tampon de lyse de l’ARN ou fixées pour l’imagerie à l’aide de paraformaldéhyde à 4 % dans du DPBS pendant 10 min à température ambiante.
- Analyse de l’orientation des cellules
- Analysez les images pour quantifier les changements d’orientation des cellules à l’aide de FIJI23 (figure supplémentaire S3).
- Ouvrez le gestionnaire de régions d’intérêt (ROI) à partir de l’onglet Analyser | Outils | Menu du gestionnaire de retour sur investissement .
- Dessinez manuellement les contours des cellules à l’aide de l’outil de sélection de polygones et ajoutez-les au gestionnaire de retour sur investissement en cliquant sur Ajouter ou en utilisant le raccourci CTRL+T .
- Mesurez l’orientation de chaque retour sur investissement en choisissant la mesure d’ellipse Ajuster dans l’onglet Analyser | Définir le menu Mesures .
- Appliquez la mesure à tous les retours sur investissement grâce à la Plus... Commande multi-mesures dans le gestionnaire de ROI.
REMARQUE : Cette étape permet d’ajuster une ellipse à chaque ROI et de générer un tableau contenant la longueur des axes de l’ellipse principale et mineure, ainsi que l’angle. - Exportez le tableau dans un fichier CSV pour l’importer dans un autre logiciel afin de le tracer.
NOTE : Le script utilisé pour les tracés est disponible à l’adresse https://gist.github.com/nicolaromano/708b3231d730ee7f70763a7cf885
0ddc.
- Analysez les images pour quantifier les changements d’orientation des cellules à l’aide de FIJI23 (figure supplémentaire S3).
Résultats
Nous décrivons ici un protocole de différenciation et de mécano-stimulation des cellules endothéliales dérivées des hiPSC qui permet d’étudier leur réponse aux signaux mécaniques (Figure 1). Ce protocole aboutit à la production de cellules endothéliales fonctionnellement mécanosensibles. Nous fournissons ici des résultats représentatifs et décrivons le phénotype attendu pour évaluer comment les cellules répondent à la stimulation des cytokines lors de la différenciation.
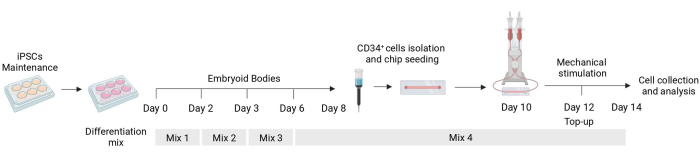
Figure 1 : Schéma du protocole de différenciation et de stimulation mécanique. Schéma du protocole de différenciation montrant la chronologie des différents mélanges de cytokines, l’isolement des cellules CD34+, l’ensemencement de la puce fluidique et l’analyse finale des cellules stimulées mécaniquement. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Culture des hiPSC
Il est important de commencer le protocole à partir de hiPSC qui se développent correctement dans des conditions d’auto-renouvellement. Une bonne indication de la qualité de la culture est la vitesse de leur croissance. Après la décongélation, les cellules peuvent avoir besoin de 2 à 3 semaines pour atteindre la phase de croissance correcte qui assurera une bonne différenciation. Lorsque les cellules peuvent être traversées deux fois par semaine dans un rapport de 1 :6 pour atteindre une confluence presque complète, c’est le moment où elles sont prêtes à être différenciées le jour même où elles doivent être traversées.
Différenciation des hiPSCs en cellules endothéliales
La première étape de la différenciation, qui consiste en la formation de corps embryoïdes (EB), dépend de la lignée cellulaire et peut nécessiter une certaine optimisation pour la lignée cellulaire spécifique utilisée. La dissociation décrite dans les étapes 1.3.2.2-1.3.2.4 du protocole peut être modifiée en réduisant ou en prolongeant l’incubation avec le réactif de dissociation et la dissociation ultérieure avec la pipette Pasteur. De plus, d’autres réactifs de dissociation peuvent être utilisés pour cette étape en plus de la dissociation physique des colonies à l’aide d’un outil de coupe ou d’une pointe de pipette P100. Les EB de bonne qualité présentent un bord défini au jour 2 de la différenciation et apparaissent clairs et brillants lorsqu’ils sont observés au microscope ; des zones plus foncées peuvent indiquer la mort cellulaire dans les EB (Figure 2).
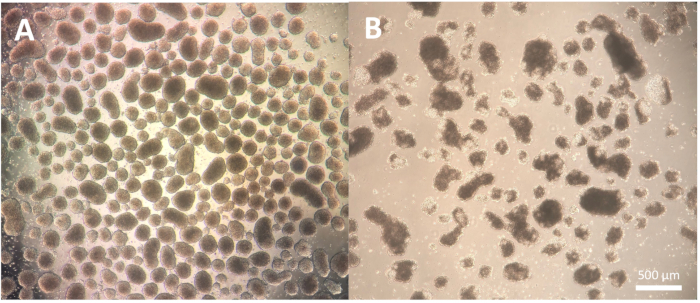
Figure 2 : Morphologie des corps embryoïdes. (A) Corps embryoïdes au jour 2 montrant des bords extérieurs bien définis et une taille constante. (B) Corps embryoïdes de mauvaise qualité au jour 2 montrant une mort cellulaire étendue conduisant à une désagrégation de la structure. Barre d’échelle = 500 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Au jour 2, l’ajout de CHIR99021 aux EB inhibe la protéine GSK-3, ce qui entraîne l’activation de la voie Wnt. Différentes lignées cellulaires ont des réponses différentes au traitement par CHIR, et cela devrait être testé en quantifiant le nombre de cellules CD34+ obtenues au jour 8 en utilisant différentes concentrations (figure 3).
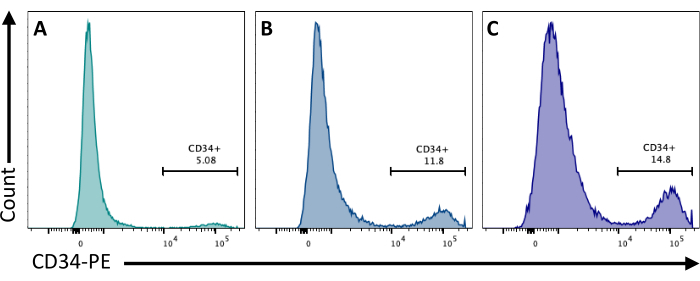
Figure 3 : Différenciation des cellules endothéliales avec différents traitements CHUR. L’engagement des cellules endothéliales a été quantifié par cytométrie en flux au jour 8 de la différenciation par l’expression membranaire de CD34, après un traitement CHIR au jour 2 à (A) 3 μM, (B) 5 μM et (C) 7 μM. Les données de cytométrie en flux ont été obtenues à l’aide de cytomètres à cinq lasers et d’un logiciel dédié (voir Tableau des matériaux). Veuillez cliquer ici pour voir une version agrandie de cette figure.
Isolement cellulaire CD34+
Il est important de valider que l’enrichissement en CD34+ à l’aide des billes magnétiques fournit au moins 80% de CD34+ après élution de la colonne. Pour assurer une pureté suffisante, une aliquote de cellules obtenues à partir de l’isolement magnétique peut être analysée par cytométrie en flux en veillant à utiliser un clone d’anticorps différent de celui utilisé pour l’enrichissement magnétique. Ici, le clone 4H11 a été utilisé et une pureté de ~85% a été atteinte après enrichissement (Figure 4).
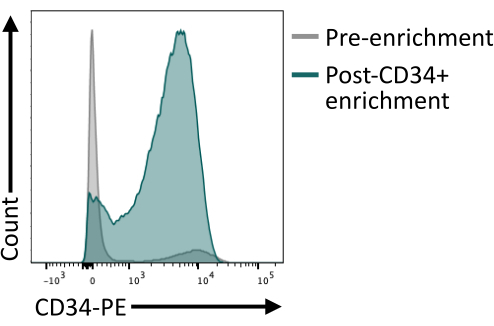
Figure 4 : Expression membranaire de CD34 avant et après enrichissement par tri magnétique. Les corps embryoïdes dissociés du jour 8 (en gris) et les cellules après enrichissement magnétique (en vert) ont été colorés pour l’expression de CD34 et analysés par cytométrie en flux, montrant un enrichissement réussi après le tri. Les données de cytométrie en flux ont été obtenues à l’aide de cytomètres à cinq lasers et d’un logiciel dédié (voir Tableau des matériaux). Veuillez cliquer ici pour voir une version agrandie de cette figure.
Ensemencement des cellules dans le canal fluidique
Lors de l’ensemencement des cellules dans le canal fluidique, il est crucial de suivre l’adhésion et la prolifération des cellules endothéliales. Après l’ensemencement, les cellules mettent ~5 h pour adhérer complètement au canal (Figure 5A). Une solution de revêtement alternative peut également être testée pour améliorer l’adhérence à ce stade. Pour confirmer que les cellules testées sont mécanosensibles et donc capables de répondre à une stimulation mécanique, l’orientation des cellules peut être testée au fil du temps. Les cellules avant la stimulation présentent une orientation aléatoire (Figure 5A et Figure 5C) et elles se réorientent parallèlement à la direction de l’écoulement (Figure 5B, C). Le protocole décrit ici permet de prélever les cellules du canal pour effectuer une analyse en aval, par exemple une cytométrie en flux, pour l’étude de leur immunophénotype membranaire, fournissant l’identité endothéliale des cellules stimulées (Figure 5D,E).
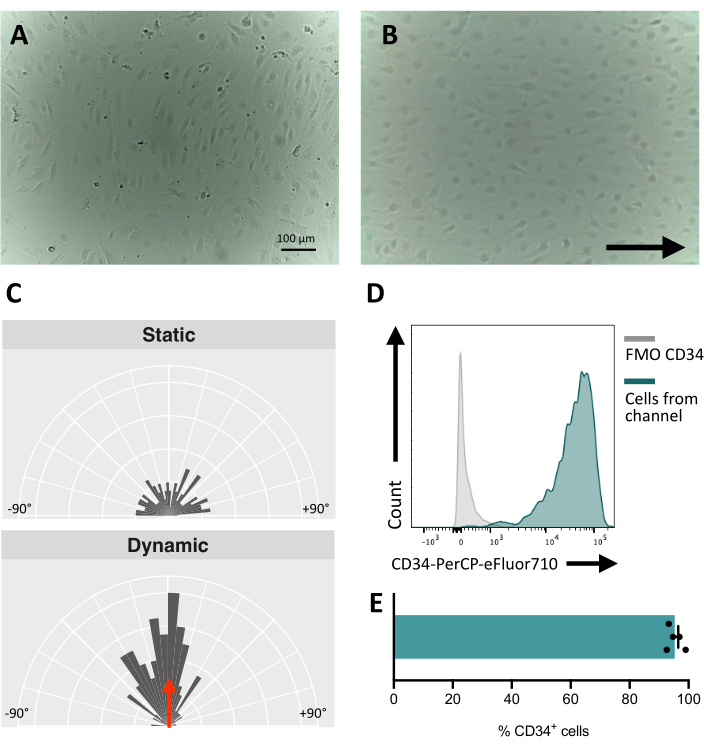
Figure 5 : Mécano-réactivité des cellules endothéliales dérivées des cellules hiPS. (A) Couche confluente de cellules CD34+ isolées 48 h après l’ensemencement. (B) Couche réorientée de cellules endothéliales 3 jours sous culture dynamique. (C) Analyse d’orientation des cellules endothéliales après 5 jours de culture dynamique. (D) Profil d’expression de CD34 de cellules cultivées sous flux pendant 5 jours. (E) Pourcentage de cellules CD34+ de la population cellulaire extraites du canal fluidique. Les images ont été prises à l’aide d’un microscope inversé dans l’incubateur ; Les données de cytométrie en flux ont été obtenues à l’aide de cytomètres à cinq lasers et d’un logiciel dédié (voir Tableau des matériaux). Barres d’échelle = 100 μm (A,B). Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Réactifs | Concentration des stocks | Volume ajouté | Concentration finale |
| Médium Dulbecco modifié d’Iscove (IMDM) | - | 333 mL | - |
| Mélange nutritif F-12 de Ham’s (F-12) | - | 167 mL | - |
| Supplément N-2 (100x) | 100 fois plus tard | 5 ml | 1 fois |
| Supplément B-27 (50x) | 50 fois plus tard | 10 ml | 1 fois |
| Acide ascorbique | 10 mg/mL | 1,25 mL | 25 μg/mL |
| α-monothioglycérol (MTG) | 11,5 millions | 19,5 μL | 448,5 μM |
| Albumine sérique humaine | 100 mg/mL | 2,5 mL | 0,5 mg/mL |
| Holo-Transferrine | 100 mg/mL | 0,75 ml | 150 μg/mL |
Tableau 1 : Composition et recette pour 500 mL de milieu de différenciation sans sérum (SFD).
| Des journées de différenciation | Mélange de cytokines | Cytokine | Concentration finale |
| Jour 0 - 2 | Mélange 1 | Le BMP4 | 20 ng/mL |
| Jour 2 | Mélange 2 | CHIR99021 | 7 μM |
| À partir du jour 3 | Mélangez 3 et 4 | Le VEGF | 15 ng/mL |
| bFGF | 5 ng/mL | ||
| À partir du jour 6 | Mélange 4 | IL6 | 10 ng/mL |
| FLT3L | 10 ng/mL | ||
| L’IGF1 | 25 ng/mL | ||
| IL11 | 5 ng/mL | ||
| SCF | 50 ng/mL | ||
| OEB | 3 U/mL | ||
| TPO | 30 ng/mL | ||
| IL3 | 30 ng/mL |
Tableau 2 : Mélanges de cytokines utilisées pour la différenciation des cellules endothéliales, jours pendant lesquels elles sont ajoutées au milieu SFD et concentration finale.
| Contrainte de cisaillement (dyn/cm2) | Temps (h) |
| 0.5 | 1 |
| 1 | 1 |
| 1.5 | 1 |
| 2 | 1 |
| 2.5 | 1 |
| 3 | 1 |
| 3.5 | 1 |
| 4 | 1 |
| 4.5 | 1 |
| 5 | Jusqu’à la fin de l’expérience |
Tableau 3 : Valeurs des contraintes de cisaillement pour la culture dynamique et la durée de leur application.
Figure supplémentaire S1 : Géométrie et dimensions de la puce et du tube utilisés pour ce protocole. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S2 : Guide étape par étape pour le logiciel de commande de la pompe à air avec une description de chaque étape. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S3 : Guide pour l’analyse d’orientation à l’aide de FIDJI montrant le dessin de la forme de la cellule, l’ajustement elliptique et la mesure finale. Veuillez cliquer ici pour télécharger ce fichier.
Tableau supplémentaire S1 : Taille de l’unité, volume de remise en suspension et concentrations de la souche pour les cytokines utilisées dans le protocole de différenciation. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Le protocole que nous décrivons ici permet de générer des cellules endothéliales mécanosensibles à partir de cellules souches pluripotentes humaines et d’étudier leur réponse à la stimulation mécanique médiée par une contrainte de cisaillement contrôlée. Ce protocole est entièrement à base de cytokines et entièrement compatible avec les réactifs GMP pour une traduction potentielle dans la production de cellules pour la thérapie cellulaire.
La dérivation des hiPSCs fournit aux scientifiques un modèle instrumental pour les premiers stades du développement embryonnaire qui permet d’étudier des processus qui sont autrement difficiles à étudier in vivo24. En fait, les tissus embryonnaires humains disponibles pour la recherche sont prélevés sur des embryons dépourvus de circulation, ce qui pourrait avoir un impact significatif sur la signature moléculaire contrôlée par des signaux mécaniques. L’approche décrite ici permet l’imagerie en direct et l’étude en temps réel de la réponse cellulaire à la contrainte de cisaillement. La combinaison des hiPSCs avec la fluidique fournit un modèle d’étude qui permet de surmonter la disponibilité limitée et l’inaccessibilité des tissus fœtaux en développement lorsque l’initiation de la circulation remodèle et contrôle l’établissement du système cardiovasculaire et sanguin 3,9,10,25.
L’une des limites du protocole est que les cellules endothéliales dérivées de ce protocole peuvent ne pas refléter les différentes identités des différentes cellules endothéliales présentes dans les tissus en développement. Pour surmonter cette limitation, une combinaison spécifique de cytokines peut être nécessaire au cours du processus de différenciation précédant la stimulation fluidique afin d’obtenir l’identité souhaitée ou le phénotype tissulairespécifique 26. L’isolement des sous-ensembles endothéliaux peut être obtenu à l’aide d’un immunophénotype plus affiné lors de l’étape d’isolement. Ce protocole isole les cellules endothéliales en se basant uniquement sur l’expression de CD34, permettant ainsi l’isolement de la colonne au lieu du tri cellulaire activé par fluorescence (FACS) ; Cela réduit la mort cellulaire et le risque de contamination. De plus, ce protocole est spécifiquement conçu pour étudier le rôle de la contrainte de cisaillement médiée par l’écoulement laminaire. D’autres approches fluidiques devront être employées pour étudier l’effet d’autres signaux mécaniques, tels que l’étirement ou la compression, ou d’autres types d’écoulement tels que l’écoulement perturbé ou perturbé.
Nous avons précédemment montré que les cellules endothéliales dérivées des CSPi imitent les identités cellulaires artérioveineuses hétérogènes27 similaires à celles observées dans l’aorte dorsale fœtale28,29,30. Ceci est particulièrement important dans le contexte du développement des vaisseaux et de la spécification cellulaire, dont on sait qu’ils sont contrôlés par la circulation sanguine. Des études dans différents modèles ont montré que l’absence de circulation entraîne une altération de la spécification artérioveineuse11,14,31. Les mécanismes qui relient les signaux mécaniques à la spécification cellulaire sont encore inconnus et le pipeline décrit ici permet d’affiner les études fonctionnelles qui n’ont pas pu être testées in vivo.
Ce pipeline décrit la production et la stimulation de cellules endothéliales dérivées de cellules hiPS à l’aide de canaux fluidiques disponibles dans le commerce, évitant ainsi la nécessité de couler les dispositifs comme pour les dispositifs en polydiméthylsiloxane (PDMS) largement utilisés12. De plus, l’utilisation de puces PDMS rend la collecte des cellules stimulées particulièrement difficile, tandis qu’avec ce protocole, les cellules peuvent être facilement récupérées du canal. Cela améliore considérablement la puissance analytique, ce qui permet des analyses ultérieures telles que des analyses protéomiques et transcriptomiques, la cytométrie en flux et des tests fonctionnels, qui peuvent nécessiter une culture supplémentaire ou des tests in vivo .
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Ces travaux ont été soutenus par la Research Advanced Grant 2021 de l’Association européenne d’hématologie, le Global Research Award 2021 de l’American Society of Hematology et le Fonds de soutien à la stratégie interne ISSF3 financé par le Welcome Trust et l’Université d’Édimbourg. Nous remercions Fiona Rossi du Flow Cytometry Facility pour son soutien dans l’analyse de la cytométrie en flux. Aux fins du libre accès, l’auteur a appliqué une licence Creative Commons Attribution (CC BY) à toute version du manuscrit accepté par l’auteur découlant de cette soumission.
matériels
| Name | Company | Catalog Number | Comments |
| 0.6 Luer uncoated slide | ibidi | IB-80186 | |
| 25% BSA | Life Technologies | A10008-01 | |
| 6-well plates | Greiner Bio-one | 657160 | |
| Accutase | Life Technologies | A1110501 | Cited as Dissociation reagent |
| Ascorbic acid | Merck | A4544-100G | |
| Aspiration pipette | Sardtedt | 86.1252.011 | |
| B27 supplement | Life Technologies | 17504044 | Cited as Neuronal cell culture supplement (50x) |
| BD FACS DIVA | BD Biosciences | Version 8.0.1 | Cited as flow cytometry software |
| BD LSR Fortessa 5 Laser | BD Biosciences | ||
| bFGF | Life Technologies | PHG0021 | |
| CD34 Microbead kit | Miltenyil Biotec | 30-046-702 | |
| CD34 PE clone 4H11 | Invitrogen | 12-0349-42 | |
| CD34 PerCP-eFluor 710 clone 4H11 | Invitrogen | 44-0349-42 | |
| Cellstar cell-repellent surface 6-well plates | Greiner Bio-one | 657970 | Cited as cell-repellent plate |
| CHIR99021 | Cayman Chemicals | 13122-1mg-CAY | |
| Cryostor CS10 cell cryopreservation | Merck | C2874-100ML | Cited as Cryopreservation solution |
| Dimethyl Sulfoxide | VWR | 200-664-3 | Cited as DMSO |
| DMEM/F-12 | Life Technologies | 10565-018 | |
| DPB Ca2+ Mg2+ | Life Technologies | 14080055 | |
| DPBS | Life Technologies | 14200075 | |
| EASY Strainer 40 μm | Greiner Bio-one | 542040 | |
| EDTA | Life technologies | 15575020 | |
| FcR Blocking Reagent | Miltenyil Biotec | 130-059-901 | |
| Fiji | Version 1.53c | ||
| Flow Jo | Version 10.7.1 | Cited as flow cytometry sanalysis oftware | |
| FLT3L | Peprotech | 300-19-10uG | |
| Fluidic unit | ibidi | 10903 | |
| GlutaMax | Life Technologies | 35050038 | Cited as L-glutamine supplement |
| Ham F-12 | Life Technologies | 11765054 | |
| Holo-transferrin | Merk | T0665-500MG | |
| Human Serum Albumin | Fujifilm UK LTD | 9988 | |
| Ibidi Pump system | ibidi | 10902 | Cited as Pump system |
| IMDM | Life Technologies | 12440053 | |
| Inverted microscope | ioLight/Thisle Scientific | IOL-IO-INVERT | Cited as inverted in-incubator microscope |
| Lyophilised BSA | Merck | A2153-100G | |
| MiniMACS Separator | Miltenyil Biotec | 130-042-102 | Cited as Magnetic separator |
| MS Columns | Miltenyil Biotec | 130-042-201 | Cited as Magnetic column |
| MTG | Merck | M6145-25ML | |
| N2 supplement | Life Technologies | 17502048 | |
| Notebook for pump system | ibidi | 10908 | |
| Paraformaldehyde 37-41% | Fisher Chemicals | F/1501/PB15 | |
| Pastette | Greiner Bio-one | 612398 | |
| Pen/Strep | Gibco | 15070063 | |
| Perfusion Set YELLOW/GREEN: 50 cm, ID 1.6 mm, 10 mL reservoirs | Ibidi | IB-10964 | Cited as Perfusion set |
| Polystyrene Round Bottom Tubes | Falcon | 352008 | Cited as Flow cytometry tubes |
| Prism 9 | Verison 9.4.0 | ||
| Pump control software | ibidi | version 1.6.1 | Cited as Pump software |
| ReLeSR | Stem cell tecchonologies | 5872 | Cited as Detaching solution |
| rhBMP4 | R&D | 314-BP-010 | |
| rhEPO | R&D | 287-TC-500 | |
| rhIGF1 | Peprotech | 100-11-100uG | |
| rhIL11 | Peprotech | 200-11-10uG | |
| rhIL3 | Peprotech | 200-03-10uG | |
| rhIL6 | R&D | 206-IL-010 | |
| rhLaminin-521 | Life technologies | A29248 | Cited as Laminin |
| rhSCF | Life Technologies | PHC2111 | |
| rhTPO | R&D | 288-TPN-025 | |
| rhVEGF | R&D | 293-VE-010 | |
| RLT Lysis Buffer | Qiagen | 79216 | |
| Serial Connector for µ-Slides: Sterile, Sterile | ibidi | IB-10830 | |
| StemPro-CD34 SFM media | Life Technologies | 10639011 | Cited as Serum-Free media for CD34+ cells (SFM-34) |
| StemPro-CD34 Nutrient Supplement | Life Technologies | 10641-025 | Cited as 34 nutrient supplement |
| StemPro hESC SFM | Life Technologies | A1000701 | Cited as Culture media |
| StemPro supplement | Life Technologies | A10006-01 | |
| Vitronectin (VTN-N) recombinant human protein, truncated | Invitrogen | A31804 | |
| Y-27632 dihydrochloride | Tocris | 1254 | Cited as iRock |
| β-Mercaptoethanol | Gibco | 21985023 |
Références
- Copp, A. J. Death before birth: clues from gene knockouts and mutations. Trends in Genetics. 11 (3), 87-93 (1995).
- Ji, R. P., et al. Onset of cardiac function during early mouse embryogenesis coincides with entry of primitive erythroblasts into the embryo proper. Circulation Research. 92 (2), 133-135 (2003).
- Peacock, H. M., Daems, M., Jones, E. A. V. Hemodynamic control of endothelial cell fates in development. Cardiac and Vascular Biology. 8, 127-166 (2021).
- Chong, D. C., Koo, Y., Xu, K., Fu, S., Cleaver, O. Stepwise arteriovenous fate acquisition during mammalian vasculogenesis. Developmental Dynamics. 240 (9), 2153-2165 (2011).
- Jaffredo, T., Gautier, R., Eichmann, A., Dieterlen-Lièvre, F. Intraaortic hemopoietic cells are derived from endothelial cells during ontogeny. Development. 125 (22), 4575-4583 (1998).
- Zovein, A. C., et al. Fate Tracing reveals the endothelial origin of hematopoietic stem cells. Cell Stem Cell. 3 (6), 625-636 (2008).
- Bertrand, J. Y., et al. Haematopoietic stem cells derive directly from aortic endothelium during development. Nature. 464 (7285), 108-111 (2010).
- Boisset, J. C., et al. In vivo imaging of haematopoietic cells emerging from the mouse aortic endothelium. Nature. 464 (7285), 116-120 (2010).
- Adamo, L., et al. Biomechanical forces promote embryonic haematopoiesis. Nature. 459 (7250), 1131-1135 (2009).
- Diaz, M. F., et al. Biomechanical forces promote blood development through prostaglandin E2 and the cAMP-PKA signaling axis. Journal of Experimental Medicine. 212 (5), 665-680 (2015).
- North, T. E., et al. Hematopoietic stem cell development is dependent on blood flow. Cell. 137 (4), 736-748 (2009).
- Lundin, V., et al. YAP regulates hematopoietic stem cell formation in response to the biomechanical forces of blood flow. Developmental Cell. 52 (4), 446.e5-460.e5 (2020).
- Li, J., et al. Mimicry of embryonic circulation enhances the hoxa hemogenic niche and human blood development. Cell Reports. 40 (11), 111339 (2022).
- Azzoni, E., et al. The onset of circulation triggers a metabolic switch required for endothelial to hematopoietic transition. Cell Reports. 37 (11), 110103 (2021).
- Li, Y. S. J., Haga, J. H., Chien, S. Molecular basis of the effects of shear stress on vascular endothelial cells. Journal of Biomechanics. 38 (10), 1949-1971 (2005).
- Batsivari, A., et al. Understanding hematopoietic stem cell development through functional correlation of their proliferative status with the intra-aortic cluster architecture. Stem Cell Reports. 8 (6), 1549-1562 (2017).
- Canu, G., et al. Analysis of endothelial-to-haematopoietic transition at the single cell level identifies cell cycle regulation as a driver of differentiation. Genome Biology. 21 (1), 157 (2020).
- Luo, W., et al. Arterialization requires the timely suppression of cell growth. Nature. 589 (7842), 437-441 (2020).
- Yang, C. -. T., et al. Activation of KLF1 enhances the differentiation and maturation of red blood cells from human pluripotent stem cells. Stem Cells. 35 (4), 886-897 (2017).
- Lopez-Yrigoyen, M., et al. A human iPSC line capable of differentiating into functional macrophages expressing ZsGreen: A tool for the study and in vivo tracking of therapeutic cells. Philosophical Transactions of the Royal Society B: Biological Sciences. 373 (1750), 20170219 (2018).
- Lopez-Yrigoyen, M., et al. Production and characterization of human macrophages from pluripotent stem cells. Journal of Visualized Experiments. 2020 (158), (2020).
- Fidanza, A., et al. Single cell analyses and machine learning define hematopoietic progenitor and HSC-like cells derived from human PSCs. Blood. 136 (25), 2893-2904 (2020).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- North, T. E., et al. Hematopoietic stem cell development is dependent on blood flow. Cell. 137 (4), 736-748 (2009).
- Nguyen, J., Lin, Y. Y., Gerecht, S. The next generation of endothelial differentiation: Tissue-specific ECs. Cell Stem Cell. 28 (7), 1188-1204 (2021).
- Petazzi, P., et al. Arterial cells support the development of human hematopoietic progenitors in vitro via secretion of IGFBP2. bioRxiv. , (2022).
- Crosse, E. I., et al. Multi-layered spatial transcriptomics identify secretory factors promoting human hematopoietic stem cell development. Cell Stem Cell. 27 (5), 822 (2020).
- Calvanese, V., et al. Mapping human haematopoietic stem cells from haemogenic endothelium to birth. Nature. 604 (7906), 534-540 (2022).
- Zeng, Y., et al. Tracing the first hematopoietic stem cell generation in human embryo by single-cell RNA sequencing. Cell Research. 29 (11), 881-894 (2019).
- Hwa, J. J., et al. Abnormal arterial-venous fusions and fate specification in mouse embryos lacking blood flow. Scientific Reports. 7 (1), 11965 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon