Method Article
Isolement de vecteurs viraux adéno-associés au moyen d’un protocole de chromatographie d’affinité d’héparine en une seule étape et semi-automatisé
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce manuscrit décrit un protocole détaillé pour la génération et la purification de vecteurs viraux adéno-associés à l’aide d’une méthode de chromatographie d’affinité optimisée basée sur l’héparine. Il s’agit d’une approche simple, évolutive et rentable, qui élimine le besoin d’ultracentrifugation. Les vecteurs résultants présentent une pureté et une activité biologique élevées, ce qui prouve leur valeur dans les études précliniques.
Résumé
Le virus adéno-associé (AAV) est devenu un vecteur de plus en plus précieux pour l’administration de gènes in vivo et fait actuellement l’objet d’essais cliniques sur l’homme. Cependant, les méthodes couramment utilisées pour purifier les AAV utilisent l’ultracentrifugation par gradient de densité au chlorure de césium ou à l’iodixanol. Malgré leurs avantages, ces méthodes prennent du temps, ont une évolutivité limitée et aboutissent souvent à des vecteurs de faible pureté. Pour pallier ces contraintes, les chercheurs s’intéressent aux techniques de chromatographie. Nous présentons ici un protocole de chromatographie d’affinité optimisé basé sur l’héparine qui sert d’étape de capture universelle pour la purification des AAV.
Cette méthode repose sur l’affinité intrinsèque du sérotype 2 de l’AAV (AAV2) pour les protéoglycanes de sulfate d’héparane. Plus précisément, le protocole implique la co-transfection de plasmides codant pour les protéines de capside AAV souhaitées avec celles d’AAV2, produisant des vecteurs AAV mosaïques qui combinent les propriétés des deux sérotypes parentaux. En bref, après la lyse des cellules productrices, un mélange contenant des particules d’AAV est directement purifié selon un protocole de chromatographie d’affinité d’héparine optimisé en une seule étape à l’aide d’un système standard de chromatographie liquide à protéines rapides (FPLC). Les particules d’AAV purifiées sont ensuite concentrées et soumises à une caractérisation complète en termes de pureté et d’activité biologique. Ce protocole offre une approche simplifiée et évolutive qui peut être réalisée sans avoir besoin d’ultracentrifugation et de gradients, ce qui permet d’obtenir des titres viraux nets et élevés.
Introduction
Le vecteur viral adéno-associé (AAV) est en train de se frayer un chemin comme l’un des systèmes d’administration les plus prometteurs dans les études actuelles sur la thérapie génique. Initialement identifié en 19651, l’AAV est un petit virus non enveloppé, avec une capside protéique icosaédrique d’environ 25 nm de diamètre, hébergeant un génome d’ADN simple brin. Les AAV appartiennent à la famille des Parvoviridae et au genre Dependoparvovirus en raison de leur dépendance unique à la co-infection avec un virus auxiliaire, tel que le virus de l’herpès simplex ou, plus fréquemment, l’adénovirus, pour compléter leur cycle lytique 2,3.
Le génome de 4,7 kilobases des AAV se compose de deux cadres de lecture ouverts (ORF) flanqués de deux répétitions terminales inversées (ITR) qui forment des extrémités en épingle à cheveux caractéristiques en forme de T4. Les ITR sont les seuls éléments cis essentiels pour l’emballage, la réplication et l’intégration des AAV, étant donc les seules séquences AAV conservées dans les vecteurs AAV recombinants (rAAV). Dans ce système, les gènes nécessaires à la production du vecteur sont fournis séparément, en trans, ce qui permet au gène d’intérêt d’être emballé à l’intérieur de la capside virale 5,6.
Chaque gène viral codifie différentes protéines par le biais d’épissages alternatifs et de codons de départ. Au sein de l’ORF Rep, quatre protéines non structurales (Rep40, Rep52, Rep68 et Rep78) sont codées, jouant un rôle crucial dans la réplication, l’intégration spécifique au site et l’encapsulation de l’ADN viral 7,8. Le Cap ORF sert de modèle pour l’expression de trois protéines structurelles différentes les unes des autres à leur N-terminale (VP1, VP2 et VP3), qui s’assemblent pour former une capside virale de 60 mères dans un rapport de 1:1:10 4,9. De plus, un ORF alternatif imbriqué dans le gène Cap avec un codon de départ CUG non conventionnel code pour une protéine d’activation de l’assemblage (AAP). Il a été démontré que cette protéine nucléaire interagit avec les protéines de capside VP1-3 nouvellement synthétisées et favorise l’assemblage de la capside10,11.
Les différences dans la séquence d’acides aminés de la capside expliquent les 11 sérotypes AAV naturels et plus de 100 variantes isolées dans les tissus humains et non humains 7,12,13. Les variations de conformation des régions structurellement variables régissent les propriétés antigéniques distinctes et les spécificités de liaison aux récepteurs des capsides de différentes souches. Il en résulte des tropismes tissulaires distincts et des efficacités de transduction entre différents organes de mammifères14.
Les premières méthodes de production des rAAV reposaient sur l’infection à adénovirusà des fins d’aide 15,16,17,18,19. Bien qu’elle soit efficace et généralement facile à produire à grande échelle, plusieurs problèmes découlent de cette infection. Même après la purification et une étape de dénaturation thermique pour l’inactivation, des particules adénovirales peuvent toujours être présentes dans les préparations AAV, ce qui constitue un problème de sécurité indésirable20. De plus, la présence de protéines adénovirales dénaturées est inacceptable pour un usage clinique. D’autres stratégies de production tirent parti des souches recombinantes du virus de l’herpès simplex conçues pour apporter le Rep/Cap et le transgène dans les cellules cibles21 ou du système cellulaire baculovirus-insecte22. Bien que ces systèmes offrent des avantages en termes d’évolutivité et de compatibilité GMP, ils sont toujours confrontés à des problèmes similaires.
La méthode de triple transfection pour la production de rAAV a été couramment adoptée pour surmonter facilement ces problèmes. En bref, l’assemblage du rAAV repose sur la transfection transitoire de cellules avec trois plasmides codant pour : 1) la cassette d’expression du transgène emballée entre les ITR du génome AAV2 de type sauvage (pITR) ; 2) les séquences Rep/Cap nécessaires à la réplication et à l’assemblage des virions (pAAV-RC) ; et 3) les protéines adénovirales minimales (E1A, E1B, E4 et E2A) ainsi que les ARN associés au virus adénoviral nécessaires à l’effet auxiliaire (pHelper)6,20,23. Bien que les méthodes de transfection plasmidique offrent simplicité et flexibilité pour la production de rAAV dans les études précliniques, ces procédures présentent des limites en termes d’évolutivité et de reproductibilité lorsqu’elles sont appliquées à une production à grande échelle. En tant qu’approche alternative, la production de rAAV peut être réalisée par l’utilisation de lignées cellulaires productrices d’AAV (à croissance adhérente et en suspension), exprimant de manière stable soit les gènes AAV Rep/Cap, soit Rep/Cap en combinaison avec des constructions vectorielles. Dans ces systèmes, les gènes adénoviraux auxiliaires sont introduits par transfection plasmidique. Même si cette stratégie améliore l’évolutivité du processus de culture cellulaire, elle est techniquement complexe et prend du temps 21,24,25.
Dans les deux cas, les cellules productrices sont ensuite lysées et soumises à une ou plusieurs étapes de purification. À l’heure actuelle, les principales méthodes de purification des rAAV comprennent l’utilisation de la centrifugation à gradient de densité à ultra-haute vitesse au chlorure de césium (CsCl) ou à l’iodixanol suivie ou non de techniques de chromatographie26. Le schéma de purification original pour la précipitation virale utilisait du sulfate d’ammonium, suivi de deux ou trois cycles d’ultracentrifugation à travers un gradient de CsCl. Les principaux avantages de ce processus comprennent la possibilité de purifier tous les sérotypes et la possibilité de séparer physiquement les particules pleines des capsides vides en fonction de leurs différentes densités. Cependant, cette méthode est élaborée, prend du temps et a une évolutivité limitée, ce qui entraîne souvent un faible rendement et une faible qualité d’échantillon 27,28,29,30. De plus, la dialyse contre un tampon physiologique est souvent nécessaire avant les études in vivo en raison des effets toxiques que le CsCl peut exercer sur les mammifères.
L’iodixanol a également été utilisé comme milieu alternatif à gradient iso-osmotique pour purifier les vecteurs rAAV, avec des avantages par rapport au CsCl du point de vue de la sécurité et de la puissance du vecteur. Cependant, comme CsCl, la méthode iodixanol présente quelques inconvénients liés à la capacité de charge du lysat de culture cellulaire (et donc à l’évolutivité de la purification rAAV) et elle reste une méthode longue et coûteuse30,31.
Pour pallier ces contraintes, les chercheurs se sont tournés vers les techniques de chromatographie. À cet égard, plusieurs approches de purification ont été développées qui intègrent des méthodes de chromatographie d’affinité, hydrophobes ou d’échange d’ions. Ces méthodes reposent sur les propriétés biochimiques d’un sérotype particulier, y compris leurs récepteurs naturels, ou sur les caractéristiques de charge de la particule virale32. Par exemple, la découverte que AAV2, AAV3, AAV6 et AAV13 se lient de préférence aux protéoglycanes de sulfate d’héparane (HSPG), a ouvert la possibilité d’utiliser l’héparine étroitement apparentée dans la purification par chromatographie d’affinité. Cependant, les sites de liaison à la HSPG peuvent différer selon les sérotypes, médiant la fixation de l’AAV et l’infection des cellules cibles de différentes manières 2,33,34,35,36. D’autre part, AAV1, AAV5 et AAV6 se lient à l’acide sialique (SA) lié à l’azote, tandis que l’AAV4 utilise le SA 2,14,34 lié à l’O. Suivant le même raisonnement, un protocole de chromatographie d’affinité en une seule étape pour la purification de rAAV5 a également été développé basé sur l’utilisation de mucine, une protéine de mammifère fortement enrichie en SA37. Comme les techniques à base d’héparine, cette purification dépend également du sérotype spécifique généré. Outre l’héparine et la mucine, d’autres ligands ont été explorés pour la chromatographie d’affinité, tels que l’anticorps monoclonal A20 et les anticorps à domaine unique des camélidés (AVB Sepharose et POROS CaptureSelect)22,23,38,39,40,41. D’autres stratégies innovantes pour améliorer les méthodes de purification existantes impliquent l’introduction de petites modifications dans la capside du rAAV pour présenter des épitopes de liaison spécifiques. Par exemple, les rAAV marqués à l’hexa-histidine ou biotinylés peuvent être purifiés à l’aide de ligands qui ciblent ces épitopes (résines de nickel nitrilotriacétique et d’avidine, respectivement)42,43,44.
Dans le but d’élargir les caractéristiques souhaitées des rAAV, les chercheurs ont travesti les virions en mélangeant leurs capsides. Ceci est accompli en fournissant le gène de la capside à partir de deux sérotypes AAV distincts dans des rapports équimolaires ou différents au cours de la production, donnant naissance à une structure de capside composée d’un mélange de sous-unités de capside de différents sérotypes 34,45,46,47,48,49,50 . Des études antérieures fournissent des preuves physiques que la co-expression des protéines de capside d’AAV2 avec AAV1 (rapport 1:1) et d’AAV2 avec AAV9 (rapport 1:1) entraîne la génération de vecteurs mosaïques rAAV1/2 et rAAV2/9, respectivement45, 46, 48. L’un des principaux avantages de la génération de rAAV en mosaïque est la capacité d’intégrer des traits avantageux de différents sérotypes d’AAV, ce qui entraîne des améliorations synergiques de l’expression des transgènes et du tropisme, tout en conservant d’autres propriétés utiles lors de la production de rAAV. Il est intéressant de noter que certaines variantes de la mosaïque présentent même de nouvelles propriétés différentes de celles des virus parentaux 46,47,49. En tirant parti de la capacité de liaison à l’héparine de l’AAV2, les vecteurs rAAV en mosaïque pourraient potentiellement être générés et purifiés en mélangeant AAV2 avec d’autres capsides AAV naturelles ou nouvelles générées par l’évolution dirigée et/ou la conception rationnelle. Néanmoins, des études antérieures ont souligné l’importance de la compatibilité des sous-unités de la capside lors de l’assemblage de vecteurs mosaïques. Par exemple, Rabinowitz et ses collègues ont démontré que bien que la transcapsidation d’AAV1, d’AAV2 et d’AAV3 conduise à un co-assemblage efficace des capsides en mosaïque, le travestissement de ces sérotypes avec AAV4 entrave la génération de virions stables 34,45,47. De plus, AAV1, AAV2 et AAV3 ont montré une faible compatibilité avec AAV5, compte tenu de la réduction des titres viraux obtenus lors du mélange de ces capsides à des ratios différents. Fait intéressant, la mosaïque rAAV2/5 a montré une diminution des propriétés de liaison à l’héparine, tout en maintenant la capacité de liaison à la mucine comme l’AAV5 parental. Cependant, rAAV3/5 à un rapport de 3:1 a préservé la double liaison à l’héparine et à la mucine. Dans l’ensemble, la génération de nouveaux rAAV en mosaïque avec une transduction améliorée, un tropisme spécifique ou une faible immunogénicité pourrait grandement bénéficier de notre compréhension de l’assemblage de la capside et des interactions récepteurs, les combinaisons spécifiques nécessitant encore une étude et une optimisation approfondies.
Dans ce travail, nous décrivons un protocole étape par étape pour la production et la purification des rAAV à l’aide d’une méthode de chromatographie d’affinité d’héparine optimisée. Les rAAV sont produits par transfection transitoire et sont purifiés à l’aide d’un système de chromatographie liquide rapide en protéines (FPLC). Après la concentration de fractions purifiées sélectionnées, les stocks viraux résultants sont caractérisés en termes de titre, de pureté, de propriétés physiques intrinsèques et d’activité biologique in vitro et in vivo. En tant que preuve de concept, nous démontrons les améliorations et l’applicabilité de ce protocole pour la génération de vecteurs mosaïques rAAV1/2 et rAAV2/9. Le choix de chaque sérotype a été basé sur leurs tropismes étonnamment différents, conférant potentiellement leurs caractéristiques uniques aux versions mosaïques. Le sérotype AAV 1, avec un tropisme global modéré pour le système nerveux central (SNC), a la capacité de transduire les neurones et la glie (dans une moindre mesure) et subit un transport axonal dans les directions antérogrades et rétrogrades in vivo 2,7,8. De plus, le sérotype 9 de l’AAV a été choisi pour sa capacité naturelle à traverser la barrière hémato-encéphalique et à cibler le SNC chez les souris néonatales et adultes51,52. Enfin, le sérotype 2 de l’AAV a été sélectionné en raison de sa capacité à se lier à l’héparine, ce qui permet une chromatographie d’affinité33. Les particules purifiées rAAV1/2 et rAAV2/9 combinent les propriétés des deux sérotypes parentaux AAV et, par conséquent, constituent des vecteurs appropriés pour la transduction du SNC 45,46,48,49.
Protocole
REMARQUE : Voir la figure 1 pour une illustration résumant le protocole. Voir le tableau des matériaux pour plus de détails sur tous les matériaux, instruments et réactifs utilisés dans ce protocole. Tous les travaux impliquant des cellules et des virus doivent être effectués dans des enceintes et des incubateurs de biosécurité dédiés, séparés de ceux régulièrement utilisés pour la maintenance des lignées cellulaires. L’équipement et les réactifs entrant en contact avec les cellules cultivées et les virus doivent être stériles. Il est essentiel que l’élimination des réactifs dangereux et des matières contaminées par des virus soit effectuée conformément aux fiches de données de sécurité et aux lois et directives nationales fournies par le bureau de l’environnement, de la santé et de la sécurité de chaque établissement. En avril 2019, les lignes directrices des NIH pour la recherche impliquant des molécules d’acide nucléique recombinantes ou synthétiques classent comme agents du groupe de risque 1 (non associés à la maladie chez les humains adultes en bonne santé) tous les sérotypes d’AAV, ainsi que les constructions d’AAV recombinantes ou synthétiques. Cette classification s’applique lorsque le transgène ne code pas pour un produit génique potentiellement tumorigène ou une toxine, et que les constructions sont produites sans virus auxiliaire.
Toutes les expériences sur des animaux ont été réalisées dans le respect de la directive de la Communauté européenne (2010/63/UE) sur le soin et l’utilisation des animaux de laboratoire, transposée dans la loi portugaise en 2013 (décret-loi 113/2013). De plus, les procédures animales ont été approuvées par l’Organisation responsable pour le bien-être animal de la Faculté de médecine et le Centre de neurosciences et de biologie cellulaire de l’animalerie agréée de l’Université de Coimbra. Les chercheurs ont reçu une formation adéquate (cours certifié par la FELASA) et une certification des autorités portugaises (Direcção Geral de Alimentação e Veterinária, Lisbonne, Portugal) pour réaliser les expériences.
1. Constructions plasmidiques
- Suivez les instructions du fabricant d’une trousse sans endotoxines maxiprep pour isoler et purifier des quantités substantielles d’ADN des plasmides suivants : i) pITR : le vecteur de transfert d’intérêt ; ii) plasmide pAAV-RC : pRV1, contenant les séquences AAV2 Rep et Cap 36 ; iii) plasmide pAAV-RC : pH21, contenant les séquences AAV1 Rep et Cap 36 ; iv) plasmide pAAV-RC : pAAV2/9n, contenant les séquences AAV2 Rep et AAV9 Cap ; v) pHelper : pFdelta6, plasmide36 auxiliaire de l’adénovirus.
- Examinez l’intégrité des plasmides générés en appliquant les restrictions enzymatiques recommandées36. Surveiller l’intégrité des plasmides pITR par digestion avec SmaI, une endonucléase de restriction, qui coupe deux fois dans une portion instable des RTI 53,54.
REMARQUE : Étant donné que les ITR sont très instables et sensibles aux délétions, l’utilisation de cellules supercompétentes SURE 2 est recommandée pour minimiser la recombinaison dans ces sites.
2. Culture cellulaire
- Cultiver un rein embryonnaire humain 293, exprimant de manière stable la lignée cellulaire du grand antigène T (HEK293T) SV40 dans le milieu modifié Eagle Medium (DMEM) de Dulbecco, avec un taux de glucose élevé de 10 % de fœtus de bovin et 1 % de pénicilline-streptomycine, à 37 °C, sous une atmosphère humidifiée contenant 5 % de CO2, comme point de départ pour les étapes suivantes.
- Sous-cultivez les cellules à l’aide d’une solution saline tamponnée au phosphate (PBS) stérile, pH 7,4, pour laver les cellules avant d’ajouter 0,05 % d’acide trypsine-éthylènediaminetétraacétique (EDTA).
REMARQUE : Évitez d’utiliser des cellules qui ont subi un nombre excessif de passages (maximum 20). Testez régulièrement les cultures cellulaires pour détecter la contamination par les mycoplasmes.
3. Production de rAAV par transfection transitoire
- Jour 1 : Placage cellulaire
- Pour chaque production virale, déposer HEK293T les cellules dans 10 boîtes de culture traitées (15 cm de diamètre) la veille de la transfection, à une densité de 10,5 × 106 cellules par boîte dans 22 mL de milieu de culture supplémenté et incuber pendant 24 h jusqu’à ce que les cellules soient confluentes à 70 % à 80 % et prêtes pour la transfection.
- Jour 2 : Transfection cellulaire à l’aide de polyéthylénimine (PEI)
- Pour chaque production virale (équivalant à 10,5 boîtes), mettre en place le mélange de transfection suivant dans un tube de microcentrifugation : 54,6 μg de pITR ; 45,675 μg de pRV1 ; 45,675 μg de pH21 ou pAAV2/9n ; 109,2 μg de pFdelta6. Mélanger en tapotant
- Ajouter le mélange à 4,557 mL de DMEM non supplémenté dans un tube à centrifuger de 50 mL. Mélanger en tapotant
- Ajouter 1,365 mL de solution stérile d’IPE à 1 mg/mL (pH 7,4), goutte à goutte. Mélanger en tapotant Incuber pendant 10 min à température ambiante pour permettre la formation de complexes ADN-PEI.
- Ajouter ce mélange à 231 ml de DMEM supplémenté préchauffé. Remplacer tout le milieu de culture de chaque plat par 22 mL de ce mélange de transfection. Incuber les cellules pendant 48 h.
REMARQUE : Cette étape doit être effectuée avec soin pour éviter le détachement des cellules.
- Jour 4 : Récolte de cellules
- Lorsque le pITR code un rapporteur fluorescent, visualisez les cellules transfectées au microscope à fluorescence.
- Recueillir le milieu de chaque plat dans des tubes à centrifuger de 50 mL et les centrifuger à 800 × g pendant 10 min. Jetez le surnageant.
REMARQUE : Cette étape est facultative et vise à récupérer les cellules transfectées qui peuvent s’être détachées en raison d’une très forte confluence. - Ajouter 10 ml de PBS préchauffé dans chaque assiette. Retirez délicatement les cellules à l’aide d’un grattoir à cellules et recueillez la suspension dans les tubes à centrifuger de 50 ml à partir de l’étape 3.3.2.
- Lavez 5 plats à la fois avec 10 ml de PBS supplémentaires et transférez la suspension dans les tubes à centrifuger de 50 ml à partir de l’étape 3.3.3. Granulez les cellules à 800 × g pendant 10 min et jetez le surnageant.
- Congelez les granulés de cellule à -80 °C.
REMARQUE : Les granulés de cellules peuvent être conservés pendant plusieurs mois (point de pause).
4. Extraction rAAV et purification FPLC
- Jour 5 : Préparation du lysat cellulaire
- Décongelez les granulés de cellules à température ambiante. Remettre en suspension les cellules prélevées dans les 10 boîtes dans 100 mL d’un tampon stérile contenant 150 mM de chlorure de sodium (NaCl) et 20 mM de Tris, pH 8,0, dans de l’eau ultrapure (type I). Mélanger la suspension par pipetage de haut en bas, pour assurer une suspension homogène.
- Ajouter 12,5 mL d’une solution stérile fraîchement préparée de désoxycholate de sodium à 10 % dans de l’eau ultrapure pour induire la lyse cellulaire. Mélanger par pipetage de haut en bas.
REMARQUE : Éliminer le désoxycholate de sodium, ainsi que les matières qui y sont en contact, conformément à la fiche signalétique et aux lignes directrices fournies par le bureau de l’hygiène et de la sécurité environnementales de l’établissement. Il est également recommandé de porter un masque facial lors de la manipulation de cette poudre. Après mélange, la solution devient très visqueuse. - Ajouter 27 μL de nucléase de benzonase au mélange précédent. Mélangez soigneusement en pipetant de haut en bas jusqu’à ce que l’échantillon ne soit plus visqueux. Incuber à 37 °C pendant 1 h, en effectuant un vortex toutes les 10 min.
REMARQUE : Cette endonucléase est capable de dégrader efficacement toutes les formes d’ADN et d’ARN sans présenter d’activité protéolytique. - Éliminer les débris cellulaires en centrifugeant le mélange à 3 000 × g pendant 60 min à 25 °C. Filtrez le surnageant à l’aide d’un filtre à seringue stérile en polyfluorure de vinylidène (PVDF) de 0,45 μm et transférez-le dans un nouveau récipient stérile.
REMARQUE : Cette étape importante garantit que la plupart des débris cellulaires sont éliminés, empêchant ainsi le colmatage des colonnes de chromatographie. Conservez une petite partie aliquote de ce mélange pour l’analyse (étape facultative).
- Jour 5 (suite) : Purification de la colonne d’héparine des rAAV
REMARQUE : L’application de l’échantillon peut être effectuée à l’aide d’une pompe d’échantillonnage ou d’un superloop de 50 ml ou 150 ml dans le cadre du système. Étant donné qu’une plus grande quantité d’air est dissoute à des températures plus basses, il est important de laisser suffisamment de temps aux tampons et aux solutions (généralement stockés à 4 °C) pour s’acclimater à la température ambiante avant de les utiliser dans le système FPLC.- Facultatif : Si le système a été stocké pendant de longues périodes, remplissez le système et toutes les entrées avec une solution de stockage fraîchement préparée (éthanol à 20 %) en suivant des instructions manuelles ou une méthode de nettoyage en place prédéfinie du système (NEP du système).
- Lavez complètement le chemin d’écoulement du liquide avec de l’eau ultrapure stérile à l’aide d’instructions manuelles ou d’une méthode CIP prédéfinie du système.
- Connectez une colonne d’héparine préemballée de 1 ml, réglez l’alarme de pression et lavez avec cinq volumes de colonne (CV) d’eau ultrapure à un débit de 1 mL/min.
- Basculez les solutions dans le bac tampon de l’eau ultrapure au tampon A (solution stérile de 100 mM de NaCl et 20 mM de Tris, pH 8, dans de l’eau ultrapure) pour l’entrée A (pompe du système A) et du tampon B (solution stérile de 500 mM de NaCl et 20 mM de Tris, pH 8, dans de l’eau ultrapure) pour l’entrée B (pompe du système B). Si le système est équipé d’une pompe d’échantillonnage, placez l’entrée du tampon de la vanne d’entrée d’échantillon dans le tampon A.
- Lavez la pompe du système B avec le tampon B et remplissez le reste du chemin d’écoulement du liquide avec le tampon A.
REMARQUE : Si nécessaire, déconnectez la colonne du chemin d’écoulement et reconnectez-la par la suite. - Insérez un tube d’entrée d’échantillon à partir de la vanne d’entrée d’échantillon, par exemple S1, dans le récipient contenant la préparation virale obtenue à l’étape 4.1.4. (à partir de la préparation de lysat cellulaire). Amorcez le chemin d’écoulement de l’entrée de l’échantillon S1 à la vanne d’injection avec la solution de l’échantillon. Vous pouvez également remplir une superloop de 50 mL ou 150 mL avec l’échantillon contenant des rAAV à l’aide d’une seringue de 50 mL.
- Équilibrer la colonne avec un volume total de cinq CV en utilisant 12,5 % de tampon B à un débit de 1 mL/min.
- Appliquez le volume total de l’échantillon dans la colonne à l’aide de la pompe d’échantillonnage (sélectionnez injecter tout l’échantillon à l’aide du capteur d’air) ou du superloop à 0,5 mL/min et recueillez l’écoulement à l’aide de l’orifice de sortie dans un nouveau récipient stérile.
REMARQUE : Lorsque la fonction de contrôle de débit pour éviter la surpression est activée, le débit diminuera automatiquement en cas de colmatage de la colonne. Si le débit descend considérablement en dessous de 0,5 mL/min, arrêtez l’application de l’échantillon, effectuez un lavage avec 2 à 5 CV de tampon A, puis reprenez l’application de l’échantillon. - Laver la colonne à 1 mL/min avec 20 CV de tampon A, en recueillant le flux à l’aide de l’orifice de sortie.
- Éluer l’échantillon à 1 mL/min avec le schéma suivant : i) gradient linéaire avec une cible de 50 % du tampon B pour cinq CV ; ii) étape avec un objectif de 90 % de la zone tampon B pendant cinq CV ; iii) étape avec un objectif de 100 % de la zone tampon B pendant cinq CV.
- Prélever l’échantillon élué en fractions de 1 mL à l’aide d’un collecteur de fractions et de tubes de microcentrifugation à faible rétention (2 mL) et les stocker à -20 °C.
REMARQUE : Les fractions rAAV peuvent être conservées pendant plusieurs semaines (point de pause). - Rééquilibrer la colonne à 1 mL/min avec 12,5 % de tampon B pour cinq CV.
- Remplacez les entrées des solutions tampons par de l’eau ultrapure et lavez la colonne à 1 mL/min pendant cinq CV.
- Passez de l’eau ultra-pure à l’éthanol à 20 % et lavez la colonne à 1 mL/min pendant cinq CV. Débranchez la colonne et stockez-la à 4 °C.
REMARQUE : Les colonnes peuvent être réutilisées plusieurs fois sans aucune autre procédure de nettoyage et d’assainissement majeure si le même sérotype et le même transgène rAAV sont utilisés. - Lavez complètement le chemin d’écoulement du liquide avec de l’éthanol à 20 % en suivant les instructions manuelles ou une méthode CIP prédéfinie du système.
5. Concentration des rAAV purifiés
- Jour 6 : Étape de concentration 1
- Concentrez les rAAV à l’aide d’une unité de filtration centrifuge de 15 mL avec une coupure de poids moléculaire de 100 kDa. Chargez les fractions souhaitées contenant des rAAV (fractions FPLC 7 à 16) dans l’unité de filtration centrifuge de 15 mL et centrifugez-la à 2 000 × g pendant 2 min à température ambiante. Assurez-vous que le volume concentré dans l’unité de filtration est d’environ 500 μL. Si le volume concentré dépasse largement 500 μL, répétez les étapes de centrifugation à intervalles de 1 minute jusqu’à ce qu’il atteigne le volume souhaité.
- Jour 6 (suite) : Échange de tampons
- Ajouter 1 mL de PBS stérile dans l’unité de filtration centrifuge contenant les rAAV. Pipetez soigneusement de haut en bas pour laver le filtre. Centrifuger à 2 000 × g par intervalles de 1 min jusqu’à ce que le volume final de 500 μL soit atteint.
- Jour 6 (suite) : Étape de concentration 2
- Transvaser les 500 μL de rAAV concentrés obtenus à l’étape précédente dans une unité de filtration centrifuge de 0,5 mL avec une coupure de poids moléculaire de 100 kDa et centrifuger à 6 000 × g pendant 1 min. Si nécessaire, répétez l’étape de centrifugation jusqu’à ce qu’un volume final inférieur à 100 μL soit atteint.
- Jour 6 (suite) : Récupération
- Pour récupérer le rAAV concentré, placez le dispositif de filtration à l’envers dans un nouveau tube de prélèvement de microcentrifugeuse. Placez le tube dans une microcentrifugeuse avec le capuchon vers le centre et effectuez une rotation prolongée à l’intérieur de la chambre d’écoulement pour transférer les rAAV concentrés de l’appareil vers le tube de la microcentrifugeuse. Vous pouvez également centrifuger à 1 000 × g pendant 2 min.
- Supplément avec du Pluronic F-68 stérile 0,001% (facultatif).
REMARQUE : Pluronic F-68 est un tensioactif non ionique approuvé pour un usage humain par la Food and Drug Administration qui est capable d’atténuer les pertes de rAAV en empêchant leurs interactions avec les surfaces des matériaux (plastiques) utilisés lors de la préparation de la dilution, du chargement de la seringue et de l’équipement d’administration55,56. - Aliquote des rAAV dans des tubes de microcentrifugation à faible rétention et stockez-les à -80 °C (point de pause).
6. Quantification des rAAV purifiés
- Jour 6 (suite) : Déterminer le titre des préparations de rAAV, exprimé en génomes viraux/μL (vg/μL), par réaction en chaîne par polymérase quantitative en temps réel (RT-qPCR) à l’aide d’un kit commercial et en suivant les instructions du fabricant.
- Incuber la solution de particules rAAV avec la DNase I à 37 °C pendant 20 min.
REMARQUE : Cette procédure favorise la digestion de l’ADN génomique libre et de l’ADN plasmidique dérivé des cellules hôtes, garantissant ainsi que seule la séquence d’acide nucléique à l’intérieur des particules intactes de rAAV est préservée. - Inactiver thermiquement la DNase I à 95 °C pendant 10 min.
- Ajouter le tampon de lyse et incuber pendant 10 min à 70 °C pour favoriser la dénaturation thermique des protéines des particules rAAV.
- Diluer la solution du génome rAAV obtenue dans un tampon de dilution avant de passer à la RT-qPCR. Préparer un ensemble d’étalons dilués en série du témoin positif (de 2 × 107 vg/μL à 2 ×10 2 vg/μL) fournis avec le kit.
- Effectuer un mélange réactionnel contenant 12,5 μL de mélange Taq II, 0,5 μL de mélange d’amorces diluées, 7 μL d’eau et 5 μL d’ADN rAAV dilué (échantillon et étalons AAV inconnus de l’étape 6.1.4.).
- Effectuez la RT-qPCR dans un système de détection PCR en temps réel, en utilisant le protocole suivant : 1 cycle à 95 °C pendant 2 min (dénaturation initiale) et 40 cycles à 95 °C pendant 5 s (dénaturation) et 60 °C pendant 30 s (recuit, extension et lecture de plaque), suivi d’une analyse de la courbe de fusion.
- Calculer la concentration absolue de l’échantillon à partir de la courbe standard (droite de régression linéaire), en tenant compte du facteur de dilution résultant de la préparation de l’échantillon rAAV.
REMARQUE : La quantification du nombre de génomes viraux est réalisée par l’amplification de la séquence ITR d’AAV2 (séquence cible des amorces fournies par le kit).
- Incuber la solution de particules rAAV avec la DNase I à 37 °C pendant 20 min.
7. SDS-PAGE, coloration au bleu Coomassie et western blot
- Dénaturer 40 μL de chaque échantillon (chaque fraction de FPLC ; le flux continu ; les échantillons de précolonne et un total de 2,3 × 1010 vg du produit concentré final) en ajoutant 6 tampons d’échantillon (0,5 M de Tris-HCl/0,4 % de dodécylsulfate de sodium (SDS) pH 6,8, 30 % de glycérol, 10 % de SDS, 0,6 M de dithiothréitol (DTT), 0,012 % de bleu de bromophénol) et en incubant les échantillons pendant 5 min à 95 °C.
- Charger les échantillons dénaturés (48 μL) dans un gel de polyacrylamide SDS (gel d’empilement à 4 % et gel de résolution à 10 %) et effectuer la séparation électrophorétique à 100 V pendant 70 min, à côté d’une échelle protéique.
- Analyse des protéines
REMARQUE : Une coloration au bleu de Coomassie ou un western blot peut être effectué.- Coloration au bleu de Coomassie
- Pour visualiser les bandes de protéines, colorez le gel pendant 10 minutes avec une solution de bleu de Coomassie G250 à 0,25 % dissoute dans du méthanol à 50 % et de l’acide acétique glacial à 10 %.
- Effectuez la décoloration du gel en le lavant plusieurs fois avec une solution contenant 25% de méthanol et 5% d’acide acétique glacial jusqu’à ce que des bandes claires avec un faible bruit de fond deviennent visibles.
- Capturez des images à l’aide d’un système d’imagerie approprié.
- Transfert Western
- Transférez les protéines sur une membrane PVDF selon les protocoles standard.
- Bloquer la membrane par incubation dans du lait écrémé à 5 % dilué dans du TBS-T (0,1 % de Tween 20 dans une solution saline tamponnée au Tris) pendant 1 h à température ambiante.
- Utiliser les anticorps primaires suivants (dilués dans une solution bloquante) pour l’incubation nocturne à 4 °C : anticorps monoclonaux anti-AAV de souris, anticorps anti-AAV monoclonaux de souris, anticorps anti-AAV VP1, VP2 (B1, 1:1 000) ou anticorps monoclonaux anti-AAV de souris, VP1, anticorps anti-VP2 (A69, 1:1 000).
- Laver les membranes pendant 3 x 15 min dans du TBS-T et incuber avec un anticorps secondaire de chèvre anti-souris lié à la phosphatase alcaline (1:10 000), pendant 2 h à température ambiante.
- Laver les membranes pendant 3 x 15 min dans du TBS-T. Ajoutez un substrat de chimifluorescence amélioré (ECF) et visualisez les bandes de protéines par imagerie par chimifluorescence.
- Coloration au bleu de Coomassie
8. Microscopie électronique à transmission (MET)
- Placez une grille de 200 mailles recouverte de carbone Formvar à l’envers sur une goutte d’échantillon rAAV et laissez-la reposer pendant 1 min.
- Lavez les grilles dans une goutte d’eau et séchez l’excès de liquide avec du papier filtre.
- Colorez négativement les grilles avec une solution d’acétate d’uranyle à 1 % (pH 7) pendant 1 min pour fixer et contraster les particules virales.
- Lavez les grilles dans une goutte d’eau et séchez l’excès de liquide avec du papier filtre.
- Examinez les échantillons au microscope électronique à transmission.
REMARQUE : Des concentrations élevées de sel peuvent avoir un impact direct sur la liaison des rAAV à la grille et conduire à la visualisation de structures cristallines.
9. Analyse consécutive de l’absorption de la lumière ultraviolette-visible, de la diffusion statique de la lumière et de la diffusion dynamique de la lumière
- Sur une plaque de quantification à 96 puits, chargez 2 μL d’un échantillon rAAV et 2 μL de PBS à utiliser comme blanc tampon (effectuez cette opération en doubles).
- Utilisez l’application AAV Quant dans le logiciel d’analyse du client, placez les noms des échantillons au bon endroit de la plaque, sélectionnez le sérotype AAV et cliquez sur Suivant.
- Chargez la plaque de quantification à 96 puits dans l’équipement dédié et procédez à la lecture de la plaque pour l’acquisition des données.
10. Essais de transduction in vitro
- Différentes lignées cellulaires peuvent être utilisées pour analyser rapidement l’efficacité de transduction des rAAV.
- Ensemencer uniformément HEK293T cellules dans des plaques à 24 puits (à une densité de 137 500 cellules/puits) et une lignée cellulaire de neuroblastome-2A de souris (Neuro2a) dans des plaques à 24 puits (50 000 cellules/puits) ou dans une lame à 8 puits (27 000 cellules/puits), en utilisant du DMEM à haute teneur en glucose, complété par 10 % de sérum de veau fœtal et 1 % de pénicilline-streptomycine, comme décrit ci-dessus. Laisser les cellules adhérer toute la nuit à 37 °C dans une atmosphère humidifiée contenant 5 % de CO2.
- Prélever le milieu conditionné de chaque puits (250 μL des plaques à 24 puits et 50 μL de la lame de chambre à 8 puits) et le stocker à 4 °C pour une utilisation ultérieure.
- Ajouter les préparations rAAV suivantes dans chaque puits et incuber les cellules pendant 24 h à 37 °C dans une atmosphère à 5 % de CO2 .
- Ajouter 50 μL des fractions F2-F16 recueillies par le FPLC et les faire circuler dans HEK293T cellules plaquées dans des plaques à 24 puits.
- Ajouter un total de 5,5 × 109 vg des rAAV concentrés dilués dans 50 μL de PBS aux cellules Neuro2a plaquées dans des plaques à 24 puits (inclure un puits de contrôle négatif, en ajoutant 50 μL de PBS aux cellules).
- Ajouter un total de 2,75 × 109 vg des rAAV concentrés dilués dans 25 μL de PBS aux cellules Neuro2a plaquées dans une lame de chambre à 8 puits (inclure la condition de contrôle négatif, en ajoutant 50 μL de PBS aux cellules).
- Ajouter le milieu conditionné préalablement stocké (étape 10.1.2) dans chaque puits et incuber pendant 24 h.
- Jetez le milieu et lavez les cellules 2 fois avec du PBS.
- Ajouter une solution de paraformaldéhyde (PFA) à 4 %, complétée par du saccharose à 4 % dans du PBS, préchauffé à 37 °C, dans chaque puits et incuber à température ambiante pendant 20 min.
- Laver 2 fois avec du PBS et conserver à 4 °C jusqu’à ce que l’imagerie soit effectuée (point de pause).
- Acquérez des images sur un microscope à fluorescence inversée équipé d’un objectif 10x/0,30, ou un microscope confocal inversé équipé d’un objectif 40x/1,4 Oil DIC.
- Pour avoir un modèle plus pertinent et plus représentatif de l’environnement in vivo , utilisez les cultures neuronales primaires comme suit :
- Préparer des cultures primaires de neurones corticaux comme décrit précédemment par Santos et al.57. En bref, ensemencer 200 000 cellules/mL dans des plaques à 12 puits et les maintenir en culture jusqu’au jour in vitro 16.
- Prélever le milieu conditionné de chaque puits (100 μL) et le stocker à 4 °C pour une utilisation ultérieure.
- Ajouter les rAAV à tester dans chaque puits : un total de 2,75 × 109 vg de rAAV concentrés dilués dans 25 μL de PBS (inclure le témoin négatif : 25 μL de PBS). Incuber pendant 24 h à 37 °C dans une atmosphère à 5% de CO2 .
- Ajouter le milieu conditionné préalablement stocké et incuber pendant 24 h.
- Jetez le milieu dans chaque puits et lavez-le 2 fois avec du PBS.
- Fixer les cellules avec 4 % de PFA/4 % de saccharose dans du PBS, comme décrit à l’étape 10.1.6. Laver 2 fois avec du PBS.
- Incuber chaque puits avec 5 μg/mL d’agglutinine de germe de blé (WGA) conjuguée à Alexa Fluor 633 pendant 10 min à température ambiante (étape facultative : effectuer plutôt une immunocytochimie). Laver 2 fois avec du PBS.
- Incuber dans 0,25% de Triton X-100 dans du PBS pendant 5 min à température ambiante. Laver avec du PBS.
- Incuber avec du 4',6-diamidino-2-phénylindole (DAPI) pendant 5 min à température ambiante. Laver 2 fois avec du PBS.
- Acquérez des images à l’aide d’un microscope à fluorescence inversée équipé d’un objectif 40x/0,95 ou d’un microscope confocal inversé équipé d’un objectif 40x/1,4 Oil DIC.
11. Expériences in vivo
REMARQUE : Les animaux ont été logés dans une pièce à température contrôlée, maintenue selon un cycle lumière/obscurité de 12 heures. De la nourriture et de l’eau ont été fournies ad libitum. Tous les efforts ont été faits pour minimiser la souffrance animale.
- Injection stéréotaxique dans le cervelet
- Anesthésier des animaux C57BL/6 âgés de 9 semaines par inhalation d’isoflurane à 2 % en présence d’oxygène (0,8 L/min) dans une chambre reliée à un vaporisateur.
- Placez l’animal anesthésié dans l’appareil stéréotaxique (sur un tampon chauffé à 35 °C) et placez le masque à l’isoflurane dans le nez de l’animal. Baissez le niveau d’isoflurane à 1,3-1,7 %.
REMARQUE : S’assurer que l’animal est correctement anesthésié avant de procéder (perte du réflexe de flexion dans les deux membres postérieurs). - Appliquez une pommade oculaire lubrifiante pour éviter le dessèchement des cornées et injectez à l’animal un analgésique approuvé.
REMARQUE : Toutes les étapes suivantes doivent être effectuées dans des conditions stériles. - Après avoir rasé la fourrure de la tête de l’animal et désinfecté la zone chirurgicale, exposez le crâne et placez l’extrémité d’une aiguille d’injection à bout émoussé de 30 G, reliée à une seringue Hamilton de 10 μL, directement sur bregma (utilisez bregma comme zéro pour le calcul des coordonnées stéréotaxiques).
- Déplacez l’aiguille à la coordonnée prévue et percez un trou dans le crâne où l’aiguille entrera.
REMARQUE : Dans le cadre de cette étude, une seule injection a été réalisée au centre du cervelet. - Injecter 4 μL d’une solution de rAAV contenant un total de 8 × 109 vg, diluée dans du PBS, à un débit de perfusion de 0,5 μL/min à l’aide d’un injecteur automatique. Utilisez les coordonnées suivantes, calculées à partir de bregma, pour effectuer une injection unique au centre du cervelet d’une souris C57BL/6 adulte : antéro-postérieure : -6,5 mm ; latéral : 0 mm ; ventrale : -2,9 mm.
REMARQUE : Ces coordonnées peuvent varier en fonction de la souche de souris, du sexe et de l’âge des animaux utilisés. - Pour minimiser le reflux et permettre la diffusion du vecteur viral, une fois la perfusion terminée, laissez l’aiguille de la seringue à ces coordonnées pendant 3 min, puis rétractez-la lentement de 0,3 mm et laissez-la rester en place pendant 2 minutes supplémentaires avant de le retirer complètement du cerveau de la souris.
- Fermez l’incision et nettoyez-la avec un agent désinfectant (par ex. 10 % de povidone iodée).
- Laissez les animaux se remettre de l’anesthésie avant de les ramener dans leurs cages d’origine.
- Prélèvement et préparation de tissus
REMARQUE : Dans cette expérience, les niveaux de transduction ont été observés 12 semaines après l’injection, mais la même procédure a pu être évaluée dès 4 semaines après l’injection.- Anesthésier les animaux en phase terminale par l’administration intrapéritonéale d’une surdose de xylazine/kétamine (8/160 mg/kg de poids corporel).
- Perfuser les animaux par voie transcardique avec du PBS glacé pendant 6 minutes à un débit de 2,5 mL/min, suivi de la perfusion avec une solution glacée de PFA à 4 % fraîchement préparée pendant 10 minutes au même rythme.
- Postfixez les cerveaux excisés dans 4% de PFA pendant la nuit à température ambiante, puis transférez-les dans une solution de saccharose/PBS à 25% pour la cryoprotection. Une fois que la cervelle coule (environ 48 h plus tard), stockez-la à -80 °C.
- Découpez en série des coupes sagittales d’une épaisseur de 30 μm à l’aide d’un cryostat à -21 °C. Pour chaque animal, prélever 96 sections sagittales d’un hémisphère cérébral dans des séries anatomiques sous forme de sections flottantes dans du PBS complété par 0,05% d’azoture de sodium. Conserver à 4 °C jusqu’à nouvel ordre.
- Immunohistochimie par fluorescence standard
- Sélectionnez huit sections sagittales par animal, à une distance de 240 μm les unes des autres.
- Incuber les sections flottantes dans une solution de blocage/perméabilisation (0,1 % de Triton X-100 contenant 10 % de sérum de chèvre normal (NGS) dans du PBS) pendant 1 h à température ambiante.
- Incuber les sections pendant la nuit à 4 °C avec l’anticorps primaire polyclonal anti-GFP de poulet (1:1 000).
- Laver 3 x 15 min dans du PBS et incuber les sections pendant 2 h à température ambiante avec l’anticorps secondaire de chèvre polyclonal anti-poulet conjugué au fluorophore Alexa Fluor 488 (1:200).
- Laver pendant 3 x 15 min dans PBS. Incuber avec du DAPI pendant 5 min à température ambiante.
- Laver pendant 3 x 15 min dans PBS. Placez les sections dans des lames recouvertes de gélatine et une lamelle avec un milieu de montage par fluorescence.
- Acquérez des images sur un microscope à fluorescence à balayage de lames équipé d’un objectif 20x/0,8.
Résultats
Dans ce travail, nous présentons un protocole détaillé pour la production, la purification et la caractérisation des rAAV en mosaïque (résumés dans la figure 1), qui ont le potentiel de cibler et de transduire le SNC (par exemple AAV1 et AAV9), étant simultanément adaptés à la purification par chromatographie d’affinité à l’héparine (AAV2). Pour y parvenir, des capsides des sérotypes AAV naturels 1, 2 et 9 ont été utilisées pour développer des vecteurs mosaïques rAAV1/2 et rAAV2/9.
Avant de commencer, l’intégrité structurelle des préparations plasmidiques a été examinée. En plus des digestions nécessaires pour valider l’insertion correcte des fragments de clonage, il est essentiel de cribler régulièrement les plasmides pITR pour détecter les délétions / insertions potentielles d’ITR. À titre d’exemple, l’intégrité des ITR dans différents clones d’un plasmide pITR a été surveillée après la digestion du plasmide avec l’enzyme de restriction SmaI (figure supplémentaire S1).
Les deux types de vecteurs mosaïques ont été générés par la co-transfection des plasmides de capside AAV respectifs dans un rapport de 1:1, selon les méthodes de transfection standard6. En bref, HEK293T cellules ont été transfectées avec i) un plasmide contenant le transgène d’intérêt emballé entre les séquences ITR (pITR), ii) un plasmide contenant les ORF du génome AAV de type sauvage Rep et Cap d’AAV2 et AAV1 ou AAV9 (plasmides pAAV-RC) et iii) un plasmide codifiant les protéines adénovirales (E1A, E1B, E4 et E2A) ainsi que les ARN associés au virus adénoviral essentiels aux fonctions auxiliaires (pHelper). Quarante-huit heures plus tard, les cellules ont été récoltées 6,36, et les rAAV ont été purifiés de l’homogénat cellulaire par chromatographie d’affinité à l’aide d’un système FPLC. Comme le montre la figure 2A, après l’équilibrage de la colonne (étape d’équilibration), le lysat de cellule contenant les rAAV a été appliqué à la colonne (chargement de l’échantillon). En raison de l’affinité naturelle du rAAV2 pour l’héparine33, les rAAV se sont liés à la résine de la colonne, tandis que d’autres composants ont été effectués dans le tampon de fonctionnement et détectés par le moniteur UV (flowthrough), ce qui a entraîné une augmentation de l’absorbance. La colonne a ensuite été lavée (étape de lavage) et les rAAV ont finalement été élués par une augmentation de la concentration en NaCl (étape d’élution). Les virus élués ont été détectés par le moniteur UV et collectés en fractions de 1 mL.
Un profil représentatif du pic d’élution de rAAV1/2 et rAAV2/9 est illustré à la figure 2B et à la figure supplémentaire S2A, respectivement, avec différents lots viraux présentant systématiquement un seul pic commençant à la fraction F7 jusqu’à F16. La hauteur des pics est variable entre les productions de rAAV, des pics plus élevés conduisant généralement à des rendements de rAAV plus élevés. Chaque fraction des rAAV1/2 et rAAV2/9 produits a ensuite été caractérisée par RT-qPCR afin d’évaluer les titres viraux (figure 2C et figure supplémentaire S2B).
Pour caractériser la pureté du matériau élué, 40 μL de chaque fraction et de l’écoulement respectif ont été examinés par électrophorèse sur gel de polyacrylamide à 10 % (figure 2D pour rAAV1/2 et figure supplémentaire S2C pour rAAV2/9). La coloration au bleu de Coomassie a révélé trois bandes principales dans les fractions F7-F16, avec des poids moléculaires correspondant aux protéines de capside VP1 (87 kDa), VP2 (72 kDa) et VP3 (62 kDa) des AAV aux rapports appropriés de 1:1:10, comme décrit précédemment par Van Vliet et ses collègues14. Dans les deux cas, et sur la base de l’absorbance UV, de la RT-qPCR et de l’intensité de la bande de gel, il est clair que la majorité des rAAV en mosaïque est présente dans les fractions F7 et F8 et commence à diminuer progressivement dans les fractions F9-F16. En plus des trois protéines de la capside virale, une autre protéine (ou des protéines) d’une taille d’environ 17 kDa a été détectée dans les fractions F8-F16.
Pour éliminer cette ou ces protéines co-épuratrices, les fractions F7-16 ont ensuite été filtrées et concentrées à l’aide d’unités de filtration centrifuge de 100 KDa et le titre final de rAAV a été déterminé par RT-qPCR (comme le montre la figure 3A,B pour rAAV1/2). Le rendement final d’une production de rAAV dépend de la longueur et de la complexité du pITR, de l’intégrité des séquences ITR, des conditions de culture cellulaire (par exemple, le nombre de passages cellulaires) et de l’efficacité de transfection 24,58,59,60,61. Néanmoins, le titre final peut être ajusté en effectuant plusieurs centrifugations de la préparation rAAV à l’aide d’unités de filtration centrifuge de 0,5 mL (étape de concentration 2). Selon ce protocole, pour un volume final compris entre 50 et 100 μL, les concentrations sont généralement comprises entre 2 × 109 et 5 × 1010 vg/μL (quantification effectuée à l’aide du kit de titrage référencé).
La pureté des préparations finales de rAAV a ensuite été évaluée sur un gel de polyacrylamide SDS à 10 %. Comme le montre la figure 3C, seules trois bandes représentant les protéines de capside du rAAV1/2 ont été observées pour la préparation du rAAV1/2 et aucune protéine co-épuratrice détectable n’a été identifiée. Ces résultats étaient cohérents avec ceux obtenus pour rAAV2/9 (figure supplémentaire S2C). Afin de confirmer l’identité et de caractériser davantage la pureté des vecteurs rAAV1/2 et rAAV2/9, les fractions virales et les stocks concentrés ont été analysés par transfert Western, avec les anticorps spécifiques B1 (figure supplémentaire S3A et figure supplémentaire S4A) et A69 (figure supplémentaire S3B et figure supplémentaire S4B). Alors que l’anticorps B1 reconnaît un épitope C-terminal commun à toutes les protéines VP de la plupart des sérotypesAAV 62, le clone A69 ne reconnaît que les épitopes de VP1 et VP263. Néanmoins, certaines bandes faibles avec des poids moléculaires inférieurs à VP3 (<62 kDa) peuvent également être détectées lors du marquage B1 et A69.
Pour caractériser la morphologie structurale et évaluer davantage la pureté des rAAV, les particules virales ont été directement visualisées par TEM. Cette technique a été la procédure standard pour évaluer l’intégrité et la pureté des échantillons viraux, car elle permet la quantification des particules rAAV vides et pleines, ainsi que l’évaluation de la contamination dans un échantillon 29,64,65,66,67. Comme le montre la figure 3D, de grandes quantités de particules rAAV, de ~25 nm de diamètre, ont pu être observées sur un fond propre. Des particules vides (flèche noire) avec un centre dense en électrons, ainsi que des vecteurs pleins (flèche blanche) ont également pu être observés dans tout le champ d’échantillon.
Nous avons également effectué le contrôle de la qualité des rAAV purifiés à l’aide de Stunner, une plate-forme qui combine la spectroscopie ultraviolet-visible (UV-Vis), la diffusion statique de la lumière (SLS) et la diffusion dynamique de la lumière (DLS)68. Pour chaque échantillon, la quantité totale de protéines, d’ADNsb, ainsi que les impuretés absorbantes et la turbidité de fond, ont été mesurées par spectroscopie UV-Vis (figure 3E et figure supplémentaire S5A). SLS et DLS ont ensuite été appliqués pour évaluer le comportement de diffusion de la lumière des capsides rAAV. Étant donné que les AAV ont un diamètre moyen de 25 nm, les particules dont le diamètre est compris entre 15 et 45 nm sont considérées comme intactes. Les particules plus grosses représentent généralement des agrégats viraux, et tout ce qui est plus petit comprend très probablement de petites particules, y compris des protéines de capside non assemblées68. Pour rAAV1/2, un seul pic correspondant à des particules de capside intactes a été observé à 30 nm (Figure 3F), avec 0 % d’intensité agrégée et 0 % d’intensité de petites particules. Pour la préparation rAAV2/9, un pic à 30 nm a également été détecté, représentant une intensité de capside de 78 % (figure supplémentaire S5B). Même si l’intensité des petites particules était de 0 %, pour cet échantillon, une intensité globale de 22 % a été mesurée (représentée en gris), la contribution majeure (19,9 %) provenant de grands agrégats d’un diamètre moyen de 620 nm (figure supplémentaire S5B). Grâce à la combinaison de la spectroscopie UV-Vis avec des informations SLS et DLS, Stunner a révélé le titre total de la capside, le titre de la capside complète, la protéine libre et agrégée, ainsi que l’ADNsb libre et agrégé pour les deux préparations virales, illustrés à la figure 3G et à la figure supplémentaire S5C (valeurs spécifiques indiquées dans la légende de chaque figure).
En parallèle, afin d’évaluer l’activité biologique des vecteurs AAV en mosaïque développés, HEK293T cellules ont été infectées avec 50 μL de chaque fraction obtenue par FPLC (F2-F16) de la préparation rAAV1/2 ou rAAV2/9. Étant donné que le vecteur rAAV1/2 code pour une protéine fluorescente verte (GFP) monocaténaire, sous le contrôle d’un promoteur du CMV (pAAV-CMV-ssGFP), et que le vecteur rAAV2/9 code pour une GFP auto-complémentaire, sous le contrôle du promoteur du CMV (pAAV-CMV-scGFP53), la fluorescence directe de la GFP a été examinée dans ces cellules 48 heures après l’infection (figure supplémentaire S6 et figure supplémentaire S7). Conformément aux observations précédentes pour la RT-qPCR, le bleu de Coomassie et le western blot, le niveau d’infectivité le plus élevé a été atteint pour les fractions virales F7 et F8, diminuant progressivement dans les fractions F9 à F16.
Pour confirmer si l’activité biologique des rAAV a été maintenue après les étapes d’ultrafiltration et de concentration, les cellules Neuro2A, plaquées à la fois dans des plaques à 24 puits et une lame de chambre à 8 puits, ont été infectées par le vecteur concentré rAAV1/2, codant pour scGFP sous le contrôle du promoteur CMV (pAAV-CMV-scGFP53). Les images en fond clair et en fluorescence ont été acquises 48 h après l’infection (figures 4A, B pour les images à plus haute résolution).
Dans le but d’explorer la capacité infectieuse des rAAV produits dans un modèle cellulaire plus pertinent et plus réfléchissant, des cultures neuronales primaires semi-denses du cortex ont été ensemencées sur une plaque à 12 puits et infectées avec le rAAV1/2 - CMV-scGFP précédemment utilisé. Quarante-huit heures après l’infection, les cellules ont été fixées et marquées avec du DAPI et du WGA conjugué avec Alexa Fluor 633, Une lectine largement utilisée pour marquer les cellules fixes. Les images présentées dans la figure 4C,D ont été acquises avec un Zeiss Axio Observer Z1 et sur un LSM 710 confocal Zeiss. Comme le montrent ces figures par fluorescence directe de la GFP, les virus en mosaïque concentrée conservent leurs propriétés de transfert de gènes pour les cellules neuronales.
Après avoir caractérisé les rAAV en mosaïque en termes de pureté, de propriétés physiques et de fonctionnalité in vitro, nous avons ensuite évalué la possibilité d’utiliser les vecteurs de mosaïque rAAV1/2 purifiés pour transduire le cervelet des souris C57BL/6. Pour cela, une injection stéréotaxique a été réalisée chez des souris âgées de 9 semaines et l’expression de la GFP a été évaluée 12 semaines plus tard. Comme prévu, les animaux ayant reçu une injection de PBS n’ont montré aucune fluorescence lors de l’immunomarquage de la GFP. Des images d’épifluorescence de souris injectées avec des vecteurs rAAV1/2 codant pour la GFP sous le contrôle du promoteur de la synapsine 1 (rAAV1/2 - Syn-ssGFP) ont révélé que les vecteurs rAAV1/2 transduisaient avec succès plusieurs régions du cervelet, à savoir la région des noyaux cérébelleux profonds (DCN), ainsi que les différents lobules du cervelet (Figure 5). Ces résultats démontrent l’expression prolongée du transgène dans le cerveau des mammifères (12 semaines).
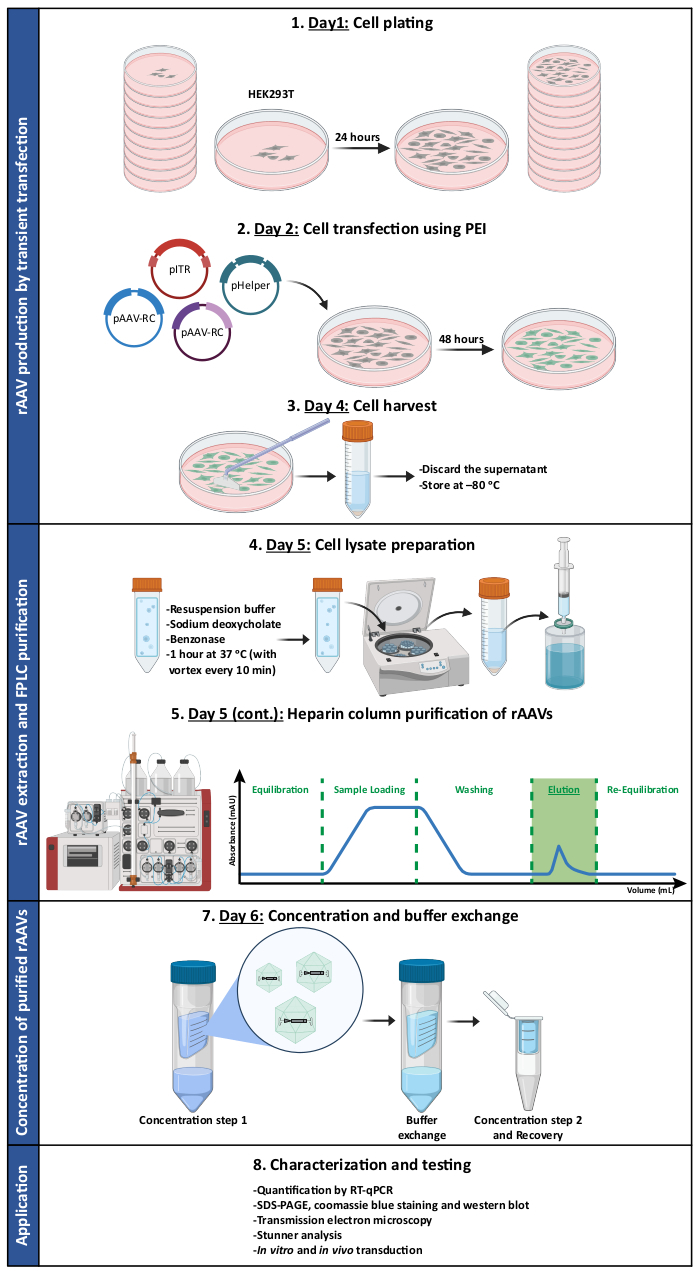
Figure 1 : Représentation schématique du protocole de production et de purification du rAAV. Les rAAV sont produits par transfection transitoire de cellules HEK293T à l’aide de polyéthylénimine (PEI). Par la suite, les cellules sont récoltées et lysées, et les rAAV sont purifiés à partir de l’homogénat cellulaire par chromatographie d’affinité. Les fractions collectées contenant des rAAV sont ensuite concentrées, et les stocks viraux finaux sont caractérisés en termes de titre, de pureté, de caractéristiques morphologiques et d’activité biologique. Abréviations : rAAV = virus adéno-associé recombinant ; PEI = polyéthylénimine ; RT-qPCR = réaction en chaîne par polymérase quantitative en temps réel ; SDS-PAGE = électrophorèse sur gel de dodécylsulfate de sodium-polyacrylamide. Veuillez cliquer ici pour voir une version agrandie de cette figure.
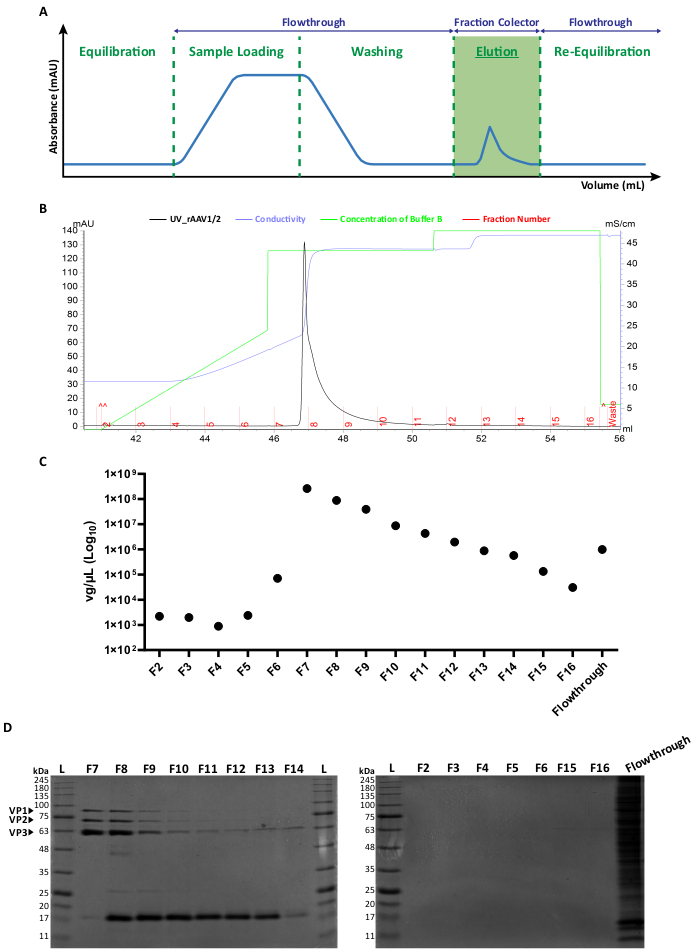
Figure 2 : Protocole de purification FPLC et profil d’élution représentatif du rAAV1/2. (A) Représentation schématique d’un profil complet de chromatogramme, montrant les différentes étapes du processus de purification du rAAV. Après une étape d’équilibrage de colonne, l’échantillon est appliqué. La colonne est ensuite lavée et l’élution est effectuée avec des concentrations croissantes de NaCl. Le matériau non lié (flux continu) et les fractions de 1 mL des virus élués sont collectés pour analyse. L’absorbance à 280 nm est exprimée en mUA et l’axe des x indique le volume en mL. (B) Chromatogramme partiel élargi montrant un pic d’élution rAAV1/2 (en noir), avec les numéros de fraction correspondants (F2-F16) et les déchets (indiqués en rouge). La concentration émise du tampon B et la conductivité (exprimée en mS/cm) sont également indiquées en vert et en violet, respectivement. (C) RT-qPCR de chaque fraction collectée lors de la purification par affinité (F2-F16) et de la diffusion par écoulement. Le titre en vg/μL est représenté sur une échelle logarithmique. (D) Analyse SDS-PAGE des fractions virales collectées. Des volumes égaux (40 μL) de chaque fraction de l’étape d’élution (F2-F16) et du flux écoulement respectif ont été chargés et résolus sur un gel de polyacrylamide SDS à 10 %. Les bandes de protéines ont été visualisées par coloration au bleu de Coomassie. Les bandes correspondant aux protéines de capside AAV VP1, VP2 et VP3 sont indiquées. L’échelle de taille standard des protéines est désignée par (L) et les poids moléculaires correspondants sont également indiqués. Abréviations : rAAV = virus adéno-associé recombinant ; RT-qPCR = réaction en chaîne par polymérase quantitative en temps réel ; SDS-PAGE = électrophorèse sur gel de dodécylsulfate de sodium-polyacrylamide. Veuillez cliquer ici pour voir une version agrandie de cette figure.
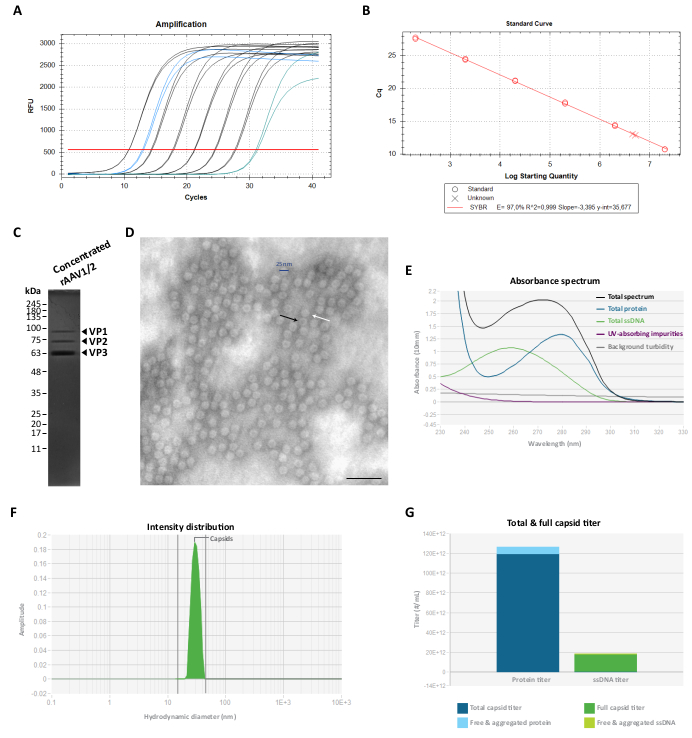
Figure 3 : Caractérisation des vecteurs rAAV1/2 concentrés. (A) Courbes d’amplification d’un échantillon concentré de rAAV1/2 (en bleu), étalons dilués en série de 2 × 107 vg/μL à 2 × 102 vg/μL (en noir) et un contrôle sans matrice (en vert), obtenues lors de la RT-qPCR. (B) Courbe standard (régression linéaire) pour la détermination du titre d’un échantillon rAAV en vg/μL. (C) Analyse SDS-PAGE des particules virales concentrées. Au total, 2,3 × 1010 vg de la matière concentrée ont été regroupés sur le gel. (D) Image de microscopie électronique en transmission de particules rAAV1/2 de ~25-30 nm de diamètre. Les particules vides avec un centre dense en électrons (mis en évidence par des flèches noires) peuvent être distinguées des capsides pleines (mises en évidence par des flèches blanches). Barre d’échelle = 100 nm. (E) Spectre d’absorbance d’une préparation rAAV1/2 mesuré par Stunner (en noir). La contribution des protéines (en bleu), de l’ADNsb (en vert), d’autres composés absorbant les UV ou des impuretés (en violet) et de la turbidité de fond (en gris) est également indiquée. (F) Distribution d’intensité DLS de rAAV1/2 avec un seul pic à 30 nm, mesurée par Stunner. Une intensité de diffusion de la capside de 100 % a été déterminée en mesurant l’aire sous la courbe de 15 à 45 nm (en vert). (G) Analyse d’étourdissement d’une préparation de vecteur rAAV1/2 présentant un titre total de capside de 1,19 × 1014 cp/mL (bleu foncé) et un titre de capside complète de 1,73 ×10 13 vg/mL (vert foncé). Une protéine libre et agrégée de 7,16 × 1012 équivalents cp/mL (bleu clair), ainsi qu’un ADNss libre et agrégé de 1,04 ×10 12 vg/mL équivalents (vert clair), ont également été mesurés. Abréviations : rAAV = virus adéno-associé recombinant ; RT-qPCR = réaction en chaîne par polymérase quantitative en temps réel ; SDS-PAGE = électrophorèse sur gel de dodécylsulfate de sodium-polyacrylamide ; ADNsb = ADN simple brin ; DLS = diffusion dynamique de la lumière. Veuillez cliquer ici pour voir une version agrandie de cette figure.
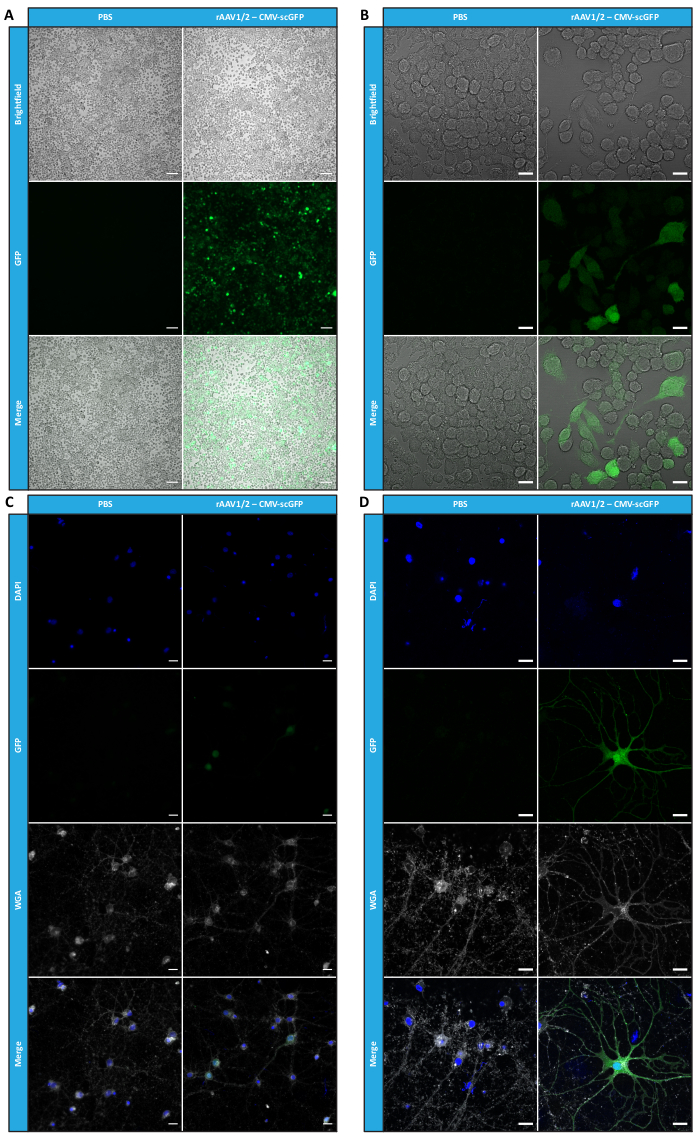
Figure 4 : Évaluation de l’infectiosité in vitro d’un échantillon concentré de rAAV1/2. (A) Les cellules Neuro2A ont été infectées par rAAV1/2 - CMV-scGFP ou incubées avec un volume équivalent de PBS, en tant que contrôle négatif. Images en fond clair et en fluorescence de cellules imagées 48 h après l’infection. Les images ont été acquises à l’aide d’un Zeiss Axio Observer Z1 (objectif 10x). Barres d’échelle = 100 μm. (B) Images détaillées des cellules Neuro2A 48 h après l’infection par rAAV1/2 - CMV-scGFP. Les images ont été acquises dans un Zeiss LSM 710 (objectif 40x). Barres d’échelle = 20 μm. (C) Cultures neuronales primaires semi-denses infectées par rAAV1/2 - CMV-scGFP ou incubées avec un volume équivalent de PBS, servant de contrôle négatif. Les cellules ont été marquées avec une coloration nucléaire (DAPI en bleu) et une coloration membranaire (WGA en blanc). Les images ont été acquises à l’aide d’un Zeiss Axio Observer Z1 (objectif 40x). Barres d’échelle = 20 μm. (D) Images détaillées de cultures neuronales primaires semi-denses 48 h après l’infection par rAAV1/2 - CMV-scGFP. Les images ont été acquises dans un Zeiss LSM 710 (objectif 40x). Barres d’échelle = 20 μm. Abréviations : rAAV = virus adéno-associé recombinant ; CMV = cytomégalovirus ; scGFP = protéine fluorescente verte auto-complémentaire ; PBS = solution saline tamponnée au phosphate ; DAPI = 4',6-diamidino-2-phénylindole ; WGA = agglutinine de germe de blé. Veuillez cliquer ici pour voir une version agrandie de cette figure.
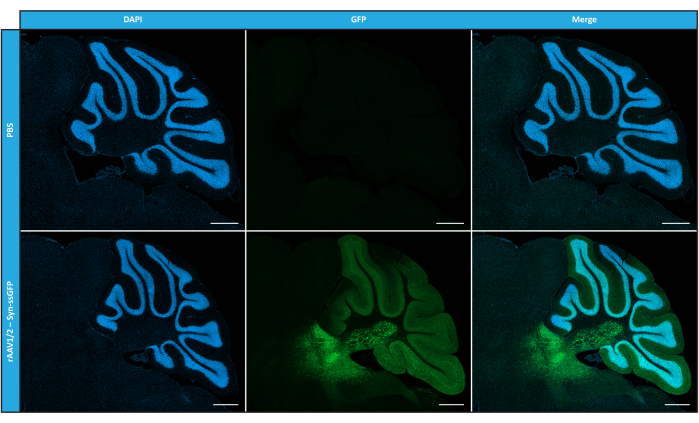
Figure 5 : Efficacité de transduction in vivo de rAAV1/2 suite à une injection intraparenchymateuse. Images représentatives d’immunofluorescence montrant l’expression généralisée de la GFP (en vert) dans tout le cervelet lors d’une injection centrale de rAAV1/2 - Syn-ssGFP dans le cervelet. Les noyaux ont été colorés au DAPI (en bleu). Barres d’échelle = 500 μm. Abréviations : rAAV = virus adéno-associé recombinant ; Syn = Synapsine 1 ; ssGFP = protéine fluorescente verte monocaténaire ; DAPI = 4',6-diamidino-2-phénylindole ; PBS = solution saline tamponnée au phosphate. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire S1 : Analyse sur gel d’agarose d’un plasmide de vecteur rAAV digéré avec SmaI. Six clones (C1-C6) d’un pITR ont été digérés avec l’enzyme de restriction SmaI (voies 2, 4, 6, 8, 10 et 12), qui coupe deux fois à l’intérieur de chaque répétition terminale inversée. Dans ce cas, une digestion complète de ce pITR devrait générer deux bandes (3 796 pb et 3 013 pb). Dans les préparations réussies (C1, C3, C4 et C5), une bande de 6809 pb, résultant d’une digestion partielle, est encore visible (~5% du total). Dans les préparations avec recombinaison ITR, les proportions sont inversées (C2), ou la digestion n’a pas eu lieu (C6). Les clones non digérés respectifs sont également présentés (couloirs 3, 5, 7, 9, 11, 13). Abréviations : rAAV = virus adéno-associé recombinant ; ITR = répétition terminale inversée. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S2 : purification du rAAV2/9 par chromatographie d’affinité à base d’héparine. (A) Profil d’élution de rAAV2/9 présentant un seul pic (en noir), suite à une augmentation de la concentration de NaCl. Les fractions collectées sont indiquées par des chiffres (2-16) en rouge au bas du graphique, l’absorbance à 280 nm est exprimée en mUA, la conductivité est exprimée en mS/cm et l’axe des x indique le volume en mL. (B) titres de rAAV quantifiés par RT-qPCR pour chaque fraction regroupée (F2-F16) et à écoulement. Les valeurs sont représentées sur une échelle logarithmique. (C) Dosage de la pureté par SDS-PAGE et coloration au bleu de Coomassie. Des volumes égaux (40 μL) de chaque fraction (F2-F16) et de la circulation correspondante ont été chargés et résolus sur une SDS-PAGE à 10 %. Le stock concentré a été quantifié par RT-qPCR et 2,3 × 1010 vg ont été dilués dans 40 μL de PBS et regroupés sur le gel. Les bandes de protéines ont été visualisées par coloration au bleu de Coomassie. Les protéines de capside AAV (VP1, VP2 et VP3) sont indiquées. L’échelle de taille standard des protéines est désignée par (L) et les poids moléculaires correspondants sont également indiqués. Abréviations : rAAV = virus adéno-associé recombinant ; RT-qPCR = réaction en chaîne par polymérase quantitative en temps réel ; SDS-PAGE = électrophorèse sur gel de dodécylsulfate de sodium-polyacrylamide. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S3 : Analyse par transfert Western de vecteurs rAAV1/2 purifiés par FPLC. (A) Les fractions collectées et les vecteurs rAAV1/2 concentrés ont été résolus sur un gel SDS-PAGE et sondés avec un anticorps monoclonal anti-AAV de souris (B1) qui reconnaît les protéines de capside VP1, VP2 et VP3. (B) Les fractions collectées et les vecteurs rAAV1/2 concentrés ont été résolus sur un gel SDS-PAGE et sondés avec un anticorps monoclonal anti-AAV de souris (A69) qui reconnaît les protéines de capside VP1 et VP2. Abréviations : rAAV = virus adéno-associé recombinant ; FPLC = chromatographie liquide protéique rapide ; SDS-PAGE = électrophorèse sur gel de dodécylsulfate de sodium-polyacrylamide ; L = échelle de taille standard des protéines. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S4 : Analyse par transfert Western de vecteurs rAAV2/9 purifiés par FPLC. (A) Les fractions collectées et les vecteurs rAAV2/9 concentrés ont été résolus sur un gel SDS-PAGE et sondés avec un anticorps monoclonal anti-AAV de souris (B1) qui reconnaît les protéines de capside VP1, VP2 et VP3. (B) Les fractions collectées et les vecteurs rAAV2/9 concentrés ont été résolus sur un gel SDS-PAGE et sondés avec un anticorps monoclonal anti-AAV de souris (A69) qui reconnaît les protéines de capside VP1 et VP2. Abréviations : rAAV = virus adéno-associé recombinant ; FPLC = chromatographie liquide protéique rapide ; SDS-PAGE = électrophorèse sur gel de dodécylsulfate de sodium-polyacrylamide ; L = échelle de taille standard des protéines. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S5 : Quantification et caractérisation du vecteur rAAV2/9 via Stunner. (A) Spectre d’absorbance (noir) d’un vecteur rAAV2/9 mesuré par Stunner. La contribution des protéines (bleu), de l’ADNsb (vert), d’autres composés absorbant les UV ou des impuretés (violet) et de la turbidité de fond (gris) est également représentée. (B) Distribution de l’intensité DLS de rAAV2/9 avec un pic majeur à 30 nm correspondant à une intensité de diffusion de la capside de 78 %, déterminée en mesurant l’aire sous la courbe de 15 à 45 nm (en vert). Une intensité globale totale de 22 % (en gris) a également été mesurée, avec une contribution principale des grands agrégats (19,9 %) avec un diamètre moyen de 620 nm. (C) Analyse d’étourdissement d’une préparation de vecteur rAAV2/9 présentant un titre de capside totale de 2,18 × 1014 cp/mL (bleu foncé) et un titre de capside complète de 2,35 ×10 13 vg/mL (vert foncé). Une protéine libre et agrégée de 2,92 × 1013 équivalents cp/mL (bleu clair), ainsi qu’un ADNss libre et agrégé de 3,14 ×10 12 équivalents vg/mL (vert clair), ont également été mesurés dans cette préparation. Abréviations : rAAV = virus adéno-associé recombinant ; ADNsb = ADN simple brin ; DLS = diffusion dynamique de la lumière. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S6 : Efficacité de la transduction in vitro et viabilité des fractions purifiées de rAAV1/2. HEK293T cellules exprimant la GFP (fluorescence directe) 48 h après la transduction avec 50 μL de fractions FPLC d’un vecteur rAAV1/2 codant pour ssGFP (rAAV1/2 - CMV-ssGFP). Barres d’échelle = 100 μm. Abréviations : rAAV = virus adéno-associé recombinant ; FPLC = chromatographie liquide protéique rapide ; ssGFP = protéine fluorescente verte simple brin. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S7 : Efficacité de la transduction in vitro et viabilité des fractions purifiées de rAAV2/9. HEK293T cellules ont été infectées par 50 μL de chaque fraction FPLC (F2-F16) ou par un flux d’un vecteur rAAV2/9 codant pour scGFP sous le contrôle du promoteur CMV. Les cellules exprimant la GFP ont été visualisées 48 h après l’infection. Barres d’échelle = 100 μm. Abréviations : rAAV = virus adéno-associé recombinant ; FPLC = chromatographie liquide protéique rapide ; scGFP = protéine fluorescente verte auto-complémentaire ; CMV = cytomégalovirus. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
La boîte à outils de vecteurs AAV, en expansion rapide, est devenue l’un des systèmes d’administration de gènes les plus prometteurs pour un large éventail de types de cellules par différentes voies d’administration. Dans ce travail, nous avons cherché à développer un protocole amélioré pour la production, la purification et la caractérisation des vecteurs rAAV en mosaïque qui pourrait faire leurs preuves dans les études précliniques. À cette fin, la génération de vecteurs mosaïques rAAV1/2 et rAAV2/9 est décrite ici, mais la procédure peut également être appliquée pour purifier les vecteurs rAAV2 standard (données non présentées).
Des rAAV en mosaïque ont été produits selon une méthode de transfection optimisée en utilisant l’IEP comme réactif de transfection. Une méthode de transfection transitoire a été choisie en raison de sa flexibilité et de sa rapidité accrues, des avantages considérables dans les études précliniques précoces. Une fois qu’un transgène et un sérotype particuliers ont été validés, le système de production peut être affiné pour obtenir une meilleure évolutivité et une meilleure rentabilité en établissant une lignée cellulaire transfectée stable qui exprime un sous-ensemble des gènes spécifiques Rep/Cap , avec des gènes supplémentaires fournis par un processus d’infection24. Par rapport à la transfection calcium-phosphate, l’IPE présente plusieurs avantages. Il s’agit d’un réactif de transfection stable et rentable qui fonctionne efficacement dans une plage de pH plus large. De plus, il n’est pas nécessaire de changer le support cellulaire après la transfection, ce qui entraîne une réduction significative des coûts et de la charge de travail69.
Dans le but de contourner certaines des limitations imposées par les gradients de CsCl ou d’iodixanol, les rAAV produits ont été récoltés et purifiés par chromatographie d’affinité. Cette stratégie offre une approche simplifiée et évolutive qui peut être réalisée sans avoir besoin d’ultracentrifugation et de gradients, ce qui permet d’obtenir des titres viraux propres et élevés. En effet, les techniques de chromatographie utilisant un système FPLC peuvent être automatisées et mises à l’échelle en emballant plus de volume de résine dans une colonne avec une hauteur de lit plus élevée. Le protocole décrit dans le présent document peut être facilement adapté pour incorporer des colonnes d’héparine HiTrap HP de 5 mL (données non présentées). De plus, les colonnes d’héparine peuvent être réutilisées plusieurs fois, ce qui contribue à la rentabilité de cette méthode.
Les rAAV purifiés ont ensuite été caractérisés en termes de titre, de pureté, de caractéristiques morphologiques et d’activité biologique. Il est intéressant de noter que dans la coloration au bleu de Coomassie, une bande d’environ 17 kDa a été détectée dans les fractions F8-F16 en plus des trois protéines typiques de la capside virale. Cependant, cette bande n’est plus présente après l’étape de concentration des rAAV. De plus, certaines bandes faibles avec des poids moléculaires inférieurs à VP3 (<62 kDa) peuvent également être détectées lors du marquage B1 et A69, suggérant qu’il pourrait s’agir de fragments des protéines de capside VP1, VP2 et VP370. Une autre possibilité est qu’il s’agit en fait d’autres protéines co-purifiantes telles que la ferritine ou d’autres protéines cellulaires avec des polypeptides qui partagent des empreintes protéiques similaires à celles des protéines de capside de l’AAV et pourraient être impliquées dans la biologie de l’AAV, comme suggéré précédemment 26,71,72.
L’analyse TEM et l’analyse des étourdissements ont également révélé la présence de particules vides à des niveaux variables entre différentes productions. De même, d’autres études antérieures ont rapporté la génération de niveaux variables et élevés (>65%) de capsides vides pour les rAAV préparés par des méthodes de transfection ou d’infection24,73. Le mécanisme à l’origine de la génération du rAAV commence par la formation rapide de capsides vides à partir de protéines VP nouvellement synthétisées, suivie d’une étape lente de limitation du taux d’empaquetage du génome dans les capsides préformées médiées par les protéines Rep74,75. Par conséquent, des capsides vides sont générées dans les productions de rAAV, bien que la proportion de capsides vides et pleines puisse varier en fonction de la taille et de la séquence du transgène d’intérêt et des conditions de culture cellulaire58,73. Les capsides vides soulèvent quelques inquiétudes car, en l’absence du génome d’intérêt, elles sont incapables de fournir un effet thérapeutique et peuvent également potentiellement augmenter une réponse immunitaire innée ou adaptative. Cependant, certains rapports ont également montré qu’en ajustant leur rapport, les capsides vides d’AAV peuvent servir de leurres très efficaces pour les anticorps neutralisants spécifiques aux AAV et, par conséquent, augmenter l’efficacité de la transduction 60,76,77. Si la présence de capsides vides est gravement préjudiciable et compte tenu du caractère légèrement moins anionique des particules vides par rapport aux vecteurs pleins, une solution potentielle pourrait impliquer la réalisation d’une deuxième étape de purification par polissage à l’aide de techniques de chromatographie par échange d’anions64.
Cette étude fournit également des preuves convaincantes que les rAAV en mosaïque générés sont capables de transduire efficacement non seulement les cultures neuronales in vitro , mais aussi le SNC lors de l’injection intracrânienne de rAAV1/2. Dans l’ensemble, ces résultats suggèrent que le protocole de production et de purification décrit permet d’obtenir des rAAV hautement purs et biologiquement actifs prêts à l’emploi en 6 jours, se présentant comme une méthode polyvalente et rentable pour la génération de rAAV dans les études précliniques.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Nous sommes reconnaissants de la collaboration, des idées et de l’assistance technique fournies par le Dr Mónica Zuzarte, de l’Institut de recherche clinique et biomédicale de Coimbra (iCBR) et du Centre de biomédecine et de biotechnologie innovantes (CIBB), concernant l’analyse TEM des rAAV. Nous exprimons notre gratitude au Dr Dominique Fernandes, du Centre de neurosciences et de biologie cellulaire de l’Université de Coimbra (CNC-UC) et de l’Institut de recherche interdisciplinaire de l’Université de Coimbra (IIIUC), pour son aide technique inestimable et ses connaissances concernant les expériences de culture neuronale primaire. Les plasmides pRV1, pH21 et pFdelta6, essentiels à cette étude, ont été généreusement fournis par la Dre Christina McClure, de l’École des sciences médicales, Collège des sciences de la vie et de la médecine, Université d’Aberdeen, pour laquelle nous sommes reconnaissants. Ce travail a été financé par le Fonds européen de développement régional (FEDER), dans le cadre du programme opérationnel régional Centro 2020 ; par le biais du programme opérationnel COMPETE 2020 pour la compétitivité et l’internationalisation, et des fonds nationaux portugais par l’intermédiaire de FCT - Fundação para a Ciência e a Tecnologia, dans le cadre des projets : UIDB/04539/2020, UIDP/04539/2020, LA/P/0058/2020, ViraVector (CENTRO-01-0145-FEDER-022095), Imagene (PTDC/BBB-NAN/0932/2014 | POCI-01-0145-FEDER-016807), ReSet - IDT-COP (CENTRO-01-0247-FEDER-070162), Fighting Sars-CoV-2 (CENTRO-01-01D2-FEDER-000002), BDforMJD (CENTRO-01-0145-FEDER-181240), ModelPolyQ2.0 (CENTRO-01-0145-FEDER-181258), MJDEDIT (CENTRO-01-0145-FEDER-181266) ; par l’American Portuguese Biomedical Research Fund (APBRF) et le Richard Chin and Lily Lock Machado-Joseph Disease Research Fund, ARDAT dans le cadre de l’accord de subvention IMI2 JU n° 945473 soutenu par l’UE et l’EFPIA ; Le projet GeneT-Teaming 101059981 soutenu par le programme Horizon Europe de l’Union européenne. M.M.L. a été soutenu par 2021.05776.BD ; C.H. était soutenu par 2021.06939.BD ; A.C.S. était soutenu par 2020.07721.BD ; et D.D.L. a été soutenu par 2020.09668.BD. La figure 1 a été créée à l’aide de BioRender.com.
matériels
| Name | Company | Catalog Number | Comments |
| 10% povidone-iodine | Medline | MDS093943 | |
| 12-well plates | Thermo Scientific | 11889684 | |
| 24-well plates | VWR | 734-2325 | |
| 4’,6-diamidino-2-phenylindole (DAPI) | Invitrogen | D1306 | |
| 96-well Stunner plate | Unchained Labs | 701-2025 | 96-well quantification plate for the consecutive ultraviolet-visible light absorption, static light scattering, and dynamic light scattering analysis of rAAV samples |
| AAVpro Titration Kit (for Real-Time PCR) Ver.2 | Takara | 6233 | For determining the titer of AAV using RT-qPCR. This kit contains DNase I, Lysis Buffer, Dilution Buffer, positive control, Taq II mix, primer forward, primer reverse, water |
| Acetic acid glacial | Fisher Chemical | A/0360/PB17 | |
| ÄKTA pure 25 | Cytiva | 29018224 | FPLC system controlled by UNICORN software, version 6.3 |
| Alkaline phosphatase-linked goat anti-mouse | Invitrogen | 31328 | |
| Amicon ultra-0.5 centrifugal filter unit | Merck Millipore | UFC5100 | |
| Amicon ultra-15 centrifugal filter unit | Merck Millipore | UFC9100 | |
| Benzonase Nuclease | Merck Millipore | E1014 | |
| Bromophenol blue | Sigma-Aldrich | B0126 | |
| CFX96 Real-Time PCR detection system | Biorad | 184-5096 | |
| ChemiDoc Touch Imaging System | Bio-Rad Laboratories | 1708370 | |
| Chicken polyclonal anti-GFP primary antibody | Abcam | ab13970 | |
| Coomassie Blue G250 | Fisher Chemical | C/P541/46 | |
| Dithiothreitol (DTT) | Fisher Bioreagents | BP17225 | |
| DMEM | Sigma-Aldrich | D5796 | |
| ECF Substrate for Western Blotting | Cytiva | RPN5785 | |
| FastDigest SmaI | Thermo Scientific | FD0663 | |
| FEI-Tecnai G2 Spirit Biotwin | FEI | Biotwin | Transmission electron microscope |
| Fetal bovine serum | Biowest | S1810 | |
| Fluorescence mounting medium | Dako | S3023 | |
| Formvar-carbon coated 200 mesh grid | TAAB Laboratories Equipment | F077/025 | |
| Gas evacuation apparatus | RWD | R546W | |
| Glycerol | Fisher BioReagents | 10021083 | |
| Goat polyclonal anti-chicken antibody, Alexa Fluor 488 | Invitrogen | A-11039 | |
| Hamilton needle 30G, Small Hub RN Needle, 25 mm, PST3 | Hamilton | 7803-07 | |
| Hamilton syringe (10 µL) | Hamilton | 7653-01 | |
| HEK293T | American Type Culture Collection | CRL-11268 | |
| HiTrap Heparin HP 1 x 5 mL | Cytiva | 17040701 | Pre-packed heparin column |
| HiTrap Heparin HP 5 x 1 mL | Cytiva | 17040601 | Pre-packed heparin column |
| Immobilon-P PVDF Membrane | Merck Millipore | IPVH00010 | |
| Isoflurane | Braun | 469860 | |
| Ketamine | Dechra Pharmaceuticals | N/A | Nimatek |
| Low-retention microcentrifuge tubes (2 mL) | Fisher Scientific | 11906965 | |
| Lunatic & Stunner Client software | Unchained Labs | N/A | Client analysis software version 8.0.1.235. Software for the consecutive ultraviolet-visible light absorption, static light scattering, and dynamic light scattering analysis of rAAV samples |
| Methanol | Fisher Chemical | M/4000/FP21 | |
| Mouse monoclonal anti-AAV, VP1, VP2 antibody (A69) | American Research Products | 03-61057 | |
| Mouse monoclonal anti-AAV, VP1, VP2, VP3 antibody (B1) | American Research Products | 03-61058 | |
| Neuro2a | American Type Culture Collection | CCL-131 | |
| Normal goat serum | Gibco | 16210064 | |
| NucleoBond Xtra Maxi EF | Macherey-Nagel | 12738422 | |
| NZYColour Protein Marker II | NZYtech | MB09002 | |
| pAAV-CMV-scGFP | Addgene | 32396 | Addgene plasmid # 32396; http://n2t.net/addgene:32396; RRID:Addgene_32396 |
| pAAV-CMV-ssGFP | Addgene | 105530 | Addgene plasmid # 105530; http://n2t.net/addgene:105530; RRID:Addgene_105530 |
| pAAV2/9n | Addgene | 112865 | Addgene plasmid # 112865; http://n2t.net/addgene:112865; RRID:Addgene_112865 |
| Paraformaldehyde | Acros Organics | 10342243 | |
| PBS | Fisher BioReagents | BP2438 | |
| Penicillin-streptomycin | Gibco | 15140-122 | |
| Pluronic F-68 Non-ionic Surfactant (100x) | Gibco | 24040032 | |
| Polyethylenimine MAX, MW 40,000 | Polysciences Europe | 24765 | |
| R500 Series Compact Small Animal Anesthesia Machine - Isoflurane | RWD | N/A | |
| Sample Inlet Valve V9-IS | Cytiva | 29027746 | |
| Sample pump P9-S | Cytiva | 29027745 | |
| Sodium azide | Sigma-Aldrich | S2002 | |
| Sodium chloride | Fisher Scientific | 10428420 | |
| Sodium deoxycholate | Sigma-Aldrich | D6750 | |
| Sodium dodecyl sulfate (SDS) | Fisher Bioreagents | BP166 | |
| Sterile PVDF syringe filter | Fisher Scientific | 15191499 | |
| Stunner Platform | Unchained Labs | 700-2002 | Equipment for the consecutive ultraviolet-visible light absorption, static light scattering, and dynamic light scattering analysis of rAAV samples |
| Superloop 150 mL | Cytiva | 18-1023-85 | |
| Superloop 50 mL | Cytiva | 18-1113-82 | |
| SURE 2 supercompetent cells | Stratagene, Agilent Technologies | HPA200152 | |
| Treated culture dishes | Corning | 734-1711 | |
| Tris base | Fisher BioReagents | BP152 | |
| Tris hydrochloride | Fisher BioReagents | BP153 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Trypsin-EDTA | Gibco | 25200-072 | |
| Wheat Germ Agglutinin (WGA) conjugated with Alexa Fluor 633 | Invitrogen | W21404 | |
| Xylazine | Dechra Pharmaceuticals | N/A | Sedaxylan |
| Zeiss Axio Observer Z1 | Carl Zeiss Microscopy GmbH | N/A | Inverted fluorescence microscope equiped with an EC Plan-Neofluar 10x/0.30 objective and a Plan-Apochromat 40x/0.95 objective |
| Zeiss Axio Scan.Z1 | Carl Zeiss Microscopy GmbH | N/A | Slide scanner fluorescence microscope equipped with a Plan-Apochromat 20x/0.8 objective |
| Zeiss LSM 710 | Carl Zeiss Microscopy GmbH | N/A | Inverted confocal microscope equipped with a Plan-Apochromat 40x/1.4 Oil DIC objective |
| µ-Slide 8 well Ibidi | Ibidi | 80826 | 8-well chamber slide |
Références
- Atchison, R. W., Casto, B. C., Hammon, W. M. Adenovirus-associated defective virus particles. Science. 149 (3685), 754-756 (1965).
- Murlidharan, G., Samulski, R. J., Asokan, A. Biology of adeno-associated viral vectors in the central nervous system. Front Mol Neurosci. 7, 1-9 (2014).
- Muzyczka, N. Use of Adeno-Associated Virus as a General Transduction Vector for Mammalian Cells. Viral Expression Vectors. 158, 97-129 (1992).
- Goncalves, M. A. F. V Adeno-associated virus: from defective virus to effective vector. Virol J. 2, 43(2005).
- Flotte, T. R. Gene therapy progress and prospects: recombinant adeno-associated virus (rAAV) vectors. Gene Ther. 11 (10), 805-810 (2004).
- Grieger, J. C., Choi, V. W., Samulski, R. J. Production and characterization of adeno-associated viral vectors. Nat Protoc. 1 (3), 1412-1428 (2006).
- Ojala, D. S., Amara, D. P., Schaffer, D. V Adeno-Associated Virus Vectors and Neurological Gene Therapy. Neuroscientist. 21 (1), 84-98 (2014).
- Saraiva, J., Nobre, R. J., Pereira de Almeida, L. Gene therapy for the CNS using AAVs: The impact of systemic delivery by AAV9. Journal of Controlled Release. 241, 94-109 (2016).
- Agbandje-McKenna, M., Kleinschmidt, J. AAV capsid structure and cell interactions. Methods Mol Biol. 807, 47-92 (2011).
- Sonntag, F., Schmidt, K., Kleinschmidt, J. A. A viral assembly factor promotes AAV2 capsid formation in the nucleolus. Proc Natl Acad Sci U S A. 107 (22), 10220-10225 (2010).
- Sonntag, F., et al. The assembly-activating protein promotes capsid assembly of different adeno-associated virus serotypes. J Virol. 85 (23), 12686-12697 (2011).
- Gao, G., et al. Clades of Adeno-associated viruses are widely disseminated in human tissues. J Virol. 78 (12), 6381-6388 (2004).
- Gao, G., Vandenberghe, L. H., Wilson, J. M. New recombinant serotypes of AAV vectors. Curr Gene Ther. 5 (3), 285-297 (2005).
- Van Vliet, K. M., Blouin, V., Brument, N., Agbandje-McKenna, M., Snyder, R. O. The role of the adeno-associated virus capsid in gene transfer. Methods Mol Biol. 437, 51-91 (2008).
- Clark, K. R., Voulgaropoulou, F., Fraley, D. M., Johnson, P. R. Cell lines for the production of recombinant adeno-associated virus. Hum Gene Ther. 6 (10), 1329-1341 (1995).
- Inoue, N., Russell, D. W. Packaging cells based on inducible gene amplification for the production of adeno-associated virus vectors. J Virol. 72 (9), 7024-7031 (1998).
- Liu, X., Voulgaropoulou, F., Chen, R., Johnson, P. R., Clark, K. R. Selective Rep-Cap gene amplification as a mechanism for high-titer recombinant AAV production from stable cell lines. Mol Ther. 2 (4), 394-403 (2000).
- Mathews, L. C., Gray, J. T., Gallagher, M. R., Snyder, R. O. [23] Recombinant adeno-associated viral vector production using stable packaging and producer cell lines. Methods Enzymol. 346, 393-413 (2002).
- Gao, G., et al. Purification of recombinant adeno-associated virus vectors by column chromatography and its performance in vivo. Hum Gene Ther. 11 (15), 2079-2091 (2000).
- Xiao, X., Li, J., Samulski, R. J. Production of high-titer recombinant adeno-associated virus vectors in the absence of helper adenovirus. J Virol. 72 (3), 2224-2232 (1998).
- Aponte-Ubillus, J. J., et al. Molecular design for recombinant adeno-associated virus (rAAV) vector production. Appl Microbiol Biotechnol. 102 (3), 1045-1054 (2018).
- Smith, R. H., Levy, J. R., Kotin, R. M. A simplified baculovirus-AAV expression vector system coupled with one-step affinity purification yields high-titer rAAV stocks from insect cells. Mol Ther. 17 (11), 1888-1896 (2009).
- Grimm, D., Kern, A., Rittner, K., Kleinschmidt, J. A. Novel tools for production and purification of recombinant adenoassociated virus vectors. Hum Gene Ther. 9 (18), 2745-2760 (1998).
- Wright, J. F. Transient transfection methods for clinical adeno-associated viral vector production. Hum Gene Ther. 20 (7), 698-706 (2009).
- Yuan, Z., Qiao, C., Hu, P., Li, J., Xiao, X. A versatile adeno-associated virus vector producer cell line method for scalable vector production of different serotypes. Hum Gene Ther. 22 (5), 613-624 (2011).
- Strobel, B., Miller, F. D., Rist, W., Lamla, T. Comparative analysis of cesium chloride- and iodixanol-based purification of recombinant adeno-associated viral vectors for preclinical applications. Hum Gene Ther Methods. 26 (4), 147-157 (2015).
- Zolotukhin, S., et al. Recombinant adeno-associated virus purification using novel methods improves infectious titer and yield. Gene Ther. 6 (6), 973-985 (1999).
- Anderson, R., Macdonald, I., Corbett, T., Whiteway, A., Prentice, H. G. A method for the preparation of highly purified adeno-associated virus using affinity column chromatography, protease digestion and solvent extraction. J Virol Methods. 85 (1-2), 23-34 (2000).
- Okada, T., et al. Scalable purification of adeno-associated virus serotype 1 (AAV1) and AAV8 vectors, using dual ion-exchange adsorptive membranes. Hum Gene Ther. 20 (9), 1013-1021 (2009).
- Guo, P., et al. Rapid and simplified purification of recombinant adeno-associated virus. J Virol Methods. 183 (2), 139-146 (2012).
- Lock, M., et al. Rapid, simple, and versatile manufacturing of recombinant adeno-associated viral vectors at scale. Hum Gene Ther. 21 (10), 1259-1271 (2010).
- Wang, L., Blouin, V., Brument, N., Bello-Roufai, M., Francois, A. Production and Purification of Recombinant Adeno-Associated Vectors. Adeno-Associated Virus: Methods and Protocols. 807, 361-404 (2011).
- Summerford, C., Samulski, R. J. Membrane-associated heparan sulfate proteoglycan is a receptor for adeno-associated virus type 2 virions. J Virol. 72 (2), 1438-1445 (1998).
- Wu, Z., Asokan, A., Samulski, R. J. Adeno-associated Virus Serotypes: Vector Toolkit for Human Gene Therapy. Molecular Therapy. 14 (3), 316-327 (2006).
- Mietzsch, M., Broecker, F., Reinhardt, A., Seeberger, P. H., Heilbronn, R. Differential adeno-associated virus serotype-specific interaction patterns with synthetic heparins and other glycans. J Virol. 88 (5), 2991-3003 (2014).
- McClure, C., Cole, K. L. H., Wulff, P., Klugmann, M., Murray, A. J. Production and titering of recombinant adeno-associated viral vectors. JoVE. (57), e3348(2011).
- Auricchio, A., O’Connor, E., Hildinger, M., Wilson, J. M. A single-step affinity column for purification of serotype-5 based adeno-associated viral vectors. Mol Ther. 4 (4), 372-374 (2001).
- Wang, Q., et al. Identification of an adeno-associated virus binding epitope for AVB sepharose affinity resin. Mol Ther Methods Clin Dev. 2, 15040(2015).
- Mietzsch, M., et al. OneBac: platform for scalable and high-titer production of adeno-associated virus serotype 1–12 vectors for gene therapy. Hum Gene Ther. 25 (3), 212-222 (2014).
- Mietzsch, M., et al. Characterization of AAV-specific affinity ligands: consequences for vector purification and development strategies. Mol Ther Methods Clin Dev. 19, 362-373 (2020).
- Florea, M., et al. High-efficiency purification of divergent AAV serotypes using AAVX affinity chromatography. Mol Ther Methods Clin Dev. 28, 146-159 (2023).
- Koerber, J. T., Jang, J. -H., Yu, J. H., Kane, R. S., Schaffer, D. V. Engineering adeno-associated virus for one-Step purification via immobilized metal affinity chromatography. Hum Gene Ther. 18 (4), 367-378 (2007).
- Zhang, H. -G., et al. Addition of six-His-tagged peptide to the C terminus of adeno-associated virus VP3 does not affect viral tropism or production. J Virol. 76 (23), 12023-12031 (2002).
- Arnold, G. S., Sasser, A. K., Stachler, M. D., Bartlett, J. S. Metabolic biotinylation provides a unique platform for the purification and targeting of multiple AAV vector serotypes. Mol Ther. 14 (1), 97-106 (2006).
- Rabinowitz, J. E., et al. Cross-dressing the virion: the transcapsidation of adeno-associated virus serotypes functionally defines subgroups. J Virol. 78 (9), 4421-4432 (2004).
- Hauck, B., Chen, L., Xiao, W. Generation and characterization of chimeric recombinant AAV vectors. Mol Ther. 7 (3), 419-425 (2003).
- Choi, V., McCarty, D., Samulski, R. AAV hybrid serotypes: improved vectors for gene delivery. Curr Gene Ther. 5 (3), 299-310 (2005).
- Nonnenmacher, M., van Bakel, H., Hajjar, R. J., Weber, T. High capsid–genome correlation facilitates creation of AAV libraries for directed evolution. Mol Ther. 23 (4), 675-682 (2015).
- Kimura, K., et al. A mosaic adeno-associated virus vector as a versatile tool that exhibits high levels of transgene expression and neuron specificity in primate brain. Nat Commun. 14 (1), 4762(2023).
- Issa, S. S., Shaimardanova, A. A., Solovyeva, V. V., Rizvanov, A. A. Various AAV serotypes and their applications in gene therapy: an overview. Cells. 12 (5), 785(2023).
- Foust, K. D., et al. Intravascular AAV9 preferentially targets neonatal neurons and adult astrocytes. Nat Biotechnol. 27 (1), 59-65 (2009).
- Lopes, M. M., et al. A new protocol for whole-brain biodistribution analysis of AAVs by tissue clearing, light-sheet microscopy and semi-automated spatial quantification. Gene Ther. 29 (12), 665-679 (2022).
- Gray, J. T., Zolotukhin, S. Design and construction of functional AAV vectors. Methods Mol Biol. 807, 25-46 (2011).
- Choi, V. W., Asokan, A., Haberman, R. A., Samulski, R. J. Production of recombinant adeno-associated viral vectors. Curr Protoc Hum Genet. , Chapter 12, Unit 12.9-12.9.21 (2007).
- Bennicelli, J., et al. Reversal of blindness in animal models of leber congenital amaurosis using optimized AAV2-mediated gene transfer. Mol Ther. 16 (3), 458-465 (2008).
- Fischer, M. D., Hickey, D. G., Singh, M. S., MacLaren, R. E. Evaluation of an optimized injection system for retinal gene therapy in human patients. Hum Gene Ther Methods. 27 (4), 150-158 (2016).
- Santos, S. D., et al. Contactin-associated protein 1 (Caspr1) regulates the traffic and synaptic content of α-amino-3-hydroxy-5-methyl-4-isoxazolepropionic acid (AMPA)-type glutamate receptors. J Biol Chem. 287 (9), 6868-6877 (2012).
- Sommer, J. M., et al. Quantification of adeno-associated virus particles and empty capsids by optical density measurement. Mol Ther. 7 (1), 122-128 (2003).
- Dong, B., Nakai, H., Xiao, W. Characterization of genome integrity for oversized recombinant AAV vector. Mol Ther. 18 (1), 87-92 (2010).
- Wright, J. F. AAV empty capsids: For better or for worse. Mol Ther. 22 (1), 1-2 (2014).
- Asaad, W., et al. AAV genome modification for efficient AAV production. Heliyon. 9 (4), e15071(2023).
- Wobus, C. E., et al. Monoclonal antibodies against the adeno-associated virus type 2 (AAV-2) capsid: epitope mapping and identification of capsid domains involved in AAV-2-cell interaction and neutralization of AAV-2 infection. J Virol. 74 (19), 9281-9293 (2000).
- Wistuba, A., Kern, A., Weger, S., Grimm, D., Kleinschmidt, J. A. Subcellular compartmentalization of adeno-associated virus type 2 assembly. J Virol. 71 (2), 1341-1352 (1997).
- Qu, G., et al. Separation of adeno-associated virus type 2 empty particles from genome containing vectors by anion-exchange column chromatography. J Virol Methods. 140 (1-2), 183-192 (2007).
- Brument, N., et al. A versatile and scalable two-step ion-exchange chromatography process for the purification of recombinant adeno-associated virus serotypes-2 and -5. Mol Ther. 6 (5), 678-686 (2002).
- Potter, M., et al. A simplified purification protocol for recombinant adeno-associated virus vectors. Mol Ther Methods Clin Dev. 1, 14034(2014).
- Dobnik, D., et al. Accurate quantification and characterization of adeno-associated viral vectors. Front Microbiol. 10, 1570(2019).
- Yang, Q. -E., et al. Rapid quality control assessment of adeno-associated virus vectors via Stunner. GEN Biotechnology. 1 (3), 300-310 (2022).
- Huang, X., et al. AAV2 production with optimized N/P ratio and PEI-mediated transfection results in low toxicity and high titer for in vitro and in vivo applications. J Virol Methods. 193 (2), 270-277 (2013).
- Salganik, M., et al. Evidence for pH-dependent protease activity in the adeno-associated virus capsid. J Virol. 86 (21), 11877-11885 (2012).
- Dong, B., et al. Proteomics analysis of co-purifying cellular proteins associated with rAAV vectors. PLoS One. 9 (2), e86453(2014).
- Grieger, J. C., Soltys, S. M., Samulski, R. J. Production of recombinant adeno-associated virus vectors using suspension HEK293 cells and continuous harvest of vector from the culture media for GMP FIX and FLT1 clinical vector. Mol Ther. 24 (2), 287-297 (2016).
- Wright, J. Product-related impurities in clinical-grade recombinant AAV vectors: characterization and risk assessment. Biomedicines. 2 (1), 80-97 (2014).
- Bennett, A., Mietzsch, M., Agbandje-McKenna, M. Understanding capsid assembly and genome packaging for adeno-associated viruses. Future Virol. 12 (6), 283-297 (2017).
- Myers, M. W., Carter, B. J. Assembly of adeno-associated virus. Virology. 102 (1), 71-82 (1980).
- Mingozzi, F., et al. Overcoming preexisting humoral immunity to AAV using capsid decoys. Sci Transl Med. 5 (194), (2013).
- Hoffman, B. E., Herzog, R. W. Covert warfare against the immune system: decoy capsids, stealth genomes, and suppressors. Mol Ther. 21 (9), 1648-1650 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon