Method Article
Essai de cytotoxicité basé sur des gouttelettes pour évaluer les lymphocytes T à récepteur de l’antigène chimérique au niveau de la cellule unique
Dans cet article
Résumé
Ici, nous décrivons une méthode de création de gouttelettes à double émulsion encapsulant un lymphocyte T et une cellule cible cancéreuse afin d’examiner la destruction cellulaire au niveau de la cellule unique. Cette méthode permet une double quantification des molécules cytotoxiques et de l’apoptose des cellules cibles au sein d’une large population de lymphocytes T.
Résumé
L’évaluation du potentiel cytotoxique des thérapies à base de lymphocytes T, telles que les traitements par lymphocytes T à récepteur antigénique chimérique (CAR), est essentielle à l’évaluation de leur efficacité et constitue une condition préalable à l’application clinique. Cependant, les tests de cytotoxicité traditionnels sont effectués sous forme de tests en vrac et ne fournissent pas d’informations détaillées sur l’hétérogénéité fonctionnelle de la population de cellules CAR-T. Dans cette étude, nous décrivons une méthode basée sur des gouttelettes à double émulsion qui permet une co-encapsulation à grande échelle de cellules CAR-T effectrices uniques avec des cellules cibles uniques tout en permettant une double quantification des molécules effectrices cytotoxiques des cellules T et de la mort cellulaire de la cellule cible. Le protocole décrit une méthode pour la génération et la purification de cellules CAR-T spécifiques de CD19, suivie de leur co-encapsulation dans des gouttelettes avec la lignée cellulaire CD19+ JeKo-1, ainsi que de réactifs pour visualiser la sécrétion de molécules effectrices cytotoxiques (Granzyme B) et la mort cellulaire (à l’aide d’iodure de propidium, PI). Nous montrons comment générer des gouttelettes contenant des cellules CAR-T uniques et des cellules cibles à l’aide d’un dispositif microfluidique disponible dans le commerce pour générer des gouttelettes à double émulsion. De plus, nous fournissons des exemples de la façon d’analyser la diversité fonctionnelle des cellules CAR-T dans les gouttelettes à l’aide d’un équipement de cytométrie en flux standard. Enfin, nous décrivons brièvement la cinétique temporelle et l’hétérogénéité de la destruction des cellules CAR-T spécifiques de CD19. Bien que cette méthode se concentre sur la mort cellulaire à la suite d’une attaque de cellules CAR-T, elle est également adaptable pour examiner d’autres types de cellules T, de cellules immunitaires cytotoxiques et de fonctions des cellules effectrices, telles que la sécrétion de cytokines.
Introduction
La thérapie par cellules T à récepteur antigénique chimérique (CAR) est un domaine en pleine expansion de l’immunothérapie cellulaire du cancer qui s’est avéré efficace contre diverses formes de leucémie, de lymphome1 et de myélome multiple2. Les cellules CAR-T sont générées en modifiant les cellules T avec un récepteur antigénique synthétique qui peut se lier sélectivement à des protéines de surface telles que CD19 ou l’antigène de maturation des cellules B (BCMA), exprimée sur les cellules B, les plasmocytes et leurs homologues malins. Les progrès récents dans la conception des CAR ont permis de cibler plusieurs antigènes simultanément3, en s’appuyant sur plusieurs signaux d’entrée ou en se liant exclusivement à des antigènes associés à des tumeurs, tels que des isoformes de protéines spécifiques4. De plus, plusieurs CARs sont actuellement testés contre des cibles de cancers non hématologiques5.
Les tests de cytotoxicité sont essentiels à la fois pour le développement de CAR et le contrôle de la qualité avant la mise sur le marché des produits cliniques 6,7,8. Cependant, la plupart des tests actuels dépendent de populations massives de cellules CAR-T effectrices ajoutées en excès aux lignées cellulaires cancéreuses, ce qui peut conduire à des résultats faussement positifs en raison d’effets de spectateur9 et entraîner une faible corrélation entre les résultats in vitro et in vivo 10. Étant donné que la prolifération et la persistance à long terme des lymphocytes T dépendent des signaux reçus lors des événements d’activation initiaux11,12, il est très intéressant d’examiner de près les événements cytotoxiques au niveau de la cellule unique.
Pour remédier aux limites des méthodologies en vrac, des études de cytotoxicité au niveau de la cellule unique peuvent être réalisées à l’aide de la cytométrie en flux 13,14,15,16,17 et des tests basés sur l’imagerie 18. Cependant, même si la cytométrie en flux standard offre une résolution unicellulaire pour quantifier et caractériser à la fois les cellules effectrices et cibles, elle ne peut pas déterminer directement la capacité cytotoxique spécifique des cellules CAR-T individuelles envers les cellules cibles. De plus, l’imagerie des cellules CAR-T individuelles interagissant avec leurs cibles respectives en vrac est difficile et prend du temps en raison de la motilité constante des cellules. Pour relever ces défis, de nouveaux outils d’analyse de cellules uniques ont été mis au point qui reposent sur l’appariement de cellules effectrices et cibles dans des espaces confinés à l’aide de réseaux de micropuits19,20,21, de réseaux de microradeaux22, de micropuces23 et de microfluidique de gouttelettes 24,25,26. Ces outils offrent une sensibilité de mesure améliorée, ce qui permet d’étudier moins de cellules en utilisant des volumes de réactifs réduits. Cependant, plusieurs défis subsistent, notamment l’appariement efficace des cellules, le nombre limité d’échantillons pouvant être analysés, le recours uniquement à l’analyse basée sur l’imagerie et les difficultés à récupérer des populations cellulaires viables pour des analyses plus approfondies.
Ici, nous utilisons un dispositif microfluidique disponible dans le commerce qui permet une approche massivement simplifiée de la microfluidique à base de gouttelettes. Ce dispositif permet d’effectuer des analyses et des dosages avancés sur cellule unique à l’aide de gouttelettes à double émulsion (DE) très stables. Comparé aux tests microfluidiques à micropuits et aux tests microfluidiques traditionnels à gouttelettes, le protocole décrit ici ne nécessite pas une expertise approfondie en microfludique.
Les gouttelettes de DE sont de petits compartiments sphériques composés d’une coquille d’huile avec un noyau aqueux en suspension dans une solution aqueuse. La gouttelette aqueuse contient et conserve les cellules, le sécrétome cellulaire, le milieu cellulaire et les réactifs de dosage, ce qui permet d’effectuer des tests complexes dans chaque compartiment. À l’aide du dispositif microfluidique, les gouttelettes de DE sont générées avec un volume défini (environ 100 pL) adapté aux essais d’interaction unicellulaire ou cellule-cellule de mammifères, qui peuvent être générés en très grand nombre (environ 750 000 gouttelettes par échantillon) et dans un court laps de temps (environ 10 min pour 8 échantillons) par des utilisateurs de laboratoire généraux sans connaissances spécialisées en microfluidique. Les gouttelettes générées peuvent être suspendues dans le milieu cellulaire, permettant ainsi la diffusion trans-couche d’O2 et de CO2 et la mise en tampon de l’intérieur tout en conservant les molécules hydrophiles et plus grosses telles que les molécules effectrices sécrétées par les cellules. Les cellules examinées sont ainsi suffisamment tamponnées, ce qui permet d’examiner au fil du temps les interactions cellule-cellule et la dynamique temporelle. Contrairement aux gouttelettes d’émulsion simples (par exemple, les gouttelettes d’eau dans l’huile)27, les gouttelettes DE sont des structures robustes qui ne fusionnent pas ou ne fusionnent pas dans des incubateurs à cellules standard. En raison de leur stabilité et d’une phase externe aqueuse, ils sont également compatibles avec les procédures d’analyse en aval telles que la cytométrie en flux traditionnelle. Ces gouttelettes de picoliter générées par le dispositif microfluidique peuvent donc être utilisées pour l’analyse unicellulaire à haut débit de la fonction cellulaire afin d’élucider l’hétérogénéité fonctionnelle cachée dans les tests en vrac traditionnels.
Ici, nous décrivons un protocole qui utilise des gouttelettes de DE pour examiner le potentiel cytotoxique des CAR spécifiques de CD19 envers les cellules de lymphome. Notre protocole permet d’analyser sur cellule unique la destruction de la cible et la sécrétion de Granzyme B (GzmB) et révèle qu’environ 20 % des cellules CAR-T examinées ici ont un potentiel de destruction immédiat.
Protocole
Les cellules CAR-T utilisées dans cette étude ont été générées par transduction lentivirale de cellules T primaires avec une construction CAR CD19scFv-CD28-CD3ζ-tNGFR dans un laboratoire certifié de biosécurité OGM de classe 2 et déclassifiées après 4 jours selon la norme de l’institut. Les lymphocytes T purifiés provenaient de matériaux excédentaires provenant de dons de sang anonymes et étaient exemptés d’autres approbations éthiques conformément à la loi danoise.
1. Génération de cellules CAR-T
REMARQUE : Le nom des réactifs utilisés ci-dessous utilise des noms génériques et abrégés. Le nom commercial complet se trouve entre parenthèses dans la Table des matériaux.
- Activation, transduction et expansion des lymphocytes T
REMARQUE : L’expression stable des cellules CAR-T CD19 a été obtenue par transduction lentivirale de cellules T humaines primaires isolées de cellules mononucléées de sang périphérique (PBMC) fraîches à l’aide d’un kit d’isolement de cellules T à sélection négative. Le CAR anti-CD19 utilisé est une construction de deuxième génération composée d’un fragment variable à chaîne unique (scFv) FMC63, d’une charnière CD28 et d’un domaine transmembranaire, d’un domaine de costimulation CD28, d’un domaine d’activation CD3ζ28 et d’une version tronquée du récepteur du facteur de croissance nerveuse (tNGFR) pour la surveillance et l’enrichissement des cellules exprimant desCAR29. Le plasmide du vecteur de transfert a été synthétisé de novo, et un lentivirus de troisième génération a été fabriqué en le combinant avec pMDLg/pRRE (plasmide Addgene #12251), pRSV-Rev (plasmide Addgene #12253) et pMD2.G (plasmide Addgene #12259)30. Les trois derniers plasmides étaient un cadeau de Didier Trono. Le virus brut a été concentré à l’aide d’un réactif à base de PEG, et la multiplicité de l’infection (MOI) a été déterminée en transduisant les cellules SUP-T1 et en mesurant le tNGFR par cytométrie en flux conformément au protocole standard31.- Transférez 2 x 106 cellules T humaines primaires dans une boîte de culture à 12 puits et stimulez-les avec des billes enrobées de CD3/CD28 à un rapport bille/cellule de 1:1 dans 1 mL de milieu RPMI-1640 complet (10 % de sérum de veau fœtal inactivé par la chaleur, 1 % de pénicilline/streptomycine) complété par 100 unités/mL d’interleukine-2 humaine recombinante (IL-2). Incuber les cellules à 37°C dans un incubateur humidifié à 5 % de CO2 pendant 24 h.
- Ajoutez des particules lentivirales aux lymphocytes T activés à un moment d’inertie de 5 à 10. Mélangez doucement et incubez les cellules pendant 72 h. Inclure les lymphocytes T non transduits (ATN) comme contrôle négatif.
- Le 3e jour après la transduction, retirez les billes d’activation CD3/CD28 en récoltant les lymphocytes T dans un tube de 1,5 mL et en plaçant le tube sur un support magnétique pendant 1 à 2 min. Transférez le surnageant contenant les cellules dans un nouveau tube de 1,5 mL.
- Effectuez un comptage de cellules à l’aide d’un compteur de cellules automatisé et ajustez la densité cellulaire à 1 x 106 cellules/ml dans un milieu RPMI-1640 complet complété par 100 U/mL d’IL-2. Continuez à développer les cellules T jusqu’à ce que le nombre total de cellules CAR-T atteigne au moins 1,5 x 106 (généralement environ 6 x 106 cellules T au total) dans la suspension avant de procéder à l’enrichissement en NGFR (voir ci-dessous).
- Surveillez le nombre de cellules tous les deux jours et ajustez la concentration à 1 x 106 cellules/ml en ajoutant un milieu frais complété par 100 unités/ml d’IL-2 pour assurer un état cellulaire optimal pendant l’expansion.
- Enrichissement des lymphocytes T exprimant des CAR-T
- Transvaser 6 x 10 6 cellulesT transductées ou plus dans un tube de 15 mL et centrifuger à 300 x g pendant 5 min. Remettre en suspension la pastille cellulaire dans 320 μL de PBS complété par 0,5 % d’albumine sérique bovine (BSA) pour fabriquer du PBSA et ajouter 40 μL de billes magnétiques anti-NGFR. Bien mélanger et incuber sur glace pendant 15 min.
REMARQUE : Ce protocole suppose une efficacité de transduction d’environ 25 %, ce qui permet d’obtenir des cellules CAR-T >1,5 x 106 après enrichissement. Nous ne recommandons pas de commencer avec moins de 6 x 10cellules 6 , car cela peut entraîner une mauvaise récupération des cellules CAR-T. - Ajouter 1640 μL de PBSA pour porter le volume final à 2 mL, puis procéder à la séparation magnétique à l’aide de colonnes de séparation selon les instructions du fabricant.
- Transvaser 6 x 10 6 cellulesT transductées ou plus dans un tube de 15 mL et centrifuger à 300 x g pendant 5 min. Remettre en suspension la pastille cellulaire dans 320 μL de PBS complété par 0,5 % d’albumine sérique bovine (BSA) pour fabriquer du PBSA et ajouter 40 μL de billes magnétiques anti-NGFR. Bien mélanger et incuber sur glace pendant 15 min.
- Détection de l’expression de CAR par cytométrie en flux
REMARQUE : Nous vous recommandons de déterminer le pourcentage de cellules exprimant le CAR et leur viabilité à ce stade, afin de vous assurer que les cellules sont correctes avant d’effectuer le test ci-dessous. Le rapport CD4 et CD8 et le phénotype de la mémoire peuvent également être déterminés à ce stade. Voir la figure 1 pour des graphiques représentatifs.- Transférez 2,5 x 10 cellulesT 5 provenant de cultures transduites et non transduites dans des tubes de cytométrie en flux séparés. Laver les cellules 2 fois avec 200 μL de PBS, en centrifugeant à 300 x g pendant 5 min après chaque lavage.
- Préparez un mélange d’anticorps contenant des anticorps anti-CD19 CAR FMC63 Idiotype PE, anti-CD3 BV480 et des anticorps anti-NGFR FITC, chacun dilué à 1:100 dans du PBS. Remettre les cellules en suspension dans 50 μL du mélange d’anticorps et incuber à 4 °C pendant 20 min.
- Laver les cellules 2 fois avec 200 μL de PBS, en centrifugeant à 300 x g pendant 5 min après chaque lavage. Remettre les cellules en suspension dans 200 μL de PBS et analyser par cytométrie en flux.
2. Génération de gouttelettes encapsulant les lymphocytes T et les cellules cibles
REMARQUE : Avant l’encapsulation, les cellules T et les cellules JeKo-1 sont colorées avec différentes couleurs cellulaires pour surveiller le contenu cellulaire des gouttelettes. Les cellules sont encapsulées avec des réactifs de dosage et incubées pendant 2 à 6 h avant que les gouttelettes ne soient analysées sur le cytomètre en flux (voir figure 2).
- Coloration des cellules avant encapsulation
- Préparez des solutions de travail de deux colorants fluorescents (nous avons utilisé un colorant violet et un colorant rouge lointain) en diluant les solutions mères (préparées selon les instructions du fabricant) à 1:5 000 dans du dPBS.
- Pour chaque donneur, transférez 2,5 cellules x 106 CAR T, 2,5 x 106 NTD T et 15 x 106 cellules JeKo-1 dans des tubes à centrifuger séparés (tubes de 15 ml ou 50 ml) et centrifugez à 300 x g pendant 5 min. Dans cette expérience, 0,5 x 106 cellules effectrices ont été encapsulées avec 1,5 x 106 cellules cibles pour chaque échantillon préparé.
REMARQUE : Le nombre de cellules transférées ici est destiné à 4 échantillons d’encapsulation avec chaque type de cellule effectrice pour chaque donneur. Réduisez le nombre de cellules et de réactifs en fonction du nombre d’échantillons nécessaires. De plus, le nombre exact de cellules par échantillon peut être mis à l’échelle en fonction des besoins expérimentaux. - Retirer les pastilles de cellules effectrices surnageantes et remises en suspension dans la solution de travail 1:5 000 du colorant fluorescent violet et les pastilles de cellules cibles (JeKo-1) dans la solution de travail 1:5 000 du colorant fluorescent rouge lointain. Remettez bien les cellules en suspension à l’aide d’une pipette et à une concentration cellulaire de 1 x 10 solution de colorationde 6 cellules/mL.
- Incuber les cellules pendant 20 min dans un incubateur de CO2 humidifié à 37 °C. Centrifuger les cellules à 300 x g pendant 5 min, retirer le surnageant et remettre en suspension dans 15 mL de milieu RPMI-1640 complet pour laver les cellules.
- Répétez le lavage et la centrifugation à 300 x g pendant 5 min. Retirer le surnageant et remettre en suspension les pastilles de cellules effectrices dans 225 μL de RPMI-1640 complet et les pastilles de cellules cibles dans 450 μL de RPMI-1640 complet.
- Ajouter 30 μL d’un milieu dégradé aux suspensions de cellules effectrices et 60 μL du même milieu dégradé aux suspensions de cellules cibles. Bien avant le pipetage, remettre en suspension la solution mère de milieu dégradé.
- Ajouter 15 μL d’un stock PI de 20 μg/mL dans les suspensions de cellules effectrices et 30 μL de stock PI dans les suspensions de cellules cibles. La concentration finale en IP sera finalement de 1 μg/mL après l’ajout du substrat Granzyme B (GzmB) ci-dessous.
- Préparez une dilution 1:10 du substrat GzmB dans le RPMI-1640 complet et ajoutez 30 μL de cette dilution de substrat GzmB à la suspension de cellules effectrices et 60 μL de celle-ci aux suspensions de cellules cibles. Bien mélanger avec la pipette.
REMARQUE : Le volume total des réactifs ajoutés à chaque pastille de cellule effectrice doit maintenant être de 300 μL et celui des pastilles de cellule cible de 600 μL. La concentration du milieu de gradient dans ces solutions doit être de 10 %, la concentration de PI de 1 μg/mL et la concentration du substrat GzmB de 1:100. - Marquage alternatif à l’aide d’anticorps CD3, CD4 et CD8
REMARQUE : Dans certains cas, il peut être intéressant de marquer davantage les cellules effectrices avant la co-encapsulation avec les cellules cibles. Nous montrons ici la preuve de principe de ce type de marquage à l’aide d’anticorps CD3, CD4 et CD8 et de PBMC (voir Figure 3).- Transvasez 2,0 x 106 PBMC dans un tube de 1,5 mL et faites tourner les cellules pendant 5 minutes à 300 x g. Retirer le surnageant et remettre en suspension la pastille cellulaire dans 200 μL de dPBS avec 0,5 % de BSA. Répartissez le contenu en deux tubes de 2 ml.
- Ajoutez 5 μL d’anti-CD3-APC et 5 μL d’anti-CD4-StarBright Violet 760 dans l’un des tubes. Ajoutez 20 μL d’anti-CD3-FITC et 5 μL d’anti-CD8-StarBright Violet 760 dans l’autre tube.
- Bien mélanger et incuber 30 min à température ambiante dans l’obscurité. Ajoutez 1 mL de tampon de lavage dans les cellules, faites tourner à 300 x g pendant 5 min, retirez le surnageant et remettez en suspension les pastilles cellulaires dans 1 mL de dPBS avec 0,5 % de BSA.
- Faites tourner les cellules à 300 x g pendant 5 min, retirez le surnageant et remettez les granules de cellules en suspension dans 150 μL de RPMI sans rouge de phénol et sans milieu à gradient de 10 %. Encapsuler comme décrit à l’étape 2.2 à l’aide d’un RPMI sans rouge de phénol, d’une solution stabilisante à 33 % pour les cellules et d’un milieu à gradient de 10 % comme milieu externe.
- Encapsulation des cellules
- Préchauffez une cartouche d’encapsulation et une solution stabilisante à température ambiante avant l’encapsulation. Préparez un stock de milieu externe composé d’un milieu RPMI-1640 complet, d’une solution stabilisante à 33 % pour les cellules et d’un milieu à gradient de 10 %.
- Pour chaque échantillon d’encapsulation, préparez une solution d’échantillon cellulaire avec des cellules effectrices à cibles mixtes. Remettre en suspension les solutions cellulaires bien préparées à l’aide d’une pipette et mélanger 65 μL de suspension de cellule effectrice préparée avec 65 μL de suspension de cellule cible préparée dans des tubes de 1,5 mL (tous deux préparés à l’étape 2.1).
- Procédez immédiatement au chargement des puits indiqués de la cartouche d’encapsulation avec des réactifs dans l’ordre suivant pour assurer une encapsulation correcte. Chargez un ensemble de puits pour chaque échantillon d’encapsulation préparé.
Bien #A : 400 μL de milieu extérieur.
Bien #D : 40 μL de support extérieur sur la petite étagère.
Bien #C : 120 μL de solution pré-mélangée avec des cellules cibles (JeKo-1) et des cellules effectrices. Remettez bien les cellules en suspension avec une pipette juste avant le chargement.
Bien #B : 250 μL d’huile.
REMARQUE : Chaque cartouche peut être chargée avec jusqu’à 8 échantillons pour l’encapsulation en parallèle. - Procédez immédiatement à l’étanchéité du joint et au chargement de la cartouche dans l’instrument tout en évitant d’incliner, de secouer ou de cogner la cartouche chargée. Fermez soigneusement le joint sur la cartouche.
- Transférez soigneusement la cartouche dans le dispositif microfluidique et démarrez l’encapsulation comme décrit dans le manuel d’utilisation.
- Les gouttelettes générées ont des densités plus élevées que le milieu extérieur environnant et se sédimentent rapidement au fond du puits de collecte (puits #D). Prélever chaque gouttelette produite (toutes les gouttelettes et le milieu externe qui les entoure) en mettant en suspension les gouttelettes sédimentées du puits #D dans le milieu de recouvrement et en les transférant dans un tube d’ADN de 2 mL à faible liaison muni d’un couvercle. Lavez Well #D avec le média extérieur restant du puits #A pour recueillir les gouttelettes restantes.
- Lorsque les gouttelettes se sont sédimentées dans les tubes collecteurs (cela prend environ 1 min), vérifiez la production de gouttelettes en les examinant au microscope à fond clair. Pour ce faire, remplissez une pointe de pipette de 10 μL avec un échantillon : env. 1/3 avec des gouttelettes de la surface de la phase gouttelette (phase blanche) et env. 2/3 avec le milieu extérieur de recouvrement pour remplir la pointe. Chargez immédiatement l’échantillon sur une lame de verre à 8 chambres et examinez les gouttelettes par microscopie à champ clair à un grossissement de 4x et 20x pour confirmer le chargement des gouttelettes avec les cellules.
REMARQUE : Il est important que les gouttelettes de cette manière soient aspirées dans un plus grand volume du milieu environnant.
- Incubation
REMARQUE : Les gouttelettes peuvent maintenant être incubées dans 2 mL de tubes à faible liaison d’ADN dans un incubateur standard humidifié à 37 °C et à 5 % de CO2. Nous vous recommandons d’utiliser ces tubes, car ils ont des propriétés de surface optimales pour la culture des gouttelettes.- À l’aide d’une aiguille à seringue (23G), percez soigneusement et en toute sécurité le couvercle du nombre requis de tubes d’ADN à faible liaison de 2 ml. Cela garantira la diffusion libre de CO2/O2 tout en empêchant l’évaporation du fluide.
- Ajouter 1 mL de milieu extérieur dans chaque tube d’incubation. Les gouttelettes doivent être incubées avec au moins 5 fois le volume du support extérieur pour assurer une bonne mise en mémoire tampon. Le volume du milieu externe ajouté peut être augmenté en fonction du métabolisme des cellules appliquées.
- Remettre en suspension les gouttelettes générées dans le milieu de recouvrement et répartir chaque production dans trois des tubes d’incubation préparés (un pour chaque mesure de point temporel). Les gouttelettes sont lourdes et rapides aux sédiments, il est donc important de remettre le stock en suspension entre chaque transfert.
REMARQUE : Le nombre de tubes d’incubation dans lesquels les productions de gouttelettes sont réparties peut varier, mais nous recommandons de diviser une production en un maximum de 4 tubes d’incubation pour s’assurer que chaque échantillon contient suffisamment d’événements pour l’analyse. - Placez les tubes à la verticale dans l’incubateur pour une incubation de 2 h, 4 h ou 6 h après la génération de gouttelettes.
3. Analyse en aval des gouttelettes
- Microscopie
- Après l’incubation, transférez un petit nombre de gouttelettes sur une lame de microscope comme décrit à l’étape 2.2.7 et analysez-les par microscopie à fond clair et à fluorescence à l’aide d’un microscope à fluorescence standard doté d’une configuration laser et d’un filtre appropriés.
REMARQUE : Les gouttelettes peuvent être détectées par microscopie à fond clair. Les cellules T colorées en violet peuvent être détectées avec un filtre DAPI, les cellules JeKo-1 colorées en rouge lointain avec un filtre APC, le signal GzmB-FAM avec un filtre FITC et le signal PI avec un filtre PE. Le signal PI d’intérêt le plus élevé peut être difficile à visualiser par microscopie, car les cellules apoptotiques précoces ont une émission de pic plus faible que les cellules apoptotiques tardives. Voir la figure 4 pour des données représentatives.
- Après l’incubation, transférez un petit nombre de gouttelettes sur une lame de microscope comme décrit à l’étape 2.2.7 et analysez-les par microscopie à fond clair et à fluorescence à l’aide d’un microscope à fluorescence standard doté d’une configuration laser et d’un filtre appropriés.
- Cytométrie en flux
- Après l’incubation, remettre en suspension chaque échantillon de gouttelettes dans le milieu de recouvrement et le transférer dans des tubes FACS de 5 ml. Analysez les gouttelettes à l’aide d’un cytomètre en flux standard en suivant les directives générales énumérées ci-dessous.
- Utilisez un cytomètre en flux avec une configuration laser et un filtre appropriés. Avec le panel sélectionné de colorations cellulaires et le dosage, la compensation de couleur n’est pas nécessaire sur le cytomètre en flux utilisé ici. Si d’autres couleurs sont appliquées ou si la configuration du cytomètre en flux appliqué est différente, une compensation peut être nécessaire.
- Utilisez le FSC-H comme seuil de déclenchement pour exclure le bruit de fond des petits événements pétroliers.
- Les gouttelettes sont lourdes et se déposent au fond des tubes FACS. Assurez-vous que le SIP atteint les gouttelettes. Les gouttelettes doivent être suspendues dans au moins 5 fois le volume de la mémoire tampon externe avant l’acquisition.
- Les événements Droplet sont acquis à grande vitesse. Enregistrer les signaux de hauteur d’intensité (H) pour le FSC et le SSC et les fluorophores examinés, car les mesures de hauteur d’intensité sont utilisées lors de l’analyse des gouttelettes. Ajustez les gains pour avoir une bonne séparation des événements positifs et négatifs.
REMARQUE : Nous avons utilisé le canal FITC pour mesurer le signal GzmB, le canal Pacific Blue pour détecter les cellules T colorées en violet, le canal APC pour détecter les cellules JeKo-1 colorées en rouge lointain et le canal PE pour détecter le signal PI. - Enregistrez suffisamment d’événements pour l’analyse en aval. Ici, nous avons enregistré 4,5 - 8,3 x 103 des gouttelettes de co-encapsulation par point temporel.
- Lavez le cytomètre en flux dans le SIP après chaque série d’acquisitions de gouttelettes, en utilisant des solutions de nettoyage et de rinçage standard. Nettoyez soigneusement le cytomètre en flux à la fin de l’expérience. Voir les figures 4 et 5 pour des données représentatives.
- Après l’incubation, remettre en suspension chaque échantillon de gouttelettes dans le milieu de recouvrement et le transférer dans des tubes FACS de 5 ml. Analysez les gouttelettes à l’aide d’un cytomètre en flux standard en suivant les directives générales énumérées ci-dessous.
- Analyse des données
- Calculez le pourcentage de lymphocytes T sécrétant GzmB en fonction du pourcentage de co-encapsulations de lymphocytes T GzmB positifs et de cellules JeKo-1 normalisés à la viabilité des lymphocytes T déterminée dans les populations de gouttelettes de lymphocytes T uniquement au point en question, comme suit :
Gouttelettes Lymphocytes T+, Jeko-1+, GzmB+ / (Gouttelettes Lymphocytes T+, Jeko-1+ - Gouttelettes Lymphocytes T morts+, Jeko-1+)
où Gouttelettes Lymphocytes T morts+, Jeko-1+ est estimé à partir de la mort en gouttelettes avec des lymphocytes T uniquement : GouttelettesLymphocytes T morts+, Jeko-1+ = GouttelettesLymphocytes T+, Jeko-1+x Gouttelettes Lymphocytes T morts+, Jeko-1-. - Calculez le pourcentage de lymphocytes T qui ont tué leurs cellules cibles comme suit :
Pourcentage de lymphocytes T cytotoxiques = (Mort observée - Mort en arrière-plan)/(100 - Mort en arrière-plan)
où la mort de fond est la mort présente dans les gouttelettes (lymphocytes T+, Jeko-1+) résultant soit d’une mort non liée à la mort, soit d’une mort survenue avant l’encapsulation. - Estimez la mort de fond due à la mort cellulaire dans les gouttelettes (lymphocytes T+, Jeko-1-) et les gouttelettes (lymphocytes T, Jeko-1+) :
Mort en arrière-plan = (1- (1- taux de mortalité dans les gouttelettesTcell-, Jeko-1+) x (1- taux de mortalité dans les gouttelettesT lymphocy+, Jeko-1-)) x 100
où le taux de mortalité est mesuré directement en tant que % de gouttelettes PI positives dans les gouttelettes uniquement des cellules T et les gouttelettes JeKo-1 uniquement, respectivement. Tous les calculs sont effectués à l’aide de chiffres spécifiques au temps d’échantillonnage.
- Calculez le pourcentage de lymphocytes T sécrétant GzmB en fonction du pourcentage de co-encapsulations de lymphocytes T GzmB positifs et de cellules JeKo-1 normalisés à la viabilité des lymphocytes T déterminée dans les populations de gouttelettes de lymphocytes T uniquement au point en question, comme suit :
Résultats
Après la transduction, les lymphocytes T ont été analysés pour l’expression de CAR par cytométrie en flux à l’aide d’anticorps anti-CD3 et anti-NGFR. La population de cellules CAR-T a ensuite été enrichie à l’aide de billes magnétiques anti-NGFR, ce qui a donné une pureté de plus de 98 % pour les deux donneurs (Figure 1A-B). Le rapport CD4/CD8 et le phénotype mémoire des cellules CAR triées utilisées ont également été quantifiés, à l’aide d’une stratégie de porte standard utilisant des anticorps CCR7 et CD45RA. Ces données montrent que le rapport CD4/CD8 des cellules CAR-T utilisées était de 0,73, et que les cellules étaient principalement de phénotype à mémoire naïve (Figure 1C-E).
Les cellules CAR-T et les cellules JeKo-1 ont été colorées avec des colorants violets et rouge lointain, respectivement, et encapsulées dans des gouttelettes de DE50 avec des réactifs de dosage, un substrat GzmB et un PI (Figure 2). Alternativement, les cellules CAR-T peuvent également être marquées à l’aide d’anticorps tels que les anti-CD4 ou anti-CD8 (Figure 3), ce qui permet une caractérisation plus détaillée des cellules T. Chaque suspension de lymphocytes T (CAR T ou NTD) a été mélangée à une suspension de cellules JeKo-1 immédiatement avant l’encapsulation à un rapport effecteur/cellule cible de 1:3 (0,5 x 106 lymphocytes T et 1,5 x 106 JeKo-1). Après encapsulation, chaque production de gouttelettes (cellules CAR-T + JeKo-1 et NTD + JeKo-1) a été divisée en trois tubes d’incubation pour une incubation de 2 h, 4 h et 6 h, respectivement. Les cellules ont été incubées à l’intérieur des gouttelettes à 37 °C dans 5 % de CO2 , puis analysées par microscopie et cytométrie en flux aux points temporels indiqués.
Nous avons réalisé une microscopie à fluorescence de gouttelettes à double émulsion après 4 h d’incubation (Figure 4). L’intensité du signal de fluorescence verte (FITC) illustre le niveau d’activité GzmB sécrétée par les cellules CAR-T ou NTD à l’intérieur des gouttelettes DE50. La figure 4B présente des images de microscopie à contraste de phase et à fluorescence d’une seule gouttelette GzmB positive contenant une cellule CAR T et une cellule cible JeKo-1 en contact étroit.
Ensuite, les gouttelettes de DE50 ont été analysées par cytométrie en flux pour quantifier le pourcentage de cellules CAR-T présentant une sécrétion précoce de GzmB et une activité de destruction des cellules cytotoxiques (Figure 5). La coloration avant incubation des cellules cibles de JeKo-1 avec un colorant rouge lointain et des cellules T effectrices avec un colorant violet avant encapsulation a facilité l’identification de trois populations distinctes de gouttelettes contenant des cellules, car les cellules sont distribuées en gouttelettes sur la base de la distribution de Poisson. Les populations de gouttelettes identifiées sont les gouttelettes avec des lymphocytes T seuls, les cellules JeKo-1 seules et les cellules T et les cellules JeKo-1 ensemble (figure 5A). Des gouttelettes co-encapsulant des lymphocytes T avec des cellules JeKo-1 ont été contrôlées et analysées pour détecter des signaux indiquant l’activité GzmB (figure 5B) et la mort cellulaire, comme indiqué par PI (figure 5C).
L’encapsulation des cellules dans les gouttelettes suit la distribution de Poisson, et quatre populations de gouttelettes différentes sont obtenues (Figure 6A). La figure 6B montre le niveau de gouttelettes GzmB positives dans toutes les populations de gouttelettes après 6 h d’incubation de gouttelettes à partir desquelles le pourcentage de lymphocytes T sécrétant spontanément de GzmB peut être déterminé. Ces données indiquent la grande spécificité de la méthode, car seules les cellules CAR-T sécrètent GzmB.
Enfin, nous avons quantifié les niveaux de GzmB et de PI à travers les points temporels, après 2 h, 4 h et 6 h de co-incubation. À titre de référence, les gouttelettes contenant uniquement des lymphocytes T et uniquement des cellules JeKo-1 ont été analysées pour examiner la mort cellulaire de fond dans chaque population (Figure 6C). La mort cellulaire de fond a été utilisée pour déterminer le pourcentage de cellules T vivantes sécrétant GzmB et tuant les cellules cibles dans la population de cellules effectrices (Figure 7), comme décrit à l’étape 3.3. Les cellules CAR-T de deux donneurs ont montré une augmentation dépendante du temps de la sécrétion de GzmB induite par les cellules cibles et de l’activité de destruction des cellules. Près de 36 % des cellules CAR-T vivantes du donneur 1 et 31 % du donneur 2 avaient sécrété GzmB après 6 heures de co-encapsulation avec la cellule cible, une augmentation significative par rapport aux cellules T témoins des ATN (Figure 7A). De même, environ 21 % des cellules CAR-T du donneur vivant 1 et 22 % des cellules CAR-T du donneur vivant 2 avaient tué des cellules cibles, comme l’indique un signal PI positif (Figure 7B). Le pourcentage de cellules CAR-T qui avaient sécrété GzmB dépassait le pourcentage de cellules cibles tuées à chaque point temporel, ce qui correspond à la séquence attendue des événements de cytotoxicité médiée par GzmB. L’ensemble de ces données montre que la méthode présentée ici permet de caractériser l’hétérogénéité de la cytotoxicité des lymphocytes T individuels au sein d’une population de cellules ainsi que des comparaisons entre différentes populations.
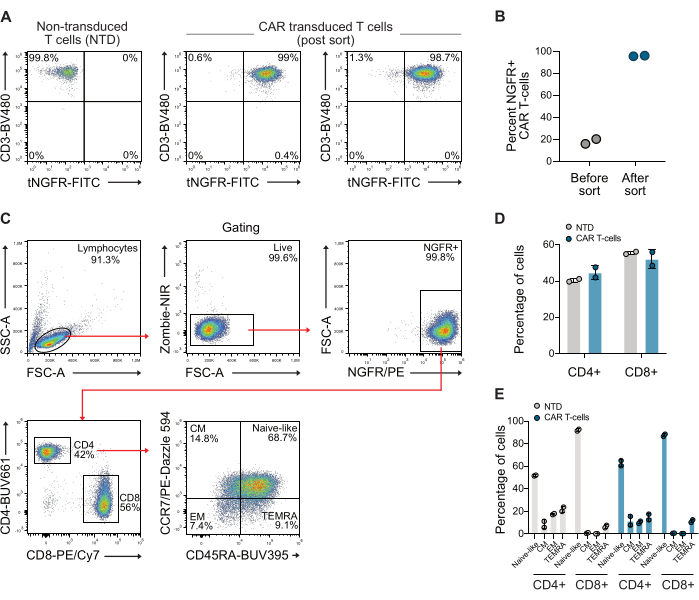
Figure 1 : Graphiques représentatifs de cytométrie en flux après tri NGFR. (A) Après environ 10 jours d’expansion, les cellules CAR-T ont été triées à l’aide de microbilles spécifiques du NGFR et d’un tri magnétique, ce qui a donné une population de cellules CAR-T composée à >98 % de cellules CAR-T. (B) Quantification des cellules CAR-T de deux donneurs utilisés dans cette étude avant et après le tri. (C) Stratégie de déclenchement utilisée pour examiner le rapport CD4/CD8 et le phénotype mémoire des lymphocytes T. (D) Quantification des CD4 et CD8 des lymphocytes T utilisés. (E) Quantification du phénotype mémoire des lymphocytes T utilisés. Abréviation : CM = mémoire centrale, EM = mémoire effectrice, NTD = non transduction, TEMRA = cellules effectrices différenciées en phase terminale. Veuillez cliquer ici pour voir une version agrandie de cette figure.
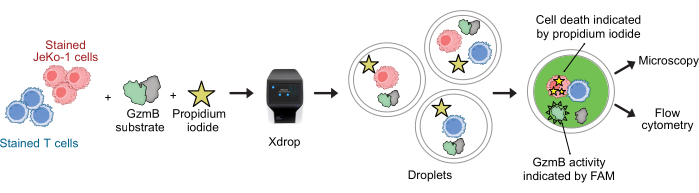
Figure 2 : Flux de travail pour le test combiné de sécrétion et de cytotoxicité de GzmB avec résolution unicellulaire en gouttelettes. Avant l’encapsulation dans des gouttelettes DE, les cellules cible et effectrice sont colorées séparément à l’aide de colorants de cellules violettes et rouges lointaines. À l’aide du dispositif microfluidique et de la cartouche d’encapsulation, les cellules effectrices sont co-encapsulées avec les cellules cibles dans des gouttelettes avec un milieu cellulaire, un PI et un substrat peptidique GzmB marqué FAM. Le dosage et l’incubation ont lieu à l’intérieur des gouttelettes. L’activité sécrétée par GzmB est indiquée par l’émission d’une fluorescence verte qui se produit après que GzmB ait clivé le substrat. La mort cellulaire est indiquée par l’IP. Après l’incubation, les gouttelettes de DE50 sont analysées par microscopie et/ou cytométrie en flux. Veuillez cliquer ici pour voir une version agrandie de cette figure.
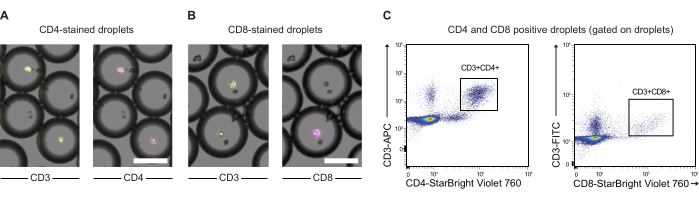
Figure 3 : Analyse de PBMC encapsulés dans des gouttelettes pré-marqués avec des anticorps anti-CD3, anti-CD4 et anti-CD8. (A) Images microscopiques de gouttelettes avec des PBMC encapsulés pré-marqués avec des anticorps anti-CD3 (APC) et anti-CD4 (NIR). Barre d’échelle = 100 μm. (B) As (A), mais avec un marquage CD3 (FITC) et anti-CD8 (NIR) à la place. Barre d’échelle = 100 μm. (C) Analyse par cytométrie en flux des mêmes gouttelettes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
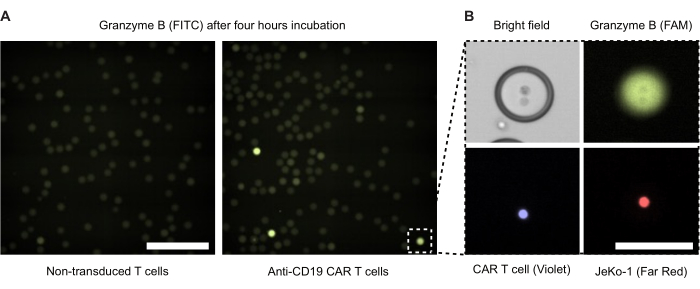
Figure 4 : Images au microscope de gouttelettes avec des cellules effectrices et cibles. Les images ont été prises après 4 h d’incubation dans un incubateur à cellules humidifiées à 37 °C et 5 % de CO2. (A) Gouttelettes d’un échantillon avec des cellules NTD et JeKo-1 encapsulées (à gauche) ou des cellules CAR T et des cellules JeKo-1 (à droite) imagées par microscopie à fluorescence à l’aide du canal FITC pour détecter les gouttelettes GzmB positives (vertes). Barre d’échelle = 500 μm. (B) Une seule gouttelette imagée par contraste de phase, FITC (GzmB), APC (cellule JeKo-1) et DAPI (cellule CAR T). Barre d’échelle = 100 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
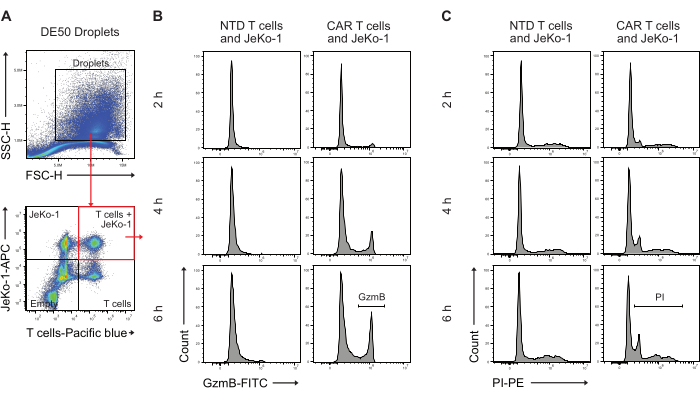
Figure 5 : Stratégie de déclenchement pour l’analyse des gouttelettes par cytométrie en flux après l’incubation. (A) Le déclenchement a été effectué par diffusion directe et latérale pour identifier les gouttelettes, suivi de la sélection de la porte contenant les gouttelettes. Les événements à l’extérieur de la porte représentent des gouttelettes d’huile produites comme sous-produit de la production de gouttelettes à double émulsion. L’analyse ultérieure par fluorescence des gouttelettes dans les canaux correspondant aux colorants cellulaires appliqués identifie quatre populations de gouttelettes : les gouttelettes contenant à la fois des cellules T et des cellules JeKo-1 (carré rouge) ; gouttelettes avec les cellules JeKo-1 seules ; gouttelettes avec les lymphocytes T seuls ; et des gouttelettes vides. (B) Histogrammes représentatifs du signal GzmB dans les gouttelettes doublement positives à travers les points temporels et entre les cellules NTD et CAR T. (C) Histogrammes représentatifs du signal PI dans les gouttelettes doublement positives à travers les points temporels et entre les cellules NTD et CAR T. Toutes les mesures de gouttelettes sont effectuées sous forme de mesures de hauteur d’intensité (H). Veuillez cliquer ici pour voir une version agrandie de cette figure.
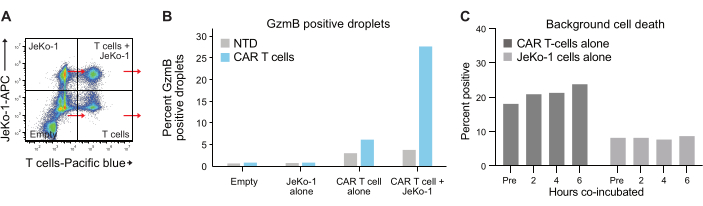
Figure 6 : Populations de contrôle interne et de référence. (A) Chacun des quatre quadrants de la figure 5A a été sélectionné, et GzmB et PI ont été mesurés dans chacun d’eux. Ce type de quantification permet de mesurer et de soustraire les signaux de fond avant qu’une analyse finale de l’efficacité des lymphocytes T ne soit effectuée. (B) Graphique montrant la fréquence des gouttelettes GzmB positives après 6 h d’incubation pour chacune des quatre populations de gouttelettes, illustrée par les données de l’échantillon CAR T du donneur 1. (C) La mort cellulaire de fond a été déterminée dans les populations de gouttelettes de contrôle de JeKo-1 uniquement et de cellules T uniquement à chaque point temporel mesuré, avec des données provenant du donneur 1. La mort cellulaire de fond est utilisée pour calculer la fréquence des cellules effectrices vivantes sécrétant et tuant les cellules GzmB illustrée à la figure 7 et expliquée à l’étape 3.3. Abréviations : Pre = encapsulation. Veuillez cliquer ici pour voir une version agrandie de cette figure.
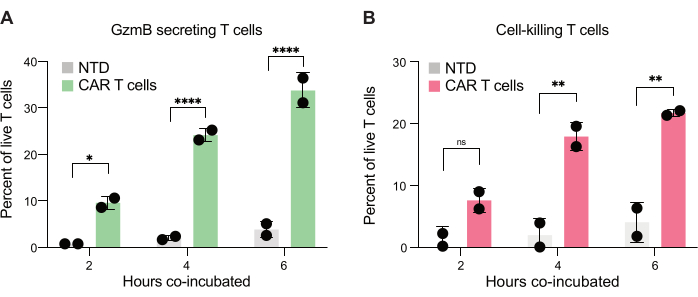
Figure 7 : Quantification des lymphocytes T capables de sécréter GzmB et de tuer les lymphocytes JeKo-1 dans des gouttelettes doublement positives. Des gouttelettes doublement positives provenant de deux donneurs ont été examinées après une co-incubation de 2 h, 4 h et 6 h dans des gouttelettes. (A) Fréquence des lymphocytes T sécrétant GzmB et comparaison entre les lymphocytes T NTD et les lymphocytes T CAR. (B) Fréquence à laquelle les lymphocytes T rencontrent la cellule cible tuant la cellule cible co-encapsulée. Abréviations : GzmB = granzyme B, NTD = non transduit, PI = iodure de propidium. * = Valeur P < 0,05, ** = Valeur P < 0,005, **** = Valeur P < 0,0001 par ANOVA à deux facteurs. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Nous présentons ici une méthode d’examen du potentiel cytotoxique des lymphocytes T au niveau de la cellule unique à l’aide d’un dispositif de microfluides facile à utiliser et disponible dans le commerce pour évaluer le potentiel cytotoxique des cellules CAR-T CD19-CD28-CD3z lors de la co-encapsulation avec la lignée cellulaire JeKo-1 du lymphome du manteau CD19-positif.
Ce protocole comporte plusieurs étapes critiques. Tout d’abord, nous recommandons de retirer les billes de stimulation des lymphocytes T au moins 48 heures avant d’effectuer tout dosage, afin d’éviter les résultats faussement positifs. Deuxièmement, l’efficacité de la transduction des lymphocytes T avec des constructions telles que les CAR ou les récepteurs des lymphocytes T peut varier considérablement. En l’absence d’une étape de purification ultérieure des lymphocytes T transduits, les résultats peuvent être difficiles à interpréter. Ici, une population pure de cellules CAR-T a été utilisée et, par conséquent, un haut degré de certitude peut être attendu lors de la caractérisation des cellules CAR-T comme étant tueuses ou non tueuses à chaque point temporel. Alternativement, le marquage des CAR avec une protéine fluorescente ou une étiquette génétique peut être appliqué pour indiquer les cellules qui sont CAR-positives, puis contrôlées lors de l’analyse des gouttelettes. Si le marquage est utilisé, les molécules fluorescentes sélectionnées ne doivent pas interférer avec les émissions fluorescentes de l’un des autres fluorophores de test. Par exemple, l’utilisation du marquage GFP interférerait avec le substrat GzmB marqué par FAM appliqué dans les expériences réalisées ici et n’est donc pas recommandée.
L’un des principaux avantages du protocole décrit ici est que la génération de gouttelettes elle-même est simplifiée et automatisée. Cependant, afin d’assurer l’encapsulation d’une seule cellule, il est important que les cellules soient complètement remises en suspension avant le chargement de la cartouche. Pour cette raison, il est recommandé de travailler à un rythme raisonnable lors de la préparation du chargement de la cartouche. Une considération similaire s’applique lors de l’ajout du substrat GzmB, car certaines cellules sont à haute sécrétion de GzmB. La remise en suspension des cellules empêchera également l’obstruction des canaux microfluidiques des petites cartouches. L’encapsulation correcte des cellules est facilement vérifiée par la microscopie, comme indiqué ci-dessus.
Parce que la gouttelette encapsule le milieu cellulaire et retient les produits cellulaires sécrétés par les cellules, d’autres cytokines, anticorps et composés peuvent être analysés. En effet, nous avons testé d’autres molécules pertinentes sécrétées par les cellules immunitaires, telles que l’IFN-γ et le TNF-α, qui nécessitent des modifications du protocole actuel. Pour la détection des cytokines, un type de format de test différent de celui que nous utilisons ici pour GzmB peut être appliqué, tel que la génération d’un sandwich ELISA à la surface de la cellule effectrice32. De plus, les colorations cellulaires et les couleurs du test peuvent être modifiées, par exemple, l’ester succinimidylique de carboxyfluorescéine (CFSE), mais il est important de s’assurer d’un saignement minimal ou nul entre les différentes combinaisons de couleurs, comme cela serait fait en cytométrie en flux standard.
De plus, ce protocole ne se limite pas à l’analyse des cellules CAR-T. Il peut également être étendu à l’étude d’autres interactions lymphocytes T/cible ou d’autres cellules immunitaires telles que les cellules CAR NK. Des expériences plus avancées peuvent également être envisagées, par exemple le marquage de sous-ensembles de lymphocytes T CD4 et CD8 avec des fluorophores supplémentaires pour effectuer un immunophénotypage fonctionnel plus large. En effet, nous montrons ici que CD4 et CD8 peuvent être détectés dans les gouttelettes, permettant ainsi une caractérisation plus poussée des lymphocytes T effecteurs et de leur capacité cytotoxique.
En optimisant ce protocole, nous avons spécifiquement visé à ce qu’il fonctionne sur des instruments de cytométrie en flux standard. Bien que la cytométrie en flux de gouttelettes ne soit pas compliquée, nous avons remarqué qu’une accumulation d’huile peut se produire si le cytomètre en flux n’est pas correctement rincé à certains intervalles ou comme recommandé ici après l’analyse des échantillons à chaque intervalle de temps. Le meilleur protocole de rinçage peut être spécifique à chaque cytomètre en flux.
L’un des avantages significatifs de cette méthode est sa capacité à haut débit, permettant de surveiller la cytotoxicité médiée par un effecteur unicellulaire contre les cellules cibles sans avoir besoin d’une expertise approfondie ou d’un équipement hautement spécialisé. Le test peut facilement être effectué à l’aide d’outils existants, tels que la cytométrie en flux ou la microscopie. Les gouttelettes à double émulsion se prêtent également au tri à l’aide de trieurs de cellules30,31, ce qui peut permettre l’isolement de cellules ayant des fonctionnalités spécifiques, par exemple, des cellules CAR-T avec et sans potentiel cytotoxique, suivi d’une analyse du transcriptome d’une seule cellule.
La technologie n’est pas sans limites. Bien que certaines lignées cellulaires tolèrent la culture en gouttelettes pendant 24 heures et au-delà, les cellules primaires peuvent avoir une viabilité nettement plus faible après 24 heures. Cela représente une contrainte de temps pour les tests en général, mais pour le test actuel, une activité notable de GzmB et de destruction des cellules peut être observée en 4 à 6 heures, probablement parce que le petit compartiment de gouttelettes assure une rencontre rapide de l’effecteur et des cellules cibles. De même, le petit volume de gouttelettes assurera une augmentation rapide de la concentration à des niveaux détectables du substrat clivé par GzmB sécrété. Une autre limitation de la technologie est l’incapacité de détecter les destructions en série par les cellules effectrices lors de l’utilisation de cytomètres en flux standard. Cependant, cela pourrait éventuellement être réalisé avec des cytomètres en flux d’images ou des technologies de cytométrie d’image, qui devront être étudiés.
Le transfert adoptif de cellules CAR-T CD19 a montré un succès remarquable dans le traitement des patients atteints d’hémopathies malignes. Malgré cela, il existe une grande variation de la réponse et une toxicité imprévisible chez les patients32, ce qui peut être dû en partie à l’hétérogénéité au sein du produit de perfusion de CAR-T. Par conséquent, il y a un intérêt croissant pour l’analyse de la composition phénotypique et de la capacité cytotoxique des cellules CAR-T individuelles au sein d’une population. Cela sera particulièrement important car la thérapie CAR est de plus en plus testée dans les maladies auto-immunes et dans d’autres formes de cancer. Le dispositif et le protocole microfluidiques décrits ici offrent une approche robuste et polyvalente pour examiner l’hétérogénéité des cellules CAR-T et d’autres thérapies cellulaires.
Déclarations de divulgation
Les auteurs déclarent les intérêts concurrents suivants : M.B.B. a reçu des honoraires de consultant de Janssen, Roche et Kite/Gilead, sans rapport avec le présent travail. D.L.P. et P.M. sont des employés de Samplix.
Remerciements
Les auteurs souhaitent remercier les membres et les employés de CITCO et de Samplix pour leurs discussions et suggestions utiles. MBB est soutenu par la bourse Early-Career Clinician Scientists' de la Fondation Lundbeck (R381-2021-1278). Ce travail est soutenu par une bourse de recherche d’élite de l’hôpital universitaire d’Odense. De plus, cette recherche a été soutenue par une subvention du Conseil européen de l’innovation Projet 190144395 à Samplix ApS.
matériels
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Blocker BSA (BSA) | Thermo Scientific | 37525 | |
| CellTrace Far Red Cell Proliferation Kit (far red fluorescent dye) | Invitrogen | C34564 | |
| CellTrace Violet Cell Proliferation Kit (violet fluorescent dye) | Invitrogen | C34557 | |
| DE Stabilizing Solution (stabilizing solution) | Samplix | REDIVSTABSOL1500 | |
| DPBS (dPBS) | Gibco | 14190-094 | |
| Dulbecco’s PBS (dPBS) | Capricorn Scientific | PBS-1A | |
| Dynabeads Human T-Activator (CD3/CD28 activation beads) | Gibco | 11132D | |
| Fetal Bovine Serum (FBS) | Capricorn Scientific | FBS-HI-12A | |
| Lenti-X Concentrator (PEG-based reagent) | Takara Bio | 631232 | |
| MACSelect LNGFR Microbeads (anti-NGFR magnetic beads) | Miltenyi Biotec | 130-091-330 | |
| OptiPrep density gradient medium (gradient medium) | Stemcell | 7820 | |
| Penicillin-Streptomycin (P/S) | Capricorn Scientific | PS-B | |
| Propidium iodide (PI) | Invitrogen | BMS500PI | |
| Recombinant Human IL-2 (IL-2) | Peprotech | 200-02 | |
| RPMI-1640 with Stable Glutamine (RPMI-1640) | Capricorn Scientific | RPMI-STA | |
| RPMI-1640 without L-Glutamine and phenol red | Gibco | 32404-014 | |
| Xdrop DE oil I (oil) | Samplix | REOILDEC1900 | |
| Xdrop Granzyme B substrate (GzmB substrate) | Samplix | REGRB100 | |
| Zombie-NIR viability dye (viability dye) | BioLegend | 423106 | |
| Plasticware etc. | |||
| 8-chamber glass slide | Chemometec | 942-0003 | |
| Cell culture plate, 12 well | TH Geyer | 7696791 | |
| DNA LoBind tube, 2 mL (DNA tube) | Eppendorf | 30108078 | |
| Eppendorf tube, 1.5 mL (1.5 mL tube) | Eppendorf | 30108051 | |
| Falcon tube, 15 mL (15 mL tube) | TPP | 91015 | |
| Falcon tube, 5 mL (5 mL tube) | Falcon (VWR) | 734-0443 | |
| Green cell suspension flasks for cell incubations (T75 flask) | Sarstedt | 148.19.22 | |
| Green cell suspension plates for cell incubations (96 well plate) | Sarstedt | 148.32.20 | |
| LS Separation Columns (separation column) | Miltenyi Biotec | 130-042-401 | |
| Xdrop DE Gaskets (gaskets) | Samplix | #GADEA100 | |
| Xdrop DE50 Cartridge (encapsulation cartridge) | Samplix | #CADE50A100 | |
| Antibodies | |||
| anti-CCR7 PE-Dazzle 594 | BioLegend | 353236 | |
| anti-CD19 CAR FMC63 Idiotype Antibody, PE | Miltenyi Biotec | 130-127-342 | |
| anti-CD3 APC | Biolegend | 300439 | |
| anti-CD3 BV480 | BD Biosciences | 566105 | |
| anti-CD3 FITC | BD Biosciences | 345763 | |
| anti-CD4 BUV661 | BD Biosciences | 612962 | |
| anti-CD4 StarBright Violet 760 | Bio-Rad | MCA1267SBV760T | |
| anti-CD45RA BUV395 | BD Biosciences | 740298 | |
| anti-CD8 PE-Cy7 | BioLegend | 344712 | |
| anti-CD8 StarBright Violet | Bio-Rad | MCA1226SBV760 | |
| anti-NGFR FITC | BioLegend | 345106 | |
| anti-NGFR PE | BioLegend | 345106 | |
| Cells | |||
| JeKo-1 Mantle-cell lymphoma cell-line (JeKo-1) | ATCC | CRL-3006 | |
| Primary peripheral blood mononuclear cells (PBMCs) | |||
| Equipment | |||
| Countess 3 Automated Cell Counter | Thermo Fisher Scientific | A50298 | |
| DynaMag-2 Magnet | Invitrogen | 12321D | |
| NovoCyte Quanteon Flow Cytometer (flow cytometer) | Agilent | 2010011AA | |
| Xdrop (microfluidics device) | Samplix | IN00110-EU |
Références
- Pasqui, D. M., Latorraca, C. D. O. C., Pacheco, R. L., Riera, R. CAR-T cell therapy for patients with hematological malignancies. A systematic review. Eur J Haematol. 109 (6), 601-618 (2022).
- Krejcik, J., et al. Harnessing the immune system to fight multiple myeloma. Cancers. 13 (18), 4546 (2021).
- Tokarew, N., Ogonek, J., Endres, S., von Bergwelt-Baildon, M., Kobold, S. Teaching an old dog new tricks: next-generation CAR T cells. British J Cancer. 120 (1), 26-37 (2019).
- Bogetofte Barnkob, M., Vitting-Seerup, K., Rønn Olsen, L. Target isoforms are an overlooked challenge and opportunity in chimeric antigen receptor cell therapy. Immunother Adv. 2 (1), ltac009 (2022).
- Daei Sorkhabi, A., et al. The current landscape of CAR T-cell therapy for solid tumors: Mechanisms, research progress, challenges, and counterstrategies. Front Immunol. 14, 1113882 (2023).
- . Considerations for the Development of Chimeric Antigen Receptor (CAR) T Cell Products Available from: https://www.fda.gov/regulatory-information/search-fda-guidance-documents/considerations-development-chimeric-antigen-receptor-car-t-cell-products (2024)
- Roddie, C., O'Reilly, M., Dias Alves Pinto, J., Vispute, K., Lowdell, M. Manufacturing chimeric antigen receptor T cells: issues and challenges. Cytotherapy. 21 (3), 327-340 (2019).
- Wang, L., et al. Improvement of in vitro potency assays by a resting step for clinical-grade chimeric antigen receptor engineered T cells. Cytotherapy. 21 (5), 566-578 (2019).
- Burrows, S. R., Fernan, A., Argaet, V., Suhrbier, A. Bystander apoptosis induced by CD8+ cytotoxic T cell (CTL) clones: implications for CTL lytic mechanisms. Int Immunol. 5 (9), 1049-1058 (1993).
- Gattinoni, L., et al. Acquisition of full effector function in vitro paradoxically impairs the in vivo antitumor efficacy of adoptively transferred CD8+ T cells. J Clin Invest. 115 (6), 1616-1626 (2005).
- Chen, G. M., et al. Integrative bulk and single-cell profiling of premanufacture T-cell populations reveals factors mediating long-term persistence of CAR T-cell therapy. Cancer Dis. 11 (9), 2186-2199 (2021).
- Frazer, G. L., Gawden-Bone, C. M., Dieckmann, N. M. G., Asano, Y., Griffiths, G. M. Signal strength controls the rate of polarization within CTLs during killing. J Cell Biol. 220 (10), e202104093 (2021).
- Hermans, I. F., et al. The VITAL assay: a versatile fluorometric technique for assessing CTL- and NKT-mediated cytotoxicity against multiple targets in vitro and in vivo. J Immunol Meth. 285 (1), 25-40 (2004).
- Zaritskaya, L., Shurin, M. R., Sayers, T. J., Malyguine, A. M. New flow cytometric assays for monitoring cell-mediated cytotoxicity. Expert Rev Vaccines. 9 (6), 601-616 (2010).
- Piccinini, C., et al. In vitro CAR-T cell killing: validation of the potency assay. Cancer Immunol Immunother CII. 73 (9), 168 (2024).
- Martinez, E. M., et al. High-throughput flow cytometric method for the simultaneous measurement of CAR-T cell characterization and cytotoxicity against solid tumor cell lines. SLAS Disc Adv Life Sci. 23 (7), 603-612 (2018).
- Jedema, I., van der Werff, N. M., Barge, R. M. Y., Willemze, R., Falkenburg, J. H. F. New CFSE-based assay to determine susceptibility to lysis by cytotoxic T cells of leukemic precursor cells within a heterogeneous target cell population. Blood. 103 (7), 2677-2682 (2004).
- Liu, L., et al. Cellular and molecular imaging of CAR-T cell-based immunotherapy. Adv Drug Delivery Rev. 203, 115135 (2023).
- Liadi, I., et al. Defining potency of CAR+ T cells: Fast and furious or slow and steady. Oncoimmunology. 8 (10), e1051298 (2019).
- Xhangolli, I., et al. Single-cell analysis of CAR-T cell activation reveals a mixed TH1/TH2 response independent of differentiation. Genom Proteom Bioinfo. 17 (2), 129-139 (2019).
- Zhou, Y., et al. Evaluation of single-cell cytokine secretion and cell-cell interactions with a hierarchical loading microwell chip. Cell Rep. 31 (4), 107574 (2020).
- LaBelle, C. A., Zhang, R. J., Hunsucker, S. A., Armistead, P. M., Allbritton, N. L. Microraft arrays for serial-killer CD19 chimeric antigen receptor T cells and single cell isolation. Cytometry J Int Soc Anal Cytol. 103 (3), 208-220 (2023).
- Hellmann, M. J., et al. Heterogeneously deacetylated chitosans possess an unexpected regular pattern favoring acetylation at every third position. Nat Comm. 15 (1), 6695 (2024).
- Wong, K. U., et al. Assessment of chimeric antigen receptor T cytotoxicity by droplet microfluidics in vitro. Antibody Therapeut. 5 (2), 85-99 (2022).
- Antona, S., Platzman, I., Spatz, J. P. Droplet-based cytotoxicity assay: Implementation of time-efficient screening of antitumor activity of natural killer cells. ACS omega. 5 (38), 24674-24683 (2020).
- Subedi, N., et al. An automated real-time microfluidic platform to probe single NK cell heterogeneity and cytotoxicity on-chip. Sci Rep. 11 (1), 17084 (2021).
- Zhang, Y., et al. Enhanced CRISPR/Cas12a-based quantitative detection of nucleic acids using double emulsion droplets. Biosens Bioelect. 257, 116339 (2024).
- Kochenderfer, J. N., et al. Construction and preclinical evaluation of an anti-CD19 chimeric antigen receptor. J Immunother. 32 (7), 689-702 (2009).
- Michaels, Y. S., et al. Precise tuning of gene expression levels in mammalian cells. Nat Comm. 10 (1), 818 (2019).
- Yuan, Y., et al. Droplet encapsulation improves accuracy of immune cell cytokine capture assays. Lab on a Chip. 20 (8), 1513-1520 (2020).
- Brower, K. K., et al. Double emulsion flow cytometry with high-throughput single droplet isolation and nucleic acid recovery. Lab on a Chip. 20 (12), 2062-2074 (2020).
- Kirouac, D. C., et al. Deconvolution of clinical variance in CAR-T cell pharmacology and response. Nat Biotechnol. 41 (11), 1606-1617 (2023).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon