Method Article
Ensaio de citotoxicidade baseado em gotículas para avaliar células T receptoras de antígeno quimérico no nível de célula única
Neste Artigo
Resumo
Aqui, descrevemos um método para criar gotículas de emulsão dupla encapsulando uma célula T e uma célula-alvo cancerígena para examinar a morte celular no nível de uma única célula. Este método permite a quantificação dupla de moléculas citotóxicas e apoptose de células-alvo em uma grande população de células T.
Resumo
A avaliação do potencial citotóxico de terapias baseadas em células T, como tratamentos com células T com receptor de antígeno quimérico (CAR), é fundamental para avaliar sua eficácia e é um pré-requisito para aplicação clínica. No entanto, os ensaios tradicionais de citotoxicidade são conduzidos como ensaios em massa e não fornecem informações detalhadas sobre a heterogeneidade funcional da população de células T CAR. Neste estudo, descrevemos um método baseado em gotículas de emulsão dupla que permite o co-encapsulamento em larga escala de células T CAR efetoras únicas com células-alvo únicas, permitindo a quantificação dupla de moléculas efetoras citotóxicas de células T e morte celular da célula-alvo. O protocolo descreve um método para a geração e purificação de células T CAR específicas para CD19, seguido por sua co-encapsulação em gotículas com a linha celular CD19+ JeKo-1, juntamente com reagentes para visualizar a secreção da molécula efetora citotóxica (Granzyme B) e morte celular (usando iodeto de propídio, PI). Demonstramos como gerar gotículas contendo CAR T único e células-alvo usando um dispositivo microfluídico disponível comercialmente para gerar gotículas de emulsão dupla. Além disso, fornecemos exemplos de como testar a diversidade funcional de células T CAR em gotículas usando equipamento de citometria de fluxo padrão. Finalmente, descrevemos brevemente a cinética temporal e a heterogeneidade da morte de células T CAR específicas para CD19. Embora esse método se concentre na morte celular após um ataque de células T CAR, ele também é adaptável para examinar outros tipos de células T, células imunes citotóxicas e funções de células efetoras, como a secreção de citocinas.
Introdução
A terapia com células T do receptor de antígeno quimérico (CAR) é um campo em rápida expansão da imunoterapia celular contra o câncer que se mostrou eficaz contra várias formas de leucemia, linfoma1 e mieloma múltiplo2. As células T CAR são geradas pela modificação de células T com um receptor de antígeno sintético que pode se ligar seletivamente a proteínas de superfície, como CD19 ou antígeno de maturação de células B (BCMA), expresso em células B, células plasmáticas e suas contrapartes malignas. Avanços recentes no design do CAR permitiram o direcionamento de vários antígenos simultaneamente3, contando com vários sinais de entrada ou ligando-se exclusivamente a antígenos associados a tumores, como isoformas de proteínas específicas4. Além disso, vários CARs estão sendo testados contra alvos de cânceres não hematológicos5.
Os ensaios de citotoxicidade são essenciais tanto para o desenvolvimento do CAR quanto para o controle de qualidade antes que os produtos clínicos sejam liberados 6,7,8. No entanto, a maioria dos ensaios atuais depende de populações em massa de células T CAR efetoras adicionadas em excesso às linhagens de células cancerígenas, o que pode levar a resultados falso-positivos devido a efeitos de espectador9 e resultar em uma correlação ruim entre os resultados in vitro e in vivo 10. Como a proliferação de células T e a persistência a longo prazo dependem dos sinais recebidos durante os eventos iniciaisde ativação 11,12, examinar de perto os eventos citotóxicos no nível de uma única célula é de grande interesse.
Para abordar as limitações das metodologias em massa, estudos de citotoxicidade no nível de célula única podem ser realizados usando citometria de fluxo 13,14,15,16,17 e ensaios baseados em imagem 18. No entanto, embora a citometria de fluxo padrão ofereça resolução de célula única para quantificar e caracterizar as células-alvo e as células-alvo, ela não pode determinar diretamente a capacidade citotóxica específica de células T CAR individuais em relação às células-alvo. Além disso, a imagem de células CAR T individuais interagindo com seus respectivos alvos em massa é desafiadora e demorada devido à motilidade consistente das células. Para enfrentar esses desafios, foram desenvolvidas novas ferramentas para análise de célula única que dependem do emparelhamento de células efetoras e alvo em espaços espacialmente confinados usando matrizes de micropoços 19,20,21, matrizes de microjangadas22, microchips23 e microfluídica de gotículas 24,25,26. Essas ferramentas fornecem sensibilidade de medição aprimorada, permitindo a investigação de menos células usando volumes reduzidos de reagente. No entanto, vários desafios permanecem, incluindo o emparelhamento eficiente de células, o número limitado de amostras que podem ser analisadas, a dependência apenas de análises baseadas em imagens e as dificuldades de recuperação de populações de células viáveis para análises posteriores.
Aqui, utilizamos um dispositivo microfluídico disponível comercialmente que permite uma abordagem massivamente simplificada para microfluídica baseada em gotículas. Este dispositivo permite que análises e ensaios avançados de célula única sejam realizados usando gotículas de emulsão dupla (DE) altamente estáveis. Em comparação com os ensaios microfluídicos de micropoços e gotículas tradicionais, o protocolo descrito aqui não requer ampla experiência em microfludicos.
As gotículas de DE são pequenos compartimentos esféricos compostos por uma casca de óleo com um núcleo aquoso suspenso em uma solução aquosa. A gotícula aquosa contém e retém células, secretoma celular, meio celular e reagentes de ensaio, permitindo a realização de ensaios complexos dentro de cada compartimento. Usando o dispositivo microfluídico, as gotículas de DE são geradas com um volume definido (aproximadamente 100 pL) adequado para ensaios de interação célula única ou célula-célula de mamíferos, que podem ser gerados em números muito altos (aproximadamente 750.000 gotículas por amostra) e em um curto período de tempo (aproximadamente 10 min para 8 amostras) por usuários de laboratório em geral sem conhecimento especializado em microfluídica. As gotículas geradas podem ser suspensas no meio celular, permitindo assim a difusão trans-shell de O2 e CO2 e tamponamento do interior, ao mesmo tempo em que retêm moléculas hidrofílicas e maiores, como moléculas efetoras secretadas por células. As células examinadas são, portanto, adequadamente tamponadas, permitindo o exame ao longo do tempo das interações célula-célula e da dinâmica temporal. Em contraste com as gotículas de emulsão única (por exemplo, gotículas de água em óleo)27, as gotículas de DE são estruturas robustas que não se fundem ou se fundem em incubadoras de células padrão. Devido à sua estabilidade e uma fase externa aquosa, eles também são compatíveis com procedimentos de análise a jusante, como a citometria de fluxo tradicional. Essas gotículas de picolitros geradas pelo dispositivo microfluídico podem, portanto, ser usadas para análise de célula única de alto rendimento da função celular para elucidar a heterogeneidade funcional oculta em ensaios tradicionais em massa.
Aqui, descrevemos um protocolo que utiliza gotículas DE para examinar o potencial citotóxico de CARs específicos para CD19 em relação às células do linfoma. Nosso protocolo permite a análise de célula única da morte de alvos e secreção de Granzyme B (GzmB) e revela que aproximadamente 20% das células T CAR examinadas aqui têm potencial de morte imediata.
Protocolo
As células T CAR usadas neste estudo foram geradas por transdução lentiviral de células T primárias com uma construção CAR CD19scFv-CD28-CD3ζ-tNGFR em um laboratório certificado de biossegurança OGM classe 2 e desclassificadas após 4 dias de acordo com o padrão do instituto. As células T purificadas eram de material excedente descartado de doações de sangue anônimas e estavam isentas de outras aprovações éticas de acordo com a lei dinamarquesa.
1. Geração de células CAR T
NOTA: O nome dos reagentes usados abaixo usa nomes genéricos e abreviados. O nome comercial completo pode ser encontrado entre parênteses na Tabela de Materiais.
- Ativação, transdução e expansão de células T
NOTA: A expressão estável de células T CAR CD19 foi alcançada através da transdução lentiviral de células T humanas primárias isoladas de células mononucleares frescas do sangue periférico (PBMCs) usando um kit de isolamento de células T de seleção negativa. O CAR anti-CD19 utilizado é uma construção de segunda geração composta por um fragmento variável de cadeia única FMC63 (scFv), um domínio de dobradiça e transmembrana CD28, um domínio coestimulatório CD28, um domínio de ativação CD3ζ28 e uma versão truncada do receptor do fator de crescimento nervoso (tNGFR) para monitoramento e enriquecimento de células que expressam CAR29. O plasmídeo vetor de transferência foi sintetizado de novo, e o lentivírus de terceira geração foi feito combinando-o com pMDLg / pRRE (plasmídeo Addgene # 12251), pRSV-Rev (plasmídeo Addgene # 12253) e pMD2.G (plasmídeo Addgene # 12259) 30. Os três plasmídeos posteriores foram um presente de Didier Trono. O vírus bruto foi concentrado usando um reagente baseado em PEG, e a multiplicidade de infecção (MOI) foi determinada pela transdução de células SUP-T1 e medição de tNGFR por citometria de fluxo de acordo com o protocolo padrão31.- Transfira 2 x 106 células T humanas primárias para uma placa de cultura de 12 poços e estimule-as com grânulos revestidos com CD3/CD28 em uma proporção de 1:1 grânulo para célula em 1 mL de meio RPMI-1640 completo (10% de soro fetal bovino inativado por calor, 1% de penicilina/estreptomicina) suplementado com 100 unidades/mL de interleucina-2 humana recombinante (IL-2). Incubar as células a 37°C em uma incubadora umidificada de CO5% 2 por 24 h.
- Adicione partículas lentivirais às células T ativadas em um MOI de 5-10. Misture delicadamente e incube as células por 72 h. Inclua células T não transduzidas (NTD) como controle negativo.
- No dia 3 pós-transdução, remova os grânulos de ativação CD3 / CD28 colhendo as células T em um tubo de 1,5 mL e colocando o tubo em um suporte magnético por 1-2 min. Transfira o sobrenadante contendo as células para um novo tubo de 1,5 ml.
- Realize uma contagem de células usando um contador de células automatizado e ajuste a densidade celular para 1 x 106 células/mL em um meio RPMI-1640 completo suplementado com 100 U/mL de IL-2. Continue expandindo as células T até que a contagem total de células T CAR atinja pelo menos 1,5 x 106 (geralmente cerca de 6 x 106 células T no total) na suspensão antes de prosseguir com o enriquecimento de NGFR (veja abaixo).
- Monitore a contagem de células em dias alternados e ajuste a concentração para 1 x 106 células/mL adicionando meio fresco suplementado com 100 unidades/mL de IL-2 para garantir o estado celular ideal durante a expansão.
- Enriquecimento de células T que expressam CAR
- Transfira 6 x 10,6 ou mais células T transduzidas para um tubo de 15 mL e centrifugue a 300 x g por 5 min. Ressuspenda o pellet celular em 320 μL de PBS suplementado com 0,5% de albumina de soro bovino (BSA) para fazer PBSA e adicione 40 μL de esferas magnéticas anti-NGFR. Misture bem e incube no gelo por 15 min.
NOTA: Este protocolo assume uma eficiência de transdução de aproximadamente 25%, resultando em >1,5 x 106 células T CAR após o enriquecimento. Não recomendamos começar com menos de 6 x 106 células, pois isso pode resultar em baixa recuperação de células T CAR. - Adicione 1640 μL de PBSA para elevar o volume final a 2 mL e, em seguida, prossiga com a separação magnética usando colunas de separação de acordo com as instruções do fabricante.
- Transfira 6 x 10,6 ou mais células T transduzidas para um tubo de 15 mL e centrifugue a 300 x g por 5 min. Ressuspenda o pellet celular em 320 μL de PBS suplementado com 0,5% de albumina de soro bovino (BSA) para fazer PBSA e adicione 40 μL de esferas magnéticas anti-NGFR. Misture bem e incube no gelo por 15 min.
- Detecção da expressão de CAR por citometria de fluxo
NOTA: Recomendamos determinar a porcentagem de células que expressam CAR e sua viabilidade neste momento, para garantir células adequadas antes de realizar o ensaio abaixo. A proporção de CD4 e CD8 e o fenótipo de memória também podem ser determinados neste ponto. Veja a Figura 1 para gráficos representativos.- Transfira 2,5 x 105 células T de culturas transduzidas e não transduzidas para tubos de citometria de fluxo separados. Lave as células 2x com 200 μL de PBS, centrifugando a 300 x g por 5 min após cada lavagem.
- Preparar uma mistura de anticorpos contendo anticorpos anti-CD19 CAR FMC63 Idiotype PE, anti-CD3 BV480 e anti-NGFR FITC, cada um diluído a 1:100 em PBS. Ressuspenda as células em 50 μL da mistura de anticorpos e incube a 4 °C por 20 min.
- Lave as células 2x com 200 μL de PBS, centrifugando a 300 x g por 5 min após cada lavagem. Ressuspenda as células em 200 μL de PBS e analise usando citometria de fluxo.
2. Geração de gotículas encapsulando células T e células-alvo
NOTA: Antes do encapsulamento, as células T e as células JeKo-1 são coradas com diferentes colorações celulares para monitorar o conteúdo celular das gotículas. As células são encapsuladas com reagentes de ensaio e incubadas por 2-6 h antes que as gotículas sejam analisadas no citômetro de fluxo (ver Figura 2).
- Coloração de células antes do encapsulamento
- Prepare soluções de trabalho de dois corantes fluorescentes (utilizamos um corante violeta e um corante vermelho distante) diluindo as soluções estoque (preparadas de acordo com as instruções do fabricante) 1:5.000 em dPBS.
- Para cada doador, transfira 2,5 x 106 CAR T, 2,5 x 106 NTD T e 15 x 106 células JeKo-1 para tubos de centrífuga separados (tubos de 15 mL ou 50 mL) e centrifugue a 300 x g por 5 min. Neste experimento, células efetoras 0,5 x 106 foram encapsuladas com 1,5 x 106 células-alvo para cada amostra preparada.
NOTA: O número de células transferidas aqui destina-se a 4x amostras de encapsulamento com cada tipo de célula efetora para cada doador. Reduza o número de células e reagentes de acordo com o número de amostras necessárias. Além disso, o número exato de células por amostra pode ser dimensionado de acordo com as necessidades experimentais. - Remova o sobrenadante e ressuspenda os pellets de células efetoras na solução de trabalho 1:5.000 do corante fluorescente violeta e dos pellets de célula-alvo (JeKo-1) na solução de trabalho 1:5.000 do corante fluorescente vermelho distante. Ressuspenda bem as células com uma pipeta e com uma concentração celular de 1 x 106 células/mL de solução de coloração.
- Incubar as células durante 20 min numa incubadora de CO2 humidificada a 37 °C. Centrifugue as células a 300 x g por 5 min, remova o sobrenadante e ressuspenda em 15 mL de meio RPMI-1640 completo para lavar as células.
- Repita a lavagem e centrifugue a 300 x g por 5 min. Remova o sobrenadante e ressuspenda os pellets de células efetoras em 225 μL de RPMI-1640 completo e os pellets de células-alvo em 450 μL de RPMI-1640 completo.
- Adicione 30 μL de um meio de gradiente às suspensões de células efetoras e 60 μL do mesmo meio de gradiente às suspensões de células-alvo. Ressuspenda a solução de estoque do meio gradiente bem antes da pipetagem.
- Adicione 15 μL de um estoque de PI de 20 μg / mL às suspensões de células efetoras e 30 μL do estoque de PI às suspensões de células-alvo. A concentração final de PI será eventualmente de 1 μg/mL após o substrato Granzyme B (GzmB) ter sido adicionado abaixo.
- Preparar uma diluição de 1:10 do substrato GzmB em RPMI-1640 completo e adicionar 30 μL desta diluição do substrato GzmB à suspensão de células efetoras e 60 μL às suspensões de células-alvo. Misture bem com a pipeta.
NOTA: O volume total dos reagentes adicionados a cada pellet de célula efetora deve agora ser de 300 μL e aos pellets de célula alvo de 600 μL. A concentração do gradiente médio nessas soluções deve ser de 10%, a concentração de PI de 1 μg/mL e a concentração de substrato de GzmB de 1:100. - Marcação alternativa usando anticorpos CD3, CD4 e CD8
NOTA: Em alguns casos, pode ser interessante rotular ainda mais as células efetoras antes do co-encapsulamento com as células-alvo. Aqui mostramos a prova de princípio para este tipo de rotulagem usando anticorpos CD3, CD4 e CD8 e PBMC (ver Figura 3).- Transfira 2,0 x 106 PBMC para um tubo de 1,5 mL e gire as células para baixo por 5 min a 300 x g. Remova o sobrenadante e ressuspenda o pellet celular em 200 μL de dPBS com 0,5% de BSA. Divida o conteúdo em dois tubos de 2 mL.
- Adicione 5 μL de anti-CD3-APC e 5 μL de anti-CD4-StarBright Violet 760 a um dos tubos. Adicione 20 μL de anti-CD3-FITC e 5 μL de anti-CD8-StarBright Violet 760 ao outro tubo.
- Misture bem e incube por 30 min em temperatura ambiente no escuro. Adicione 1 mL de tampão de lavagem às células, gire a 300 x g por 5 min, remova o sobrenadante e ressuspenda os grânulos celulares em 1 mL de dPBS com 0,5% de BSA.
- Gire as células a 300 x g por 5 min, remova o sobrenadante e ressuspenda os grânulos celulares em 150 μL de RPMI sem vermelho de fenol e meio de gradiente de 10%. Encapsular conforme descrito na etapa 2.2 usando RPMI sem vermelho de fenol, solução estabilizadora a 33% para células e meio gradiente a 10% como meio externo.
- Encapsulando as células
- Pré-aqueça um cartucho de encapsulamento e uma solução estabilizadora à temperatura ambiente antes do encapsulamento. Prepare um estoque de meio externo consistindo de meio RPMI-1640 completo, solução estabilizadora de 33% para células e meio de gradiente de 10%.
- Para cada amostra de encapsulamento, prepare uma solução de amostra de célula com células efetoras alvo mistas. Ressuspenda as soluções celulares bem preparadas com uma pipeta e misture 65 μL de suspensão de células efetoras preparadas com 65 μL de suspensão de células-alvo preparadas em tubos de 1,5 mL (ambos preparados na etapa 2.1).
- Prossiga imediatamente com o carregamento dos poços indicados do cartucho de encapsulamento com reagentes na seguinte ordem para garantir o encapsulamento adequado. Carregue um conjunto de poços para cada amostra de encapsulamento preparada.
Bem #A: 400 μL de mídia externa.
Bem #D: 40 μL de mídia externa na prateleira pequena.
Bem #C: 120 μL de solução pré-misturada com células-alvo (JeKo-1) e células efetoras. Ressuspenda bem as células com uma pipeta antes de carregar.
Poço #B: 250 μL de óleo.
NOTA: Cada cartucho pode ser carregado com até 8 amostras para encapsulamento em paralelo. - Prossiga imediatamente com a vedação da gaxeta e o carregamento do cartucho no instrumento, evitando inclinar, sacudir ou bater no cartucho carregado. Sele cuidadosamente a junta no cartucho.
- Transfira cuidadosamente o cartucho para o dispositivo microfluídico e inicie o encapsulamento conforme descrito no manual do usuário.
- As gotículas geradas têm densidades mais altas do que o meio externo circundante e sedimentam rapidamente no fundo do poço de coleta (Poço #D). Colete cada produção de gotículas (todas as gotículas e seu meio externo circundante) ressuspendendo as gotículas sedimentadas do Well #D no meio sobreposto e transferindo-as para um tubo de DNA de baixa ligação de 2 mL com tampa. Lave o Well #D com o meio externo restante do Well #A para coletar as gotículas restantes.
- Quando as gotículas tiverem sedimentado nos tubos de coleta (leva aproximadamente 1 min), verifique a produção de gotículas examinando-as em um microscópio de campo claro. Para fazer isso, encha uma ponteira de pipeta de 10 μL com uma amostra: cerca de 1/3 com gotículas da superfície da fase de gotículas (fase branca) e cerca de 2/3 com o meio externo sobreposto para preencher a ponteira. Carregue imediatamente a amostra em uma lâmina de vidro de 8 câmaras e examine as gotículas por microscopia de campo claro com ampliação de 4x e 20x para confirmar o carregamento de gotículas com células.
NOTA: É importante que as gotículas desta forma sejam aspiradas em um volume maior do meio circundante.
- Incubação
NOTA: As gotículas agora podem ser incubadas em 2 mL de tubos de baixa ligação de DNA em uma incubadora padrão umidificada a 37 ° C, 5% de CO2. Recomendamos o uso desses tubos, pois eles têm propriedades de superfície ideais para o cultivo de gotículas.- Com uma agulha de seringa (23G), perfure com cuidado e segurança a tampa do número necessário de tubos de DNA de baixa ligação de 2 mL. Isso garantirá a difusão livre de CO2/O2 , evitando a evaporação do meio.
- Adicione 1 mL de meio externo a cada tubo de incubação. As gotículas devem ser incubadas com pelo menos 5x o volume de meio externo para garantir o tamponamento adequado. O volume do meio externo adicionado pode ser aumentado dependendo do metabolismo das células aplicadas.
- Ressuspenda as gotículas geradas no meio sobreposto e divida cada produção em três dos tubos de incubação preparados (um para cada medição de ponto de tempo). As gotículas são pesadas e sedimentam rapidamente e, portanto, é importante ressuspender o estoque entre cada transferência.
NOTA: O número de tubos de incubação nos quais as produções de gotículas são divididas pode ser variado, mas recomendamos dividir uma produção em no máximo 4 tubos de incubação para garantir que cada amostra contenha eventos suficientes para análise. - Coloque os tubos na posição vertical na incubadora por 2 h, 4 h ou 6 h de incubação após a geração de gotículas.
3. Análise a jusante de gotículas
- Microscopia
- Após a incubação, transfira um pequeno número de gotículas para uma lâmina de microscópio conforme descrito na etapa 2.2.7 e analise-as por microscopia de campo claro e fluorescência usando um microscópio de fluorescência padrão com configuração apropriada de laser e filtro.
NOTA: As gotículas podem ser detectadas por microscopia de campo claro. As células T coradas com violeta podem ser detectadas com um filtro DAPI, as células JeKo-1 coradas de vermelho distante com um filtro APC, o sinal GzmB-FAM com um filtro FITC e o sinal PI com um filtro PE. O sinal PI de maior interesse pode ser difícil de visualizar por microscopia, uma vez que as células apoptóticas precoces têm um pico de emissão menor do que as células apoptóticas tardias. Consulte a Figura 4 para obter dados representativos.
- Após a incubação, transfira um pequeno número de gotículas para uma lâmina de microscópio conforme descrito na etapa 2.2.7 e analise-as por microscopia de campo claro e fluorescência usando um microscópio de fluorescência padrão com configuração apropriada de laser e filtro.
- Citometria de fluxo
- Após a incubação, ressuspenda cada amostra de gotículas no meio sobreposto e transfira-a para tubos FACS de 5 mL. Analise as gotículas usando um citômetro de fluxo padrão seguindo as diretrizes gerais listadas abaixo.
- Use um citômetro de fluxo com configuração apropriada de laser e filtro. Com o painel selecionado de colorações celulares e ensaio, a compensação de cor não é necessária no citômetro de fluxo usado aqui. Se outras cores forem aplicadas ou a configuração do citômetro de fluxo aplicado for diferente, pode ser necessária uma compensação.
- Use o FSC-H como o gatilho de limite para excluir o ruído de fundo de pequenos eventos de óleo.
- As gotículas são pesadas e sedimentam no fundo dos tubos FACS. Certifique-se de que o SIP atinja as gotículas. As gotículas devem ser suspensas em pelo menos 5x o volume de tampão externo para gotículas antes da aquisição.
- Os eventos de gotículas são adquiridos usando alta velocidade. Registre os sinais de altura de intensidade (H) para FSC e SSC e os fluoróforos examinados, pois as medições de altura de intensidade são usadas ao analisar gotículas. Ajuste os ganhos para ter uma separação adequada de eventos positivos e negativos.
NOTA: Usamos o canal FITC para medir o sinal GzmB, o canal azul do Pacífico para detectar as células T coradas com violeta, o canal APC para detectar as células JeKo-1 coradas de vermelho distante e o canal PE para detectar o sinal PI. - Registre eventos suficientes para análise downstream. Aqui, registramos 4,5 - 8,3 x 103 das gotículas co-encapsulantes por ponto de tempo.
- Lave o citômetro de fluxo através do SIP após cada série de aquisições de gotículas, usando soluções padrão de limpeza e enxágue. Limpar bem o citómetro de fluxo no final da experiência. Consulte a Figura 4 e a Figura 5 para obter dados representativos.
- Após a incubação, ressuspenda cada amostra de gotículas no meio sobreposto e transfira-a para tubos FACS de 5 mL. Analise as gotículas usando um citômetro de fluxo padrão seguindo as diretrizes gerais listadas abaixo.
- Análise dos dados
- Calcule a porcentagem de células T secretoras de GzmB com base na porcentagem de coencapsulamentos de células T positivas para GzmB e células JeKo-1 normalizadas para a viabilidade de células T determinada nas populações de gotículas somente de células T no momento em questão, da seguinte forma:
Gotículas T cell+, Jeko-1+, GzmB+ / (Gotículas T cell+, Jeko-1+ - Gotículas mortas T cell+, Jeko-1+)
onde Gotículas de células T mortas+, Jeko-1+ é estimada a partir da morte em gotículas apenas com células T: Gotículasde células T mortas+, Jeko-1+ = Células T de gotículas+, Jeko-1+ x Células T mortas+, Jeko-1- / GotículasTcell+, Jeko-1-. - Calcule a porcentagem de células T que mataram suas células-alvo da seguinte forma:
Porcentagem de células T citotóxicas = (Morte observada - Morte de fundo)/(100 - Morte de fundo)
onde a morte de fundo é a morte presente dentro de (célula T +, Jeko-1 +) gotículas resultantes de morte não relacionada à morte ou morte ocorrida antes do encapsulamento. - Estime a morte de fundo por morte celular em gotículas (células T+, Jeko-1-) e gotículas (células T-, Jeko-1+):
Morte de fundo = (1- (1- taxa de mortalidade em GotículasTcell-, Jeko-1+) x (1- taxa de mortalidade em GotículasT cell+, Jeko-1-)) x 100
onde a taxa de mortalidade é medida diretamente como a gotícula % PI positiva em gotículas somente de células T e gotículas somente de células JeKo-1, respectivamente. Todos os cálculos são realizados com amostragem de números específicos do tempo.
- Calcule a porcentagem de células T secretoras de GzmB com base na porcentagem de coencapsulamentos de células T positivas para GzmB e células JeKo-1 normalizadas para a viabilidade de células T determinada nas populações de gotículas somente de células T no momento em questão, da seguinte forma:
Resultados
Após a transdução, as células T foram analisadas quanto à expressão de CAR por citometria de fluxo usando anticorpos anti-CD3 e anti-NGFR. A população de células T CAR foi posteriormente enriquecida usando esferas magnéticas anti-NGFR, resultando em uma pureza de mais de 98% para ambos os doadores (Figura 1A-B). A relação CD4/CD8 e o fenótipo de memória das células CAR classificadas usadas também foram quantificados, usando uma estratégia de porta padrão utilizando anticorpos CCR7 e CD45RA. Esses dados mostram que a proporção CD4 / CD8 das células T CAR usadas foi de 0,73, e as células eram principalmente de fenótipo de memória ingênuo (Figura 1C-E).
As células T CAR e as células JeKo-1 foram coradas com corantes violeta e vermelho-distante, respectivamente, e encapsuladas em gotículas DE50 juntamente com os reagentes de ensaio substrato GzmB e PI (Figura 2). Alternativamente, as células T CAR também podem ser marcadas usando anticorpos como anti-CD4 ou anti-CD8 (Figura 3), permitindo uma caracterização mais detalhada das células T. Cada suspensão de células T (CAR T ou NTD) foi misturada com uma suspensão de células JeKo-1 imediatamente antes do encapsulamento em uma proporção efetor: célula-alvo de 1:3 (0,5 x 106 células T e 1,5 x 106 JeKo-1). Após o encapsulamento, cada produção de gotículas (células T CAR + JeKo-1 e NTD + JeKo-1) foi dividida em três tubos de incubação por 2 h, 4 h e 6 h de incubação, respectivamente. As células foram incubadas dentro das gotículas a 37 °C em CO2 a 5% e, em seguida, analisadas por microscopia e citometria de fluxo nos momentos indicados.
Realizamos microscopia de fluorescência de gotículas de dupla emulsão após 4 h de incubação (Figura 4). A intensidade do sinal de fluorescência verde (FITC) ilustra o nível de atividade secretada de GzmB de células T CAR ou NTDs dentro das gotículas DE50. A Figura 4B apresenta imagens de contraste de fase e microscopia de fluorescência de uma única gotícula positiva para GzmB contendo uma célula CAR T e uma célula-alvo JeKo-1 em contato próximo.
Em seguida, as gotículas de DE50 foram analisadas por citometria de fluxo para quantificar a porcentagem de células T CAR com secreção precoce de GzmB e atividade de morte de células citotóxicas (Figura 5). A coloração pré-incubação de células-alvo JeKo-1 com um corante vermelho distante e células T efetoras com um corante violeta antes do encapsulamento facilitou a identificação de três populações distintas de gotículas contendo células, pois as células são distribuídas em gotículas com base na distribuição de Poisson. As populações de gotículas identificadas são gotículas apenas com células T, células JeKo-1 sozinhas e células T e células JeKo-1 juntas (Figura 5A). Células T co-encapsuladas por gotículas com células JeKo-1 foram fechadas e analisadas quanto a sinais que indicam atividade de GzmB (Figura 5B) e morte celular, conforme indicado por PI (Figura 5C).
O encapsulamento de células em gotículas segue a distribuição de Poisson, e quatro populações de gotículas diferentes são obtidas (Figura 6A). A Figura 6B mostra o nível de gotículas positivas para GzmB em todas as populações de gotículas após 6 h de incubação de gotículas, a partir das quais a porcentagem de células T secretoras espontâneas de GzmB pode ser determinada. Esses dados indicam a alta especificidade do método, pois apenas as células T CAR secretam GzmB.
Finalmente, quantificamos os níveis de GzmB e PI em pontos de tempo, após 2 h, 4 h e 6 h de co-incubação. Para referência, as gotículas contendo apenas células T e apenas células JeKo-1 foram analisadas para examinar a morte celular de fundo em cada população (Figura 6C). A morte celular de fundo foi usada para determinar a porcentagem de células T vivas secretoras de GzmB e matadoras de células-alvo na população de células efetoras (Figura 7), conforme descrito na etapa 3.3. As células CAR T de dois doadores exibiram um aumento dependente do tempo na secreção de GzmB induzida pela célula-alvo e na atividade de morte celular. Quase 36% das células T CAR vivas do Doador 1 e 31% do Doador 2 secretaram GzmB após 6 h de co-encapsulamento com a célula-alvo, um aumento significativo em comparação com as células T de controle NTD (Figura 7A). Da mesma forma, cerca de 21% das células T CAR vivas do Doador 1 e 22% das células CAR T vivas do Doador 2 mataram células-alvo, conforme indicado por um sinal PI positivo (Figura 7B). A porcentagem de células T CAR que secretaram GzmB excedeu a porcentagem de células-alvo mortas em cada ponto de tempo, consistente com a sequência esperada de eventos na citotoxicidade mediada por GzmB. Em conjunto, esses dados mostram que o método aqui apresentado permite a caracterização da heterogeneidade na citotoxicidade de células T individuais dentro de uma população de células, bem como comparações entre diferentes populações.
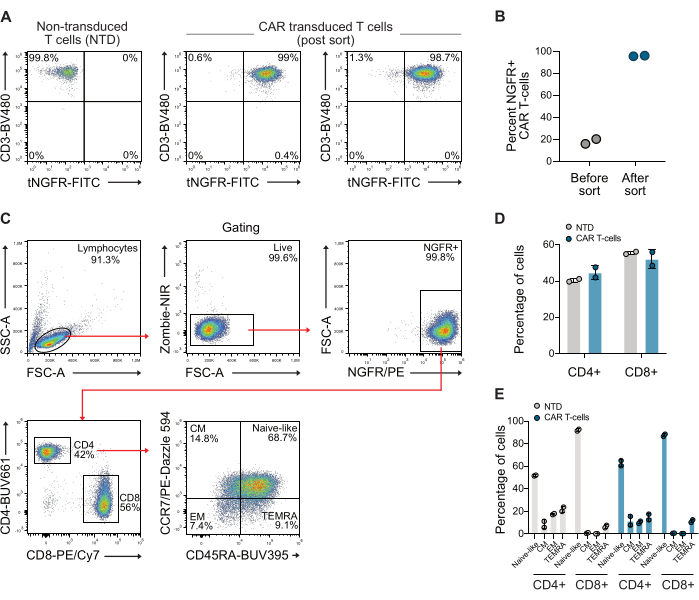
Figura 1: Gráficos de citometria de fluxo representativos após a classificação NGFR. (A) Após aproximadamente 10 dias de expansão, as células T CAR foram classificadas usando microesferas específicas para NGFR e classificação magnética, resultando em uma população de células T CAR que era >98% de células T CAR. (B) Quantificação de células T CAR de dois doadores usados neste estudo antes e depois da triagem. (C) Estratégia de bloqueio usada para examinar a relação CD4 / CD8 e o fenótipo de memória das células T. (D) Quantificação de CD4 e CD8 de células T utilizadas. (E) Quantificação do fenótipo de memória das células T utilizadas. Abreviatura: CM = memória central, EM = memória efetora, NTD = não transduzida, TEMRA = células efetoras terminalmente diferenciadas. Clique aqui para ver uma versão maior desta figura.
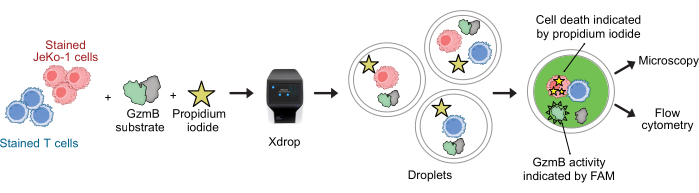
Figura 2: Fluxo de trabalho para o ensaio combinado de secreção e citotoxicidade de GzmB com resolução de célula única em gotículas. Antes do encapsulamento em gotículas DE, as células-alvo e efetoras são coradas separadamente usando coragens de células violetas e vermelhas distantes. Usando o dispositivo microfluídico e o cartucho de encapsulamento, as células efetoras são co-encapsuladas com células-alvo em gotículas junto com meio celular, PI e substrato peptídico GzmB marcado com FAM. O ensaio e a incubação ocorrem dentro das gotículas. A atividade secretada de GzmB é indicada pela emissão de fluorescência verde que ocorre após a clivagem do substrato de GzmB. A morte celular é indicada por PI. Após a incubação, as gotículas de DE50 são analisadas por microscopia e/ou citometria de fluxo. Clique aqui para ver uma versão maior desta figura.
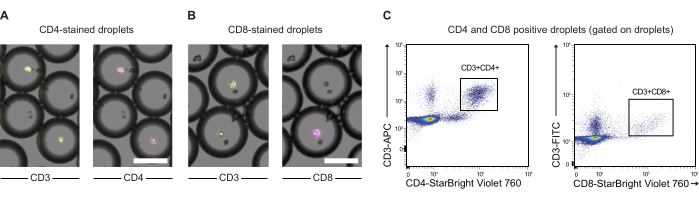
Figura 3: Análise de PBMCs encapsuladas em gotículas pré-marcadas com anticorpos anti-CD3, anti-CD4 e anti-CD8. (A) Imagens de microscopia de gotículas com PBMCs encapsuladas pré-marcadas com anticorpos anti-CD3 (APC) e anti-CD4 (NIR). Barra de escala = 100 μm. (B) Como (A), mas com marcação CD3 (FITC) e anti-CD8 (NIR). Barra de escala = 100 μm. (C) Análise por citometria de fluxo das mesmas gotículas. Clique aqui para ver uma versão maior desta figura.
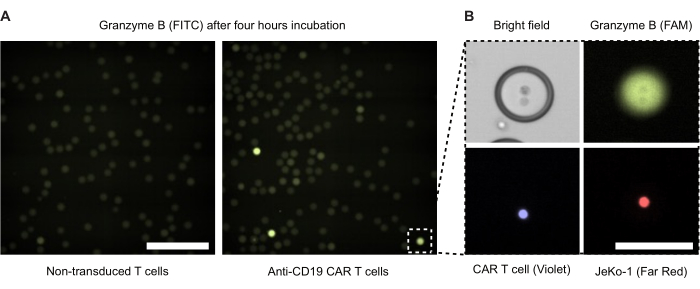
Figura 4: Imagens microscópicas de gotículas com células efetoras e alvo. As imagens foram tiradas após 4 h de incubação em uma incubadora de células umidificadas padrão de 37 °C, 5% de CO2 . (A) Gotículas de uma amostra com células NTD e JeKo-1 encapsuladas (esquerda) ou células CAR T e células JeKo-1 (direita) fotografadas por microscopia de fluorescência usando o canal FITC para detectar gotículas GzmB positivas (verde). Barra de escala = 500 μm. (B) Uma única gotícula fotografada por contraste de fase, FITC (GzmB), APC (célula JeKo-1) e DAPI (célula T CAR). Barra de escala = 100 μm. Clique aqui para ver uma versão ampliada desta figura.
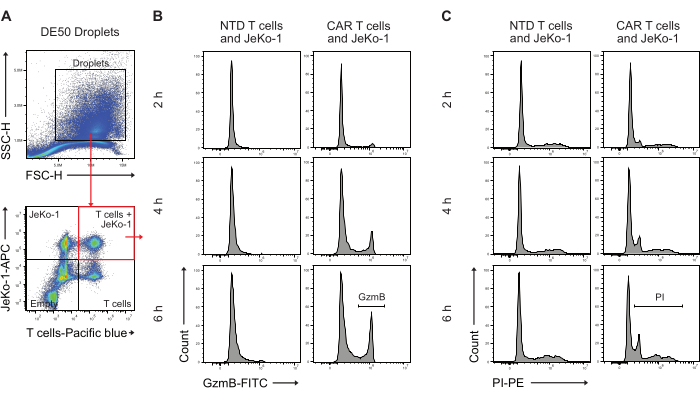
Figura 5: Estratégia de gating para análise de gotículas por citometria de fluxo após a incubação. (A) O gating foi feito por dispersão direta e lateral para identificar gotículas, seguido pela seleção da porta contendo as gotículas. Os eventos fora do portão representam gotículas de óleo produzidas como subproduto da produção de gotículas de emulsão dupla. A análise de fluorescência subsequente de gotículas em canais correspondentes às colorações celulares aplicadas identifica quatro populações de gotículas: gotículas contendo células T e células JeKo-1 (quadrado vermelho); gotículas apenas com células JeKo-1; gotículas apenas com células T; e gotículas vazias. (B) Histogramas representativos para o sinal GzmB em gotículas duplamente positivas em pontos de tempo e entre células NTD e CAR T. (C) Histogramas representativos para o sinal PI em gotículas duplamente positivas em pontos de tempo e entre células T NTD e CAR. Todas as medições de gotículas são realizadas como medições de altura de intensidade (H). Clique aqui para ver uma versão maior desta figura.
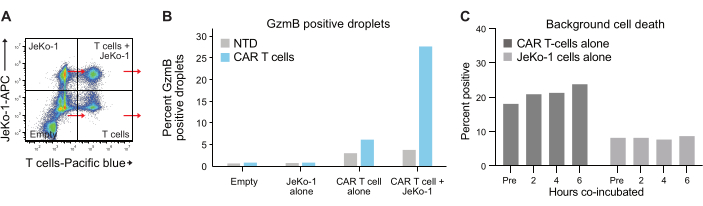
Figura 6: Controle interno e populações de referência. (A) Cada um dos quatro quadrantes da Figura 5A foi selecionado, e GzmB e PI foram medidos em cada um. Esse tipo de quantificação permite que os sinais de fundo sejam medidos e subtraídos antes que uma análise final da eficácia das células T seja realizada. (B) Gráfico mostrando a frequência de gotículas positivas para GzmB após 6 h de incubação para cada uma das quatro populações de gotículas, exemplificada com dados da amostra CAR T do Doador 1. (C) A morte celular de fundo foi determinada em populações de gotículas de controle somente JeKo-1 e somente células T em cada ponto de tempo medido, com dados do doador 1. A morte celular de fundo é usada para calcular a frequência de células efetoras secretoras e matadoras de células GzmB vivas mostradas na Figura 7 e explicadas na etapa 3.3. Abreviaturas: Pré = encapsulamento. Clique aqui para ver uma versão maior desta figura.
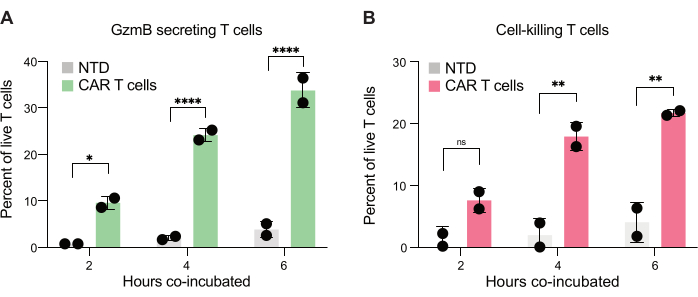
Figura 7: Quantificação de células T capazes de secretar GzmB e matar células JeKo-1 em gotículas duplamente positivas. Gotículas duplamente positivas de dois doadores foram examinadas após 2 h, 4 h e 6 h de co-incubação em gotículas. (A) Frequência de células T que encontram células-alvo secretando GzmB e comparação entre células T NTD e células T CAR. (B) Frequência de células T que encontram células-alvo matando a célula-alvo co-encapsulada. Abreviaturas: GzmB = granzima B, NTD = não transduzido, PI = iodeto de propídio. * = Valor de P < 0,05, ** = Valor de P < 0,005, **** = Valor de P < 0,0001 por ANOVA de duas vias. Clique aqui para ver uma versão maior desta figura.
Discussão
Aqui, apresentamos um método para examinar o potencial citotóxico das células T no nível de célula única usando um dispositivo de microfluidos disponível comercialmente fácil de usar para avaliar o potencial citotóxico das células T CAR CD19-CD28-CD3z após o co-encapsulamento com a linha celular de linfoma de células do manto CD19-positiva JeKo-1.
Existem várias etapas críticas neste protocolo. Primeiro, recomendamos remover os grânulos de estimulação das células T pelo menos 48 h antes de realizar qualquer ensaio, para evitar resultados falsos positivos. Em segundo lugar, a eficiência da transdução de células T com construções como CARs ou receptores de células T pode variar consideravelmente. Sem uma etapa de purificação subsequente das células T transduzidas, os resultados podem ser difíceis de interpretar. Aqui, uma população pura de células T CAR foi usada e, portanto, um alto grau de certeza pode ser esperado ao caracterizar as células T CAR como assassinas ou não assassinas em cada ponto de tempo. Alternativamente, a marcação de CARs com proteína fluorescente ou marca genética pode ser aplicada para indicar células que são positivas para CAR e, em seguida, bloqueadas ao analisar gotículas. Se a marcação for usada, as moléculas fluorescentes selecionadas não devem interferir nas emissões fluorescentes de nenhum dos outros fluoróforos do ensaio. Por exemplo, o uso da marcação GFP interferiria no substrato GzmB marcado com FAM aplicado nos experimentos realizados aqui e, portanto, não é recomendado.
Uma grande vantagem do protocolo descrito aqui é que a própria geração de gotículas é simplificada e automatizada. No entanto, para garantir o encapsulamento de célula única, é importante que as células ressuspendam completamente antes do carregamento do cartucho. Por esse motivo, recomenda-se trabalhar em um ritmo razoável ao se preparar para carregar o cartucho. Uma consideração semelhante se aplica ao adicionar o substrato GzmB, pois algumas células são altamente secretas de GzmB. A ressuspensão das células também evitará o entupimento de pequenos canais microfluídicos de cartucho. O encapsulamento adequado das células é facilmente verificado por microscopia, conforme descrito acima.
Como a gotícula encapsula o meio celular e retém produtos celulares secretados pelas células, outras citocinas, anticorpos e compostos podem ser analisados. De fato, testamos outras moléculas secretadas por células imunes relevantes, como IFN-γ e TNF-α, que requerem modificações no protocolo atual. Para a detecção de citocinas, um tipo diferente de formato de ensaio do que utilizamos aqui para GzmB pode ser aplicado, como a geração de um sanduíche ELISA na superfície da célula efetora32. Além disso, as colorações celulares e as cores do ensaio podem ser alteradas, por exemplo, éster succinimidílico de carboxifluoresceína (CFSE), mas é importante garantir o mínimo ou nenhum sangramento em diferentes combinações de cores, como seria feito na citometria de fluxo padrão.
Além disso, este protocolo não se limita à análise de células T CAR. Também pode ser estendido para estudar outras interações célula T / alvo ou outras células imunes, como células CAR NK. Experimentos mais avançados também podem ser previstos, por exemplo, marcando subconjuntos de células T CD4 e CD8 com fluoróforos adicionais para realizar imunofenotipagem funcional mais ampla. De fato, aqui mostramos que CD4 e CD8 podem ser detectados nas gotículas, permitindo assim uma maior caracterização das células T efetoras e sua capacidade citotóxica.
Otimizando este protocolo, visamos especificamente que ele funcionasse em instrumentos de citometria de fluxo padrão. Embora a citometria de fluxo de gotículas não seja complicada, notamos que o acúmulo de óleo pode ocorrer se o citômetro de fluxo não for enxaguado adequadamente em determinados intervalos ou conforme recomendado aqui após a análise das amostras em cada intervalo de tempo. O melhor protocolo de enxágue pode ser específico para cada citômetro de fluxo individual.
Uma das vantagens significativas deste método é sua capacidade de alto rendimento, permitindo o monitoramento da citotoxicidade mediada por efetor de célula única contra células-alvo sem a necessidade de ampla experiência ou equipamento altamente especializado. O ensaio pode ser feito facilmente usando ferramentas existentes, como citometria de fluxo ou microscopia. Gotículas de emulsão dupla também são passíveis de classificação usando classificadores de células30,31, o que pode permitir o isolamento de células com funcionalidades específicas, por exemplo, células T CAR com e sem potencial citotóxico, seguido de análise de transcriptoma de célula única.
A tecnologia não é isenta de limitações. Embora algumas linhagens celulares tolerem a cultura em gotículas por 24 h ou mais, as células primárias podem ter viabilidade significativamente menor após 24 h. Isso representa uma restrição de tempo nos ensaios em geral, mas para o ensaio atual, GzmB perceptível e atividade de morte celular podem ser observados dentro de 4-6 h, provavelmente porque o pequeno compartimento de gotículas garante um encontro rápido entre as células efetoras e alvo. Da mesma forma, o pequeno volume de gotículas garantirá um rápido acúmulo de concentração para níveis detectáveis do substrato secretado clivado por GzmB. Outra limitação da tecnologia é a incapacidade de detectar mortes em série por células efetoras ao usar citômetros de fluxo padrão. No entanto, isso pode ser possível com citômetros de fluxo de imagem ou tecnologias de citometria de imagem, que terão que ser investigadas.
A transferência adotiva de células T CAR CD19 mostrou notável sucesso no tratamento de pacientes com neoplasias hematológicas. Apesar disso, há uma grande variação de resposta e toxicidade imprevisível em pacientes32, o que pode ser em parte devido à heterogeneidade dentro do produto de infusão de CAR T. Como resultado, há um interesse crescente em analisar a composição fenotípica e a capacidade citotóxica de células T CAR individuais dentro de uma população. Isso será particularmente importante, pois a terapia CAR é cada vez mais testada em condições autoimunes e em outras formas de câncer. O dispositivo e o protocolo microfluídicos descritos aqui oferecem uma abordagem robusta e versátil para examinar a heterogeneidade das células T CAR e outras terapias baseadas em células.
Divulgações
Os autores declaram os seguintes interesses conflitantes: M.B.B. recebeu honorários de consultoria da Janssen, Roche e Kite/Gilead, não relacionados ao presente trabalho. D.L.P. e P.M. são funcionários da Samplix.
Agradecimentos
Os autores gostariam de agradecer aos membros e funcionários da CITCO e da Samplix pelas discussões e sugestões úteis. MBB é apoiado pela bolsa de estudos de cientistas clínicos em início de carreira da Fundação Lundbeck (R381-2021-1278). Este trabalho é apoiado por uma bolsa de pesquisa de elite do Hospital Universitário de Odense. Além disso, esta pesquisa foi apoiada por uma bolsa do Projeto do Conselho Europeu de Inovação 190144395 à Samplix ApS.
Materiais
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Blocker BSA (BSA) | Thermo Scientific | 37525 | |
| CellTrace Far Red Cell Proliferation Kit (far red fluorescent dye) | Invitrogen | C34564 | |
| CellTrace Violet Cell Proliferation Kit (violet fluorescent dye) | Invitrogen | C34557 | |
| DE Stabilizing Solution (stabilizing solution) | Samplix | REDIVSTABSOL1500 | |
| DPBS (dPBS) | Gibco | 14190-094 | |
| Dulbecco’s PBS (dPBS) | Capricorn Scientific | PBS-1A | |
| Dynabeads Human T-Activator (CD3/CD28 activation beads) | Gibco | 11132D | |
| Fetal Bovine Serum (FBS) | Capricorn Scientific | FBS-HI-12A | |
| Lenti-X Concentrator (PEG-based reagent) | Takara Bio | 631232 | |
| MACSelect LNGFR Microbeads (anti-NGFR magnetic beads) | Miltenyi Biotec | 130-091-330 | |
| OptiPrep density gradient medium (gradient medium) | Stemcell | 7820 | |
| Penicillin-Streptomycin (P/S) | Capricorn Scientific | PS-B | |
| Propidium iodide (PI) | Invitrogen | BMS500PI | |
| Recombinant Human IL-2 (IL-2) | Peprotech | 200-02 | |
| RPMI-1640 with Stable Glutamine (RPMI-1640) | Capricorn Scientific | RPMI-STA | |
| RPMI-1640 without L-Glutamine and phenol red | Gibco | 32404-014 | |
| Xdrop DE oil I (oil) | Samplix | REOILDEC1900 | |
| Xdrop Granzyme B substrate (GzmB substrate) | Samplix | REGRB100 | |
| Zombie-NIR viability dye (viability dye) | BioLegend | 423106 | |
| Plasticware etc. | |||
| 8-chamber glass slide | Chemometec | 942-0003 | |
| Cell culture plate, 12 well | TH Geyer | 7696791 | |
| DNA LoBind tube, 2 mL (DNA tube) | Eppendorf | 30108078 | |
| Eppendorf tube, 1.5 mL (1.5 mL tube) | Eppendorf | 30108051 | |
| Falcon tube, 15 mL (15 mL tube) | TPP | 91015 | |
| Falcon tube, 5 mL (5 mL tube) | Falcon (VWR) | 734-0443 | |
| Green cell suspension flasks for cell incubations (T75 flask) | Sarstedt | 148.19.22 | |
| Green cell suspension plates for cell incubations (96 well plate) | Sarstedt | 148.32.20 | |
| LS Separation Columns (separation column) | Miltenyi Biotec | 130-042-401 | |
| Xdrop DE Gaskets (gaskets) | Samplix | #GADEA100 | |
| Xdrop DE50 Cartridge (encapsulation cartridge) | Samplix | #CADE50A100 | |
| Antibodies | |||
| anti-CCR7 PE-Dazzle 594 | BioLegend | 353236 | |
| anti-CD19 CAR FMC63 Idiotype Antibody, PE | Miltenyi Biotec | 130-127-342 | |
| anti-CD3 APC | Biolegend | 300439 | |
| anti-CD3 BV480 | BD Biosciences | 566105 | |
| anti-CD3 FITC | BD Biosciences | 345763 | |
| anti-CD4 BUV661 | BD Biosciences | 612962 | |
| anti-CD4 StarBright Violet 760 | Bio-Rad | MCA1267SBV760T | |
| anti-CD45RA BUV395 | BD Biosciences | 740298 | |
| anti-CD8 PE-Cy7 | BioLegend | 344712 | |
| anti-CD8 StarBright Violet | Bio-Rad | MCA1226SBV760 | |
| anti-NGFR FITC | BioLegend | 345106 | |
| anti-NGFR PE | BioLegend | 345106 | |
| Cells | |||
| JeKo-1 Mantle-cell lymphoma cell-line (JeKo-1) | ATCC | CRL-3006 | |
| Primary peripheral blood mononuclear cells (PBMCs) | |||
| Equipment | |||
| Countess 3 Automated Cell Counter | Thermo Fisher Scientific | A50298 | |
| DynaMag-2 Magnet | Invitrogen | 12321D | |
| NovoCyte Quanteon Flow Cytometer (flow cytometer) | Agilent | 2010011AA | |
| Xdrop (microfluidics device) | Samplix | IN00110-EU |
Referências
- Pasqui, D. M., Latorraca, C. D. O. C., Pacheco, R. L., Riera, R. CAR-T cell therapy for patients with hematological malignancies. A systematic review. Eur J Haematol. 109 (6), 601-618 (2022).
- Krejcik, J., et al. Harnessing the immune system to fight multiple myeloma. Cancers. 13 (18), 4546 (2021).
- Tokarew, N., Ogonek, J., Endres, S., von Bergwelt-Baildon, M., Kobold, S. Teaching an old dog new tricks: next-generation CAR T cells. British J Cancer. 120 (1), 26-37 (2019).
- Bogetofte Barnkob, M., Vitting-Seerup, K., Rønn Olsen, L. Target isoforms are an overlooked challenge and opportunity in chimeric antigen receptor cell therapy. Immunother Adv. 2 (1), ltac009 (2022).
- Daei Sorkhabi, A., et al. The current landscape of CAR T-cell therapy for solid tumors: Mechanisms, research progress, challenges, and counterstrategies. Front Immunol. 14, 1113882 (2023).
- . Considerations for the Development of Chimeric Antigen Receptor (CAR) T Cell Products Available from: https://www.fda.gov/regulatory-information/search-fda-guidance-documents/considerations-development-chimeric-antigen-receptor-car-t-cell-products (2024)
- Roddie, C., O'Reilly, M., Dias Alves Pinto, J., Vispute, K., Lowdell, M. Manufacturing chimeric antigen receptor T cells: issues and challenges. Cytotherapy. 21 (3), 327-340 (2019).
- Wang, L., et al. Improvement of in vitro potency assays by a resting step for clinical-grade chimeric antigen receptor engineered T cells. Cytotherapy. 21 (5), 566-578 (2019).
- Burrows, S. R., Fernan, A., Argaet, V., Suhrbier, A. Bystander apoptosis induced by CD8+ cytotoxic T cell (CTL) clones: implications for CTL lytic mechanisms. Int Immunol. 5 (9), 1049-1058 (1993).
- Gattinoni, L., et al. Acquisition of full effector function in vitro paradoxically impairs the in vivo antitumor efficacy of adoptively transferred CD8+ T cells. J Clin Invest. 115 (6), 1616-1626 (2005).
- Chen, G. M., et al. Integrative bulk and single-cell profiling of premanufacture T-cell populations reveals factors mediating long-term persistence of CAR T-cell therapy. Cancer Dis. 11 (9), 2186-2199 (2021).
- Frazer, G. L., Gawden-Bone, C. M., Dieckmann, N. M. G., Asano, Y., Griffiths, G. M. Signal strength controls the rate of polarization within CTLs during killing. J Cell Biol. 220 (10), e202104093 (2021).
- Hermans, I. F., et al. The VITAL assay: a versatile fluorometric technique for assessing CTL- and NKT-mediated cytotoxicity against multiple targets in vitro and in vivo. J Immunol Meth. 285 (1), 25-40 (2004).
- Zaritskaya, L., Shurin, M. R., Sayers, T. J., Malyguine, A. M. New flow cytometric assays for monitoring cell-mediated cytotoxicity. Expert Rev Vaccines. 9 (6), 601-616 (2010).
- Piccinini, C., et al. In vitro CAR-T cell killing: validation of the potency assay. Cancer Immunol Immunother CII. 73 (9), 168 (2024).
- Martinez, E. M., et al. High-throughput flow cytometric method for the simultaneous measurement of CAR-T cell characterization and cytotoxicity against solid tumor cell lines. SLAS Disc Adv Life Sci. 23 (7), 603-612 (2018).
- Jedema, I., van der Werff, N. M., Barge, R. M. Y., Willemze, R., Falkenburg, J. H. F. New CFSE-based assay to determine susceptibility to lysis by cytotoxic T cells of leukemic precursor cells within a heterogeneous target cell population. Blood. 103 (7), 2677-2682 (2004).
- Liu, L., et al. Cellular and molecular imaging of CAR-T cell-based immunotherapy. Adv Drug Delivery Rev. 203, 115135 (2023).
- Liadi, I., et al. Defining potency of CAR+ T cells: Fast and furious or slow and steady. Oncoimmunology. 8 (10), e1051298 (2019).
- Xhangolli, I., et al. Single-cell analysis of CAR-T cell activation reveals a mixed TH1/TH2 response independent of differentiation. Genom Proteom Bioinfo. 17 (2), 129-139 (2019).
- Zhou, Y., et al. Evaluation of single-cell cytokine secretion and cell-cell interactions with a hierarchical loading microwell chip. Cell Rep. 31 (4), 107574 (2020).
- LaBelle, C. A., Zhang, R. J., Hunsucker, S. A., Armistead, P. M., Allbritton, N. L. Microraft arrays for serial-killer CD19 chimeric antigen receptor T cells and single cell isolation. Cytometry J Int Soc Anal Cytol. 103 (3), 208-220 (2023).
- Hellmann, M. J., et al. Heterogeneously deacetylated chitosans possess an unexpected regular pattern favoring acetylation at every third position. Nat Comm. 15 (1), 6695 (2024).
- Wong, K. U., et al. Assessment of chimeric antigen receptor T cytotoxicity by droplet microfluidics in vitro. Antibody Therapeut. 5 (2), 85-99 (2022).
- Antona, S., Platzman, I., Spatz, J. P. Droplet-based cytotoxicity assay: Implementation of time-efficient screening of antitumor activity of natural killer cells. ACS omega. 5 (38), 24674-24683 (2020).
- Subedi, N., et al. An automated real-time microfluidic platform to probe single NK cell heterogeneity and cytotoxicity on-chip. Sci Rep. 11 (1), 17084 (2021).
- Zhang, Y., et al. Enhanced CRISPR/Cas12a-based quantitative detection of nucleic acids using double emulsion droplets. Biosens Bioelect. 257, 116339 (2024).
- Kochenderfer, J. N., et al. Construction and preclinical evaluation of an anti-CD19 chimeric antigen receptor. J Immunother. 32 (7), 689-702 (2009).
- Michaels, Y. S., et al. Precise tuning of gene expression levels in mammalian cells. Nat Comm. 10 (1), 818 (2019).
- Yuan, Y., et al. Droplet encapsulation improves accuracy of immune cell cytokine capture assays. Lab on a Chip. 20 (8), 1513-1520 (2020).
- Brower, K. K., et al. Double emulsion flow cytometry with high-throughput single droplet isolation and nucleic acid recovery. Lab on a Chip. 20 (12), 2062-2074 (2020).
- Kirouac, D. C., et al. Deconvolution of clinical variance in CAR-T cell pharmacology and response. Nat Biotechnol. 41 (11), 1606-1617 (2023).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados