A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
העצרת ומעקב אחר פיתוח הקהילה מיקרוביאלית בתוך פלטפורמת מערך Microell
In This Article
Summary
הפיתוח של קהילות חיידקים תלוי בשילוב של גורמים, כולל אדריכלות סביבתית, שפע חבר, תכונות, ואינטראקציות. פרוטוקול זה מתאר סביבה סינתטי, microfabricated למעקב בו זמנית של אלפי קהילות הכלול בארות femtoliter, שבו גורמים מרכזיים כגון גודל נישה כליאה ניתן בקירוב.
Abstract
הפיתוח של קהילות חיידקים תלוי בשילוב של גורמים דטרמיניסטיים וסטוכסטיים מורכבים שיכולים לשנות באופן דרמטי את ההתפלגות המרחבית ואת פעילותם של חברי הקהילה. פיתחנו פלטפורמת מערך microell שניתן להשתמש בה במהירות להרכיב ולעקוב אחר אלפי קהילות חיידקים במקביל. פרוטוקול זה מדגיש את השירות של הפלטפורמה ומתאר את השימוש שלה עבור ניטור אופטי של פיתוח של קהילות פשוטות, שתי חברות בתוך אנסמבל של מערכים בתוך הפלטפורמה. הדגמה זו משתמשת בשני מוטציות של Pseudomonas aeruginosa , חלק מסדרה של מוטציות שפותחו כדי לחקור פתוגניות של סוג VI. הוספת כרומוזומלית של גנים mCherry או GFP או להקל על הביטוי המכונן של חלבונים ניאון עם אורכי גל פליטה שונים שניתן להשתמש בהם כדי לפקח על שפע הקהילה ואת המיקום בתוך microwell. פרוטוקול זה מתאר מת 'מפורטד להרכבת תערובות של חיידקים לתוך הבארות של המערך ושימוש זמן לשגות הדמיה פלואורסציה ניתוח כמותי התמונה כדי למדוד את הצמיחה היחסית של כל אוכלוסייה חבר לאורך זמן. זריעה והרכבה של פלטפורמת microwell, ההליכים הדמיה הכרחי לניתוח כמותי של קהילות חיידקים בתוך המערך, ואת השיטות שניתן להשתמש בהם כדי לחשוף אינטראקציות בין מינים באזור מינים כל דנו.
Introduction
קהילות מיקרוביאליות מעוצבות על ידי גורמים דטרמיניסטיים, כגון מבנה הסביבה ותהליכים סטוכסטיים, הקשורים למוות תאים, חלוקה, ריכוז חלבון, מספר האברונים, ומוטציה 1 . בתוך הסביבה הטבעית, זה כמעט בלתי אפשרי לנתח את ההשפעה הפרטנית של השפעות אלה על הרכב הקהילה ופעילותה. המסתתרים על ידי מבנים טבעיים ונקברים בתוך סביבה כימית וביולוגית, זיהוי חברי הקהילה והמשך פתרון הפיצול הספטיוטמפוראלי שלהם בתוך הסביבה הטבעית הם מאתגרים ביותר. עם זאת, המאמצים האחרונים הדגישו את חשיבותו של הארגון המרחבי על תפקוד הקהילה והצביעו על הצורך לתת דין וחשבון על שפע הארגון וארגונו במחקרים מתמשכים 2 , 3 , 4 .
זהברור כי הסביבה הכימית המקומית ( כלומר , הזמינות של חומרים מזינים וחילוף חומרים משני), המבנה הפיזי ( למשל, ארכיטקטורה של אדמה, שורשי צמחים, חלקיקי האוקיינוס או מיקרוביילי המעי), נוכחות או היעדר חמצן והכנסת מינים פתוגניים משפיעים על הרכב, ארכיטקטורה ותפקוד של קהילות מיקרוביאליות 5 , 6 , 7 , 8 , 9 , 10 , 11 . עם זאת, טכניקות מסורתיות עבור תרבויות הזנחה ללכוד גורמים אלה ממשיכים לגבור. הרכב הקהילה ( למשל, נוכחות של מינים תלויי תלות), התקשרות פיזית, ריכוז מולקולה איתות, קשר ישיר תא סלולרי הם כל הגורמים החשובים לעיצוב קהילה חיידקים והוא יכול ללכת לאיבוד גתנאי התרבות האונבנציונלית. מאפיינים אלה קשה לשכפל בתרבות נוזלי בתפזורת או על צלחת אגר. הזמינות של טכניקות microfluidic, micropatterning ו- nanofabrication המאפשרות שכפול של תכונות פיסיקליות וכימיות עיקריות של סביבות טבעיות אפשרה לחוקרים רבים לבנות קהילות חיידקים כדי לחקור את האינטראקציות שלהם 12 , 13 , 14 ולפתח סביבות מלאכותיות לחקות את התנאים הטבעיים 4 , 15 , 16 , 17 , 18 , 19 , 20 .
פרוטוקול זה מתאר שיטה לפברק מכשיר מערך microell ומספק נהלים ניסיוניים מפורט שניתן להשתמש בהם כדי פונקציונליE wells במערך לגדל חיידקים, הן כמושבות יחיד מינים בקהילות מרובות חברים. עבודה זו גם ממחישה כיצד חיידקים שונה כדי לייצר חלבונים הכתב ניאון ניתן להשתמש כדי לפקח על צמיחה חיידקים בתוך בארות לאורך זמן. מערך דומה הוצג בעבר הראה כי ניתן לעקוב אחר הצמיחה של מינים בודדים מינים של Pseudomonas aeruginosa ( פ aeruginosa) ב microwells. על ידי שינוי גודל וצפיפות זריעה היטב, התנאים ההתחלתיים של אלפי ניסויים הצמיחה יכול להיות מגוונות במקביל כדי לקבוע כיצד תנאי חיסון ראשוני להשפיע על היכולת של חיידקים לגדול 21 . העבודה הנוכחית משתמשת בגירסה שונה מעט של מערך microwell אשר בונה על העבודה הקודמת בכך שהוא מאפשר השוואה בו זמנית של מערכים מרובים באמצעות פרוטוקול ניסיוני חזק יותר. המערך שבו נעשה שימוש בעבודה זו מכיל מספר תת-מערכות משנה, או מערך אנסםBles, המכיל בארות בגדלים שונים, הנעים בין 15-100 מיקרומטר בקוטר, אשר מסודרים על שלוש זפות שונות ( כלומר 2x, 3x, ו 4x קוטר). מערכים הם חרוט לתוך סיליקון, ואת הצמיחה של חיידקים seeded במערכי הסיליקון מאופשר על ידי איטום מערכים עם coverslip כי כבר מצופה עם ג'ל agarose בינונית חדורים. P. aeruginosa מוטנטים שנועדו ללמוד את מערכת הפרשת סוג VI משמשים בהפגנה זו.
התוצאות המוצגות כאן לבנות לקראת המטרה האולטימטיבית של ניתוח קהילות multimember בתוך מערכים microwell, המאפשר לחוקרים לפקח על שפע וארגון של חיידקים באתרם תוך שליטה וחוקר הסביבה הכימית. זה צריך בסופו של דבר לספק תובנות את "הכללים" אשר קובעים פיתוח הקהילה ואת הירושה.
Protocol
1. Silicon Microwell- מערך ייצור
- ציפוי פרילין
- הפקדה בין 1-1.5 מיקרומטר של parylene N על ופלים סיליקון באמצעות מערכת ציפוי parylene זמין מסחרית על פי מפרטים של היצרן והוראות (הגדרות: מאדה להגדיר נקודה = 160 ° C; תנור להגדיר נקודת = 650 ° C).
הערה: כ 6 גרם של parylene N נטען לתוך ציפויים תשומות קאמרית 1-1.5 מיקרומטר עבה.
- הפקדה בין 1-1.5 מיקרומטר של parylene N על ופלים סיליקון באמצעות מערכת ציפוי parylene זמין מסחרית על פי מפרטים של היצרן והוראות (הגדרות: מאדה להגדיר נקודה = 160 ° C; תנור להגדיר נקודת = 650 ° C).
- פוטוליטוגרפיה
- ספין מעיל ופרילין N- מצופה ופלים עם האמרגן הידבקות, 20% hexamethyldisilazane (HMDS), ו 80% פרופילן גליקול monomethyl אתר אצטט (PGMEA) (ראה טבלה של חומרים ) ב 3000 סל"ד במשך 45 שניות. ממלאים פיפטה העברת 2 מ"ל עם האמרגן הידבקות ומפזרים אותו על פרוסות כולו. אפשר פרוסות לשבת על כ 10 s לפני ספינינג אותו יבש.
- מילוי פיפטה העברת 2 מ"ל עם חיובי לNe photoresist (ראה טבלה של חומרים ) ו לוותר על photoresist במרכז רקיק. ספין בסל"ד 3,000 עבור 45 ים להניב ציפוי להתנגד כי הוא כ 1.5 מיקרומטר עבה.
- רך לאפות את דגימות על hotplate ב 115 מעלות צלזיוס למשך 1 דקות.
- השתמש aligner קשר photomask עם דפוס היטב הרצוי לחשוף את המדגם לאור אולטרה סגול. לחשוף את רקיק מצופה ספין דרך photomask בדוגמת במשך 6 שניות, נותן מינון משוער של 60-80 mJ / cm 2 נמדד ב 365 ננומטר.
- פיתוח דפוס על ידי שקוע המדגם מפתח (<3% tetramethyl amroium hydroxide במים, לראות את לוח החומרים ) במשך 2 דקות. לשטוף עם מים DI יבש עם חנקן נקי, יבש.
הערה: שטחי photoresist חשופים UV יש לנקות במהלך הפיתוח.
- ריאקציה יונית תגובתי
- השתמש לחרוט פלזמה חמצן כדי להסיר את parylene חשוףכל הדרך למצע הסיליקון.
הערה: המתכון יכול להיות מאופנן כדי לשנות את שיעור חרוט של parylene. עבור עובי parylene בין 1 ל 5 מיקרומטר, השתמש מתכון עם 60 mTorr, 20 ° C, 100 Sccm O 2 , 10 W RF, ו 2000 W ICP על תגובתי יון תחריט (RIE) כלי. לאחר חריטה והסרה של שכבת parylene חשופים, אזור בדוגמת ( כלומר, הסיליקון חשוף) צריך להיראות מבריק וכסף. - השתמש עמוק RIE (DRIE, למשל, Bosch DRIE) תהליך לחרוט כדי לחרוט לתוך הסיליקון.
הערה: קצב חריץ ומשך יקבע את עומק היטב. מחזור אחד שלם של תהליך בוש (שלב 3 בתצהיר: 20 mTorr, 15 ° C, 140 sccm C 4 F 8 , 10 W RF, ו 1,750 W ICP ואחריו תהליך 10 etch: 20 mTorr, 15 ° C , 120 sccm SF 6 , 8 W RF, ו 1,750 W ICP) מתאים כ 1 מיקרומטר של עומק חרוט. הבארות המשמשים בהפגנה זו נע בין 3 - 3.5 מיקרומטר עמוק. - אשרלהקים את עומק חרוט באמצעות profilometry פיזית.
- טען את המדגם לתוך profilometer פיזית (ראה טבלה של חומרים ).
- הפעל את ואקום המדגם ולחץ על לחצן העומס הידני.
- פוקוס על המערכת על ידי לחיצה על כפתור "פוקוס". מקם תכונה מתאימה למדידה במסך התצוגה.
- סרוק את המדגם. שלב את הפרופיל ולמדוד את עומק התכונה.
- הקלט את קצב חריץ ו לווסת פעמים etch הבאים כדי להשיג את העומק הרצוי.
הערה: המדידות יכללו את עומק הסיליקון היטב, את עובי parylene שהופקדו, ואת עובי photoresist. אימות עובי של כל שכבה לאורך ההליך הוא הכרחי כדי להשיג עומק מדויק מדויק.
- השתמש לחרוט פלזמה חמצן כדי להסיר את parylene חשוףכל הדרך למצע הסיליקון.
2. תרבות חיידקים זריעת ( איור 1 א )
- מושבות התחל על Luria Broth (LB) אגר צלחות מניות גליצרול ולהשתמש בתוך שבועיים. בחר מושבות של הזנים הרצוי של צלחות אגר LB ולהתחיל תרבויות לילה של פ aeruginosa. דגירה תרבויות לילה במשך כ 18 שעות ב 37 ° C תוך רעד ב 220 סל"ד בינוני R2A.
הערה: המושבות צריך להיבחר בתוך שבועיים של ציפוי על מנת להבטיח את המוטציות ואת גנים כתב ניאון נשמרים. כל עבודה P. aeruginosa צריך להיעשות בתנאים BSL-2. - השתמש בסופר היהלום כדי לפרק את פרוסות הסיליקון לתוך שבבים בודדים המכילים את ההרכבים בגדלים שונים מערכים המגרש היטב. ודא כי כל שבב מכיל את מלוא השלמה של גדלים היטב ואת המגרש עבור המחקר.
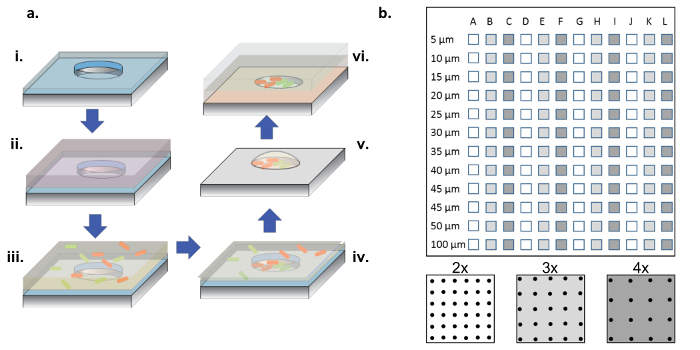
איור 1: ייצור ו Cell זרע נוהל. ( א ) מערכי Microell אנחרט מחדש לתוך פרוסות סיליקון מצופה בשכבה דקה של parylene (i). כדי להרטיב את הבארות ו / או פונקציונלי את פני השטח, פתרון חלבון מתווסף טיפה על גבי מערכים (ii). הפתרון חלבון מוסר, ופלים מיובשים, וכן פתרון חדש המכיל את החיידקים הרצוי נוסף (iii). הפתרון החיידקי מוסר לאחר תקופת הדגירה, ופלים מותר להתייבש, משאיר מאחוריו חיידקים בבארות ועל פני השטח (iv). החיידקים הקשורים משטח מוסרים עם parylene להרים- off, משאירים מאחור חיידקים seeded נקי microells ועדיין קיימא בשל בינוני גליצרול 2%, אשר מסייע לשמור על בארות hydrated (V). שבבי סיליקון ממוקמים מכן בצד מערך למטה על agarose ג'ל מצופה coverslip זכוכית, אשר מזין צמיחה חיידקים microwells (vi). ( ב ) פריסת תת מערכים על מכשיר סיליקון יחיד. כל מערך משנה מכיל קבוצה של בארות זהות. הקוטר של microwells בכל תת-אRrays טווח בקוטר של 5-100 מיקרומטר מאורגנים ב 2x, 3x, או 4x את הקוטר היטב המגרש, אשר מסומן על ידי לבן כדי אפור כהה צבעים על לוחית הסכימה התחתונה. כאשר מעמקי הבאר הם רדודים (<10 מיקרומטר), 5 ו 10 מיקרומטר היטב בקטרים מועילים לעתים נדירות, בדרך כלל בגלל חוסר תאים ליישב אלה בארות קטנות מאוד. בעבודה זו, רק את הנתונים מבארות עם 15-100 קוטר מיקרומטר נותחו. אנא לחץ כאן כדי להציג גרסה גדולה יותר של דמות זו.
הערה: כפי שמוצג בתרשים 1b , שבב מלא מכיל מערכי משנה של בארות, בקוטר שבין 5 ל 100 מיקרומטר, עם שלוש זפות שונות ( כלומר 2x, 3x, 4x קוטר) חוזר 4 פעמים.
- מניחים 150 droplet μL של 500 מיקרוגרם / מ"ל אלבומין בסרום שור (BSA) ב PBS פתרוןעל גבי המערך כדי להרטיב את microwells. דגירה הפתרון BSA עבור 1 שעה על השבב ב RT בחדר לח.
- יצירת החדר על ידי מילוי החלק התחתון של תיבת פיפטה קצה ריק עם פוספט שנאגרו מלוחים (PBS).
הערה: חומרים אחרים, כגון lectins ספציפיים, ניתן להשתמש במקום BSA כדי לתפקד את פני השטח של microwells.
- יצירת החדר על ידי מילוי החלק התחתון של תיבת פיפטה קצה ריק עם פוספט שנאגרו מלוחים (PBS).
- בעוד דגירה שבבי סיליקון עם פתרון BSA, צנטריפוגה התרבויות בסל"ד 2,500 (המקביל לממוצע של 950 XG) במשך 5 דקות ולאחר מכן resuspend אותם μL 500 של מדיום R2A טרי עם גליצרול 2%.
- לקבוע את OD של התרבות באמצעות ספקטרומטר UV-vis ב 600 ננומטר. התאם אותו OD של 0.02 באמצעות בינוני 2% גליצרול R2A.
הערה: גליצרול מסייע למנוע את הבארות מתייבש במהלך parylene להרים- off.
- לקבוע את OD של התרבות באמצעות ספקטרומטר UV-vis ב 600 ננומטר. התאם אותו OD של 0.02 באמצעות בינוני 2% גליצרול R2A.
- לאחר הדגירה, להסיר את הפתרון BSA ולשטוף 3x עם PBS על ידי הסרת והחלפת נוזל נוזללא מכסה את מערך סיליקון microwell. יבש בחנקן.
- הוסף 150 μL של 0.02 תרבויות OD לכל אחד מערכים יבש להציב תא לח. לדגור על 1 שעות ב 4 ° C כדי לאפשר את החיידקים לדבוק קירות היטב.
הערה: קירור אינו נדרש עבור הדגירה. 4 ° C דגירה זמן ניתן להשתמש כדי למנוע את הצמיחה של חיידקים לפני הדמיה מתחילה, כך שניתן לדמיין את הארגון המרחבי של הקהילות לפני הצמיחה. ניתן להשתמש בדגירת טמפרטורת החדר. שני הפרוטוקולים לגרום עקומות הצמיחה דומה.
3. מיקרוסקופ הגדרת
- לפני תחילת הדגירה חיידקים על שבבי סיליקון, להדליק את הבמה העליונה בקרת הסביבה קאמרית (עיין בטבלה של חומרים) ולהתאים את ההגדרות על תיבת הבקרה כך הלחות (~ 100%) והטמפרטורה (30-32 ° C, ראה שלב 3.2) יכול לאזן לפני הוספת דוגמאות.
- רמה את בעל המדגם ואת קו פנימה Terior סביב המדגם עם מגבונים PBS ספוג מעבדה (ראה טבלה של חומרים ) כדי להגדיל את הלחות בחדר לנקודת טל. הגדר את הטמפרטורה של החדר ל 30 מעלות צלזיוס וכי מכסה תא ל 32 מעלות צלזיוס כדי להפחית את עיבוי על המטוס הדמיה.
הערה: מחזיק שקופיות נכנס לחדר תא חי עם אטם כי הוא כ 1 ס"מ עובי. מחזיק המדגם הוא מפולס בסיוע ברמה בועה כי הוא הניח על גבי המדגם. בעל המדגם יכול להיות מוטה מעט ולהישאר אטום אטם לרמה. - בעוד תרבויות הן incubating על שבבי סיליקון, ידני להפעיל את מתג ההפעלה עבור מנורת כספית לפחות 30 דקות לפני הדמיה. הפעל ידנית את המצלמה ואת הבמה המיקרוסקופ האוטומטית. פתח את התוכנה המשמשת לשלוט במיקרוסקופ וציוד היקפי ולוודא כי הציוד מוכר על ידי התוכנה.
הערה: ההגדלה היא 10X עם 0.3 = NA.
Class = "jove_title"> 4. הכנת Agarose מצופה זכוכית Coverslips
- מיקרוגל שהוכנו בעבר agarose פתרונות ( כלומר 2% agarose במדיום R2A) עד למצב נוזלי הוא הגיע, כ 60 s.
- רטוב בחלק האחורי של 75 מ"מ x 22 מ"מ, # 1.5 זכוכית coverslip עם אתנול ומניחים אותו לאורכו, במרכז, על פני שקופית זכוכית 2 x 3 "(50 x 75 מ"מ) .מיקום שני מרווחי PDMS (עובי של 1 מ"מ) לאורך הקצוות הארוכים של coverslip ולהעביר את coverslip זכוכית כך בערך 1 מ"מ של coverslip הוא overhanging את קצה השקופית.
- יוצקים 5 מ"ל של תמיסת agarose נוזלית על גבי coverslip זכוכית, רק מספיק כדי לכסות אותו לחלוטין, במקום השני 2 x 3 "שקופיות זכוכית על גבי הרכבה כדי" כריך "אגר נוזלי בין coverslip ו השקופית.
הערה: זה שולט בעומק agarose, מה שהופך את עובי הכולל של coverslip ו agarose מוקשה שווה עובי למרווחים PDMS. - אפשר זכוכית שקופית-coverslip-agarose ג'ל זכוכית שקופית כריך להגדיר עד הפתרון agarose מתחיל לחזק; ואז, להעביר אותו למקרר. לאחר 15 דקות, להסיר את agarose מוצק עודף לחתוך סביב coverslip זכוכית. מעבירים אותו לתבנית נקייה ומניחים אותו במקרר עד לשימוש.
5. איטום וולס עם Coverslip ו- Agarose מצופה הדמיה
- לאחר תקופת הדגירה חיידקים מוחלטת, להסיר את coverslip מצופה agarose מהמקרר ולהכין את שבבי סיליקון, כדלקמן.
- לטבול את שבבי הסיליקון במים ultrapure, אחד בכל פעם, במשך 10 שניות כל אחד. הגדר אותם על הקצוות שלהם על מעבדה לנגב או רקמות עד שרוב הנוזלים העודפים יש מרוקן מקצוות של הצ 'יפס.
- חותכים חתיכת קלטת כדי להתאים את קצה קצה של כל שבב סיליקון. מניחים את הקלטת על parylene כי הוא מכסה את הסיליקון ולהשתמש בו כדי לקלף במהירות את ציפוי parylene.
- Immedלהפוך את כל שבב מקולף ומקום כל שבב כזה בצד microwell מערך פונה (ועושה קשר עם) בצד agarose מצופה של coverslip מצופה agarose. הקפד לא לזוז או להזיז את השבב לאחר שהוא נוגע agarose כדי למנוע את הצמיחה של חיידקים מחוץ הבארות.
- מניחים את מערך microwell התאספו / coverslip agarose מחזיק השקופית של הבמה העליונה בקרת הסביבה קאמרית על miscroscope.
- השתמש באורות הסביבה או באור מכוון ( למשל, פנס) כדי לאתר מערכי עניין. השתמש בתוכנה המסחרית השולטת על הבמה האוטומטית כדי לשמור על מיקומים אלה (ראה טבלת החומרים). כבה את האור הסביבתי או המכוון לאחר שהמיקומים מאוחסנים.
- בתוכנה המסחרית, פתח את החלונית "ND רכישה".
הערה: לוח זה כולל תפריט לשמירה אוטומטית לספרייה מסוימת, כמו גם לרכישת תמונה לתכנות. עבור ניסויים אלה, התפריטים "זמן", "XY" ו- "λ" משמשים. - כדי לשמור את המיקומים בתוכנה, לחץ על "תפריט XY" ולאחר מכן סמן תיבה ריקה בצד שמאל עבור כל מיקום שיש לשמור. כמו כן, לחץ על הלחצן "כלול Z".
- לרכוש תמונות לאורך זמן באורכי גל הרצוי 10 הגדלה באמצעות קוביות מסנן הקרינה המתאים (ראה טבלה של חומרים).
- השתמש בתוכנות הבקרה ושמור את מיקומי המעברים כדי לעבור לכל מיקום שנשמר ולהתמקד בבארות. לחץ על כל מיקום XY ברשימה השמורה ולהתאים את המיקוד באמצעות מסנן חלבון הירוק (GFP) מסנן. שמור את מיקום z החדש על ידי לחיצה על החץ המצביע על מיקום z.
הערה: תהליך זה עשוי לגזול זמן רב. שקול לנקוט באמצעי זהירות של הגדלת הרווח באמצעות מסנן צפיפות נייטרלית כדי להפחית את עוצמת האור כדי למנוע photobleaching. - לקבוע את המרחק בין ציר הציר בין מטוסים מוקד לכל אורך גל על ידי ציון ההבדל במצב z- ציר כאשר התמקדו על פני השטח של המערך. בחר 2-3 מיקומים מהמערך עם האוכלוסייה מעורבת אדום / ירוק חיידקים ולהתמקד באמצעות פילטר הקרינה האדום (RFP) מסנן.
- הפחת את המרחק בין המטוסים מוקד באמצעות GFP ו RFP הקרינה מסננים ולהוסיף כי התאמה המטוס מוקד תחת תפריט "λ".
הערה: לדוגמה, אם המערך נראה ממוקד בערוץ ה- GFP במיקום z של 50 מיקרומטר, ואת המערך אותו נראה ממוקד בערוץ RFP ב 55 מיקרומטר, הוסף +5 ליד תצורה אופטי RFP ב "λ בתפריט.
- הפחת את המרחק בין המטוסים מוקד באמצעות GFP ו RFP הקרינה מסננים ולהוסיף כי התאמה המטוס מוקד תחת תפריט "λ".
- להתחיל זמן לשגות רכישת התמונה.
הערה: עבור הניסויים המוצגים כאן, RFP ותמונות GFP נרכשו עבור כל מיקום מערך במרווחים 30 דקות באמצעות רכישת תמונה מרובת מאפיינים באמצעות תוכנה מסחרית השולטתאת המצלמה, תריס, גלגל המסנן, ואת הבמה ממונע.- הגדר את "מרווח" ל 30 דקות ואת "משך הניסוי" ל 24 שעות תחת תפריט "זמן". לחץ על "הפעל כעת".
הערה: עם הזמן "תיבה", "XY" ו "λ" תיבות מסומנת, הפעלת התוכנית תעביר את הבמה לתמונה כל מיקום ( כלומר את מיקומי XYZ שנשמרו), לקחת תמונה באורך גל אחד, להזיז את המיקום z כדי להסביר את ההבדלים בין המטוס הפוקוס ( כלומר, lambda או אורך הגל שליטה), לקחת את התמונה השנייה, לעבור אל מיקום המערך הבא (multipoint), ואת לולאה זה ב 30 דקות אינטרוולים (זמן לשגות).
- הגדר את "מרווח" ל 30 דקות ואת "משך הניסוי" ל 24 שעות תחת תפריט "זמן". לחץ על "הפעל כעת".
- השתמש בתוכנות הבקרה ושמור את מיקומי המעברים כדי לעבור לכל מיקום שנשמר ולהתמקד בבארות. לחץ על כל מיקום XY ברשימה השמורה ולהתאים את המיקוד באמצעות מסנן חלבון הירוק (GFP) מסנן. שמור את מיקום z החדש על ידי לחיצה על החץ המצביע על מיקום z.
- לרכוש תמונות בקרת תאורה.
הערה: השתמש בתפריטים "ND רכישה", "זמן" ו- "XY" כדי לצלם תמונות של 4 מיקומים, 25x כל אחת.- קח סדרה של 100 "darkfield" תמונות על ידי כיבוי כל מקורות האור לוקח"תמונה" של שקופית רגילה. תמונות אלה יצלמו את רעש המצלמה. השתמש זמן חשיפה הארוך ביותר בשימוש במהלך timelapse (שלב 5.3.3).
- קח סדרה של 100 "שדה תאורה" תמונות על ידי הדמיה שקופית סטנדרטית ( כלומר RFP אחיד או עוצמת ה- GFP) בכמה מיקומים שונים כדי ללכוד את תאורה אחידה בתנאים הניסוי נתון. בחר זמן חשיפה שממקסם את האות מבלי להגיע לרוויה.
6. ניתוח
- לעבד את התמונה ערימות באמצעות תוכנת ניתוח תמונה ( למשל, ImageJ).
- להמיר את התמונות שנרכשו לפורמט קובץ tiff באמצעות תוכנה מסחרית. העלה תמונות לתוכנת ניתוח התמונות על ידי לחיצה על "קובץ"> "ייבוא"> "רצף תמונות".
- יצירת "תיקון תמונה" על ידי ממוצעים של כל "darkfield" ו "שדה תאורה" תמונות. תת-סמךT התמונה הממוצעת "darkfield" תמונה ממוצעת "שדה תאורה" על ידי בחירת "תהליך"> "מחשבון תמונה". בחר את שתי התמונות, "Image1" ו- "Image2" ולאחר מכן "Subtract" בשדה "מבצע". לחץ על "אישור".
- עבור ממוצע, טען את תמונות התיקון (או darkfield), לחץ על "תמונה"> "ערימות"> "פרוייקט Z"> "תחזית ממוצעת".
- בצע רישום תמונה במידת הצורך. לאחר מכן, בצע חיסור רקע על-ידי לחיצה על 'עיבוד'> 'הפחתת רקע'. הזן רדיוס ( למשל, 125) בשדה "רדיוס" ובחר "פרבולואיד הזזה".
- בצע תיקון תאורה באמצעות "תהליך"> "מחשבון פלוס". בחר את הפרמטרים הבאים: פעולה, מחלק; I1, גם תמונה; I2, תמונת תיקון; K1, תיקון תמונה מתכוון; ו k2, 0. לחץ על "Creאכלו חלון חדש ".
הערה: סט נתונים זה לא דורש רישום, אבל בעבודה אחרת, ImageJ Plugin StackReg שימש עם טרנספורמציה "תרגום". עבור חיסור רקע, להשתמש באותו רדיוס paraboloid הזזה עבור כל תמונה מוגדר. לדוגמה, אם בארות הגדול ביותר צילמו יש רדיוס פיקסל של 100, להשתמש ברדיוס גדול מ -100 ( למשל, 125) עבור כל תמונה להגדיר.
- לקבוע את הצמיחה של כל זן microwells.
- בחר אזורים של עניין (ROIs) סביב microwell בכל המערכים הרצוי באמצעות ImageJ "MicroArray" תוסף.
- בתפריט "מפה", לחץ על "אפס רשת". ציין שורות, עמודות וקוטר (בהתאם לגודל ולמספר במערך, ראה איור 1 ב ). בחר "מעגל" מהתפריט "צורה ROI".
- החזק את מקש "Alt" בעת בחירת ROI השמאלי העליון עם העכבר כדי לעבור tהוא ROI מערך. החזק את המקש "Shift" בעת בחירת החזר ה- ROI השמאלי התחתון כדי לשנות את גודל המערך. החזק את המקש "Shift" בעת בחירת החזר ROI מהצד הימני של המערך, אך לא בפינות, כדי לשנות את המרווחים של החזר ההשקעה (ROI).
- השתמש בפקודות לעיל כדי להתאים את מערך ההחזר על ההשקעה על הבארות בתמונה. לחץ על "מדידת RT".
הערה: הפלאגין ייצא את המדידות הרצויות מכל החזר ROI. השתמש שלושה גדלים ROI, יצירת טבעות קונצנטריות סביב הבארות כדי לאסוף המקומי את האות הרקע ( כלומר, האות מן הטבעת באמצע מופחת מן הטבעת החיצונית) ואת מדידות הקרינה ( כלומר, האות מן הטבעת הפנימית).
- לאסוף את הנתונים בתוכנה גיליון אלקטרוני ולחשב את האות הרקע. לייבא אותו תוכנת scripting מותאמת אישית לניתוח נוסף.
- בחר אזורים של עניין (ROIs) סביב microwell בכל המערכים הרצוי באמצעות ImageJ "MicroArray" תוסף.
- ארגון נתונים וניתוח
- ייבא את הנתונים וארגן את הנתוניםשנאספו ב- ImageJ למטריצה בסדר הבא לכל הזמנים: עמודה 1, מספר מערך משנה; טור 2, שורה טובה; עמודה 3, עמודה טובה; טור 4, עוצמת מתכוון; עמודה 5, עוצמת הרקע; ואת עמודה 6, עוצמת אינטנסיביות - עוצמת הרקע.
- להפריד את התוצאות של הרכישה mCherry ו GFP לתוך מטריצות שונות. אחסן את התוצאות מכל מערך משנה וכל צבע בתא אחר במערך תאים.
הערה: ארגון זה מקל על מעבר הלוך ושוב בין נתוני תמונות ותוצאות מדידה, ניקוי הנתונים ולוודא שהמדידות מייצגות במדויק את הנתונים.
- להפריד את התוצאות של הרכישה mCherry ו GFP לתוך מטריצות שונות. אחסן את התוצאות מכל מערך משנה וכל צבע בתא אחר במערך תאים.
- התאם עבור autofluorescence של פ aeruginosa .
הערה: בניסויים מעורבים שיתוף תרבות של GFP ו mCherry זנים, שבב mCherry בלבד צריך להיות מנותח כדי לוודא את הקשר בין mCherry ו autofluorescence ירוק.- מגרש את האות mCherry לעומת GFP מכל mCherry ΔretS &# 916; tse / i1-6 בארות בכל עת נקודות כדי לקבוע את הקשר בין האות mCherry ו autofluorescence בערוץ ה- GFP. הפחת את האות autofluorescence מן שיתוף תרבויות.
- מגרש את המסלולים ולהתאים משוואה לוגיסטית שונה לכל מסלול כדי לחלץ פרמטרים באמצעות ריבועים לפחות התאמת או תוכנת גיליון אלקטרוני או תוכנה מותאמת אישית scripting.
- חפש קורלציות בין לבין הפרמטרים מסלול GFP ו mCherry.
- ייבא את הנתונים וארגן את הנתוניםשנאספו ב- ImageJ למטריצה בסדר הבא לכל הזמנים: עמודה 1, מספר מערך משנה; טור 2, שורה טובה; עמודה 3, עמודה טובה; טור 4, עוצמת מתכוון; עמודה 5, עוצמת הרקע; ואת עמודה 6, עוצמת אינטנסיביות - עוצמת הרקע.
תוצאות
הפלטפורמה הניסויית המוצגת כאן מיועדת לתפוקה גבוהה ולחקר תוכן גבוה של קהילות חיידקים. התכנון מאפשר לנתח בו זמנית אלפי קהילות, הגדלות בארות בגדלים שונים. עם עיצוב מערך microwell זה, את התלות של הרכב הקהילה הסופי על צפיפות זריעת הראשונית, גם גודל, סביבה כימ?...
Discussion
מאמר זה הציג מכשיר מערך microell ופרוטוקולים ניסיוניים שנועדו לאפשר תפוקה גבוהה תוכן גבוהה לחיות תא הדמיה מבוסס ניתוח של פיתוח הקהילה חיידקים. בעוד שמיקוד ההפגנה כאן היה לבחון את ההשפעות של הפרשת סוג 6 על פיתוח הקהילה, המערכים נועדו להיות גמישים ולהתאים את המחקר למגוון ר...
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
מערכים Microwell היו מפוברקות מאופיין במרכז חומרים Nanophase מדעי משתמש מתקני החטיבה, משרד בסיסי מדעי האנרגיה, מחלקת האנרגיה של ארה"ב. תמיכה כספית לעבודה זו נמסרה באמצעות Oak Ridge National Laboratory מנהל מחקר ופיתוח הקרן. המחברים גם רוצים להודות למעבדה ג 'יי מוגז (אוניברסיטת וושינגטון, סיאטל, WA) על אספקת זנים פ aeruginosa המשמשים מחקרים אלה.
Materials
| Name | Company | Catalog Number | Comments |
| Parylene N | Specialty Coating Systems | CAS NO.:1633-22-3 | |
| Parylene coater | Specialty Coating Systems | Labcoter 2 Parylene Deposition Unit PDS2010 | |
| Silicon Wafer | WRS Materials | 100mm diameter, 500-550μm thickness, Prime, 10-20 resistivity, N/Phos<100>, | |
| adhesion promoter | Shin-Etsu Microsci | MicroPrime P20 adhesion promoter | |
| postive tone photoresist | Rohm and Haas Electronics Materials LLC (Owned by Dow) | Microposit S1818 Positive Photoresist (code 10018357) | |
| Quintel Contact Aligner | Neutronix Quintel Corp | NXQ 7500 Mask Aligner | |
| Reactive Ion Etching Tool | Oxford Instruments | Plasmalab System 100 Reactive Ion Etcher | |
| R2A Broth | TEKnova | R0005 | |
| Bovine Serum Albumin | Sigma | A9647 | |
| Multimode Plate Reader | Perkin Elmer | Enspire, 2300-0000 | |
| Fluorescent Microscope | Nikon | Eclipse Ti-U | |
| Automated Stage | Prior | ProScan III | |
| CCD camera | Nikon | DS-QiMc | |
| Stage-top environmental control chamber | In Vivo Scientific | STEV ECU-HOC | |
| Phosphate Buffered Saline | ThermoFisher Scientific | 14190144 | |
| UltraPure Agarose | ThermoFisher Scientific | 16500500 | |
| 25 x 75 mm No. 1.5 coverslip | Nexterion | High performance #1.5H coverslips | |
| Fluorescence Reference Slides | Ted Pella | 2273 | |
| Physical Stylus Profilometer | KLA Tencor | P-6 | |
| lab wipes | Kimberly Clark | Kimipe KIMTECH SCIENCE Brand, 34155 | |
| commercial software | Nikon | NIS Elements | |
| Zeiss 710 Confocal Microscope | Zeiss | ||
| filter cubes | Nikon | Nikon FITC (96311), Nikon Texas Red(96313) |
References
- Zhou, J., Deng, Y., et al. Stochasticity, succession, and environmental perturbations in a fluidic ecosystem. Proc Natl Acad Sci. 111, E836-E845 (2014).
- Valm, A. M., Welch, J. L. M., et al. Systems-level analysis of microbial community organization through combinatorial labeling and spectral imaging. Proc Natl Acad Sci USA. 108 (10), 4152-4157 (2011).
- Satoh, H., Miura, Y., Tsushima, I., Okabe, S. Layered structure of bacterial and archaeal communities and their in situ activities in anaerobic granules. Appl Environ Microbiol. 73 (22), 7300-7307 (2007).
- Kim, H. J., Boedicker, J. Q., Choi, J. W., Ismagilov, R. F. Defined spatial structure stabilizes a synthetic multispecies bacterial community. Proc Natl Acad Sci USA. 105 (47), 18188-18193 (2008).
- Nunan, N., Wu, K., Young, I. M., Crawford, J. W., Ritz, K. Spatial distribution of bacterial communities and their relationships with the micro-architecture of soil. FEMS Microbiol Ecol. 44, 203-215 (2003).
- Grundmann, G. L. Spatial scales of soil bacterial diversity - The size of a clone. FEMS Microbiol Ecol. 48, 119-127 (2004).
- Langenheder, S., Lindstrom, E. S., Tranvik, L. J. Structure and Function of Bacterial Communities Emerging from Different Sources under Identical Conditions. Appl Environ Microbiol. 72 (1), 212-220 (2006).
- Camp, J. G., Kanther, M., Semova, I., Rawls, J. F. Patterns and Scales in Gastrointestinal Microbial Ecology. Gastroenterology. 136 (6), 1989-2002 (2009).
- Renner, L. D., Weibel, D. B. Physicochemical regulation of biofilm formation. MRS Bull. 36 (5), 347-355 (2011).
- Wessel, A. K., Hmelo, L., Parsek, M. R., Whiteley, M. Going local: technologies for exploring bacterial microenvironments. Nat Rev Microbiol. 11 (5), 337-348 (2013).
- Stacy, A., McNally, L., Darch, S. E., Brown, S. P., Whiteley, M. The biogeography of polymicrobial infection. Nat Rev Microbiol. 14 (2), 93-105 (2015).
- Hansen, R. R., Shubert, K. R., Morrell-Falvey, J. L., Lokitz, B. S., Doktycz, M. J., Retterer, S. T. Microstructured block copolymer surfaces for control of microbe adhesion and aggregation. Biosensors. 4 (1), 63-75 (2014).
- Hansen, R. R., Hinestrosa, J. P., et al. Lectin-functionalized poly(glycidyl methacrylate)- block -poly(vinyldimethyl azlactone) surface scaffolds for high avidity microbial capture. Biomacromolecules. 14 (10), 3742-3748 (2013).
- Timm, C. M., Hansen, R. R., Doktycz, M. J., Retterer, S. T., Pelletier, D. A. Microstencils to generate defined, multi-species patterns of bacteria. Biomicrofluidics. 9 (6), (2015).
- Keymer, J. E., Galajda, P., Muldoon, C., Park, S., Austin, R. H. Bacterial metapopulations in nanofabricated landscapes. Proc Natl Acad Sci USA. 103 (46), 17290-17295 (2006).
- Zhang, Q., Lambert, G., et al. Acceleration of Emergence of Bacterial Antibiotic Resistance in Connected Microenvironments. Science. 333 (6050), 1764-1767 (2011).
- Friedlander, R. S., Vlamakis, H., Kim, P., Khan, M., Kolter, R., Aizenberg, J. Bacterial flagella explore microscale hummocks and hollows to increase adhesion. Proc Natl Acad Sci USA. 110 (14), 5624-5629 (2013).
- Zhou, J., Liu, W., et al. Stochastic Assembly Leads to Alternative Communities with Distinct Functions in a Bioreactor Microbial Community. MBio. 4 (2), 1-8 (2013).
- van Vliet, S., Hol, F. J., Weenink, T., Galajda, P., Keymer, J. E. The effects of chemical interactions and culture history on the colonization of structured habitats by competing bacterial populations. BMC Microbiol. 14 (1), 116 (2014).
- Niepa, T. H. R., Hou, L., et al. Microbial Nanoculture as an Artificial Microniche. Sci Rep. 6, 30578 (2016).
- Hansen, R. H., Timm, A. C., et al. Stochastic Assembly of Bacteria in Microwell Arrays Reveals the Importance of Confinement in Community Development. PLoS ONE. 11 (5), e0155080 (2016).
- Hood, R. D., Singh, P., et al. A Type VI Secretion System of Pseudomonas aeruginosa Targets a Toxin to Bacteria. Cell Host Microbe. 7 (1), 25-37 (2010).
- LeRoux, M., Ja De Leon, ., et al. Quantitative single-cell characterization of bacterial interactions reveals type VI secretion is a double-edged sword. Proc Natl Acad Sci. 109 (48), 19804-19809 (2012).
- Whitney, J. C., Beck, C. M., et al. Genetically distinct pathways guide effector export through the type VI secretion system. Mol Microbiol. 92 (3), 529-542 (2014).
- Warrick, J. W., Timm, A., Swick, A., Yin, J. Tools for Single-Cell Kinetic Analysis of Virus-Host Interactions. PLoS ONE. 11 (1), e0145081 (2016).
- Zwietering, M. H., Jongenburger, I., Rombouts, F. M., Van't Riet, K. Modeling of the Bacterial Growth Curve. Appl Environ Microbiol. 56 (6), 1875-1881 (1990).
- Halsted, M., Wilmoth, J. L., et al. Development of transparent microwell arrays for optical monitoring and dissection of microbial communities. J Vac Sci Technol B Nanotechnol Microelectron. 34 (6), 06KI03 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved