JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
마이크로 웰 어레이 플랫폼 내 미생물 공동체 개발의 구성 및 추적
요약
미생물 군집의 개발은 환경 구조, 구성원의 풍부 성, 형질 및 상호 작용을 포함하는 여러 요소의 조합에 달려있다. 이 프로토콜은 틈새 크기 및 감금과 같은 핵심 요소가 근사 될 수있는 femtoliter 우물에 포함 된 수천 개의 공동체를 동시에 추적하기위한 합성, 미세 제작 환경을 설명합니다.
초록
미생물 군집의 개발은 공동체 구성원의 공간 분포와 활동을 극적으로 바꿀 수있는 복잡한 결정 론적 요인과 확률 론적 요인의 조합에 달려있다. 우리는 수천 개의 세균 공동체를 신속하게 조립하고 추적하는 데 사용할 수있는 마이크로 웰 어레이 플랫폼을 개발했습니다. 이 프로토콜은 플랫폼의 유용성을 강조하고 플랫폼 내의 배열 앙상블 내에서 간단한 2 인 커뮤니티의 개발을 광학적으로 모니터링하기위한 용도를 설명합니다. 이 시연은 유형 VI 분비 병원성을 연구하기 위해 개발 된 일련의 돌연변이 체의 일부인 Pseudomonas aeruginosa 의 두 가지 돌연변이 체를 사용합니다. mCherry 또는 GFP 유전자의 염색체 삽입물은 각 마이크로 웰 내에서 커뮤니티 구성원의 풍부 성과 위치를 모니터링하는 데 사용할 수있는 별개의 방출 파장을 지닌 형광 단백질의 구성 적 발현을 촉진합니다. 이 프로토콜은 상세한 메토d 어레이의 우물에 박테리아의 혼합물을 조립하고 시간 경과에 따른 각 구성원의 상대적 성장을 측정하기 위해 시간 경과 형광 이미징 및 정량적 이미지 분석을 사용합니다. 마이크로 웰 플랫폼의 파종 및 조립, 어레이 내 미생물 군집의 정량 분석에 필요한 이미징 절차 및 모두 논의 된 미생물 종간의 상호 작용을 밝힐 수있는 방법.
서문
미생물 군집은 환경의 구조, 세포 사멸, 분열, 단백질 농도, 세포 소기관 수 및 돌연변이와 관련된 확률 적 과정과 같은 결정 론적 요인에 의해 형성된다. 자연 환경에서 이러한 영향이 지역 사회의 구성과 활동에 미치는 영향을 분석하는 것은 거의 불가능합니다. 자연 구조에 의해 가려지고 화학 및 생물학적 환경에 묻혀 지역 공동체 구성원을 확인하고 자연 환경 내에서 시공간적 분포를 해결하는 것은 매우 어렵습니다. 그럼에도 불구하고 최근의 노력은 지역 사회 기능에 대한 공간 조직의 중요성을 강조하고 진행중인 연구 2 , 3 , 4 에서 회원 수와 조직 모두를 설명 할 필요성을 지적했다.
그것( 예 : 토양 구조, 식물 뿌리, 해양 입자 또는 장 미세 덩어리), 산소의 존재 또는 부재, 그리고 미생물의 도입과 같은 화학적 환경 ( 즉 , 영양소와 이차 대사 산물의 이용 가능성) 병원성 종은 모두 미생물 군집의 구성, 구조 및 기능에 영향을 미친다. 5 , 6 , 7 , 8 , 9 , 10 , 11 . 그럼에도 불구하고 이러한 요소를 포착하는 것을 게을리하는 전통 문화 기술은 계속해서 널리 퍼집니다. 공동체 구성 ( 예 : 공존하는 종의 존재), 물리적 부착, 신호 분자 농도 및 직접 세포 - 세포 접촉은 모두 미생물 군집 형성에 중요한 요소이며 c예방 적 문화 조건. 이러한 특성은 대량 액체 배양 또는 한천 플레이트에서 복제하기가 어렵습니다. 그러나 자연 환경의 주요 물리적 및 화학적 특성을 복제 할 수있는 미세 유체, 미세 패터닝 및 나노 제작 기술을 이용할 수있게 됨으로써 많은 연구자들이 박테리아 공동체를 구축하여 상호 작용을 연구하고 상호 작용을 연구하고 합성 환경을 개발할 수있었습니다. 자연 조건 4 , 15 , 16 , 17 , 18 , 19 , 20을 모방한다.
이 프로토콜은 microwell 배열 장치를 조작하는 방법을 설명하고 th 기능화하는 데 사용할 수있는 자세한 실험 절차를 제공합니다e 웰을 배열하고 박테리아를 단일 종의 식민지와 다중 구성원 공동체로 성장시킬 수 있습니다. 이 연구는 또한 형광 리포터 단백질을 생산하기 위해 변형 된 박테리아가 시간이 지남에 따라 웰 내의 박테리아 성장을 모니터링하는 데 어떻게 사용될 수 있는지를 보여줍니다. 유사한 배열이 이전에 제시되었고 마이크로 웰에서 녹농균 ( Pseudomonas aeruginosa) ( P. aeruginosa) 의 단일 종 콜로니의 성장을 추적하는 것이 가능하다는 것을 보여 주었다. 웰 크기와 파종 밀도를 조절함으로써 초기 접종 조건이 박테리아의 성장 능력에 어떻게 영향을 주는지를 결정하기 위해 수 천 번의 성장 실험의 시작 조건을 병렬로 변화시킬 수 있습니다 21 . 현재 연구는 다중 배열의 동시 비교를 가능하게하고보다 견고한 실험 프로토콜을 사용함으로써 이전 연구를 기반으로하는 마이크로 웰 어레이의 약간 수정 된 버전을 사용합니다. 이 저작물에 사용 된 배열은 여러 개의 하위 배열 또는 배열 ensem을 포함합니다.(직경 2x, 3x 및 4x의 우물 직경)로 배열 된 직경이 15 - 100 μm 인 다양한 크기의 우물을 포함하고 있습니다. 어레이는 실리콘으로 에칭되고, 실리콘 어레이에 시드 된 박테리아의 증식은 매체 주입 아가로 오스 겔로 코팅 된 커버 슬립으로 어레이를 밀봉함으로써 가능해진다. Type Ⅵ 분비 시스템을 연구하기 위해 설계된 P. aeruginosa 돌연변이 체는이 시연에서 사용됩니다.
여기에 제시된 결과는 마이크로 웰 어레이 내에서 다중 구성원 커뮤니티를 분석하는 궁극적 인 목표를 향해 만들어졌으며 연구원은 화학 환경을 제어 및 프로빙하면서 현장 에서 박테리아의 풍부 성과 조직을 모니터링 할 수있었습니다. 이것은 궁극적으로 지역 사회 개발과 승계를 지배하는 "규칙"에 대한 통찰력을 제공해야합니다.
프로토콜
1. 실리콘 마이크로 웰 어레이 제작
- 파릴 렌 코팅
- 시판중인 파릴 렌 코팅 시스템을 사용하여 제조사의 사양 및 지침 (설정 : 기화기 설정 포인트 = 160 ° C, 퍼니스 설정 포인트 = 650 ° C)에 따라 실리콘 웨이퍼에 1-1.5 μm의 파릴 렌 N을 입금하십시오.
참고 : 약 6 g의 파릴 렌 N을 챔버에로드하면 1-1.5 μm 두께의 코팅이 생성됩니다.
- 시판중인 파릴 렌 코팅 시스템을 사용하여 제조사의 사양 및 지침 (설정 : 기화기 설정 포인트 = 160 ° C, 퍼니스 설정 포인트 = 650 ° C)에 따라 실리콘 웨이퍼에 1-1.5 μm의 파릴 렌 N을 입금하십시오.
- 포토 리소그래피
- 접착 촉진제 인 20 % 헥사 메틸 디 실라 잔 (HMDS)과 80 % 프로필렌 글리콜 모노 메틸 에테르 아세테이트 (PGMEA) ( 표 3 참조)를 사용하여 파릴 렌 N- 코팅 웨이퍼를 3,000 rpm으로 45 초 동안 스핀 코팅 하십시오. 접착 촉진제와 함께 2 ML 전송 피펫을 채우고 전체 웨이퍼에 뿌려. 회전시키기 전에 웨이퍼를 약 10 초 동안 그대로 두십시오.
- 양성 - 양성으로 2 mL 이동 피펫을 채 웁니다.ne photoresist ( 표 2 참조) 웨이퍼의 중앙에 포토 레지스트를 분배합니다. 약 1.5 μm 두께의 레지스트 코팅을 생산하기 위해 45 초 동안 3,000 rpm으로 스핀합니다.
- 115 ° C에서 핫 플레이트에 1 분간 샘플을 소프트 베이킹합니다.
- 원하는 패턴으로 콘택트 얼 라이너와 포토 마스크를 사용하여 샘플을 자외선에 노출시킵니다. 스핀 코팅 된 웨이퍼를 패턴 화 된 포토 마스크를 통해 6 초 동안 노출시켜 365 nm에서 측정 한 약 60-80 mJ / cm2의 대략적인 양을 제공합니다.
- 현상액에 샘플을 담그고 (물에 3 % 미만의 수산화 테트라 메틸 암모늄, 표 2 참조) 2 분 동안 패턴을 현상하십시오. 탈 이온수로 헹구고 깨끗하고 건조한 질소로 건조하십시오.
참고 : UV에 노출 된 포토 레지스트 영역은 현상 중에 제거되어야합니다.
- 반응성 이온 에칭
- 노출 된 파릴 렌을 제거하기 위해 산소 플라즈마 에칭을 사용하십시오.실리콘 기판까지.
참고 : 조리법은 파릴 렌의 에칭 속도를 변경하기 위해 변조 될 수 있습니다. 1에서 5 μm 사이의 파릴 렌 두께의 경우 RIE (Reactive Ion Etching) 도구에서 60 mTorr, 20 ° C, 100 sccm O 2 , 10 W RF 및 2,000 W ICP를 사용하십시오. 노출 된 파릴 렌 층을 에칭하고 제거한 후에, 패터닝 된 영역 ( 즉 , 노출 된 실리콘)은 반짝이는은으로 보일 것입니다. - 깊은 RIE (DRIE, 예 : Bosch DRIE) 에칭 공정을 사용하여 실리콘을 에칭합니다.
참고 : 에칭 속도와 지속 시간에 따라 웰 깊이가 결정됩니다. Bosch 공정의 1 사이클 (3 초 증착 단계 : 20 mTorr, 15 ° C, 140 sccm C 4 F 8 , 10 W RF 및 1,750 W ICP 다음에 10 s 에칭 프로세스 : 20 mTorr, 15 ° C , 120sccm SF6, 8WRF, 및 1,750W ICP)는 대략 1㎛의 에칭 깊이에 해당한다. 이 데모에 사용 된 우물은 3 - 3.5 μm 깊이입니다. - V물리적 인 profilometry를 사용하여 에칭 깊이를 분석하십시오.
- 물리적 프로파일 미터에 샘플을로드하십시오 ( 재료 표 참조).
- 샘플 진공을 켜고 수동로드 버튼을 누릅니다.
- "Focus"버튼을 눌러 시스템을 샘플에 초점을 맞 춥니 다. 뷰 화면에 측정을위한 적절한 피쳐를 놓습니다.
- 샘플을 스캔하십시오. 프로파일 레벨을 지정하고 피쳐 깊이를 측정하십시오.
- 에칭 속도를 기록하고 후속 에칭 시간을 조절하여 원하는 깊이를 달성하십시오.
참고 : 측정에는 실리콘 웰의 깊이, 증착 된 파릴 렌의 두께 및 포토 레지스트의 두께가 포함됩니다. 정확한 깊이를 얻기 위해서는 각 층의 두께를 확인하는 것이 필요합니다.
- 노출 된 파릴 렌을 제거하기 위해 산소 플라즈마 에칭을 사용하십시오.실리콘 기판까지.
2. 세균 배양 및 씨앗 뿌리기 ( 그림 1a )
- Luria Broth (LB) 글리세롤 주식의 한천 접시와 2 주 이내에 사용. LB 한천 플레이트에서 원하는 균주의 식민지를 선택하고 P. aeruginosa 의 밤새 배양을 시작 하십시오. R2A 매체에서 220 rpm으로 흔들면서 37 ° C에서 약 18 시간 동안 밤새 문화를 품어.
참고 : 돌연변이 및 형광 리포터 유전자가 유지되도록 도금 후 2 주 이내에 콜로니를 골라야합니다. P. aeruginosa의 모든 작업은 BSL-2 조건 하에서 수행되어야합니다. - 다이아몬드 스크라이브를 사용하여 실리콘 웨이퍼를 다양한 크기와 피치 - 우 배열의 앙상블을 포함하는 개별 칩으로 분할합니다. 각 칩에 연구용 웰 크기와 피치가 모두 포함되어 있는지 확인하십시오.
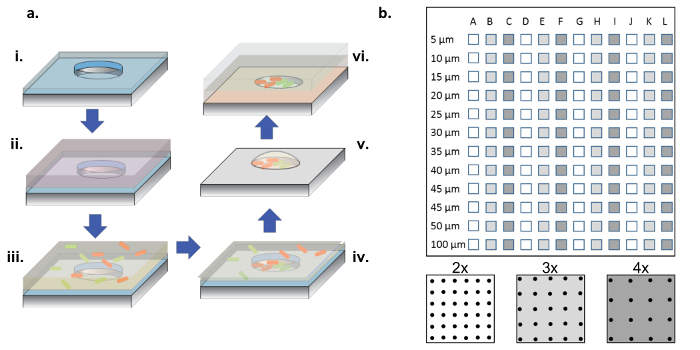
그림 1 : 제작 및 세포 시드 절차. ( a ) 마이크로 웰 어레이 a파릴 렌 (i)의 얇은 층으로 코팅 된 실리콘 웨이퍼로 다시 에칭된다. 웰을 적시거나 표면을 기능화시키기 위해, 단백질 용액을 어레이 (ii) 위에 액체 방울로 첨가한다. 단백질 용액을 제거하고, 웨이퍼를 건조시키고, 원하는 박테리아를 함유하는 새로운 용액을 첨가한다 (iii). 잠복기 후에 박테리아 용액을 제거하고 웨이퍼를 건조시켜 웰과 표면에 박테리아를 남깁니다 (iv). 표면 관련 박테리아는 파릴 리프트 리프트 오프로 제거되어 마이크로 웰에 깨끗하게 파종 된 박테리아를 남기고 웰을 수화 된 상태로 유지하는 데 도움이되는 2 % 글리세롤 매질로 인해 여전히 생존 할 수 있습니다. 실리콘 칩은 다음 microwells에서 박테리아 성장 피드 아가로 오스 겔 코팅 유리 coverslip에 배열 쪽을 아래로 배치됩니다 (vi). ( b ) 단일 실리콘 소자에 서브 어레이의 배치. 각 하위 어레이에는 동일한 우물 세트가 들어 있습니다. 모든 sub-a에 걸친 마이크로 웰의 직경rrays는 5-100 μm의 지름 범위를 가지며 바닥 패널의 회로도에서 흰색에서 어두운 회색까지의 색으로 표시되는 우물 직경 피치의 2 배, 3 배 또는 4 배로 구성됩니다. 우물 깊이가 얕 으면 (<10 μm), 5 ~ 10 μm의 우물 직경은 거의 유용하지 않습니다. 일반적으로이 작은 우물을 식민지로하는 세포가 없기 때문입니다. 이 작업에서는 직경이 15-100 μm 인 우물의 데이터 만 분석했습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
참고 : 그림 1b 에서 볼 수 있듯이 전체 칩에는 직경이 5 ~ 100 μm이고 세 가지 피치 ( 즉, 2x, 3x 및 4x 직경)가 4 번 반복되는 우물의 하위 배열이 포함됩니다.
- PBS 용액에 500 μg / mL Bovine Serum Albumin (BSA)의 150 μL 방울을 놓습니다.어레이 위에 마이크로 웰을 적시십시오. 습도가 높은 챔버에서 실온에서 칩 위에 BSA 용액을 1 시간 동안 인큐베이션하십시오.
- 인산염 완충 식염수 (PBS)로 빈 피펫 팁 상자의 하단을 작성하여 챔버를 만듭니다.
참고 : 특정 렉틴과 같은 다른 물질을 마이크로 웰 표면을 기능화하기 위해 BSA 대신 사용할 수 있습니다.
- 인산염 완충 식염수 (PBS)로 빈 피펫 팁 상자의 하단을 작성하여 챔버를 만듭니다.
- 실리콘 칩을 BSA 솔루션과 함께 incubate하는 동안 5 분 동안 2,500 rpm (평균 950 xg에 해당)에서 배양 한 다음 2 % 글리세롤이 첨가 된 신선한 R2A 배지 500 μL에 resuspend하십시오.
- 600 nm에서 UV-vis 분광계를 사용하여 배양액의 OD를 측정하십시오. 2 % 글리세롤 R2A 배지를 사용하여 0.02의 OD로 조절한다.
참고 : 글리세롤은 파릴 리프트 리프트 오프 동안 우물이 건조되는 것을 방지합니다.
- 600 nm에서 UV-vis 분광계를 사용하여 배양액의 OD를 측정하십시오. 2 % 글리세롤 R2A 배지를 사용하여 0.02의 OD로 조절한다.
- 부화 후 BSA 용액을 제거하고 액체 방울을 제거하고 대체하여 PBS로 3 회 헹구십시오t는 실리콘 마이크로 웰 어레이를 덮고있다. 질소 하에서 건조시킨다.
- 습도가 높은 챔버에 배치 된 건조 어레이 각각에 0.02 OD 배양액 150 μL를 첨가하십시오. 박테리아가 우물 벽에 부착 수 있도록 4 ° C에서 1 시간 품어.
참고 : 배양에는 냉장이 필요하지 않습니다. 4 ℃의 배양 시간은 이미징이 시작되기 전에 박테리아의 성장을 방지하여 성장 이전에 커뮤니티의 공간적 구성을 시각화 할 수 있습니다. 상온 배양을 사용할 수도 있습니다. 두 프로토콜 모두 비슷한 성장 곡선을 나타냅니다.
3. 현미경 셋업
- 실리콘 칩에서 세균 배양을 시작하기 전에 스테이지 상단 환경 제어 챔버 (재료 표 참조)를 켜고 습도 (~ 100 %) 및 온도 (30-32 ℃)가되도록 제어 상자의 설정을 조정하십시오 ° C, 단계 3.2 참조)는 시료를 첨가하기 전에 평형을 이룰 수 있습니다.
- 샘플 홀더의 수평을 맞추고시험관 주변에 PBS가 담긴 실험용 물티슈 ( 표 15 참조)를 사용하여 이슬점으로 챔버의 습도를 높입니다. 이미징 평면에서 응축을 줄이려면 챔버 온도를 30 ° C로, 챔버 뚜껑 온도는 32 ° C로 설정하십시오.
참고 : 슬라이드 홀더는 약 1cm 두께의 개스킷이있는 라이브 셀 챔버에 맞습니다. 샘플 홀더는 샘플 홀더의 상부에 놓인 버블 레벨의 도움으로 수평을 유지합니다. 샘플 홀더는 약간 기울어 질 수 있으며 개스킷에서 수평을 유지하도록 밀봉되어 있습니다. - 배양액이 실리콘 칩 위에서 배양되는 동안 이미징 전 적어도 30 분 동안 수은 램프의 전원 스위치를 수동으로 켜십시오. 수동으로 카메라와 자동 현미경 스테이지를 켭니다. 현미경 및 주변 장치를 제어하고 소프트웨어가 소프트웨어를 인식하는지 확인하는 데 사용되는 소프트웨어를 엽니 다.
참고 : NA = 0.3 인 경우 배율은 10X입니다.
- 마이크로 웨이브는 액체 상태에 도달 할 때까지 약 60 초 동안 미리 아가 로스 용액 ( 즉 , R2A 배지에서 2 % 아가로 오스)을 준비했습니다.
- 에탄올로 75mm x 22mm, # 1.5 유리 커버 슬립의 뒷면을 적시고 2x3 "(50x75mm) 유리 슬라이드에 세로로 가운데 놓습니다 .PDMS 스페이서 (두께 1mm 이하) coverslip의 긴 가장자리를 따라하고 coverslip의 약 1mm가 슬라이드의 가장자리를 돌출되도록 유리 coverslip을 이동합니다.
- 커버 유리와 슬라이드 사이에 액체 한천을 "샌드위치"어셈블리의 상단에 두 번째 2 X 3 "유리 슬라이드를 배치하고, 그것을 커버하고, 충분히 커버 유리 coverslip 상단에 액체 아가로 오스 솔루션 5 ML을 붓는다.
참고 : 이것은 아가로 오스의 깊이를 조절하여 PDMS 스페이서와 동일한 두께로 커버 슬립과 경화 된 아가로 오스의 전체 두께를 만듭니다. - 유리 슬라이드 coverslip - 아가로 오스 겔 유리 슬라이드 샌드위치가 아가로 오스 솔루션이 응고하기 시작할 때까지 설정할 수 있습니다; 그런 다음 냉장고에 옮기십시오. 15 분 후, 과도한 고체 아가로 오스를 제거하고 유리 coverslip 주위를 잘라. 이것을 깨끗한 접시에 옮기고 사용할 때까지 냉장고에 보관하십시오.
5. 아가로 오스 코팅 Coverslip 및 이미지로 우물을 밀봉
- 박테리아 잠복기가 완료되면, 다음과 같이 냉장고에서 아가로 오스 코팅 coverslip을 제거하고 실리콘 칩을 준비합니다.
- 초순수에 실리콘 칩을 한 번에 하나씩 10 초씩 담근다. 과도한 액체가 칩의 가장자리에서 흘러 나올 때까지 실험실 와이퍼 나 티슈에 가장자리를 대십시오.
- 각 실리콘 칩의 가장자리 길이에 맞게 테이프 조각을 자릅니다. 실리콘을 덮고있는 파릴 렌 (parylene) 위에 테이프를 놓고 파릴 렌 코팅을 빠르게 벗겨냅니다.
- Immed순차적으로 각 벗겨 칩을 반전하고 microwell - 배열 측면이 아가로 오스 - 코팅 coverslip의 아가로 오스 - 코팅 측면 (직면하고 접촉) 있도록 각 칩을 놓습니다. 우물 외부의 박테리아의 성장을 막기 위해 아가로 오스에 닿은 후에 칩을 움직이거나 이동하지 않도록주의하십시오.
- 조립 된 마이크로 웰 어레이 / 아가로 오스 coverslip을 miscroscope의 stage-top 환경 제어 챔버의 슬라이드 홀더에 놓습니다.
- 주위 조명이나 지시등 ( 예 : 손전등)을 사용하여 원하는 어레이를 찾으십시오. 자동화 된 스테이지를 제어하는 상용 소프트웨어를 사용하여 위치를 저장하십시오 (재료 표 참조). 위치를 저장 한 후 주위 또는 지시등을 끕니다.
- 상용 소프트웨어에서 "ND Acquisition"패널을 엽니 다.
참고 :이 패널에는 프로그램 가능한 이미지 수집뿐만 아니라 특정 디렉토리에 자동 저장하기위한 메뉴가 포함되어 있습니다. 이 실험의 경우, "시간", "XY"및 "λ"메뉴가 사용됩니다. - 소프트웨어에서 위치를 저장하려면 "XY 메뉴"를 클릭 한 다음 저장해야하는 각 위치의 왼쪽에있는 빈 상자를 선택하십시오. 또한 "Z 포함"버튼을 클릭하십시오.
- 적절한 형광 필터 큐브를 사용하여 원하는 파장 및 10 배율로 시간 경과에 따라 이미지를 수집합니다 (재료 표 참조).
- 제어 소프트웨어와 저장된 어레이 위치를 사용하여 저장된 각 위치로 이동하고 우물에 집중하십시오. 저장된 목록에서 각 XY 위치를 클릭하고 녹색 형광 단백질 (GFP) 필터를 사용하여 초점을 조정하십시오. z 위치를 가리키는 화살표를 클릭하여 새로운 z 위치를 저장하십시오.
참고 :이 프로세스는 시간이 오래 걸릴 수 있습니다. 게인을 높이는 예방 조치를 취하고 광 침지를 방지하기 위해 광도를 줄이기 위해 중립 밀도 필터를 사용하는 것을 고려하십시오. - 배열의 표면에 초점을 맞출 때 z 축 위치의 차이를 지적하여 각 파장에 대한 초점 평면 간의 z 축 거리를 결정합니다. 혼합 된 적색 / 녹색 박테리아 개체군으로 배열에서 2-3 위치를 선택하고 RFP (Red Fluorescence Protein) 필터를 사용하여 초점을 맞 춥니 다.
- GFP 및 RFP 형광 필터를 사용하여 초점 평면 사이의 거리를 빼고 "λ"메뉴 아래에 초점 평면 조정을 추가하십시오.
참고 : 예를 들어, 어레이가 50μm의 z 위치에서 GFP 채널에 초점이 맞춰져 있고 동일한 어레이가 55μm의 RFP 채널에 초점을 맞추고있는 것처럼 보일 경우 RFP 광학 구성 옆에 +5를 "λ "메뉴.
- GFP 및 RFP 형광 필터를 사용하여 초점 평면 사이의 거리를 빼고 "λ"메뉴 아래에 초점 평면 조정을 추가하십시오.
- 시간 경과 이미지 수집을 시작하십시오.
참고 : 여기에 표시된 실험의 경우 RFP 및 GFP 이미지를 제어하는 상용 소프트웨어를 통해 다차원 이미지 수집을 사용하여 30 분 간격으로 모든 어레이 위치에 대해 수집했습니다.카메라, 셔터, 필터 휠 및 전동 스테이지가 있습니다.- '시간'메뉴에서 '간격'을 30 분으로, '실험 기간'을 24 시간으로 설정하십시오. "지금 실행"을 클릭하십시오.
참고 : "시간", "XY"및 "λ"상자를 선택한 상태에서 프로그램을 실행하면 스테이지가 각 위치 ( 예 : 저장된 XYZ 위치)로 이동하고, 한 파장으로 이미지를 촬영하고, z 위치로 이동합니다 초점 평면 차이 ( 즉, 람다 또는 파장 제어)를 설명하고 두 번째 이미지를 가져 와서 다음 배열 위치 (다 지점)로 이동 한 다음 30 분 간격으로이를 반복합니다 (시간 경과).
- '시간'메뉴에서 '간격'을 30 분으로, '실험 기간'을 24 시간으로 설정하십시오. "지금 실행"을 클릭하십시오.
- 제어 소프트웨어와 저장된 어레이 위치를 사용하여 저장된 각 위치로 이동하고 우물에 집중하십시오. 저장된 목록에서 각 XY 위치를 클릭하고 녹색 형광 단백질 (GFP) 필터를 사용하여 초점을 조정하십시오. z 위치를 가리키는 화살표를 클릭하여 새로운 z 위치를 저장하십시오.
- 조명 제어 이미지를 얻습니다.
참고 : "ND Acquisition", "Time"및 "XY"메뉴를 사용하여 각각 25x 4 위치의 이미지를 찍습니다.- 모든 광원을 끄고 촬영하여 100 개의 "darkfield"이미지를 연속으로 촬영하십시오.표준 슬라이드의 "이미지". 이 이미지는 카메라의 노이즈를 캡처합니다. 시간 경과 동안 사용 된 가장 긴 노출 시간을 사용하십시오 (5.3.3 절).
- 주어진 실험 조건에서 고르지 않은 조명을 포착하기 위해 표준 슬라이드 ( 즉 균일 RFP 또는 GFP 강도)를 몇 개의 다른 위치에서 이미징하여 100 개의 "조명 필드"이미지를 가져옵니다. 채도에 도달하지 않고 신호를 최대화하는 노출 시간을 선택하십시오.
6. 분석
- 이미지 분석 소프트웨어 ( 예 : ImageJ)를 사용하여 이미지 스택을 처리합니다.
- 상용 소프트웨어를 사용하여 획득 한 이미지를 tiff 파일 형식으로 변환하십시오. "파일"> "가져 오기"> "이미지 시퀀스"를 클릭하여 이미지 분석 소프트웨어에 이미지를 업로드하십시오.
- 모든 "darkfield"및 "illumination field"이미지를 평균화하여 "Correction Image"를 만듭니다. 서브 트랙"프로세스"> "이미지 계산기"를 선택하여 평균 "조명 필드"이미지의 평균 "암실"이미지를 만듭니다. "Image1"과 "Image2"두 이미지를 선택한 다음 "Operation"필드에서 "Subtract"를 선택하십시오. "확인"을 클릭하십시오.
- 평균화하려면 보정 (또는 암실 이미지) 이미지를로드하고 '이미지'> '스택'> 'Z 프로젝트'> '평균 투영'을 클릭하십시오.
- 필요한 경우 이미지 등록을 수행하십시오. 그런 다음 "처리"> "배경 빼기"를 클릭하여 배경 빼기를 수행하십시오. "반경"필드에 반경 ( 예 : 125)을 입력하고 "슬라이딩 포물선"을 선택하십시오.
- "프로세스"> "계산기 플러스"를 사용하여 조명 보정을 수행하십시오. 다음 매개 변수를 선택하십시오 : operation, divide; i1, 우물 이미지; i2, 정정 화상; k1, 보정 영상 평균; 및 k2, 0. "Cre새로운 창을 먹었다. "
참고 :이 데이터 세트에는 등록이 필요하지 않지만 다른 작업에서는 ImageJ Plugin StackReg가 "변환"변환과 함께 사용되었습니다. 배경 빼기의 경우 모든 이미지 세트에 동일한 슬라이딩 포물선 반경을 사용하십시오. 예를 들어, 이미지화 된 가장 큰 웰의 픽셀 반경이 100 인 경우 모든 이미지 세트에 대해 100보다 큰 반경 ( 예 : 125)을 사용하십시오.
- 마이크로 웰에서 각 균주의 성장을 결정하십시오.
- ImageJ "MicroArray"플러그인을 사용하여 원하는 어레이의 각 마이크로 웰 주변의 관심 영역 (ROI)을 선택하십시오.
- "MAP"메뉴에서 "Reset Grid"를 클릭하십시오. 행, 열 및 직경을 지정하십시오 (어레이의 우물 크기 및 수에 따라, 그림 1b 참조). "ROI 모양"메뉴에서 "원"을 선택하십시오.
- "Alt"키를 누른 채로 마우스 왼쪽 상단 ROI를 선택하여 t를 움직입니다.그는 ROI 배열. "shift"키를 누른 채로 왼쪽 하단 ROI를 선택하여 배열의 크기를 변경하십시오. 배열의 오른쪽에서 ROI를 선택하는 동안 "Shift"키를 누르고 ROI의 간격을 변경하려면 모서리가 아닌 ROI를 선택하십시오.
- 위의 명령을 사용하여 이미지의 우물 위에 ROI 어레이를 맞 춥니 다. '측정 RT'를 클릭하십시오.
참고 : 플러그인은 각 ROI에서 원하는 측정 값을 내 보냅니다. 3 개의 ROI 크기를 사용하여 우물 주위에 동심원 고리를 만들어 백그라운드 신호 ( 즉, 외부 링에서 중간 링을 뺀 신호)와 형광 측정 ( 즉, 내부 링으로부터의 신호)을 수집합니다.
- 스프레드 시트 소프트웨어에서 데이터를 수집하고 백그라운드 신호를 계산하십시오. 추가 분석을 위해 사용자 정의 스크립팅 소프트웨어로 가져옵니다.
- ImageJ "MicroArray"플러그인을 사용하여 원하는 어레이의 각 마이크로 웰 주변의 관심 영역 (ROI)을 선택하십시오.
- 데이터 구성 및 분석
- 데이터 가져 오기 및 데이터 구성모든 시간 동안 ImageJ에서 다음 순서로 행렬로 수집됩니다 : 열 1, 하위 배열 번호; 칼럼 2, 웰 행; 컬럼 3, 웰 컬럼; 열 4, 평균 강도; 열 5, 배경 강도; 6 열, 평균 강도 - 배경 강도.
- mCherry 및 GFP 획득의 결과를 다른 행렬로 분리하십시오. 각 하위 배열 및 각 색상의 결과를 셀 배열의 다른 셀에 저장합니다.
참고 :이 조직을 사용하면 이미지 데이터와 측정 결과간에 쉽게 이동하고 데이터를 정리하고 측정 값이 데이터를 정확하게 나타낼 수 있습니다.
- mCherry 및 GFP 획득의 결과를 다른 행렬로 분리하십시오. 각 하위 배열 및 각 색상의 결과를 셀 배열의 다른 셀에 저장합니다.
- P. aeruginosa의자 가 형광을 조정합니다.
참고 : GFP와 mCherry 균주의 공동 배양과 관련된 실험에서, mCherry만의 칩을 분석하여 mCherry와 녹색 자기 형광 사이의 관계를 확인해야합니다.- 모든 mCherry로부터 mCherry 대 GFP 신호를 플롯합니다. ΔretS &# 916; tse / i1-6 웰을 사용하여 mCherry 신호와 GFP 채널의자가 형광 사이의 관계를 결정합니다. 공동 문화에서 autofluorescence 신호를 뺍니다.
- 궤적을 플롯하고 수정 된 물류 방정식을 각 궤적에 맞추어 스프레드 시트 소프트웨어 또는 사용자 정의 스크립팅 소프트웨어에서 최소 제곱을 사용하여 매개 변수를 추출합니다.
- GFP와 mCherry 궤도 매개 변수 사이의 상관 관계를 찾습니다.
- 데이터 가져 오기 및 데이터 구성모든 시간 동안 ImageJ에서 다음 순서로 행렬로 수집됩니다 : 열 1, 하위 배열 번호; 칼럼 2, 웰 행; 컬럼 3, 웰 컬럼; 열 4, 평균 강도; 열 5, 배경 강도; 6 열, 평균 강도 - 배경 강도.
결과
여기에 제시된 실험 플랫폼은 박테리아 공동체에 대한 높은 처리량과 높은 함량의 연구를 위해 고안되었습니다. 이 설계로 인해 다양한 크기의 우물에서 자라는 수천 개의 공동체를 동시에 분석 할 수 있습니다. 이 마이크로 웰 어레이 디자인을 사용하면 초기 종자 밀도, 우물 크기 및 화학적 환경에 대한 최종 커뮤니티 구성의 의존성을 결정할 수 있습니다. 이 작업은 마...
토론
이 기사에서는 박테리아 공동체 개발에 대한 높은 처리량 및 고감도 생체 이미징 기반 분석을 가능하게하도록 설계된 마이크로 웰 어레이 장치 및 실험 프로토콜을 발표했습니다. 여기 시위의 초점은 지역 사회 개발에 접촉 매개 형 VI 형 분비의 효과를 연구하는 것이었지만, 배열은 유연하고 다양한 미생물 군집 및 미생물 - 미생물 상호 작용에 대한 연구를 수용하도록 설계되었습니다. 여기에서...
공개
저자는 공개 할 것이 없습니다.
감사의 말
Microwell 배열은 미국 에너지 부 기본 에너지 과학 사무실의 Nanophase Materials Sciences 사용자 시설 부문 센터에서 제조 및 특성 분석되었습니다. 이 연구에 대한 재정 지원은 Oak Ridge 국립 연구소의 연구 개발 기금을 통해 제공되었습니다. 저자들은 또한이 연구에 사용 된 P. aeruginosa 균주의 공급에 대해 J. Mougous Laboratory (Washington, Seattle, WA)에 감사 드리고자 합니다.
자료
| Name | Company | Catalog Number | Comments |
| Parylene N | Specialty Coating Systems | CAS NO.:1633-22-3 | |
| Parylene coater | Specialty Coating Systems | Labcoter 2 Parylene Deposition Unit PDS2010 | |
| Silicon Wafer | WRS Materials | 100mm diameter, 500-550μm thickness, Prime, 10-20 resistivity, N/Phos<100>, | |
| adhesion promoter | Shin-Etsu Microsci | MicroPrime P20 adhesion promoter | |
| postive tone photoresist | Rohm and Haas Electronics Materials LLC (Owned by Dow) | Microposit S1818 Positive Photoresist (code 10018357) | |
| Quintel Contact Aligner | Neutronix Quintel Corp | NXQ 7500 Mask Aligner | |
| Reactive Ion Etching Tool | Oxford Instruments | Plasmalab System 100 Reactive Ion Etcher | |
| R2A Broth | TEKnova | R0005 | |
| Bovine Serum Albumin | Sigma | A9647 | |
| Multimode Plate Reader | Perkin Elmer | Enspire, 2300-0000 | |
| Fluorescent Microscope | Nikon | Eclipse Ti-U | |
| Automated Stage | Prior | ProScan III | |
| CCD camera | Nikon | DS-QiMc | |
| Stage-top environmental control chamber | In Vivo Scientific | STEV ECU-HOC | |
| Phosphate Buffered Saline | ThermoFisher Scientific | 14190144 | |
| UltraPure Agarose | ThermoFisher Scientific | 16500500 | |
| 25 x 75 mm No. 1.5 coverslip | Nexterion | High performance #1.5H coverslips | |
| Fluorescence Reference Slides | Ted Pella | 2273 | |
| Physical Stylus Profilometer | KLA Tencor | P-6 | |
| lab wipes | Kimberly Clark | Kimipe KIMTECH SCIENCE Brand, 34155 | |
| commercial software | Nikon | NIS Elements | |
| Zeiss 710 Confocal Microscope | Zeiss | ||
| filter cubes | Nikon | Nikon FITC (96311), Nikon Texas Red(96313) |
참고문헌
- Zhou, J., Deng, Y., et al. Stochasticity, succession, and environmental perturbations in a fluidic ecosystem. Proc Natl Acad Sci. 111, E836-E845 (2014).
- Valm, A. M., Welch, J. L. M., et al. Systems-level analysis of microbial community organization through combinatorial labeling and spectral imaging. Proc Natl Acad Sci USA. 108 (10), 4152-4157 (2011).
- Satoh, H., Miura, Y., Tsushima, I., Okabe, S. Layered structure of bacterial and archaeal communities and their in situ activities in anaerobic granules. Appl Environ Microbiol. 73 (22), 7300-7307 (2007).
- Kim, H. J., Boedicker, J. Q., Choi, J. W., Ismagilov, R. F. Defined spatial structure stabilizes a synthetic multispecies bacterial community. Proc Natl Acad Sci USA. 105 (47), 18188-18193 (2008).
- Nunan, N., Wu, K., Young, I. M., Crawford, J. W., Ritz, K. Spatial distribution of bacterial communities and their relationships with the micro-architecture of soil. FEMS Microbiol Ecol. 44, 203-215 (2003).
- Grundmann, G. L. Spatial scales of soil bacterial diversity - The size of a clone. FEMS Microbiol Ecol. 48, 119-127 (2004).
- Langenheder, S., Lindstrom, E. S., Tranvik, L. J. Structure and Function of Bacterial Communities Emerging from Different Sources under Identical Conditions. Appl Environ Microbiol. 72 (1), 212-220 (2006).
- Camp, J. G., Kanther, M., Semova, I., Rawls, J. F. Patterns and Scales in Gastrointestinal Microbial Ecology. Gastroenterology. 136 (6), 1989-2002 (2009).
- Renner, L. D., Weibel, D. B. Physicochemical regulation of biofilm formation. MRS Bull. 36 (5), 347-355 (2011).
- Wessel, A. K., Hmelo, L., Parsek, M. R., Whiteley, M. Going local: technologies for exploring bacterial microenvironments. Nat Rev Microbiol. 11 (5), 337-348 (2013).
- Stacy, A., McNally, L., Darch, S. E., Brown, S. P., Whiteley, M. The biogeography of polymicrobial infection. Nat Rev Microbiol. 14 (2), 93-105 (2015).
- Hansen, R. R., Shubert, K. R., Morrell-Falvey, J. L., Lokitz, B. S., Doktycz, M. J., Retterer, S. T. Microstructured block copolymer surfaces for control of microbe adhesion and aggregation. Biosensors. 4 (1), 63-75 (2014).
- Hansen, R. R., Hinestrosa, J. P., et al. Lectin-functionalized poly(glycidyl methacrylate)- block -poly(vinyldimethyl azlactone) surface scaffolds for high avidity microbial capture. Biomacromolecules. 14 (10), 3742-3748 (2013).
- Timm, C. M., Hansen, R. R., Doktycz, M. J., Retterer, S. T., Pelletier, D. A. Microstencils to generate defined, multi-species patterns of bacteria. Biomicrofluidics. 9 (6), (2015).
- Keymer, J. E., Galajda, P., Muldoon, C., Park, S., Austin, R. H. Bacterial metapopulations in nanofabricated landscapes. Proc Natl Acad Sci USA. 103 (46), 17290-17295 (2006).
- Zhang, Q., Lambert, G., et al. Acceleration of Emergence of Bacterial Antibiotic Resistance in Connected Microenvironments. Science. 333 (6050), 1764-1767 (2011).
- Friedlander, R. S., Vlamakis, H., Kim, P., Khan, M., Kolter, R., Aizenberg, J. Bacterial flagella explore microscale hummocks and hollows to increase adhesion. Proc Natl Acad Sci USA. 110 (14), 5624-5629 (2013).
- Zhou, J., Liu, W., et al. Stochastic Assembly Leads to Alternative Communities with Distinct Functions in a Bioreactor Microbial Community. MBio. 4 (2), 1-8 (2013).
- van Vliet, S., Hol, F. J., Weenink, T., Galajda, P., Keymer, J. E. The effects of chemical interactions and culture history on the colonization of structured habitats by competing bacterial populations. BMC Microbiol. 14 (1), 116 (2014).
- Niepa, T. H. R., Hou, L., et al. Microbial Nanoculture as an Artificial Microniche. Sci Rep. 6, 30578 (2016).
- Hansen, R. H., Timm, A. C., et al. Stochastic Assembly of Bacteria in Microwell Arrays Reveals the Importance of Confinement in Community Development. PLoS ONE. 11 (5), e0155080 (2016).
- Hood, R. D., Singh, P., et al. A Type VI Secretion System of Pseudomonas aeruginosa Targets a Toxin to Bacteria. Cell Host Microbe. 7 (1), 25-37 (2010).
- LeRoux, M., Ja De Leon, ., et al. Quantitative single-cell characterization of bacterial interactions reveals type VI secretion is a double-edged sword. Proc Natl Acad Sci. 109 (48), 19804-19809 (2012).
- Whitney, J. C., Beck, C. M., et al. Genetically distinct pathways guide effector export through the type VI secretion system. Mol Microbiol. 92 (3), 529-542 (2014).
- Warrick, J. W., Timm, A., Swick, A., Yin, J. Tools for Single-Cell Kinetic Analysis of Virus-Host Interactions. PLoS ONE. 11 (1), e0145081 (2016).
- Zwietering, M. H., Jongenburger, I., Rombouts, F. M., Van't Riet, K. Modeling of the Bacterial Growth Curve. Appl Environ Microbiol. 56 (6), 1875-1881 (1990).
- Halsted, M., Wilmoth, J. L., et al. Development of transparent microwell arrays for optical monitoring and dissection of microbial communities. J Vac Sci Technol B Nanotechnol Microelectron. 34 (6), 06KI03 (2016).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유