Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Bir Mikro Kuyruklu Dizi Platformunda Mikrobik Topluluk Geliştirmenin Montajı ve Takibi
Bu Makalede
Özet
Mikrobiyal toplulukların gelişimi, çevresel mimari, üye bolluğu, özellikleri ve etkileşimleri de içeren faktörlerin bir kombinasyonuna bağlıdır. Bu protokol, femtolitör kuyularda bulunan binlerce topluluğun eşzamanlı olarak izlenebilmesi için sentetik, mikrofabrik bir ortam tanımlamakta olup niş boyut ve sınırlama gibi anahtar faktörler yaklaşık olarak belirlenebilmektedir.
Özet
Mikrobiyal toplulukların gelişimi, topluluk üyelerinin mekansal dağılımını ve faaliyetlerini önemli ölçüde değiştirebilen karmaşık deterministik ve stokastik faktörlerin bir kombinasyonuna bağlıdır. Binlerce bakteri topluluğunu paralel olarak hızlı bir şekilde bir araya getirmek ve izlemek için kullanılabilecek bir mikro kollu dizi platformu geliştirdik. Bu protokol, platformun kullanımını vurgular ve platform içindeki dizilerin bir topluluğunda basit, iki üyeli toplulukların gelişimini optik olarak izlemek için kullanımını açıklar. Bu gösteri, Tip VI salgı patojenisitesini incelemek üzere geliştirilen bir dizi mutantın bir parçası olan Pseudomonas aeruginosa'nın iki mutantını kullanıyor. MCherry veya GFP genlerinin kromozomal girdileri, her bir mikrowell içindeki topluluk üyelerinin bolluğunu ve konumunu izlemek için kullanılabilen farklı emisyon dalga boylarına sahip floresan proteinlerin yapılı ifadesini kolaylaştırır. Bu protokol ayrıntılı bir metodu açıklamaktadır.D, bakteri karışımlarını dizinin kuyularına monte etmek ve zaman içindeki tüm üye popülasyonun göreli büyümesini ölçmek için zaman atlamalı floresans görüntüleme ve niceliksel görüntü analizi kullanmak için kullanılmıştır. Mikro platformun tohumlanması ve montajı, dizi içindeki mikrobik toplulukların kantitatif analizi için gerekli görüntüleme prosedürleri ve mikrobik tür alanları arasındaki etkileşimleri ortaya çıkarmak için kullanılabilecek yöntemlerin hepsi tartışılmıştır.
Giriş
Mikrobiyal topluluklar, hem hücre ölümü, bölünme, protein konsantrasyonu, organel sayısı ve mutasyon 1 ile ilişkili olan ortamın yapısı ve stokastik süreçler gibi belirleyici faktörler tarafından şekillendirilir 1 . Doğal ortamda, bu etkilerin toplumdaki bileşimi ve etkinliği üzerindeki bireysel etkisini çözümlemek neredeyse imkansız olabilir. Doğal yapılar tarafından örtülmüş ve kimyasal ve biyolojik bir ortamda gömülmüş, topluluk üyelerinin tanımlanması ve doğal çevre içerisinde zaman-zaman dağılımının daha da çözülmesi son derece zorlu bir konudur. Bununla birlikte, son zamanlardaki çabalar, mekânsal organizasyonun topluluk işlevi üzerindeki önemini vurgulamış ve devam eden çalışmalarda hem üye bolluğunu hem de örgütlenmeyi hesaba katma ihtiyacına işaret etmektedir 2 , 3 , 4 .
O, Yerel kimyasal çevrenin (besin maddelerinin ve sekonder metabolitlerin mevcudiyeti), fiziksel yapının ( örneğin toprak yapısı, bitki kökleri, okyanus parçacıkları veya bağırsak mikrovillikleri), oksijenin varlığı veya yokluğunun ve Patojenik türlerin hepsi mikrobiyal toplulukların kompozisyonu, mimarisi ve fonksiyonunu etkiler 5 , 6 , 7 , 8 , 9 , 10 , 11 . Bununla birlikte, kültürler için bu faktörleri yakalamayı ihmal eden geleneksel teknikler hakim olmaya devam etmektedir. Topluluk bileşimi ( örneğin birlikte bağımlı türlerin varlığı), fiziksel bağlanma, sinyal molekül konsantrasyonu ve direkt hücre-hücre teması, mikrobiyal bir topluluğun şekillenmesi için önemli faktörlerdir ve cÖnceki kültür koşulları. Bu özelliklerin bir toplu sıvı kültüründe veya bir agar plakasında kopyalanması zordur. Bununla birlikte, doğal ortamların önemli fiziksel ve kimyasal özelliklerinin çoğaltılmasına izin veren mikroakışkan, mikro-modelleme ve nanofabrikasyon teknikleri, birçok araştırmacıya, 12 , 13 , 14 arasındaki etkileşimleri incelemek için bakteriyel topluluklar inşa ettirdi ve Doğal koşulları 4 , 15 , 16 , 17 , 18 , 19 , 20'yi taklit edin.
Bu protokol, bir mikrodellek dizilimi cihazı imal etmek için bir yöntem açıklamaktadır ve bu yöntemin işlevselleştirilmesi için kullanılabilecek ayrıntılı deneysel prosedürler sağlamaktadır.Hem kuyuları hem de tek tür kolonileri olarak ve çok üyeli topluluklarda bakteri yetiştirmek için kullanıldı. Bu çalışma aynı zamanda, floresan muhabir proteinleri üretmek üzere modifiye edilen bakterilerin, zaman içinde kuyu içerisindeki bakterilerin büyümesini izlemek için nasıl kullanılabileceğini göstermektedir. Benzer bir dizi daha önce sunulmuş ve mikrowelllerde Pseudomonas aeruginosa ( P. aeruginosa) tek tür kolonilerin büyümesini izlemek mümkün olduğunu göstermiştir. Kuyu boyutu ve ekim yoğunluğunu modüle ederek, binlerce büyüme deneyinin başlangıç koşulları, ilk inokülasyon koşullarının bakterilerin yetişme yeteneğini nasıl etkilediğini belirlemek için paralel olarak değiştirilebilir. Mevcut çalışma, çoklu dizilerin eşzamanlı karşılaştırılmasını sağlayarak ve daha sağlam bir deney protokolü kullanarak önceki çalışmaya dayanan mikrodevre dizisinin biraz değiştirilmiş bir sürümünü kullanıyor. Bu çalışmada kullanılan dizi, çoklu alt sıralar veya dizi ensem içerirÇapları 15 - 100 μm arasında değişen, üç farklı aralıklarla ( yani , kuyu çapında 2x, 3x ve 4x) düzenlenmiş farklı boyutlarda oyuklar içeren oyuklar. Diziler silikona kazınır ve silikon dizilerde tohumlanan bakterilerin büyümesi, orta-enfüze bir agaroz jeli ile kaplanmış bir lamel ile dizileri sızdırmaz hale getirerek etkinleştirilir. Bu demonstrasyonda Tip VI sekresyon sistemini incelemek üzere tasarlanmış P. aeruginosa mutantları kullanılır.
Burada sunulan sonuçlar, mikrowell dizilerdeki çok elemanlı toplulukların analizinin nihai hedefine doğru inşa ederek, araştırmacıların kimyasal çevrenin kontrol edilmesi ve araştırılması sırasında yerinde bakterilerin bolluğunu ve organizasyonunu izlemesini sağlıyor. Bu, nihayetinde topluluk gelişimini ve halefiyetini yöneten "kurallar" hakkında fikir verir.
Protokol
1. Silicon Microwell-array Fabrikasyon
- Parylene kaplama
- Üreticinin spesifikasyonlarına ve talimatlarına (ayar: buharlaştırıcı set değeri = 160 ° C; fırın ayar noktası = 650 ° C) göre ticari olarak mevcut bir parylene kaplama sistemi kullanılarak silisyum levhalar üzerine 1-1.5 um parylene N arasında birikim yapın.
NOT: Bir odaya yüklenen yaklaşık 6 gr parylene N, 1-1.5 μm kalınlığında kaplamalar üretir.
- Üreticinin spesifikasyonlarına ve talimatlarına (ayar: buharlaştırıcı set değeri = 160 ° C; fırın ayar noktası = 650 ° C) göre ticari olarak mevcut bir parylene kaplama sistemi kullanılarak silisyum levhalar üzerine 1-1.5 um parylene N arasında birikim yapın.
- Fotolitografi
- Parylene N kaplı gofretleri 45 s süreyle 3.000 rpm'de yapışma arttırıcı,% 20 heksametildisilazan (HMDS) ve% 80 propilen glikol monometil eter asetat (PGMEA) (bkz . Malzeme Tablosu) ile spin-kaplama . Yapışma arttırıcı ile 2 mL transfer pipet doldurun ve tüm gofret üzerine serpiştirin. Kurutmadan önce gofretin yaklaşık 10 saniye beklemesine izin verin.
- Pozitif geçişli 2 mL transfer pipetini doldurun(Malzeme Tablosuna bakınız) ve fotorezisti gofretin ortasına dağıtıyoruz. Yaklaşık 1.5 um kalınlığında bir rezistans kaplaması elde etmek için 45 saniye boyunca 3.000 devirde döndürün.
- Numuneleri 115 ° C'de 1 dakika boyunca bir ısı plakası üzerine yumuşak fırında pişirin.
- Numuneyi mor ötesi ışığa maruz bırakmak için istenen kuyu modeline sahip bir kontak hizalayıcı ve fotokask kullanın. Spin kaplı gofreti, 365 nm'de ölçülen yaklaşık 60-80 mJ / cm2'lik bir doz vererek 6 saniye süreyle desenli fotomaskdan maruz bırakın.
- Numuneyi, geliştirici içinde suya batırarak desende (suda <% 3 tetrametil amonyum hidroksit; malzeme tablolarına bakınız ) 2 dakika daldırarak deseni geliştirin . DI su ile durulayın ve temiz, kuru azot ile kurutun.
NOT: UV'ye maruz bırakılan fotorezistlerin alanları geliştirme sırasında temizlenmelidir.
- Reaktif iyon aşındırma
- Maruz kalmış parylene'yi çıkarmak için bir oksijen plazma aşındırma kullanınSilikon alt tabakaya kadar.
NOT: Tarif, parylene'nin etit hızını değiştirmek için modüle edilebilir. 1 ila 5 μm arasındaki parylene kalınlıkları için, Reaktif İyon Etching (RIE) aleti üzerinde 60 mTorr, 20 ° C, 100 sccm O2, 10 W RF ve 2.000 W ICP içeren bir reçete kullanın. Aşındırma ve parylene katmanını çıkardıktan sonra desenli alan ( yani maruz kalmış silikon) parlak ve gümüş görünmelidir. - Silisyum içine girmek için derin bir RIE (DRIE, örneğin Bosch DRIE) aşındırma işlemi kullanın.
NOT: Etki hızı ve süresi iyi derinliği belirleyecektir. Bosch prosesinin bir tam devri (3 s çöktürme adımı: 20 mTorr, 15 ° C, 140 sccm C4F8, 10 W RF ve 1,750 W ICP ardından 10 saniyelik bir aşındırma işlemi: 20 mTorr, 15 ° C , 120 sccm SF 6 , 8 W RF ve 1,750 W ICP) yaklaşık 1 um aşındırma derinliğine karşılık gelir. Bu gösteri sırasında kullanılan kuyular 3 - 3,5 μm derinliğe kadar değişir. - VFiziksel profilometri yardımıyla kazınmış derinliği düzeltin.
- Numuneyi fiziksel bir profilometreye yükleyin ( Malzeme Tablosuna bakın ).
- Numune vakumunu açın ve manuel yük düğmesine basın.
- "Odak" düğmesine basarak sistemi numuneye odaklayın. Görüntüleme ekranında ölçüm için uygun bir konum belirleyin.
- Numuneyi tara. Profili düzleştirin ve özellik derinliğini ölçün.
- İstenen derinliği elde etmek için etit hızını kaydedin ve sonraki etit sürelerini modüle edin.
NOT: Ölçümler, silikon kuyunun derinliğini, depolanan parylene kalınlığını ve fotorezistin kalınlığını içerecektir. Prosedür boyunca her katmanın kalınlığının doğrulanması, doğru derinlikte derinlik elde etmek için gereklidir.
- Maruz kalmış parylene'yi çıkarmak için bir oksijen plazma aşındırma kullanınSilikon alt tabakaya kadar.
2. Bakteriyel Kültür ve Tohumlama ( Şekil 1a )
- Luria Broth'ta kolonileri başlatın (LB) gliserol stoklarından elde edilen agar plakalar ve iki hafta içinde kullanın. LB agar plakalarından istenen suşların kolonilerini seçin ve P. aeruginosa'nın gece kültürlerini başlatın. 220 ° C'de R2A ortamında çalkalarken gece boyunca kültürleri 37 ° C'de yaklaşık 18 saat inkübe edin.
NOT: Koloniler, mutasyonların ve floresan raportör genlerinin muhafaza edilmesini sağlamak için kaplamanın iki hafta içinde seçilmelidir. Tüm P. aeruginosa çalışmaları BSL-2 koşulları altında yapılmalıdır. - Silisyum gofreti, farklı ebat ve perde-kuyruk dizileri topluluklarını içeren ayrı cipslere bölmek için bir elmas kuramı kullanın. Her çipin, çalışma için iyi boyutların ve sahaların tam tamamlayıcısını içerdiğinden emin olun.
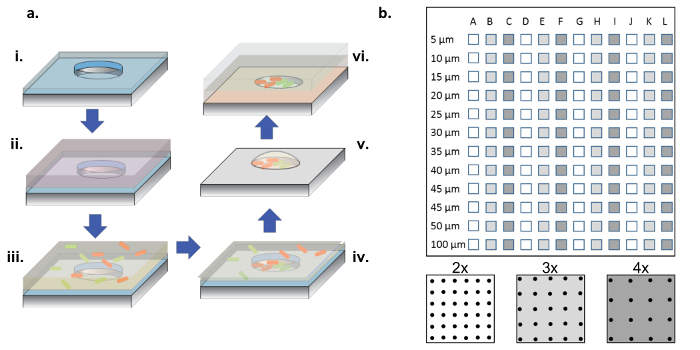
Şekil 1: Fabrikasyon ve Hücre Tohumlama Prosedürü. ( A ) Mikroeleme dizileriInce bir parylene katmanı (i) ile kaplanmış silikon waferlara yeniden kazınır. Kuyuları ıslatmak ve / veya yüzeyi fonksiyonelleştirmek için, bir protein çözeltisi, dizilerin üstündeki bir damla içine eklenir (ii). Protein çözeltisi çıkarılır, gofretler kurutulur ve arzu edilen bakterileri içeren yeni bir solüsyon eklenir (iii). Bakteri çözeltisi inkübasyon periyodundan sonra çıkarılır ve gofretlerin kurumasına izin verilir, kuyularda ve yüzeyde (iv) bakteri bırakılır. Yüzeye bağlı bakteri parylene kaldırma ile çıkarılır, bakterilerin arkasında mikrodalgalara temiz şekilde serpilmiş ve kuyuları sulu tutmaya yardımcı olan% 2 gliserol ortamı nedeniyle halen yaşayabilir (v). Silisyum cips daha sonra mikrodaklarda bakteri üremesini sağlayan bir agaroz jel kaplı cam lameline dizi tarafında yerleştirilir (vi). ( B ) Alt dizilerin tek bir silikon aygıta yerleştirilmesi. Her bir alt dizide, aynı kuyucuklar kümesi bulunur. Tüm alt-a boyunca mikrodalganın çapıRöleler çapı 5-100 μm arasındadır ve alt panel şemasında beyaz ila koyu gri renkleri ile gösterilen kuyu çapı adımında 2x, 3x veya 4x olarak düzenlenmiştir. Kuyu derinlikleri sığ olduğunda (<10 μm), 5 ve 10 μm'lik kuyu çapları nadiren faydalıdır, genelde bu küçük kuyuları kolonize eden hücrelerin eksikliği nedeniyle. Bu çalışmada, yalnızca 15-100 μm çaplı kuyulardan alınan veriler analiz edilmiştir. Bu rakamın daha büyük bir versiyonunu görmek için lütfen tıklayınız.
NOT: Şekil 1b'de gösterildiği gibi, komple bir çip, çapları 5 ila 100 μm arasında değişen ve ç farklı yivli ( yani çapı 2x, 3x ve 4x çaplı) dört kez yinelenen kuyu alt dizileri içerir.
- PBS solüsyonunda 500 μg / mL Sığır Serumu Albümini (BSA) 150 uL damla yerleştirinMikrodalgaları ıslatmak için dizinin üstünde. BSA çözeltisini, nemli bir odada oda sıcaklığında çipte 1 saat inkübe edin.
- Boş bir pipet uç kutusunun alt kısmını Fosfat Tamponlu Tuz (PBS) ile doldurarak bölmeyi oluşturun.
NOT: Mikro kuyucukların yüzeyini işlevselleştirmek için özel lektin gibi diğer maddeler BSA yerine kullanılabilir.
- Boş bir pipet uç kutusunun alt kısmını Fosfat Tamponlu Tuz (PBS) ile doldurarak bölmeyi oluşturun.
- BSA çözeltisi ile silikon yongaları inkübe ederken, 5 dakika süreyle 2.500 rpm (ortalama 950 xg karşılık gelen) kültürleri santrifüj ve daha sonra% 2 gliserol ile taze R2A orta 500 mcL tekrar süspanse edin.
- 600 nm'de bir UV-vis spektrometresi kullanarak kültür OD'sini belirleyin. % 2 gliserol R2A ortamı kullanarak 0,02'ye ayarlayın.
NOT: Gliserol, parylene kaldırma sırasında kuyuların kurumasını önlemeye yardımcı olur.
- 600 nm'de bir UV-vis spektrometresi kullanarak kültür OD'sini belirleyin. % 2 gliserol R2A ortamı kullanarak 0,02'ye ayarlayın.
- İnkübasyondan sonra, BSA solüsyonunu çıkarın ve sıvı boşaltımı çıkarıp yerine koyarak PBS ile 3 kez durulayınT silikon mikrodalga dizisini kapsıyor. Azot altında kurutun.
- Nemli bir odaya yerleştirilen kuru dizilerin her birine 150 μL 0.02 OD kültür ekleyin. Bakterilerin kuyu duvarlarına yapışmasına izin vermek için 1 saat boyunca 4 ° C'de inkübe edin.
NOT: Kuluçka için soğutma gerekmez. 4 ° C inkübasyon süresi, görüntüleme başlamadan önce bakterilerin büyümesini önlemek için kullanılabilir; böylece büyümeden önce toplulukların mekânsal organizasyonunu görselleştirebilir. Oda sıcaklığında kuluçka da kullanılabilir. Her iki protokol benzer büyüme eğrileri ile sonuçlanır.
3. Mikroskop Set-up
- Silikon cips üzerinde bakteri inkübasyonuna başlamadan önce sahne üstü çevresel kontrol odasını açın (malzeme tablosuna bakın) ve kontrol kutusundaki ayarları nem (~% 100) ve sıcaklık (30-32 ° C) olacak şekilde ayarlayın ° C, bkz. Adım 3.2) örnek eklemeden önce dengelenebilir.
- Numune tutucusunu düzleştirin ve inÇamur noktasına odadaki nemi arttırmak için PBS ile ıslatılmış laboratuar mendilleri (Malzeme Tablosuna bakınız ) ile numunenin çevresine bakınız. Görüntüleme düzlemindeki yoğuşmayı azaltmak için odanın sıcaklığını 30 ° C'ye ve odanın kapağını 32 ° C'ye ayarlayın.
NOT: Sürgü, yaklaşık 1 cm kalınlığında bir conta ile canlı hücrenin bölmesine sığar. Numune tutacağı, numune tutacağının üzerine yerleştirilen bir kabarcık seviyesinin yardımıyla düzleştirilir. Numune tutucu hafifçe eğilebilir ve contaya düz bir şekilde mühürlü kalır. - Kültürler silikon cips üzerinde kuluçka iken, elle görüntüleme en az 30 dakika önce cıva lambası için güç anahtarı açın. Elle kamerayı ve otomatik mikroskop sahnesini açın. Mikroskopu ve çevre ekipmanını kontrol etmek için kullanılan yazılımı açın ve ekipmanın yazılım tarafından tanıtıldığından emin olun.
NOT: Büyütme NA = 0.3 olduğunda 10X'dir.
- Mikrodalga, önceden sıvı haline gelene kadar yaklaşık 60 s. Agaroz çözeltileri ( yani % 2 agaroz, R2A ortamı) hazırladı.
- 75 mm x 22 mm, # 1.5 cam lamelinin etanol ile ıslatılması ve ortalanmış olarak, 2 x 3 "(50 x 75 mm) cam slayt boyunca uzunlamasına yerleştirilmesi İki PDMS ara parçası yerleştirin (~ 1 mm kalınlık) Lamel uzun kenarları boyunca ve cam lamel kaydırın, lamel kabaca 1 mm slayt kenarına sarkıtacak şekilde.
- Cam örtüsünün üstünde 5 mL sıvı agaroz çözeltisi dökün, tamamen kaplayacak kadar kalın ve lamel ile slayt arasında sıvı agar "sandviç" için montajın üzerine ikinci bir 2 x 3 "cam slayt yerleştirin.
NOT: Bu, Agarozun derinliğini kontrol eder ve lamellerin ve sertleştirilmiş agarozun toplam kalınlığını PDMS aralıklarına eşit kalınlıkta yaparak verir. - Cam kaydırma-lamel-agaroz jel-cam slayt sandviç agaroz çözüm katılaşıncaya kadar ayarlamak için izin verin; Sonra, bir buzdolabına aktarın. 15 dakika sonra aşırı katı agarozu alın ve cam lamelini kesin. Bunu kullandıkça temiz bir çanağa taşıyın ve bir buzdolabına koyun.
5. Bir agaroz kaplı Coverslip ve Görüntüleme ile Kuyuları Sızdırmazlık
- Bakteri inkübasyon periyodu tamamlandıktan sonra, agaroz kaplı lamel buzdolabından çıkarın ve aşağıdaki gibi silikon çipleri hazırlayın.
- Silisyum çipleri her biri 10 saniye boyunca, birer birer olmak üzere ultra saf suyun içine daldırın. Fazla sıvının büyük kısmı yonganın kenarlarından boşaltılana kadar onları bir laboratuar silin veya dokusunda kenarlarına yerleştirin.
- Her bir silikon çipin kenar uzunluğuna uyacak şekilde bir bant kes. Bantları, silikayı kaplayan parylene üzerine yerleştirin ve parylene kaplamayı hızla soymak için kullanın.
- immedSoyulmuş her çipi tersine çevirin ve her yongayı mikrodalga düzen tarafı bir agaroz kaplı lamelin agarozla kaplanmış tarafıyla yüzleşecek şekilde (ve bununla temas kuracak şekilde) yerleştirin. Kuyuların dışındaki bakterilerin büyümesini önlemek için çipi agaroza dokunduktan sonra hareket ettirmemeye veya yer değiştirmemeye özen gösterin.
- Mikrosel dizi / agaroz lamelini, miscroscope'daki sahne üstü çevresel kontrol odasının slayt tutucusuna yerleştirin.
- İlgilendiğiniz dizileri bulmak için ortam ışığını veya yönlendirilmiş ışığı ( örn. Bir el feneri) kullanın. Bu pozisyonları kaydetmek için otomatik aşamayı kontrol eden ticari yazılımı kullanın (materyal tablosuna bakın). Konumlar saklandıktan sonra çevre ışığı veya yönlendirilmiş ışığı kapatın.
- Ticari yazılımda, "ND Acquisition" panelini açın.
NOT: Bu panel belirli bir dizine otomatik olarak kaydedilebilecek bir menünün yanı sıra programlanabilir görüntü elde etmeyi de içerir. Bu deneyler için, "Zaman", "XY" ve "λ" menüleri kullanılır. - Yazılımdaki konumları kaydetmek için, "XY menüsü" nü tıklayın ve daha sonra kaydedilmesi gereken her konum için sol taraftaki boş bir kutuyu işaretleyin. Ayrıca, "Z'yi Dahil Et" düğmesini tıklayın.
- İstenilen dalga boylarında ve uygun floresans filtre küplerini kullanarak 10 büyütmede zamanla görüntü elde edin (Bkz. Malzeme Tablosu).
- Kaydedilen her bir yere taşınmak ve kuyularda odaklanmak için kontrol yazılımını ve kaydedilmiş dizi konumlarını kullanın. Kaydedilen listedeki her XY konumunu tıklayın ve Green Flourescence Protein (GFP) filtresini kullanarak odağı ayarlayın. Yeni z-konumunu, z-konumunu gösteren oku tıklatarak kaydedin.
NOT: Bu işlem zaman alıcı olabilir. Kazancın arttırılmasına yönelik önlemi almayı ve photobleaching'i önlemek için ışık yoğunluğunu azaltmak için nötr yoğunluk filtresini kullanmayı düşünün. - Dizinin yüzeyine odaklandığında z ekseni konumundaki farkı not ederek, her dalga boyu için odak düzlemleri arasındaki z ekseni mesafesini belirleyin. Karışık kırmızı / yeşil bakteri popülasyonlu dizi arasından 2-3 yer seçin ve Kırmızı Floresan Protein (RFP) filtresini kullanarak odaklama yapın.
- GFP ve RFP floresans filtrelerini kullanarak odak düzlemleri arasındaki mesafeyi çıkarın ve "λ" menüsü altında odak düzlemi ayarını ekleyin.
NOT: Örneğin, dizi 50 μm'lik bir z-konumunda GFP kanalında odaklanmış görünüyorsa ve aynı dizi 55 μm'de RFP kanalında odaklanmış görünüyorsa, "λ" da RFP optik yapılandırmasının yanında + 5'i ekleyin "Menüsünden.
- GFP ve RFP floresans filtrelerini kullanarak odak düzlemleri arasındaki mesafeyi çıkarın ve "λ" menüsü altında odak düzlemi ayarını ekleyin.
- Zaman atlamalı görüntü elde etmeye başlayın.
NOT: Burada gösterilen deneyler için, RFP ve GFP görüntüleri, her dizi konumu için, kontrol eden ticari bir yazılım aracılığıyla çok boyutlu görüntü elde edilmesi yoluyla 30 dakika aralıklarla edinilmiştirKamera, deklanşör, filtre tekerleği ve motorlu sahne.- "Aralık" ı 30 dk ve "Deneme süresi" "Zaman" menüsü altında 24 saate ayarlayın. "Şimdi Çalıştır" ı tıklayın.
NOT: "Zaman", "XY" ve "λ" kutuları işaretlendiğinde, programın çalıştırılmasıyla sahne her konuma ( yani kaydedilmiş XYZ konumlarına) taşır, bir dalga boyunda görüntü çeker, z konumunu hareket ettirir ( Yani lambda veya dalga boyu kontrolü) hesaba katmak için ikinci görüntüyü alın, bir sonraki dizi konumuna (çok nokta) geçin ve bunu 30 dakikalık aralıklarla (zaman aşımı) tekrarlayın.
- "Aralık" ı 30 dk ve "Deneme süresi" "Zaman" menüsü altında 24 saate ayarlayın. "Şimdi Çalıştır" ı tıklayın.
- Kaydedilen her bir yere taşınmak ve kuyularda odaklanmak için kontrol yazılımını ve kaydedilmiş dizi konumlarını kullanın. Kaydedilen listedeki her XY konumunu tıklayın ve Green Flourescence Protein (GFP) filtresini kullanarak odağı ayarlayın. Yeni z-konumunu, z-konumunu gösteren oku tıklatarak kaydedin.
- Aydınlatma kontrol görüntülerini edinin.
NOT: Her biri 25x4 konumlu görüntüleri çekmek için "ND Acquisition", "Time" ve "XY" menülerini kullanın.- Tüm ışık kaynaklarını kapatarak 100 "karanlık alan" görüntüsü serisi alınStandart bir slaydın bir "görüntüsü". Bu görüntüler kamera gürültüsünü yakalar. Zaman atlaması sırasında kullanılan en uzun pozlama süresini kullanın (adım 5.3.3).
- Verilen deneysel koşullarda pürüzlü aydınlatmayı yakalamak için birkaç farklı yerde standart bir slayt ( yani tekbiçimli RFP veya GFP yoğunluğu) görüntüleyerek bir dizi 100 "aydınlatma alanı" görüntüsü alın. Doygunluğa erişmeden sinyali en üst düzeye çıkaran bir pozlama zamanı seçin.
6. Analiz
- Bir görüntü analiz yazılımını (ImageJ gibi) kullanarak görüntü yığınlarını işleyin.
- Elde edilen görüntüleri ticari yazılımları kullanarak tiff dosya biçimine dönüştürün. "Dosya"> "İçe Aktarma"> "Resim Sırası" nı tıklayarak görüntü analiz yazılımına görüntü yükleyin.
- Tüm "karanlık alan" ve "aydınlatma alanı" görüntülerinin ortalaması alınarak bir "Düzeltme Resmi" oluşturun. Subtrac"İşlem"> "Görüntü Hesaplayıcı" yı seçerek ortalama "aydınlatma alanı" görüntüsünden ortalama "karanlık alan" görüntüsü. "Görüntü1" ve "Görüntü2" ve "İşlem" alanındaki "Çıkarma" olmak üzere iki görüntüyü seçin. "Tamam" ı tıklayın.
- Ortalama için düzeltme (veya karanlık alan) resimlerini yükleyin, "Resim"> "Yığınlar"> "Z Projesi"> "Ortalama Projeksiyon" u tıklayın.
- Gerekirse görüntü kaydı gerçekleştirin. Ardından, "İşlem"> "Arka Planı Çıkart" ı tıklayarak arka payı çıkarma işlemini gerçekleştirin. "Yarıçap" alanına bir yarıçap girin ( ör. 125) ve "sürgülü paraboloid" i seçin.
- Aydınlatma düzeltme işlemini "İşlem"> "Hesap Makinesi Artı" ile gerçekleştirin. Aşağıdaki parametreleri seçin: işlem, bölme; I1, iyi görüntü; I2, düzeltme görüntüsü; K1, düzeltme imgesi ortalaması; Ve k2, 0. "Cre" i tıklayın.Yeni Porsiyon yedi. "
NOT: Bu veri kümesi kayıt gerektirmez, ancak diğer çalışmalarda ImageJ Plugin StackReg "Çeviri" dönüşümü ile birlikte kullanılmıştır. Arka arkaya çıkarma için, her resim seti için aynı kayan paraboloid yarıçapını kullanın. Örneğin, görüntülenecek en büyük oyukların piksel yarıçapı 100 olduğunda, her görüntü kümesi için 100'den ( örneğin 125) büyük bir yarıçap kullanın.
- Mikro kuyucuktaki her suşun büyümesini belirleyin.
- ImageJ "MicroArray" eklentisini kullanarak, istenen dizideki her bir mikrodalga çevresinde ilgi bölgeleri (ROI'ler) seçin.
- "HARİTA" menüsünde, "Şebekeyi Sıfırla" yı tıklayın. Satırları, sütunları ve çapları belirtin (dizi üzerindeki kuyu boyutuna ve sayısına göre; bkz. Şekil 1b ). "ROI şekli" menüsünden "daire" seçin.
- "Alt" tuşunu basılı tutarken sol üst ROI'yi fareyle seçerek fareyi hareket ettirin tROI dizisi. Dizinin boyutunu değiştirmek için sol alt ROI'yı seçerken "shift" tuşunu basılı tutun. ROI'lerin aralığını değiştirmek için dizinin sağ tarafından bir ROI seçerken, köşelerde değilse "shift" tuşunu basılı tutun.
- ROI dizisini bir görüntünün kuyularının üzerine oturtmak için yukarıdaki komutları kullanın. "RT ölçümü" nü tıklayın.
NOT: Eklenti istenen ölçümleri her ROI'den verecektir. Arka plan sinyalini ( diğer bir deyişle, dış halkadan çıkarılmış orta halkadan gelen sinyal) ve flüoresan ölçümlerini ( yani, iç halkadan gelen sinyal) toplamak için kuyuların etrafında konsantrik halkalar oluşturarak üç ROI ebadını kullanın.
- Verileri bir elektronik tablo yazılımında toplayın ve arka plan sinyalini hesaplayın. Daha fazla analiz için özel bir komut dosyası yazılımına aktarın.
- ImageJ "MicroArray" eklentisini kullanarak, istenen dizideki her bir mikrodalga çevresinde ilgi bölgeleri (ROI'ler) seçin.
- Veri organizasyonu ve analizi
- Verileri içe aktarın ve verileri düzenleyinImageJ'de her zaman aşağıdaki sıra ile bir matriste toplanmıştır: sütun 1, alt dizi numarası; Sütun 2, kuyu sırası; Sütun 3, kuyu sütunu; Sütun 4, ortalama yoğunluk; Sütun 5, arka plan yoğunluğu; Ve sütun 6, ortalama yoğunluk - arka plan yoğunluğu.
- MCherry ve GFP ediniminin sonuçlarını farklı matriste ayırın. Her alt dizideki sonuçları ve her rengi bir hücre dizisindeki farklı bir hücrede saklayın.
NOT: Bu organizasyon, görüntü verileri ve ölçüm sonuçları arasında ileri-geri hareket etmeyi, verileri temizlemeyi ve ölçümlerin verileri doğru şekilde temsil etmeyi sağlamayı kolaylaştırır.
- MCherry ve GFP ediniminin sonuçlarını farklı matriste ayırın. Her alt dizideki sonuçları ve her rengi bir hücre dizisindeki farklı bir hücrede saklayın.
- P. aeruginosa'nın otofluoresansı için ayarlayın .
NOT: GFP ve mCherry suşlarının birlikte-kültürünü içeren deneylerde, mCherry ile yeşil otofloresans arasındaki ilişkiyi saptamak için bir mCherry çipi analiz edilmelidir.- Tüm mCherry ΔretS'den mCherry-versus-GFP sinyalini çizin &# 916; tse / i1-6 kuyuları, GFP kanalındaki mCherry sinyali ve otofloresans arasındaki ilişkiyi belirlemek için tüm zaman noktalarında. Otofloresans sinyalini birlikte kültürlerden çıkartın.
- Yörüngeleri çizin ve her yörüngeye değiştirilmiş bir lojistik denklemi sığdırarak en basit kareler kullanarak hesap çizelgesini veya bir çizelge yazılımına uyan parametreleri çıkarın.
- GFP ve mCherry yörünge parametreleri arasındaki korelasyonlar arayın.
- Verileri içe aktarın ve verileri düzenleyinImageJ'de her zaman aşağıdaki sıra ile bir matriste toplanmıştır: sütun 1, alt dizi numarası; Sütun 2, kuyu sırası; Sütun 3, kuyu sütunu; Sütun 4, ortalama yoğunluk; Sütun 5, arka plan yoğunluğu; Ve sütun 6, ortalama yoğunluk - arka plan yoğunluğu.
Sonuçlar
Burada sunulan deney platformu, bakteri topluluklarının yüksek verimli ve yüksek içerikli çalışmaları için tasarlanmıştır. Tasarım, çeşitli büyüklükteki kuyulardan büyüyen binlerce topluluğun aynı anda analiz edilmesini sağlıyor. Bu mikrodellek dizilimi tasarımı ile, nihai topluluk bileşiminin ilk ekim yoğunlukları, iyi boyut ve kimyasal çevre üzerindeki bağımlılığı belirlenebilir. Bu çalışma, mikrodalgalı dizide iki üyeli bir toplumun büyümes...
Tartışmalar
Bu makale, bakteri topluluğunun gelişiminin yüksek verimli ve yüksek içerikli canlı hücre görüntüleme tabanlı analizini mümkün kılmak için tasarlanmış bir mikrodalkaralı dizi cihazı ve deney protokollerini sundu. Buradaki gösterinin odağı, temas yoluyla aracılık edilen Tip VI sekresyonun topluluk gelişimine etkilerini incelemek iken, diziler esnek olacak şekilde tasarlandı ve geniş bir mikrobik topluluklar mikrobik-mikrop etkileşimleri çalışmasına uyacak şekilde tasarlandı. Buradaki ç...
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Mikrokoşa diziler, Nanofaze Malzeme Bilimleri Merkezi Kullanıcı Tesisleri Bölümü, Temel Enerji Bilimleri Ofisi, ABD Enerji Bakanlığı tarafından imal edildi ve karakterize edildi. Bu çalışma için maddi destek Oak Ridge Ulusal Laboratuar Direktörü Araştırma ve Geliştirme Fonu aracılığıyla sağlandı. Yazarlar ayrıca, bu araştırmalarda kullanılan P. aeruginosa suşlarının temini için J. Mougous Laboratuvarına (Washington Üniversitesi, Seattle, WA) teşekkür etmek istiyorlar .
Malzemeler
| Name | Company | Catalog Number | Comments |
| Parylene N | Specialty Coating Systems | CAS NO.:1633-22-3 | |
| Parylene coater | Specialty Coating Systems | Labcoter 2 Parylene Deposition Unit PDS2010 | |
| Silicon Wafer | WRS Materials | 100mm diameter, 500-550μm thickness, Prime, 10-20 resistivity, N/Phos<100>, | |
| adhesion promoter | Shin-Etsu Microsci | MicroPrime P20 adhesion promoter | |
| postive tone photoresist | Rohm and Haas Electronics Materials LLC (Owned by Dow) | Microposit S1818 Positive Photoresist (code 10018357) | |
| Quintel Contact Aligner | Neutronix Quintel Corp | NXQ 7500 Mask Aligner | |
| Reactive Ion Etching Tool | Oxford Instruments | Plasmalab System 100 Reactive Ion Etcher | |
| R2A Broth | TEKnova | R0005 | |
| Bovine Serum Albumin | Sigma | A9647 | |
| Multimode Plate Reader | Perkin Elmer | Enspire, 2300-0000 | |
| Fluorescent Microscope | Nikon | Eclipse Ti-U | |
| Automated Stage | Prior | ProScan III | |
| CCD camera | Nikon | DS-QiMc | |
| Stage-top environmental control chamber | In Vivo Scientific | STEV ECU-HOC | |
| Phosphate Buffered Saline | ThermoFisher Scientific | 14190144 | |
| UltraPure Agarose | ThermoFisher Scientific | 16500500 | |
| 25 x 75 mm No. 1.5 coverslip | Nexterion | High performance #1.5H coverslips | |
| Fluorescence Reference Slides | Ted Pella | 2273 | |
| Physical Stylus Profilometer | KLA Tencor | P-6 | |
| lab wipes | Kimberly Clark | Kimipe KIMTECH SCIENCE Brand, 34155 | |
| commercial software | Nikon | NIS Elements | |
| Zeiss 710 Confocal Microscope | Zeiss | ||
| filter cubes | Nikon | Nikon FITC (96311), Nikon Texas Red(96313) |
Referanslar
- Zhou, J., Deng, Y., et al. Stochasticity, succession, and environmental perturbations in a fluidic ecosystem. Proc Natl Acad Sci. 111, E836-E845 (2014).
- Valm, A. M., Welch, J. L. M., et al. Systems-level analysis of microbial community organization through combinatorial labeling and spectral imaging. Proc Natl Acad Sci USA. 108 (10), 4152-4157 (2011).
- Satoh, H., Miura, Y., Tsushima, I., Okabe, S. Layered structure of bacterial and archaeal communities and their in situ activities in anaerobic granules. Appl Environ Microbiol. 73 (22), 7300-7307 (2007).
- Kim, H. J., Boedicker, J. Q., Choi, J. W., Ismagilov, R. F. Defined spatial structure stabilizes a synthetic multispecies bacterial community. Proc Natl Acad Sci USA. 105 (47), 18188-18193 (2008).
- Nunan, N., Wu, K., Young, I. M., Crawford, J. W., Ritz, K. Spatial distribution of bacterial communities and their relationships with the micro-architecture of soil. FEMS Microbiol Ecol. 44, 203-215 (2003).
- Grundmann, G. L. Spatial scales of soil bacterial diversity - The size of a clone. FEMS Microbiol Ecol. 48, 119-127 (2004).
- Langenheder, S., Lindstrom, E. S., Tranvik, L. J. Structure and Function of Bacterial Communities Emerging from Different Sources under Identical Conditions. Appl Environ Microbiol. 72 (1), 212-220 (2006).
- Camp, J. G., Kanther, M., Semova, I., Rawls, J. F. Patterns and Scales in Gastrointestinal Microbial Ecology. Gastroenterology. 136 (6), 1989-2002 (2009).
- Renner, L. D., Weibel, D. B. Physicochemical regulation of biofilm formation. MRS Bull. 36 (5), 347-355 (2011).
- Wessel, A. K., Hmelo, L., Parsek, M. R., Whiteley, M. Going local: technologies for exploring bacterial microenvironments. Nat Rev Microbiol. 11 (5), 337-348 (2013).
- Stacy, A., McNally, L., Darch, S. E., Brown, S. P., Whiteley, M. The biogeography of polymicrobial infection. Nat Rev Microbiol. 14 (2), 93-105 (2015).
- Hansen, R. R., Shubert, K. R., Morrell-Falvey, J. L., Lokitz, B. S., Doktycz, M. J., Retterer, S. T. Microstructured block copolymer surfaces for control of microbe adhesion and aggregation. Biosensors. 4 (1), 63-75 (2014).
- Hansen, R. R., Hinestrosa, J. P., et al. Lectin-functionalized poly(glycidyl methacrylate)- block -poly(vinyldimethyl azlactone) surface scaffolds for high avidity microbial capture. Biomacromolecules. 14 (10), 3742-3748 (2013).
- Timm, C. M., Hansen, R. R., Doktycz, M. J., Retterer, S. T., Pelletier, D. A. Microstencils to generate defined, multi-species patterns of bacteria. Biomicrofluidics. 9 (6), (2015).
- Keymer, J. E., Galajda, P., Muldoon, C., Park, S., Austin, R. H. Bacterial metapopulations in nanofabricated landscapes. Proc Natl Acad Sci USA. 103 (46), 17290-17295 (2006).
- Zhang, Q., Lambert, G., et al. Acceleration of Emergence of Bacterial Antibiotic Resistance in Connected Microenvironments. Science. 333 (6050), 1764-1767 (2011).
- Friedlander, R. S., Vlamakis, H., Kim, P., Khan, M., Kolter, R., Aizenberg, J. Bacterial flagella explore microscale hummocks and hollows to increase adhesion. Proc Natl Acad Sci USA. 110 (14), 5624-5629 (2013).
- Zhou, J., Liu, W., et al. Stochastic Assembly Leads to Alternative Communities with Distinct Functions in a Bioreactor Microbial Community. MBio. 4 (2), 1-8 (2013).
- van Vliet, S., Hol, F. J., Weenink, T., Galajda, P., Keymer, J. E. The effects of chemical interactions and culture history on the colonization of structured habitats by competing bacterial populations. BMC Microbiol. 14 (1), 116 (2014).
- Niepa, T. H. R., Hou, L., et al. Microbial Nanoculture as an Artificial Microniche. Sci Rep. 6, 30578 (2016).
- Hansen, R. H., Timm, A. C., et al. Stochastic Assembly of Bacteria in Microwell Arrays Reveals the Importance of Confinement in Community Development. PLoS ONE. 11 (5), e0155080 (2016).
- Hood, R. D., Singh, P., et al. A Type VI Secretion System of Pseudomonas aeruginosa Targets a Toxin to Bacteria. Cell Host Microbe. 7 (1), 25-37 (2010).
- LeRoux, M., Ja De Leon, ., et al. Quantitative single-cell characterization of bacterial interactions reveals type VI secretion is a double-edged sword. Proc Natl Acad Sci. 109 (48), 19804-19809 (2012).
- Whitney, J. C., Beck, C. M., et al. Genetically distinct pathways guide effector export through the type VI secretion system. Mol Microbiol. 92 (3), 529-542 (2014).
- Warrick, J. W., Timm, A., Swick, A., Yin, J. Tools for Single-Cell Kinetic Analysis of Virus-Host Interactions. PLoS ONE. 11 (1), e0145081 (2016).
- Zwietering, M. H., Jongenburger, I., Rombouts, F. M., Van't Riet, K. Modeling of the Bacterial Growth Curve. Appl Environ Microbiol. 56 (6), 1875-1881 (1990).
- Halsted, M., Wilmoth, J. L., et al. Development of transparent microwell arrays for optical monitoring and dissection of microbial communities. J Vac Sci Technol B Nanotechnol Microelectron. 34 (6), 06KI03 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır