A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
ניתוח של היסטון נוגדנים היסטון עם Microprays פפטיד
In This Article
Summary
כתב היד מתאר שיטות להחלת הטכנולוגיה microarray פפטיד פרופיל פרופיליות של נוגדנים כי לזהות היסטונים ושינויים שלאחר translational שלהם.
Abstract
לאחר שינויים טרנסלציוניים (PTM) על חלבונים היסטון נחקרים באופן נרחב עבור תפקידים שלהם ויסות מבנה הכרומטין ביטוי גנים. ייצור המוני והפצה של נוגדנים ספציפיים PTMs היסטון יש להקל מאוד מחקר על סימנים אלה. כמו נוגדנים PTM היסטון הם ריאגנטים מפתח עבור יישומים ביוכימיה רבים הכרומטין, ניתוח קפדני של נוגדנים הספציפיות נחוץ לפרש נתונים מדויק והמשך ההתקדמות בתחום. פרוטוקול זה מתאר צינור משולב עבור ייצור, ייצור ושימוש של microprays פפטיד עבור אפיון הספציפיות של נוגדנים היסטון. עיצוב וניתוח היבטים של הליך זה הם הקלו על ידי ArrayNinja, קוד פתוח וחבילת תוכנה אינטראקטיבית פיתחנו לאחרונה כדי לייעל את ההתאמה האישית של פורמטים להדפיס microarray. צינור זה שימש למסך מספר רב של antibodie PTM זמין מסחרית בשימוש נרחבS, ונתונים שנוצרו בניסויים אלה זמינים באופן חופשי דרך האינטרנט והרחבת ההיסטון נוגדנים היסטון מסד נתונים. מעבר היסטונים, המתודולוגיה הכללית המתוארת כאן ניתן ליישם באופן רחב לניתוח של נוגדנים ספציפיים PTM.
Introduction
הדנ"א הגנומי הוא ארוז באלגנטיות בתוך גרעין התא האוקריוטים עם חלבונים היסטון ליצירת הכרומטין. יחידת המשנה של הכרומטין היא הנוקלאוזום, המורכב מ -147 זוגות בסיס של דנ"א עטופים סביב ליבה אוקטמית של חלבונים היסטוניים - H2A, H2B, H3 ו- H4 1 . הכרומטין מאורגן באופן כללי לתוך euchromatin ארוז רופף ותחומים heterochromatin ארוז בחוזקה. מידת הדחיסה הכרומטין מסדירה את המידה שבה machineries חלבון יכול לגשת DNA הבסיסית לבצע תהליכים בסיסיים DNA תבניות כמו שכפול, שעתוק, ותיקון.
רגולטורים עיקריים של נגישות הגנום בהקשר של הכרומטין הם PTM על זנב בלתי מובנה ותחומי הליבה של חלבונים היסטון 2 , 3 . Histone PTMs לתפקד ישירות על ידי השפעה על המבנה של הכרומטין 4 בעקיפין througH גיוס של חלבונים הקורא מתחמי macromolecular הקשורים להם כירומטין שיפוץ, פעילויות אנזימטיות, פיגומים 5 . מחקרים של פונקציה PTM היסטון במהלך שני העשורים האחרונים להצביע על הסימנים האלה לשחק תפקידים מרכזיים בוויסות גורל התא, התפתחות אורגניזמים, ואת תחילת המחלה / התקדמות. מונעת על ידי ההתקדמות של הטכנולוגיה מבוסס proteomic מבוססי ספקטרומטריית מסה, יותר מ -20 PTMs היסטון ייחודי על יותר מ 80 שאריות היסטון שונים התגלו 6 . יש לציין כי שינויים אלה מתרחשים לעיתים קרובות בשילובים, ועולים בקנה אחד עם ההשערה "קוד היסטון", מחקרים רבים מציעים כי חלבונים הקורא ממוקדות אזורים נפרדים של הכרומטין באמצעות הכרה של שילובים ספציפיים של PTMs היסטון 7 , 8 , 9 . אתגר מפתח להתקדם יהיה להקצות פונקציות לגררשימה של htone PTMs ולקבוע כיצד שילובים ספציפיים של PTMs היסטון לתזמר את הפונקציות דינמי הקשורים הכרומטין.
נוגדנים הם ריאגנטים lynchpin עבור זיהוי של PTMs היסטון. ככזה, יותר מ -1,000 נוגדנים ספציפיים PTM ספציפי היסטון פותחו מסחרית לשימוש במחקר ביוכימיה הכרומטין. עם התפתחות מהירה של התפוקה גבוהה דנ"א טכנולוגיית רצף, ריאגנטים אלה נמצאים בשימוש נרחב על ידי חוקרים בודדים בקנה מידה גדול epigenomics "מפת הדרכים" יוזמות ( למשל , ENCODE ו BLUEPRINT) ב שבב- seq (כרום immunoprecipitation הכרומטין עם הדור הבא רצף ) צינורות ליצור ברזולוציה גבוהה מפות מרחביות של היסטון PTM היסטון גנום רחב 10 , 11 . עם זאת, מחקרים שנעשו לאחרונה הראו כי הספציפיות של נוגדנים PTM היסטון יכול להיות משתנה מאוד וכי ריאגנטים אלה התערוכה unf תכונות אבורייביות כגון הכרה ב- epitope מחוץ להשפעה, השפעה חיובית ושלילית חזקה על-ידי PTM שכנות וקושי בהבחנה של סדר השינוי על שאריות מסוימת ( לדוגמה , מונו-די- או טרי-מתילייזין) 12 , 13 , 14 , 15 , 16 , 17 , 18 . לכן, בקרת איכות קפדנית של ריאגנטים נוגדנים ספציפיים היסטון PTM יש צורך לפרש במדויק נתונים שנוצרו עם אלה ריאגנטים יקר.
טכנולוגיית Microarray מאפשרת חקירה סימולטנית של אלפי אינטראקציות macromolecular בתפוקה גבוהה, לשחזור, ואת פורמט ממוזער. מסיבה זו, מגוון של פלטפורמות microarray נוצרו לנתח חלבון DNA 19 ,"20, חלבון 21 חלבון, אינטראקציות חלבונים פפטיד 22. אכן, microstrays פפטיד היסטון צמחו כפלטפורמה גילוי אינפורמטיבי עבור מחקר ביוכימיה הכרומטין, המאפשר פרופיל התפוקה גבוהה של סופרים, מחקים, ו הקוראים של PTMs היסטון 15 , 23 , 24 , וכן לניתוח של נוגדנים הנוגדן היסטון 17 , 25. מעבר ליישום שלהם במחקר הכרומטין ו epigenetics, מערכי פפטיד היסטון יש פוטנציאל השירות כמו אבחון / בדיקה פרוגנוסטית זאבת מערכתית אריתמטוס ומחלות אוטואימוניות אחרות שבו אנטי- נוגדנים עצמיים הכרומטין נוצרים 26 , 27 .
כאן, אנו מתארים צינור משולב שפיתחנו עבור עיצוב, בודה, queRyan histone microptrays פפטיד כדי ליצור פרופילים ספציפיות עבור נוגדנים לזהות histones ו PTM שלהם. צינור הוא הקל על ידי ArrayNinja, קוד פתוח, יישום תוכנה אינטראקטיבית כי פיתחנו לאחרונה, המשלבת את שלבי התכנון והניתוח של ניסויים microarray 28 . ArrayNinja עובד הכי טוב ב- Google Chrome. בקצרה, מדפסת microarray קשר רובוטית משמש להפקיד ספריה של פפטידים היסטון ביוטין מצומדות במיקומים מוגדרים על שקופיות מיקרוסקופ זכוכית streptavidin מצופה. מערכים לאחר מכן ניתן להשתמש בפורמט assay תחרותי במקביל לחקור אינטראקציות נוגדן-epitope ( איור 1 ). הספריה פפטיד מורכב מאות פפטידים סינתטיים ייחודי מחסה PTMs (ליזין acetylation, ליזין / מתילציה ארגינין, ו serine / threonine זרחון) לבד בשילובים רלוונטיים נגזר בעיקר מן הנתונים datasets proteomics. שיטות סינתזה פפטיד ואימות מפורטים במקום אחר 23. נתונים שנוצרו על ידי המתמשך שלנו histone PTM נוגדנים בדיקות הסינון באמצעות פלטפורמת מערך זה מאוחסנים על משאב אינטרנט ציבורי, היסטון נוגדנים היסטון מסד נתונים (www.histoneantibodies.com). יש לציין, כי microarrays היסטון פפטיד מפוברק עם וריאציות של פרוטוקול זה שימשו גם בהרחבה כדי לאפיין את פעילותם של תחומים הקורא PTM היסטון 8 , 29 , 30 , 31 , 32 , 33 , 34 , 35 , 36 , 37 ולאחרונה פרופיל היסטון PTM סופר ומחק פעילויות 24 .
/files/ftp_upload/55912/55912fig1.jpg "/>
איור 1: תיאור קריקטורה של נוהל שלב של הקרנת נוגדנים על micropray פפטיד היסטון. פפטידים היסטון Biotinylated מחסה שינויים שלאחר טרנסלציה מוגדרים (עיגולים אדומים וכחולים) מודפסים בשיתוף עם biotin-fluorescein על זכוכית מצופה streptavidin. אינטראקציות חיוביות הם דמיינו כמו פלואורסצנטי אדום. אנא לחץ כאן כדי להציג גרסה גדולה יותר של דמות זו.
Protocol
1. התקנה והפעלת ArrayNinja
- הורד והתקן את Oracle Virtual Box מתוך www.virtualbox.org.
- הורד או לבטל את הדחיסה של מכונה וירטואלית ArrayNinja (VM) מ http://research.vai.org/Tools/arrayninja.
- פתח Virtual Box ולהוסיף את ArrayNinja VM על ידי לחיצה על 'מחשב', 'הוסף', ובחר arrayninja.vbox מהתיקייה שבה נשמר ArrayNinja VM.
- התחל ArrayNinja על ידי בחירת אותו בתוך Virtual Box ולחיצה על החץ 'התחל' ירוק.
- Virtual Box יפתח חלון חדש ויציג הודעה כי VM ניתן לגשת על ידי ניווט בדפדפן האינטרנט כדי localhost: 2080.60; הערה: גרסה של containerized של ArrayNinja זמינה גם דרך hub.docker.com/r/bradley.dickson/arrayninja/
2. עיצוב מערך שקופית ואת מקור פלייט פריסה
- תחת "תוכנית פריסה שקופית" כותרת על ממשק ArrayNinja, לחץ על הקישור המתאים למדפסת microarray בשימוש.
הערה: ArrayNinja כבר מתוכנת לחקות את התנועה הרובוטית של שני מדפסות microarray נפוץ (ראה טבלה1). תאימות עם arrayers אחרים ניתן להגדיר לפי בקשה. - לחץ בתוך תיבת הדו-שיח 'טען צלחת', הקלד "ריק" ולחץ על 'Enter'. ראה איור 2 עבור צילום מסך של מודול העיצוב ArrayNinja.
- להתאים את קוטר ספוט ל 275 מיקרומטר ואת המרווח נקודה ל 375 מיקרומטר. התאם את ההגדרות הנותרות (בלוקים צלחת / שורה צלחת, סה"כ שורות צלחת, משכפל, תכונות y, מערכים סופר, פאדג 'סופר, ראה איור 2 ) כדי להתאים אישית את התכונות יופיעו על השקופית microarray.
הערה: קוטר הנקודה נקבע על ידי גודל של סיכת microarray. כמו הגדרות אלה מותאמים, שקופית קריקטורה יעדכן בזמן אמת. השתמש בקריקטורה זו כדי לראות כיצד כל הגדרה משנה את פריסת השקופית הסופית. - לאחר פריסת התכונות בשקופית הסופית, העכבר מעל כל תכונה ייחודית והזן את מזהה התכונה בתיבת הדו-שיח המוקפצת.
הערה: מזהי תכונות יכולים להכיל מספרים, אותיות או שילובים. זה נדרש רק עבור תכונות ייחודיות, תיבת הדו שיח לא יופיע כאשר משוכפלים מסומנים. - לאחר שכל התכונות הייחודיות הוקצו למזהה, לחץ על 'מאכלס'. הזן שם לפריסת השקפים ולחץ על 'הדפס את הצלחת' כדי לשמור. דף חדש יפתח המציג את מספר 384-well המקור לוחות צורך לפברק את השקופית שקופית נבחר טבלה מיפוי המיקום הפיזי של כל תכונה להיות נטען בצלחת המקור (ים).
הערה: זכור את השם הזה, שכן הוא ישמש כדי לזכור עיצוב זה בעת ניתוח נתוני microarray (ראה סעיף 6.2). לחיצה על "הדפס את הצלחת שלך שומר את הפריסה בתוך ArrayNinja.

איור 2: מודול עיצוב ArrayNinja. צילום מסך של מודול העיצוב ArrayNinja מוצג בשורה מנוקדת. לוח הבקרה (למעלה) מציג את כל הפרמטרים שניתן לשנות במדפסת microarray. כמו פרמטרים אלה מותאמים, התמונה הקריקטורה של פריסת השקופית (למטה משמאל) עדכונים בזמן אמת. לאחר הפריסה, המשתמש יכול לעבור על נקודות בודדות כדי להזין מזהי תכונות ייחודיים. ArrayNinja בונה קלט משתמש זה מפה של המיקום של כל תכונה בצלחת המקור (ים) (מימין למטה) צורך לפברק פריסה שקופית microarray שצוין. אנא לחץ כאן כדי להציג גרסה גדולה יותר של דמות זו.
3. בודה Microarrays
- הכנת לוח המקור
- השתמש המפה שנוצר עם ArrayNinja בסעיף 2.5 כדי ליצור את הצלחת 384 גם מקור (ים).
הערה: תיאורים מפורטים של פפטידים queried על פלטפורמה זו ניתן למצוא במקום אחרS = "xref"> 17. - הפקדה 1 - 2 μL של כל תכונה ( למשל , פפטיד היסטון biotinylated) לתוך הבאר הנכון של צלחת 384 גם מקור (ים).
הערה: פפטידים היסטון Biotinylated מופקדים בדרך כלל מ 200-400 פתרונות מניות מיקרומטר, אשר משווה את 10 עד 25 פי הטבעת עודף של פפטיד לאתרים streptavidin מחייב במקום מערך אחד. זה מחושב באמצעות המשוואה: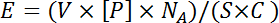
כאשר V הוא הנפח מועבר על ידי סיכה, [טז] הוא הריכוז של התכונה מודפס, N A הוא מספר אבוגדרו, ו- S הוא שטח הפנים של נקודה. C הוא הכיסוי של השקופית, לידי ביטוי כמספר המולקולות streptavidin ליחידת שטח כפול שלוש (המספר הממוצע של אתרי מחייב streptavidin זמין). V ו- C מתקבלים על ידי היצרן בהתאמה. תכונות אחרות עשויות להיותדורשים ריכוזים שונים בהתאם לגודל של ביומולקולה שבו הצפיפות עשויה להיות דאגה. מגוון של ריכוזי הדפסה עבור כל סוג חדש של תכונה צריך להיבדק באופן אמפירי כדי לקבוע את הריכוז הדפוס האופטימלי. - לדלל כל תכונה 10 פי עם חיץ הדפסה 1x בתוספת 1% אלבומין בסרום שור (BSA). ספין את צלחת המקור (ים) ב 500 xg למשך 2 דקות בטמפרטורת החדר.
הערה: הכללה של biotin שכותרתו fluorescein (5 מיקרוגרם / μL) במאגר ההדפסה מומלץ כבקרת תצפית וכסיוע חזותי כדי להקל על יישור מערך תקין במהלך ניתוח (ראה איור 4 ).
- השתמש המפה שנוצר עם ArrayNinja בסעיף 2.5 כדי ליצור את הצלחת 384 גם מקור (ים).
- פרוטוקול הדפסה ( איור 3 א - ב ).
- הכן את המערך על ידי ריקון מיכל איסוף פסולת ומלא את מיכל פתרון לשטוף ומיכל אדים עם מים מזוקקים סטריליים.
- הזן את הפרמטרים uSed לעצב את השקופית ArrayNinja (סעיף 2 ואיור 2 ) לתוך תוכנית בקרת מדפסת microarray.
- באמצעות תוכנית בקרת מדפסת microarray, להגדיר את הליך הכביסה לשטוף 1 עם טבילה אחת. הגדר את ההגדרות לאחר כביסה כדי לטבול מחדש את הפינים 5 פעמים לאחר כל לשטוף. הגדר את הלחות ל -60%.
הערה: תצורת השטיפה האופטימלית עשויה להשתנות בהתאם למדפסת microarray בשימוש. תצורת הטבילה האופטימלית לאחר השטיפה עשויה להשתנות בהתאם למדפסת microarray בשימוש. - הכנס שקופיות פונקציונלי ( למשל , זכוכית streptavidin מצופה) לתוך לוחות המצע ומכניסים את כל platens לתוך המעלית platen. הכנס את צלחת המקור (ים) לתוך מחזיק הצלחת (ים) והכנס למעלית המקלחת.
- לחץ על 'הדפס'. לפקח על תהליך ההדפסה עבור כמה סיבובים של בתצהיר תכונה כדי להבטיח את כל הגדרות לשטוף לטבול נכונים. לאחר השלמת ההדפסה, הסראת המצע platens מן המערך.
הערה: כאשר ריצות הדפסה גדולות אוסרים על השלמת שלבי חסימה בתוך יום אחד, שקופיות מודפס יכול להיות מודגרת בתא humidified ב 4 מעלות צלזיוס למשך הלילה. דגירה את השקופיות ליד כוס קטנה של מים בתוך קופסת קרטון חתום בעטיפה פלסטיק. - לחסום את השקפים עם חיץ חסימת למשך 30 דקות בטמפרטורת החדר עם ערבוב.
- שטפו את השקופיות 2 x 10 דקות בטמפרטורת החדר ב פוספט שנאגרו מלוחים (PBS), pH 7.6 עם ערבוב. יבש את השקופיות על ידי ספינינג צנטריפוגה שקופיות microarray במשך 30 s בטמפרטורת החדר.
הערה: לעיבוד מספר גדול של שקופיות בבת אחת, תפוקה גבוהה מיקרוסקופ שקופית כביסה החדר ניתן להשתמש, המאפשר 50 שקופיות להישטף במקביל. - עבור שקופיות שנועדו להיות מחולקת עם שעווה, המשך לסעיף 4.1. עבור כל עיצובים אחרים, שקופיות חנות ב 4 ° C מוגן מפני אור ולחות.
הערה: מודפס פפטידים היסטון biotinylatedיציבים במשך לפחות 6 חודשים כאשר מאוחסנים בדרך זו.
4. מחיצות Microarray שקופיות
- הידרופובי עט שעווה ( איור 3 ג )
- החל שעווה סביב אזורים המכילים תכונות באמצעות עט שעווה. אפשר שעווה לאוויר יבש במשך 5 דקות לפני שתמשיך סעיף 5.
הערה: לאחר חסימה, נקודות המערך יכול להיות קשה מאוד לדמיין לפי העין. עיצוב שקופית מ ArrayNinja ניתן להדפיס בקנה מידה ומשמש מדריך עבור החלת שעווה.
- החל שעווה סביב אזורים המכילים תכונות באמצעות עט שעווה. אפשר שעווה לאוויר יבש במשך 5 דקות לפני שתמשיך סעיף 5.
- אטם הסיליקון ( איור 3D )
- לקלף את הסרט ברור את החלק האחורי של אטם מערך במקום בצד דבק על גבי השקופית microarray.
- החזק את האטם במקום למשך 5 שניות לפני שתמשיך לסעיף 5.
- שעווה חותם ( איור 3E )
- עבור שקופיות שנועדו להיות partitioned על ידי שעווה, להדפיס שקופית הבדיקה על זכוכית רגילה באמצעות BSA 10%. השתמש בשקופית הבדיקה הזו כדי לייעל את המדריכים imminter שעווה או הגדרות מערך כדי להבטיח את כל התכונות יהיו בתוך החדרים עובש שעווה.
- מחממים את השעווה microarray שעווה ל 85 מעלות צלזיוס עד כל שעווה הוא נמסה לחלוטין, כ 30 דקות.
- הכנס את השקופית עם הצד המודפס פונה כלפי מטה ולדחוף את השקופית כל הדרך אל המדריך הנכון על ההופעה microarray. משוך את הידית כדי להביא את התבנית במגע עם פני השטח של השקופית. החזק במשך 2 שניות.
הערה: זמן ההחזקה יכול להיות שונה כדי להשיג עובי גבול שעווה אופטימלי. - להסיר במהירות את השקופית ואת חזותית לבדוק את גבולות השעווה כדי להבטיח את כל הבארות סגורים כי הגבולות הם לא כל כך עבה כי הם לחרוק על כתמים. שקופיות חנות ב 4 ° C מוגן מפני לחות ואור.
הערה: ניתן להגדיל או להקטין את זמן ההחזקה כדי להגיע לגבולות עבים או דקים יותר.

איור 3: ייצור Microarray. (א) Histone פפטיד microarray ייצור על שקופיות מיקרוסקופ streptavidin מצופה באמצעות מדפסת microarray מגע. (ב) Microarrays מפוברק עם 3 subrarays של 48 x 48 רשת של תכונות פפטיד. הפרדה של ג ( 3 ) תת-שערים עם עט שעווה הידרופובי, (D) 2 תת-דומייתי עם דבק סיליקון, ו- (E) 48 תת-שכבות עם טביעת שעווה. כל microarrays מוצגים מפוברקים באמצעות שקופיות 25 x 75 מ"מ מיקרוסקופ. אנא לחץ כאן כדי להציג גרסה גדולה יותר של דמות זו.
5. הכלאה של נוגדנים PTM היסטון עם Microarray פפטיד
- הכן חיץ הכלאה (PBS, pH 7.6, BSA 5%, 0.1% Tween-20).
- לאזן את השקופית במאגר הכלאהבאמצעות כלי הכלאה. לגמרי לכסות את השקופית כולה חיץ הכלאה דגירה במשך 30 דקות ב 4 מעלות צלזיוס על שייקר מסלולית במהירות נמוכה.
- הכן פתרון המכיל נוגדנים PTM נוגדן היסטון במאגר הכלאה.
הערה: בנתונים לדוגמה, הן נוגדנים היסטון # 1 ונוגדן # 2 היו מדולל 1: 1,000 במאגר הכלאה. טווח דילול דומה לזה המשמש immunoblotting מומלץ כנקודת התחלה. - לדגור את המערך עם פתרון נוגדנים עבור 1 שעות ב 4 ° C. הסר את הפתרון נוגדנים לשטוף את המערך 3 פעמים במשך 5 דקות ב 4 מעלות צלזיוס עם PBS קר, pH 7.6.
- הכן 1: 5000 - 1: 10,000 דילול של נוגדן משני פלואורסצנטי מצומדות לצבוע במאגר הכלאה.
- לדגור את המערך עם פתרון נוגדנים משני עבור 30 דקות ב 4 ° C מוגן מפני האור. הסר את הפתרון נוגדנים משני לשטוף את השקופית microarray 3 פעמים במשך 5 דקות על 4 מעלות צלזיוס עם PBS, pH 7.6. לִטבּוֹלMicroarray בצינור חרוטי 50 מ"ל המכיל 0.1x PBS, pH 7.6 להסיר מלח עודף בטמפרטורת החדר. יבש את השקופית בצנטריפוגה שקופיות microarray בטמפרטורת החדר.
- סרוק את השקופית עם סורק microarray ברזולוציה 25 מיקרומטר או גבוה יותר, בעקבות פרוטוקול הסריקה המומלץ של היצרן microarray סורק.
הערה: אם נותב biotin שכותרתו פלואורסצין קיים, סרוק את הערוץ הירוק (לדוגמה: 488 ננומטר, em: 509 ננומטר) והערוץ המתאים לצבע הנוגדנים המשניים של צבע ניאון פלואורסצנטי, בדרך כלל אדום (לדוגמה: 635 ננומטר, em : 677 ננומטר). מטרת הסריקה היא להשיג קבצי .tif ערוץ יחיד שניתן למזג לקובץ .png יחיד (כמתואר בסעיף 6.1).
6. ניתוח של נתונים Microarray באמצעות ArrayNinja
- הכנת תמונה microarray ממוזג
הערה: מטרתו של סעיף זה היא ליצור קובץ תמונה .png שממזג את שני קבצי .tif החד-ערוציים (שהתקבלו בסעיף 5). זֶההוא פורמט התמונה היחיד התואם את המודול ניתוח ArrayNinja. ההוראות הבאות מייצגות דרך אחת להשיג קובץ תמונה ממוזג. עם זאת, פתרונות אחרים זמינים ( למשל , ImageJ חופשית).- משורת הפקודה במסוף bash של מחשב עם תוכנת ImageMagick התקינה, נווט לתיקיה שמכילה את הקבצים .tif הערוץ היחיד, להעתיק / להדביק את השלבים הבאים, להכות 'Enter' בין כל צעד (6.1.4 - 6.1 .7).
הערה: יש להחליף את שמות הקבצים באותיות רישיות ( לדוגמה , RED_CHANNEL) בשם הקובץ של תמונות השקופיות של microarray. - במידת הצורך, תחילה להפוך את התמונות באמצעות הפקודה 'להמיר INPUT.tif -Negate OUTPUT.tif'. פעולה זו נדרשת אם הסורק חוסך קבצים .tif עם האות הלבן והרקע בשחור.
- המרה -depth 16 RED_CHANNEL.TIF -clone 0 -channel GB-resaluation set 0 -delete 0 out.png 2> error.file.
- המרותErt - עומק 16 CONTROL_CHANNEL.TIF -clone 0-ערוץ RB- הערכה מחדש להגדיר 0 -delete 0 outa.png 2> error.file.
- המרת -depth 16 CONTROL_CHANNEL.TIF -clone 0 -channel RG-Revaluate set 0 -delete 0 outB.png 2> error.file.
- המרת out.png outa.png outB.png -set colorpace RGV -combine merged.png.
הערה: קובץ בשם 'merged.png' יישמר באותה תיקייה כמו קבצי tif המקוריים.
- משורת הפקודה במסוף bash של מחשב עם תוכנת ImageMagick התקינה, נווט לתיקיה שמכילה את הקבצים .tif הערוץ היחיד, להעתיק / להדביק את השלבים הבאים, להכות 'Enter' בין כל צעד (6.1.4 - 6.1 .7).
- כימות נתונים באמצעות ArrayNinja
- פתח ArrayNinja ולחץ על הקישור המתאים מדפסת microarray תחת "כדי לכמת תמונות אשר יש צלחת מקור ידוע." הקלד את שם עיצוב השקפים שנשמרו (שלב 2.5) בתיבת הדו-שיח "טעינת עומס" ולחץ על 'Enter'. ראה איור 4 עבור צילום מסך של מודול ניתוח ArrayNinja.
- לחץ על 'בחר קובץ' וניווט ובחר את הקובץ 'merged.png' שנוצר בסעיף 6.1. הדמיון הממוזגE יהיה לטעון כמו גם רשת עבור פריסת השקופית.
- השתמש במחוונים בחלק התחתון של לוח הבקרה ArrayNinja כדי להתאים את הניגודיות, את הבהירות ואת נקודת האמצע.
הערה: התאמות אלה מיועדות להדמיה בלבד ואין להן השפעה על הכימות. - בחר "רזולוציה" והזן את הערך התואם את הרזולוציה של התמונה הסרוקה. השתמש "בצד השוליים" ו "שולי הדף" כדי להזיז את הרשת לתוך יישור עם כתמים על המערך. התאם לפי הצורך כדי לקבל את הרשת כפי מיושר מקרוב על כל נקודה ככל האפשר.
- לחץ על 'חיפוש ספוט' והמתן עד שהלחצן יחזור לצבע האפור המקורי.
הערה: ניתן לחזור על זה מספר פעמים כדי למרכז את עיגולי הרשת בנקודות מסוימות. הפונקציה Seek מרגיעה את הרשת לעבר כל נקודה כדי לכוונן את היישור. - שנה את הערך "Super Arrays" ל - "1" כדי לעבוד על כל לוח מערך משנה בנפרד (אם תרצה). אם באמצעות 4X 12 שעווה טביעת פריסה שקופית ( איור 3E ), להשתמש "iPin" "jPin" ו "subA" שולטת כדי לכבות תכונות לנתח את כל השילוב הרצוי של 4 x 12 בארות. לדוגמה, כדי לנתח את בארות ימין למעלה ולמטה העליון כמו משכפלים זה לזה, הזן "1 4" לתוך iPin, "1 1" לתוך jPin ו "1 4" לתוך תת. לחץ אנטר'.
- העבר את העכבר מעל נקודות מסוימות כדי להציג את הזהות של תכונה זו.
- לחץ על 'החלף זום' כדי לסקור תכונות ביתר קפדנות. בחר נקודות התייחסות הרקע על ידי לחיצה על מקש 'R' בזמן העכבר הוא מעל נקודה. נקודות ייחוס יודגשו בכתום.
הערה: במצב "מעבר לזום", תמונה מוגדלת של התכונה שהעכבר נגמר מוצגת בפינה הימנית העליונה. דיון מפורט של תכונות נוספות ברקע תיקון ArrayNinja נדון במקומות אחרים 28 . - החלף את המקומות (עם מורפולוגיה ספורטיבית מגוונת או פסולת שתשפיע על כימות מדויק) על ידי לחיצה על מקש 'A' תוך כדי ריחוף העכבר מעל הנקודות המושפעות. כתמים בלתי פעילים יהפכו לבנים.
- לחץ על 'מאכלס' ולאחר מכן על 'שלח' כדי לכמת את הנקודות.
הערה: כרטיסייה חדשה תפתח ותציג תרשים עמודות של נתונים מנורמל לממוצע הנקודה הבהיר ביותר. טבלה מתחת לתרשים העמודות מכילה הן את ערכי הנתונים המנורמלים והן את הנתונים הגולמיים. ניתן להעתיק טבלה זו לגיליון אלקטרוני לצורך אחסון בארכיון וניתוח נוסף.
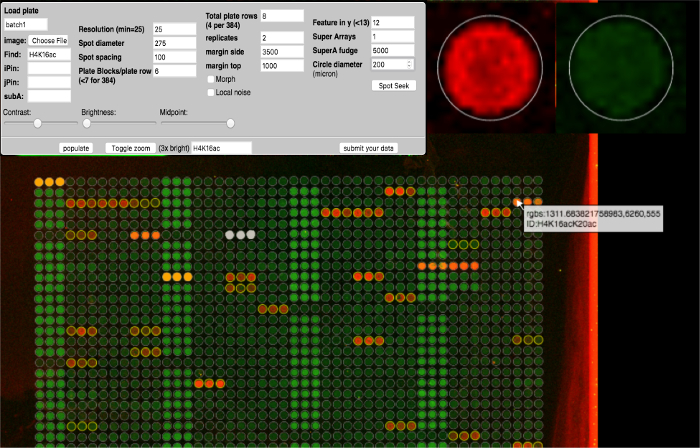
איור 4: מודול ניתוח ArrayNinja. צילום מסך של מודול ניתוח ArrayNinja מוצג. לוח הבקרה (משמאל למעלה) מציג את כל הפרמטרים שניתן לכוונן כדי להציג את המערך, למצוא נקודות, וליישר רשת מעלתמונת מערך. מעבר עם העכבר מעל לתכונה מציג תצוגה מוגדלת (מימין למעלה) ומציג חלון קופץ שמכיל את פרטי הזיהוי המשויכים לתכונה זו (למטה). נקודות ייחוס שנבחרו עבור תיקון רקע הן כתום. תכונות להיות מחוץ לניתוח downstream הם לבנים. ArrayNinja מכיל תכונת חיפוש מבוססת טקסט המדגישה תכונות תואמות בצהוב, כפי שמוצג בדוגמה עבור H4K16. אנא לחץ כאן כדי להציג גרסה גדולה יותר של דמות זו.
תוצאות
פרוטוקול זה שימש לעיצוב לפברק פלטפורמת microarray פפטיד לניתוח של סגוליות נוגדנים PTM היסטון. מערך שאילתות ספריה של יותר מ -300 תכונות פפטיד ייחודי (20 - 40 שאריות באורך) המייצג רבים של שילובים ידוע של PTMs למצוא על הליבה חלבונים היסטון חלופה 38 . צינו...
Discussion
אמינות נוגדנים ביישומים רפואיים ביו-רפואיים היא בעלת חשיבות עליונה 46 , 47 . זה נכון במיוחד בביוכימיה הכרומטין בהתחשב בעמדה של נוגדנים כמו כלי מפתח עבור רוב הטכניקות שפותחו כדי לאפיין את שפע והפצה של PTMs היסטון. הפרוטוקול המוצג כאן פרטים ?...
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
עבודה זו נתמכה בחלקו על ידי מכון המחקר ואן אנדל ומענק מחקר של המכונים הלאומיים לבריאות (CA181343) כדי SBR
Materials
| Name | Company | Catalog Number | Comments |
| Printing Buffer | ArrayIt | PPB | |
| BSA | Omnipure | 2390 | |
| Streptavidin-coated glass microscope slides | Greiner Bio-one | 439003-25 | |
| polypropylene 384 well plate | Greiner Bio-one | 784201 | |
| Biotin-fluorescein | Sigma | 53608 | |
| contact microarray printer | Aushon | 2470 | Aushon 2470 Microarray Printer |
| contact microarray printer | Gene Machines | OmniGrid 100 | OmniGrid Microarray Printer |
| PBS | Invitrogen | 14190 | |
| Blocking Buffer | ArrayIt | SBB | |
| Hydrophobic wax pen | Vector Labs | H-4000 | ImmEdge Hydrophobic Barrier PAP Pen |
| Silicon Gasket | Grace Bio-labs | 622511 | |
| Hybridization Vessel | Thermo Scientific | 267061 | or similar vessel |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21244 | Alexa Fluor 647 (anti-rabbit) |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21235 | Alexa Fluor 647 (anti-mouse) |
| Wax Imprinter | ArrayIt | MSI48 | |
| Tween-20 | Omnipure | 9490 | |
| Microarray Scanner | Innopsys | InnoScan 1100AL | or equivalent microarray scanner |
| EipTitan Histone Peptide Microarray | Epicypher | 112001 | |
| AbSurance Pro Histone Peptide Microarray | Millipore | 16668 | |
| MODified Histone Peptide Array | Active Motif | 13001 | |
| Histone Code Peptide Microarrays | JPT | His_MA_01 | |
| Wax | Royal Oak | GulfWax | for wax imprinter |
| Humidified Microarray Slide Hybridization Chamber | VWR | 97000-284 | |
| High throughput microscope slide washing chamber | ArrayIt | HTW | |
| Microscope slide centrifuge | VWR | 93000-204 | |
| Antibody 1 | Abcam | 8898 | |
| Antibody 2 | Millipore | 07-473 | |
| Biotinylated histone peptide | EpiCypher | 12-0001 | Example peptide. Similar peptides with various modifications are available from several commercial sources. |
| ImageMagick | https://www.imagemagick.org/script/index.php | ||
| ArrayNinja | https://rothbartlab.vai.org/tools/ |
References
- van Steensel, B. Chromatin: constructing the big picture. EMBO J. 30 (10), 1885-1895 (2011).
- Kouzarides, T. Chromatin modifications and their function. Cell. 128 (4), 693-705 (2007).
- Rothbart, S. B., Strahl, B. D. Interpreting the language of histone and DNA modifications. Biochim Biophys Acta. 1839 (8), 627-643 (2014).
- Shogren-Knaak, M., Ishii, H., Sun, J. -. M., Pazin, M. J., Davie, J. R., Peterson, C. L. Histone H4-K16 acetylation controls chromatin structure and protein interactions. Science. 311 (5762), 844-847 (2006).
- Musselman, C. A., Lalonde, M. -. E., Côté, J., Kutateladze, T. G. Perceiving the epigenetic landscape through histone readers. Nat Struct Mol Biol. 19 (12), 1218-1227 (2012).
- Huang, H., Sabari, B. R., Garcia, B. A., Allis, C. D., Zhao, Y. SnapShot: Histone Modifications. Cell. 159 (2), 458 (2014).
- Strahl, B. D., Allis, C. D. The language of covalent histone modifications. Nature. 403 (6765), 41-45 (2000).
- Rothbart, S. B., Krajewski, K., et al. Association of UHRF1 with methylated H3K9 directs the maintenance of DNA methylation. Nat Struct Mol Biol. 19 (11), 1155-1160 (2012).
- Wang, Z., Zang, C., et al. Combinatorial patterns of histone acetylations and methylations in the human genome. Nat Genet. 40 (7), 897-903 (2008).
- Stunnenberg, H. G., Hirst, M. The International Human Epigenome Consortium: A Blueprint for Scientific Collaboration and Discovery. Cell. 167 (5), 1145-1149 (2016).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Egelhofer, T. A., Minoda, A., et al. An assessment of histone-modification antibody quality. Nat Struct Mol Biol. 18 (1), 91-93 (2011).
- Bock, I., Dhayalan, A., Kudithipudi, S., Brandt, O., Rathert, P., Jeltsch, A. Detailed specificity analysis of antibodies binding to modified histone tails with peptide arrays. Epigenetics. 6 (2), 256-263 (2011).
- Busby, M., Xue, C., et al. Systematic comparison of monoclonal versus polyclonal antibodies for mapping histone modifications by ChIP-seq. Epigenetics Chromatin. 9, 49 (2016).
- Fuchs, S. M., Krajewski, K., Baker, R. W., Miller, V. L., Strahl, B. D. Influence of combinatorial histone modifications on antibody and effector protein recognition. Curr Biol. 21 (1), 53-58 (2011).
- Kungulovski, G., Jeltsch, A. Quality of histone modification antibodies undermines chromatin biology research. F1000Research. 4, 1160 (2015).
- Rothbart, S. B., Dickson, B. M., et al. An Interactive Database for the Assessment of Histone Antibody Specificity. Mol Cell. 59 (3), 502-511 (2015).
- Rothbart, S. B., Lin, S., et al. Poly-acetylated chromatin signatures are preferred epitopes for site-specific histone H4 acetyl antibodies. Sci Rep. 2, 489 (2012).
- Berger, M. F., Bulyk, M. L. Universal protein-binding microarrays for the comprehensive characterization of the DNA-binding specificities of transcription factors. Nat Protoc. 4 (3), 393-411 (2009).
- Hu, S., Wan, J., et al. DNA methylation presents distinct binding sites for human transcription factors. eLife. 2, e00726 (2013).
- Moore, C. D., Ajala, O. Z., Zhu, H. Applications in high-content functional protein microarrays. Curr Opin Chem Biol. 30, 21-27 (2016).
- MacBeath, G., Schreiber, S. L. Printing proteins as microarrays for high-throughput function determination. Science. 289 (5485), 1760-1763 (2000).
- Rothbart, S. B., Krajewski, K., Strahl, B. D., Fuchs, S. M. Peptide microarrays to interrogate the "histone code" . Methods Enzymol. 512, 107-135 (2012).
- Cornett, E. M., Dickson, B. M., et al. Substrate Specificity Profiling of Histone-Modifying Enzymes by Peptide Microarray. Methods Enzymol. 574, 31-52 (2016).
- Nady, N., Min, J., Kareta, M. S., Chédin, F., Arrowsmith, C. H. A SPOT on the chromatin landscape? Histone peptide arrays as a tool for epigenetic research. Trends Biochem Sci. 33 (7), 305-313 (2008).
- Dieker, J., Berden, J. H., et al. Autoantibodies against Modified Histone Peptides in SLE Patients Are Associated with Disease Activity and Lupus Nephritis. PLoS ONE. 11 (10), (2016).
- Price, J. V., Tangsombatvisit, S., et al. "On silico" peptide microarrays for high-resolution mapping of antibody epitopes and diverse protein-protein interactions. Nat Med. 18 (9), 1434-1440 (2012).
- Dickson, B. M., Cornett, E. M., Ramjan, Z., Rothbart, S. B. ArrayNinja: An Open Source Platform for Unified Planning and Analysis of Microarray Experiments. Methods Enzymol. 574, 53-77 (2016).
- Gatchalian, J., Fütterer, A., et al. Dido3 PHD modulates cell differentiation and division. Cell Rep. 4 (1), 148-158 (2013).
- Cai, L., Rothbart, S. B., et al. An H3K36 methylation-engaging Tudor motif of polycomb-like proteins mediates PRC2 complex targeting. Mol Cell. 49 (3), 571-582 (2013).
- Rothbart, S. B., Dickson, B. M., et al. Multivalent histone engagement by the linked tandem Tudor and PHD domains of UHRF1 is required for the epigenetic inheritance of DNA methylation. Genes Dev. 27 (11), 1288-1298 (2013).
- Ali, M., Rincón-Arano, H., et al. Molecular basis for chromatin binding and regulation of MLL5. Proc Natl Acad Sci U S A. 110 (28), 11296-11301 (2013).
- Kinkelin, K., Wozniak, G. G., Rothbart, S. B., Lidschreiber, M., Strahl, B. D., Cramer, P. Structures of RNA polymerase II complexes with Bye1, a chromatin-binding PHF3/DIDO homologue. Proc Natl Acad Sci U S A. 110 (38), 15277-15282 (2013).
- Klein, B. J., Piao, L., et al. The histone-H3K4-specific demethylase KDM5B binds to its substrate and product through distinct PHD fingers. Cell Rep. 6 (2), 325-335 (2014).
- Kim, H. -. S., Mukhopadhyay, R., et al. Identification of a BET family bromodomain/casein kinase II/TAF-containing complex as a regulator of mitotic condensin function. Cell Rep. 6 (5), 892-905 (2014).
- Greer, E. L., Beese-Sims, S. E., et al. A histone methylation network regulates transgenerational epigenetic memory in C. elegans. Cell Rep. 7 (1), 113-126 (2014).
- Andrews, F. H., Tong, Q., et al. Multivalent Chromatin Engagement and Inter-domain Crosstalk Regulate MORC3 ATPase. Cell Rep. 16 (12), 3195-3207 (2016).
- Sidoli, S., Lin, S., Karch, K. R., Garcia, B. A. Bottom-Up and Middle-Down Proteomics Have Comparable Accuracies in Defining Histone Post-Translational Modification Relative Abundance and Stoichiometry. Anal Chem. 87 (6), 3129-3133 (2015).
- Tsukada, Y., Ishitani, T., Nakayama, K. I. KDM7 is a dual demethylase for histone H3 Lys 9 and Lys 27 and functions in brain development. Genes Dev. 24 (5), 432-437 (2010).
- Tachibana, M., Sugimoto, K., Fukushima, T., Shinkai, Y. Set domain-containing protein, G9a, is a novel lysine-preferring mammalian histone methyltransferase with hyperactivity and specific selectivity to lysines 9 and 27 of histone H3. J Biol Chem. 276 (27), 25309-25317 (2001).
- Wu, H., Chen, X., et al. Histone methyltransferase G9a contributes to H3K27 methylation in vivo. Cell Res. 21 (2), 365-367 (2011).
- Koch, C. M., Andrews, R. M., et al. The landscape of histone modifications across 1% of the human genome in five human cell lines. Genome Res. 17 (6), 691-707 (2007).
- Okitsu, C. Y., Hsieh, J. C. F., Hsieh, C. -. L. Transcriptional Activity Affects the H3K4me3 Level and Distribution in the Coding Region. Mol Cell Biol. 30 (12), 2933-2946 (2010).
- Zentner, G. E., Tesar, P. J., Scacheri, P. C. Epigenetic signatures distinguish multiple classes of enhancers with distinct cellular functions. Genome Res. 21 (8), 1273-1283 (2011).
- Garske, A. L., Oliver, S. S., et al. Combinatorial profiling of chromatin binding modules reveals multisite discrimination. Nat Chem Biol. 6 (4), 283-290 (2010).
- Baker, M. Reproducibility crisis: Blame it on the antibodies. Nature. 521 (7552), 274-276 (2015).
- Bradbury, A., Plückthun, A. Reproducibility: Standardize antibodies used in research. Nature. 518 (7537), 27-29 (2015).
- Nguyen, U. T. T., Bittova, L., et al. Accelerated chromatin biochemistry using DNA-barcoded nucleosome libraries. Nat Methods. 11 (8), 834-840 (2014).
- Frank, R. Spot-synthesis: an easy technique for the positionally addressable, parallel chemical synthesis on a membrane support. Tetrahedron. 48 (42), 9217-9232 (1992).
- Hilpert, K., Winkler, D. F. H., Hancock, R. E. W. Peptide arrays on cellulose support: SPOT synthesis, a time and cost efficient method for synthesis of large numbers of peptides in a parallel and addressable fashion. Nat Protoc. 2 (6), 1333-1349 (2007).
- Kudithipudi, S., Kusevic, D., Weirich, S., Jeltsch, A. Specificity analysis of protein lysine methyltransferases using SPOT peptide arrays. J Vis Exp. (93), e52203 (2014).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved