Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Histon Antikor Özgüllüğünün Peptit Mikroarraylerle Analizi
Bu Makalede
Özet
Bu el yazısı, histonları ve bunların translasyon sonrası modifikasyonlarını tanıyan antikorların spesifiklik profiline peptid mikroarray teknolojisinin uygulanması için yöntemleri açıklamaktadır.
Özet
Histon proteinleri üzerindeki translasyon sonrası modifikasyonlar (PTM'ler), kromatin yapısını ve gen ekspresyonunu düzenleme rolleri için geniş çapta incelenmektedir. Histon PTM'lerine spesifik antikorların seri olarak üretilmesi ve dağıtımı, bu işaretler üzerinde araştırmayı büyük ölçüde kolaylaştırdı. Histon PTM antikorları, birçok kromatin biyokimya uygulaması için önemli reaktifler olduğundan, doğru veri yorumlaması ve sahadaki ilerlemenin devam etmesi için antikor özgüllüğünün titiz analizi gereklidir. Bu protokol, histone antikorlarının spesifikliğini profillemek için peptit mikrodizilerinin tasarım, imalat ve kullanımı için entegre bir boru hattı tarif eder. Bu prosedürün tasarım ve analiz yönleri ArrayNinja tarafından kolaylaştırılmış ve açık kaynaklı ve etkileşimli bir yazılım paketi, son zamanlarda mikroarray baskı formatlarının uyarlanmasını hızlandırmak için geliştirdi. Bu boru hattı, piyasada bulunan ve yaygın olarak kullanılan histon PTM antibodisinin çok sayıda taranması için kullanılmıştırS ve bu deneylerden elde edilen veriler çevrimiçi ve genişletici bir Histon Antikor Özgüllilik Veritabanı aracılığıyla serbestçe elde edilebilir. Histonların ötesinde, burada açıklanan genel metod, PTM'ye spesifik antikorların analizine genel olarak uygulanabilir.
Giriş
Genomik DNA, kromatin oluşturmak için ökaryotik hücre çekirdeğinin içinde histon proteinleri ile zarif bir şekilde paketlenmiştir. Kromatin tekrarlayan altbirimi, H2A, H2B, H3 ve H4 1 oktamerik bir çekirdek histon proteinleri etrafında sarılan 147 baz çift DNA'dan oluşan nükleozomdur. Kromatin genel olarak gevşek biçimde paketlenmiş ökromatin ve yoğun sıkıştırılmış heterokromatin alanlar halinde düzenlenir. Kromatin sıkıştırma derecesi, protein makineleri, kopyalama, kopyalama ve onarım gibi temel DNA şablonlu işlemleri gerçekleştirmek için altta yatan DNA'ya ne ölçüde erişebildiğini düzenler.
Genetik erişilebilirliğin kromatin bağlamındaki kilit düzenleyicileri histon proteinlerinin yapılandırılmamış kuyruk ve çekirdek alanlarındaki 2 , 3 PTM'leridir. Histon PTM'leri doğrudan kromatin 4'ün yapısını etkiler ve dolaylı olarak işlev görürlerh okuyucu proteinleri ve kromatin remodeling, enzimatik ve iskele aktivitelerini 5 sahip ilişkili makromoleküler kompleksler işe. Son iki on yılda histon PTM fonksiyonu üzerine yapılan çalışmalar, bu işaretlerin hücre akıbetini, organizma gelişimini ve hastalık başlatma / ilerlemesini düzenleyen önemli rol oynadığını göstermektedir. Kütle spektrometresi tabanlı proteomik teknolojideki ilerlemelerle beslenen 80'den fazla farklı histon artıkları üzerinde 20'den fazla benzersiz histon PTM'si keşfedildi 6 . Özellikle, bu modifikasyonlar sıklıkla kombinasyon halinde ortaya çıkmakta ve "histon kodu" hipotezi ile tutarlı bir şekilde sayısız araştırma, histon PTM'lerinin 7 , 8 , 9'un spesifik kombinasyonlarının tanınması yoluyla okuyucu proteinlerinin farklı kromatin bölgelerini hedef aldığını göstermektedir. Önemli bir zorluk, işlevleri GrHistone PTM'lerinin listesine bağlı olarak ve belirli histone PTM kombinasyonlarının kromatin ile ilişkili dinamik fonksiyonları nasıl düzenlediğini belirlemek için kullandı.
Antikorlar, histon PTM'lerinin tespiti için linç pinin reaktifleridir. Bu nedenle, kromatin biyokimya araştırmasında kullanılmak üzere 1.000'den fazla histon PTM'ye spesifik antikor ticari olarak geliştirilmiştir. Yüksek verimli DNA dizileme teknolojisinin hızlı gelişimi ile bu reaktifler, ChIP-seq'deki bireysel araştırmacılar ve büyük ölçekli epigenomik "yol haritası" girişimleri ( örn. , ENCODE ve BLUEPRINT) tarafından geniş ölçüde kullanılmaktadır (kromatin immüno çökeltme, yeni nesil sıralama ) Histonun yüksek çözünürlüklü mekânsal haritalarını üretmek için boru hatları PTM dağıtım genomu genişliği 10 , 11 . Bununla birlikte, son çalışmalar histon PTM antikorlarının özgüllüğünün oldukça değişken olduğunu ve bu reaktiflerin unf Hedef dışı epitop tanıma, komşu PTM'ler tarafından güçlü pozitif ve negatif etki ve belirli bir kalıntı ( örn. , Mono-, di- veya tri-metillisin) 12 , 13 , 14 , 15'deki modifikasyon sırasını ayırt etme zorluğu gibi avorable özellikler , 16 , 17 , 18 . Bu nedenle, bu değerli reaktiflerle üretilen verileri doğru bir şekilde yorumlamak için histon PTM'ye özgü antikor reaktiflerinin titiz kalite kontrolü gereklidir.
Mikroarray teknolojisi, binlerce makromoleküler etkileşimin aynı anda yüksek verimli, tekrarlanabilir ve minyatür formatta sorgulanmasını sağlar. Bu nedenle protein-DNA 19'u analiz etmek için çeşitli mikrodizim platformları yaratılmıştır ,"> 20, protein-protein 21 ve protein-peptid etkileşimlerinin 22. Gerçekten de, histon peptid mikrodizileri yazarlar, silgi yüksek verimli profil sağlayan kromatin biyokimya araştırma için bilgi keşif platformu olarak ortaya çıktı ve histonun okuyucuları PTMS 15 adres , 23 , 24 ve ayrıca histone antikor özgünlüğünün analizi için 17 , 25. Histon peptit dizileri, kromatin ve epigenetik araştırmalarının ötesinde, sistemik lupus eritematosus ve diğer otoimmün hastalıkların teşhis / prognostik testi olarak potansiyel bir kullanıma sahiptir; kromatin otoantikorları, 27 26 oluşturulur.
Burada, tasarım, imalat ve que için geliştirdiğimiz entegre bir boru hattı tanımlıyoruzHistonları ve PTM'lerini tanıyan antikorlar için özgüllük profilleri üretmek için histon peptidi mikrodizileri kullandı. Boru hattı, yakın zamanda geliştirdiğimiz, mikroarray deneylerinin tasarım ve analiz aşamalarını bütünleştiren, açık kaynaklı ve interaktif bir yazılım uygulaması olan ArrayNinja tarafından kolaylaştırıldı 28 . ArrayNinja, Google Chrome'da en iyi şekilde çalışır. Kısacası, streptavidin kaplı cam mikroskop lamlarında belirli bir konumda biyotinle konjuge edilmiş histone peptidlerinin bir kütüphanesinin depolanması için bir robotik temas mikrodizisi yazıcı kullanılır. Ardından diziler, antikor-epitop etkileşimlerini sorgulamak için rekabetçi ve paralel bir tahlil formunda kullanılabilir ( Şekil 1 ). Peptid kütüphanesi, tek başına PTM'leri (lizin asetilasyonu, lisin / arginin metilasyonu ve serin / treonin fosforilasyonu) içeren yüzlerce benzersiz sentetik peptid ve büyük oranda proteomik veri kümelerinden türetilen ilgili kombinasyonlardan oluşur. Peptid sentezi ve geçerliliği için yöntemler Başka yerlerde detaylandırılmıştır 23 . Bu dizilim platformunu kullanarak halen devam eden histon PTM antikor tarama çabalarımızdan elde edilen veriler, halka açık bir web kaynağı, Histone Antikor Özgüllilik Veritabanı (www.histoneantibodies.com) üzerinde arşivlenir. Özellikle, bu protokolün çeşitlemeleri ile imal edilen histon peptit mikroarrayleri, histon PTM okuyucu alanlarının 8 , 29 , 30 , 31 , 32 , 33 , 34 , 35 , 36 , 37 ve daha yakın zamanda profil histonuna aktivitesini karakterize etmek için yaygın bir şekilde kullanılmıştır PTM yazar ve silgi faaliyetleri 24 .
/files/ftp_upload/55912/55912fig1.jpg "/>
Şekil 1: Bir Histone Peptid mikroarrayinde Antikor Taramasının Kademeli Prosedürünün Çizgi Film Tasvirleri. Tanımlanmış translasyon sonrası modifikasyonları (kırmızı ve mavi daireler) barındıran biyotinlenmiş histon peptidleri, streptavidin kaplı cam üzerinde biyotin-floresein ile birlikte basılmıştır. Pozitif etkileşimler kırmızı floresans olarak görselleştirilir. Bu rakamın daha büyük bir versiyonunu görmek için lütfen tıklayınız.
Protokol
1. ArrayNinja'nın Kurulumu ve Çalıştırılması
- Www.virtualbox.org adresinden Oracle Virtual Box'u indirip yükleyin.
- ArrayNinja sanal makinesini (VM) http://research.vai.org/Tools/arrayninja adresinden indirin ve açın.
- Sanal Kutuyu açın ve 'Makine'yi,' 'Ekle'yi tıklayarak ArrayNinja VM'yi ekleyin ve ArrayNinja VM'nin kaydedildiği klasörden arrayninja.vbox'ı seçin.
- ArrayNinja'yı Virtual Box içinde seçip yeşil 'start' okunu tıklayarak başlatın.
- Virtual Box, yeni bir pencere açar ve web tarayıcısında localhost: 2080 adresine giderek VM'ye erişilebileceğini bildiren bir mesaj görüntüler.60; NOT: ArrayNinja bir konteynerle versiyonu hub.docker.com/r/bradley.dickson/arrayninja/ yoluyla da mevcuttur
2. Dizi Slaydı ve Kaynak Pleyt Düzeni Tasarlama
- ArrayNinja arayüzünde 'Plan slayt düzeni' başlığı altında, kullanılan mikro-dizilim yazıcıya karşılık gelen bağlantıyı tıklayın.
NOT: ArrayNinja, yaygın olarak kullanılan iki mikrodizim makinesinin robot hareketini taklit edecek şekilde programlanmıştır (bkz. Tablo1). Diğer diziciler ile uyumluluk isteğe bağlı olarak yapılandırılabilir. - 'Plaka yükle' iletişim kutusununun içine tıklayın, "boş" yazıp "enter" düğmesini tıklayın. ArrayNinja tasarım modülünün ekran görüntüsü için Şekil 2'ye bakın.
- Nokta çapını 275 μm ve nokta aralığı 375 μm olarak ayarlayın. Özelliklerin mikroarray slaytta nasıl görüneceğini özelleştirmek için kalan ayarları (Plaka Blokları / plakalı satır, Toplam plaka Satırları, çoğaltır, y'deki özellikleri, Süper Diziler, SuperA bulanıklığı; bkz. Şekil 2 ) ayarlayın.
NOT: Nokta çapı mikrodizin iğnesinin boyutuna göre belirlenir. Bu ayarlar değiştirildiğinde, karikatür slaytları gerçek zamanlı olarak güncellenecektir. Bu karikatürü, her ayarın nihai slayt yerleşimini nasıl değiştirdiğini önizlemek için kullanın. - Slaytdaki özelliklerin düzenini tamamladıktan sonra, benzersiz her özelliğin üzerine gelin ve açılır iletişim kutusuna özellik tanımlayıcısını girin.
NOT: Özellik tanımlayıcıları sayı, harf veya kombinasyondan oluşabilir. Bu sadece benzersiz özellikler için gereklidir ve çoğaltmalar seçildiğinde bir iletişim kutusu görünmez. - Tüm benzersiz özelliklere bir tanımlayıcı atandıktan sonra, 'doldur' düğmesini tıklayın. Slayt yerleşimi için bir ad girin ve kaydetmek için 'tablonuzu yazdır' ı tıklayın. Seçilen slayt tasarımını yapmak için gereken 384 kuyulu kaynak plakalarının sayısını gösteren yeni bir sayfa ve kaynak plakala (lara) yüklenecek her bir özelliğin fiziksel konumunu gösteren bir tablo açılır.
NOT: Bu ismi hatırla, çünkü mikroarray verilerini analiz ederken bu tasarımı geri çağırmak için kullanılacaktır (bkz. Bölüm 6.2). 'Tablonuzu basmak' tıklandığında düzen ArrayNinja'ya kaydedilir.

Şekil 2: ArrayNinja Tasarım Modülü. Ekran görüntüsü ArrayNinja tasarım modülü noktalı çizgiyle gösterilir. Kontrol paneli (üstte), mikro-dizi yazıcıda değiştirilebilen tüm parametreleri gösterir. Bu parametreler ayarlandığında, slayt düzeninin karikatür resmi (sol alt) gerçek zamanlı olarak güncellenir. Düzen ayarlandıktan sonra, kullanıcı benzersiz özellik tanımlayıcılarını girmek için tek tek noktalar üzerine fareyle dokunabilir. Bu kullanıcı girişinden ArrayNinja yapıları, belirlenen bir mikrodalga slayt düzenini oluşturmak için gereken kaynak plakasındaki her bir parçanın konumunu (sağ alt kısmında) gösteren bir haritayı girer. Bu rakamın daha büyük bir versiyonunu görmek için lütfen tıklayınız.
3. Mikroarray Yapımı
- Kaynak Tabakasını Hazırlama
- 384-kuyulu kaynak plak (lar) ı yaratmak için bölüm 2.5'te ArrayNinja ile üretilen haritayı kullanın.
NOT: Bu platformda sorgulanan peptitlerin ayrıntılı açıklaması başka yerlerde bulunabilirS = "xref"> 17. - Her bir özelliğin 1 - 2 uL'lik ( örn. , Biyotinlenmiş histon peptidi), 384 kuyulu kaynak plakalarının (veyaların) doğru kuyusuna yerleştirilir.
NOT: Biyotinlenmiş histon peptidleri tipik olarak, tek bir diziliş noktasında streptavidin bağlanma bölgelerine peptitten 10 ila 25 misli fazlalık eşdeğer olan 200-400 uM stok solüsyonlarından toplanır. Bu denklem kullanılarak hesaplanır: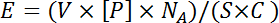
Burada V bir pim tarafından verilen hacim, [ P ] basılan özellik konsantrasyonu, N A Avogadro sayısı ve S bir noktanın yüzey alanıdır. C , birim alan başına streptavidin moleküllerinin sayısı üç ile çarpılarak (mevcut streptavidin bağlanma alanlarının ortalama sayısı olarak ifade edilen) slaydın kapsamıdır. V ve C , ilgili üreticiler tarafından elde edilir. Diğer özelliklerKalabalığın endişe verici olduğu biyomolekülün büyüklüğüne bağlı olarak farklı konsantrasyonlar gerektirir. Her yeni özellik türü için bir dizi baskı yoğunluğu, optimum baskı konsantrasyonunu belirlemek için ampirik olarak test edilmelidir. - Her bir özelliği,% 1 sığır serumu albumin (BSA) ile takviye edilmiş 1x baskı tamponu ile 10 kat sulandırın. Kaynak plakalarını oda sıcaklığında 2 dakika 500 x g'de döndürün.
NOT: Baskı tamponuna flöresanlı etiketli biotin (5 μg / μL) dahil edilmesi, analiz esnasında uygun dizi hizalamasını kolaylaştırmak için lekelenme kontrolü ve görsel bir yardımcı olarak önerilir (bkz. Şekil 4 ).
- 384-kuyulu kaynak plak (lar) ı yaratmak için bölüm 2.5'te ArrayNinja ile üretilen haritayı kullanın.
- Baskı Protokolü ( Şekil 3A - B ).
- Atık toplama kabını boşaltarak ve yıkama çözeltisi kabını ve nemlendirici kabını steril damıtılmış su ile doldurarak dizi düzenleyiciyi hazırlayın.
- Parametrelerini u girinArrayNinja'daki slaydı (bölüm 2 ve Şekil 2 ) mikroarray yazıcı kontrol programına tasarlamak için sed kullandı.
- Mikroarray yazıcı kontrol programını kullanarak yıkama prosedürünü bir batırma ile 1 s yıkamaya ayarlayın. Her yıkamayı takiben pimleri 5 kez tekrar daldıracak şekilde yıkama sonrası ayarlarını yapın. Nemi% 60'a ayarlayın.
NOT: En uygun yıkama konfigürasyonu, kullanılan microarray yazıcısına bağlı olarak değişebilir. En iyi post-yıkama pimi daldırma konfigürasyonu kullanılan mikro-yazıcıya bağlı olarak değişiklik gösterebilir. - Fonksiyonlu slaytları ( örn. , Streptavidin kaplı cam) alt tabaka plakalarına yerleştirin ve tüm plakları plaka asansörüne yerleştirin. Kaynak plakasını / plakalarını tutucuya (lığa) yerleştirin ve kaynak plakası asansöre yerleştirin.
- 'Baskıya tıklayın. Tüm yıkama ve dip ayarlarının doğru olmasını sağlamak için birkaç tur mermi için baskı işlemini izleyin. Yazdırma işlemi tamamlandığında kaldırınAlt tabaka, dizi düzenleyiciden.
NOT: Büyük baskılar, bir gün içinde engelleme adımlarının tamamlanmasını yasakladığında, yazdırılan slaytlar gece boyunca 4 ° C'de nemli bir odada inkübe edilebilir. Slaytları, plastik bir sargı ile mühürlenmiş bir karton kutu içindeki küçük bir su bardağının yanına kuluçkalayın. - Slaytları, karıştırma ile oda sıcaklığında 30 dakika bloke edici tampon ile bloke edin.
- Slaytları 2 x 10 dakika oda sıcaklığında fosfat tamponlu salin (PBS), pH 7.6 içinde karıştırarak yıkayın. Slaytları, oda sıcaklığında 30 saniye mikroarray slayt santrifüjüyle döndürerek kurutun.
NOT: Bir kerede çok sayıda slaydın işlenmesi için, 50 slaytın paralel olarak yıkanmasına izin veren yüksek verimli mikroskoplı bir slayt yıkama odası kullanılabilir. - Balya ile bölünecek şekilde tasarlanmış slaytlar için, bölüm 4.1'e geçin. Diğer tüm tasarımlar için, slaytları 4 ° C'de ışıktan ve nemden koruyarak saklayın.
NOT: Baskılı biyotinlenmiş histon peptidleriBu şekilde depolandığında en az 6 ay boyunca stabildir.
4. Bölme Microarray Slaytları
- Hidrofobik Wax Kalemi ( Şekil 3C )
- Bir mum kalemi kullanarak özellikleri içeren alanların etrafına balmumu uygulayın. Bölüm 5'e geçmeden önce balmumu havada 5 dakika kurumaya bırakın.
NOT: Bloke olduktan sonra, dizi noktaları gözle görselleştirilmesi çok zor olabilir. ArrayNinja'nın slayt tasarımı ölçekli olarak basılabilir ve balmumu uygulama kılavuzu olarak kullanılabilir.
- Bir mum kalemi kullanarak özellikleri içeren alanların etrafına balmumu uygulayın. Bölüm 5'e geçmeden önce balmumu havada 5 dakika kurumaya bırakın.
- Silikon conta ( Şekil 3D )
- Seri contanın arkasındaki berrak filmi soyun ve yapışkan tarafı mikrodizi slaydının üzerine yerleştirin.
- 5. bölüme geçmeden önce contayı 5 saniye tutun.
- Wax Baskısı ( Şekil 3E )
- Balya ile bölünecek şekilde tasarlanan slaytlar için düz cam üzerine% 10 BSA kullanarak bir test slaydı basın. Tüm özelliklerin mum kalıp hazneleri içinde olmasını sağlamak için balmumu imprintör kılavuzlarını veya dizi ayarlarını optimize etmek için bu test slaydını kullanın.
- Mikro dizi balmumu yazıcıyı, tüm mum tamamen eriyene kadar 85 ° C'ye ısıtın, yaklaşık 30 dakika.
- Slaytları basılı taraf aşağı bakacak şekilde yerleştirin ve kaydırma çubuğunu mikroarray yazıcıdaki sağdaki kılavuza tamamen itin. Kalıbı slaydın yüzeyi ile temas ettirmek için kolu yukarı doğru çekin. 2 saniye bekleyin.
NOT: Optimum mum sınırı kalınlığını elde etmek için tutma süresi değiştirilebilir. - Kuyu kapatıldığından ve kenarların o noktalara tırmanacak kadar kalın olmadığından emin olmak için slaydı hızla çıkarın ve mum sınırlarını görsel olarak inceleyin. Slaytları 4 ° C'de nem ve ışıktan koruyarak saklayın.
NOT: Daha kalın veya daha ince kenarlıklar elde etmek için tutma süresi artırılabilir veya azaltılabilir.

Şekil 3: Mikroarray Yapımı. (A) Bir temas mikrodizisi yazıcısı kullanılarak streptavidin kaplı mikroskop lamlarında histoprotein mikroarray üretimi. (B) 48 x 48 ızgara peptit özellikli 3 alt diziyle imal edilen mikroarrayler. (C) 3 subarray'lerin hidrofobik bir balmumu kalemle, (D) silikon yapıştırıcı içeren 2 alt düzen ve (E) 48 balmumu ile subarray'lerin ayrılması. Gösterilen tüm mikrodiziler 25 x 75 mm mikroskop lamı kullanılarak üretilmektedir. Bu rakamın daha büyük bir versiyonunu görmek için lütfen tıklayınız.
5. Bir Histone PTM Antikorunun Peptid Mikroarray ile Hibridlenmesi
- Hibridizasyon tamponu (PBS, pH 7.6,% 5 BSA,% 0.1 Tween-20) hazırlayın.
- Sürgüyü hibridizasyon tamponunda dengeleyinBir hibridizasyon teknesi kullanarak. Hibridizasyon tamponundaki tüm slaytı tamamen kaplayın ve düşük hızda bir orbital çalkalayıcı üzerinde 4 ° C'de 30 dakika inkübe edin.
- Hibridizasyon tamponunda seyreltilmiş histon PTM antikoru içeren bir solüsyon hazırlayın.
NOT: Örnek verilerde, hem histon Antikor # 1 hem Antikor # 2 melezleme tamponunda 1: 1000 oranında seyreltilmiştir. Bir başlangıç noktası olarak immünoblotlama için kullanılana benzer seyreltme aralığı önerilir. - Dizi 4 ° C'de 1 saat boyunca antikor çözeltisi ile inkübe edin. Antikor çözeltisini çıkarın ve diziyi, soğuk PBS, pH 7.6 ile 4 ° C'de 5 dakika 3 kez yıkayın.
- Hibridizasyon tamponunda 1: 5,000 - 1: 10,000 seyreltme floresan boya konjuge sekonder antikor hazırlayın.
- Işık korunan 4 ° C'de 30 dakika ikincil antikor çözümü ile dizi inkübe edin. İkincil antikor çözeltisini çıkarın ve 4 ° C'de PBS, pH 7.6 ile 5 dakika microarray slaydını 3 kez yıkayın. daldırmaMikroarray, oda sıcaklığında fazla miktarda tuzu uzaklaştırmak için 0.1x PBS, pH 7.6 içeren 50 mL'lik konik bir tüpe yerleştirilir. Slayt, oda sıcaklığında bir mikrodizi slayt santrifüjde kurulayın.
- Mikroarray tarayıcı üreticisinin önerilen tarama protokolünü takiben, slaydı 25 μm çözünürlükte veya daha yüksek bir mikrodizil tarayıcı ile tarayın.
NOT: Floreseinle etiketlenmiş biyotin izleyici varsa, hem yeşil kanalı (ör. 488 nm, em: 509 nm) hem de floresan boya konjuge sekonder antikoruna karşılık gelen kanal, genellikle kırmızı (örn. 635 nm, em : 677 nm). Taramanın amacı, tek bir .pif dosyasına birleştirilebilen tek kanallı .tif dosyalar elde etmektir (bölüm 6.1'de açıklandığı gibi).
6. ArrayNinja kullanarak Mikroarray Verilerinin Analizi
- Birleştirilmiş mikro-dizi görüntü hazırlama
Not: Bu bölümün amacı, iki tek kanallı .tif dosyalarını birleştiren bir .png resim dosyası oluşturmaktır (bölüm 5'te elde edilir). BuArrayNinja analiz modülü ile uyumlu tek görüntü formatıdır. Aşağıdaki talimatlar birleştirilmiş bir resim dosyası elde etmek için olası bir yolu temsil etmektedir. Bununla birlikte, diğer çözümler mevcuttur ( örn. , Ücretsiz ImageJ).- Ücretsiz ImageMagick yüklü bir bilgisayarın bash terminalindeki komut satırından, tek kanallı .tif dosyalarını içeren klasöre gidin ve aşağıdaki adımları kopyalayıp yapıştırın ve her adımı (6.1.4 - 6.1) arasında 'enter' tuşuna basarak .7).
NOT: Büyük harflerdeki dosya isimleri ( örn . RED_CHANNEL), mikrodizi slayt görüntülerinin dosya adına değiştirilmelidir. - Gerekirse, önce 'convert INPUT.tif -negate OUTPUT.tif' komutunu kullanarak görüntüleri ters çevirin. Tarayıcı, .tif dosyaları sinyalle birlikte beyaz ve arka plan olarak siyah olarak kaydederse gereklidir.
- Dönüştürmek -depth 16 RED_CHANNEL.TIF -clone 0 -kanal GB-değerlendirmek 0 -delete 0 out.png 2> hata.file.
- dönüşümErt - derinlik 16 CONTROL_CHANNEL.TIF - klibi 0 - kanal RB - değerlendirilme 0 -delete 0 outa.png 2> hata.file.
- Dönüştürmek-derinliği 16 CONTROL_CHANNEL.TIF -clone 0-kanal RG-değerlendirmek set 0 -delete 0 outB.png 2> error.file.
- Out.png outa.png outB.png -set renk aralığı RGV -combine merged.png.
NOT: 'merged.png' adlı bir dosya orijinal .tif dosyalarıyla aynı klasöre kaydedilir.
- Ücretsiz ImageMagick yüklü bir bilgisayarın bash terminalindeki komut satırından, tek kanallı .tif dosyalarını içeren klasöre gidin ve aşağıdaki adımları kopyalayıp yapıştırın ve her adımı (6.1.4 - 6.1) arasında 'enter' tuşuna basarak .7).
- ArrayNinja kullanarak verilerin nicelenmesi
- ArrayNinja'yı açın ve "Bilinen bir kaynak plakasına sahip görüntülerin miktarını belirlemek için" başlığının altındaki uygun mikrodizil yazıcı bağlantısını tıklayın. Kaydedilen slayt tasarımının adını (adım 2.5) "plaka yükleme" iletişim kutusuna yazın ve 'gir'i tıklayın. ArrayNinja analiz modülünün bir ekran görüntüsü için Şekil 4'e bakın.
- 'Dosya Seç' düğmesini tıklayın ve bölüm 6.1'de oluşturulan 'merged.png' dosyasına gidin ve seçin. Birleştirilmiş imgelerSlayt düzeni için bir kılavuz da yüklenecektir.
- Kontrastı, parlaklığı ve orta noktayı ayarlamak için ArrayNinja kontrol panelinin altındaki kaydırıcıları kullanın.
NOT: Bu ayarlamalar yalnızca görsel amaçlıdır ve miktar tayini üzerinde herhangi bir etkisi yoktur. - "Özünürlük" seçin ve taranan görüntünün çözünürlüğü ile eşleşen değeri girin. Izgarayı dizideki noktalarla hizaya getirmek için "marj kenarı" ve "kenar boşluğu" nu kullanın. Izgiyi olabildiğince her noktada hizalamak için gerektiği gibi ayarlayın.
- 'Spot Seek'i tıklayın ve düğme orijinal gri renge dönünceye kadar bekleyin.
NOT: Bu, ızgara dairelerini tek tek noktalarda ortalamak için birkaç kez tekrar edilebilir. Seek işlevi, hizalamada ince ayar yapmak için ızgarayı her bir yere doğru rahatlatır. - Her alt dizi panelini ayrı olarak (eğer isterseniz) çalışmak için "Süper Diziler" değerini "1" olarak değiştirin. 4X 12 mum baskı slayt düzeni ( Şekil 3E ), 4 x 12 kuyucukların herhangi bir kombinasyonunu analiz etmek için özellikleri kapatmak için "iPin" "jPin" ve "subA" kontrollerini kullanın. Örneğin, sol üst ve üst sağ kuyuları birbirlerinin çoğaltmaları olarak analiz etmek için iPin'e "1 4", jPin'e "1 1" ve altA'ya "1 4" girin. Enter'a bas'.
- Bu özelliğin kimliğini görüntülemek için farenizi tek tek noktalar üzerine getirin.
- Özelliklerini daha dikkatli bir şekilde incelemek için 'Yakınlaştırmayı değiştir' düğmesini tıklayın. Fare bir noktanın üzerinde iken 'R' tuşuna basarak arka plan referans noktalarını seçin. Referans noktaları turuncu renkte vurgulanır.
NOT: "toggle zoom" modunda, sağ üst köşede fare üzerinde bulunan özelliğin büyütülmüş bir görüntüsü görüntülenir. ArrayNinja'daki ek arka plan düzeltme özellikleri ile ilgili ayrıntılı bir tartışma, başka yerlerde 28 tartışılmıştır. - Fareyi etkilenen noktaların üzerine getirirken 'A' tuşuna basarak lekeleri (doğru miktarı etkileyebilecek çok çeşitli noktalarda morfoloji veya enkaz ile birlikte) açın. Aktive edilmemiş lekeler beyaza dönecektir.
- Nokta sayısını belirlemek için 'doldur' ve ardından 'Gönder'i tıklayın.
NOT: Yeni bir sekme açılır ve en parlak nokta ortalamasına normalleştirilmiş bir çubuk grafik grafiği görüntülenir. Çubuk grafiğin altında bir tablo normalleştirilmiş ve ham veri değerlerini içerir. Bu tablo, arşivleme ve daha ayrıntılı analiz için bir elektronik tabloya kopyalanabilir.
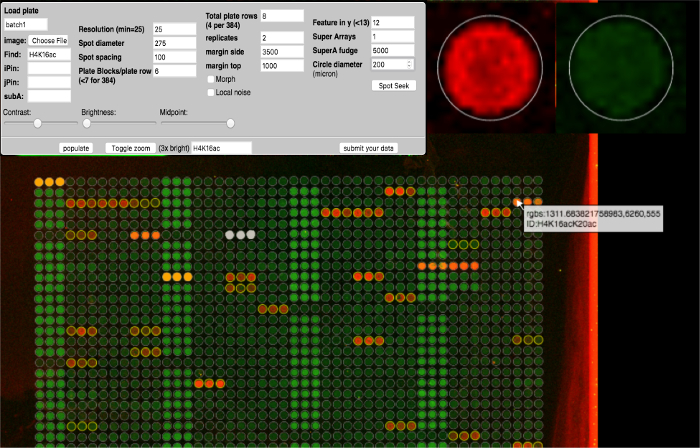
Şekil 4: ArrayNinja Analiz Modülü. ArrayNinja analiz modülünün bir ekran görüntüsü gösterilir. Kontrol paneli (sol üst), diziyi görselleştirmek, lekeler bulmak ve bir ızgaranın üzerine dizmek için ayarlanabilen tüm parametreleri gösterirDizi görüntüsü. Fareyi bir özellik üzerine getirmek, yakınlaştırılmış bir görünümü (sağ üst) gösterir ve o özellikle (altta) ilgili tanımlama bilgisini içeren bir pop-up görüntüler. Arka plan düzeltmesi için seçilen referans noktalar turuncu renktedir. Aşağı akış analizinden çıkarılacak özellikler beyazdır. ArrayNinja, H4K16 örneğinde gösterildiği gibi eşleşen özellikleri sarı olarak vurgulayan, metin tabanlı bir arama özelliğine sahiptir. Bu rakamın daha büyük bir versiyonunu görmek için lütfen tıklayınız.
Sonuçlar
Bu protokol, histone PTM antikor özgünlüğünün analizi için bir peptid mikroarray platformu tasarlamak ve üretmek için kullanılmıştır. Çekirdek ve varyant histon proteinleri 38 üzerinde bulunan PTMS bilinen kombinasyonların birçok temsil - (uzunluğu 40 kalıntıları 20) dizi 300'den benzersiz peptid özellikleri bir kütüphane sorgular. Bu boru hattı, yaygın olarak kullanılan ve piyasada mevcut olan histon PTM antikorlarının taranması ...
Tartışmalar
Biyomedikal araştırma uygulamalarında antikor güvenilirliği en iyisi 46 , 47 . Bu, özellikle histamin PTM'lerinin bolluğu ve dağılımını karakterize etmek için geliştirilen teknikler için anahtar araçlar olarak antikorların bulunduğu kromatin biyokimyasında geçerlidir. Burada sunulan protokol, histon PTM antikor özgünlüğünü analiz etmek için peptit mikrodizilerinin tasarım, imalat ve kullanımı için optimize edilmiş bir boru hat...
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Bu çalışma kısmen Van Andel Araştırma Enstitüsü ve Ulusal Sağlık Enstitüsünden (CA181343) SBR'e verilen bir araştırma ödeneği ile desteklendi
Malzemeler
| Name | Company | Catalog Number | Comments |
| Printing Buffer | ArrayIt | PPB | |
| BSA | Omnipure | 2390 | |
| Streptavidin-coated glass microscope slides | Greiner Bio-one | 439003-25 | |
| polypropylene 384 well plate | Greiner Bio-one | 784201 | |
| Biotin-fluorescein | Sigma | 53608 | |
| contact microarray printer | Aushon | 2470 | Aushon 2470 Microarray Printer |
| contact microarray printer | Gene Machines | OmniGrid 100 | OmniGrid Microarray Printer |
| PBS | Invitrogen | 14190 | |
| Blocking Buffer | ArrayIt | SBB | |
| Hydrophobic wax pen | Vector Labs | H-4000 | ImmEdge Hydrophobic Barrier PAP Pen |
| Silicon Gasket | Grace Bio-labs | 622511 | |
| Hybridization Vessel | Thermo Scientific | 267061 | or similar vessel |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21244 | Alexa Fluor 647 (anti-rabbit) |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21235 | Alexa Fluor 647 (anti-mouse) |
| Wax Imprinter | ArrayIt | MSI48 | |
| Tween-20 | Omnipure | 9490 | |
| Microarray Scanner | Innopsys | InnoScan 1100AL | or equivalent microarray scanner |
| EipTitan Histone Peptide Microarray | Epicypher | 112001 | |
| AbSurance Pro Histone Peptide Microarray | Millipore | 16668 | |
| MODified Histone Peptide Array | Active Motif | 13001 | |
| Histone Code Peptide Microarrays | JPT | His_MA_01 | |
| Wax | Royal Oak | GulfWax | for wax imprinter |
| Humidified Microarray Slide Hybridization Chamber | VWR | 97000-284 | |
| High throughput microscope slide washing chamber | ArrayIt | HTW | |
| Microscope slide centrifuge | VWR | 93000-204 | |
| Antibody 1 | Abcam | 8898 | |
| Antibody 2 | Millipore | 07-473 | |
| Biotinylated histone peptide | EpiCypher | 12-0001 | Example peptide. Similar peptides with various modifications are available from several commercial sources. |
| ImageMagick | https://www.imagemagick.org/script/index.php | ||
| ArrayNinja | https://rothbartlab.vai.org/tools/ |
Referanslar
- van Steensel, B. Chromatin: constructing the big picture. EMBO J. 30 (10), 1885-1895 (2011).
- Kouzarides, T. Chromatin modifications and their function. Cell. 128 (4), 693-705 (2007).
- Rothbart, S. B., Strahl, B. D. Interpreting the language of histone and DNA modifications. Biochim Biophys Acta. 1839 (8), 627-643 (2014).
- Shogren-Knaak, M., Ishii, H., Sun, J. -. M., Pazin, M. J., Davie, J. R., Peterson, C. L. Histone H4-K16 acetylation controls chromatin structure and protein interactions. Science. 311 (5762), 844-847 (2006).
- Musselman, C. A., Lalonde, M. -. E., Côté, J., Kutateladze, T. G. Perceiving the epigenetic landscape through histone readers. Nat Struct Mol Biol. 19 (12), 1218-1227 (2012).
- Huang, H., Sabari, B. R., Garcia, B. A., Allis, C. D., Zhao, Y. SnapShot: Histone Modifications. Cell. 159 (2), 458 (2014).
- Strahl, B. D., Allis, C. D. The language of covalent histone modifications. Nature. 403 (6765), 41-45 (2000).
- Rothbart, S. B., Krajewski, K., et al. Association of UHRF1 with methylated H3K9 directs the maintenance of DNA methylation. Nat Struct Mol Biol. 19 (11), 1155-1160 (2012).
- Wang, Z., Zang, C., et al. Combinatorial patterns of histone acetylations and methylations in the human genome. Nat Genet. 40 (7), 897-903 (2008).
- Stunnenberg, H. G., Hirst, M. The International Human Epigenome Consortium: A Blueprint for Scientific Collaboration and Discovery. Cell. 167 (5), 1145-1149 (2016).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Egelhofer, T. A., Minoda, A., et al. An assessment of histone-modification antibody quality. Nat Struct Mol Biol. 18 (1), 91-93 (2011).
- Bock, I., Dhayalan, A., Kudithipudi, S., Brandt, O., Rathert, P., Jeltsch, A. Detailed specificity analysis of antibodies binding to modified histone tails with peptide arrays. Epigenetics. 6 (2), 256-263 (2011).
- Busby, M., Xue, C., et al. Systematic comparison of monoclonal versus polyclonal antibodies for mapping histone modifications by ChIP-seq. Epigenetics Chromatin. 9, 49 (2016).
- Fuchs, S. M., Krajewski, K., Baker, R. W., Miller, V. L., Strahl, B. D. Influence of combinatorial histone modifications on antibody and effector protein recognition. Curr Biol. 21 (1), 53-58 (2011).
- Kungulovski, G., Jeltsch, A. Quality of histone modification antibodies undermines chromatin biology research. F1000Research. 4, 1160 (2015).
- Rothbart, S. B., Dickson, B. M., et al. An Interactive Database for the Assessment of Histone Antibody Specificity. Mol Cell. 59 (3), 502-511 (2015).
- Rothbart, S. B., Lin, S., et al. Poly-acetylated chromatin signatures are preferred epitopes for site-specific histone H4 acetyl antibodies. Sci Rep. 2, 489 (2012).
- Berger, M. F., Bulyk, M. L. Universal protein-binding microarrays for the comprehensive characterization of the DNA-binding specificities of transcription factors. Nat Protoc. 4 (3), 393-411 (2009).
- Hu, S., Wan, J., et al. DNA methylation presents distinct binding sites for human transcription factors. eLife. 2, e00726 (2013).
- Moore, C. D., Ajala, O. Z., Zhu, H. Applications in high-content functional protein microarrays. Curr Opin Chem Biol. 30, 21-27 (2016).
- MacBeath, G., Schreiber, S. L. Printing proteins as microarrays for high-throughput function determination. Science. 289 (5485), 1760-1763 (2000).
- Rothbart, S. B., Krajewski, K., Strahl, B. D., Fuchs, S. M. Peptide microarrays to interrogate the "histone code" . Methods Enzymol. 512, 107-135 (2012).
- Cornett, E. M., Dickson, B. M., et al. Substrate Specificity Profiling of Histone-Modifying Enzymes by Peptide Microarray. Methods Enzymol. 574, 31-52 (2016).
- Nady, N., Min, J., Kareta, M. S., Chédin, F., Arrowsmith, C. H. A SPOT on the chromatin landscape? Histone peptide arrays as a tool for epigenetic research. Trends Biochem Sci. 33 (7), 305-313 (2008).
- Dieker, J., Berden, J. H., et al. Autoantibodies against Modified Histone Peptides in SLE Patients Are Associated with Disease Activity and Lupus Nephritis. PLoS ONE. 11 (10), (2016).
- Price, J. V., Tangsombatvisit, S., et al. "On silico" peptide microarrays for high-resolution mapping of antibody epitopes and diverse protein-protein interactions. Nat Med. 18 (9), 1434-1440 (2012).
- Dickson, B. M., Cornett, E. M., Ramjan, Z., Rothbart, S. B. ArrayNinja: An Open Source Platform for Unified Planning and Analysis of Microarray Experiments. Methods Enzymol. 574, 53-77 (2016).
- Gatchalian, J., Fütterer, A., et al. Dido3 PHD modulates cell differentiation and division. Cell Rep. 4 (1), 148-158 (2013).
- Cai, L., Rothbart, S. B., et al. An H3K36 methylation-engaging Tudor motif of polycomb-like proteins mediates PRC2 complex targeting. Mol Cell. 49 (3), 571-582 (2013).
- Rothbart, S. B., Dickson, B. M., et al. Multivalent histone engagement by the linked tandem Tudor and PHD domains of UHRF1 is required for the epigenetic inheritance of DNA methylation. Genes Dev. 27 (11), 1288-1298 (2013).
- Ali, M., Rincón-Arano, H., et al. Molecular basis for chromatin binding and regulation of MLL5. Proc Natl Acad Sci U S A. 110 (28), 11296-11301 (2013).
- Kinkelin, K., Wozniak, G. G., Rothbart, S. B., Lidschreiber, M., Strahl, B. D., Cramer, P. Structures of RNA polymerase II complexes with Bye1, a chromatin-binding PHF3/DIDO homologue. Proc Natl Acad Sci U S A. 110 (38), 15277-15282 (2013).
- Klein, B. J., Piao, L., et al. The histone-H3K4-specific demethylase KDM5B binds to its substrate and product through distinct PHD fingers. Cell Rep. 6 (2), 325-335 (2014).
- Kim, H. -. S., Mukhopadhyay, R., et al. Identification of a BET family bromodomain/casein kinase II/TAF-containing complex as a regulator of mitotic condensin function. Cell Rep. 6 (5), 892-905 (2014).
- Greer, E. L., Beese-Sims, S. E., et al. A histone methylation network regulates transgenerational epigenetic memory in C. elegans. Cell Rep. 7 (1), 113-126 (2014).
- Andrews, F. H., Tong, Q., et al. Multivalent Chromatin Engagement and Inter-domain Crosstalk Regulate MORC3 ATPase. Cell Rep. 16 (12), 3195-3207 (2016).
- Sidoli, S., Lin, S., Karch, K. R., Garcia, B. A. Bottom-Up and Middle-Down Proteomics Have Comparable Accuracies in Defining Histone Post-Translational Modification Relative Abundance and Stoichiometry. Anal Chem. 87 (6), 3129-3133 (2015).
- Tsukada, Y., Ishitani, T., Nakayama, K. I. KDM7 is a dual demethylase for histone H3 Lys 9 and Lys 27 and functions in brain development. Genes Dev. 24 (5), 432-437 (2010).
- Tachibana, M., Sugimoto, K., Fukushima, T., Shinkai, Y. Set domain-containing protein, G9a, is a novel lysine-preferring mammalian histone methyltransferase with hyperactivity and specific selectivity to lysines 9 and 27 of histone H3. J Biol Chem. 276 (27), 25309-25317 (2001).
- Wu, H., Chen, X., et al. Histone methyltransferase G9a contributes to H3K27 methylation in vivo. Cell Res. 21 (2), 365-367 (2011).
- Koch, C. M., Andrews, R. M., et al. The landscape of histone modifications across 1% of the human genome in five human cell lines. Genome Res. 17 (6), 691-707 (2007).
- Okitsu, C. Y., Hsieh, J. C. F., Hsieh, C. -. L. Transcriptional Activity Affects the H3K4me3 Level and Distribution in the Coding Region. Mol Cell Biol. 30 (12), 2933-2946 (2010).
- Zentner, G. E., Tesar, P. J., Scacheri, P. C. Epigenetic signatures distinguish multiple classes of enhancers with distinct cellular functions. Genome Res. 21 (8), 1273-1283 (2011).
- Garske, A. L., Oliver, S. S., et al. Combinatorial profiling of chromatin binding modules reveals multisite discrimination. Nat Chem Biol. 6 (4), 283-290 (2010).
- Baker, M. Reproducibility crisis: Blame it on the antibodies. Nature. 521 (7552), 274-276 (2015).
- Bradbury, A., Plückthun, A. Reproducibility: Standardize antibodies used in research. Nature. 518 (7537), 27-29 (2015).
- Nguyen, U. T. T., Bittova, L., et al. Accelerated chromatin biochemistry using DNA-barcoded nucleosome libraries. Nat Methods. 11 (8), 834-840 (2014).
- Frank, R. Spot-synthesis: an easy technique for the positionally addressable, parallel chemical synthesis on a membrane support. Tetrahedron. 48 (42), 9217-9232 (1992).
- Hilpert, K., Winkler, D. F. H., Hancock, R. E. W. Peptide arrays on cellulose support: SPOT synthesis, a time and cost efficient method for synthesis of large numbers of peptides in a parallel and addressable fashion. Nat Protoc. 2 (6), 1333-1349 (2007).
- Kudithipudi, S., Kusevic, D., Weirich, S., Jeltsch, A. Specificity analysis of protein lysine methyltransferases using SPOT peptide arrays. J Vis Exp. (93), e52203 (2014).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır