JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
펩티드 Microarrays를 가진 Histone 항체 특이성의 분석
요약
이 원고는 히스톤 및 그 번역 후 변형을 인식하는 항체의 특이성 프로파일 링에 펩타이드 마이크로 어레이 기술을 적용하는 방법을 설명합니다.
초록
히스톤 단백질에 대한 번역 후 변형 (PTMs)은 염색질 구조 및 유전자 발현 조절에서의 역할에 대해 널리 연구되고있다. 히스톤 PTM에 특이적인 항체의 대량 생산 및 분포는 이러한 표지에 대한 연구를 크게 촉진시켰다. 히스톤 PTM 항체는 많은 염색질 생화학 분야의 주요 시약이기 때문에 항체 특이성의 엄격한 분석이 정확한 데이터 해석과 현장에서의 지속적인 진보에 필요합니다. 이 프로토콜은 히스톤 항체의 특이성을 프로파일 링하기위한 펩타이드 마이크로 어레이의 설계, 제조 및 사용을위한 통합 파이프 라인을 설명합니다. 이 절차의 설계 및 분석 측면은 Microarray 인쇄 형식의 사용자 정의를 간소화하기 위해 최근 개발 한 오픈 소스 및 대화 형 소프트웨어 패키지 인 ArrayNinja를 사용하여 쉽게 수행 할 수 있습니다. 이 파이프 라인은 상업적으로 이용되고 널리 사용되고있는 히스톤 PTM 항체이 실험에서 생성 된 데이터는 온라인 및 확장 Histone Antibody Specificity Database를 통해 자유롭게 사용할 수 있습니다. 히스톤을 넘어, 여기에 기술 된 일반적인 방법론은 PTM 특이 적 항체의 분석에 광범위하게 적용될 수 있습니다.
서문
게놈 DNA는 히스톤 단백질이있는 진핵 세포 핵 내부에 우아하게 포장되어 염색질을 형성합니다. 염색질의 반복적 인 아 단위는 히스톤 단백질 (H2A, H2B, H3 및 H4 1) 의 8 량체 핵 주위에 감싸 진 147 염기쌍의 DNA로 구성된 뉴 소솜 (nucleosome)이다. 크로마 틴은 느슨하게 패킹 된 진균 염색체와 단단히 채워진 헤테로 크로마 틴 도메인으로 광범위하게 구성됩니다. 염색질 압축 정도는 단백질 기계가 복제, 전사 및 수리와 같은 기본적인 DNA 템플릿 공정을 수행하기 위해 기본 DNA에 접근 할 수있는 정도를 조절합니다.
염색체의 맥락에서 게놈 접근성의 주요 조절 인자는 히스톤 단백질 2 , 3 의 구조화되지 않은 꼬리와 핵심 부위의 PTM입니다. 히스톤 PTM은 염색질 4 의 구조에 직접적으로 영향을 미치고 간접적으로 기능합니다.염색질 개질, 효소 및 스캐 폴딩 활동을하는 독자 단백질 및 관련 거대 분자 복합체의 모집 5 . 지난 20 년간 히스톤 PTM 기능에 대한 연구는 세포의 운명, 유기체 발달 및 질병의 시작 / 진행을 조절하는 데 중요한 역할을한다는 것을 압도적으로 제안합니다. 질량 분석 기반 단백체 기술의 발전에 힘 입어, 80 명 이상의 서로 다른 히스톤 잔류 물에 20 개 이상의 고유 한 히스톤 PTMS는 6 발견되었다. 주목할 만하게,이 수정은 수시로 조합에서 생기고, "히스톤 부호"가설과 일치하여, 수많은 학문은 독자 단백질이 히스톤 PTMs의 특정한 조합의 승인을 통해 염색질의 불연속 지역에 표적으로 한 ㄴ다는 것을 건의한다 7 , 8 , 9 . 앞으로 나아갈 핵심 과제는 gr에 함수를 할당하는 것입니다.히스톤 PTM의 목록을 작성하고 히스톤 PTM의 특정 조합이 염색질과 관련된 동적 기능을 조율하는 방법을 결정할 수 있습니다.
항체는 히스톤 PTM의 검출을위한 lynchpin 시약입니다. 이와 같이, 염색질 생화학 연구에 사용하기 위해 1,000 개 이상의 히스톤 PTM 특이 적 항체가 상업적으로 개발되었습니다. 고효율 DNA 시퀀싱 기술의 급속한 발전으로이 시약은 ChIP-seq (염색질 면역 침전 및 차세대 시퀀싱)에서 개별 조사자와 대규모 후성 유전체학 "로드맵"이니셔티브 ( 예 : ENCODE 및 BLUEPRINT)에 광범위하게 사용되고 있습니다 ) 파이프 라인은 히스톤 PTM 분포 게놈 전체의 고해상도 공간 맵을 생성합니다 10 , 11 . 그러나 최근의 연구에 따르면 히스톤 PTM 항체의 특이성은 매우 다양 할 수 있으며 이러한 시약은 unfones 오프 - 타겟 에피토프 인식, 이웃 PTM에 의한 강한 긍정 및 부정적인 영향, 특정 잔기 ( 예 : 모노 -, 디 - 또는 트리 - 메틸 리신)의 변형 순서 식별 어려움과 같은 유용한 특성 12 , 13 , 14 , 15 , 16 , 17 , 18 . 따라서 히스톤 PTM 특이 적 항체 시약의 엄격한 품질 관리가 이러한 유용한 시약으로 생성 된 데이터를 정확하게 해석하는 데 필요합니다.
Microarray 기술은 높은 처리량, 재현성 및 소형화 된 포맷으로 수천 개의 거대 분자 상호 작용을 동시에 조사 할 수있게합니다. 이러한 이유로, 단백질 -DNA 19 을 분석하기 위해 다양한 마이크로 어레이 플랫폼이 만들어졌으며 ,"> (20), 단백질 - 단백질 21 단백질 펩타이드의 상호 작용 (22). 실제로, 히스톤 펩타이드 마이크로 어레이는 작가, 지우개의 높은 처리량 프로파일 링을 가능하게 염색질 생화학 연구에 대한 정보를 검색 플랫폼으로 등장, 그리고 히스톤의 독자 PTMS 15있다 , 23, 24, 또한 히스톤 항체 특이성의 분석을 위해 17, 25. 염색질과 후성 유전학 연구에 자신의 응용 프로그램을 넘어, 히스톤 펩타이드 배열은 전신성 홍 반성 루푸스 등의자가 면역 질환에 대한 진단 / 예후 테스트 잠재적 인 유틸리티가 어디 방지 염색질자가 항체가 생성됩니다 26 , 27 .
여기서는 설계, 제작 및 큐를 위해 개발 한 통합 파이프 라인을 설명합니다.히스톤 및 그 PTM을 인식하는 항체에 대한 특이성 프로파일을 생성하기 위해 히스톤 펩타이드 마이크로 어레이를 제조한다. 파이프 라인은 ArrayNinja, 마이크로 어레이 실험 (28)의 설계 및 분석 단계를 통합하는 오픈 소스, 우리는 최근에 개발 된 대화 형 소프트웨어 응용 프로그램에 의해 촉진된다. ArrayNinja는 Google 크롬에서 가장 잘 작동합니다. 간단히 말하면, 로봇 접촉 마이크로 어레이 프린터를 사용하여 스트렙 타비 딘 - 코팅 된 유리 현미경 슬라이드상의 한정된 위치에 비오틴 - 컨쥬 게이트 된 히스톤 펩타이드의 라이브러리를 침착시킨다. 그런 다음 항체 - 항원 결정기 상호 작용을 조사하기 위해 경쟁적이고 평행 한 분석 형식으로 배열을 사용할 수 있습니다 ( 그림 1 ). 펩타이드 라이브러리는 PTM (라이신 아세틸 화, 라이신 / 아르기닌 메틸화, 및 세린 / 트레오닌 인산화)이있는 수백 가지의 독특한 합성 펩타이드와 프로테오믹스 데이터 세트에서 주로 유래 된 적절한 조합으로 구성됩니다. 펩타이드 합성 및 검증 방법 다른 곳에서 (23)에 자세히 설명되어 있습니다. 이 어레이 플랫폼을 사용하는 진행중인 히스톤 PTM 항체 스크리닝 작업에서 생성 된 데이터는 공개 웹 리소스 인 Histone Antibody Specificity Database (www.histoneantibodies.com)에 보관됩니다. 특히,이 프로토콜의 변형으로 제작 된 히스톤 펩타이드 마이크로 어레이는 히스톤 PTM 판독기 도메인 8 , 29 , 30 , 31 , 32 , 33 , 34 , 35 , 36 , 37 의 활성을 특성화하기 위해 광범위하게 사용되어왔다. PTM 작성자 및 지우개 활동 24 .
/files/ftp_upload/55912/55912fig1.jpg "/>
그림 1 : 히스톤 펩타이드 마이크로 어레이에서 항체 스크리닝을위한 Stepwise 절차의 카툰 묘사. 정의 된 번역 후 변형 (적색 및 청색 원)을 갖는 비 오티 닐화 히스톤 펩타이드는 스트렙 타비 딘 - 코팅 된 유리상에 바이오틴 - 플루오 레세 인과 함께 인쇄된다. 양성 상호 작용은 적색 형광으로 시각화됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
1. ArrayNinja 설치 및 실행
- www.virtualbox.org에서 Oracle Virtual Box를 다운로드하여 설치하십시오.
- http://research.vai.org/Tools/arrayninja에서 ArrayNinja 가상 시스템 (VM)을 다운로드하고 압축을 푸십시오.
- VirtualBox를 열고 'Machine', 'Add'를 클릭하여 ArrayNinja VM을 추가하고 ArrayNinja VM이 저장된 폴더에서 arrayninja.vbox를 선택하십시오.
- 가상 박스에서 ArrayNinja를 선택하고 초록색 '시작'화살표를 클릭하여 ArrayNinja를 시작하십시오.
- Virtual Box는 새 창을 열고 웹 브라우저에서 localhost : 2080으로 이동하여 VM에 액세스 할 수 있다는 메시지를 표시합니다.60; 참고 : ArrayNinja의 컨테이너 버전 hub.docker.com/r/bradley.dickson/arrayninja/를 통해도 가능합니다
2. 배열 슬라이드 및 소스 플레이트 레이아웃 디자인
- ArrayNinja 인터페이스의 '슬라이드 레이아웃 계획'아래에서 사용중인 마이크로 어레이 프린터에 해당하는 링크를 클릭하십시오.
참고 : ArrayNinja는 일반적으로 사용되는 두 개의 마이크로 어레이 프린터의 로봇 움직임을 모방하도록 프로그래밍되었습니다 ( 표 참조).1). 요청에 따라 다른 배열 자와의 호환성을 구성 할 수 있습니다. - 'Load plate'대화 상자 내부를 클릭하고 'empty'를 입력 한 다음 'enter'를 클릭하십시오. ArrayNinja 디자인 모듈의 스크린 샷은 그림 2 를 참조하십시오.
- 스폿 직경을 275 μm로 조정하고 스폿 간격을 375 μm로 조정하십시오. 나머지 설정 (플레이트 블록 / 플레이트 행, 전체 플레이트 행, 복제, y의 기능, 수퍼 어레이, SuperA 퍼지, 그림 2 참조)을 조정하여 기능이 마이크로 어레이 슬라이드에 표시되는 방법을 사용자 정의하십시오.
참고 : 스팟 지름은 마이크로 어레이 핀의 크기에 의해 결정됩니다. 이러한 설정이 조정되면 실시간으로 만화 슬라이드가 업데이트됩니다. 이 만화를 사용하여 각 설정이 최종 슬라이드 레이아웃을 수정하는 방법을 미리 봅니다. - 슬라이드의 피쳐 레이아웃이 완료되면 각 고유 피쳐 위로 마우스를 이동하고 팝업 대화 상자에 피쳐 식별자를 입력하십시오.
참고 : 기능 식별자는 숫자, 문자 또는 조합으로 구성 될 수 있습니다. 이 기능은 고유 기능에만 필요하며 복제를 선택하면 대화 상자가 나타나지 않습니다. - 모든 고유 기능에 식별자가 지정되면 '채우기'를 클릭하십시오. 슬라이드 레이아웃의 이름을 입력하고 '인쇄판 인쇄'를 클릭하여 저장하십시오. 선택한 슬라이드 디자인을 제작하는 데 필요한 384 개의 소스 플레이트 개수와 소스 플레이트에로드 할 각 피쳐의 물리적 위치를 매핑하는 테이블이 표시된 새 페이지가 열립니다.
참고 :이 이름은 마이크로 어레이 데이터를 분석 할 때이 디자인을 불러오는 데 사용되므로이 이름을 기억하십시오 (6.2 절 참조). '인쇄판 인쇄'를 클릭하면 ArrayNinja 내에 레이아웃이 저장됩니다.

그림 2 : ArrayNinja 디자인 모듈. 스크린 샷 ArrayNinja 디자인 모듈은 점선으로 표시됩니다. 제어판 (상단)은 마이크로 어레이 프린터에서 변경할 수있는 모든 매개 변수를 보여줍니다. 이 매개 변수가 조정되면 슬라이드 레이아웃의 만화 이미지 (왼쪽 하단)가 실시간으로 업데이트됩니다. 레이아웃이 설정되면 사용자는 개별 스폿을 마우스로 클릭하여 고유 한 피쳐 식별자를 입력 할 수 있습니다. ArrayNinja는 지정된 마이크로 어레이 슬라이드 레이아웃을 제작하는 데 필요한 소스 플레이트 (오른쪽 아래)의 각 기능 위치 맵을이 사용자 입력으로부터 구성합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
3. 제작 Microarrays
- 소스 플레이트 준비
- 섹션 2.5의 ArrayNinja로 생성 된 맵을 사용하여 384-well source plate를 생성하십시오.
참고 :이 플랫폼에서 질문하는 펩타이드에 대한 자세한 설명은 다른 곳에서 찾을 수 있습니다.s = "xref"> 17. - 1 ~ 2 μL의 각 특징 ( 예 : biotinylated histone peptide)을 384-well source plate (s)의 올바른 well에 넣는다.
참고 : Biotinylated 히스톤 펩타이드는 일반적으로 하나의 배열 자리에 streptavidin 결합 사이트에 펩타이드의 10 ~ 25 배 몰 과량에 해당하는 200 - 400 μm의 주식 솔루션에서 입금됩니다. 이것은 다음 방정식을 사용하여 계산됩니다.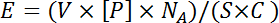
여기서 V 는 핀에 의해 전달되는 부피, [ P ]는 인쇄되는 형상의 농도, N A 는 아보가드로의 수, S 는 스폿의 표면적입니다. C 는 단위 면적당 스트렙 타비 딘 분자의 수에 3을 곱한 (사용 가능한 스트렙 타비 딘 결합 사이트의 평균 수)로 나타낸 슬라이드의 적용 범위입니다. V 와 C 는 각각의 제조업 자에 의해 얻어진다. 다른 기능은군집이 우려되는 생체 분자의 크기에 따라 다른 농도가 필요합니다. 새로운 유형의 피쳐 각각에 대한 인쇄 농도의 범위는 최적의 인쇄 농도를 결정하기 위해 경험적으로 테스트해야합니다. - 1 % 소 혈청 알부민 (BSA)이 보충 된 1x 인쇄 버퍼로 각 기능을 10 배 희석하십시오. 상온에서 2 분 동안 500xg에서 소스 플레이트를 돌립니다.
참고 : 인쇄 버퍼에 fluorescein-labeled biotin (5 μg / μL)을 포함시키는 것이 spotting control 및 분석 중에 알맞은 배열 정렬을 돕기위한 시각 보조 장치로 권장됩니다 ( 그림 4 참조).
- 섹션 2.5의 ArrayNinja로 생성 된 맵을 사용하여 384-well source plate를 생성하십시오.
- 인쇄 프로토콜 ( 그림 3A -B ).
- 폐기물 수집 용기를 비우고 세척 용액 용기와 가습기 용기에 멸균 증류수를 채워서 어레이를 준비하십시오.
- u 매개 변수를 입력하십시오.ArrayNinja (섹션 2 및 그림 2 )의 슬라이드를 마이크로 어레이 프린터 제어 프로그램에 맞게 설계하십시오.
- microarray 프린터 제어 프로그램을 사용하여 한 번의 침지로 1 초 세척을위한 세척 절차를 설정하십시오. 각 세척 후 5 번 핀을 다시 흘리기위한 세척 후 설정을 설정하십시오. 습도를 60 %로 설정하십시오.
참고 : 최적의 세척 구성은 사용되는 마이크로 어레이 프린터에 따라 다를 수 있습니다. 최적의 세척 후 핀 딥 구성은 사용되는 마이크로 어레이 프린터에 따라 다를 수 있습니다. - 기능성 슬라이드 ( 예 : 스트렙 타비 딘 코팅 유리)를 기판 플래 튼에 삽입하고 모든 플래 튼을 플래 턴 엘리베이터에 놓습니다. 소스 플레이트를 플레이트 홀더에 넣고 소스 플레이트 엘리베이터에 넣습니다.
- '인쇄'를 클릭하십시오. 모든 세탁 및 딥 설정이 올바른지 확인하기 위해 몇 차례의 기능 증착을 위해 인쇄 프로세스를 모니터링하십시오. 인쇄 작업이 완료되면기판은 어레이 장치로부터 플래 튼 (platens)된다.
참고 : 큰 인쇄 작업으로 1 일 이내에 차단 단계가 완료되지 않으면 인쇄 된 슬라이드를 가습 챔버에서 4 ° C 밤새 인큐 베이트 할 수 있습니다. 플라스틱 랩으로 밀봉 된 골판지 상자 안의 작은 비이커 옆에 슬라이드를 품습니다. - 섞어 실온에서 30 분 동안 차단 완충액으로 슬라이드를 차단하십시오.
- 슬라이드를 실온에서 인산 완충 식염수 (PBS) (pH 7.6)와 2 분간 혼합하여 씻으십시오. 실온에서 30 초 동안 microarray 슬라이드 원심 분리기에서 회전하여 슬라이드를 건조.
참고 : 한 번에 많은 수의 슬라이드를 처리하기 위해 높은 처리량의 현미경 슬라이드 세척 챔버를 사용하여 50 개의 슬라이드를 동시에 씻을 수 있습니다. - 왁스로 분할되도록 설계된 슬라이드의 경우 4.1 절을 계속 진행하십시오. 다른 모든 디자인의 경우 빛과 습기로부터 보호 된 4 ° C에서 슬라이드를 보관하십시오.
참고 : 인쇄 된 biotinylated 히스톤 펩타이드이런 방식으로 저장하면 적어도 6 개월 동안 안정적입니다.
4. 분할 Microarray 활주
- 소수성 왁스 펜 ( 그림 3C )
- 왁스 펜을 사용하여 피처가 포함 된 영역 주위에 왁스를 바릅니다. 섹션 5로 진행하기 전에 왁스를 5 분간 공기 건조시킵니다.
참고 : 차단 후 어레이 스폿은 눈으로 시각화하기가 매우 어려울 수 있습니다. ArrayNinja의 슬라이드 디자인은 축척으로 인쇄 할 수 있으며 왁스를 적용 할 때 가이드로 사용할 수 있습니다.
- 왁스 펜을 사용하여 피처가 포함 된 영역 주위에 왁스를 바릅니다. 섹션 5로 진행하기 전에 왁스를 5 분간 공기 건조시킵니다.
- 실리콘 가스켓 ( 그림 3D )
- 어레이 가스켓 뒷면의 투명 필름을 떼어 내고 접착면을 마이크로 어레이 슬라이드 위에 놓습니다.
- 5 절을 계속하기 전에 5 초 동안 가스켓을 제 위치에 두십시오.
- 왁스 인쇄물 ( 그림 3E )
- 왁스로 분할하도록 설계된 슬라이드의 경우 10 % BSA를 사용하여 일반 유리에 시험 슬라이드를 인쇄하십시오. 이 테스트 슬라이드를 사용하여 모든 기능이 왁스 몰드 챔버 내에 있는지 확인하기 위해 왁스 임 프린터 가이드 또는 배열 설정을 최적화하십시오.
- 모든 왁스가 완전히 녹을 때까지 약 30 분 동안 마이크로 어레이 왁스 임 프린터를 85 ° C로 가열하십시오.
- 인쇄 된면을 아래로 향하게하여 슬라이드를 삽입하고 슬라이드를 마이크로 어레이 임 프린터의 오른쪽 가이드까지 끝까지 밀어 넣으십시오. 몰드가 슬라이드의 표면에 닿도록 레버를 당깁니다. 2 초간 기다리십시오.
참고 : 최적의 왁스 경계 두께를 달성하기 위해 유지 시간을 변경할 수 있습니다. - 슬라이드를 신속하게 제거하고 왁스 경계를 육안으로 검사하여 모든 우물이 밀폐되어 있고 테두리가 너무 두껍지 않아서 그 지점을 침범 할 수 없도록하십시오. 수분과 빛으로부터 보호되는 4 ° C에서 슬라이드를 보관하십시오.
참고 : 두껍거나 얇은 테두리를 얻으려면 보류 시간을 늘리거나 줄일 수 있습니다.

그림 3 : Microarray Fabrication. 접촉 microarray 프린터를 사용하여 streptavidin - 코팅 현미경 슬라이드 (A) 히스톤 펩티드 microarray 제작. (B) 48 × 48 격자의 펩타이드 특징의 3 개의 서브 어레이로 제조 된 마이크로 어레이. 소수성 왁스 펜으로 (C) 3 개의 서브 어레이, (D) 실리콘 접착제로 2 개의 서브 어레이, (E) 왁스 임프린트로 48 개의 서브 어레이를 분리. 표시된 모든 마이크로 어레이는 25 x 75 mm 현미경 슬라이드를 사용하여 제작됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
5. 히스톤 PTM 항체와 펩타이드 마이크로 어레이의 혼성화
- 하이브 리다이 제이션 버퍼 (PBS, 산도 7.6, 5 % BSA, 0.1 % 트윈 -20)를 준비합니다.
- 하이브리드 화 버퍼에서 슬라이드 평형하이브리드 화 용기를 사용하여. 하이브 리다이 제이션 완충액에서 전체 슬라이드를 완전히 덮고 저속에서 궤도 진탕 기에서 4 ° C 30 분 동안 품을 수 있습니다.
- 하이브 리다이 제이션 버퍼에 희석 히스톤 PTM 항체가 들어있는 용액을 준비합니다.
참고 : 예제 데이터에서 histone Antibody # 1과 Antibody # 2는 하이브 리다이 제이션 버퍼에서 1 : 1,000으로 희석했습니다. 면역 블로 팅에 사용 된 것과 유사한 희석 범위가 출발점으로 권장됩니다. - 4 ° C에서 1 시간 항체 용액으로 어레이를 품어 라. 항체 용액을 제거하고 4 ° C에서 5 분간 3 번 어레이를 3 번 씻으십시오 (차가운 PBS, pH 7.6).
- 하이브 리다이 제이션 버퍼에 형광 염료가 접합 된 2 차 항체의 1 : 5,000 - 1 : 10,000 희석액을 준비하십시오.
- 4 ° C에서 30 분 동안 이차 항체 솔루션과 배열을 품어 ° C 빛으로부터 보호. 2 차 항체 솔루션을 제거하고 PBS, 산도 7.6 4 ° C에서 5 분 3 번 microarray 슬라이드를 씻으십시오. 담그다실온에서 과량의 염을 제거하기 위해 0.1x PBS, pH 7.6을 함유하는 50-mL 원뿔 튜브 내의 마이크로 어레이. 실온에서 microarray 슬라이드 원심 분리기에서 슬라이드를 건조.
- Microarray 스캐너 제조업체의 권장 스캔 프로토콜에 따라 25 μm 이상의 해상도로 마이크로 어레이 스캐너로 슬라이드를 스캔하십시오.
참고 : fluorescein-labeled biotin tracer가있는 경우, 녹색 채널 (예 : 488 nm, 509 nm)과 형광 염료가 결합 된 2 차 항체 (일반적으로 적색 (예 : 635 nm, em) : 677 nm). 스캔의 목적은 단일 .png 파일로 병합 할 수있는 단일 채널 .tif 파일을 얻는 것입니다 (6.1 절에서 설명).
6. ArrayNinja를 이용한 마이크로 어레이 데이터 분석
- 병합 된 마이크로 어레이 이미지 준비하기
참고 :이 섹션의 목표는 두 개의 단일 채널 .tif 파일 (섹션 5에서 얻음)을 병합하는 .png 이미지 파일을 만드는 것입니다. 이ArrayNinja 분석 모듈과 호환되는 유일한 이미지 형식입니다. 다음 지침은 병합 된 이미지 파일을 얻는 한 가지 방법을 나타냅니다. 그러나 다른 솔루션도 사용할 수 있습니다 ( 예 : 프리웨어 ImageJ).- 프리웨어 ImageMagick이 설치된 컴퓨터의 bash 터미널에있는 명령 줄에서 단일 채널 .tif 파일이 들어있는 폴더로 이동하여 각 단계 (6.1.4 - 6.1) 사이에 'enter'키를 눌러 다음 단계를 복사하여 붙여 넣으십시오. .7).
참고 : 대문자로 된 파일 이름 ( 예 : RED_CHANNEL)은 마이크로 어레이 슬라이드 이미지의 파일 이름으로 대체해야합니다. - 필요한 경우 'convert INPUT.tif -negate OUTPUT.tif'명령을 사용하여 이미지를 반전하십시오. 스캐너가 신호가 흰색이고 배경이 검은 색 인 .tif 파일을 저장하는 경우 필요합니다.
- 변환 -depth 16 RED_CHANNEL.TIF -clone 0- 채널 GB - 집합 0 평가 -delete 0 out.png 2> error.file.
- 전환ert - 깊이 16 CONTROL_CHANNEL.TIF - 클론 0 채널 RB - 세트 0 측정 -delete 0 outa.png 2> error.file.
- 변환 - 깊이 16 CONTROL_CHANNEL.TIF - 복제 0 - 채널 RG - 집합 0 평가 -delete 0 outB.png 2> 오류. 파일.
- convert out.png outa.png outB.png -set colorspace RGV -combine merged.png.
참고 : 'merged.png'라는 파일은 원본 .tif 파일과 동일한 폴더에 저장됩니다.
- 프리웨어 ImageMagick이 설치된 컴퓨터의 bash 터미널에있는 명령 줄에서 단일 채널 .tif 파일이 들어있는 폴더로 이동하여 각 단계 (6.1.4 - 6.1) 사이에 'enter'키를 눌러 다음 단계를 복사하여 붙여 넣으십시오. .7).
- ArrayNinja를 사용하여 데이터 정량화
- ArrayNinja를 열고 "알려진 원본 플레이트가있는 이미지를 정량화하려면"에서 적절한 마이크로 어레이 프린터 링크를 클릭하십시오. 로드 플레이트 대화 상자에 저장된 슬라이드 디자인의 이름을 입력하고 (2.5 단계) '입력'을 클릭하십시오. ArrayNinja 분석 모듈의 스크린 샷은 그림 4 를 참조하십시오.
- '파일 선택'을 클릭하고 6.1 절에서 생성 한 'merged.png'파일을 찾아 선택하십시오. 병합 된 이미지e뿐만 아니라 슬라이드 레이아웃에 대한 그리드를로드합니다.
- ArrayNinja 제어판 아래에있는 슬라이더를 사용하여 대비, 밝기 및 중간 점을 조정하십시오.
참고 :이 조정은 시각화 목적으로 만 사용되며 수량화에는 영향을 미치지 않습니다. - "해상도"를 선택하고 스캔 한 이미지의 해상도와 일치하는 값을 입력하십시오. "margin side"와 "margin top"을 사용하여 그리드를 배열의 스팟들과 정렬시킵니다. 가능한 한 각 지점에서 그리드가 밀접하게 정렬되도록 필요한만큼 조정합니다.
- 'Spot Seek'을 클릭하고 버튼이 원래 회색 색상으로 돌아갈 때까지 기다립니다.
참고 :이 작업을 여러 번 반복하여 그리드 원을 개별 스팟의 중심에 배치 할 수 있습니다. Seek 기능은 각 지점을 향해 그리드를 완화하여 정렬을 미세 조정합니다. - "Super Arrays"값을 "1"로 변경하여 각 하위 배열 패널을 개별적으로 작업하십시오 (원하는 경우). 4x 12 왁스 임프린트 슬라이드 레이아웃 ( 그림 3E ), "iPin" "jPin"및 "subA"컨트롤을 사용하여 4 x 12 웰의 원하는 조합을 분석하는 기능을 끄십시오. 예를 들어 왼쪽 위 우물과 우물을 각각 복제물로 분석하려면 iPin에 "1 4", jPin에 "1 1"을 입력하고 subA에 "1 4"를 입력하십시오. 'enter'키를 누릅니다.
- 마우스를 개별 스폿 위로 이동하면 해당 기능의 ID를 볼 수 있습니다.
- 기능을보다 신중하게 검토하려면 '토글 줌'을 클릭하십시오. 마우스가 자리 위에있을 때 'R'키를 눌러 배경 참조 점을 선택하십시오. 참조 점이 주황색으로 강조 표시됩니다.
참고 : "토글 줌"모드에서는 마우스 오른쪽 위에 마우스가있는 기능의 확대 이미지가 표시됩니다. ArrayNinja의 추가 배경 보정 기능에 대한 자세한 설명은 다른 곳에서 28 설명되어 있습니다. - 영향을받는 지점 위로 마우스를 가져 가면서 'A'키를 눌러 스폿을 전환 (정확한 정량화에 영향을주는 광범위하게 변하는 스폿 형태 또는 파편이 있음) 할 수 있습니다. 비활성화 된 스폿은 흰색으로 변합니다.
- 'populate'다음에 'Submit'을 클릭하여 스팟을 정량화하십시오.
참고 : 새 탭이 열리고 가장 밝은 스팟 평균으로 정규화 된 데이터의 막대 그래프가 표시됩니다. 막대 그래프 아래의 표는 정규화 된 값과 원시 데이터 값을 모두 포함합니다. 이 테이블은 보관 및 추가 분석을 위해 스프레드 시트에 복사 할 수 있습니다.
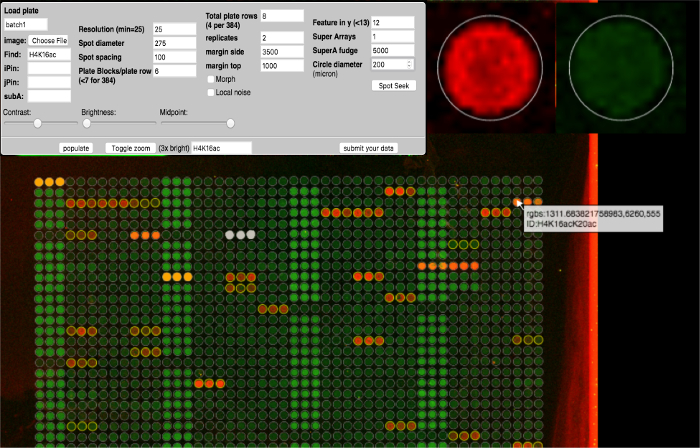
그림 4 : ArrayNinja 분석 모듈. ArrayNinja 분석 모듈의 스크린 샷이 표시됩니다. 제어판 (왼쪽 위)에는 배열을 시각화하고, 반점을 찾고, 그 위에 격자를 정렬하기 위해 조정할 수있는 모든 매개 변수가 표시됩니다배열 이미지. 기능 위로 마우스를 가져 가면 확대 된보기 (오른쪽 위)가 표시되고 해당 기능과 관련된 식별 정보 (아래)가 포함 된 팝업이 표시됩니다. 배경 보정을 위해 선택된 기준점은 주황색입니다. 다운 스트림 분석에서 제외 할 기능은 흰색입니다. ArrayNinja에는 H4K16의 예제와 같이 일치하는 기능을 노란색으로 강조 표시하는 텍스트 기반 검색 기능이 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
이 프로토콜은 히스톤 PTM 항체 특이성의 분석을위한 펩타이드 마이크로 어레이 플랫폼을 설계하고 제조하는데 사용되었습니다. 이 어레이는 핵심 및 변형 히스톤 단백질에서 발견되는 PTM의 많은 조합을 나타내는 300 개 이상의 고유 한 펩타이드 기능 (20-40 잔기 길이) 라이브러리를 조회합니다 38 . 이 파이프 라인은 널리 사용되고 상업적으로 이용 가능?...
토론
생물 의학 연구 분야의 항체 신뢰도가 가장 중요합니다 46 , 47 . 이것은 히스톤 PTM의 풍부성과 분포를 특성화하기 위해 개발 된 대부분의 기술에 대한 핵심 도구로서 항체의 위치를 고려할 때 염색질 생화학에서 특히 그렇습니다. 여기에 제시된 프로토콜은 히스톤 PTM 항체 특이성을 분석하기위한 펩타이드 마이크로 어레이의 설계, 제조 및 사용을?...
공개
저자는 공개 할 것이 없습니다.
감사의 말
이 연구는 반 안델 연구소 (Van Andel Research Institute)와 국립 보건원 (CA181343)에서 SBR
자료
| Name | Company | Catalog Number | Comments |
| Printing Buffer | ArrayIt | PPB | |
| BSA | Omnipure | 2390 | |
| Streptavidin-coated glass microscope slides | Greiner Bio-one | 439003-25 | |
| polypropylene 384 well plate | Greiner Bio-one | 784201 | |
| Biotin-fluorescein | Sigma | 53608 | |
| contact microarray printer | Aushon | 2470 | Aushon 2470 Microarray Printer |
| contact microarray printer | Gene Machines | OmniGrid 100 | OmniGrid Microarray Printer |
| PBS | Invitrogen | 14190 | |
| Blocking Buffer | ArrayIt | SBB | |
| Hydrophobic wax pen | Vector Labs | H-4000 | ImmEdge Hydrophobic Barrier PAP Pen |
| Silicon Gasket | Grace Bio-labs | 622511 | |
| Hybridization Vessel | Thermo Scientific | 267061 | or similar vessel |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21244 | Alexa Fluor 647 (anti-rabbit) |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21235 | Alexa Fluor 647 (anti-mouse) |
| Wax Imprinter | ArrayIt | MSI48 | |
| Tween-20 | Omnipure | 9490 | |
| Microarray Scanner | Innopsys | InnoScan 1100AL | or equivalent microarray scanner |
| EipTitan Histone Peptide Microarray | Epicypher | 112001 | |
| AbSurance Pro Histone Peptide Microarray | Millipore | 16668 | |
| MODified Histone Peptide Array | Active Motif | 13001 | |
| Histone Code Peptide Microarrays | JPT | His_MA_01 | |
| Wax | Royal Oak | GulfWax | for wax imprinter |
| Humidified Microarray Slide Hybridization Chamber | VWR | 97000-284 | |
| High throughput microscope slide washing chamber | ArrayIt | HTW | |
| Microscope slide centrifuge | VWR | 93000-204 | |
| Antibody 1 | Abcam | 8898 | |
| Antibody 2 | Millipore | 07-473 | |
| Biotinylated histone peptide | EpiCypher | 12-0001 | Example peptide. Similar peptides with various modifications are available from several commercial sources. |
| ImageMagick | https://www.imagemagick.org/script/index.php | ||
| ArrayNinja | https://rothbartlab.vai.org/tools/ |
참고문헌
- van Steensel, B. Chromatin: constructing the big picture. EMBO J. 30 (10), 1885-1895 (2011).
- Kouzarides, T. Chromatin modifications and their function. Cell. 128 (4), 693-705 (2007).
- Rothbart, S. B., Strahl, B. D. Interpreting the language of histone and DNA modifications. Biochim Biophys Acta. 1839 (8), 627-643 (2014).
- Shogren-Knaak, M., Ishii, H., Sun, J. -. M., Pazin, M. J., Davie, J. R., Peterson, C. L. Histone H4-K16 acetylation controls chromatin structure and protein interactions. Science. 311 (5762), 844-847 (2006).
- Musselman, C. A., Lalonde, M. -. E., Côté, J., Kutateladze, T. G. Perceiving the epigenetic landscape through histone readers. Nat Struct Mol Biol. 19 (12), 1218-1227 (2012).
- Huang, H., Sabari, B. R., Garcia, B. A., Allis, C. D., Zhao, Y. SnapShot: Histone Modifications. Cell. 159 (2), 458 (2014).
- Strahl, B. D., Allis, C. D. The language of covalent histone modifications. Nature. 403 (6765), 41-45 (2000).
- Rothbart, S. B., Krajewski, K., et al. Association of UHRF1 with methylated H3K9 directs the maintenance of DNA methylation. Nat Struct Mol Biol. 19 (11), 1155-1160 (2012).
- Wang, Z., Zang, C., et al. Combinatorial patterns of histone acetylations and methylations in the human genome. Nat Genet. 40 (7), 897-903 (2008).
- Stunnenberg, H. G., Hirst, M. The International Human Epigenome Consortium: A Blueprint for Scientific Collaboration and Discovery. Cell. 167 (5), 1145-1149 (2016).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Egelhofer, T. A., Minoda, A., et al. An assessment of histone-modification antibody quality. Nat Struct Mol Biol. 18 (1), 91-93 (2011).
- Bock, I., Dhayalan, A., Kudithipudi, S., Brandt, O., Rathert, P., Jeltsch, A. Detailed specificity analysis of antibodies binding to modified histone tails with peptide arrays. Epigenetics. 6 (2), 256-263 (2011).
- Busby, M., Xue, C., et al. Systematic comparison of monoclonal versus polyclonal antibodies for mapping histone modifications by ChIP-seq. Epigenetics Chromatin. 9, 49 (2016).
- Fuchs, S. M., Krajewski, K., Baker, R. W., Miller, V. L., Strahl, B. D. Influence of combinatorial histone modifications on antibody and effector protein recognition. Curr Biol. 21 (1), 53-58 (2011).
- Kungulovski, G., Jeltsch, A. Quality of histone modification antibodies undermines chromatin biology research. F1000Research. 4, 1160 (2015).
- Rothbart, S. B., Dickson, B. M., et al. An Interactive Database for the Assessment of Histone Antibody Specificity. Mol Cell. 59 (3), 502-511 (2015).
- Rothbart, S. B., Lin, S., et al. Poly-acetylated chromatin signatures are preferred epitopes for site-specific histone H4 acetyl antibodies. Sci Rep. 2, 489 (2012).
- Berger, M. F., Bulyk, M. L. Universal protein-binding microarrays for the comprehensive characterization of the DNA-binding specificities of transcription factors. Nat Protoc. 4 (3), 393-411 (2009).
- Hu, S., Wan, J., et al. DNA methylation presents distinct binding sites for human transcription factors. eLife. 2, e00726 (2013).
- Moore, C. D., Ajala, O. Z., Zhu, H. Applications in high-content functional protein microarrays. Curr Opin Chem Biol. 30, 21-27 (2016).
- MacBeath, G., Schreiber, S. L. Printing proteins as microarrays for high-throughput function determination. Science. 289 (5485), 1760-1763 (2000).
- Rothbart, S. B., Krajewski, K., Strahl, B. D., Fuchs, S. M. Peptide microarrays to interrogate the "histone code" . Methods Enzymol. 512, 107-135 (2012).
- Cornett, E. M., Dickson, B. M., et al. Substrate Specificity Profiling of Histone-Modifying Enzymes by Peptide Microarray. Methods Enzymol. 574, 31-52 (2016).
- Nady, N., Min, J., Kareta, M. S., Chédin, F., Arrowsmith, C. H. A SPOT on the chromatin landscape? Histone peptide arrays as a tool for epigenetic research. Trends Biochem Sci. 33 (7), 305-313 (2008).
- Dieker, J., Berden, J. H., et al. Autoantibodies against Modified Histone Peptides in SLE Patients Are Associated with Disease Activity and Lupus Nephritis. PLoS ONE. 11 (10), (2016).
- Price, J. V., Tangsombatvisit, S., et al. "On silico" peptide microarrays for high-resolution mapping of antibody epitopes and diverse protein-protein interactions. Nat Med. 18 (9), 1434-1440 (2012).
- Dickson, B. M., Cornett, E. M., Ramjan, Z., Rothbart, S. B. ArrayNinja: An Open Source Platform for Unified Planning and Analysis of Microarray Experiments. Methods Enzymol. 574, 53-77 (2016).
- Gatchalian, J., Fütterer, A., et al. Dido3 PHD modulates cell differentiation and division. Cell Rep. 4 (1), 148-158 (2013).
- Cai, L., Rothbart, S. B., et al. An H3K36 methylation-engaging Tudor motif of polycomb-like proteins mediates PRC2 complex targeting. Mol Cell. 49 (3), 571-582 (2013).
- Rothbart, S. B., Dickson, B. M., et al. Multivalent histone engagement by the linked tandem Tudor and PHD domains of UHRF1 is required for the epigenetic inheritance of DNA methylation. Genes Dev. 27 (11), 1288-1298 (2013).
- Ali, M., Rincón-Arano, H., et al. Molecular basis for chromatin binding and regulation of MLL5. Proc Natl Acad Sci U S A. 110 (28), 11296-11301 (2013).
- Kinkelin, K., Wozniak, G. G., Rothbart, S. B., Lidschreiber, M., Strahl, B. D., Cramer, P. Structures of RNA polymerase II complexes with Bye1, a chromatin-binding PHF3/DIDO homologue. Proc Natl Acad Sci U S A. 110 (38), 15277-15282 (2013).
- Klein, B. J., Piao, L., et al. The histone-H3K4-specific demethylase KDM5B binds to its substrate and product through distinct PHD fingers. Cell Rep. 6 (2), 325-335 (2014).
- Kim, H. -. S., Mukhopadhyay, R., et al. Identification of a BET family bromodomain/casein kinase II/TAF-containing complex as a regulator of mitotic condensin function. Cell Rep. 6 (5), 892-905 (2014).
- Greer, E. L., Beese-Sims, S. E., et al. A histone methylation network regulates transgenerational epigenetic memory in C. elegans. Cell Rep. 7 (1), 113-126 (2014).
- Andrews, F. H., Tong, Q., et al. Multivalent Chromatin Engagement and Inter-domain Crosstalk Regulate MORC3 ATPase. Cell Rep. 16 (12), 3195-3207 (2016).
- Sidoli, S., Lin, S., Karch, K. R., Garcia, B. A. Bottom-Up and Middle-Down Proteomics Have Comparable Accuracies in Defining Histone Post-Translational Modification Relative Abundance and Stoichiometry. Anal Chem. 87 (6), 3129-3133 (2015).
- Tsukada, Y., Ishitani, T., Nakayama, K. I. KDM7 is a dual demethylase for histone H3 Lys 9 and Lys 27 and functions in brain development. Genes Dev. 24 (5), 432-437 (2010).
- Tachibana, M., Sugimoto, K., Fukushima, T., Shinkai, Y. Set domain-containing protein, G9a, is a novel lysine-preferring mammalian histone methyltransferase with hyperactivity and specific selectivity to lysines 9 and 27 of histone H3. J Biol Chem. 276 (27), 25309-25317 (2001).
- Wu, H., Chen, X., et al. Histone methyltransferase G9a contributes to H3K27 methylation in vivo. Cell Res. 21 (2), 365-367 (2011).
- Koch, C. M., Andrews, R. M., et al. The landscape of histone modifications across 1% of the human genome in five human cell lines. Genome Res. 17 (6), 691-707 (2007).
- Okitsu, C. Y., Hsieh, J. C. F., Hsieh, C. -. L. Transcriptional Activity Affects the H3K4me3 Level and Distribution in the Coding Region. Mol Cell Biol. 30 (12), 2933-2946 (2010).
- Zentner, G. E., Tesar, P. J., Scacheri, P. C. Epigenetic signatures distinguish multiple classes of enhancers with distinct cellular functions. Genome Res. 21 (8), 1273-1283 (2011).
- Garske, A. L., Oliver, S. S., et al. Combinatorial profiling of chromatin binding modules reveals multisite discrimination. Nat Chem Biol. 6 (4), 283-290 (2010).
- Baker, M. Reproducibility crisis: Blame it on the antibodies. Nature. 521 (7552), 274-276 (2015).
- Bradbury, A., Plückthun, A. Reproducibility: Standardize antibodies used in research. Nature. 518 (7537), 27-29 (2015).
- Nguyen, U. T. T., Bittova, L., et al. Accelerated chromatin biochemistry using DNA-barcoded nucleosome libraries. Nat Methods. 11 (8), 834-840 (2014).
- Frank, R. Spot-synthesis: an easy technique for the positionally addressable, parallel chemical synthesis on a membrane support. Tetrahedron. 48 (42), 9217-9232 (1992).
- Hilpert, K., Winkler, D. F. H., Hancock, R. E. W. Peptide arrays on cellulose support: SPOT synthesis, a time and cost efficient method for synthesis of large numbers of peptides in a parallel and addressable fashion. Nat Protoc. 2 (6), 1333-1349 (2007).
- Kudithipudi, S., Kusevic, D., Weirich, S., Jeltsch, A. Specificity analysis of protein lysine methyltransferases using SPOT peptide arrays. J Vis Exp. (93), e52203 (2014).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유