Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Analyse de la spécificité d'anticorps d'histone avec des microarrays de peptides
Dans cet article
Résumé
Ce manuscrit décrit des méthodes pour l'application de la technologie des micro-aigres peptidiques au profilage spécifique des anticorps qui reconnaissent les histones et leurs modifications post-traductionnelles.
Résumé
Les modifications post-traductionnelles (PTM) sur les protéines d'histones sont largement étudiées pour leur rôle dans la régulation de la structure de la chromatine et de l'expression des gènes. La production en masse et la distribution d'anticorps spécifiques aux PTM des histones ont considérablement facilité la recherche sur ces marques. Comme les anticorps PTM des histones sont des réactifs clés pour de nombreuses applications de biochimie de la chromatine, une analyse rigoureuse de la spécificité des anticorps est nécessaire pour une interprétation précise des données et des progrès continus sur le terrain. Ce protocole décrit une pipeline intégrée pour la conception, la fabrication et l'utilisation de microarrays de peptides pour le profilage de la spécificité des anticorps histoniques. Les aspects de conception et d'analyse de cette procédure sont facilités par ArrayNinja, un logiciel logiciel ouvert et interactif que nous avons développé récemment pour rationaliser la personnalisation des formats d'impression microarray. Ce pipeline a été utilisé pour dépister un grand nombre d'antibodie d'histone PTM commercialement disponibles et largement utiliséesS, et les données générées à partir de ces expériences sont disponibles gratuitement via une base de données de spécificité d'anticorps Histone en ligne et en expansion. Au-delà des histones, la méthodologie générale décrite ici peut être largement appliquée à l'analyse des anticorps spécifiques de PTM.
Introduction
L'ADN génomique est emballé élégamment à l'intérieur du noyau de cellules eucaryotes avec des protéines d'histone pour former la chromatine. La sous-unité récurrente de la chromatine est le nucléosome, qui comprend 147 paires de bases d'ADN enroulées autour d'un noyau octamerique de protéines d'histones - H2A, H2B, H3 et H4 1 . La chromatine est globalement organisée en euchromatine lâchement emballée et dans les domaines de l'hétérochromatine étroitement emballés. Le degré de compactage de la chromatine régule la mesure dans laquelle les machines à protéines peuvent accéder à l'ADN sous-jacent pour mener à bien des processus fondés sur l'ADN fondamentaux comme la réplication, la transcription et la réparation.
Les principaux régulateurs de l'accessibilité du génome dans le contexte de la chromatine sont les PTM sur la queue non structurée et les domaines de base des protéines histoniques 2 , 3 . Les PTM d'histone fonctionnent directement en influençant la structure de la chromatine 4 et indirectement à traversH le recrutement de protéines lecteur et leurs complexes macromoléculaires associés qui ont des activités de remodelage de la chromatine, enzymatiques et échafaudages 5 . Les études de la fonction PTM des histones au cours des deux dernières décennies suggèrent largement que ces marques jouent un rôle clé dans la régulation du devenir cellulaire, du développement organique et de l'initiation / progression de la maladie. Alimenté par des progrès dans la technologie protéomique à base de spectrométrie de masse, plus de 20 PTM d'histones uniques sur plus de 80 résidus d'histones distinctes ont été découverts 6 . Notamment, ces modifications se produisent souvent dans des combinaisons et, conformément à l'hypothèse du "code histone", de nombreuses études suggèrent que les protéines de lecteur sont ciblées sur des régions discrètes de la chromatine en reconnaissant des combinaisons spécifiques d'histones PTM 7 , 8 , 9 . Un défi majeur pour aller de l'avant sera d'assigner des fonctions à la grEn raison de la liste des PTM des histones et pour déterminer comment des combinaisons spécifiques de PTM d'histones orchestrent les fonctions dynamiques associées à la chromatine.
Les anticorps sont les réactifs lynchpin pour la détection des PTM des histones. En tant que tel, plus de 1 000 anticorps spécifiques des PTM d'histone ont été commercialement développés pour être utilisés dans la recherche sur la biochimie de la chromatine. Avec le développement rapide de la technologie de séquençage d'ADN à haut débit, ces réactifs sont largement utilisés par des chercheurs individuels et des initiatives de "feuille de route" d'épigénomènes à grande échelle ( p . Ex . ENCODE et BLUEPRINT) dans ChIP-seq (immunoprécipitation de la chromatine couplée au séquençage de la prochaine génération ) Pipelines pour générer des cartes spatiales à haute résolution de la distribution de PTM d'histones dans l'ensemble du génome 10 , 11 . Cependant, des études récentes ont montré que la spécificité des anticorps anti-histone PTM peut être très variable et que ces réactifs ne présentent pas Des propriétés avorables telles que la reconnaissance de l'epitope hors cible, une forte influence positive et négative par les PTM voisins et une difficulté à discriminer l'ordre de modification sur un résidu particulier ( par exemple , mono-, di- ou tri-méthyllysine) 12 , 13 , 14 , 15 , 16 , 17 , 18 . Par conséquent, un contrôle rigoureux de la qualité des réactifs d'anticorps spécifiques des PTM d'histones est nécessaire pour interpréter avec précision les données générées avec ces réactifs précieux.
La technologie Microarray permet l'interrogation simultanée de milliers d'interactions macromoléculaires dans un format à haut débit, reproductible et miniaturisé. Pour cette raison, une variété de plates-formes de microarray ont été créées pour analyser l'ADN protéique 19 ,"> 20, protéine-protéine 21 et interactions protéine-peptide 22. En effet, les microarrays de peptides histoniques sont apparus comme une plate-forme de découverte informative pour la recherche sur la biochimie de la chromatine, permettant le profilage à haut débit des écrivains, des effaces et des lecteurs d'histones PTM 15 , 23 , 24 , et aussi pour l'analyse de la spécificité des anticorps d'histone 17 , 25. Au-delà de leur application dans la recherche de la chromatine et de l'épigénétique, les tableaux de peptides histoniques ont une utilité potentielle comme test diagnostique / pronostique pour le lupus érythémateux systémique et d'autres maladies auto-immunes, Les autoanticorps de chromatine sont générés 26 , 27 .
Nous décrivons ici un pipeline intégré que nous avons développé pour concevoir, fabriquer et quitter.Des microarrays de peptides d'histone qui agissent pour générer des profils de spécificité pour les anticorps qui reconnaissent les histones et leurs PTM. Le pipeline est facilité par ArrayNinja, une application logicielle interactive ouverte, que nous avons développée récemment, qui intègre les étapes de conception et d'analyse des expériences microarray 28 . ArrayNinja fonctionne mieux dans Google Chrome. En bref, une imprimante à microarray de contact robotique est utilisée pour déposer une bibliothèque de peptides d'histones conjugués à la biotine à des positions définies sur des lames de microscope en verre revêtues de streptavidine. Les tableaux peuvent ensuite être utilisés dans un format de dosage compétitif et parallèle pour interroger les interactions anticorps-épitope ( Figure 1 ). La bibliothèque de peptides se compose de centaines de peptides synthétiques uniques hébergeant des PTM (acétylation de lysine, méthylation de lysine / arginine et phosphorylation de serine / thréonine) et en combinaisons pertinentes dérivées en grande partie de jeux de données protéiques. Méthodes de synthèse et de validation des peptides Sont détaillés ailleurs 23 . Les données générées à partir de nos efforts actuels de dépistage d'anticorps PTM d'histone utilisant cette plate-forme de réseau sont archivées sur une ressource Web publique, la base de données de spécificité d'anticorps Histone (www.histoneantibodies.com). Notamment, les microarrays de peptides d'histones fabriqués avec des variations de ce protocole ont également été largement utilisés pour caractériser l'activité des domaines de lecteur de PTM d'histone 8 , 29 , 30 , 31 , 32 , 33 , 34 , 35 , 36 , 37 et plus récemment à l'histone de profil Activités d'écrivain et de gomme à effet PTM 24 .
/files/ftp_upload/55912/55912fig1.jpg "/>
Figure 1: Traçage de la Procédure Stepwise pour le dépistage d'anticorps sur un microarray de peptide histone. Les peptides d'histone biotinylés abritant des modifications post-traductionnelles définies (cercles rouges et bleus) sont co-imprimés avec de la biotine-fluorescéine sur du verre revêtu de streptavidine. Les interactions positives sont visualisées sous forme de fluorescence rouge. Cliquez ici pour voir une version plus grande de ce chiffre.
Protocole
1. Installation et exécution de ArrayNinja
- Téléchargez et installez Oracle Virtual Box à partir de www.virtualbox.org.
- Téléchargez et décompressez la machine virtuelle ArrayNinja (VM) de http://research.vai.org/Tools/arrayninja.
- Ouvrez la boîte virtuelle et ajoutez la machine virtuelle ArrayNinja en cliquant sur 'Machine', 'Ajouter' et sélectionnez arrayninja.vbox dans le dossier où ArrayNinja VM a été enregistré.
- Commencez ArrayNinja en le sélectionnant dans la boîte virtuelle et en cliquant sur la flèche verte 'start'.
- La boîte virtuelle ouvrira une nouvelle fenêtre et affichera un message permettant d'accéder à la VM en naviguant sur le navigateur Web vers localhost: 2080.60; REMARQUE: une version conteneurisée de ArrayNinja est également disponible via hub.docker.com/r/bradley.dickson/arrayninja/
2. Conception du tableau de bord et de la mise en page de la plaque d'origine
- Dans le cadre «Planifier une mise en page de diapositive» sur l'interface ArrayNinja, cliquez sur le lien correspondant à l'imprimante à microarray utilisée.
REMARQUE: ArrayNinja a été programmé pour imiter le mouvement robotique de deux imprimantes microarray couramment utilisées (voir tableau1). La compatibilité avec les autres configurateurs peut être configurée sur demande. - Cliquez dans la boîte de dialogue "Charger la plaque", tapez "vide", puis cliquez sur "entrer". Voir la Figure 2 pour une capture d'écran du module de conception ArrayNinja.
- Réglez le diamètre du spot à 275 μm et l'espacement des taches à 375 μm. Réglez les réglages restants (blocs de plaques / rangées de plaques, rangées de plaques totales, répliques, fonctions dans y, Super Arrays, SuperA fudge, voir la figure 2 ) pour personnaliser la façon dont les fonctionnalités apparaîtront sur la diapositive en microarrains.
REMARQUE: Le diamètre du spot est déterminé par la taille de la broche de microarray. À mesure que ces paramètres sont ajustés, la diapositive animée sera mise à jour en temps réel. Utilisez ce dessin animé pour prévisualiser comment chaque paramètre modifie la disposition finale des diapositives. - Une fois la mise en page des fonctionnalités de la diapositive terminée, passez la souris sur chaque caractéristique unique et entrez l'identifiant de la fonction dans la boîte de dialogue contextuelle.
REMARQUE: les identificateurs de fonctions peuvent être composés de nombres, de lettres ou de combinaisons. Ceci n'est requis que pour les fonctionnalités uniques, et une boîte de dialogue n'apparaît pas lorsque les répliques sont sélectionnées. - Une fois que toutes les fonctionnalités uniques ont été attribuées à un identifiant, cliquez sur "remplir". Entrez un nom pour la disposition des diapositives et cliquez sur 'imprimer votre plaque' pour sauvegarder. Une nouvelle page s'ouvrira affichant le nombre de plaques de source de 384 puits nécessaires pour fabriquer la conception de la diapositive choisie et une table de cartographie de l'emplacement physique de chaque caractéristique à charger dans la (les) plaque (s) source (s).
REMARQUE: rappelez-vous ce nom, car il sera utilisé pour rappeler cette conception lors de l'analyse des données microarraintes (voir la section 6.2). En cliquant sur «imprimer votre assiette», vous enregistrez la mise en page dans ArrayNinja.

Figure 2: Module de conception ArrayNinja. Une capture d'écran Le module de conception ArrayNinja est représenté en pointillé. Le panneau de commande (en haut) affiche tous les paramètres qui peuvent être modifiés sur l'imprimante à microarray. À mesure que ces paramètres sont ajustés, l'image animée de la disposition des diapositives (en bas à gauche) est mise à jour en temps réel. Une fois la mise en page configurée, l'utilisateur peut se déplacer sur des points individuels pour entrer des identifiants de caractéristiques uniques. Les constructions ArrayNinja de cet utilisateur entrent une carte de la position de chaque caractéristique dans la (les) plaque (s) de source (en bas à droite) nécessaire pour fabriquer une disposition de diapositives de microarray spécifiée. Cliquez ici pour voir une version plus grande de ce chiffre.
3. Fabrication de microarrays
- Préparation de la plaque de base
- Utilisez la carte générée avec ArrayNinja dans la section 2.5 pour créer la (les) plaque (s) source (s) de 384 puits.
REMARQUE: Des descriptions détaillées des peptides interrogés sur cette plate-forme peuvent être trouvées ailleursS = "xref"> 17. - Déposer 1 - 2 μL de chaque caractéristique ( p . Ex. , Peptide d'histone biotinylé) dans le puits correct de la (les) plaque (s) de source de 384 puits.
NOTE: Les peptides d'histone biotinylés sont typiquement déposés à partir de solutions stock de 200 à 400 uM, ce qui équivaut à un excès molaire de 10 à 25 fois du peptide dans des sites de liaison de streptavidine dans un seul point de matrice. Ceci est calculé en utilisant l'équation: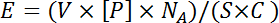
Où V est le volume délivré par une broche, [ P ] est la concentration de la fonction en cours d'impression, N A est le numéro d'Avogadro et S est la surface d'un point. C est la couverture de la glissière, exprimée en nombre de molécules de streptavidine par unité de surface multipliées par trois (le nombre moyen de sites de liaison de streptavidine disponibles). V et C sont obtenus par les fabricants respectifs. D'autres fonctionnalités peuventNécessitent des concentrations différentes en fonction de la taille de la biomolécule où l'encombrement peut être une préoccupation. Une gamme de concentrations d'impression pour chaque nouveau type de fonctionnalité devrait être testée de manière empirique pour déterminer la concentration d'impression optimale. - Diluer chaque caractéristique 10 fois avec 1x tampon d'impression complété par 1% d'albumine de sérum bovin (BSA). Faites tourner la (les) plaque (s) source (s) à 500 xg pendant 2 min à température ambiante.
REMARQUE: l'inclusion de la biotine marquée par fluorescéine (5 μg / μL) dans le tampon d'impression est recommandée en tant que contrôle de tache et comme aide visuelle pour faciliter l'alignement correct du tableau lors de l'analyse (voir la figure 4 ).
- Utilisez la carte générée avec ArrayNinja dans la section 2.5 pour créer la (les) plaque (s) source (s) de 384 puits.
- Protocole d'impression ( Figure 3A -B ).
- Préparez l'arrayer en vidant le récipient de collecte des déchets et en remplissant le récipient de solution de lavage et le récipient d'humidificateur avec de l'eau distillée stérile.
- Entrez les paramètres que vous avezPour concevoir la diapositive dans ArrayNinja (section 2 et Figure 2 ) dans le programme de contrôle de l'imprimante microarray.
- À l'aide du programme de contrôle de l'imprimante microarray, réglez la procédure de lavage pour un lavage de 1 s avec une immersion. Réglez les paramètres de post-lavage pour replonger les broches 5 fois après chaque lavage. Réglez l'humidité à 60%.
REMARQUE: la configuration de lavage optimale peut varier en fonction de l'imprimante à microarray utilisée. La configuration optimale de la pince post-lavage peut varier en fonction de l'imprimante à microarray utilisée. - Insérez des diapositives fonctionnalisées ( p . Ex. , Verre revêtu de streptavidine) dans les plateaux du substrat et placez tous les plateaux dans l'élévateur de la platine. Insérez la (s) plaque (s) de la source dans le (s) support (s) de la plaque et placez-les dans l'élévateur de la plaque d'origine.
- Cliquez sur 'imprimer'. Surveillez le processus d'impression pour quelques rondes de dépôt de fonctionnalités pour s'assurer que tous les réglages de lavage et de plongée sont corrects. Lorsque l'exécution de l'impression est terminée, supprimezLes plaques de substrat de l'arrayer.
REMARQUE: Lorsque les tirages importants interdisent l'achèvement des étapes de blocage dans un jour, les diapositives imprimées peuvent être incubées dans une chambre humidifiée à 4 ° C pendant la nuit. Incuber les diapositives à côté d'un petit bécher d'eau dans une boîte en carton scellée avec une pellicule en plastique. - Bloquez les diapositives avec un tampon de blocage pendant 30 minutes à température ambiante avec mélange.
- Laver les diapositives 2 x 10 min à température ambiante dans une solution salée tamponnée au phosphate (PBS), pH 7,6 avec mélange. Séchez les glissières en faisant tourner dans une centrifugeuse à microarray pendant 30 s à température ambiante.
REMARQUE: pour traiter un grand nombre de diapositives à la fois, une chambre de lavage à glissière à microscope à haut débit peut être utilisée, ce qui permet de laver 50 glissières en parallèle. - Pour les diapositives conçues pour être partagées avec de la cire, passez à la section 4.1. Pour tous les autres modèles, rangez les diapositives à 4 ° C protégés de la lumière et de l'humidité.
REMARQUE: peptides d'histone biotinylés imprimésSont stables pendant au moins 6 mois lorsqu'ils sont stockés de cette façon.
4. Partitionnement des diapositives en microarrains
- Stylo de cire hydrophobe ( figure 3C )
- Appliquer de la cire autour des zones qui contiennent des caractéristiques à l'aide d'un stylo à cire. Laisser la cire sécher à l'air pendant 5 minutes avant de passer à la section 5.
REMARQUE: après le blocage, les points de matrice peuvent être très difficiles à visualiser à l'œil. La conception de la diapositive de ArrayNinja peut être imprimée à l'échelle et utilisée comme guide pour l'application de la cire.
- Appliquer de la cire autour des zones qui contiennent des caractéristiques à l'aide d'un stylo à cire. Laisser la cire sécher à l'air pendant 5 minutes avant de passer à la section 5.
- Joint de silicone ( Figure 3D )
- Épluchez le film transparent à l'arrière du joint d'étanchéité et placez le côté adhésif vers le bas sur le toboggan à micro-arme.
- Tenez le joint en place pendant 5 s avant de procéder à l'article 5.
- Empreinte de cire ( figure 3E )
- Pour les diapositives conçues pour être divisées par de la cire, imprimez une diapositive de test sur un verre ordinaire à l'aide de 10% de BSA. Utilisez cette diapositive de test pour optimiser les guides d'imprimeur de cire ou les paramètres de réseau afin de s'assurer que toutes les fonctionnalités se situent dans les chambres à moule en cire.
- Faire chauffer l'imprimeuse à la cire microarray à 85 ° C jusqu'à ce que toute la cire soit complètement fondue, environ 30 min.
- Insérez la diapositive avec le côté imprimé vers le bas et enfoncez la glissière tout droit vers le bon guide sur l'imprimante à microarray. Tirez le levier pour mettre le moule en contact avec la surface de la glissière. Tenir pendant 2 s.
REMARQUE: Le temps de maintien peut être modifié pour obtenir une épaisseur optimale de la bordure de la cire. - Retirez rapidement la glissière et inspectez visuellement les bords de cire pour s'assurer que tous les puits sont inclus et que les bordures ne sont pas si épaisses qu'elles empiètent sur les taches. Rangez les diapositives à 4 ° C contre l'humidité et la lumière.
REMARQUE: le temps de maintien peut être augmenté ou diminué pour obtenir des bordures plus épaisses ou plus minces.

Figure 3: Fabrication de microarray. (A) Fabrication de microarray de peptides d'histone sur des lames de microscope recouvertes de streptavidine à l'aide d'une imprimante à microarray de contact. (B) Microarrays fabriqués avec 3 sous-réseaux d'une grille 48 x 48 de caractéristiques peptidiques. Séparation des sous-réseaux (C) 3 avec un stylo à cire hydrophobe, (D) 2 sous-arborescences avec un adhésif au silicium, et (E) 48 sous-arborescences avec empreinte de cire. Toutes les microarrays montrées sont fabriquées en utilisant des lames de microscope 25 x 75 mm. Cliquez ici pour voir une version plus grande de ce chiffre.
5. Hybridation d'un anticorps PTM Histone avec un micro-aigre peptidique
- Préparer un tampon d'hybridation (PBS, pH 7,6, 5% de BSA, 0,1% de Tween-20).
- Equilibrer la glissière dans le tampon d'hybridationEn utilisant un vaisseau d'hybridation. Recouvrez complètement la totalité de la glissière dans le tampon d'hybridation et incupez pendant 30 min à 4 ° C sur un agitateur orbitaire à faible vitesse.
- Préparez une solution contenant un anticorps PTM d'histone dilué dans un tampon d'hybridation.
REMARQUE: Dans les exemples de données, l'anticorps histone n ° 1 et l'anticorps n ° 2 ont été dilués 1: 1 000 dans un tampon d'hybridation. Une gamme de dilution similaire à celle utilisée pour immunoblot est recommandée comme point de départ. - Incuber le réseau avec la solution d'anticorps pendant 1 h à 4 ° C. Enlever la solution d'anticorps et laver le réseau 3 fois pendant 5 min à 4 ° C avec du PBS froid, pH 7,6.
- Préparez une dilution 1: 5,000 - 1: 10 000 d'anticorps secondaire conjugué à un colorant fluorescent dans un tampon d'hybridation.
- Incuber le réseau avec une solution d'anticorps secondaire pendant 30 minutes à 4 ° C protégé de la lumière. Retirez la solution d'anticorps secondaire et lavez le toboggan à micro-arme 3 fois pendant 5 min à 4 ° C avec du PBS, pH 7,6. TremperLe microarray dans un tube conique de 50 ml contenant 0,1 x PBS, pH 7,6 pour éliminer l'excès de sel à température ambiante. Séchez la glissière dans une centrifugeuse à micro-arme à température ambiante.
- Numérisez la diapositive avec un scanner à microarray à une résolution de 25 μm ou plus, en suivant le protocole de balayage recommandé par le fabricant du scanner à microarrains.
REMARQUE: si le traceur de biotine marqué par la fluorescéine est présent, numérisez à la fois le canal vert (ex: 488 nm, em: 509 nm) et le canal qui correspond à l'anticorps secondaire conjugué à un colorant fluorescent, typiquement rouge (ex: 635 nm, em : 677 nm). L'objectif de la numérisation est d'obtenir les fichiers .tif à une seule chaîne qui peuvent être fusionnés dans un seul fichier .png (comme décrit dans la section 6.1).
6. Analyse des données de microarray à l'aide de ArrayNinja
- Préparation d'une image de microarray fusionnée
REMARQUE: L'objectif de cette section est de créer un fichier image .png qui fusionne les deux fichiers .tif monocanal (obtenus dans la section 5). CeEst le seul format d'image compatible avec le module d'analyse ArrayNinja. Les instructions suivantes représentent un moyen possible d'obtenir un fichier image fusionné. Cependant, d'autres solutions sont disponibles ( par exemple , le freeware ImageJ).- À partir de la ligne de commande dans un terminal bash d'un ordinateur avec le Freeware ImageMagick installé, accédez au dossier contenant les fichiers .tif à une seule chaîne et copiez / collez les étapes suivantes, en appuyant sur 'entrer' entre chaque étape (6.1.4 - 6.1 .7).
REMARQUE: les noms de fichiers en majuscules ( p . Ex . , RED_CHANNEL) doivent être remplacés par le nom de fichier des images de diapositives à microarrains. - Si nécessaire, inversez d'abord les images en utilisant la commande 'convert INPUT.tif -negate OUTPUT.tif'. Ceci est nécessaire si le scanner enregistre les fichiers .tif avec le signal en blanc et l'arrière-plan en noir.
- Convert -depth 16 RED_CHANNEL.TIF -clone 0 -channel GB -évaluer l'ensemble 0 -delete 0 out.png 2> error.file.
- ConvErt - profondeur 16 CONTROL_CHANNEL.TIF -clone 0 -channel RB -évaluer l'ensemble 0 -delete 0 outa.png 2> error.file.
- Convert -depth 16 CONTROL_CHANNEL.TIF -clone 0 -channel RG -évaluer l'ensemble 0 -protéger 0 outB.png 2> error.file.
- Convert out.png outa.png outB.png -set zone de couleurs RGV -combine fusioned.png.
REMARQUE: un fichier nommé 'fusioned.png' sera enregistré dans le même dossier que les fichiers .tif originaux.
- À partir de la ligne de commande dans un terminal bash d'un ordinateur avec le Freeware ImageMagick installé, accédez au dossier contenant les fichiers .tif à une seule chaîne et copiez / collez les étapes suivantes, en appuyant sur 'entrer' entre chaque étape (6.1.4 - 6.1 .7).
- Quantifier les données en utilisant ArrayNinja
- Ouvrez ArrayNinja et cliquez sur le lien d'imprimante microarray approprié sous "Pour quantifier les images qui ont une plaque source connue". Tapez le nom de la conception de la diapositive enregistrée (étape 2.5) dans la boîte de dialogue "plaque de charge" et cliquez sur "entrer". Voir la Figure 4 pour une capture d'écran du module d'analyse ArrayNinja.
- Cliquez sur "Choisir un fichier" et accédez à et sélectionnez le fichier 'fusionné.png' créé à la section 6.1. L'imagerie fusionnéeE chargera aussi bien qu'une grille pour la disposition des diapositives.
- Utilisez les curseurs au bas du panneau de commande ArrayNinja pour régler le contraste, la luminosité et le point médian.
REMARQUE: ces ajustements sont uniquement à des fins de visualisation et n'ont aucun impact sur la quantification. - Sélectionnez "résolution" et entrez la valeur correspondant à la résolution de l'image scannée. Utilisez le "côté de la marge" et le "haut de la marge" pour déplacer la grille dans l'alignement avec les points du tableau. Ajustez au besoin pour que la grille soit alignée sur chaque point le plus possible.
- Cliquez sur 'Spot Seek' et attendez que le bouton revienne à la couleur grise originale.
REMARQUE: cela peut être répété plusieurs fois pour centrer les cercles de grille sur des points individuels. La fonction Seek relâche la grille vers chaque point pour affiner l'alignement. - Modifiez la valeur "Super Arrays" à "1" pour effectuer chaque panneau de sous-tableau individuellement (si vous le souhaitez). Si vous utilisez un 4Disposition de la diapositive d'impression de cire x 12 ( Figure 3E ), utilisez les contrôles "iP" "jPin" et "subA" pour désactiver les fonctionnalités pour analyser toute combinaison désirée de 4 x 12 puits. Par exemple, pour analyser les puits supérieur gauche et supérieur droit comme répliques les uns des autres, entrez "1 4" dans iPin, "1 1" dans jPin et "1 4" en sousA. Appuyez sur Entrée'.
- Passez la souris sur des points individuels pour voir l'identité de cette fonctionnalité.
- Cliquez sur 'Toggle zoom' pour examiner les fonctionnalités plus attentivement. Sélectionnez les points de référence en arrière-plan en appuyant sur la touche 'R' lorsque la souris est sur un point. Les points de référence seront mis en surbrillance en orange.
REMARQUE: en mode "basculer le zoom", une image agrandie de la fonction sur laquelle la souris est terminée s'affiche dans le coin supérieur droit. Une discussion détaillée des fonctionnalités supplémentaires de correction de fond dans ArrayNinja est discutée ailleurs 28 . - Déplacer les taches (avec une morphologie des taches très variée ou des débris qui affecteraient une quantification précise) en appuyant sur la touche 'A' tout en situant la souris sur les points affectés. Les taches inactivées deviennent de couleur blanche.
- Cliquez sur 'remplir' suivi de 'Soumettre' pour quantifier les points.
REMARQUE: Un nouvel onglet s'ouvre et affiche un graphique à barres de données normalisées à la moyenne spot la plus brillante. Un tableau ci-dessous le graphique à barres contient à la fois les valeurs de données normalisées et brutes. Ce tableau peut être copié dans une feuille de calcul pour l'archivage et l'analyse ultérieure.
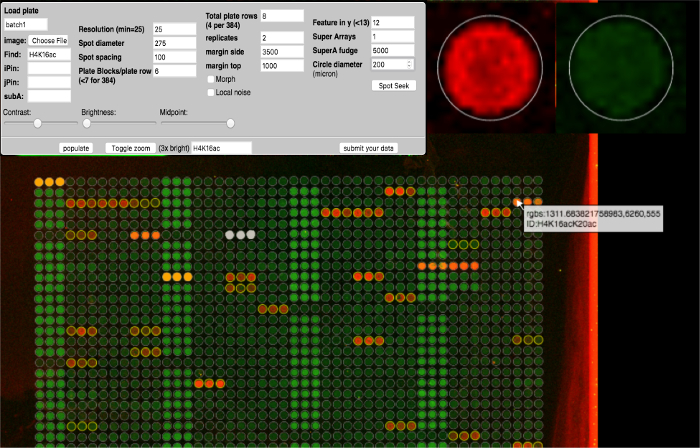
Figure 4: Module d'analyse ArrayNinja. Une capture d'écran du module d'analyse ArrayNinja est affichée. Le panneau de commande (en haut à gauche) affiche tous les paramètres qui peuvent être ajustés pour visualiser le tableau, trouver des points et aligner une grille sur leImage de tableau. Passer la souris sur une fonctionnalité affiche une vue agrandie (en haut à droite) et affiche une fenêtre contextuelle contenant les informations d'identification associées à cette fonction (en bas). Les points de référence sélectionnés pour la correction d'arrière-plan sont orange. Les caractéristiques à exclure de l'analyse en aval sont blanches. ArrayNinja contient une fonction de recherche basée sur le texte qui met en évidence les fonctionnalités correspondantes en jaune, comme indiqué dans l'exemple de H4K16. Cliquez ici pour voir une version plus grande de ce chiffre.
Résultats
Ce protocole a été utilisé pour concevoir et fabriquer une plate-forme de microarray peptidique pour l'analyse de la spécificité d'anticorps PTM des histones. Le tableau interroge une bibliothèque de plus de 300 caractéristiques peptidiques uniques (20 à 40 résidus de longueur) représentant une grande partie des combinaisons connues de PTM trouvés sur les protéines histone de coeur et variantes 38 . Ce pipeline a été un cheval de bataille pou...
Discussion
La fiabilité des anticorps dans les applications de recherche biomédicale est primordiale 46 , 47 . Ceci est particulièrement vrai dans la biochimie de la chromatine étant donné la position des anticorps comme outils clés pour la majorité des techniques développées pour caractériser l'abondance et la distribution des PTM des histones. Le protocole présenté ici détaille un pipeline optimisé pour la conception, la fabrication et l'utilisation...
Déclarations de divulgation
Les auteurs n'ont rien à dévoiler.
Remerciements
Ce travail a été soutenu en partie par l'Institut de recherche Van Andel et une subvention de recherche des Instituts nationaux de santé (CA181343) à SBR
matériels
| Name | Company | Catalog Number | Comments |
| Printing Buffer | ArrayIt | PPB | |
| BSA | Omnipure | 2390 | |
| Streptavidin-coated glass microscope slides | Greiner Bio-one | 439003-25 | |
| polypropylene 384 well plate | Greiner Bio-one | 784201 | |
| Biotin-fluorescein | Sigma | 53608 | |
| contact microarray printer | Aushon | 2470 | Aushon 2470 Microarray Printer |
| contact microarray printer | Gene Machines | OmniGrid 100 | OmniGrid Microarray Printer |
| PBS | Invitrogen | 14190 | |
| Blocking Buffer | ArrayIt | SBB | |
| Hydrophobic wax pen | Vector Labs | H-4000 | ImmEdge Hydrophobic Barrier PAP Pen |
| Silicon Gasket | Grace Bio-labs | 622511 | |
| Hybridization Vessel | Thermo Scientific | 267061 | or similar vessel |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21244 | Alexa Fluor 647 (anti-rabbit) |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21235 | Alexa Fluor 647 (anti-mouse) |
| Wax Imprinter | ArrayIt | MSI48 | |
| Tween-20 | Omnipure | 9490 | |
| Microarray Scanner | Innopsys | InnoScan 1100AL | or equivalent microarray scanner |
| EipTitan Histone Peptide Microarray | Epicypher | 112001 | |
| AbSurance Pro Histone Peptide Microarray | Millipore | 16668 | |
| MODified Histone Peptide Array | Active Motif | 13001 | |
| Histone Code Peptide Microarrays | JPT | His_MA_01 | |
| Wax | Royal Oak | GulfWax | for wax imprinter |
| Humidified Microarray Slide Hybridization Chamber | VWR | 97000-284 | |
| High throughput microscope slide washing chamber | ArrayIt | HTW | |
| Microscope slide centrifuge | VWR | 93000-204 | |
| Antibody 1 | Abcam | 8898 | |
| Antibody 2 | Millipore | 07-473 | |
| Biotinylated histone peptide | EpiCypher | 12-0001 | Example peptide. Similar peptides with various modifications are available from several commercial sources. |
| ImageMagick | https://www.imagemagick.org/script/index.php | ||
| ArrayNinja | https://rothbartlab.vai.org/tools/ |
Références
- van Steensel, B. Chromatin: constructing the big picture. EMBO J. 30 (10), 1885-1895 (2011).
- Kouzarides, T. Chromatin modifications and their function. Cell. 128 (4), 693-705 (2007).
- Rothbart, S. B., Strahl, B. D. Interpreting the language of histone and DNA modifications. Biochim Biophys Acta. 1839 (8), 627-643 (2014).
- Shogren-Knaak, M., Ishii, H., Sun, J. -. M., Pazin, M. J., Davie, J. R., Peterson, C. L. Histone H4-K16 acetylation controls chromatin structure and protein interactions. Science. 311 (5762), 844-847 (2006).
- Musselman, C. A., Lalonde, M. -. E., Côté, J., Kutateladze, T. G. Perceiving the epigenetic landscape through histone readers. Nat Struct Mol Biol. 19 (12), 1218-1227 (2012).
- Huang, H., Sabari, B. R., Garcia, B. A., Allis, C. D., Zhao, Y. SnapShot: Histone Modifications. Cell. 159 (2), 458 (2014).
- Strahl, B. D., Allis, C. D. The language of covalent histone modifications. Nature. 403 (6765), 41-45 (2000).
- Rothbart, S. B., Krajewski, K., et al. Association of UHRF1 with methylated H3K9 directs the maintenance of DNA methylation. Nat Struct Mol Biol. 19 (11), 1155-1160 (2012).
- Wang, Z., Zang, C., et al. Combinatorial patterns of histone acetylations and methylations in the human genome. Nat Genet. 40 (7), 897-903 (2008).
- Stunnenberg, H. G., Hirst, M. The International Human Epigenome Consortium: A Blueprint for Scientific Collaboration and Discovery. Cell. 167 (5), 1145-1149 (2016).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Egelhofer, T. A., Minoda, A., et al. An assessment of histone-modification antibody quality. Nat Struct Mol Biol. 18 (1), 91-93 (2011).
- Bock, I., Dhayalan, A., Kudithipudi, S., Brandt, O., Rathert, P., Jeltsch, A. Detailed specificity analysis of antibodies binding to modified histone tails with peptide arrays. Epigenetics. 6 (2), 256-263 (2011).
- Busby, M., Xue, C., et al. Systematic comparison of monoclonal versus polyclonal antibodies for mapping histone modifications by ChIP-seq. Epigenetics Chromatin. 9, 49 (2016).
- Fuchs, S. M., Krajewski, K., Baker, R. W., Miller, V. L., Strahl, B. D. Influence of combinatorial histone modifications on antibody and effector protein recognition. Curr Biol. 21 (1), 53-58 (2011).
- Kungulovski, G., Jeltsch, A. Quality of histone modification antibodies undermines chromatin biology research. F1000Research. 4, 1160 (2015).
- Rothbart, S. B., Dickson, B. M., et al. An Interactive Database for the Assessment of Histone Antibody Specificity. Mol Cell. 59 (3), 502-511 (2015).
- Rothbart, S. B., Lin, S., et al. Poly-acetylated chromatin signatures are preferred epitopes for site-specific histone H4 acetyl antibodies. Sci Rep. 2, 489 (2012).
- Berger, M. F., Bulyk, M. L. Universal protein-binding microarrays for the comprehensive characterization of the DNA-binding specificities of transcription factors. Nat Protoc. 4 (3), 393-411 (2009).
- Hu, S., Wan, J., et al. DNA methylation presents distinct binding sites for human transcription factors. eLife. 2, e00726 (2013).
- Moore, C. D., Ajala, O. Z., Zhu, H. Applications in high-content functional protein microarrays. Curr Opin Chem Biol. 30, 21-27 (2016).
- MacBeath, G., Schreiber, S. L. Printing proteins as microarrays for high-throughput function determination. Science. 289 (5485), 1760-1763 (2000).
- Rothbart, S. B., Krajewski, K., Strahl, B. D., Fuchs, S. M. Peptide microarrays to interrogate the "histone code" . Methods Enzymol. 512, 107-135 (2012).
- Cornett, E. M., Dickson, B. M., et al. Substrate Specificity Profiling of Histone-Modifying Enzymes by Peptide Microarray. Methods Enzymol. 574, 31-52 (2016).
- Nady, N., Min, J., Kareta, M. S., Chédin, F., Arrowsmith, C. H. A SPOT on the chromatin landscape? Histone peptide arrays as a tool for epigenetic research. Trends Biochem Sci. 33 (7), 305-313 (2008).
- Dieker, J., Berden, J. H., et al. Autoantibodies against Modified Histone Peptides in SLE Patients Are Associated with Disease Activity and Lupus Nephritis. PLoS ONE. 11 (10), (2016).
- Price, J. V., Tangsombatvisit, S., et al. "On silico" peptide microarrays for high-resolution mapping of antibody epitopes and diverse protein-protein interactions. Nat Med. 18 (9), 1434-1440 (2012).
- Dickson, B. M., Cornett, E. M., Ramjan, Z., Rothbart, S. B. ArrayNinja: An Open Source Platform for Unified Planning and Analysis of Microarray Experiments. Methods Enzymol. 574, 53-77 (2016).
- Gatchalian, J., Fütterer, A., et al. Dido3 PHD modulates cell differentiation and division. Cell Rep. 4 (1), 148-158 (2013).
- Cai, L., Rothbart, S. B., et al. An H3K36 methylation-engaging Tudor motif of polycomb-like proteins mediates PRC2 complex targeting. Mol Cell. 49 (3), 571-582 (2013).
- Rothbart, S. B., Dickson, B. M., et al. Multivalent histone engagement by the linked tandem Tudor and PHD domains of UHRF1 is required for the epigenetic inheritance of DNA methylation. Genes Dev. 27 (11), 1288-1298 (2013).
- Ali, M., Rincón-Arano, H., et al. Molecular basis for chromatin binding and regulation of MLL5. Proc Natl Acad Sci U S A. 110 (28), 11296-11301 (2013).
- Kinkelin, K., Wozniak, G. G., Rothbart, S. B., Lidschreiber, M., Strahl, B. D., Cramer, P. Structures of RNA polymerase II complexes with Bye1, a chromatin-binding PHF3/DIDO homologue. Proc Natl Acad Sci U S A. 110 (38), 15277-15282 (2013).
- Klein, B. J., Piao, L., et al. The histone-H3K4-specific demethylase KDM5B binds to its substrate and product through distinct PHD fingers. Cell Rep. 6 (2), 325-335 (2014).
- Kim, H. -. S., Mukhopadhyay, R., et al. Identification of a BET family bromodomain/casein kinase II/TAF-containing complex as a regulator of mitotic condensin function. Cell Rep. 6 (5), 892-905 (2014).
- Greer, E. L., Beese-Sims, S. E., et al. A histone methylation network regulates transgenerational epigenetic memory in C. elegans. Cell Rep. 7 (1), 113-126 (2014).
- Andrews, F. H., Tong, Q., et al. Multivalent Chromatin Engagement and Inter-domain Crosstalk Regulate MORC3 ATPase. Cell Rep. 16 (12), 3195-3207 (2016).
- Sidoli, S., Lin, S., Karch, K. R., Garcia, B. A. Bottom-Up and Middle-Down Proteomics Have Comparable Accuracies in Defining Histone Post-Translational Modification Relative Abundance and Stoichiometry. Anal Chem. 87 (6), 3129-3133 (2015).
- Tsukada, Y., Ishitani, T., Nakayama, K. I. KDM7 is a dual demethylase for histone H3 Lys 9 and Lys 27 and functions in brain development. Genes Dev. 24 (5), 432-437 (2010).
- Tachibana, M., Sugimoto, K., Fukushima, T., Shinkai, Y. Set domain-containing protein, G9a, is a novel lysine-preferring mammalian histone methyltransferase with hyperactivity and specific selectivity to lysines 9 and 27 of histone H3. J Biol Chem. 276 (27), 25309-25317 (2001).
- Wu, H., Chen, X., et al. Histone methyltransferase G9a contributes to H3K27 methylation in vivo. Cell Res. 21 (2), 365-367 (2011).
- Koch, C. M., Andrews, R. M., et al. The landscape of histone modifications across 1% of the human genome in five human cell lines. Genome Res. 17 (6), 691-707 (2007).
- Okitsu, C. Y., Hsieh, J. C. F., Hsieh, C. -. L. Transcriptional Activity Affects the H3K4me3 Level and Distribution in the Coding Region. Mol Cell Biol. 30 (12), 2933-2946 (2010).
- Zentner, G. E., Tesar, P. J., Scacheri, P. C. Epigenetic signatures distinguish multiple classes of enhancers with distinct cellular functions. Genome Res. 21 (8), 1273-1283 (2011).
- Garske, A. L., Oliver, S. S., et al. Combinatorial profiling of chromatin binding modules reveals multisite discrimination. Nat Chem Biol. 6 (4), 283-290 (2010).
- Baker, M. Reproducibility crisis: Blame it on the antibodies. Nature. 521 (7552), 274-276 (2015).
- Bradbury, A., Plückthun, A. Reproducibility: Standardize antibodies used in research. Nature. 518 (7537), 27-29 (2015).
- Nguyen, U. T. T., Bittova, L., et al. Accelerated chromatin biochemistry using DNA-barcoded nucleosome libraries. Nat Methods. 11 (8), 834-840 (2014).
- Frank, R. Spot-synthesis: an easy technique for the positionally addressable, parallel chemical synthesis on a membrane support. Tetrahedron. 48 (42), 9217-9232 (1992).
- Hilpert, K., Winkler, D. F. H., Hancock, R. E. W. Peptide arrays on cellulose support: SPOT synthesis, a time and cost efficient method for synthesis of large numbers of peptides in a parallel and addressable fashion. Nat Protoc. 2 (6), 1333-1349 (2007).
- Kudithipudi, S., Kusevic, D., Weirich, S., Jeltsch, A. Specificity analysis of protein lysine methyltransferases using SPOT peptide arrays. J Vis Exp. (93), e52203 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon