È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Analisi della specificità degli anticorpi istonici con microarray di peptide
In questo articolo
Riepilogo
Questo manoscritto descrive i metodi per applicare la tecnologia del microarray peptide alla profilazione di specificità degli anticorpi che riconoscono gli istoni e le loro modificazioni post-traslazionali.
Abstract
Le modificazioni post-traduzionali (PTMs) sulle proteine dell'istone sono ampiamente studiate per i loro ruoli nella regolazione della struttura cromatica e dell'espressione genica. La produzione di massa e la distribuzione di anticorpi specifici ai PTM istonici ha facilitato notevolmente la ricerca su questi segni. Poiché gli anticorpi PTM dell'istone sono reagenti chiave per molte applicazioni di biochimica cromatica, è necessaria un'analisi rigorosa della specificità degli anticorpi per un'accurata interpretazione dei dati e un progresso continuo nel campo. Questo protocollo descrive una pipeline integrata per la progettazione, la fabbricazione e l'uso di microarray peptidi per la profilazione della specificità degli anticorpi istoni. Gli aspetti di progettazione e analisi di questa procedura sono facilitati da ArrayNinja, un pacchetto software open source e interattivo che abbiamo recentemente sviluppato per semplificare la personalizzazione dei formati di stampa con microarray. Questa pipeline è stata utilizzata per la visualizzazione di un gran numero di anticorpi anti-PTM di istone commercialmente disponibili e ampiamente utilizzatiE i dati generati da questi esperimenti sono liberamente disponibili tramite un database di specificità anticorpo istante in linea e in espansione. Al di là degli istoni, la metodologia generale descritta qui può essere applicata ampiamente all'analisi degli anticorpi PTM-specifici.
Introduzione
Il DNA genomico è imballato elegantemente all'interno del nucleo delle cellule eucariotiche con proteine dell'istone per formare la cromatina. La subunità ripetizione della cromatina è il nucleosoma, che consiste di 147 coppie di basi di DNA avvolto attorno ad un nucleo octameric di proteine istoni H2A, - H2B, H3, H4 e 1. La cromatinica è ampiamente organizzata in euchromatin pienamente ricoperti e domini eterocromatina strettamente confezionati. Il grado di compattazione della cromatina regola la misura in cui i macchinari di proteine possono accedere al DNA sottostante per eseguire processi fondamentali del DNA come la replica, la trascrizione e la riparazione.
I regolatori chiave dell'accessibilità del genoma nel contesto della cromatina sono PTM sui nodi non strutturati e nuclei delle proteine istone 2 , 3 . Histon PTMs funziona direttamente influenzando la struttura della cromatina 4 e indirettamenteH l'assunzione di proteine di lettura e dei loro complessi macromolecolari associati con attività di ricostruzione cromatica, enzimatica e di ponteggi 5 . Studi sulla funzione istone PTM negli ultimi due decenni suggeriscono in maniera massiccia che questi segni svolgono ruoli chiave nel regolare il destino cellulare, lo sviluppo organico e l'iniziazione / progressione delle malattie. Alimentata dai progressi della tecnologia proteomica basata sulla spettrometria di massa, sono stati scoperti più di 20 PTM istonici unici su più di 80 residui istoniali distinti 6 . Notevolmente, queste modifiche spesso si verificano in combinazioni e coerenti con l'ipotesi del codice istone, numerosi studi suggeriscono che le proteine del lettore sono destinate a regioni discrete di cromatina attraverso il riconoscimento di combinazioni specifiche di istone PTMs 7 , 8 , 9 . Una sfida fondamentale in avanti sarà attribuire funzioni al grGrazie alla lista degli istoni PTMs e per determinare come combinazioni specifiche di PTM istoni orchestrino le funzioni dinamiche associate alla cromatina.
Gli anticorpi sono i reagenti lynchpin per la rilevazione di PTM istonico. In quanto tale, più di 1.000 anticorpi specifici PTM-istone sono stati commercialmente sviluppati per l'uso nella ricerca biochimica cromatina. Con il rapido sviluppo di high-throughput tecnologia di sequenziamento del DNA, questi reagenti sono stati ampiamente utilizzati dai singoli ricercatori e epigenomics grandi "calendario" iniziative (ad esempio, codificare e progetto), in ChIP-seq (cromatina immunoprecipitazione accoppiato con sequenziamento di prossima generazione ) Per generare mappe spaziali ad alta risoluzione della distribuzione istone PTM a livello genomico 10 , 11 . Tuttavia, recenti studi hanno dimostrato che la specificità degli anticorpi PTM dell'istone può essere altamente variabile e che questi reagenti presentano Proprietà avorabili come il riconoscimento epitopico fuori bersaglio, forte influenza positiva e negativa da parte dei PTM vicini e difficoltà a discriminare l'ordine di modifica su un determinato residuo ( ad esempio mono-, di- o tri-metililossina) 12 , 13 , 14 , 15 , 16 , 17 , 18 . Pertanto, è necessario un controllo rigoroso della qualità dei reagenti anticorpo specifico PTM-istone per interpretare con precisione i dati generati con questi reagenti preziosi.
La tecnologia Microarray consente di interrogare simultaneamente migliaia di interazioni macromolecolari in un formato ad alta produttività, riproducibile e miniaturizzato. Per questo motivo, sono state create diverse piattaforme di microarray per analizzare il DNA proteico 19 ,"> 20, proteine-proteine 21 e proteine-peptide interazioni 22. In effetti, i microarray di peptide dell'istone sono emerse come una piattaforma di scoperta informativa per la ricerca di biochimica cromatica, consentendo una profilazione ad alta velocità degli scrittori, cancellatori e lettori di istone PTMs 15 , 23 , 24 e anche per l'analisi della specificità degli anticorpi degli istoni 17 , 25. Oltre alla loro applicazione nella ricerca di cromatina e epigenetica, gli array di peptone istonico hanno potenziale utilità come test diagnostico / prognostico per il lupus eritematoso sistemico e altre malattie autoimmuni, Gli anticorpi cromatici vengono generati 26 , 27 .
Qui descriviamo una pipeline integrata che abbiamo sviluppato per la progettazione, la fabbricazione e la queMicroarrays di peptide istone per generare profili di specificità per gli anticorpi che riconoscono gli istoni e le loro PTM. Il gasdotto è facilitata da ArrayNinja, un open-source, un'applicazione software interattivo che abbiamo recentemente sviluppato, che integra le fasi di progettazione e di analisi di esperimenti di microarray 28. ArrayNinja funziona meglio in Google Chrome. In breve, viene utilizzata una stampante microarray a contatto robotizzato per depositare una biblioteca di peptidi istoni coniugati biotina in posizioni definite su vetrini a microscopio vetrato con streptavidina. Le matrici possono quindi essere utilizzate in un modo conciso e in parallelo di test per interrogare le interazioni con gli anticorpi-epitopi ( Figura 1 ). La biblioteca del peptide è costituita da centinaia di peptidi sintetici unici che ospitano PTM (acetilazione lisina, metilazione lisina / arginina e fosforilazione serina / treonina) da soli e in combinazioni pertinenti in gran parte derivate da set di dati proteomici. Metodi per la sintesi e la convalida dei peptidi Sono dettagliate altrove 23 . I dati generati dagli sforzi di screening degli anticorpi PTH istantanei in corso utilizzando questa piattaforma di array sono archiviati in una risorsa web pubblica, il database di specificità degli anticorpi Histone (www.histoneantibodies.com). In particolare, i microarray di peptide istone fabbricati con variazioni di questo protocollo sono stati ampiamente utilizzati per caratterizzare l'attività dei domini di lettore di istone PTM 8 , 29 , 30 , 31 , 32 , 33 , 34 , 35 , 36 , 37 e più recentemente all'istone del profilo PTM scrittore e attività gomma 24 .
/files/ftp_upload/55912/55912fig1.jpg "/>
Figura 1: Disposizione del fumetto della procedura stepwise per la screening degli anticorpi su un microarray di peptide dell'istone. I peptidi biotinilati di istone che ospitano modifiche post-traslazionali definite (cerchi rossi e blu) sono stampati con biotina-fluoresceina sul vetro rivestito con streptavidina. Le interazioni positive sono visualizzate come fluorescenza rossa. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
1. Installazione e esecuzione di ArrayNinja
- Scaricare e installare Oracle Virtual Box da www.virtualbox.org.
- Scaricare e decomprimere la macchina virtuale ArrayNinja (VM) da http://research.vai.org/Tools/arrayninja.
- Aprire Virtual Box e aggiungere l'ArrayNinja VM facendo clic su "Machine", "Aggiungi" e selezionando arrayninja.vbox dalla cartella in cui è stato salvato ArrayNinja VM.
- Avviare ArrayNinja selezionandolo all'interno di Virtual Box e facendo clic sulla freccia verde 'start'.
- Virtual Box apre una nuova finestra e visualizza un messaggio che consente di accedere alla VM navigando il browser web a localhost: 2080.60; NOTA: Una versione containerizzata di ArrayNinja è disponibile anche tramite hub.docker.com/r/bradley.dickson/arrayninja/
2. Progettazione del layout di scorrimento dell'albero e della piastrina di origine
- Sotto l'intestazione "Pianificare un layout di diapositive" nell'interfaccia ArrayNinja, fare clic sul collegamento corrispondente alla stampante microarray utilizzata.
NOTA: ArrayNinja è stato programmato per simulare il movimento robotico di due stampanti microarray comunemente usate (vedere tabella1). La compatibilità con gli altri array può essere configurata su richiesta. - Fai clic all'interno della finestra di dialogo 'Carica piastra', digita "vuoto" e fai clic su "entra". Vedere la Figura 2 per uno screenshot del modulo di progettazione ArrayNinja.
- Regolare il diametro del punto a 275 μm e la spaziatura a 375 μm. Regolare le impostazioni rimanenti (Plate Blocks / riga piastra, Righe totali, replica, caratteristiche in y, Super array, SuperA fudge, vedere la Figura 2 ) per personalizzare come le funzioni appaiono sulla diapositiva microarray.
NOTA: Il diametro del punto è determinato dalla dimensione del perno microarray. Poiché queste impostazioni sono regolate, la diapositiva dei cartoni animati verrà aggiornata in tempo reale. Utilizza questo cartone per visualizzare l'anteprima di come ogni impostazione sta modificando il layout di diapositive finale. - Dopo aver completato il layout delle funzioni sulla diapositiva, toccare sopra ogni funzione unica e immettere l'identificatore delle funzionalità nella finestra di dialogo a comparsa.
NOTA: Gli identificatori delle funzioni possono essere costituiti da numeri, lettere o combinazioni. Ciò è richiesto solo per funzioni uniche e una finestra di dialogo non verrà visualizzata quando vengono selezionati i replicati. - Dopo che tutte le funzionalità uniche sono state assegnate un identificatore, fai clic su "popolare". Inserisci un nome per il layout della diapositiva e fai clic su "stampa la tua piastra" per salvare. Viene aperta una nuova pagina che visualizza il numero di piastre di origine da 384 pozzetti necessarie per realizzare il disegno scelto e una tabella che traccia la posizione fisica di ciascuna funzionalità da caricare nella piastra di origine.
NOTA: Ricorda questo nome, in quanto verrà utilizzato per richiamare questo disegno quando si analizza i dati di microarray (vedere la sezione 6.2). Cliccando su "stampa la tua piastra" salva la disposizione all'interno di ArrayNinja.

Figura 2: Modulo di progettazione ArrayNinja. Un colpo di schermo del Il modulo di progettazione ArrayNinja è mostrato nella linea punteggiata. Il pannello di controllo (in alto) mostra tutti i parametri che possono essere modificati sulla stampante microarray. Quando questi parametri vengono regolati, l'immagine del fumetto del layout di diapositive (in basso a sinistra) viene aggiornata in tempo reale. Dopo aver impostato il layout, l'utente può sfiorare singoli punti per immettere identificatori di funzionalità univoche. ArrayNinja costruisce da questo utente una mappa della posizione di ciascuna funzione nella piastra (i) di origine (in basso a destra) necessaria per realizzare un layout di diapositiva specificato di microarray. Clicca qui per visualizzare una versione più grande di questa figura.
3. Microarrays Fabricating
- Preparazione della Source Plate
- Utilizzare la mappa generata con ArrayNinja nella sezione 2.5 per creare le piastre della sorgente a 384 pozzetti.
NOTA: Descrizioni dettagliate dei peptidi interrogati su questa piattaforma possono essere trovati altroveS = "xref"> 17. - Depurare 1 - 2 μL di ciascuna caratteristica ( ad esempio , peptone biotinilato di istone) nel pozzetto corretto delle piastre di fonte di 384 pozzetti.
NOTA: I peptidi biotinilati di istone sono tipicamente depositati da soluzioni di stock di 200-400 μM, che equivale a 10 a 25 volte l'eccesso molare di peptide ai siti di legame di streptavidina in un unico punto di matrice. Questo viene calcolato utilizzando l'equazione: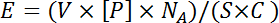
Dove V è il volume erogato da un perno, [ P ] è la concentrazione della caratteristica stampata, N A è il numero di Avogadro e S è la superficie di un punto. C è la copertura della diapositiva, espressa come numero di molecole di streptavidina per area unitaria moltiplicata per tre (il numero medio di siti di legame streptavidici disponibili). V e C sono ottenuti dai rispettivi produttori. Altre caratteristiche possonoRichiedono diverse concentrazioni a seconda della dimensione della biomolecola in cui l'affollamento può essere una preoccupazione. Una gamma di concentrazioni di stampa per ogni nuovo tipo di caratteristica deve essere testata empiricamente per determinare la concentrazione ottimale di stampa. - Diluire ogni caratteristica 10 volte con tampone di stampa 1x integrato con 1% di albumina bovina del siero (BSA). Spingere la targhetta (s) a 500 xg per 2 min a temperatura ambiente.
NOTA: L'inclusione di biotina (5 μg / μL) con etichettatura fluoresceina nel tampone di stampa è raccomandata come un controllo di macchia e come aiuto visivo per facilitare l'allineamento corretto dell'array durante l'analisi (vedere la figura 4 ).
- Utilizzare la mappa generata con ArrayNinja nella sezione 2.5 per creare le piastre della sorgente a 384 pozzetti.
- Protocollo di stampa ( Figura 3A - B ).
- Preparare l'allineamento svuotando il contenitore della raccolta e riempendo il contenitore della soluzione di lavaggio e il contenitore dell'umidificatore con acqua distillata sterile.
- Immettere i parametri uSed per progettare la diapositiva in ArrayNinja (sezione 2 e figura 2 ) nel programma di controllo della stampante microarray.
- Utilizzando il programma di controllo della stampante microarray, impostare la procedura di lavaggio per una lavata da 1 s con una sola immersione. Impostare le impostazioni di post-wash per rimettere nuovamente i pin 5 volte dopo ogni lavaggio. Impostare l'umidità al 60%.
NOTA: La configurazione ottimale del lavaggio può variare a seconda della stampante microarray utilizzata. La configurazione ottimale del dipinto a pin di lavaggio può variare a seconda della stampante microarray utilizzata. - Inserire le diapositive funzionalizzate ( ad es . Vetro rivestito con streptavidina) nelle piastre del substrato e posizionare tutte le piastre nell'elevatore a piastra. Inserire la piastra (s) nella piastra (i) e collocare nell'elevatore della piastra di sorgente.
- Fare clic su "stampa". Monitorare il processo di stampa per alcuni cicli di deposizione delle funzioni per assicurare che tutte le impostazioni di lavaggio e immer- sione siano corrette. Al termine della stampa, rimuoviLe piastre del substrato dall'allegato.
NOTA: quando le operazioni di stampa di grandi dimensioni vietano di completare i passaggi di blocco entro un giorno, le diapositive stampate possono essere incubate in una camera umidificata a 4 ° C durante la notte. Incubare le diapositive accanto ad un piccolo bicchiere d'acqua all'interno di una scatola di cartone sigillata con involucro in plastica. - Bloccare le diapositive con blocco di blocco per 30 minuti a temperatura ambiente con miscelazione.
- Lavare le diapositive 2 x 10 min a temperatura ambiente in soluzione salina fosfata (PBS), pH 7,6 con miscelazione. Asciugare le diapositive ruotando in una centrifuga a microarray per 30 s a temperatura ambiente.
NOTA: Per l'elaborazione di un gran numero di diapositive contemporaneamente, è possibile utilizzare una camera di lavaggio a scorrimento a microscopio ad alta capacità, consentendo la lavatura di 50 diapositive in parallelo. - Per le diapositive progettate per essere partizionate con cera, procedere alla sezione 4.1. Per tutti gli altri disegni, conservare le diapositive a 4 ° C protette dalla luce e dall'umidità.
NOTA: Peptidi stampati di istone biotinilatiSono stabili per almeno 6 mesi quando vengono memorizzati in questo modo.
4. Partizione delle diapositive Microarray
- Penna cera idrofoba ( figura 3C )
- Applicare la cera intorno alle aree che contengono caratteristiche utilizzando una penna a cera. Lasciare asciugare la cera per 5 minuti prima di procedere alla sezione 5.
NOTA: dopo il blocco, i punti array possono essere molto difficili da visualizzare per occhio. Il disegno a scorrimento da ArrayNinja può essere stampato in scala e utilizzato come guida per l'applicazione della cera.
- Applicare la cera intorno alle aree che contengono caratteristiche utilizzando una penna a cera. Lasciare asciugare la cera per 5 minuti prima di procedere alla sezione 5.
- Guarnizione in silicone ( Figura 3D )
- Sbucciate la pellicola trasparente sul retro della guarnizione di matrice e posizionate il lato adesivo sullo scivolo microarray.
- Tenere la guarnizione in posizione per 5 s prima di procedere alla sezione 5.
- Stampa di cera ( Figura 3E )
- Per le diapositive progettate per essere suddivise in cera, stampare una diapositiva di prova su vetro normale utilizzando il 10% BSA. Utilizzare questa diapositiva di prova per ottimizzare le guide dell'impronta della cera o le impostazioni dell'array per assicurare che tutte le funzioni saranno all'interno delle camere della muffa.
- Riscaldare l'impregnatrice a cera di microarray a 85 ° C finché tutta la cera non sia completamente fusa, circa 30 min.
- Inserire la diapositiva con il lato stampato rivolto verso il basso e spingere la diapositiva fino alla guida destra dell'etichettatrice microarray. Tirare la leva per portare lo stampo in contatto con la superficie dello scivolo. Tenere per 2 s.
NOTA: il tempo di attesa può essere modificato per ottenere lo spessore ottimale del bordo della cera. - Rimuovere rapidamente la diapositiva e ispezionare visivamente i bordi della cera per assicurare che tutti i pozzetti siano chiusi e che i bordi non siano così grossi che penetrano nei punti. Conservare le diapositive a 4 ° C protette da umidità e luce.
NOTA: il tempo di attesa può essere aumentato o diminuito per ottenere bordi più spessi o più sottili.

Figura 3: Fabbricazione microarray. (A) Fabbricazione di microarray di peptide dell'istone su vetrini con microscopio rivestito con streptavidina utilizzando una stampante a microarray di contatto. (B) Microarray fabbricate con 3 subarray di una griglia di 48 x 48 delle caratteristiche del peptide. Separazione di (C) 3 subarray con una penna di cera idrofobica, (D) 2 subarray con un adesivo in silicone e (E) 48 subarray con impronta di cera. Tutti i microarray mostrati sono fabbricati con diapositive a microscopio da 25 x 75 mm. Clicca qui per visualizzare una versione più grande di questa figura.
5. Ibridizzazione di un anticorpo di Histon PTM con un microarray di peptide
- Preparare il tampone di ibridazione (PBS, pH 7,6, 5% BSA, 0,1% Tween-20).
- Equilibrare la diapositiva nel buffer di ibridazioneUtilizzando un recipiente di ibridazione. Completamente coprire tutta la diapositiva nel tampone di ibridazione e incubare per 30 minuti a 4 ° C su un agitatore orbitale a bassa velocità.
- Preparare una soluzione contenente anticorpo PTM istone diluito in tampone di ibridazione.
NOTA: Nei dati di esempio, sia l'anticorpo # 1 che l'anticorpo # 2 di istone sono stati diluiti 1: 1.000 in tampone di ibridazione. Si consiglia come campo di partenza una gamma di diluizione simile a quella utilizzata per immunoblotting. - Incubare l'array con la soluzione anticorpale per 1 h a 4 ° C. Rimuovere la soluzione anticorpale e lavare l'array 3 volte per 5 min a 4 ° C con PBS freddo, pH 7,6.
- Preparare una diluizione 1: 5000 - 1: 10.000 di anticorpo secondario coniugato con fluorescenza colorante nel tampone di ibridazione.
- Incubare l'array con soluzione anticorpale secondaria per 30 min a 4 ° C protetta dalla luce. Rimuovere la soluzione anticorpale secondaria e lavare il vetrino microarray 3 volte per 5 min a 4 ° C con PBS, pH 7,6. TuffoIl microarray in un tubo conico da 50 mL contenente 0,1 x PBS, pH 7,6 per rimuovere il sale in eccesso a temperatura ambiente. Asciugare la diapositiva in una centrifuga a microarray a temperatura ambiente.
- Scansiona la diapositiva con uno scanner microarray a una risoluzione di 25 μm o superiore, seguendo il protocollo di scansione consigliato dal produttore dello scanner di microarray.
NOTA: se è presente un indicatore di biotina con fluoresceina, analizzare sia il canale verde (es: 488 nm, em: 509 nm) sia il canale corrispondente all'anticorpo secondario coniugato fluorescente colorato, tipicamente rosso (es: 635 nm, em 677 nm). L'obiettivo della scansione è quello di ottenere file .tif di singolo canale che possono essere fusi in un unico file png (come descritto nella sezione 6.1).
6. Analisi dei dati Microarray utilizzando ArrayNinja
- Preparazione di un'immagine unita di microarray
NOTA: L'obiettivo di questa sezione è quello di creare un file di immagine .png che unisce i due file .tif di singolo canale (ottenuto nella sezione 5). QuestoÈ l'unico formato di immagine compatibile con il modulo di analisi di ArrayNinja. Le seguenti istruzioni rappresentano un modo possibile per ottenere un file di immagine unito. Tuttavia, sono disponibili altre soluzioni ( ad esempio , l'ImageJ freeware).- Dalla riga di comando in un terminale di bash di un computer con il freeware ImageMagick installato, passare alla cartella che contiene i file .tif di singolo canale e copiare / incollare i passaggi seguenti, colpendo "entrare" tra ogni passaggio (6.1.4-6.1 0,7).
NOTA: I nomi dei file in lettere maiuscole ( ad esempio , RED_CHANNEL) dovrebbero essere sostituiti con il nome del file delle diapositive microarray. - Se necessario, invertire prima le immagini usando il comando 'convert INPUT.tif -negate OUTPUT.tif'. Ciò è necessario se lo scanner salva i file .tif con il segnale in bianco e nero in background.
- Convertire -depth 16 RED_CHANNEL.TIF -clone 0 -channel GB -valuta il set 0 -delete 0 out.png 2> error.file.
- convProfondità 16 CONTROL_CHANNEL.TIF -clone 0 -canale RB -valuta il set 0 -delete 0 outa.png 2> error.file.
- Convertire -depth 16 CONTROL_CHANNEL.TIF -clone 0 -canale RG -valuta il set 0 -delete 0 outB.png 2> error.file.
- Convertire fuori.png outa.png outB.png -set color space RGV -combine merged.png.
NOTA: Un file denominato "merged.png" verrà salvato nella stessa cartella dei file .tif originali.
- Dalla riga di comando in un terminale di bash di un computer con il freeware ImageMagick installato, passare alla cartella che contiene i file .tif di singolo canale e copiare / incollare i passaggi seguenti, colpendo "entrare" tra ogni passaggio (6.1.4-6.1 0,7).
- Quantificare i dati utilizzando ArrayNinja
- Apri ArrayNinja e fai clic sul collegamento appropriato della stampante microarray sotto "Per quantificare le immagini con una nota piastra di origine". Digitare il nome del disegno di diapositio salvato (punto 2.5) nella finestra di dialogo "piastra di carico" e fare clic su "entra". Vedere la Figura 4 per uno screenshot del modulo di analisi ArrayNinja.
- Fai clic su "Scegli il file" e vai a e seleziona il file 'merged.png' creato nella sezione 6.1. L'immaginario fusoE caricherà così come una griglia per il layout della diapositiva.
- Utilizzare i cursori nella parte inferiore del pannello di controllo ArrayNinja per regolare il contrasto, la luminosità e il punto medio.
NOTA: Queste regolazioni sono solo a scopo di visualizzazione e non hanno alcun impatto sulla quantificazione. - Selezionare "risoluzione" e inserire il valore corrispondente alla risoluzione dell'immagine acquisita. Utilizza il "margine" e "il margine superiore" per spostare la griglia in allineamento con i punti dell'array. Regolare come necessario per ottenere la griglia più strettamente allineata su ogni punto possibile.
- Fai clic su "Spot Seek" e attendi che il pulsante torni al colore grigio originale.
NOTA: questo può essere ripetuto più volte per centrare i cerchi di griglia su singoli punti. La funzione Seek rilassa la griglia verso ogni punto per ottimizzare l'allineamento. - Modificare il valore "Super Arrays" a "1" per elaborare singolarmente ciascun pannello di array secondario (se lo si desidera). Se si utilizza un 4X 12 layout di diapositiva a cera ( Figura 3E ), utilizzare i comandi "iPin" "jPin" e "subA" per disattivare le funzionalità per analizzare qualsiasi combinazione desiderata di 4 x 12 pozzetti. Ad esempio, per analizzare i pozzetti in alto a sinistra e in alto a destra come replicati l'uno all'altro, digitare "1 4" in iPin, "1 1" in jPin e "1 4" in subA. Premere Invio'.
- Spostare il mouse su singoli punti per visualizzare l'identità di quella funzionalità.
- Fai clic su "Toggle zoom" per esaminare le funzionalità più attentamente. Selezionare i punti di riferimento di sfondo premendo il tasto 'R' mentre il mouse è sopra un punto. I punti di riferimento verranno evidenziati in arancione.
NOTA: nella modalità "zoom ingrandita", nell'angolo superiore destro viene visualizzata un'immagine ingrandita della funzione che il mouse ha superato. Una discussione dettagliata sulle funzionalità aggiuntive di correzione dello sfondo in ArrayNinja è discussa altrove 28 . - Spostare i punti (con una morfologia spot o un detriti molto diversi che potrebbero influenzare la quantificazione accurata) premendo il tasto "A" mentre si aziona il mouse sui punti interessati. I punti inattivati diventeranno bianchi.
- Fai clic su "popolazione" seguito da "Invia" per quantificare i punti.
NOTA: una nuova scheda si apre e visualizza un grafico a barre di dati normalizzato alla media spot più luminosa. Una tabella sotto il grafico a barre contiene sia i valori di dati normalizzati che quelli raw. Questa tabella può essere copiata in un foglio di calcolo per archiviare e analizzare ulteriormente.
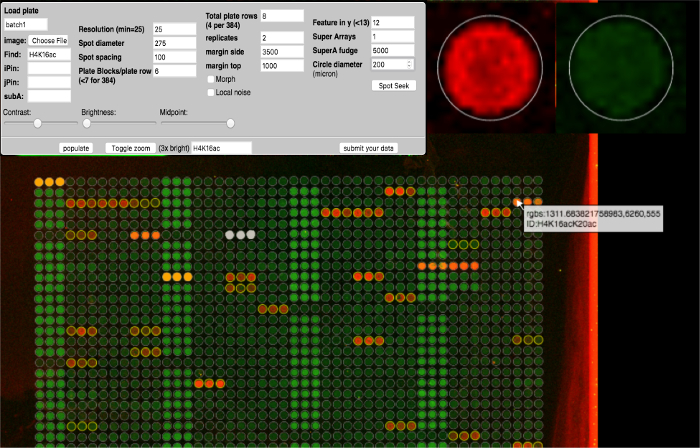
Figura 4: Modulo di analisi ArrayNinja. Viene visualizzato un colpo di schermo del modulo di analisi ArrayNinja. Il pannello di controllo (in alto a sinistra) mostra tutti i parametri che possono essere regolati per visualizzare l'array, trovare macchie e allineare una grigliaImmagine di matrice. Spostando il mouse su una funzione mostra una vista ingrandita (in alto a destra) e visualizza un popup che contiene le informazioni di identificazione associate a quella funzionalità (in basso). I punti di riferimento selezionati per la correzione dello sfondo sono arancione. Le caratteristiche da escludere dall'analisi a valle sono bianche. ArrayNinja contiene una funzionalità di ricerca basata sul testo che evidenzia le caratteristiche di corrispondenza in giallo, come mostrato nell'esempio di H4K16. Clicca qui per visualizzare una versione più grande di questa figura.
Risultati
Questo protocollo è stato utilizzato per progettare e realizzare una piattaforma microarray peptide per l'analisi della specificità degli anticorpi PTM. L'array richiede una libreria di più di 300 funzionalità peptide uniche (20-40 residui di lunghezza) che rappresentano molte delle combinazioni note di PTM trovate sulle proteine di istone del nucleo e delle varianti 38 . Questa pipeline è stata un cavallo di lavoro per lo screening di molti ant...
Discussione
L'affidabilità degli anticorpi nelle applicazioni di ricerca biomedica è fondamentale 46 , 47 . Ciò è particolarmente vero nella biochimica cromatica data la posizione degli anticorpi come strumenti chiave per la maggior parte delle tecniche sviluppate per caratterizzare l'abbondanza e la distribuzione di PTM istoniche. Il protocollo qui presentato illustra una pipeline ottimizzata per la progettazione, la fabbricazione e l'uso di microarray pept...
Divulgazioni
Gli autori non hanno niente da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto in parte da Van Andel Research Institute e da una borsa di studio degli Istituti Nazionali di Sanità (CA181343) a SBR
Materiali
| Name | Company | Catalog Number | Comments |
| Printing Buffer | ArrayIt | PPB | |
| BSA | Omnipure | 2390 | |
| Streptavidin-coated glass microscope slides | Greiner Bio-one | 439003-25 | |
| polypropylene 384 well plate | Greiner Bio-one | 784201 | |
| Biotin-fluorescein | Sigma | 53608 | |
| contact microarray printer | Aushon | 2470 | Aushon 2470 Microarray Printer |
| contact microarray printer | Gene Machines | OmniGrid 100 | OmniGrid Microarray Printer |
| PBS | Invitrogen | 14190 | |
| Blocking Buffer | ArrayIt | SBB | |
| Hydrophobic wax pen | Vector Labs | H-4000 | ImmEdge Hydrophobic Barrier PAP Pen |
| Silicon Gasket | Grace Bio-labs | 622511 | |
| Hybridization Vessel | Thermo Scientific | 267061 | or similar vessel |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21244 | Alexa Fluor 647 (anti-rabbit) |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21235 | Alexa Fluor 647 (anti-mouse) |
| Wax Imprinter | ArrayIt | MSI48 | |
| Tween-20 | Omnipure | 9490 | |
| Microarray Scanner | Innopsys | InnoScan 1100AL | or equivalent microarray scanner |
| EipTitan Histone Peptide Microarray | Epicypher | 112001 | |
| AbSurance Pro Histone Peptide Microarray | Millipore | 16668 | |
| MODified Histone Peptide Array | Active Motif | 13001 | |
| Histone Code Peptide Microarrays | JPT | His_MA_01 | |
| Wax | Royal Oak | GulfWax | for wax imprinter |
| Humidified Microarray Slide Hybridization Chamber | VWR | 97000-284 | |
| High throughput microscope slide washing chamber | ArrayIt | HTW | |
| Microscope slide centrifuge | VWR | 93000-204 | |
| Antibody 1 | Abcam | 8898 | |
| Antibody 2 | Millipore | 07-473 | |
| Biotinylated histone peptide | EpiCypher | 12-0001 | Example peptide. Similar peptides with various modifications are available from several commercial sources. |
| ImageMagick | https://www.imagemagick.org/script/index.php | ||
| ArrayNinja | https://rothbartlab.vai.org/tools/ |
Riferimenti
- van Steensel, B. Chromatin: constructing the big picture. EMBO J. 30 (10), 1885-1895 (2011).
- Kouzarides, T. Chromatin modifications and their function. Cell. 128 (4), 693-705 (2007).
- Rothbart, S. B., Strahl, B. D. Interpreting the language of histone and DNA modifications. Biochim Biophys Acta. 1839 (8), 627-643 (2014).
- Shogren-Knaak, M., Ishii, H., Sun, J. -. M., Pazin, M. J., Davie, J. R., Peterson, C. L. Histone H4-K16 acetylation controls chromatin structure and protein interactions. Science. 311 (5762), 844-847 (2006).
- Musselman, C. A., Lalonde, M. -. E., Côté, J., Kutateladze, T. G. Perceiving the epigenetic landscape through histone readers. Nat Struct Mol Biol. 19 (12), 1218-1227 (2012).
- Huang, H., Sabari, B. R., Garcia, B. A., Allis, C. D., Zhao, Y. SnapShot: Histone Modifications. Cell. 159 (2), 458 (2014).
- Strahl, B. D., Allis, C. D. The language of covalent histone modifications. Nature. 403 (6765), 41-45 (2000).
- Rothbart, S. B., Krajewski, K., et al. Association of UHRF1 with methylated H3K9 directs the maintenance of DNA methylation. Nat Struct Mol Biol. 19 (11), 1155-1160 (2012).
- Wang, Z., Zang, C., et al. Combinatorial patterns of histone acetylations and methylations in the human genome. Nat Genet. 40 (7), 897-903 (2008).
- Stunnenberg, H. G., Hirst, M. The International Human Epigenome Consortium: A Blueprint for Scientific Collaboration and Discovery. Cell. 167 (5), 1145-1149 (2016).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Egelhofer, T. A., Minoda, A., et al. An assessment of histone-modification antibody quality. Nat Struct Mol Biol. 18 (1), 91-93 (2011).
- Bock, I., Dhayalan, A., Kudithipudi, S., Brandt, O., Rathert, P., Jeltsch, A. Detailed specificity analysis of antibodies binding to modified histone tails with peptide arrays. Epigenetics. 6 (2), 256-263 (2011).
- Busby, M., Xue, C., et al. Systematic comparison of monoclonal versus polyclonal antibodies for mapping histone modifications by ChIP-seq. Epigenetics Chromatin. 9, 49 (2016).
- Fuchs, S. M., Krajewski, K., Baker, R. W., Miller, V. L., Strahl, B. D. Influence of combinatorial histone modifications on antibody and effector protein recognition. Curr Biol. 21 (1), 53-58 (2011).
- Kungulovski, G., Jeltsch, A. Quality of histone modification antibodies undermines chromatin biology research. F1000Research. 4, 1160 (2015).
- Rothbart, S. B., Dickson, B. M., et al. An Interactive Database for the Assessment of Histone Antibody Specificity. Mol Cell. 59 (3), 502-511 (2015).
- Rothbart, S. B., Lin, S., et al. Poly-acetylated chromatin signatures are preferred epitopes for site-specific histone H4 acetyl antibodies. Sci Rep. 2, 489 (2012).
- Berger, M. F., Bulyk, M. L. Universal protein-binding microarrays for the comprehensive characterization of the DNA-binding specificities of transcription factors. Nat Protoc. 4 (3), 393-411 (2009).
- Hu, S., Wan, J., et al. DNA methylation presents distinct binding sites for human transcription factors. eLife. 2, e00726 (2013).
- Moore, C. D., Ajala, O. Z., Zhu, H. Applications in high-content functional protein microarrays. Curr Opin Chem Biol. 30, 21-27 (2016).
- MacBeath, G., Schreiber, S. L. Printing proteins as microarrays for high-throughput function determination. Science. 289 (5485), 1760-1763 (2000).
- Rothbart, S. B., Krajewski, K., Strahl, B. D., Fuchs, S. M. Peptide microarrays to interrogate the "histone code" . Methods Enzymol. 512, 107-135 (2012).
- Cornett, E. M., Dickson, B. M., et al. Substrate Specificity Profiling of Histone-Modifying Enzymes by Peptide Microarray. Methods Enzymol. 574, 31-52 (2016).
- Nady, N., Min, J., Kareta, M. S., Chédin, F., Arrowsmith, C. H. A SPOT on the chromatin landscape? Histone peptide arrays as a tool for epigenetic research. Trends Biochem Sci. 33 (7), 305-313 (2008).
- Dieker, J., Berden, J. H., et al. Autoantibodies against Modified Histone Peptides in SLE Patients Are Associated with Disease Activity and Lupus Nephritis. PLoS ONE. 11 (10), (2016).
- Price, J. V., Tangsombatvisit, S., et al. "On silico" peptide microarrays for high-resolution mapping of antibody epitopes and diverse protein-protein interactions. Nat Med. 18 (9), 1434-1440 (2012).
- Dickson, B. M., Cornett, E. M., Ramjan, Z., Rothbart, S. B. ArrayNinja: An Open Source Platform for Unified Planning and Analysis of Microarray Experiments. Methods Enzymol. 574, 53-77 (2016).
- Gatchalian, J., Fütterer, A., et al. Dido3 PHD modulates cell differentiation and division. Cell Rep. 4 (1), 148-158 (2013).
- Cai, L., Rothbart, S. B., et al. An H3K36 methylation-engaging Tudor motif of polycomb-like proteins mediates PRC2 complex targeting. Mol Cell. 49 (3), 571-582 (2013).
- Rothbart, S. B., Dickson, B. M., et al. Multivalent histone engagement by the linked tandem Tudor and PHD domains of UHRF1 is required for the epigenetic inheritance of DNA methylation. Genes Dev. 27 (11), 1288-1298 (2013).
- Ali, M., Rincón-Arano, H., et al. Molecular basis for chromatin binding and regulation of MLL5. Proc Natl Acad Sci U S A. 110 (28), 11296-11301 (2013).
- Kinkelin, K., Wozniak, G. G., Rothbart, S. B., Lidschreiber, M., Strahl, B. D., Cramer, P. Structures of RNA polymerase II complexes with Bye1, a chromatin-binding PHF3/DIDO homologue. Proc Natl Acad Sci U S A. 110 (38), 15277-15282 (2013).
- Klein, B. J., Piao, L., et al. The histone-H3K4-specific demethylase KDM5B binds to its substrate and product through distinct PHD fingers. Cell Rep. 6 (2), 325-335 (2014).
- Kim, H. -. S., Mukhopadhyay, R., et al. Identification of a BET family bromodomain/casein kinase II/TAF-containing complex as a regulator of mitotic condensin function. Cell Rep. 6 (5), 892-905 (2014).
- Greer, E. L., Beese-Sims, S. E., et al. A histone methylation network regulates transgenerational epigenetic memory in C. elegans. Cell Rep. 7 (1), 113-126 (2014).
- Andrews, F. H., Tong, Q., et al. Multivalent Chromatin Engagement and Inter-domain Crosstalk Regulate MORC3 ATPase. Cell Rep. 16 (12), 3195-3207 (2016).
- Sidoli, S., Lin, S., Karch, K. R., Garcia, B. A. Bottom-Up and Middle-Down Proteomics Have Comparable Accuracies in Defining Histone Post-Translational Modification Relative Abundance and Stoichiometry. Anal Chem. 87 (6), 3129-3133 (2015).
- Tsukada, Y., Ishitani, T., Nakayama, K. I. KDM7 is a dual demethylase for histone H3 Lys 9 and Lys 27 and functions in brain development. Genes Dev. 24 (5), 432-437 (2010).
- Tachibana, M., Sugimoto, K., Fukushima, T., Shinkai, Y. Set domain-containing protein, G9a, is a novel lysine-preferring mammalian histone methyltransferase with hyperactivity and specific selectivity to lysines 9 and 27 of histone H3. J Biol Chem. 276 (27), 25309-25317 (2001).
- Wu, H., Chen, X., et al. Histone methyltransferase G9a contributes to H3K27 methylation in vivo. Cell Res. 21 (2), 365-367 (2011).
- Koch, C. M., Andrews, R. M., et al. The landscape of histone modifications across 1% of the human genome in five human cell lines. Genome Res. 17 (6), 691-707 (2007).
- Okitsu, C. Y., Hsieh, J. C. F., Hsieh, C. -. L. Transcriptional Activity Affects the H3K4me3 Level and Distribution in the Coding Region. Mol Cell Biol. 30 (12), 2933-2946 (2010).
- Zentner, G. E., Tesar, P. J., Scacheri, P. C. Epigenetic signatures distinguish multiple classes of enhancers with distinct cellular functions. Genome Res. 21 (8), 1273-1283 (2011).
- Garske, A. L., Oliver, S. S., et al. Combinatorial profiling of chromatin binding modules reveals multisite discrimination. Nat Chem Biol. 6 (4), 283-290 (2010).
- Baker, M. Reproducibility crisis: Blame it on the antibodies. Nature. 521 (7552), 274-276 (2015).
- Bradbury, A., Plückthun, A. Reproducibility: Standardize antibodies used in research. Nature. 518 (7537), 27-29 (2015).
- Nguyen, U. T. T., Bittova, L., et al. Accelerated chromatin biochemistry using DNA-barcoded nucleosome libraries. Nat Methods. 11 (8), 834-840 (2014).
- Frank, R. Spot-synthesis: an easy technique for the positionally addressable, parallel chemical synthesis on a membrane support. Tetrahedron. 48 (42), 9217-9232 (1992).
- Hilpert, K., Winkler, D. F. H., Hancock, R. E. W. Peptide arrays on cellulose support: SPOT synthesis, a time and cost efficient method for synthesis of large numbers of peptides in a parallel and addressable fashion. Nat Protoc. 2 (6), 1333-1349 (2007).
- Kudithipudi, S., Kusevic, D., Weirich, S., Jeltsch, A. Specificity analysis of protein lysine methyltransferases using SPOT peptide arrays. J Vis Exp. (93), e52203 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon