Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Analyse der Histon-Antikörper-Spezifität mit Peptid-Microarrays
In diesem Artikel
Zusammenfassung
Dieses Manuskript beschreibt Methoden zur Anwendung der Peptid-Microarray-Technologie auf die Spezifitätsprofilierung von Antikörpern, die Histone und ihre posttranslationalen Modifikationen erkennen.
Zusammenfassung
Posttranslationale Modifikationen (PTMs) auf Histonproteinen sind weitgehend untersucht für ihre Rollen bei der Regulierung der Chromatinstruktur und der Genexpression. Die Massenproduktion und -verteilung von Antikörpern, die spezifisch für Histon-PTMs sind, hat die Forschung auf diesen Markierungen stark erleichtert. Als Histon-PTM-Antikörper sind Schlüsselreagenzien für viele Chromatin-Biochemie-Anwendungen, eine rigorose Analyse der Antikörperspezifität ist notwendig für eine genaue Dateninterpretation und fortgesetzte Fortschritte auf dem Gebiet. Dieses Protokoll beschreibt eine integrierte Pipeline für die Konstruktion, Herstellung und Verwendung von Peptid-Mikroarrays zur Profilierung der Spezifität von Histon-Antikörpern. Die Design- und Analyseaspekte dieses Verfahrens werden durch ArrayNinja, ein Open-Source- und interaktives Softwarepaket, das wir vor kurzem entwickelt haben, um die Anpassung von Microarray-Druckformaten zu optimieren, erleichtert. Diese Pipeline wurde verwendet, um eine große Anzahl von kommerziell verfügbaren und weit verbreiteten Histon-PTM-Antibiotika zu screenenS und Daten aus diesen Experimenten sind frei verfügbar durch eine Online-und erweiterte Histone Antibody Specificity Database. Jenseits von Histonen kann die hier beschriebene allgemeine Methodik weitgehend auf die Analyse von PTM-spezifischen Antikörpern angewendet werden.
Einleitung
Genomische DNA ist elegant im eukaryotischen Zellkern mit Histonproteinen verpackt, um Chromatin zu bilden. Die sich wiederholende Untereinheit von Chromatin ist das Nukleosom, das aus 147 Basenpaaren von DNA besteht, die um einen oktameren Kern von Histonproteinen - H2A, H2B, H3 und H4 1 gewickelt sind. Chromatin ist weitgehend in lose gepackten Euchromatin und fest verpackten Heterochromatin-Domänen organisiert. Der Grad der Chromatin-Verdichtung regelt das Ausmaß, in dem Protein-Maschineries auf die zugrunde liegende DNA zugreifen können, um fundamentale DNA-templated Prozesse wie Replikation, Transkription und Reparatur durchzuführen.
Schlüsselregulatoren der Genomzugänglichkeit im Rahmen von Chromatin sind PTMs auf dem unstrukturierten Schwanz und Kerndomänen der Histonproteine 2 , 3 . Histone-PTMs funktionieren direkt durch die Beeinflussung der Struktur von Chromatin 4 und indirekt durchH die Rekrutierung von Leserproteinen und deren assoziierten makromolekularen Komplexen, die Chromatin-Remodeling-, Enzym- und Gerüstaktivitäten haben 5 . Studien der Histon-PTM-Funktion in den vergangenen zwei Jahrzehnten überwiegend vorschlagen, dass diese Markierungen spielen Schlüsselrollen bei der Regulierung der Zelle Schicksal, organismen Entwicklung und Krankheit Einleitung / Progression. Angetrieben durch Fortschritte in der Massenspektrometrie-basierten Proteomtechnologie wurden mehr als 20 einzigartige Histon-PTMs auf mehr als 80 verschiedenen Histonresten entdeckt 6 . Bemerkenswert ist, dass diese Modifikationen häufig in Kombinationen auftreten und im Einklang mit der "Histon-Code" -Hypothese sind, zeigen zahlreiche Studien, dass Leserproteine auf diskrete Bereiche des Chromatins durch Erkennung spezifischer Kombinationen von Histon-PTMs 7 , 8 , 9 gerichtet sind . Eine zentrale Herausforderung, die vorwärts geht, wird es sein, dem grEine Liste von Histon-PTMs und zu bestimmen, wie spezifische Kombinationen von Histon-PTMs die mit Chromatin verbundenen dynamischen Funktionen orchestrieren.
Antikörper sind die Lynchpin-Reagenzien für den Nachweis von Histon-PTMs. Als solche wurden mehr als 1.000 Histon-PTM-spezifische Antikörper kommerziell für den Einsatz in der Chromatin-Biochemie-Forschung entwickelt. Mit der rasanten Entwicklung der DNA-Sequenzierungstechnologie mit hohem Durchsatz werden diese Reagenzien ausgiebig von einzelnen Forschern und großflächigen epigenomischen "Roadmap" -Initiativen ( zB ENCODE und BLUEPRINT) in ChIP-seq (Chromatin-Immunpräzipitation gepaart mit Sequenzierung der nächsten Generation) eingesetzt ) Pipelines zur Erzeugung hochauflösender räumlicher Karten der Histon-PTM-Verteilung genomweit 10 , 11 . Allerdings haben jüngste Studien gezeigt, dass die Spezifität von Histon-PTM-Antikörpern sehr variabel sein kann und dass diese Reagenzien unf Austretende Eigenschaften wie z. B. Off-Target-Epitop-Erkennung, starker positiver und negativer Einfluss durch benachbarte PTMs und Schwierigkeiten, die Modifikationsreihenfolge an einem bestimmten Rest ( z. B. Mono-, Di- oder Tri-Methyllysin) 12 , 13 , 14 , 15 zu unterscheiden , 16 , 17 , 18 Daher ist eine rigorose Qualitätskontrolle von Histon-PTM-spezifischen Antikörperreagenzien notwendig, um die mit diesen wertvollen Reagenzien erzeugten Daten genau zu interpretieren.
Die Microarray-Technologie ermöglicht die gleichzeitige Abfrage von Tausenden von makromolekularen Wechselwirkungen in einem hochdurchsatz-, reproduzierbaren und miniaturisierten Format. Aus diesem Grund wurde eine Vielzahl von Microarray-Plattformen geschaffen, um die Protein-DNA 19 zu analysieren ,„> 20, Protein-Protein - 21 und Protein-Peptid - Wechselwirkungen 22. In der Tat, Histon - Peptidchips haben als informative Plattform zur Entdeckung für Chromatin - Biochemie Forschung entstanden, Hochdurchsatz - Profilierung der Autoren, Radiergummis ermöglicht, und die Leser von Histon PTM 15 , 23 , 24 und auch für die Analyse der Histon-Antikörper-Spezifität 17 , 25. Über ihre Anwendung in der Chromatin- und Epigenetikforschung hinaus haben Histon-Peptid-Arrays potentielle Nutzen als diagnostischer / prognostischer Test für systemischen Lupus erythematodes und andere Autoimmunkrankheiten, bei denen Anti- Chromatin-Autoantikörper werden erzeugt 26 , 27 .
Hier beschreiben wir eine integrierte Pipeline, die wir für die Konstruktion, Fertigung und Ausführung entwickelt habenRindende Histon-Peptid-Mikroarrays zur Erzeugung von Spezifitätsprofilen für Antikörper, die Histone und ihre PTMs erkennen. Die Pipeline wird von ArrayNinja erleichtert, ein Open-Source, interaktive Software - Anwendung , die wir vor kurzem entwickelt, die die Design- und Analysestufen Microarray - Experimente 28 integriert. ArrayNinja funktioniert am besten in Google Chrome. Kurz gesagt wird ein Roboter-Kontakt-Mikroarray-Drucker verwendet, um eine Bibliothek von Biotin-konjugierten Histon-Peptiden an definierten Positionen auf Streptavidin-beschichteten Glasmikroskop-Objektträgern abzuscheiden. Arrays können dann in einem kompetitiven und parallelen Assayformat verwendet werden, um Antikörper-Epitop-Wechselwirkungen abzufragen ( Abbildung 1 ). Die Peptidbibliothek besteht aus Hunderten von einzigartigen synthetischen Peptiden, die PTMs (Lysinacetylierung, Lysin / Arginin-Methylierung und Serin / Threonin-Phosphorylierung) allein und in relevanten Kombinationen, die weitgehend von Proteomik-Datensätzen abgeleitet sind, beherbergen. Methoden zur Peptidsynthese und Validierung Sind an anderer Stelle bekannt 23 . Daten, die aus unseren laufenden Histon-PTM-Antikörper-Screening-Bemühungen unter Verwendung dieser Array-Plattform generiert werden, werden auf einer öffentlichen Webressource, der Histone Antibody Specificity Database (www.histoneantibodies.com) archiviert. Bemerkenswerterweise wurden auch Histon-Peptid-Mikroarrays, die mit Variationen dieses Protokolls hergestellt wurden, ausgiebig verwendet, um die Aktivität der Histon-PTM-Leserdomänen 8 , 29 , 30 , 31 , 32 , 33 , 34 , 35 , 36 , 37 zu charakterisieren und in jüngerer Zeit Histon zu profilieren PTM-Schriftsteller und Radiergummi-Aktivitäten 24 .
/files/ftp_upload/55912/55912fig1.jpg "/>
Abbildung 1: Karikatur Darstellung der schrittweisen Vorgehensweise zur Antikörper-Screening auf einem Histon-Peptid-Mikroarray. Biotinylierte Histon-Peptide, die definierte posttranslationale Modifikationen (rote und blaue Kreise) beherbergen, werden mit Biotin-Fluorescein auf Streptavidin-beschichtetem Glas co-gedruckt. Positive Wechselwirkungen werden als rote Fluoreszenz visualisiert. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Protokoll
1. Installieren und Ausführen von ArrayNinja
- Downloaden und installieren Sie Oracle Virtual Box von www.virtualbox.org.
- Laden und entpacken Sie die ArrayNinja virtuelle Maschine (VM) von http://research.vai.org/Tools/arrayninja.
- Öffnen Sie die virtuelle Box und fügen Sie die ArrayNinja VM hinzu, indem Sie auf "Maschine", "Hinzufügen" klicken und arrayninja.vbox aus dem Ordner auswählen, in dem ArrayNinja VM gespeichert wurde.
- Starten Sie ArrayNinja, indem Sie es in der virtuellen Box auswählen und auf den grünen Pfeil "Start" klicken.
- Virtual Box öffnet ein neues Fenster und zeigt eine Meldung an, auf die die VM zugreifen kann, indem sie den Webbrowser zum localhost navigiert: 2080.60; HINWEIS: Eine Containerversion von ArrayNinja ist auch über hub.docker.com/r/bradley.dickson/arrayninja/ verfügbar.
2. Entwerfen des Array Slide und Source Plate Layouts
- Klicken Sie unter dem "Plan ein Folienlayout" auf der ArrayNinja-Oberfläche auf den Link, der dem verwendeten Microarray-Drucker entspricht.
HINWEIS: ArrayNinja wurde programmiert, um die Roboterbewegung von zwei häufig verwendeten Microarray-Druckern nachzuahmen (siehe Tabelle1). Die Kompatibilität mit den anderen Arradern kann auf Anfrage konfiguriert werden. - Klicken Sie in das Dialogfeld 'Leertafel', geben Sie "leer" ein und klicken Sie auf "Enter". Siehe Abbildung 2 für einen Screenshot des ArrayNinja Designmoduls.
- Den Fleckdurchmesser auf 275 μm einstellen und den Abstand auf 375 μm stellen. Passen Sie die verbleibenden Einstellungen an (Plattenblöcke / Plattenreihe, Gesamtplatte Zeilen, Replikate, Funktionen in y, Super Arrays, SuperA Fudge, siehe Abbildung 2 ), um anzupassen, wie die Features auf der Microarray Folie erscheinen.
HINWEIS: Der Spotdurchmesser wird durch die Größe des Microarray-Pins bestimmt. Da diese Einstellungen angepasst sind, wird die Karikatur-Folie in Echtzeit aktualisiert. Verwenden Sie diese Karikatur, um zu sehen, wie jede Einstellung das endgültige Folienlayout modifiziert. - Nachdem das Layout der Features auf der Folie abgeschlossen ist, Maus über jede einzelne Funktion und geben Sie die Feature-ID in das Pop-up-Dialogfeld.
HINWEIS: Funktionskennungen können aus Zahlen, Buchstaben oder Kombinationen bestehen. Dies ist nur für eindeutige Funktionen erforderlich, und ein Dialogfeld wird nicht angezeigt, wenn Replikate ausgewählt sind. - Nachdem alle eindeutigen Funktionen eine Kennung zugewiesen wurden, klicken Sie auf 'befüllen'. Geben Sie einen Namen für das Folienlayout ein und klicken Sie auf "Drucken Sie Ihren Teller", um zu speichern. Es wird eine neue Seite geöffnet, in der die Anzahl der 384-Well-Quellplatten angezeigt wird, die für die Herstellung des gewählten Diaplaydesigns erforderlich sind, und eine Tabelle, die die physikalische Position jedes Merkmals, das in die Quellplatte (n) geladen werden soll, abbildet.
HINWEIS: Denken Sie daran, diesen Namen zu verwenden, da er bei der Analyse von Microarray-Daten verwendet wird (siehe Abschnitt 6.2). Klicken Sie auf "print your plate" speichert das Layout in ArrayNinja.

Abbildung 2: ArrayNinja Design Modul. Ein Screenshot von der Das ArrayNinja Designmodul ist in der gepunkteten Linie dargestellt. Das Bedienfeld (oben) zeigt alle Parameter an, die auf dem Microarray-Drucker verändert werden können. Da diese Parameter eingestellt sind, aktualisiert sich das Karikaturbild des Folienlayouts (unten links) in Echtzeit. Nachdem das Layout eingestellt ist, kann der Benutzer die Maus über einzelne Punkte bewegen, um eindeutige Merkmalskennungen einzugeben. ArrayNinja konstruiert von diesem Benutzer eine Karte der Position jedes Merkmals in der Quellplatte (n) (unten rechts), die benötigt wird, um ein bestimmtes Mikroarray-Folienlayout herzustellen. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
3. Herstellung von Microarrays
- Vorbereiten der Quellplatte
- Verwenden Sie die mit ArrayNinja erstellte Karte in Abschnitt 2.5, um die 384-Well-Quellplatte (n) zu erstellen.
HINWEIS: Detaillierte Beschreibungen von Peptiden, die auf dieser Plattform abgefragt werden, können an anderer Stelle gefunden werdenS = "xref"> 17 - Ablagerung von 1 - 2 & mgr; l von jedem Merkmal ( z. B. biotinyliertes Histonpeptid) in die korrekte Vertiefung der 384-Well-Quellenplatte (n).
HINWEIS: Biotinylierte Histonpeptide werden typischerweise aus 200 - 400 μM Stammlösungen abgeschieden, was einem 10 bis 25fachen molaren Überschuss an Peptid zu Streptavidinbindungsstellen in einem einzigen Arraypunkt entspricht. Dies wird nach folgender Gleichung berechnet: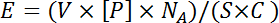
Wobei V das Volumen ist, das von einem Stift geliefert wird, [ P ] ist die Konzentration des zu druckenden Merkmals, N A ist die Nummer von Avogadro und S ist die Fläche eines Flecks. C ist die Abdeckung der Folie, ausgedrückt als Anzahl der Streptavidin-Moleküle pro Flächeneinheit multipliziert mit drei (die durchschnittliche Anzahl der verfügbaren Streptavidin-Bindungsstellen). V und C werden von den jeweiligen Herstellern erhalten. Andere Eigenschaften könnenErfordern unterschiedliche Konzentrationen in Abhängigkeit von der Größe des Biomoleküls, wo die Verdrängung ein Anliegen sein kann. Eine Reihe von Druckkonzentrationen für jede neue Art von Merkmal sollte empirisch getestet werden, um die optimale Druckkonzentration zu bestimmen. - Verdünnen Sie jedes Merkmal 10-fach mit 1x Druckpuffer, ergänzt mit 1% Rinderserumalbumin (BSA). Die Quellplatte (n) bei 500 xg für 2 min bei Raumtemperatur drehen.
HINWEIS: Die Einbeziehung von Fluorescein-markiertem Biotin (5 μg / μL) im Druckpuffer wird als Spotting-Kontrolle und als visuelle Hilfe empfohlen, um eine korrekte Array-Ausrichtung während der Analyse zu ermöglichen (siehe Abbildung 4 ).
- Verwenden Sie die mit ArrayNinja erstellte Karte in Abschnitt 2.5, um die 384-Well-Quellplatte (n) zu erstellen.
- Druckprotokoll ( Abbildung 3A - B ).
- Bereiten Sie den Arrayer vor, indem Sie den Müllsammelbehälter entleeren und den Waschlösungsbehälter und den Befeuchterbehälter mit sterilem destilliertem Wasser füllen.
- Geben Sie die Parameter u einUm die Folie in ArrayNinja (Abschnitt 2 und Abbildung 2 ) in das Mikroarray-Druckersteuerprogramm zu entwerfen.
- Verwenden Sie das Mikroarray-Druckersteuerprogramm, stellen Sie den Waschvorgang für eine 1-s-Waschung mit einem Eintauchen ein. Setzen Sie die Nachwäsche-Einstellungen, um die Stifte 5 mal nach jeder Wäsche wieder zu tauchen. Stellen Sie die Feuchtigkeit auf 60% ein.
HINWEIS: Die optimale Waschkonfiguration kann je nach verwendetem Microarray-Drucker variieren. Die optimale Nachwasch-Pin-Dip-Konfiguration kann je nach verwendetem Microarray-Drucker variieren. - Setzen Sie funktionalisierte Objektträger ( z. B. Streptavidin-beschichtetes Glas) in die Substratplatten ein und legen Sie alle Platten in den Plattenaufzug. Setzen Sie die Quellplatte (n) in den Plattenhalter (s) ein und legen Sie sie in den Quellplattenheber.
- Klicken Sie auf 'drucken'. Überwachen Sie den Druckvorgang für ein paar Runden von Feature Deposition, um sicherzustellen, dass alle Wasch- und Dip-Einstellungen korrekt sind. Wenn die Druckauflage abgeschlossen ist, entfernen SieDie Substratplatten aus dem Arrayer.
HINWEIS: Wenn große Druckaufträge die Beendigung der Sperrschritte innerhalb eines Tages beenden, können die bedruckten Folien in einer befeuchteten Kammer bei 4 ° C über Nacht inkubiert werden. Inkubieren Sie die Folien neben einem kleinen Becher Wasser in einem Karton mit Plastikfolie versiegelt. - Blockieren Sie die Objektträger mit Blockierpuffer für 30 min bei Raumtemperatur unter Mischen.
- Waschen der Objektträger 2 x 10 min bei Raumtemperatur in phosphatgepufferter Kochsalzlösung (PBS), pH 7,6 unter Mischen. Trocknen Sie die Objektträger durch Spinnen in einer Microarray-Objektträgerzentrifuge für 30 s bei Raumtemperatur.
HINWEIS: Für die Bearbeitung einer großen Anzahl von Folien auf einmal kann eine Hochdurchsatz-Mikroskop-Objektträgerwaschkammer verwendet werden, so dass 50 Folien parallel gewaschen werden können. - Für Objektträger, die mit Wachs partitioniert werden sollen, fahren Sie mit Abschnitt 4.1 fort. Für alle anderen Designs, lassen Sie Dias bei 4 ° C geschützt vor Licht und Feuchtigkeit.
HINWEIS: Gedruckte biotinylierte HistonpeptideSind mindestens 6 Monate stabil, wenn sie auf diese Weise gelagert werden.
4. Partitionieren von Microarray-Folien
- Hydrophobes Wachsstift ( Abbildung 3C )
- Wenden Sie Wachs um die Bereiche, die Merkmale mit einem Wachsstift enthalten. Lassen Sie das Wachs 5 Minuten lang an der Luft trocknen, bevor Sie mit Abschnitt 5 fortfahren.
HINWEIS: Nach dem Blockieren können die Array-Spots sehr schwer durch das Auge sichtbar werden. Die Dia-Design von ArrayNinja kann gedruckt und als Anleitung für die Anwendung von Wachs verwendet werden.
- Wenden Sie Wachs um die Bereiche, die Merkmale mit einem Wachsstift enthalten. Lassen Sie das Wachs 5 Minuten lang an der Luft trocknen, bevor Sie mit Abschnitt 5 fortfahren.
- Siliziumdichtung ( Abbildung 3D )
- Ziehen Sie den klaren Film von der Rückseite der Array-Dichtung und legen Sie die Klebstoffseite nach unten auf die Microarray-Folie.
- Halten Sie die Dichtung für 5 s vor dem Fortfahren auf Abschnitt 5.
- Wachs Impressum ( Abbildung 3E )
- Für Objektträger, die durch Wachs partitioniert werden sollen, drucken Sie einen Testschieber auf Normalglas mit 10% BSA. Verwenden Sie diese Testfolie, um die Wachs-Imprinter-Guides oder Array-Einstellungen zu optimieren, um sicherzustellen, dass alle Funktionen innerhalb der Wachsformkammern liegen.
- Den Mikroarray-Wachs-Imprinter auf 85 ° C erhitzen, bis das gesamte Wachs vollständig geschmolzen ist, ca. 30 min.
- Setzen Sie die Folie mit der bedruckten Seite nach unten und schieben Sie die Folie bis hin zur richtigen Führung auf dem Microarray-Imprinter. Ziehen Sie den Hebel hoch, um die Form mit der Oberfläche des Objektträgers in Berührung zu bringen. Halten Sie für 2 s.
HINWEIS: Die Haltezeit kann geändert werden, um eine optimale Wachsgrenzdicke zu erreichen. - Entfernen Sie schnell die Folie und visuell überprüfen Sie die Wachsgrenzen, um sicherzustellen, dass alle Brunnen eingeschlossen sind und dass die Ränder nicht so dick sind, dass sie auf den Flecken greifen. Schaufeln bei 4 ° C vor Feuchtigkeit und Licht schützen.
HINWEIS: Die Haltezeit kann erhöht oder verringert werden, um dickere oder dünnere Ränder zu erhalten.

Abbildung 3: Microarray-Fertigung. (A) Histon-Peptid-Mikroarray-Herstellung auf Streptavidin-beschichteten Mikroskop-Objektträgern unter Verwendung eines Kontakt-Mikroarray-Druckers. (B) Mikroarrays, die mit 3 Subarrays eines 48 x 48 Gitter von Peptidmerkmalen hergestellt wurden. Trennung von (C) 3 Subarrays mit einem hydrophoben Wachsstift, (D) 2 Subarrays mit Siliziumkleber und (E) 48 Subarrays mit Wachsabdruck. Alle dargestellten Mikroarrays werden mit 25 x 75 mm Objektträgern hergestellt. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
5. Hybridisierung eines Histon-PTM-Antikörpers mit einem Peptid-Microarray
- Hybridisierungspuffer (PBS, pH 7,6, 5% BSA, 0,1% Tween-20) vorbereiten.
- Äquilibrieren Sie die Folie in HybridisierungspufferMit einem Hybridisierungsgefäß. Die gesamte Folie im Hybridisierungspuffer vollständig abdecken und für 30 min bei 4 ° C auf einem Orbitalschüttler bei niedriger Geschwindigkeit inkubieren.
- Vorbereitung einer Lösung, die verdünnten Histon-PTM-Antikörper in Hybridisierungspuffer enthält.
HINWEIS: In den Beispieldaten wurden sowohl Histon-Antikörper # 1 als auch Antikörper # 2 1: 1000 in Hybridisierungspuffer verdünnt Als Ausgangspunkt empfiehlt sich ein Verdünnungsbereich ähnlich dem, der für das Immunoblotting verwendet wird. - Inkubieren Sie das Array mit der Antikörperlösung für 1 h bei 4 ° C. Entfernen Sie die Antikörperlösung und waschen Sie das Array 3 mal für 5 min bei 4 ° C mit kaltem PBS, pH 7,6.
- Vorbereitung einer 1: 5.000 - 1: 10.000 Verdünnung des fluoreszierenden Farbstoff-konjugierten sekundären Antikörpers in Hybridisierungspuffer.
- Inkubieren Sie das Array mit sekundärer Antikörperlösung für 30 min bei 4 ° C vor Licht geschützt. Entfernen Sie die sekundäre Antikörperlösung und waschen Sie die Mikroarray-Folie 3 mal für 5 min bei 4 ° C mit PBS, pH 7,6. TauchenDas Mikroarray in einem 50-ml-konischen Röhrchen mit 0,1x PBS, pH 7,6, um überschüssiges Salz bei Raumtemperatur zu entfernen. Trocknen Sie den Objektträger in einer Mikroarray-Gleitzentrifuge bei Raumtemperatur.
- Scannen Sie die Folie mit einem Microarray-Scanner mit einer Auflösung von 25 μm oder höher nach dem empfohlenen Scan-Protokoll des Microarray-Scanners.
HINWEIS: Wenn Fluorescein-markierter Biotin-Tracer vorhanden ist, scannen Sie sowohl den grünen Kanal (zB: 488 nm, em: 509 nm) als auch den Kanal, der dem fluoreszierenden Farbstoff-konjugierten sekundären Antikörper entspricht, typischerweise Rot (ex: 635 nm, em : 677 nm). Das Ziel des Scannens ist es, Einzelkanal-TIF-Dateien zu erhalten, die zu einer einzigen .png-Datei zusammengeführt werden können (wie in Abschnitt 6.1 beschrieben).
6. Analyse von Microarray-Daten mit ArrayNinja
- Vorbereiten eines zusammengesetzten Mikroarraybildes
HINWEIS: Das Ziel dieses Abschnitts ist es, eine .png-Bilddatei zu erstellen, die die beiden einkanaligen .tif-Dateien zusammenführt (erhalten in Abschnitt 5). DiesIst das einzige Bildformat, das mit dem ArrayNinja Analysemodul kompatibel ist. Die folgenden Anweisungen stellen eine Möglichkeit dar, eine zusammengeführte Bilddatei zu erhalten. Es stehen jedoch auch andere Lösungen zur Verfügung ( zB die Freeware ImageJ).- Von der Kommandozeile in einem bash-Terminal eines Computers mit der Freeware ImageMagick installiert, navigieren Sie zu dem Ordner, der die einzelnen Kanal-TIF-Dateien enthält, und kopieren / fügen Sie die folgenden Schritte ein, indem Sie zwischen jedem Schritt "eingegeben" (6.1.4 - 6.1) .7).
HINWEIS: Dateinamen in Großbuchstaben ( zB RED_CHANNEL) sollten durch den Dateinamen der Microarray-Folienbilder ersetzt werden. - Bei Bedarf zuerst die Bilder mit dem Befehl 'convert INPUT.tif -negate OUTPUT.tif' invertieren. Dies ist erforderlich, wenn der Scanner die .tif-Dateien mit dem Signal in Weiß und Hintergrund in Schwarz aufnimmt.
- Konvertieren -tiefe 16 RED_CHANNEL.TIF -clone 0-kanal GB -evaluate set 0 -delete 0 out.png 2> error.file.
- ConvErt - Tiefe 16 CONTROL_CHANNEL.TIF -clone 0 -Kanal RB -Evaluate set 0 -delete 0 outa.png 2> error.file.
- Konvertieren -tiefe 16 CONTROL_CHANNEL.TIF -clone 0-kanal RG -evaluate set 0 -delete 0 outB.png 2> error.file.
- Konvertieren out.png outa.png outB.png -set Farbraum RGV-combine merged.png.
HINWEIS: Eine Datei mit dem Namen 'merged.png' wird im selben Ordner wie die Original-TIF-Dateien gespeichert.
- Von der Kommandozeile in einem bash-Terminal eines Computers mit der Freeware ImageMagick installiert, navigieren Sie zu dem Ordner, der die einzelnen Kanal-TIF-Dateien enthält, und kopieren / fügen Sie die folgenden Schritte ein, indem Sie zwischen jedem Schritt "eingegeben" (6.1.4 - 6.1) .7).
- Quantifizierung von Daten mit ArrayNinja
- Öffnen Sie ArrayNinja und klicken Sie auf den entsprechenden Microarray-Drucker-Link unter "Um Bilder zu quantifizieren, die eine bekannte Quellplatte haben". Geben Sie den Namen des gespeicherten Dia-Designs (Schritt 2.5) im Dialogfeld "Lastplatte" ein und klicken Sie auf "Enter". Siehe Abbildung 4 für einen Screenshot des ArrayNinja-Analysemoduls.
- Klicken Sie auf "Datei auswählen" und navigieren Sie zu der in Abschnitt 6.1 erstellten Datei 'merged.png'. Das fusionierte sich vorE wird geladen sowie ein Raster für die Folie Layout.
- Verwenden Sie die Schieberegler am unteren Rand des ArrayNinja Bedienfeldes, um den Kontrast, die Helligkeit und den Mittelpunkt einzustellen.
HINWEIS: Diese Anpassungen dienen nur der Visualisierung und haben keinen Einfluss auf die Quantifizierung. - Wählen Sie "Auflösung" und geben Sie den Wert ein, der mit der Auflösung des gescannten Bildes übereinstimmt. Benutze die "Margin Side" und "Margin Top", um das Raster in die Ausrichtung mit den Spots auf dem Array zu verschieben. Passen Sie nach Bedarf an, um das Raster so genau wie möglich über jede Stelle zu bringen.
- Klicken Sie auf 'Spot Seek' und warten Sie, bis die Schaltfläche auf die ursprüngliche graue Farbe zurückkehrt.
HINWEIS: Dies kann mehrmals wiederholt werden, um die Gitterkreise auf einzelnen Stellen zu zentrieren. Die Seek-Funktion entspannt das Gitter auf jeden Punkt, um die Ausrichtung fein abzustimmen. - Ändern Sie den Wert "Super Arrays" auf "1", um jedes Sub-Array-Panel einzeln zu bearbeiten (falls gewünscht). Bei Verwendung eines 4X 12 Wachsabdruck-Folienlayout ( Abbildung 3E ), verwenden Sie die Bedienelemente "iPin" "jPin" und "subA", um die Funktionen auszuschalten, um eine beliebige Kombination von 4 x 12 Vertiefungen zu analysieren. Um zum Beispiel die obersten linken und oberen rechten Brunnen als Replikate voneinander zu analysieren, geben Sie "1 4" in iPin, "1 1" in jPin und "1 4" in subA ein. Drücken Sie Enter'.
- Bewege die Maus über einzelne Punkte, um die Identität dieser Funktion zu sehen.
- Klicken Sie auf "Toggle Zoom", um die Funktionen sorgfältiger zu überprüfen. Wählen Sie Hintergrundreferenzpunkte, indem Sie die Taste 'R' drücken, während sich die Maus über einem Punkt befindet. Referenzpunkte werden in orange hervorgehoben.
HINWEIS: Im "toggle zoom" -Modus wird in der oberen rechten Ecke ein vergrößertes Bild der Funktion angezeigt, die die Maus überschreitet. Eine detaillierte Diskussion über zusätzliche Hintergrundkorrekturmerkmale in ArrayNinja werden an anderer Stelle diskutiert 28 . - Umschalten von Flecken (mit vielfältiger Fleckmorphologie oder Trümmer, die eine genaue Quantifizierung beeinträchtigen), indem man die Taste "A" drückt, während man die Maus über die betroffenen Stellen schwebt. Inaktivierte Flecken werden weiß.
- Klicken Sie auf 'befüllen', gefolgt von 'Submit', um die Spots zu quantifizieren.
HINWEIS: Eine neue Registerkarte öffnet und zeigt ein Balkendiagramm von Daten an, die auf den hellsten Punktdurchschnitt normalisiert sind. Eine Tabelle unterhalb des Balkendiagramms enthält sowohl die normierten als auch die Rohdatenwerte. Diese Tabelle kann in eine Tabellenkalkulation für die Archivierung und weitere Analyse kopiert werden.
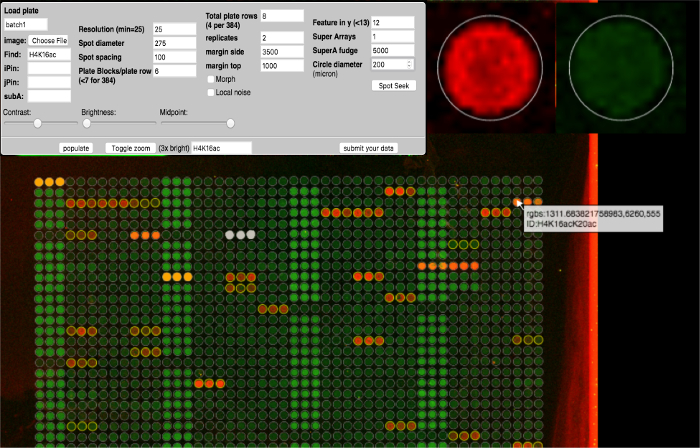
Abbildung 4: ArrayNinja Analysemodul. Ein Screenshot des ArrayNinja Analysemoduls wird gezeigt. Das Bedienfeld (oben links) zeigt alle Parameter an, die angepasst werden können, um das Array zu visualisieren, Spots zu finden und ein Raster über das Raster auszurichtenArray-Bild. Wenn Sie die Maus über eine Funktion schweben, erscheint eine vergrößerte Ansicht (oben rechts) und zeigt ein Popup an, das die mit dieser Funktion verknüpften Identifikationsinformationen enthält (unten). Referenzpunkte, die für die Hintergrundkorrektur ausgewählt wurden, sind orange. Merkmale, die von der nachgelagerten Analyse ausgeschlossen werden sollen, sind weiß. ArrayNinja enthält eine textbasierte Suchfunktion, die übereinstimmende Funktionen in gelb hervorhebt, wie im Beispiel für H4K16 gezeigt. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Ergebnisse
Dieses Protokoll wurde verwendet, um eine Peptid-Mikroarray-Plattform für die Analyse der Histon-PTM-Antikörperspezifität zu entwerfen und herzustellen. Das Array fragt eine Bibliothek von mehr als 300 einzigartigen Peptidmerkmalen (20 - 40 Reste in der Länge) ab, die viele der bekannten Kombinationen von PTMs darstellen, die auf Kern- und Variantenhistonproteinen gefunden wurden 38 . Diese Pipeline war ein Arbeitspferd für das Screening von vielen weit verbr...
Diskussion
Die Zuverlässigkeit der Antikörper in biomedizinischen Forschungsanwendungen liegt im Mittelpunkt 46 , 47 . Dies trifft besonders für die Chromatin-Biochemie zu, da die Position von Antikörpern als Schlüsselinstrumente für die Mehrheit der Techniken zur Charakterisierung der Häufigkeit und Verteilung von Histon-PTMs entwickelt wurde. Das hier vorgestellte Protokoll beschreibt eine optimierte Pipeline für die Konstruktion, Herstellung und Verwendung von P...
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Diese Arbeit wurde zum Teil vom Van Andel Research Institute und einem Forschungsstipendium der National Institutes of Health (CA181343) an SBR unterstützt
Materialien
| Name | Company | Catalog Number | Comments |
| Printing Buffer | ArrayIt | PPB | |
| BSA | Omnipure | 2390 | |
| Streptavidin-coated glass microscope slides | Greiner Bio-one | 439003-25 | |
| polypropylene 384 well plate | Greiner Bio-one | 784201 | |
| Biotin-fluorescein | Sigma | 53608 | |
| contact microarray printer | Aushon | 2470 | Aushon 2470 Microarray Printer |
| contact microarray printer | Gene Machines | OmniGrid 100 | OmniGrid Microarray Printer |
| PBS | Invitrogen | 14190 | |
| Blocking Buffer | ArrayIt | SBB | |
| Hydrophobic wax pen | Vector Labs | H-4000 | ImmEdge Hydrophobic Barrier PAP Pen |
| Silicon Gasket | Grace Bio-labs | 622511 | |
| Hybridization Vessel | Thermo Scientific | 267061 | or similar vessel |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21244 | Alexa Fluor 647 (anti-rabbit) |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21235 | Alexa Fluor 647 (anti-mouse) |
| Wax Imprinter | ArrayIt | MSI48 | |
| Tween-20 | Omnipure | 9490 | |
| Microarray Scanner | Innopsys | InnoScan 1100AL | or equivalent microarray scanner |
| EipTitan Histone Peptide Microarray | Epicypher | 112001 | |
| AbSurance Pro Histone Peptide Microarray | Millipore | 16668 | |
| MODified Histone Peptide Array | Active Motif | 13001 | |
| Histone Code Peptide Microarrays | JPT | His_MA_01 | |
| Wax | Royal Oak | GulfWax | for wax imprinter |
| Humidified Microarray Slide Hybridization Chamber | VWR | 97000-284 | |
| High throughput microscope slide washing chamber | ArrayIt | HTW | |
| Microscope slide centrifuge | VWR | 93000-204 | |
| Antibody 1 | Abcam | 8898 | |
| Antibody 2 | Millipore | 07-473 | |
| Biotinylated histone peptide | EpiCypher | 12-0001 | Example peptide. Similar peptides with various modifications are available from several commercial sources. |
| ImageMagick | https://www.imagemagick.org/script/index.php | ||
| ArrayNinja | https://rothbartlab.vai.org/tools/ |
Referenzen
- van Steensel, B. Chromatin: constructing the big picture. EMBO J. 30 (10), 1885-1895 (2011).
- Kouzarides, T. Chromatin modifications and their function. Cell. 128 (4), 693-705 (2007).
- Rothbart, S. B., Strahl, B. D. Interpreting the language of histone and DNA modifications. Biochim Biophys Acta. 1839 (8), 627-643 (2014).
- Shogren-Knaak, M., Ishii, H., Sun, J. -. M., Pazin, M. J., Davie, J. R., Peterson, C. L. Histone H4-K16 acetylation controls chromatin structure and protein interactions. Science. 311 (5762), 844-847 (2006).
- Musselman, C. A., Lalonde, M. -. E., Côté, J., Kutateladze, T. G. Perceiving the epigenetic landscape through histone readers. Nat Struct Mol Biol. 19 (12), 1218-1227 (2012).
- Huang, H., Sabari, B. R., Garcia, B. A., Allis, C. D., Zhao, Y. SnapShot: Histone Modifications. Cell. 159 (2), 458 (2014).
- Strahl, B. D., Allis, C. D. The language of covalent histone modifications. Nature. 403 (6765), 41-45 (2000).
- Rothbart, S. B., Krajewski, K., et al. Association of UHRF1 with methylated H3K9 directs the maintenance of DNA methylation. Nat Struct Mol Biol. 19 (11), 1155-1160 (2012).
- Wang, Z., Zang, C., et al. Combinatorial patterns of histone acetylations and methylations in the human genome. Nat Genet. 40 (7), 897-903 (2008).
- Stunnenberg, H. G., Hirst, M. The International Human Epigenome Consortium: A Blueprint for Scientific Collaboration and Discovery. Cell. 167 (5), 1145-1149 (2016).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Egelhofer, T. A., Minoda, A., et al. An assessment of histone-modification antibody quality. Nat Struct Mol Biol. 18 (1), 91-93 (2011).
- Bock, I., Dhayalan, A., Kudithipudi, S., Brandt, O., Rathert, P., Jeltsch, A. Detailed specificity analysis of antibodies binding to modified histone tails with peptide arrays. Epigenetics. 6 (2), 256-263 (2011).
- Busby, M., Xue, C., et al. Systematic comparison of monoclonal versus polyclonal antibodies for mapping histone modifications by ChIP-seq. Epigenetics Chromatin. 9, 49 (2016).
- Fuchs, S. M., Krajewski, K., Baker, R. W., Miller, V. L., Strahl, B. D. Influence of combinatorial histone modifications on antibody and effector protein recognition. Curr Biol. 21 (1), 53-58 (2011).
- Kungulovski, G., Jeltsch, A. Quality of histone modification antibodies undermines chromatin biology research. F1000Research. 4, 1160 (2015).
- Rothbart, S. B., Dickson, B. M., et al. An Interactive Database for the Assessment of Histone Antibody Specificity. Mol Cell. 59 (3), 502-511 (2015).
- Rothbart, S. B., Lin, S., et al. Poly-acetylated chromatin signatures are preferred epitopes for site-specific histone H4 acetyl antibodies. Sci Rep. 2, 489 (2012).
- Berger, M. F., Bulyk, M. L. Universal protein-binding microarrays for the comprehensive characterization of the DNA-binding specificities of transcription factors. Nat Protoc. 4 (3), 393-411 (2009).
- Hu, S., Wan, J., et al. DNA methylation presents distinct binding sites for human transcription factors. eLife. 2, e00726 (2013).
- Moore, C. D., Ajala, O. Z., Zhu, H. Applications in high-content functional protein microarrays. Curr Opin Chem Biol. 30, 21-27 (2016).
- MacBeath, G., Schreiber, S. L. Printing proteins as microarrays for high-throughput function determination. Science. 289 (5485), 1760-1763 (2000).
- Rothbart, S. B., Krajewski, K., Strahl, B. D., Fuchs, S. M. Peptide microarrays to interrogate the "histone code" . Methods Enzymol. 512, 107-135 (2012).
- Cornett, E. M., Dickson, B. M., et al. Substrate Specificity Profiling of Histone-Modifying Enzymes by Peptide Microarray. Methods Enzymol. 574, 31-52 (2016).
- Nady, N., Min, J., Kareta, M. S., Chédin, F., Arrowsmith, C. H. A SPOT on the chromatin landscape? Histone peptide arrays as a tool for epigenetic research. Trends Biochem Sci. 33 (7), 305-313 (2008).
- Dieker, J., Berden, J. H., et al. Autoantibodies against Modified Histone Peptides in SLE Patients Are Associated with Disease Activity and Lupus Nephritis. PLoS ONE. 11 (10), (2016).
- Price, J. V., Tangsombatvisit, S., et al. "On silico" peptide microarrays for high-resolution mapping of antibody epitopes and diverse protein-protein interactions. Nat Med. 18 (9), 1434-1440 (2012).
- Dickson, B. M., Cornett, E. M., Ramjan, Z., Rothbart, S. B. ArrayNinja: An Open Source Platform for Unified Planning and Analysis of Microarray Experiments. Methods Enzymol. 574, 53-77 (2016).
- Gatchalian, J., Fütterer, A., et al. Dido3 PHD modulates cell differentiation and division. Cell Rep. 4 (1), 148-158 (2013).
- Cai, L., Rothbart, S. B., et al. An H3K36 methylation-engaging Tudor motif of polycomb-like proteins mediates PRC2 complex targeting. Mol Cell. 49 (3), 571-582 (2013).
- Rothbart, S. B., Dickson, B. M., et al. Multivalent histone engagement by the linked tandem Tudor and PHD domains of UHRF1 is required for the epigenetic inheritance of DNA methylation. Genes Dev. 27 (11), 1288-1298 (2013).
- Ali, M., Rincón-Arano, H., et al. Molecular basis for chromatin binding and regulation of MLL5. Proc Natl Acad Sci U S A. 110 (28), 11296-11301 (2013).
- Kinkelin, K., Wozniak, G. G., Rothbart, S. B., Lidschreiber, M., Strahl, B. D., Cramer, P. Structures of RNA polymerase II complexes with Bye1, a chromatin-binding PHF3/DIDO homologue. Proc Natl Acad Sci U S A. 110 (38), 15277-15282 (2013).
- Klein, B. J., Piao, L., et al. The histone-H3K4-specific demethylase KDM5B binds to its substrate and product through distinct PHD fingers. Cell Rep. 6 (2), 325-335 (2014).
- Kim, H. -. S., Mukhopadhyay, R., et al. Identification of a BET family bromodomain/casein kinase II/TAF-containing complex as a regulator of mitotic condensin function. Cell Rep. 6 (5), 892-905 (2014).
- Greer, E. L., Beese-Sims, S. E., et al. A histone methylation network regulates transgenerational epigenetic memory in C. elegans. Cell Rep. 7 (1), 113-126 (2014).
- Andrews, F. H., Tong, Q., et al. Multivalent Chromatin Engagement and Inter-domain Crosstalk Regulate MORC3 ATPase. Cell Rep. 16 (12), 3195-3207 (2016).
- Sidoli, S., Lin, S., Karch, K. R., Garcia, B. A. Bottom-Up and Middle-Down Proteomics Have Comparable Accuracies in Defining Histone Post-Translational Modification Relative Abundance and Stoichiometry. Anal Chem. 87 (6), 3129-3133 (2015).
- Tsukada, Y., Ishitani, T., Nakayama, K. I. KDM7 is a dual demethylase for histone H3 Lys 9 and Lys 27 and functions in brain development. Genes Dev. 24 (5), 432-437 (2010).
- Tachibana, M., Sugimoto, K., Fukushima, T., Shinkai, Y. Set domain-containing protein, G9a, is a novel lysine-preferring mammalian histone methyltransferase with hyperactivity and specific selectivity to lysines 9 and 27 of histone H3. J Biol Chem. 276 (27), 25309-25317 (2001).
- Wu, H., Chen, X., et al. Histone methyltransferase G9a contributes to H3K27 methylation in vivo. Cell Res. 21 (2), 365-367 (2011).
- Koch, C. M., Andrews, R. M., et al. The landscape of histone modifications across 1% of the human genome in five human cell lines. Genome Res. 17 (6), 691-707 (2007).
- Okitsu, C. Y., Hsieh, J. C. F., Hsieh, C. -. L. Transcriptional Activity Affects the H3K4me3 Level and Distribution in the Coding Region. Mol Cell Biol. 30 (12), 2933-2946 (2010).
- Zentner, G. E., Tesar, P. J., Scacheri, P. C. Epigenetic signatures distinguish multiple classes of enhancers with distinct cellular functions. Genome Res. 21 (8), 1273-1283 (2011).
- Garske, A. L., Oliver, S. S., et al. Combinatorial profiling of chromatin binding modules reveals multisite discrimination. Nat Chem Biol. 6 (4), 283-290 (2010).
- Baker, M. Reproducibility crisis: Blame it on the antibodies. Nature. 521 (7552), 274-276 (2015).
- Bradbury, A., Plückthun, A. Reproducibility: Standardize antibodies used in research. Nature. 518 (7537), 27-29 (2015).
- Nguyen, U. T. T., Bittova, L., et al. Accelerated chromatin biochemistry using DNA-barcoded nucleosome libraries. Nat Methods. 11 (8), 834-840 (2014).
- Frank, R. Spot-synthesis: an easy technique for the positionally addressable, parallel chemical synthesis on a membrane support. Tetrahedron. 48 (42), 9217-9232 (1992).
- Hilpert, K., Winkler, D. F. H., Hancock, R. E. W. Peptide arrays on cellulose support: SPOT synthesis, a time and cost efficient method for synthesis of large numbers of peptides in a parallel and addressable fashion. Nat Protoc. 2 (6), 1333-1349 (2007).
- Kudithipudi, S., Kusevic, D., Weirich, S., Jeltsch, A. Specificity analysis of protein lysine methyltransferases using SPOT peptide arrays. J Vis Exp. (93), e52203 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten