Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Анализ специфичности гистонового антитела с пептидными микрочипами
В этой статье
Резюме
В этой рукописи описаны методы применения пептидной микрочиповой технологии для профилирования специфичности антител, которые распознают гистоны и их посттрансляционные модификации.
Аннотация
Пост-трансляционные модификации (ПТМ) на белках гистонов широко изучены для их роли в регулировании структуры хроматина и экспрессии генов. Массовое производство и распределение антител, специфичных к ПТМ гистонов, значительно облегчило исследование этих признаков. Поскольку гистоновые ПТМ-антитела являются ключевыми реагентами для многих применений биохимии хроматина, необходим тщательный анализ специфичности антител для точной интерпретации данных и дальнейшего прогресса в этой области. Этот протокол описывает интегрированный конвейер для проектирования, изготовления и использования пептидных микрочипов для профилирования специфичности гистоновых антител. Аспекты дизайна и анализа этой процедуры облегчаются с помощью ArrayNinja, открытого программного обеспечения и интерактивного программного пакета, который мы недавно разработали для упрощения настройки форматов печати микрочипов. Этот конвейер использовался для скрининга большого количества коммерчески доступного и широко используемого гистонового PTM-антителаS, а данные, полученные в результате этих экспериментов, свободно доступны через онлайн-базу данных Спецификации антител Histone Antibody. Помимо гистонов, общая методика, описанная здесь, может быть широко применена для анализа специфичных к ПТМ антител.
Введение
Геномная ДНК изящно упакована внутри ядра эукариотической клетки с белками гистонов с образованием хроматина. Повторяющейся субъединицей хроматина является нуклеосома, которая состоит из 147 пар оснований ДНК, обернутых вокруг октамерного ядра гистоновых белков - H2A, H2B, H3 и H4 1 . Хроматин широко организован в свободно упакованный эухроматин и плотно упакованные домены гетерохроматинов. Степень уплотнения хроматина регулирует степень, в которой белковые механизмы могут получить доступ к основной ДНК для осуществления фундаментальных процессов, связанных с ДНК, таких как репликация, транскрипция и восстановление.
Ключевыми регуляторами доступности генома в контексте хроматина являются ПТМ на неструктурированных хвостовых и основных доменах белков гистонов 2 , 3 . Гистоновые ПТМ функционируют напрямую, влияя на структуру хроматина 4 и косвенно проникаяH - набор белков-считывателей и связанных с ними макромолекулярных комплексов, которые имеют ремоделирование, ферментативную и лессификацию хроматина. 5 . Исследования функции гистонового ПТМ в течение последних двух десятилетий в подавляющем большинстве предполагают, что эти знаки играют ключевую роль в регулировании судьбы клеток, развития организма и инициирования / прогрессирования заболевания. Питаемые достижения в области масс - спектрометрия на основе протеомики технологии, более 20 уникальных гистоны PTMs на более чем 80 различных остатках гистонов были обнаружены 6. Примечательно, что эти модификации часто встречаются в комбинациях и согласуются с гипотезой «гистонового кода», многочисленные исследования показывают, что белки-считыватели нацелены на дискретные области хроматина посредством распознавания специфических комбинаций гистоновых ПТМ 7 , 8 , 9 . Ключевой задачей продвижения вперед будет назначение функций grКоторый содержит список гистоновых ПТМ и определяет, как конкретные комбинации гистоновых ПТМ организуют динамические функции, связанные с хроматином.
Антитела - это линчпиновые реагенты для обнаружения гистоновых ПТМ. Таким образом, более 1000 гистоновых ПТМ-специфических антител были коммерчески разработаны для использования в исследованиях биохимии хроматина. С быстрым развитием высокопроизводительной технологии секвенирования ДНК эти реагенты широко используются отдельными исследователями и крупномасштабными инициативами «дорожной карты» эпигеномики ( например , ENCODE и BLUEPRINT) в ChIP-seq (иммунопреципитация хроматина в сочетании с последовательностью следующего поколения ) Для создания пространственных карт с высоким разрешением распределения генома гистонов в геноме 10 , 11 . Однако недавние исследования показали, что специфичность антител к гистону ПТМ может быть очень переменной и что эти реагенты проявляют Приемлемые свойства, такие как распознавание нецелевого эпитопа, сильное положительное и отрицательное влияние со стороны соседних ПТМ и трудности, определяющие порядок модификации на определенном остатке ( например , моно-, ди- или триметиллизине) 12 , 13 , 14 , 15 , 16 , 17 , 18 . Поэтому для точной интерпретации данных, полученных с помощью этих ценных реагентов, необходим строгий контроль качества реагентов антител, специфичных к гистону ПТМ.
Технология Microarray позволяет одновременный опрос тысяч макромолекулярных взаимодействий в высокопроизводительном, воспроизводимом и миниатюрном формате. По этой причине были созданы различные платформы для микрочипов для анализа белковой ДНК 19 ,«> 20, белок-белок 21 и белок-пептидные взаимодействия 22. Действительно, гистоновые пептидные микрочипы появились как информационная платформа для исследования биохимических свойств хроматина, позволяющая высокопрофильное профилирование писателей, ластиков и считывателей гистоновых ПТМ 15 , 23 , 24 , а также для анализа специфичности антител к гистону 17 , 25. Вне их применения в исследованиях хроматина и эпигенетики гистоновые пептидные массивы имеют потенциальную полезность в качестве диагностического / прогностического теста для системной красной волчанки и других аутоиммунных заболеваний, Хроматиновые аутоантитела генерируются 26 , 27 .
Здесь мы описываем интегрированный конвейер, который мы разработали для проектирования, изготовления иДля получения микротипов гистоновых пептидов для получения профилей специфичности для антител, которые распознают гистоны и их ПТМ. Конвейер облегчается благодаря недавно разработанному ArrayNinja, интерактивному программному приложению с открытым исходным кодом, которое объединяет стадии проектирования и анализа экспериментов с микрочипами 28 . ArrayNinja работает лучше всего в Google Chrome. Вкратце, роботизированный контактный микрочиповый принтер используется для осаждения библиотеки биотин-конъюгированных гистоновых пептидов в определенных положениях на стеклянных микроскопах, покрытых стрептавидином. Затем массивы можно использовать в конкурентном и параллельном анализе для опроса антитело-эпитопных взаимодействий ( рис. 1 ). Библиотека пептидов состоит из сотен уникальных синтетических пептидов, содержащих PTM (ацетилирование лизина, метилирование лизина / аргинина и фосфорилирование серина / треонина) и в соответствующих комбинациях, в основном полученных из наборов данных протеомики. Методы синтеза и валидации пептидов Подробно описаны в других разделах 23 . Данные, полученные в ходе наших текущих скрининговых исследований скрининга антител к гистону, использующих эту платформу массивов, архивируются на общедоступном веб-ресурсе, базе данных специфичности антител Histone (www.histoneantibodies.com). Примечательно, что гистоновые пептидные микрочипы, изготовленные с вариациями этого протокола, также широко использовались для характеристики активности досок 8 , 29 , 30 , 31 , 32 , 33 , 34 , 35 , 36 , 37 гистоновых ПТМ-считывателей для профилирования гистона Запись PTT и ластик 24 .
/files/ftp_upload/55912/55912fig1.jpg "/>
Рисунок 1: Мультяшное изображение пошаговой процедуры скрининга антител на микрочипе гистонового пептида. Биотинилированные гистоновые пептиды, содержащие определенные посттрансляционные модификации (красные и синие круги), совместно печатаются с биотин-флуоресцеином на стекле, покрытом стрептавидином. Положительные взаимодействия визуализируются как красная флуоресценция. Нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
протокол
1. Установка и запуск ArrayNinja
- Загрузите и установите Oracle Virtual Box с сайта www.virtualbox.org.
- Загрузите и распакуйте виртуальную машину ArrayNinja (VM) с сайта http://research.vai.org/Tools/arrayninja.
- Откройте Virtual Box и добавьте виртуальную машину ArrayNinja, нажав «Машина», «Добавить» и выберите arrayninja.vbox из папки, в которой была сохранена копия ArrayNinja.
- Запустите ArrayNinja, выбрав его в Virtual Box и нажав зеленую стрелку «начать».
- Виртуальный ящик откроет новое окно и отобразит сообщение о доступе виртуальной машины, перейдя по веб-браузеру на localhost: 2080.60; ПРИМЕЧАНИЕ. Контейнерную версию ArrayNinja также можно получить через hub.docker.com/r/bradley.dickson/arrayninja/
2. Проектирование макета слайда массива и исходной пластины
- В разделе «Планирование макета слайдов» на интерфейсе ArrayNinja щелкните ссылку, соответствующую используемому принтеру микрочипов.
ПРИМЕЧАНИЕ. ArrayNinja был запрограммирован так, чтобы имитировать роботизированное перемещение двух обычно используемых микрочиповых принтеров (см. Таблицу1). Совместимость с другими серверами может быть настроена по запросу. - Нажмите внутри диалогового окна «Загрузить табличку», введите «пусто» и нажмите «войти». См. Рисунок 2 для скриншота проектного модуля ArrayNinja.
- Отрегулируйте диаметр пятна до 275 мкм и расстояние между точками до 375 мкм. Отрегулируйте остальные настройки (Plate Blocks / plate row, Total Plates Rows, replicates, features in y, Super Arrays, SuperA fudge, см. Рисунок 2 ), чтобы настроить, как будут отображаться функции на слайде микрочипа.
ПРИМЕЧАНИЕ. Диаметр пятна определяется размером штифта микрочипа. По мере того, как эти настройки будут скорректированы, слайд мультфильма будет обновляться в режиме реального времени. Используйте этот мультфильм, чтобы просмотреть, как каждый параметр изменяет окончательный макет слайдов. - После завершения компоновки функций на слайде наведите курсор на каждую уникальную функцию и введите идентификатор функции во всплывающем диалоговом окне.
ПРИМЕЧАНИЕ. Идентификаторы функций могут состоять из цифр, букв или комбинаций. Это требуется только для уникальных функций, и при выборе реплик диалоговое окно не появляется. - После того, как всем уникальным функциям был присвоен идентификатор, нажмите «заполнить». Введите имя макета слайда и нажмите «распечатать свою тарелку» для сохранения. Открывается новая страница, отображающая количество 384-луночных колонок источника, необходимых для изготовления выбранного дизайна слайда, и таблицу, отображающую физическое местоположение каждой функции, которая должна быть загружена в исходную пластину (-ы).
ПРИМЕЧАНИЕ. Помните это имя, так как оно будет использоваться для вызова этого проекта при анализе данных микрочипов (см. Раздел 6.2). Нажатие «распечатать вашу пластину» сохраняет макет внутри ArrayNinja.

Рисунок 2: Модуль проектирования ArrayNinja. Снимок экрана Конструктивный модуль ArrayNinja показан пунктирной линией. На панели управления (вверху) отображаются все параметры, которые могут быть изменены на микрочиповом принтере. По мере изменения этих параметров изображение мультфильма макета слайда (внизу слева) обновляется в реальном времени. После установки макета пользователь может навести курсор на отдельные точки, чтобы ввести уникальные идентификаторы функций. Конструкции ArrayNinja от этого пользователя вводят карту положения каждой функции в исходной пластине (внизу справа), необходимой для изготовления заданной макетной карты микрочипов. Нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
3. Изготовление микрочипов
- Подготовка исходной пластины
- Используйте карту, сгенерированную с помощью ArrayNinja в разделе 2.5, чтобы создать 384-луночный источник (ы).
ПРИМЕЧАНИЕ. Подробное описание пептидов, запрошенных на этой платформе, можно найти в другом местеS = "xref"> 17. - Депозит 1 - 2 мкл каждого признака ( например , биотинилированного гистонового пептида) в правильную лунку 384-луночного источника (-ов).
ПРИМЕЧАНИЕ. Биотинилированные гистоновые пептиды обычно осаждаются из исходных растворов 200-400 мкМ, что соответствует 10-25-кратным молярным избыткам пептида в сайты связывания стрептавидина в одном месте. Это вычисляется с использованием уравнения: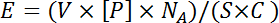
Где V - объем, подаваемый штифтом, [ P ] - концентрация печатаемого объекта, N A - число Авогадро, S - площадь поверхности пятна. C - покрытие слайда, выраженное как количество молекул стрептавидина на единицу площади, умноженное на три (среднее количество доступных мест связывания стрептавидина). V и C получены соответствующими изготовителями. Другие функции могутТребуют разных концентраций в зависимости от размера биомолекулы, где может возникнуть проблема с толпой. Для определения оптимальной концентрации печати необходимо эмпирически проверять диапазон концентраций печати для каждого типа нового типа. - Разбавьте каждую функцию в 10 раз с помощью 1х печатного буфера, дополненного 1% бычьим сывороточным альбумином (БСА). Вращайте исходную пластину (ы) при 500 мкг в течение 2 мин при комнатной температуре.
ПРИМЕЧАНИЕ. Включение меченого флуоресцеином биотина (5 мкг / мкл) в буфер печати рекомендуется в качестве контроля пятна и в качестве визуального средства для обеспечения правильного выравнивания массива во время анализа (см. Рисунок 4 ).
- Используйте карту, сгенерированную с помощью ArrayNinja в разделе 2.5, чтобы создать 384-луночный источник (ы).
- Протокол печати ( рисунок 3A- B ).
- Подготовьте устройство, опустошив контейнер для сбора отходов и заполнив контейнер для промывочного раствора и контейнер для увлажнителя стерильной дистиллированной водой.
- Введите параметры uSed для проектирования слайда в ArrayNinja (раздел 2 и рисунок 2 ) в программу управления принтером микрочипов.
- Используя программу управления принтером микрочипов, установите процедуру стирки на 1-секундную промывку одним погружением. Установите параметры после стирки, чтобы повторно опустить штифты 5 раз после каждой стирки. Установите влажность до 60%.
ПРИМЕЧАНИЕ. Оптимальная конфигурация мойки может варьироваться в зависимости от используемого принтера микрочипов. Оптимальная конфигурация погружного штифта после мойки может меняться в зависимости от используемого принтера микрочипов. - Вставьте функционализированные слайды ( например , стекло, покрытое стрептавидином) в плиты подложки, и поместите все плиты в лифт лифта. Вставьте исходную пластину (-ы) в держатель (-ы) пластины и положите в лифте-листы источника.
- Нажмите «Печать». Следите за процессом печати в течение нескольких раундов отложения свойств, чтобы убедиться, что все настройки мойки и погружения верны. Когда печать завершена, удалитеПодложка из плиты.
ПРИМЕЧАНИЕ. Когда большие тиражи запрещают завершение шагов блокировки в течение одного дня, печатные слайды можно инкубировать в увлажненной камере при температуре 4 ° C в течение ночи. Инкубируйте слайды рядом с небольшим стаканом воды внутри картонной коробки, запечатанной пластиковой пленкой. - Блокируйте слайды с блокирующим буфером в течение 30 мин при комнатной температуре с перемешиванием.
- Промойте слайды 2 × 10 мин при комнатной температуре в забуференном фосфатом физиологическом растворе (PBS), pH 7,6 с перемешиванием. Высушите слайды путем вращения в центрифуге с микрочипом в течение 30 с при комнатной температуре.
ПРИМЕЧАНИЕ. Для обработки большого количества слайдов одновременно можно использовать камеру для мойки с высокой пропускной способностью микроскопа, позволяющую параллельно промывать 50 слайдов. - Для слайдов, предназначенных для разделения на воск, перейдите к разделу 4.1. Для всех других конструкций храните слайды при температуре 4 ° C, защищенные от света и влаги.
ПРИМЕЧАНИЕ. Печатные биотинилированные гистоновые пептидыСтабильны в течение не менее 6 месяцев при хранении таким образом.
4. Разделение слайдов Microarray
- Гидрофобная восковая ручка ( рисунок 3C )
- Нанесите воск вокруг областей, содержащих функции, используя восковую ручку. Дайте воску высохнуть на воздухе в течение 5 минут, прежде чем переходить к разделу 5.
ПРИМЕЧАНИЕ. После блокировки пятна массива могут быть очень трудно визуализироваться на глаз. Конструкция слайда от ArrayNinja может быть напечатана в масштабе и использована в качестве руководства для применения воска.
- Нанесите воск вокруг областей, содержащих функции, используя восковую ручку. Дайте воску высохнуть на воздухе в течение 5 минут, прежде чем переходить к разделу 5.
- Силиконовая прокладка ( рисунок 3D )
- Очистите прозрачную пленку с задней части прокладки массива и положите клейкую сторону вниз на микрочиповую ползун.
- Удерживайте прокладку на месте в течение 5 с перед переходом к разделу 5.
- Отпечаток воска ( рисунок 3Е )
- Для слайдов, предназначенных для разделения воском, напечатайте тестовый слайд на обычном стекле с использованием 10% BSA, Используйте этот тестовый слайд, чтобы оптимизировать направляющие для впечатывания воска или параметры массива, чтобы гарантировать, что все функции будут находиться в камерах из восковой формы.
- Нагрейте вольфрам микрочипов до 85 ° C, пока весь воск не будет полностью расплавлен, приблизительно 30 минут.
- Вставьте слайд печатной стороной вниз и надавите на ползунок до упора справа на микроприемнике. Потяните рычаг, чтобы привести пресс-форму в контакт с поверхностью слайда. Держите в течение 2 с.
ПРИМЕЧАНИЕ. Время удержания может быть изменено для достижения оптимальной толщины границы воска. - Быстро снимите ползунок и визуально осмотрите границы воска, чтобы обеспечить закрытие всех колодцев и чтобы границы были не такими толстыми, чтобы они посягали на пятна. Храните слайды при температуре 4 ° C, защищенные от влаги и света.
ПРИМЕЧАНИЕ. Время удержания можно увеличить или уменьшить, чтобы получить более толстые или более тонкие границы.

Рисунок 3: Изготовление микрочипов. (A) Изготовление микрочипов гистонового пептида на покрытых стрептавидином микроскопах с использованием контактного микрочипового принтера. (B) Микроматрицы, изготовленные с 3 подмассивами сетки 48 x 48 пептидных признаков. Разделение (C) 3 подмассивов с помощью гидрофобного воскового пера, (D) 2 подмассива с кремниевым клеем и (E) 48 подмассивов с восковым отпечатком. Все показанные микрочипы изготовлены с использованием микроскопов размером 25 х 75 мм. Нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
5. Гибридизация антитела гистонового ПТМ с пептидным микрочипом
- Подготовьте буфер для гибридизации (PBS, pH 7,6, 5% BSA, 0,1% Tween-20).
- Уравновешивание слайда в буфере для гибридизацииИспользуя сосуд для гибридизации. Полностью покрыть весь слайд в буфере для гибридизации и инкубировать в течение 30 мин при 4 ° С на орбитальном шейкере с низкой скоростью.
- Подготовьте раствор, содержащий разбавленное гистоновое ПТМ-антитело в буфере для гибридизации.
ПРИМЕЧАНИЕ. В данных примера оба гистонового антитела # 1 и антитела # 2 разводились в буфере гибридизации 1: 1000. В качестве отправной точки рекомендуется использовать диапазон разбавления, аналогичный используемому для иммуноблоттинга. - Инкубируйте матрицу с раствором антитела в течение 1 часа при 4 ° C. Удаляют раствор антитела и промывают матрицу 3 раза в течение 5 мин при 4 ° С с холодным PBS, pH 7,6.
- Подготовьте 1: 5000 - 1: 10000 разведение флуоресцентного конъюгированного с красителем вторичного антитела в буфере для гибридизации.
- Инкубируйте массив со вторичным раствором антитела в течение 30 мин при 4 ° C, защищенном от света. Удалите раствор вторичного антитела и промойте горшок микрочипом 3 раза в течение 5 мин при 4 ° С с PBS, pH 7,6. наклонениеМикрочипе в 50-миллилитровой конической трубке, содержащей 0,1х PBS, pH 7,6 для удаления избытка соли при комнатной температуре. Просушите слайд в центрифуге с микрочипом при комнатной температуре.
- Сканирование слайда с помощью микрочипового сканера с разрешением 25 мкм или выше, следуя рекомендованному протоколом сканирования производителя микрочипов.
ПРИМЕЧАНИЕ. Если присутствует меченый меченым флуоресцеином биотин, сканируйте как зеленый канал (например: 488 нм, em: 509 нм), так и канал, соответствующий второму антителу с флуоресцентным конъюгированным с красителем красным (например: 635 нм, em : 677 нм). Целью сканирования является получение одноканальных файлов .tif, которые могут быть объединены в один .png-файл (как описано в разделе 6.1).
6. Анализ данных Microarray с использованием ArrayNinja
- Подготовка объединенного изображения микрочипов
ПРИМЕЧАНИЕ. Цель этого раздела - создать файл изображения .png, который объединяет два одноканальных файла .tif (полученные в разделе 5). ЭтаЯвляется единственным форматом изображения, совместимым с модулем анализа ArrayNinja. Следующие инструкции представляют собой один из возможных способов получения объединенного файла изображения. Однако доступны другие решения ( например , бесплатный ImageJ).- В командной строке на терминале bash компьютера с установленным программным обеспечением ImageMagick перейдите в папку, содержащую файлы с одним каналом .tif, и скопируйте / вставьте следующие шаги, нажав «enter» между каждым шагом (6.1.4 - 6.1 +0,7).
ПРИМЕЧАНИЕ. Имена файлов заглавными буквами ( например , RED_CHANNEL) следует заменить на имя файла изображений слайдов микрочипов. - При необходимости сначала инвертируйте изображения, используя команду «convert INPUT.tif -negate OUTPUT.tif». Это необходимо, если сканер сохраняет файлы .tif с белым и фоновым сигналом в черном цвете.
- Convert -depth 16 RED_CHANNEL.TIF -clone 0 -channel GB -определить набор 0 -delete 0 out.png 2> error.file.
- конвErt - depth 16 CONTROL_CHANNEL.TIF -clone 0 -channel RB -определить набор 0 -delete 0 outa.png 2> error.file.
- Convert -depth 16 CONTROL_CHANNEL.TIF -clone 0 -channel RG -определить набор 0 -delete 0 outB.png 2> error.file.
- Convert out.png outa.png outB.png -set цветовое пространство RGV -combine merged.png.
ПРИМЕЧАНИЕ. Файл с именем «merged.png» будет сохранен в той же папке, что и исходные .tif-файлы.
- В командной строке на терминале bash компьютера с установленным программным обеспечением ImageMagick перейдите в папку, содержащую файлы с одним каналом .tif, и скопируйте / вставьте следующие шаги, нажав «enter» между каждым шагом (6.1.4 - 6.1 +0,7).
- Количественная оценка данных с использованием ArrayNinja
- Откройте ArrayNinja и нажмите соответствующую ссылку на микрочиповый принтер в разделе «Для количественного определения изображений с известной исходной табличкой». Введите имя сохраненной конструкции слайда (шаг 2.5) в диалоговом окне «Загрузочная табличка» и нажмите «войти». См. Рисунок 4 для скриншота модуля анализа ArrayNinja.
- Нажмите «Выбрать файл» и перейдите к файлу «merged.png», который был создан в разделе 6.1. Объединенный образE будет загружаться, а также сетка для макета слайдов.
- Используйте ползунки в нижней части панели управления ArrayNinja, чтобы настроить контрастность, яркость и среднюю точку.
ПРИМЕЧАНИЕ. Эти корректировки предназначены только для визуализации и не влияют на количественную оценку. - Выберите «разрешение» и введите значение, соответствующее разрешению отсканированного изображения. Используйте «сторону поля» и «край поля», чтобы переместить сетку в соответствие с пятнами на массиве. Отрегулируйте по мере необходимости, чтобы сетка была максимально точно совмещена с каждым местом.
- Нажмите «Spot Seek» и дождитесь, пока кнопка вернется к исходному серому цвету.
ПРИМЕЧАНИЕ. Это можно повторить несколько раз, чтобы центрировать круги сетки на отдельных местах. Функция Seek ослабляет сетку в каждом месте, чтобы точно настроить выравнивание. - Измените значение «Super Arrays» на «1», чтобы индивидуально обрабатывать каждую панель под массива (при желании). Если вы используете 4X 12 ( рис. 3E ), используйте элементы управления «iPin» «jPin» и «subA», чтобы отключить функции для анализа любой желаемой комбинации из 4 x 12 колодцев. Например, чтобы проанализировать верхние левые и верхние правые лунки в качестве реплик друг друга, введите «1 4» в iPin, «1 1» в jPin и «1 4» в subA. Нажмите Ввод'.
- Наведите указатель мыши на отдельные точки, чтобы просмотреть личность этой функции.
- Нажмите «Переключить масштабирование», чтобы более тщательно просмотреть функции. Выберите фоновые опорные точки, нажав клавишу «R», когда мышь находится над пятном. Опорные точки будут выделены оранжевым цветом.
ПРИМЕЧАНИЕ. В режиме «переключать масштабирование» в верхнем правом углу отображается увеличенное изображение функции, над которой находится мышь. Подробное обсуждение дополнительных функций коррекции фона в ArrayNinja обсуждается в другом месте 28 . - Переключить пятна (с очень разнообразной морфологией пятна или мусором, которые могут повлиять на точную количественную оценку), нажав клавишу «A», наводя указатель мыши на пораженные участки. Инактивированные пятна станут белыми.
- Нажмите «заполнить», а затем «Отправить», чтобы количественно определить места.
ПРИМЕЧАНИЕ. Открывается новая вкладка и отображается гистограмма данных, нормализованная до самого яркого среднего пятна. Таблица ниже столбчатой диаграммы содержит как нормированные, так и необработанные значения данных. Эта таблица может быть скопирована в электронную таблицу для архивирования и дальнейшего анализа.
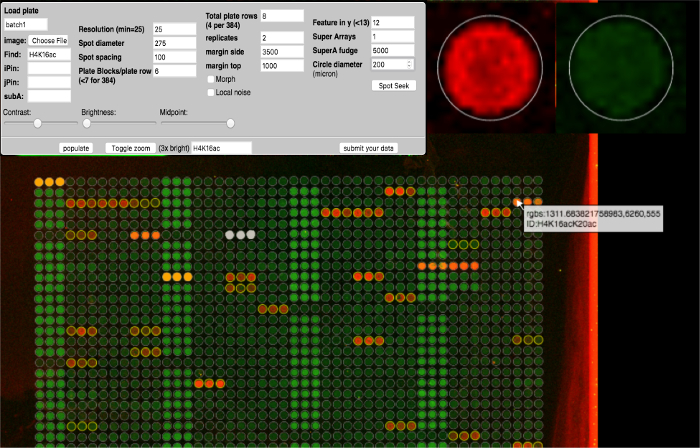
Рисунок 4: Модуль анализа ArrayNinja. Показан снимок экрана анализатора ArrayNinja. Панель управления (вверху слева) показывает все параметры, которые можно настроить для визуализации массива, поиска пятен и выравнивания сетки надОбраз массива. Наведение мыши над функцией показывает увеличенное изображение (вверху справа) и отображает всплывающее окно, которое содержит идентификационную информацию, связанную с этой функцией (внизу). Исходные точки, выбранные для коррекции фона, оранжевые. Функции, которые должны быть исключены из анализа ниже по потоку, являются белыми. ArrayNinja содержит текстовую функцию поиска, которая выделяет соответствующие функции желтым цветом, как показано в примере для H4K16. Нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Результаты
Этот протокол использовался для разработки и изготовления платформы пептидного микрочипа для анализа специфичности антитела к гистону ПТМ. Массив запрашивает библиотеку из более чем 300 уникальных пептидных объектов (20-40 остатков в длину), представляющих многие изве?...
Обсуждение
Надежность антител в приложениях для биомедицинских исследований имеет первостепенное значение 46 , 47 . Это особенно актуально в биохимии хроматина, учитывая положение антител в качестве ключевых инструментов для большинства методов, разработанных для ...
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Эта работа была частично поддержана Научно-исследовательским институтом Ван Анделя и грантом исследований Национального института здоровья (CA181343) на SBR
Материалы
| Name | Company | Catalog Number | Comments |
| Printing Buffer | ArrayIt | PPB | |
| BSA | Omnipure | 2390 | |
| Streptavidin-coated glass microscope slides | Greiner Bio-one | 439003-25 | |
| polypropylene 384 well plate | Greiner Bio-one | 784201 | |
| Biotin-fluorescein | Sigma | 53608 | |
| contact microarray printer | Aushon | 2470 | Aushon 2470 Microarray Printer |
| contact microarray printer | Gene Machines | OmniGrid 100 | OmniGrid Microarray Printer |
| PBS | Invitrogen | 14190 | |
| Blocking Buffer | ArrayIt | SBB | |
| Hydrophobic wax pen | Vector Labs | H-4000 | ImmEdge Hydrophobic Barrier PAP Pen |
| Silicon Gasket | Grace Bio-labs | 622511 | |
| Hybridization Vessel | Thermo Scientific | 267061 | or similar vessel |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21244 | Alexa Fluor 647 (anti-rabbit) |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21235 | Alexa Fluor 647 (anti-mouse) |
| Wax Imprinter | ArrayIt | MSI48 | |
| Tween-20 | Omnipure | 9490 | |
| Microarray Scanner | Innopsys | InnoScan 1100AL | or equivalent microarray scanner |
| EipTitan Histone Peptide Microarray | Epicypher | 112001 | |
| AbSurance Pro Histone Peptide Microarray | Millipore | 16668 | |
| MODified Histone Peptide Array | Active Motif | 13001 | |
| Histone Code Peptide Microarrays | JPT | His_MA_01 | |
| Wax | Royal Oak | GulfWax | for wax imprinter |
| Humidified Microarray Slide Hybridization Chamber | VWR | 97000-284 | |
| High throughput microscope slide washing chamber | ArrayIt | HTW | |
| Microscope slide centrifuge | VWR | 93000-204 | |
| Antibody 1 | Abcam | 8898 | |
| Antibody 2 | Millipore | 07-473 | |
| Biotinylated histone peptide | EpiCypher | 12-0001 | Example peptide. Similar peptides with various modifications are available from several commercial sources. |
| ImageMagick | https://www.imagemagick.org/script/index.php | ||
| ArrayNinja | https://rothbartlab.vai.org/tools/ |
Ссылки
- van Steensel, B. Chromatin: constructing the big picture. EMBO J. 30 (10), 1885-1895 (2011).
- Kouzarides, T. Chromatin modifications and their function. Cell. 128 (4), 693-705 (2007).
- Rothbart, S. B., Strahl, B. D. Interpreting the language of histone and DNA modifications. Biochim Biophys Acta. 1839 (8), 627-643 (2014).
- Shogren-Knaak, M., Ishii, H., Sun, J. -. M., Pazin, M. J., Davie, J. R., Peterson, C. L. Histone H4-K16 acetylation controls chromatin structure and protein interactions. Science. 311 (5762), 844-847 (2006).
- Musselman, C. A., Lalonde, M. -. E., Côté, J., Kutateladze, T. G. Perceiving the epigenetic landscape through histone readers. Nat Struct Mol Biol. 19 (12), 1218-1227 (2012).
- Huang, H., Sabari, B. R., Garcia, B. A., Allis, C. D., Zhao, Y. SnapShot: Histone Modifications. Cell. 159 (2), 458 (2014).
- Strahl, B. D., Allis, C. D. The language of covalent histone modifications. Nature. 403 (6765), 41-45 (2000).
- Rothbart, S. B., Krajewski, K., et al. Association of UHRF1 with methylated H3K9 directs the maintenance of DNA methylation. Nat Struct Mol Biol. 19 (11), 1155-1160 (2012).
- Wang, Z., Zang, C., et al. Combinatorial patterns of histone acetylations and methylations in the human genome. Nat Genet. 40 (7), 897-903 (2008).
- Stunnenberg, H. G., Hirst, M. The International Human Epigenome Consortium: A Blueprint for Scientific Collaboration and Discovery. Cell. 167 (5), 1145-1149 (2016).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Egelhofer, T. A., Minoda, A., et al. An assessment of histone-modification antibody quality. Nat Struct Mol Biol. 18 (1), 91-93 (2011).
- Bock, I., Dhayalan, A., Kudithipudi, S., Brandt, O., Rathert, P., Jeltsch, A. Detailed specificity analysis of antibodies binding to modified histone tails with peptide arrays. Epigenetics. 6 (2), 256-263 (2011).
- Busby, M., Xue, C., et al. Systematic comparison of monoclonal versus polyclonal antibodies for mapping histone modifications by ChIP-seq. Epigenetics Chromatin. 9, 49 (2016).
- Fuchs, S. M., Krajewski, K., Baker, R. W., Miller, V. L., Strahl, B. D. Influence of combinatorial histone modifications on antibody and effector protein recognition. Curr Biol. 21 (1), 53-58 (2011).
- Kungulovski, G., Jeltsch, A. Quality of histone modification antibodies undermines chromatin biology research. F1000Research. 4, 1160 (2015).
- Rothbart, S. B., Dickson, B. M., et al. An Interactive Database for the Assessment of Histone Antibody Specificity. Mol Cell. 59 (3), 502-511 (2015).
- Rothbart, S. B., Lin, S., et al. Poly-acetylated chromatin signatures are preferred epitopes for site-specific histone H4 acetyl antibodies. Sci Rep. 2, 489 (2012).
- Berger, M. F., Bulyk, M. L. Universal protein-binding microarrays for the comprehensive characterization of the DNA-binding specificities of transcription factors. Nat Protoc. 4 (3), 393-411 (2009).
- Hu, S., Wan, J., et al. DNA methylation presents distinct binding sites for human transcription factors. eLife. 2, e00726 (2013).
- Moore, C. D., Ajala, O. Z., Zhu, H. Applications in high-content functional protein microarrays. Curr Opin Chem Biol. 30, 21-27 (2016).
- MacBeath, G., Schreiber, S. L. Printing proteins as microarrays for high-throughput function determination. Science. 289 (5485), 1760-1763 (2000).
- Rothbart, S. B., Krajewski, K., Strahl, B. D., Fuchs, S. M. Peptide microarrays to interrogate the "histone code" . Methods Enzymol. 512, 107-135 (2012).
- Cornett, E. M., Dickson, B. M., et al. Substrate Specificity Profiling of Histone-Modifying Enzymes by Peptide Microarray. Methods Enzymol. 574, 31-52 (2016).
- Nady, N., Min, J., Kareta, M. S., Chédin, F., Arrowsmith, C. H. A SPOT on the chromatin landscape? Histone peptide arrays as a tool for epigenetic research. Trends Biochem Sci. 33 (7), 305-313 (2008).
- Dieker, J., Berden, J. H., et al. Autoantibodies against Modified Histone Peptides in SLE Patients Are Associated with Disease Activity and Lupus Nephritis. PLoS ONE. 11 (10), (2016).
- Price, J. V., Tangsombatvisit, S., et al. "On silico" peptide microarrays for high-resolution mapping of antibody epitopes and diverse protein-protein interactions. Nat Med. 18 (9), 1434-1440 (2012).
- Dickson, B. M., Cornett, E. M., Ramjan, Z., Rothbart, S. B. ArrayNinja: An Open Source Platform for Unified Planning and Analysis of Microarray Experiments. Methods Enzymol. 574, 53-77 (2016).
- Gatchalian, J., Fütterer, A., et al. Dido3 PHD modulates cell differentiation and division. Cell Rep. 4 (1), 148-158 (2013).
- Cai, L., Rothbart, S. B., et al. An H3K36 methylation-engaging Tudor motif of polycomb-like proteins mediates PRC2 complex targeting. Mol Cell. 49 (3), 571-582 (2013).
- Rothbart, S. B., Dickson, B. M., et al. Multivalent histone engagement by the linked tandem Tudor and PHD domains of UHRF1 is required for the epigenetic inheritance of DNA methylation. Genes Dev. 27 (11), 1288-1298 (2013).
- Ali, M., Rincón-Arano, H., et al. Molecular basis for chromatin binding and regulation of MLL5. Proc Natl Acad Sci U S A. 110 (28), 11296-11301 (2013).
- Kinkelin, K., Wozniak, G. G., Rothbart, S. B., Lidschreiber, M., Strahl, B. D., Cramer, P. Structures of RNA polymerase II complexes with Bye1, a chromatin-binding PHF3/DIDO homologue. Proc Natl Acad Sci U S A. 110 (38), 15277-15282 (2013).
- Klein, B. J., Piao, L., et al. The histone-H3K4-specific demethylase KDM5B binds to its substrate and product through distinct PHD fingers. Cell Rep. 6 (2), 325-335 (2014).
- Kim, H. -. S., Mukhopadhyay, R., et al. Identification of a BET family bromodomain/casein kinase II/TAF-containing complex as a regulator of mitotic condensin function. Cell Rep. 6 (5), 892-905 (2014).
- Greer, E. L., Beese-Sims, S. E., et al. A histone methylation network regulates transgenerational epigenetic memory in C. elegans. Cell Rep. 7 (1), 113-126 (2014).
- Andrews, F. H., Tong, Q., et al. Multivalent Chromatin Engagement and Inter-domain Crosstalk Regulate MORC3 ATPase. Cell Rep. 16 (12), 3195-3207 (2016).
- Sidoli, S., Lin, S., Karch, K. R., Garcia, B. A. Bottom-Up and Middle-Down Proteomics Have Comparable Accuracies in Defining Histone Post-Translational Modification Relative Abundance and Stoichiometry. Anal Chem. 87 (6), 3129-3133 (2015).
- Tsukada, Y., Ishitani, T., Nakayama, K. I. KDM7 is a dual demethylase for histone H3 Lys 9 and Lys 27 and functions in brain development. Genes Dev. 24 (5), 432-437 (2010).
- Tachibana, M., Sugimoto, K., Fukushima, T., Shinkai, Y. Set domain-containing protein, G9a, is a novel lysine-preferring mammalian histone methyltransferase with hyperactivity and specific selectivity to lysines 9 and 27 of histone H3. J Biol Chem. 276 (27), 25309-25317 (2001).
- Wu, H., Chen, X., et al. Histone methyltransferase G9a contributes to H3K27 methylation in vivo. Cell Res. 21 (2), 365-367 (2011).
- Koch, C. M., Andrews, R. M., et al. The landscape of histone modifications across 1% of the human genome in five human cell lines. Genome Res. 17 (6), 691-707 (2007).
- Okitsu, C. Y., Hsieh, J. C. F., Hsieh, C. -. L. Transcriptional Activity Affects the H3K4me3 Level and Distribution in the Coding Region. Mol Cell Biol. 30 (12), 2933-2946 (2010).
- Zentner, G. E., Tesar, P. J., Scacheri, P. C. Epigenetic signatures distinguish multiple classes of enhancers with distinct cellular functions. Genome Res. 21 (8), 1273-1283 (2011).
- Garske, A. L., Oliver, S. S., et al. Combinatorial profiling of chromatin binding modules reveals multisite discrimination. Nat Chem Biol. 6 (4), 283-290 (2010).
- Baker, M. Reproducibility crisis: Blame it on the antibodies. Nature. 521 (7552), 274-276 (2015).
- Bradbury, A., Plückthun, A. Reproducibility: Standardize antibodies used in research. Nature. 518 (7537), 27-29 (2015).
- Nguyen, U. T. T., Bittova, L., et al. Accelerated chromatin biochemistry using DNA-barcoded nucleosome libraries. Nat Methods. 11 (8), 834-840 (2014).
- Frank, R. Spot-synthesis: an easy technique for the positionally addressable, parallel chemical synthesis on a membrane support. Tetrahedron. 48 (42), 9217-9232 (1992).
- Hilpert, K., Winkler, D. F. H., Hancock, R. E. W. Peptide arrays on cellulose support: SPOT synthesis, a time and cost efficient method for synthesis of large numbers of peptides in a parallel and addressable fashion. Nat Protoc. 2 (6), 1333-1349 (2007).
- Kudithipudi, S., Kusevic, D., Weirich, S., Jeltsch, A. Specificity analysis of protein lysine methyltransferases using SPOT peptide arrays. J Vis Exp. (93), e52203 (2014).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены