このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ペプチドマイクロアレイによるヒストン抗体特異性の解析
要約
この原稿では、ヒストンとその翻訳後修飾を認識する抗体の特異性プロファイリングにペプチドマイクロアレイ技術を適用する方法が記載されています。
要約
ヒストンタンパク質の翻訳後修飾(PTM)は、クロマチン構造および遺伝子発現の調節におけるそれらの役割について広く研究されている。ヒストンPTMに特異的な抗体の大量生産および分布は、これらのマークの研究を非常に容易にした。ヒストンPTM抗体は多くのクロマチン生化学アプリケーションにとって重要な試薬であるため、正確なデータ解釈と現場での継続的な進歩には抗体特異性の厳密な解析が必要です。このプロトコルは、ヒストン抗体の特異性をプロファイリングするためのペプチドマイクロアレイの設計、製作および使用のための統合されたパイプラインを記載している。この手順の設計と分析の側面は、マイクロアレイ印刷フォーマットのカスタマイズを合理化するために最近開発したオープンソースのインタラクティブなソフトウェアパッケージであるArrayNinjaによって容易になります。このパイプラインは、市販されて広く使用されている多数のヒストンPTM抗体のスクリーニングに使用されているこれらの実験から生成されたデータは、オンラインおよび拡大Histone Antibody Specificity Databaseを通じて自由に入手できます。ヒストンを越えて、本明細書に記載の一般的な方法論は、PTM特異的抗体の分析に広く適用することができる。
概要
ゲノムDNAは、ヒストンタンパク質を含む真核細胞の核内にエレガントにパッケージングされ、クロマチンを形成する。クロマチンの反復サブユニットはヌクレオソームであり、これはヒストンタンパク質の八量体コア(H2A、H2B、H3、およびH4 1)のまわりに包まれた147塩基対のDNAからなる。クロマチンは、ゆるやかに詰まったユークロマチンおよび密に詰まったヘテロクロマチンドメインに広く組織されている。クロマチン圧縮の程度は、複製、転写、および修復のような基本的なDNA鋳型プロセスを実行するために、タンパク質機械が根底にあるDNAにアクセスできる程度を調節する。
クロマチンの文脈におけるゲノムアクセシビリティの重要な調節因子は、ヒストンタンパク質2、3の構造化されていない尾とコアドメイン上のPTMです。ヒストンPTMは、クロマチン4の構造に直接的に作用し、間接的に作用する染色体リモデリング、酵素的および足場活性を有するリーダータンパク質および関連する巨大分子複合体の動員5 。過去20年にわたるヒストンPTM機能の研究は、これらのマークが細胞運命、生物発達、および疾患の開始/進行の調節において重要な役割を果たすことを圧倒的に示唆している。質量分析に基づくプロテオーム技術の進歩により、80以上の異なるヒストン残基に20以上のユニークなヒストンPTMが発見されました6 。注目すべきことに、これらの修飾は、しばしば組み合わせで発生し、「ヒストンコード」仮説と一致して、多くの研究は、リーダタンパク質がヒストンのPTM 7、8、9の特定の組み合わせの認識を介してクロマチンの別個の領域を標的とすることを示唆しています。重要な課題は、関数をgrに割り当てることですヒストンPTMの特定の組み合わせがどのようにしてクロマチンに関連する動的機能を調整するかを決定することができる。
抗体は、ヒストンPTMの検出のためのリンチピン試薬である。このように、クロマチン生化学研究に使用するために、1,000を超えるヒストンPTM特異的抗体が商業的に開発されている。ハイスループットDNA配列決定技術の急速な発展に伴い、これらの試薬は、チップ配列の個々の研究者および大規模エピゲノミクス「ロードマップ」の取り組み( 例えば 、ENCODEおよび青写真)によって広く使用されている(次世代シークエンシングと結合クロマチン免疫沈降)パイプラインヒストンPTM分布ゲノムワイド10、11の高解像度空間マップを生成します。しかし、最近の研究により、ヒストンPTM抗体の特異性は非常に可変であり得、これらの試薬は非特定の残基に変更順序を識別ようなオフターゲットエピトープ認識、隣接のPTMによる強い正および負の影響、及び困難としてavorable特性( 例えば 、モノ- 、ジ- 、またはトリ-メチルリジン)12、13、14、15 、16、17、18。したがって、これらの貴重な試薬で生成されたデータを正確に解釈するには、ヒストンPTM特異的抗体試薬の厳密な品質管理が必要です。
マイクロアレイ技術は、ハイスループットで、再現性があり、かつ小型化されたフォーマットで、何千もの高分子相互作用の同時検査を可能にする。このため、マイクロアレイプラットフォームの様々なタンパク質-DNA 19を分析するために作成されています、「> 20、タンパク質-タンパク質21、及びタンパク質-ペプチド相互作用22。実際、ヒストンペプチドマイクロアレイは消しゴム、作家のハイスループットプロファイリングを可能にする、クロマチン生化学研究のための有益な発見プラットフォームとして浮上し、そしてヒストンのPTM 15の読者ました、23、24、またヒストン抗体特異性17、25の分析のため。クロマチンとエピジェネティクス研究におけるそれらの用途を超えて、ヒストンペプチド配列は、抗全身性エリテマトーデスおよび他の自己免疫疾患の診断/予後診断検査としての潜在的有用性を有しますクロマチン自己抗体は26、27を生成しています。
ここでは、設計、製造、および開発のために開発した統合パイプラインについて説明しますヒストンおよびそれらのPTMを認識する抗体の特異性プロファイルを生成するためにヒストンペプチドマイクロアレイをライニングする。このパイプラインは、我々が最近開発したオープンソースのインタラクティブなソフトウェアアプリケーションであるArrayNinjaによって容易にされ、マイクロアレイ実験の設計と解析段階を統合している28 。 ArrayNinjaはGoogle Chromeで最適に動作します。簡単に述べると、ロボット接触マイクロアレイプリンターを用いて、ストレプトアビジン被覆ガラス顕微鏡スライド上の規定された位置にビオチン結合ヒストンペプチドのライブラリーを沈着させる。次いで、抗体 - エピトープ相互作用を調べるために、競合的および並行アッセイ形式でアレイを使用することができる( 図1 )。ペプチドライブラリーは、PTM(リジンアセチル化、リシン/アルギニンメチル化、およびセリン/スレオニンリン酸化)を単独で、プロテオミクスデータセットに主に由来する関連する組み合わせを有する何百ものユニークな合成ペプチドからなる。ペプチド合成および検証のための方法他の場所で詳述されている23 。このアレイプラットフォームを利用した、現在行われているヒストンPTM抗体スクリーニングの取り組みから得られたデータは、公開されているWebリソース、Histone Antibody Specificity Database(www.histoneantibodies.com)に保存されています。注目すべきことに、このプロトコルのバリエーションで製造ヒストンペプチドマイクロアレイはまた、ヒストンPTMリーダードメイン8、29、30、31、32、33、34、35、36、37の活性を特徴づけるために広く使用されており、より最近ヒストンをプロファイルしますPTMライターと消しゴムの活動24 。
/files/ftp_upload/55912/55912fig1.jpg "/>
図1:ヒストンペプチドマイクロアレイ上の抗体スクリーニングのための段階的手順の漫画の描写。定義された翻訳後修飾(赤色および青色の円)を有するビオチン化ヒストンペプチドを、ストレプトアビジン被覆ガラス上のビオチン - フルオレセインと同時プリントする。陽性の相互作用は、赤色蛍光として視覚化される。 この図の拡大版を見るには、ここをクリックしてください。
プロトコル
1. ArrayNinjaのインストールと実行
- www.virtualbox.orgからOracle Virtual Boxをダウンロードしてインストールします。
- http://research.vai.org/Tools/arrayninjaからArrayNinja仮想マシン(VM)をダウンロードして解凍します。
- バーチャルボックスを開き、 'Machine'、 'Add'をクリックしてArrayNinja VMを追加し、ArrayNinja VMが保存されていたフォルダからarrayninja.vboxを選択します。
- 仮想ボックス内でArrayNinjaを選択し、緑色の「開始」矢印をクリックしてArrayNinjaを起動します。
- バーチャルボックスは新しいウィンドウを開き、Webブラウザからlocalhost:2080にナビゲートしてVMにアクセスできることを示すメッセージを表示します。60;注:ArrayNinjaのコンテナ化バージョンは、hub.docker.com/r/bradley.dickson/arrayninja/ からも入手できます。
アレイスライドとソースプレートレイアウトの設計
- ArrayNinjaインターフェースの「スライドレイアウトを計画する」の下で、使用しているマイクロアレイプリンタに対応するリンクをクリックします。
注:ArrayNinjaは、一般的に使用されている2つのマイクロアレイプリンタのロボット動作を模倣するようにプログラムされています( 表1)。要求に応じて、他のアレイとの互換性を設定することができます。 - 'プレートをロード'ダイアログボックスの内部をクリックし、「空」と入力して「入力」をクリックします。 ArrayNinjaデザインモジュールのスクリーンショットについては、 図2を参照してください。
- スポット径を275μmに、スポット間隔を375μmに調整します。残りの設定(Plate Blocks / Plate Row、Total plate Rows、Replicates、yの機能、Super Arrays、SuperA fudge; 図2参照)を調整して、マイクロアレイスライドに機能がどのように表示されるかをカスタマイズします。
注:スポット径は、マイクロアレイピンのサイズによって決まります。これらの設定が調整されると、漫画スライドがリアルタイムで更新されます。この漫画を使用して、各設定が最終スライドレイアウトを変更する方法をプレビューします。 - スライド上のフィーチャのレイアウトが確定したら、各固有フィーチャにマウスを移動し、ポップアップダイアログボックスにフィーチャ識別子を入力します。
注:機能識別子は、数字、文字、または組み合わせで構成できます。これは一意のフィーチャにのみ必要であり、複製が選択されてもダイアログボックスは表示されません。 - すべての固有の機能に識別子が割り当てられたら、[入力]をクリックします。スライドレイアウトの名前を入力し、[プレートを印刷]をクリックして保存します。選択したスライドデザインを作成するために必要な384ウェルソースプレートの数とソースプレートにロードする各フィーチャーの物理的位置をマッピングするテーブルが表示された新しいページが開きます。
注:マイクロアレイデータを分析するときにこのデザインを呼び出すために使用されるので、この名前を覚えておいてください(セクション6.2を参照)。 'あなたのプレートを印刷する'をクリックすると、ArrayNinja内にレイアウトが保存されます。

図2:ArrayNinjaデザインモジュールスクリーンショット ArrayNinjaデザインモジュールは点線で示されています。コントロールパネル(上部)には、マイクロアレイプリンタで変更可能なすべてのパラメータが表示されます。これらのパラメータが調整されると、スライドレイアウトの漫画画像(左下)がリアルタイムで更新されます。レイアウトが設定された後、ユーザーは個々のスポットにマウスを置いて一意のフィーチャ識別子を入力できます。 ArrayNinjaは、指定されたマイクロアレイのスライドレイアウトを作成するために必要なソースプレート(右下)の各フィーチャーの位置のマップをこのユーザー入力から構築します。 この図の拡大版を見るには、ここをクリックしてください。
3.マイクロアレイの作製
- ソースプレートの準備
- セクション2.5のArrayNinjaで生成されたマップを使用して、384ウェルのソースプレートを作成します。
注:このプラットフォームで検索されたペプチドの詳細な説明は、他の場所で見つけることができますs = "xref"> 17。 - 1〜2μLの各フィーチャー( 例えばビオチン化ヒストンペプチド)を384ウェルソースプレートの正しいウェルに入れる。
注:ビオチン化されたヒストンペプチドは、典型的には、200〜400μMのストック溶液から沈着され、これは、単一のアレイスポットにおいて10〜25倍モル過剰のペプチド対ストレプトアビジン結合部位に等しい。これは、次の式を使用して計算されます。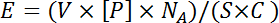
ここで、 Vはピンによって供給される体積であり、[ P ]は印刷されるフィーチャの濃度であり、 N Aはアボガドロの数であり、 Sはスポットの表面積である。 Cは単位面積当たりのストレプトアビジン分子数に3を掛けた数(利用可能なストレプトアビジン結合部位の平均数)として表されるスライドのカバレッジである。 VおよびCはそれぞれの製造業者によって得られる。その他の機能はクラウディングが懸念される生体分子のサイズに応じて異なる濃度を必要とする。新しいタイプのフィーチャのそれぞれについての印刷濃度の範囲は、最適な印刷濃度を決定するために経験的に試験されるべきである。 - 1%ウシ血清アルブミン(BSA)を添加した1x印刷バッファーで各フィーチャーを10倍に希釈する。ソースプレートを室温で2分間500 xgで回転させます。
注:蛍光バッファー中にフルオレセイン標識ビオチン(5μg/μL)を含めることは、スポッティングコントロールとして、また分析中の適切なアレイアライメントを容易にするための視覚的補助として推奨されます( 図4参照)。
- セクション2.5のArrayNinjaで生成されたマップを使用して、384ウェルのソースプレートを作成します。
- 印刷プロトコル( 図3A -B )。
- 廃棄物収集容器を空にし、洗浄液容器と加湿器容器に滅菌蒸留水を満たしてアレイを準備する。
- パラメータuを入力してくださいArrayNinja(セクション2と図2 )のスライドをマイクロアレイのプリンタ制御プログラムに設計することができました。
- マイクロアレイプリンタ制御プログラムを使用して、1回の浸漬で1秒間の洗浄のための洗浄手順を設定する。各洗浄後に5回ピンを再浸漬するように洗浄後の設定を行います。湿度を60%に設定します。
注:最適な洗浄構成は、使用されているマイクロアレイプリンタによって異なる場合があります。最適な洗浄後ピンディップ構成は、使用されているマイクロアレイプリンタによって異なる場合があります。 - 官能基化されたスライド( 例えば 、ストレプトアビジン被覆ガラス)を基材プラテンに挿入し、全てのプラテンをプラテンエレベータに入れる。ソースプレートをプレートホルダーに挿入し、ソースプレートエレベーターの中に置きます。
- [印刷]をクリックします。すべての洗浄と浸漬の設定が正しいことを確認するために、数回の機能析出の印刷プロセスを監視します。印刷実行が完了したら、基板はアレイ装置からプラテンされる。
注:大量の印刷が1日以内にブロックステップの完了を禁止する場合、プリントされたスライドは、加湿チャンバー内で4℃で一晩インキュベートすることができます。プラスチックラップでシールされた段ボール箱の内側に小さなビーカーの横にスライドをインキュベートする。 - ブロッキングバッファーでスライドを混合しながら室温で30分間ブロックする。
- スライドを、リン酸緩衝生理食塩水(PBS)(pH7.6)中、室温で2×10分間、混合しながら洗浄する。室温で30秒間、マイクロアレイスライド遠心分離機で回転させることによってスライドを乾燥させる。
注:一度に多数のスライドを処理するには、ハイスループットの顕微鏡スライド洗浄チャンバーを使用して、50枚のスライドを並行して洗浄することができます。 - ワックスで区画されるように設計されたスライドについては、セクション4.1に進んでください。他のすべてのデザインでは、光と湿気から保護された4℃でスライドを保管してください。
注:プリントされたビオチン化ヒストンペプチドこの方法で保管した場合、少なくとも6ヶ月間安定です。
4.マイクロアレイスライドの分割
- 疎水性ワックスペン( 図3C )
- ワックスペンを使用してフィーチャーを含む領域の周りにワックスを塗布します。ワックスを5分間空気乾燥させてから、セクション5に進んでください。
注:ブロッキング後、アレイスポットは目で視覚化するのが非常に難しい場合があります。 ArrayNinjaのスライドデザインは、スケールに従って印刷し、ワックス塗布のガイドとして使用することができます。
- ワックスペンを使用してフィーチャーを含む領域の周りにワックスを塗布します。ワックスを5分間空気乾燥させてから、セクション5に進んでください。
- シリコンガスケット( 図3D )
- アレイのガスケットの裏側から透明フィルムを剥がし、接着剤の面をマイクロアレイのスライドの上に置きます。
- セクション5に進む前に、ガスケットを5秒間定位置に保持してください。
- ワックスインプリント( 図3E )
- ワックスで区画されるように設計されたスライドの場合、10%BSAを用いてプレーングラス上に試験スライドを印刷する。このテストスライドを使用して、ワックスインプリンターガイドまたはアレイ設定を最適化して、すべてのフィーチャーがワックス成形チャンバー内に収まるようにします。
- すべてのワックスが完全に融解するまで、約30分、マイクロアレイワックスインプリンターを85℃に加熱する。
- 印刷された面を下にしてスライドを挿入し、スライドをマイクロアレイインプリンターの右側のガイドまで押してください。レバーを引き上げて、金型をスライドの表面に接触させます。 2秒間保持する。
注記:最適なワックスボーダー厚さを達成するために保持時間を変更することができます。 - スライドをすばやく外し、ワックスの境界線を目視検査して、すべての井戸が囲まれていること、境界が侵入していることを確認してください。水分と光から保護された4℃でスライドを保管してください。
注記:ホールドタイムを長くしたり減らしたりすることで、より太いまたは薄いボーダーを得ることができます。

図3:マイクロアレイ作製。 (A)接触マイクロアレイプリンターを用いたストレプトアビジン被覆顕微鏡スライド上のヒストンペプチドマイクロアレイ作製。 (B) 48×48グリッドのペプチドフィーチャーの3つのサブアレイで作製されたマイクロアレイ。 (C) 3個のサブアレイを疎水性ワックスペンで、 (D) 2個のサブアレイをシリコン接着剤で、(E)48個のサブアレイをワックスインプリントで分離する。示された全てのマイクロアレイは、25×75mmの顕微鏡スライドを用いて作製される。 この図の拡大版を見るには、ここをクリックしてください。
5.ヒストンPTM抗体とペプチドマイクロアレイとのハイブリッド形成
- ハイブリダイゼーションバッファー(PBS、pH 7.6,5%BSA、0.1%Tween-20)を調製する。
- スライドをハイブリダイゼーションバッファーで平衡化するハイブリダイゼーション容器を用いて行った。ハイブリダイゼーションバッファー中のスライド全体を完全に覆い、低速でオービタルシェーカーで4℃で30分間インキュベートする。
- ハイブリダイゼーションバッファーに希釈ヒストンPTM抗体を含む溶液を調製する。
注:サンプルデータでは、ヒストン抗体#1と抗体#2の両方をハイブリダイゼーションバッファーで1:1,000に希釈しました。イムノブロッティングに似た希釈範囲が出発点として推奨されます。 - 4℃で1時間、抗体溶液とアレイをインキュベートする。抗体溶液を除去し、冷PBS、pH 7.6で4℃で5分間3回アレイを洗浄する。
- ハイブリダイゼーション緩衝液中の蛍光色素結合二次抗体の1:5,000 - 1:10,000希釈液を調製する。
- 光から保護された4℃で30分間、二次抗体溶液とアレイをインキュベートする。二次抗体溶液を除去し、PBS、pH 7.6で4℃で5分間3回マイクロアレイスライドを洗浄する。浸漬マイクロアレイを、室温で過剰の塩を除去するために0.1xPBS、pH7.6を含む50mLコニカルチューブに入れた。スライドを室温でマイクロアレイスライド遠心分離機で乾燥させる。
- マイクロアレイスキャナーの推奨スキャンプロトコルに従い、25μm以上の解像度でマイクロアレイスキャナーでスライドをスキャンします。
注:フルオレセイン標識ビオチントレーサーが存在する場合、緑色チャネル(例:488 nm、em:509 nm)および蛍光色素結合二次抗体に対応するチャネル(通常は赤色(例:635 nm、em :677nm)。スキャンの目的は、単一の.pngファイルにマージできる単一チャネルの.tifファイルを取得することです(6.1項を参照)。
ArrayNinjaを用いたマイクロアレイデータ解析
- マージされたマイクロアレイ画像の準備
注:このセクションの目的は、2つのシングルチャネル.tifファイル(セクション5で取得)をマージする.pngイメージファイルを作成することです。このArrayNinja分析モジュールと互換性のある唯一の画像フォーマットです。以下の手順は、マージされたイメージファイルを取得する方法の1つです。しかし、他の解決法が利用可能である( 例えばフリーウェアのImageJ)。- フリーウェアのImageMagickがインストールされているコンピュータのbashターミナルのコマンドラインから、単一チャンネルの.tifファイルを含むフォルダに移動し、次のステップをコピー/ペーストし、各ステップ(6.1.4 - 6.1 。7)。
注:大文字のファイル名( 例: RED_CHANNEL)は、マイクロアレイのスライド画像のファイル名に置き換えてください。 - 必要に応じて、 'convert INPUT.tif -negate OUTPUT.tif'コマンドを使用して、まず画像を反転させます。これは、スキャナーが信号が白と黒の背景の.tifファイルを保存する場合に必要です。
- 変換-depth 16 RED_CHANNEL.TIF - クローン0 - チャネルGB - セット0を削除 - 削除0 out.png 2> error.file。
- コンバージョンert - depth 16 CONTROL_CHANNEL.TIF - クローン0 - チャネルRB - セット0を削除 - アウトレット0 outa.png 2> error.file。
- 変換 - 深さ16 CONTROL_CHANNEL.TIF - クローン0 - チャンネルRG - セット0を削除 - 削除0 outB.png 2> error.file。
- convert out.png outa.png outB.png -set colorspace RGV -combine merged.png。
注: 'merged.png'という名前のファイルは、元の.tifファイルと同じフォルダに保存されます。
- フリーウェアのImageMagickがインストールされているコンピュータのbashターミナルのコマンドラインから、単一チャンネルの.tifファイルを含むフォルダに移動し、次のステップをコピー/ペーストし、各ステップ(6.1.4 - 6.1 。7)。
- ArrayNinjaを使用したデータの数量化
- ArrayNinjaを開き、「既知のソースプレートを有する画像を定量する」の下にある適切なマイクロアレイプリンタリンクをクリックしてください。 [ロードプレート]ダイアログボックスで保存したスライドデザインの名前を入力し(ステップ2.5)、[入力]をクリックします。 ArrayNinja解析モジュールのスクリーンショットについては、 図4を参照してください。
- [ファイルを選択]をクリックし、セクション6.1で作成した 'merged.png'ファイルに移動して選択します。マージされたイメージeはスライドレイアウト用のグリッドと同様に読み込まれます。
- ArrayNinjaコントロールパネルの下部にあるスライダを使用して、コントラスト、輝度、および中間点を調整します。
注:これらの調整は視覚化の目的のみであり、定量化には影響しません。 - 「解像度」を選択し、スキャンした画像の解像度に一致する値を入力します。グリッドをアレイ上のスポットと整列させるには、「マージン側」と「マージントップ」を使用します。必要に応じて調整して、可能な限り各スポット上でグリッドを緊密に整列させます。
- 'Spot Seek'をクリックし、ボタンが元の灰色に戻るまで待ちます。
注:これは、グリッド円を個々のスポットの中心に合わせるために何度か繰り返すことができます。 Seek機能は、グリッドを各スポットに向かって緩やかにしてアラインメントを微調整します。 - スーパーアレイの値を「1」に変更して、各サブアレイパネルを個別に処理します(必要な場合)。 4を使用する場合x 12ワックスインプリントスライドレイアウト( 図3E )、「iPin」「jPin」および「subA」コントロールを使用してフィーチャをオフにして、4 x 12ウェルの任意の組み合わせを分析します。例えば、左上のウェルと右上のウェルをお互いの複製として分析するには、iPinに「1 4」、jPinに「1 1」、subAに「1 4」を入力します。 'enter'を押します。
- マウスを個々のスポットに合わせると、そのフィーチャのアイデンティティが表示されます。
- 機能をより慎重に確認するには、[Toggle zoom]をクリックしてください。マウスがある場所にいるときに 'R'キーを押して、背景参照スポットを選択します。基準点はオレンジ色で強調表示されます。
注記:「トグルズーム」モードでは、マウスの上にある機能の拡大画像が右上隅に表示されます。 ArrayNinjaに追加のバックグラウンド補正機能の詳細な議論は別の場所で28説明されています。 - 影響を受けたスポットの上にマウスを置いている間に「A」キーを押すことにより、(正確に定量化に影響を及ぼす広範囲に変化したスポット形態または残骸を有する)スポットをトグルオフする。不活性化したスポットは白くなります。
- スポットを定量化するには、「投入」をクリックしてから「送信」をクリックします。
注記:新しいタブが開き、最も明るいスポットの平均値に正規化されたデータの棒グラフが表示されます。棒グラフの下の表には、正規化されたデータ値と生データ値の両方が含まれています。この表は、アーカイブおよび詳細分析のためにスプレッドシートにコピーできます。
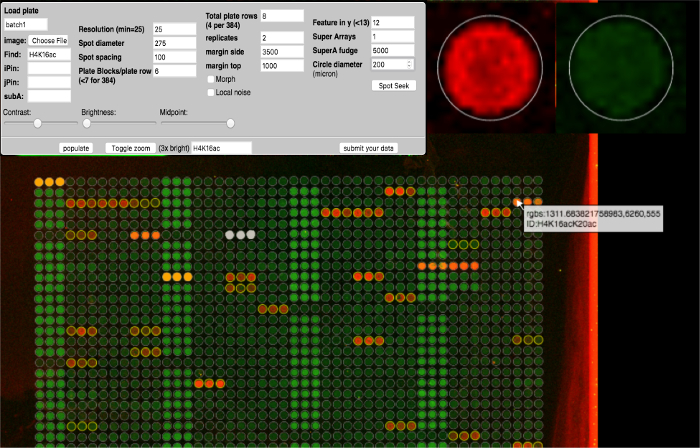
図4:ArrayNinja解析モジュール ArrayNinja解析モジュールのスクリーンショットが表示されます。コントロールパネル(左上)には、アレイを視覚化したり、スポットを見つけたり、グリッドをアレイ画像。フィーチャーの上にマウスを置くと、ズームインされたビュー(右上)が表示され、そのフィーチャーに関連付けられた識別情報(下)を含むポップアップが表示されます。バックグラウンド補正用に選択された基準点はオレンジ色です。下流分析から除外される機能は白色です。 ArrayNinjaには、H4K16の例に示すように、一致する機能を黄色で強調表示するテキストベースの検索機能が含まれています。 この図の拡大版を見るには、ここをクリックしてください。
結果
このプロトコルは、ヒストンPTM抗体特異性の分析のためのペプチドマイクロアレイプラットフォームを設計し、製造するために使用されてきた。このアレイは、コアおよびバリアントヒストンタンパク質38に見られるPTMの既知の組み合わせの多くを表す、300を超える独自のペプチド機能(長さ20〜40残基)のライブラリを照会します38
ディスカッション
生物医学研究用途における抗体の信頼性が最優先事項46、47です。これは、ヒストンPTMの存在量および分布を特徴付けるために開発された技術の大部分のための重要なツールとしての抗体の位置を考慮すると、クロマチン生化学において特に当てはまります。ここに示されたプロトコルは、ヒストンPTM抗体特異性を分析するためのペプチドマイクロ?...
開示事項
著者は何も開示することはない。
謝辞
この研究は、部分的には、Van Andel Research Instituteによって支援され、国立衛生研究所(CA181343)からSBR
資料
| Name | Company | Catalog Number | Comments |
| Printing Buffer | ArrayIt | PPB | |
| BSA | Omnipure | 2390 | |
| Streptavidin-coated glass microscope slides | Greiner Bio-one | 439003-25 | |
| polypropylene 384 well plate | Greiner Bio-one | 784201 | |
| Biotin-fluorescein | Sigma | 53608 | |
| contact microarray printer | Aushon | 2470 | Aushon 2470 Microarray Printer |
| contact microarray printer | Gene Machines | OmniGrid 100 | OmniGrid Microarray Printer |
| PBS | Invitrogen | 14190 | |
| Blocking Buffer | ArrayIt | SBB | |
| Hydrophobic wax pen | Vector Labs | H-4000 | ImmEdge Hydrophobic Barrier PAP Pen |
| Silicon Gasket | Grace Bio-labs | 622511 | |
| Hybridization Vessel | Thermo Scientific | 267061 | or similar vessel |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21244 | Alexa Fluor 647 (anti-rabbit) |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21235 | Alexa Fluor 647 (anti-mouse) |
| Wax Imprinter | ArrayIt | MSI48 | |
| Tween-20 | Omnipure | 9490 | |
| Microarray Scanner | Innopsys | InnoScan 1100AL | or equivalent microarray scanner |
| EipTitan Histone Peptide Microarray | Epicypher | 112001 | |
| AbSurance Pro Histone Peptide Microarray | Millipore | 16668 | |
| MODified Histone Peptide Array | Active Motif | 13001 | |
| Histone Code Peptide Microarrays | JPT | His_MA_01 | |
| Wax | Royal Oak | GulfWax | for wax imprinter |
| Humidified Microarray Slide Hybridization Chamber | VWR | 97000-284 | |
| High throughput microscope slide washing chamber | ArrayIt | HTW | |
| Microscope slide centrifuge | VWR | 93000-204 | |
| Antibody 1 | Abcam | 8898 | |
| Antibody 2 | Millipore | 07-473 | |
| Biotinylated histone peptide | EpiCypher | 12-0001 | Example peptide. Similar peptides with various modifications are available from several commercial sources. |
| ImageMagick | https://www.imagemagick.org/script/index.php | ||
| ArrayNinja | https://rothbartlab.vai.org/tools/ |
参考文献
- van Steensel, B. Chromatin: constructing the big picture. EMBO J. 30 (10), 1885-1895 (2011).
- Kouzarides, T. Chromatin modifications and their function. Cell. 128 (4), 693-705 (2007).
- Rothbart, S. B., Strahl, B. D. Interpreting the language of histone and DNA modifications. Biochim Biophys Acta. 1839 (8), 627-643 (2014).
- Shogren-Knaak, M., Ishii, H., Sun, J. -. M., Pazin, M. J., Davie, J. R., Peterson, C. L. Histone H4-K16 acetylation controls chromatin structure and protein interactions. Science. 311 (5762), 844-847 (2006).
- Musselman, C. A., Lalonde, M. -. E., Côté, J., Kutateladze, T. G. Perceiving the epigenetic landscape through histone readers. Nat Struct Mol Biol. 19 (12), 1218-1227 (2012).
- Huang, H., Sabari, B. R., Garcia, B. A., Allis, C. D., Zhao, Y. SnapShot: Histone Modifications. Cell. 159 (2), 458 (2014).
- Strahl, B. D., Allis, C. D. The language of covalent histone modifications. Nature. 403 (6765), 41-45 (2000).
- Rothbart, S. B., Krajewski, K., et al. Association of UHRF1 with methylated H3K9 directs the maintenance of DNA methylation. Nat Struct Mol Biol. 19 (11), 1155-1160 (2012).
- Wang, Z., Zang, C., et al. Combinatorial patterns of histone acetylations and methylations in the human genome. Nat Genet. 40 (7), 897-903 (2008).
- Stunnenberg, H. G., Hirst, M. The International Human Epigenome Consortium: A Blueprint for Scientific Collaboration and Discovery. Cell. 167 (5), 1145-1149 (2016).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Egelhofer, T. A., Minoda, A., et al. An assessment of histone-modification antibody quality. Nat Struct Mol Biol. 18 (1), 91-93 (2011).
- Bock, I., Dhayalan, A., Kudithipudi, S., Brandt, O., Rathert, P., Jeltsch, A. Detailed specificity analysis of antibodies binding to modified histone tails with peptide arrays. Epigenetics. 6 (2), 256-263 (2011).
- Busby, M., Xue, C., et al. Systematic comparison of monoclonal versus polyclonal antibodies for mapping histone modifications by ChIP-seq. Epigenetics Chromatin. 9, 49 (2016).
- Fuchs, S. M., Krajewski, K., Baker, R. W., Miller, V. L., Strahl, B. D. Influence of combinatorial histone modifications on antibody and effector protein recognition. Curr Biol. 21 (1), 53-58 (2011).
- Kungulovski, G., Jeltsch, A. Quality of histone modification antibodies undermines chromatin biology research. F1000Research. 4, 1160 (2015).
- Rothbart, S. B., Dickson, B. M., et al. An Interactive Database for the Assessment of Histone Antibody Specificity. Mol Cell. 59 (3), 502-511 (2015).
- Rothbart, S. B., Lin, S., et al. Poly-acetylated chromatin signatures are preferred epitopes for site-specific histone H4 acetyl antibodies. Sci Rep. 2, 489 (2012).
- Berger, M. F., Bulyk, M. L. Universal protein-binding microarrays for the comprehensive characterization of the DNA-binding specificities of transcription factors. Nat Protoc. 4 (3), 393-411 (2009).
- Hu, S., Wan, J., et al. DNA methylation presents distinct binding sites for human transcription factors. eLife. 2, e00726 (2013).
- Moore, C. D., Ajala, O. Z., Zhu, H. Applications in high-content functional protein microarrays. Curr Opin Chem Biol. 30, 21-27 (2016).
- MacBeath, G., Schreiber, S. L. Printing proteins as microarrays for high-throughput function determination. Science. 289 (5485), 1760-1763 (2000).
- Rothbart, S. B., Krajewski, K., Strahl, B. D., Fuchs, S. M. Peptide microarrays to interrogate the "histone code" . Methods Enzymol. 512, 107-135 (2012).
- Cornett, E. M., Dickson, B. M., et al. Substrate Specificity Profiling of Histone-Modifying Enzymes by Peptide Microarray. Methods Enzymol. 574, 31-52 (2016).
- Nady, N., Min, J., Kareta, M. S., Chédin, F., Arrowsmith, C. H. A SPOT on the chromatin landscape? Histone peptide arrays as a tool for epigenetic research. Trends Biochem Sci. 33 (7), 305-313 (2008).
- Dieker, J., Berden, J. H., et al. Autoantibodies against Modified Histone Peptides in SLE Patients Are Associated with Disease Activity and Lupus Nephritis. PLoS ONE. 11 (10), (2016).
- Price, J. V., Tangsombatvisit, S., et al. "On silico" peptide microarrays for high-resolution mapping of antibody epitopes and diverse protein-protein interactions. Nat Med. 18 (9), 1434-1440 (2012).
- Dickson, B. M., Cornett, E. M., Ramjan, Z., Rothbart, S. B. ArrayNinja: An Open Source Platform for Unified Planning and Analysis of Microarray Experiments. Methods Enzymol. 574, 53-77 (2016).
- Gatchalian, J., Fütterer, A., et al. Dido3 PHD modulates cell differentiation and division. Cell Rep. 4 (1), 148-158 (2013).
- Cai, L., Rothbart, S. B., et al. An H3K36 methylation-engaging Tudor motif of polycomb-like proteins mediates PRC2 complex targeting. Mol Cell. 49 (3), 571-582 (2013).
- Rothbart, S. B., Dickson, B. M., et al. Multivalent histone engagement by the linked tandem Tudor and PHD domains of UHRF1 is required for the epigenetic inheritance of DNA methylation. Genes Dev. 27 (11), 1288-1298 (2013).
- Ali, M., Rincón-Arano, H., et al. Molecular basis for chromatin binding and regulation of MLL5. Proc Natl Acad Sci U S A. 110 (28), 11296-11301 (2013).
- Kinkelin, K., Wozniak, G. G., Rothbart, S. B., Lidschreiber, M., Strahl, B. D., Cramer, P. Structures of RNA polymerase II complexes with Bye1, a chromatin-binding PHF3/DIDO homologue. Proc Natl Acad Sci U S A. 110 (38), 15277-15282 (2013).
- Klein, B. J., Piao, L., et al. The histone-H3K4-specific demethylase KDM5B binds to its substrate and product through distinct PHD fingers. Cell Rep. 6 (2), 325-335 (2014).
- Kim, H. -. S., Mukhopadhyay, R., et al. Identification of a BET family bromodomain/casein kinase II/TAF-containing complex as a regulator of mitotic condensin function. Cell Rep. 6 (5), 892-905 (2014).
- Greer, E. L., Beese-Sims, S. E., et al. A histone methylation network regulates transgenerational epigenetic memory in C. elegans. Cell Rep. 7 (1), 113-126 (2014).
- Andrews, F. H., Tong, Q., et al. Multivalent Chromatin Engagement and Inter-domain Crosstalk Regulate MORC3 ATPase. Cell Rep. 16 (12), 3195-3207 (2016).
- Sidoli, S., Lin, S., Karch, K. R., Garcia, B. A. Bottom-Up and Middle-Down Proteomics Have Comparable Accuracies in Defining Histone Post-Translational Modification Relative Abundance and Stoichiometry. Anal Chem. 87 (6), 3129-3133 (2015).
- Tsukada, Y., Ishitani, T., Nakayama, K. I. KDM7 is a dual demethylase for histone H3 Lys 9 and Lys 27 and functions in brain development. Genes Dev. 24 (5), 432-437 (2010).
- Tachibana, M., Sugimoto, K., Fukushima, T., Shinkai, Y. Set domain-containing protein, G9a, is a novel lysine-preferring mammalian histone methyltransferase with hyperactivity and specific selectivity to lysines 9 and 27 of histone H3. J Biol Chem. 276 (27), 25309-25317 (2001).
- Wu, H., Chen, X., et al. Histone methyltransferase G9a contributes to H3K27 methylation in vivo. Cell Res. 21 (2), 365-367 (2011).
- Koch, C. M., Andrews, R. M., et al. The landscape of histone modifications across 1% of the human genome in five human cell lines. Genome Res. 17 (6), 691-707 (2007).
- Okitsu, C. Y., Hsieh, J. C. F., Hsieh, C. -. L. Transcriptional Activity Affects the H3K4me3 Level and Distribution in the Coding Region. Mol Cell Biol. 30 (12), 2933-2946 (2010).
- Zentner, G. E., Tesar, P. J., Scacheri, P. C. Epigenetic signatures distinguish multiple classes of enhancers with distinct cellular functions. Genome Res. 21 (8), 1273-1283 (2011).
- Garske, A. L., Oliver, S. S., et al. Combinatorial profiling of chromatin binding modules reveals multisite discrimination. Nat Chem Biol. 6 (4), 283-290 (2010).
- Baker, M. Reproducibility crisis: Blame it on the antibodies. Nature. 521 (7552), 274-276 (2015).
- Bradbury, A., Plückthun, A. Reproducibility: Standardize antibodies used in research. Nature. 518 (7537), 27-29 (2015).
- Nguyen, U. T. T., Bittova, L., et al. Accelerated chromatin biochemistry using DNA-barcoded nucleosome libraries. Nat Methods. 11 (8), 834-840 (2014).
- Frank, R. Spot-synthesis: an easy technique for the positionally addressable, parallel chemical synthesis on a membrane support. Tetrahedron. 48 (42), 9217-9232 (1992).
- Hilpert, K., Winkler, D. F. H., Hancock, R. E. W. Peptide arrays on cellulose support: SPOT synthesis, a time and cost efficient method for synthesis of large numbers of peptides in a parallel and addressable fashion. Nat Protoc. 2 (6), 1333-1349 (2007).
- Kudithipudi, S., Kusevic, D., Weirich, S., Jeltsch, A. Specificity analysis of protein lysine methyltransferases using SPOT peptide arrays. J Vis Exp. (93), e52203 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved