É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Análise da especificidade de anticorpo com histona com microarrays de péptidos
Neste Artigo
Resumo
Este manuscrito descreve métodos para a aplicação de tecnologia de microarrays de péptidos para o perfil específico de anticorpos que reconhecem histonas e suas modificações pós-tradução.
Resumo
As modificações pós-translacionais (PTMs) nas proteínas das histonas são amplamente estudadas quanto aos seus papéis na regulação da estrutura da cromatina e da expressão gênica. A produção em massa e a distribuição de anticorpos específicos para as PTM das histonas facilitaram grandemente a pesquisa nessas marcas. Como os anticorpos PTM das histonas são reagentes-chave para muitas aplicações de bioquímica de cromatina, a análise rigorosa da especificidade do anticorpo é necessária para a interpretação precisa dos dados e o progresso contínuo no campo. Este protocolo descreve uma tubagem integrada para o projeto, fabricação e uso de microarrays de péptidos para perfilar a especificidade de anticorpos de histonas. Os aspectos de design e análise deste procedimento são facilitados pelo ArrayNinja, um pacote de software de código aberto e interativo desenvolvido recentemente para simplificar a personalização de formatos de impressão em microarray. Este pipeline foi usado para exibir um grande número de antibióticos de histona PTM, amplamente utilizados e comercializados.S, e os dados gerados a partir dessas experiências estão disponíveis gratuitamente através de um banco de dados de especificações de anticorpos de histona online e em expansão. Além das histonas, a metodologia geral aqui descrita pode ser aplicada amplamente à análise de anticorpos específicos de PTM.
Introdução
O DNA genômico é embalado elegantemente dentro do núcleo da célula eucariótica com proteínas histonas para formar cromatina. A subunidade de repetição da cromatina é o nucleossoma, que consiste em 147 pares de bases de DNA envolvidos em torno de um núcleo octamerico de proteínas histonas - H2A, H2B, H3 e H4 1 . A cromatina é amplamente organizada em eucromatina vagamente compactada e domínios de heterocromatina bem embalados. O grau de compactação da cromatina regula a medida em que as máquinas de proteínas podem acessar o DNA subjacente para realizar processos fundamentais de DNA, tais como replicação, transcrição e reparo.
Os principais reguladores da acessibilidade do genoma no contexto da cromatina são PTMs na cauda não estruturada e nos domínios principais das proteínas das histonas 2 , 3 . Os PTM Histone funcionam diretamente influenciando a estrutura da cromatina 4 e indiretamente através daH o recrutamento de proteínas leitoras e seus complexos macromoleculares associados que possuem atividades de remodelação da cromatina, enzima e andaimes 5 . Estudos sobre a função das histonas PTM nas últimas duas décadas sugerem esmagadoramente que essas marcas desempenham papéis fundamentais na regulação do destino das células, do desenvolvimento organizacional e da iniciação / progressão da doença. Alimentado pelos avanços na tecnologia proteômica baseada em espectrometria de massa, foram descobertas mais de 20 PTM de histonas únicas em mais de 80 resíduos de histonas distintas 6 . Notavelmente, essas modificações geralmente ocorrem em combinações e consistentes com a hipótese do "código histônico", numerosos estudos sugerem que as proteínas leitoras são direcionadas para regiões discretas de cromatina através do reconhecimento de combinações específicas de histonas PTM 7 , 8 , 9 . Um desafio chave para avançar será atribuir funções ao grDevido a lista de PTM de histonas e para determinar como combinações específicas de PTM de histonas orquestra as funções dinâmicas associadas à cromatina.
Os anticorpos são os reagentes lynchpin para a detecção de PTM de histonas. Como tal, mais de 1.000 anticorpos específicos de PTM de histonas foram comercialmente desenvolvidos para uso em pesquisa de bioquímica de cromatina. Com o rápido desenvolvimento da tecnologia de sequenciação de DNA de alto rendimento, esses reagentes estão sendo amplamente utilizados por pesquisadores individuais e iniciativas de "roteiro" de epigenômica em larga escala ( por exemplo , ENCODE e BLUEPRINT) em ChIP-seq (imunoprecipitação de cromatina, juntamente com sequenciação da próxima geração ) Pipelines para gerar mapas espaciais de alta resolução da distribuição de histonas PTM em todo o genoma 10 , 11 . No entanto, estudos recentes mostraram que a especificidade dos anticorpos PTM das histonas pode ser altamente variável e que esses reagentes exibem falta Propriedades avoráveis, como o reconhecimento do epitopo fora do alvo, forte influência positiva e negativa dos PTM vizinhos e dificuldade em discriminar a ordem de modificação sobre um resíduo particular ( por exemplo , mono-, di- ou trimetilisina) 12 , 13 , 14 , 15 , 16, 17, 18. Portanto, o controle de qualidade rigoroso dos reagentes de anticorpos específicos de PTM de histonas é necessário para interpretar com precisão os dados gerados com esses reagentes valiosos.
A tecnologia Microarray permite a interrogação simultânea de milhares de interações macromoleculares em um formato de alto rendimento, reprodutível e miniaturizado. Por esta razão, uma variedade de plataformas de microarray foram criadas para analisar proteína-DNA 19 ,", 20, proteína-proteína 21 e interações proteína-péptido 22. Na verdade, as microarrays de péptidos de histonas emergiram como uma plataforma de descoberta informativa para pesquisa de bioquímica de cromatina, permitindo o perfil de alto rendimento dos escritores, borrachas e leitores de histonas PTMs 15 , 23 , 24 e também para a análise da especificidade do anticorpo das histonas 17 , 25. Além da sua aplicação na pesquisa de cromatina e epigenética, os arrays de péptidos de histona possuem utilidade potencial como teste diagnóstico / prognóstico para lúpus eritematoso sistêmico e outras doenças autoimunes em que os anticorpos anti- Os autoanticorpos da cromatina são gerados 26 , 27 .
Aqui, descrevemos um pipeline integrado que desenvolvemos para projetar, fabricar e queGerando microarrays de péptidos de histona para gerar perfis de especificidade para anticorpos que reconhecem histonas e seus PTMs. O pipeline é facilitado pela ArrayNinja, uma aplicação de software de código aberto e interativa que desenvolvemos recentemente, que integra os estágios de design e análise das experiências de microarrays 28 . ArrayNinja funciona melhor no Google Chrome. Resumidamente, uma impressora de microarray de contato robotizado é usada para depositar uma biblioteca de péptidos de histona conjugados com biotina em posições definidas em lâminas de microscópio de vidro revestidas com estreptavidina. As matrizes podem então ser usadas em um formato de ensaio competitivo e paralelo para interrogar interações anticorpo-epítopo ( Figura 1 ). A biblioteca de péptidos consiste em centenas de péptidos sintéticos únicos que abrigam PTMs (acetilação de lisina, metilação de lisina / arginina e fosforilação de serina / treonina) e em combinações relevantes em grande parte derivadas de conjuntos de dados proteômicos. Métodos para síntese e validação de péptidos São detalhados em outro lugar 23 . Os dados gerados a partir dos nossos atuais esforços de rastreamento de anticorpos de PTM de histonas utilizando esta plataforma de matriz são arquivados em um recurso público da Web, a Histone Antibody Specificity Database (www.histoneantibodies.com). Notavelmente, as microarrays de péptidos de histonas fabricadas com variações deste protocolo também foram utilizadas amplamente para caracterizar a atividade dos domínios de leitor PTM de histona 8 , 29 , 30 , 31 , 32 , 33 , 34 , 35 , 36 , 37 e, mais recentemente, para histona de perfil Atividades de gravador e borracha PTM 24 .
/files/ftp_upload/55912/55912fig1.jpg "/>
Figura 1: Desenho dos desenhos animados do procedimento Stepwise para triagem de anticorpos em um microarray de péptido de histona. Os péptidos de histona biotinilados que abrigam modificações pós-translacionais definidas (círculos vermelho e azul) são co-impressos com biotina-fluoresceína em vidro revestido com estreptavidina. As interações positivas são visualizadas como fluorescência vermelha. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. Instalando e executando ArrayNinja
- Baixe e instale o Oracle Virtual Box de www.virtualbox.org.
- Baixe e descomprima a máquina virtual ArrayNinja (VM) de http://research.vai.org/Tools/arrayninja.
- Abra a caixa virtual e adicione a VM ArrayNinja clicando em 'Máquina', 'Adicionar' e selecione arrayninja.vbox na pasta onde ArrayNinja VM foi salvo.
- Inicie o ArrayNinja selecionando-o dentro da Caixa Virtual e clicando na seta verde 'Iniciar'.
- A Caixa Virtual abrirá uma nova janela e exibirá uma mensagem de que a VM pode ser acessada navegando no navegador da Web para localhost: 2080.60; NOTA: uma versão em contentor do ArrayNinja também está disponível via hub.docker.com/r/bradley.dickson/arrayninja/
2. Projetando o Array Slide e Layout da Fonte de Placa
- Sob o cabeçalho 'Planejar um layout de slide' na interface ArrayNinja, clique no link que corresponde à impressora de microarray que está sendo usada.
NOTA: ArrayNinja foi programado para imitar o movimento robótico de duas impressoras de microarrays de uso comum (ver Tabela1). A compatibilidade com os outros distribuidores pode ser configurada mediante solicitação. - Clique dentro da caixa de diálogo 'Carregar a placa', digite "vazio" e clique em "Inserir". Veja a Figura 2 para uma captura de tela do módulo de design ArrayNinja.
- Ajuste o diâmetro do ponto para 275 μm e espaçador de pontos para 375 μm. Ajuste as configurações restantes (Blocos de placa / linha de placa, Fileiras de placa total, repetições, recursos em y, Super Arrays, SuperA fudge, veja a Figura 2 ) para personalizar como os recursos aparecerão no slide de microarray.
NOTA: O diâmetro do ponto é determinado pelo tamanho do pino de microarray. À medida que essas configurações são ajustadas, o slide de desenho animado será atualizado em tempo real. Use este desenho para visualizar como cada configuração está modificando o layout final do slide. - Depois que o layout dos recursos no slide for finalizado, passe o mouse sobre cada recurso exclusivo e insira o identificador do recurso na caixa de diálogo pop-up.
NOTA: Os identificadores de recursos podem consistir em números, letras ou combinações. Isso só é necessário para recursos exclusivos e uma caixa de diálogo não aparecerá quando as réplicas forem selecionadas. - Depois que todos os recursos exclusivos foram atribuídos a um identificador, clique em "preencher". Digite um nome para o layout do slide e clique em 'imprimir o seu prato' para salvar. Uma nova página abrirá exibindo o número de placas de origem de 384 poços necessárias para fabricar o design de slide escolhido e uma tabela que mapeie a localização física de cada recurso para ser carregada na (s) placa (s) de origem.
NOTA: Lembre-se deste nome, pois ele será usado para recuperar esse design ao analisar dados de microarrays (ver seção 6.2). Ao clicar em 'imprimir o seu prato', ele salva o layout dentro do ArrayNinja.

Figura 2: Módulo de projeto ArrayNinja. Uma captura de tela do O módulo de design ArrayNinja é mostrado na linha pontilhada. O painel de controle (parte superior) mostra todos os parâmetros que podem ser alterados na impressora microarray. À medida que esses parâmetros são ajustados, a imagem de desenho animado do layout de slide (inferior esquerda) atualiza em tempo real. Depois que o layout é definido, o usuário pode passar o mouse sobre pontos individuais para inserir identificadores de recursos exclusivos. As construções ArrayNinja deste usuário introduzem um mapa da posição de cada característica na (s) placa (s) de origem (inferior direita) necessária para fabricar um layout de slide de microarrays especificado. Clique aqui para ver uma versão maior desta figura.
3. Fabricação de microarrays
- Preparando a Placa da Fonte
- Use o mapa gerado com ArrayNinja na seção 2.5 para criar a (s) fonte (s) de fonte de 384 poços.
NOTA: descrições detalhadas de péptidos consultados nesta plataforma podem ser encontradas em outro lugarS = "xref"> 17. - Deposite 1 - 2 μL de cada característica ( por exemplo , péptido de histona biotinilada) no poço correto da (s) placa (s) de origem de 384 poços.
NOTA: Os péptidos de histona biotinilados são tipicamente depositados a partir de soluções de estoque de 200 a 400 μM, o que equivale a um excesso molar de 10 a 25 vezes de péptido para locais de ligação de estreptavidina em um único ponto de matriz. Isso é calculado usando a equação: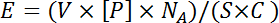
Onde V é o volume entregue por um pino, [ P ] é a concentração da característica que está sendo impressa, N A é o número da Avogadro e S é a superfície de um ponto. C é a cobertura do slide, expressa como o número de moléculas de estreptavidina por unidade de área multiplicada por três (o número médio de locais de ligação de estreptavidina disponíveis). V e C são obtidos pelos respectivos fabricantes. Outras características podemRequerem diferentes concentrações dependendo do tamanho da biomolécula onde o aglomerado pode ser uma preocupação. Uma gama de concentrações de impressão para cada novo tipo de recurso deve ser testada empiricamente para determinar a concentração de impressão ideal. - Diluir cada característica 10 vezes com 1x tampão de impressão suplementado com 1% de albumina de soro bovino (BSA). Gire a (s) placa (s) de origem a 500 xg durante 2 min à temperatura ambiente.
NOTA: A inclusão de biotina marcada com fluoresceína (5 μg / μL) no buffer de impressão é recomendada como um controle de manchas e como um auxílio visual para facilitar o alinhamento adequado da matriz durante a análise (ver Figura 4 ).
- Use o mapa gerado com ArrayNinja na seção 2.5 para criar a (s) fonte (s) de fonte de 384 poços.
- Protocolo de impressão ( Figura 3A -B ).
- Prepare o arrayer esvaziando o recipiente de coleta de resíduos e enchendo o recipiente da solução de lavagem e o recipiente do umidificador com água destilada estéril.
- Digite os parâmetros vocêSed para projetar o slide no ArrayNinja (seção 2 e Figura 2 ) no programa de controle da impressora de microarray.
- Usando o programa de controle da impressora microarray, defina o procedimento de lavagem para uma lavagem de 1 s com uma imersão. Defina as configurações de pós-lavagem para re-mergulhar os pinos 5 vezes após cada lavagem. Ajuste a umidade para 60%.
NOTA: A configuração de lavagem ideal pode variar de acordo com a impressora de microarray sendo usada. A configuração de inclinação do pino pós-lavagem ótima pode variar de acordo com a impressora de microarray que está sendo usada. - Insira slides funcionalizados ( por exemplo , vidro revestido com estreptavidina) nos pratos do substrato e coloque todos os pratos no elevador da platina. Insira a (s) placa (s) de origem no (s) suporte (s) da placa e coloque no elevador da placa fonte.
- Clique em 'imprimir'. Monitore o processo de impressão para algumas rodadas de deposição de recursos para garantir que todas as configurações de lavagem e mergulho estejam corretas. Quando a execução estiver completa, removaOs pratos de substratos do arrayer.
NOTA: Quando grandes lançamentos de impressão proíbem a conclusão das etapas de bloqueio dentro de um dia, as lâminas impressas podem ser incubadas em uma câmara humidificada a 4 ° C durante a noite. Incube os slides ao lado de um pequeno copo de água dentro de uma caixa de papelão selada com plástico. - Bloqueie os slides com tampão de bloqueio durante 30 minutos à temperatura ambiente com a mistura.
- Lave os slides 2 x 10 min à temperatura ambiente em solução salina tamponada com fosfato (PBS), pH 7,6 com mistura. Secar as lâminas girando em uma centrífuga deslizante de microarrays durante 30 s à temperatura ambiente.
NOTA: Para processar um grande número de lâminas de uma vez, pode usar-se uma câmara de lavagem de microscópio de alto rendimento, permitindo que 50 lâminas sejam lavadas em paralelo. - Para os slides projetados para serem divididos com cera, vá para a seção 4.1. Para todos os outros desenhos, armazene slides a 4 ° C protegidos da luz e da umidade.
NOTA: Peptídeos de histona biotinilados impressosSão estáveis por pelo menos 6 meses quando armazenados desta forma.
4. Particionamento de slides de microarray
- Caneta de cera hidrófoba ( Figura 3C )
- Aplique cera em torno das áreas que contêm recursos usando uma caneta de cera. Deixe a cera secar ao ar por 5 minutos antes de seguir a seção 5.
NOTA: Após o bloqueio, os pontos da matriz podem ser muito difíceis de visualizar por olho. O design do slide do ArrayNinja pode ser impresso em escala e usado como guia para aplicação de cera.
- Aplique cera em torno das áreas que contêm recursos usando uma caneta de cera. Deixe a cera secar ao ar por 5 minutos antes de seguir a seção 5.
- Junta de silicone ( Figura 3D )
- Retire o filme transparente da parte traseira da junta da armação e coloque o lado adesivo para baixo no slide do microarray.
- Segure a junta no lugar por 5 s antes de prosseguir para a seção 5.
- Impressão de cera ( Figura 3E )
- Para slides projetados para serem divididos por cera, imprima um slide de teste em vidro simples usando 10% de BSA. Use este slide de teste para otimizar as guias de impressão de cera ou configurações de matriz para garantir que todos os recursos estejam dentro das câmaras de molde de cera.
- Aqueça a impressora de cera de microarray até 85 ° C até que toda a cera esteja completamente derretida, aproximadamente 30 min.
- Insira o slide com o lado impresso voltado para baixo e empurre o slide até o guia direito na impressora microarray. Puxe a alavanca para colocar o molde em contato com a superfície do slide. Segure por 2 s.
NOTA: O tempo de espera pode ser alterado para obter uma espessura de borda de cera ideal. - Remova rapidamente o slide e inspecione visualmente as bordas de cera para garantir que todos os poços estejam fechados e que as bordas não sejam tão espessas que invadirem os pontos. Armazene as lâminas a 4 ° C protegidas da umidade e luz.
NOTA: O tempo de espera pode ser aumentado ou diminuído para obter bordas mais grossas ou mais finas.

Figura 3: Fabricação de microarray. (A) Fabricação de microarrays de péptidos de histona em lâminas de microscópio revestidas com estreptavidina usando uma impressora de microarray de contato. (B) Microarrays fabricados com 3 subarranjos de uma grade 48 x 48 de características peptídicas. Separação de subarrays (C) 3 com uma caneta de cera hidrofóbica, (D) 2 subarrays com um adesivo de silício e (E) 48 subarrays com impressão de cera. Todos os microarrays mostrados são fabricados usando lâminas de microscópio de 25 x 75 mm. Clique aqui para ver uma versão maior desta figura.
5. Hibridando um anticorpo de Histone PTM com um microarray de péptido
- Preparar tampão de hibridação (PBS, pH 7,6, 5% de BSA, 0,1% de Tween-20).
- Equilibre o slide no buffer de hibridaçãoUsando um navio de hibridação. Cobre completamente todo o slide no tampão de hibridação e incube durante 30 min a 4 ° C em um agitador orbital a baixa velocidade.
- Preparar uma solução contendo anticorpo de PTM de histona diluída em tampão de hibridação.
NOTA: Nos dados de exemplo, tanto o anticorpo de histona # 1 quanto o Anticorpo # 2 foram diluídos 1: 1000 em tampão de hibridação. Uma faixa de diluição semelhante à utilizada para imunotransferência é recomendada como ponto de partida. - Incubar a matriz com a solução de anticorpo durante 1 h a 4 ° C. Remova a solução de anticorpo e lave a matriz 3 vezes durante 5 min a 4 ° C com PBS frio, pH 7,6.
- Prepare uma diluição 1: 5.000 - 1: 10 000 de anticorpo secundário conjugado com corante fluorescente em tampão de hibridação.
- Incubar a matriz com solução de anticorpo secundário durante 30 min a 4 ° C protegido da luz. Remova a solução de anticorpo secundário e lave o filtro de microarray 3 vezes durante 5 min a 4 ° C com PBS, pH 7,6. MergulhoO microarray em um tubo cônico de 50 mL contendo 0,1x PBS, pH 7,6 para remover o excesso de sal à temperatura ambiente. Secar a corrediça em uma centrífuga de microarray a temperatura ambiente.
- Digitalize o slide com um scanner de microarray com uma resolução de 25 μm ou superior, seguindo o protocolo de digitalização recomendado pelo fabricante do scanner de microarrays.
NOTA: Se o marcador de biotina marcado com fluoresceína estiver presente, verifique o canal verde (ex: 488 nm, em: 509 nm) eo canal que corresponde ao anticorpo secundário conjugado com corante fluorescente, tipicamente vermelho (ex: 635 nm, em : 677 nm). O objetivo da digitalização é obter arquivos .tif de canal único que podem ser mesclados em um único arquivo .png (conforme descrito na seção 6.1).
6. Análise de Dados Microarray usando ArrayNinja
- Preparando uma imagem de microarrays mesclada
NOTA: O objetivo desta seção é criar um arquivo de imagem .png que funde os dois arquivos .tif de canal único (obtidos na seção 5). esteÉ o único formato de imagem compatível com o módulo de análise ArrayNinja. As instruções a seguir representam uma maneira possível de obter um arquivo de imagem mesclado. No entanto, outras soluções estão disponíveis ( por exemplo , o freeware ImageJ).- A partir da linha de comando em um terminal bash de um computador com o freeware ImageMagick instalado, navegue até a pasta que contém os arquivos .tif de canal único e copie / cole as seguintes etapas, pressionando 'enter' entre cada etapa (6.1.4 - 6.1 .7).
NOTA: Os nomes dos arquivos em letras maiúsculas ( por exemplo , RED_CHANNEL) devem ser substituídos pelo nome do arquivo das imagens do slide do microarray. - Se necessário, primeiro inverta as imagens usando o comando 'convert INPUT.tif -negate OUTPUT.tif'. Isso é necessário se o scanner salvar arquivos .tif com o sinal em branco e fundo em preto.
- Convert -depth 16 RED_CHANNEL.TIF -clone 0 -channel GB -evalate set 0 -delete 0 out.png 2> error.file.
- ConvErt - depth 16 CONTROL_CHANNEL.TIF -clone 0 -channel RB -evaluate set 0 -delete 0 outa.png 2> error.file.
- Convert -depth 16 CONTROL_CHANNEL.TIF -clone 0 -channel RG -valor de avaliação 0 -delete 0 outB.png 2> error.file.
- Convert out.png outa.png outB.png -set colorspace RGV -combine fusioned.png.
NOTA: Um arquivo chamado 'fusioned.png' será salvo na mesma pasta que os arquivos .tif originais.
- A partir da linha de comando em um terminal bash de um computador com o freeware ImageMagick instalado, navegue até a pasta que contém os arquivos .tif de canal único e copie / cole as seguintes etapas, pressionando 'enter' entre cada etapa (6.1.4 - 6.1 .7).
- Quantificando dados usando o ArrayNinja
- Abra ArrayNinja e clique no link de impressora de microarray apropriado em "Para quantificar imagens que possuem uma fonte conhecida". Digite o nome do design do slide salvo (passo 2.5) na caixa de diálogo "carregar a placa" e clique em 'digitar'. Veja a Figura 4 para uma captura de tela do módulo de análise ArrayNinja.
- Clique em 'Escolher arquivo' e navegue até e selecione o arquivo 'fusionado.png' criado na seção 6.1. A imagem combinadaE irá carregar, bem como uma grade para o layout de slide.
- Use os controles deslizantes na parte inferior do painel de controle ArrayNinja para ajustar o contraste, o brilho e o ponto intermediário.
NOTA: Estes ajustes são apenas para fins de visualização e não têm impacto na quantificação. - Selecione "resolução" e digite o valor que corresponde à resolução da imagem digitalizada. Use o "lado da margem" e a "margem superior" para mover a grade para o alinhamento com os pontos da matriz. Ajuste conforme necessário para obter a grade tão alinhada sobre cada ponto quanto possível.
- Clique em 'Spot Seek' e aguarde até que o botão volte à cor cinza original.
NOTA: Isso pode ser repetido várias vezes para centrar os círculos da grade em pontos individuais. A função Seek relaxa a grade em direção a cada ponto para ajustar o alinhamento. - Altere o valor "Super Arrays" para "1" para trabalhar cada painel de sub-painel individualmente (se desejado). Se estiver usando um 4X 12 layout de slide de impressão de cera ( Figura 3E ), use os controles "iPin" "jPin" e "subA" para desligar os recursos para analisar qualquer combinação desejada de 4 x 12 poços. Por exemplo, para analisar os poços superior esquerdo e superior direito como repetições um do outro, digite "1 4" na iPin, "1 1" em jPin e "1 4" em subA. Pressione Enter'.
- Passe o mouse sobre os pontos individuais para ver a identidade desse recurso.
- Clique em 'Alternar zoom' para analisar os recursos com mais cuidado. Selecione os pontos de referência de fundo pressionando a tecla 'R' enquanto o mouse estiver sobre um ponto. Os pontos de referência serão destacados em laranja.
NOTA: No modo "alternar zoom", uma imagem ampliada do recurso que o mouse acabou é exibida no canto superior direito. Uma discussão detalhada dos recursos de correção de fundo adicionais em ArrayNinja é discutida em outro lugar 28 . - Alternar pontos (com uma morfologia de manchas amplamente variada ou detritos que afetariam a quantificação precisa), pressionando a tecla 'A' ao passar o mouse sobre os pontos afetados. As manchas inativadas ficam brancas.
- Clique em 'preencher' seguido de 'Enviar' para quantificar os pontos.
NOTA: Uma nova guia será aberta e exibirá um gráfico de barras de dados normalizados para a média de pontos mais brilhantes. Uma tabela abaixo do gráfico de barras contém os valores de dados normalizados e brutos. Esta tabela pode ser copiada para uma planilha para arquivamento e análise posterior.
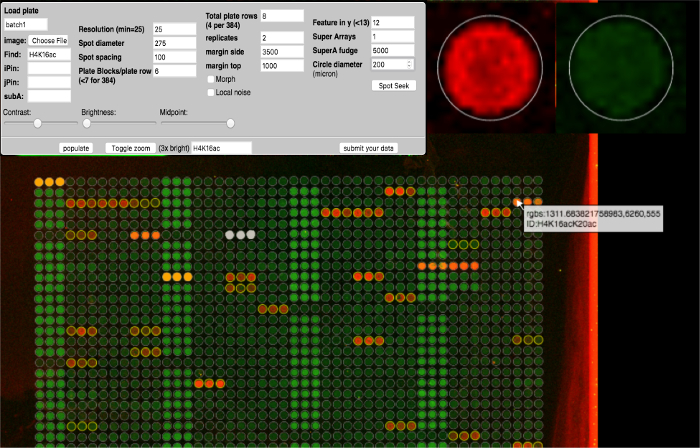
Figura 4: Módulo de análise ArrayNinja. Uma tela do módulo de análise ArrayNinja é mostrada. O painel de controle (superior esquerdo) mostra todos os parâmetros que podem ser ajustados para visualizar a matriz, encontrar pontos e alinhar uma grade ao longo daImagem da matriz. O deslocamento do mouse sobre um recurso mostra uma visualização ampliada (canto superior direito) e exibe um popup que contém as informações de identificação associadas a esse recurso (parte inferior). Os pontos de referência selecionados para a correção de fundo são laranja. Os recursos a serem excluídos da análise a jusante são brancos. ArrayNinja contém um recurso de pesquisa baseado em texto que destaca recursos correspondentes em amarelo, como mostrado no exemplo para H4K16. Clique aqui para ver uma versão maior desta figura.
Resultados
Este protocolo tem sido usado para projetar e fabricar uma plataforma de microarrays de péptidos para a análise da especificidade de anticorpos de PTM das histonas. A matriz consulta uma biblioteca de mais de 300 características peptídicas únicas (20 a 40 resíduos de comprimento) representando muitas das combinações conhecidas de PTMs encontradas nas proteínas histonas núcleo e variante 38 . Este pipeline tem sido um campo de trabalho para a triagem de m...
Discussão
A confiabilidade de anticorpos em aplicações de pesquisa biomédica é primordial 46 , 47 . Isto é especialmente verdadeiro na bioquímica da cromatina, dada a posição dos anticorpos como ferramentas-chave para a maioria das técnicas desenvolvidas para caracterizar a abundância e distribuição de PTM de histonas. O protocolo apresentado aqui detalha um pipeline otimizado para o projeto, fabricação e uso de microarrays de péptidos para analisar a espec...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho foi apoiado em parte pelo Van Andel Research Institute e uma bolsa de pesquisa dos Institutos Nacionais de Saúde (CA181343) para SBR
Materiais
| Name | Company | Catalog Number | Comments |
| Printing Buffer | ArrayIt | PPB | |
| BSA | Omnipure | 2390 | |
| Streptavidin-coated glass microscope slides | Greiner Bio-one | 439003-25 | |
| polypropylene 384 well plate | Greiner Bio-one | 784201 | |
| Biotin-fluorescein | Sigma | 53608 | |
| contact microarray printer | Aushon | 2470 | Aushon 2470 Microarray Printer |
| contact microarray printer | Gene Machines | OmniGrid 100 | OmniGrid Microarray Printer |
| PBS | Invitrogen | 14190 | |
| Blocking Buffer | ArrayIt | SBB | |
| Hydrophobic wax pen | Vector Labs | H-4000 | ImmEdge Hydrophobic Barrier PAP Pen |
| Silicon Gasket | Grace Bio-labs | 622511 | |
| Hybridization Vessel | Thermo Scientific | 267061 | or similar vessel |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21244 | Alexa Fluor 647 (anti-rabbit) |
| Fluorescent-dye conjugated secondary antibody | Life Technologies | A-21235 | Alexa Fluor 647 (anti-mouse) |
| Wax Imprinter | ArrayIt | MSI48 | |
| Tween-20 | Omnipure | 9490 | |
| Microarray Scanner | Innopsys | InnoScan 1100AL | or equivalent microarray scanner |
| EipTitan Histone Peptide Microarray | Epicypher | 112001 | |
| AbSurance Pro Histone Peptide Microarray | Millipore | 16668 | |
| MODified Histone Peptide Array | Active Motif | 13001 | |
| Histone Code Peptide Microarrays | JPT | His_MA_01 | |
| Wax | Royal Oak | GulfWax | for wax imprinter |
| Humidified Microarray Slide Hybridization Chamber | VWR | 97000-284 | |
| High throughput microscope slide washing chamber | ArrayIt | HTW | |
| Microscope slide centrifuge | VWR | 93000-204 | |
| Antibody 1 | Abcam | 8898 | |
| Antibody 2 | Millipore | 07-473 | |
| Biotinylated histone peptide | EpiCypher | 12-0001 | Example peptide. Similar peptides with various modifications are available from several commercial sources. |
| ImageMagick | https://www.imagemagick.org/script/index.php | ||
| ArrayNinja | https://rothbartlab.vai.org/tools/ |
Referências
- van Steensel, B. Chromatin: constructing the big picture. EMBO J. 30 (10), 1885-1895 (2011).
- Kouzarides, T. Chromatin modifications and their function. Cell. 128 (4), 693-705 (2007).
- Rothbart, S. B., Strahl, B. D. Interpreting the language of histone and DNA modifications. Biochim Biophys Acta. 1839 (8), 627-643 (2014).
- Shogren-Knaak, M., Ishii, H., Sun, J. -. M., Pazin, M. J., Davie, J. R., Peterson, C. L. Histone H4-K16 acetylation controls chromatin structure and protein interactions. Science. 311 (5762), 844-847 (2006).
- Musselman, C. A., Lalonde, M. -. E., Côté, J., Kutateladze, T. G. Perceiving the epigenetic landscape through histone readers. Nat Struct Mol Biol. 19 (12), 1218-1227 (2012).
- Huang, H., Sabari, B. R., Garcia, B. A., Allis, C. D., Zhao, Y. SnapShot: Histone Modifications. Cell. 159 (2), 458 (2014).
- Strahl, B. D., Allis, C. D. The language of covalent histone modifications. Nature. 403 (6765), 41-45 (2000).
- Rothbart, S. B., Krajewski, K., et al. Association of UHRF1 with methylated H3K9 directs the maintenance of DNA methylation. Nat Struct Mol Biol. 19 (11), 1155-1160 (2012).
- Wang, Z., Zang, C., et al. Combinatorial patterns of histone acetylations and methylations in the human genome. Nat Genet. 40 (7), 897-903 (2008).
- Stunnenberg, H. G., Hirst, M. The International Human Epigenome Consortium: A Blueprint for Scientific Collaboration and Discovery. Cell. 167 (5), 1145-1149 (2016).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Egelhofer, T. A., Minoda, A., et al. An assessment of histone-modification antibody quality. Nat Struct Mol Biol. 18 (1), 91-93 (2011).
- Bock, I., Dhayalan, A., Kudithipudi, S., Brandt, O., Rathert, P., Jeltsch, A. Detailed specificity analysis of antibodies binding to modified histone tails with peptide arrays. Epigenetics. 6 (2), 256-263 (2011).
- Busby, M., Xue, C., et al. Systematic comparison of monoclonal versus polyclonal antibodies for mapping histone modifications by ChIP-seq. Epigenetics Chromatin. 9, 49 (2016).
- Fuchs, S. M., Krajewski, K., Baker, R. W., Miller, V. L., Strahl, B. D. Influence of combinatorial histone modifications on antibody and effector protein recognition. Curr Biol. 21 (1), 53-58 (2011).
- Kungulovski, G., Jeltsch, A. Quality of histone modification antibodies undermines chromatin biology research. F1000Research. 4, 1160 (2015).
- Rothbart, S. B., Dickson, B. M., et al. An Interactive Database for the Assessment of Histone Antibody Specificity. Mol Cell. 59 (3), 502-511 (2015).
- Rothbart, S. B., Lin, S., et al. Poly-acetylated chromatin signatures are preferred epitopes for site-specific histone H4 acetyl antibodies. Sci Rep. 2, 489 (2012).
- Berger, M. F., Bulyk, M. L. Universal protein-binding microarrays for the comprehensive characterization of the DNA-binding specificities of transcription factors. Nat Protoc. 4 (3), 393-411 (2009).
- Hu, S., Wan, J., et al. DNA methylation presents distinct binding sites for human transcription factors. eLife. 2, e00726 (2013).
- Moore, C. D., Ajala, O. Z., Zhu, H. Applications in high-content functional protein microarrays. Curr Opin Chem Biol. 30, 21-27 (2016).
- MacBeath, G., Schreiber, S. L. Printing proteins as microarrays for high-throughput function determination. Science. 289 (5485), 1760-1763 (2000).
- Rothbart, S. B., Krajewski, K., Strahl, B. D., Fuchs, S. M. Peptide microarrays to interrogate the "histone code" . Methods Enzymol. 512, 107-135 (2012).
- Cornett, E. M., Dickson, B. M., et al. Substrate Specificity Profiling of Histone-Modifying Enzymes by Peptide Microarray. Methods Enzymol. 574, 31-52 (2016).
- Nady, N., Min, J., Kareta, M. S., Chédin, F., Arrowsmith, C. H. A SPOT on the chromatin landscape? Histone peptide arrays as a tool for epigenetic research. Trends Biochem Sci. 33 (7), 305-313 (2008).
- Dieker, J., Berden, J. H., et al. Autoantibodies against Modified Histone Peptides in SLE Patients Are Associated with Disease Activity and Lupus Nephritis. PLoS ONE. 11 (10), (2016).
- Price, J. V., Tangsombatvisit, S., et al. "On silico" peptide microarrays for high-resolution mapping of antibody epitopes and diverse protein-protein interactions. Nat Med. 18 (9), 1434-1440 (2012).
- Dickson, B. M., Cornett, E. M., Ramjan, Z., Rothbart, S. B. ArrayNinja: An Open Source Platform for Unified Planning and Analysis of Microarray Experiments. Methods Enzymol. 574, 53-77 (2016).
- Gatchalian, J., Fütterer, A., et al. Dido3 PHD modulates cell differentiation and division. Cell Rep. 4 (1), 148-158 (2013).
- Cai, L., Rothbart, S. B., et al. An H3K36 methylation-engaging Tudor motif of polycomb-like proteins mediates PRC2 complex targeting. Mol Cell. 49 (3), 571-582 (2013).
- Rothbart, S. B., Dickson, B. M., et al. Multivalent histone engagement by the linked tandem Tudor and PHD domains of UHRF1 is required for the epigenetic inheritance of DNA methylation. Genes Dev. 27 (11), 1288-1298 (2013).
- Ali, M., Rincón-Arano, H., et al. Molecular basis for chromatin binding and regulation of MLL5. Proc Natl Acad Sci U S A. 110 (28), 11296-11301 (2013).
- Kinkelin, K., Wozniak, G. G., Rothbart, S. B., Lidschreiber, M., Strahl, B. D., Cramer, P. Structures of RNA polymerase II complexes with Bye1, a chromatin-binding PHF3/DIDO homologue. Proc Natl Acad Sci U S A. 110 (38), 15277-15282 (2013).
- Klein, B. J., Piao, L., et al. The histone-H3K4-specific demethylase KDM5B binds to its substrate and product through distinct PHD fingers. Cell Rep. 6 (2), 325-335 (2014).
- Kim, H. -. S., Mukhopadhyay, R., et al. Identification of a BET family bromodomain/casein kinase II/TAF-containing complex as a regulator of mitotic condensin function. Cell Rep. 6 (5), 892-905 (2014).
- Greer, E. L., Beese-Sims, S. E., et al. A histone methylation network regulates transgenerational epigenetic memory in C. elegans. Cell Rep. 7 (1), 113-126 (2014).
- Andrews, F. H., Tong, Q., et al. Multivalent Chromatin Engagement and Inter-domain Crosstalk Regulate MORC3 ATPase. Cell Rep. 16 (12), 3195-3207 (2016).
- Sidoli, S., Lin, S., Karch, K. R., Garcia, B. A. Bottom-Up and Middle-Down Proteomics Have Comparable Accuracies in Defining Histone Post-Translational Modification Relative Abundance and Stoichiometry. Anal Chem. 87 (6), 3129-3133 (2015).
- Tsukada, Y., Ishitani, T., Nakayama, K. I. KDM7 is a dual demethylase for histone H3 Lys 9 and Lys 27 and functions in brain development. Genes Dev. 24 (5), 432-437 (2010).
- Tachibana, M., Sugimoto, K., Fukushima, T., Shinkai, Y. Set domain-containing protein, G9a, is a novel lysine-preferring mammalian histone methyltransferase with hyperactivity and specific selectivity to lysines 9 and 27 of histone H3. J Biol Chem. 276 (27), 25309-25317 (2001).
- Wu, H., Chen, X., et al. Histone methyltransferase G9a contributes to H3K27 methylation in vivo. Cell Res. 21 (2), 365-367 (2011).
- Koch, C. M., Andrews, R. M., et al. The landscape of histone modifications across 1% of the human genome in five human cell lines. Genome Res. 17 (6), 691-707 (2007).
- Okitsu, C. Y., Hsieh, J. C. F., Hsieh, C. -. L. Transcriptional Activity Affects the H3K4me3 Level and Distribution in the Coding Region. Mol Cell Biol. 30 (12), 2933-2946 (2010).
- Zentner, G. E., Tesar, P. J., Scacheri, P. C. Epigenetic signatures distinguish multiple classes of enhancers with distinct cellular functions. Genome Res. 21 (8), 1273-1283 (2011).
- Garske, A. L., Oliver, S. S., et al. Combinatorial profiling of chromatin binding modules reveals multisite discrimination. Nat Chem Biol. 6 (4), 283-290 (2010).
- Baker, M. Reproducibility crisis: Blame it on the antibodies. Nature. 521 (7552), 274-276 (2015).
- Bradbury, A., Plückthun, A. Reproducibility: Standardize antibodies used in research. Nature. 518 (7537), 27-29 (2015).
- Nguyen, U. T. T., Bittova, L., et al. Accelerated chromatin biochemistry using DNA-barcoded nucleosome libraries. Nat Methods. 11 (8), 834-840 (2014).
- Frank, R. Spot-synthesis: an easy technique for the positionally addressable, parallel chemical synthesis on a membrane support. Tetrahedron. 48 (42), 9217-9232 (1992).
- Hilpert, K., Winkler, D. F. H., Hancock, R. E. W. Peptide arrays on cellulose support: SPOT synthesis, a time and cost efficient method for synthesis of large numbers of peptides in a parallel and addressable fashion. Nat Protoc. 2 (6), 1333-1349 (2007).
- Kudithipudi, S., Kusevic, D., Weirich, S., Jeltsch, A. Specificity analysis of protein lysine methyltransferases using SPOT peptide arrays. J Vis Exp. (93), e52203 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados