A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
ניתוח אלקטרופורטי של שכפול באמצעות DNA נוטה למבנה חוזר בתוך אפיזום אנושי מבוסס SV40
In This Article
Summary
כאן, אנו מתארים את ההליך לניתוח התקדמות השכפול באמצעות חזרות פתוגניות, נוטות למבנה באמצעות אלקטרופורזה ג'ל דו מימדית.
Abstract
אלקטרופורזה ג'ל נייטרלי/נייטרלי דו-ממדי (2DGE) התפתחה כטכניקת אמת מידה לניתוח שכפול DNA דרך מכשולים טבעיים. פרוטוקול זה מתאר כיצד לנתח את התקדמות מזלג השכפול באמצעות חזרות DNA מובנות וניתנות להרחבה בתוך אפיזום מבוסס וירוס סימיאן 40 (SV40) בתאים אנושיים. בקצרה, עם טרנספקציה פלסמיד לתאים אנושיים, מתווכי השכפול מבודדים על ידי פרוטוקול הירט שונה ומטופלים באנזים הגבלת DpnI כדי להסיר DNA שאינו משוכפל. לאחר מכן מתווכים מתעכלים על ידי אנזימי הגבלה מתאימים כדי למקם את החזרה על העניין בתוך המחצית המוצאת-דיסטלית של מקטע DNA באורך 3-5 קילובייט. מתווכי השכפול מופרדים לשני ממדים ניצבים, תחילה לפי גודל ולאחר מכן לפי צורה. בעקבות הכלאת הכתם הדרומי, גישה זו מאפשרת לחוקרים לצפות בהשתקעות מזלג בחזרות יוצרות מבנה שונות במחצית היורדת של קשת Y-השכפול. יתר על כן, מיקום זה של אתר הדוכן מאפשר הדמיה של תוצאות שונות של הזדקרות מזלג בתיווך חוזר, כגון היפוך מזלג, הופעת מזלג מתכנס, והפעלה מחדש של מזלג רקומבינלי.
Introduction
חזרות טנדם קצרות (STR) הן רצפים קטנים, בדרך כלל 2-9 זוגות בסיסים (bp), רצפים חוזרים של דנ"א המהווים כ-3% מהגנום האנושי1. STR ממלא תפקיד חשוב בבקרת גנים2; עם זאת, הרכבם החוזר מותיר אותם מועדים להיווצרות מבנה משני של DNA לא קנוני וחוסר יציבות גנטית 3,4. החל מסלילים שמאליים, דרך סיכות ראש/צלבים, וכלה בסלילים בעלי שלושה וארבעה גדילים, מבני דנ"א חלופיים אלה גורמים לאתגרים מהותיים עבור הרפליזום. תנאי מוקדם טבעי להיווצרות מבנה משני הוא שחרור DNA, שהוא תנאי מוקדם לשכפול DNA. זה מציג חידה ייחודית לתפקוד הגנום מכיוון שרבים מהמבנים הללו יכולים להיווצר במהלך השכפול, לעכב את התקדמות השכפול ובסופו של דבר לגרום לעיכוב מזלג השכפול 5,6,7, או במקרים חמורים, קריסת מזלג ושבירת DNA 8,9. הן הפעלה מחדש של מזלגות תקועים והן מסלולי תיקון דנ"א הוכחו כמובילים לחוסר יציבות חוזרת, כגון הרחבות חוזרות10,11 וסידורי גנום מורכבים (CGR)12,13. אירועים אלה יכולים לגרום להתפתחות של כ -60 מחלות אנושיות הידועות כהפרעות התפשטות חוזרות, כולל תסמונת X שביר, מחלת הנטינגטון, אטקסיה של פרידרייך ואחרות14,15 כמו גם מחלות CGR, כגון תסמונת עמנואל16. לכן, כדי להבין טוב יותר את מנגנוני המחלה האנושית המונעים על ידי חוסר יציבות חוזרת, הכרחי ללמוד את הפרטים של התקדמות מזלג השכפול באמצעות חזרות אלה.
טכניקה לחקר התקדמות השכפול התפתחה באמצע שנות ה-80 של המאה ה-20, כאשר ברואר ופאנגמן ביקשו לספק ראיות ישירות לכך שהתחלת השכפול ב-Saccharomyces cerevisiae מתרחשת באלמנטים17 של רצף שכפול אוטונומי (הידוע בכינויו ARS). בעשותם כן, הם הפרידו מבנים של מתווכי שכפול שמרים באגרוז, תוך אימוץ שיטה מוקדמת יותר של בל וביירס הידועה בשם אלקטרופורזה ג'ל נייטרלי/נייטרלי דו-ממדי (2DGE)18. טכניקה זו ניצלה את העובדה שדנ"א לא ליניארי נע באופן שונה בג'ל אגרוז מאשר המקבילה הליניארית שלו לאותה מסה. באופן ספציפי יותר, ב-2DGE, דנ"א מבודד מופרד בשני ממדים ניצבים, תחילה בעיקר לפי גודל ולאחר מכן בעיקר לפי צורה, כדי ליצור מפה מקיפה של שכפול באזור עניין מסוים. במאמרם המקורי, ברואר ופאנגמן הדגימו זאת כקשת המורכבת ממבני "Y פשוטים" או מזלגות שכפול המגשרים על דנ"א לא משוכפל לעמיתיהם המשוכפלים. הם גם מתארים מתווכים נצפים אחרים כ"בועות" ו"Ys כפולים", המייצגים מקורות שכפול ומזלגות מתכנסים, בהתאמה.
ניתן להשתמש ב-2DGE כדי לחקור את האוכלוסיות היחסיות של מתווכי שכפול דנ"א בזמן נתון. לכן, אם אוכלוסיית ביניים אחת נפוצה יותר מאחרת, הדבר יתברר בהדמיה. זה הופך את 2DGE לכלי שימושי במיוחד לחקר התקדמות השכפול באמצעות רצפים מאתגרים, כמו חזרות יוצרות מבנה. לדוגמה, אם האזור שנותח מכיל רצף שמסוגל לגרום להזדקרות מזלג שכפול, זה יופיע כבליטה בקשת (איור 1A), מה שמצביע על הצטברות של מזלגות שכפול באותו לוקוס. ניתן לראות זאת עם שכפול של רצפים חוזרים יוצרי סיכות שיער בשמרים 19,20,21 וחזרות יוצרות טריפלקס בתאים אנושיים 22,23,24. בנוסף להשתהות, ניתן להשתמש ב-2DGE כדי לצפות במבני דנ"א שאינם תואמים את ה-Y הפשוט הסטנדרטי שנוצר במהלך השכפול, כמו במקרה של מתווכים רקומביננטיים25. למזלגות ביניים אלה יש מבנה כבד ומסועף יותר בצורת X, ולכן הם נעים לאט יותר הן בממד הראשון והן בממד השני מאשר מזלגות שכפול סטנדרטיים. תוצאות דומות ניתן לראות גם לגבי היפוך מזלג שכפול 20,24,26. בתגובה ללחץ שכפול חזק, תאים אאוקריוטים הוכחו כמנצלים היפוך מזלג שכפול כדי להציל מזלגות תקועים. מזלגות הפוכים אלה הם בעלי משקל מולקולרי דומה למזלגות תקועים; עם זאת, מבנה רגל העוף שלהם גורם לניידות אלקטרופורטית איטית יותר בממד השני ביחס למשלימים בצורת Y שלהם, וכתוצאה מכך מתרחבת למעלה והחוצה מהקשת.
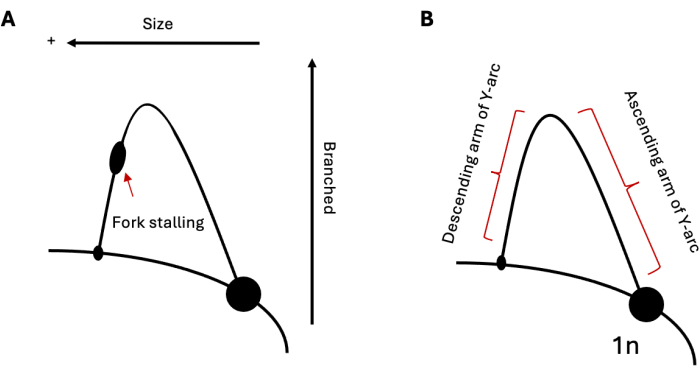
איור 1: ניתוח אלקטרופורזה של ג'ל דו-ממדי של שכפול דנ"א. (A) סכמטי של 2DGE טיפוסי המתאר שכפול באמצעות חזרה יוצרת מבנה המסוגלת לגרום להיתקעות מזלג. גודל ומבנה ביניים ישפיעו על ניידות אלקטרופורטית. (B) דגימה של קשת Y עם זרועות עולות ויורדות בהתאמה. קיצור: 2DGE = אלקטרופורזה ג'ל נייטרלי/נייטרלי דו-מימדי. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
באופן טבעי, אחד ההיבטים החשובים ביותר של 2DGE נוגע לאיכות וכמות מתווכי השכפול. עם זאת, הרזולוציה של ניתוח 2DGE של שכפול באמצעות אתרים אנדוגניים בתאי יונקים אינה מספיקה לרצף מטרה של עותק יחיד בתוך הגנום האנושי הדיפלואידי 6 × 109 bp, אם כי היא נעשתה עבור גנים מרובי עותקים, כגון מוקד DHFRמוגבר מאוד 27 או RNA ריבוזומלי28. שכפול מבוסס SV40 הוא אמצעי יעיל ומאופיין היטב לחקר שכפול בתאים איקריוטים29. הוא מספק מודל אמין של שכפול אאוקריוטי המנצל את רוב מכונות השכפול של הפונדקאי כדי לשכפל את הגנום הנגיפי, אשר מחולק לנוקלאוזומים עם זיהום30,31. שני יוצאים מן הכלל בולטים מרפליזום היונקים הם שאנטיגן T (Tag), במקום קומפלקס CMG המארח, משמש כמנחת דנ"א משוכפל, ודלתא של DNA פולימראז מסנתז גדילי DNA מובילים ומפגרים32. ניצלנו מערכת זו על ידי הצבת קטעים פתוגניים של חזרות יוצרות מבנה במורד הזרם ממקור שכפול SV40 בתוך פלסמיד שנוצר במקור במעבדת מאסימו לופס22. חשוב לציין, פלסמיד זה מכיל גם את הגן המקודד לתג עצמו, ובכך גורם לשכפול המכונן והחזק ביותר שלו עם טרנספקציה למגוון תאים אנושיים בתרבית. תכונה זו מולידה כמות גדולה של מוצרים, אידיאלי לניתוח 2DGE של מתווכים שנוצרו במהלך ובתגובה לשכפול של חזרות פתוגניות בתאים אנושיים. כאן, אנו מתארים שיטה מפורטת להדמיה של שכפול חזרות יוצרות מבנה בתוך אפיזום אנושי מבוסס SV40 באמצעות אלקטרופורזה ג'ל דו-ממדית.
Access restricted. Please log in or start a trial to view this content.
Protocol
הערה: הפלסמיד שתוכנן עבור ניתוח 2DGE המתואר שלנו בתאי יונקים צריך להכיל מקור SV40 של שכפול כמה קילובייט במעלה הזרם של חזרות המועדות למבנה (איור 2). יש לזכור סינתזה מובילה ומפגרת בעת בחירת הכיוון ביחס למקור יש לשכפל את החזרות לפלסמיד.

איור 2: עיכול פלסמיד המכיל חזרה עבור ניתוח 2DGE. חזרות המועדות למבנה מתוארות מספר קילו-בתים במורד הזרם ממזלג השכפול הנע ימינה. עיכול עם חותכים ייחודיים 1 ו-2 יניח את הרצף החוזר על הזרוע היורדת של קשת Y, בהינתן הרצף מעבר לנקודת האמצע של המקטע המעוכל. קיצור: 2DGE = אלקטרופורזה ג'ל נייטרלי/נייטרלי דו-מימדי. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
1. טרנספקציה פלסמידית לתאי יונקים
- זרעו 600,000 HEK293T תאים בצלחת תרבית רקמה בקוטר 10 ס"מ לפני ההעברה. אפשר לתאים להתאושש ב 37 ° C בן לילה.
הערה: ניתן להשתמש בקווי תאים רבים בניסוי זה, אם כי תאים המכילים את התג SV40 מומלצים לקבלת תוצאות מיטביות. זהירות: HEK293T התאים נחשבים BSL-2 וכל עבודת הטיפוח צריכה להתבצע בארון בטיחות ביולוגית תוך שימוש בטכניקה אספטית מתאימה וציוד הגנה אישי מתאים. - כאשר התאים מגיעים למפגש של 60%, יש להדביק 8 מיקרוגרם של DNA פלסמיד המכיל חומר חוזר לתוך תאי הזרעים באמצעות ריאגנטים מתאימים בהתאם לפרוטוקול היצרן.
- אם לא מבודדים מתווכים בנקודת זמן זו, שאפו מדיה ישנה והחליפו ב -10 מ"ל של מדיה טרייה לאחר 24 שעות.
- התחל לאסוף תאים 24-48 שעות לאחר הטרנספקציה.
- יש לשאוף מדיה ולשטוף בזהירות עם 10 מ"ל של מלח חוצץ פוספט (PBS). נתק ואסוף תאים באמצעות 0.5 מ"ל של טריפסין וסובב למטה ב 340 × גרם במשך 4 דקות.
- שאפו את הסופרנאטנט ושטפו את כדורי התא עם PBS. סובבו שוב ב 340 × גרם במשך 4 דקות ושאפו את הסופרנטנט.
הערה: ניתן להשהות את הניסוי כאן על ידי הקפאת כדורי תאים ב -80 ° C. מצאנו את הרזולוציה הטובה ביותר בידוד שכפול ביניים 48 שעות לאחר transfection; עם זאת, 24 שעות הניבו תוצאות בנות קיימא.
2. בידוד מתווכי שכפול
- להשהות מחדש את התאים ב 1.5 מ"ל של חיץ הירט ליזה שונה [10 mM tris-HCl (pH 7.5), 10 mM ethylenediaminetetraacetic חומצה (EDTA)] ב 50 מ"ל צינורות חרוטי ולהתחיל ליזה התא.
- הוסף סודיום דודציל סולפט (SDS) לריכוז סופי של 0.6% (כ 650 מיקרוליטר של מלאי 2% SDS) ופרוטאינאז K לריכוז סופי של 100 מיקרוגרם / מ"ל (כ -10 מיקרוליטר של מלאי 20 מ"ג / מ"ל פרוטאינאז K) כדי להסיר נוקלאזות.
- מערבבים בעדינות על ידי פיפטינג עד לקבלת תערובת הומוגנית ודוגרים על התערובת ב-37°C למשך 90 דקות לפחות.
- יש להגדיל את ריכוז NaCl ל-1 M (כ-540 μL של מלאי 5 M NaCl) ולערבב בעדינות עד לקבלת מרקם הומוגני. לדגור לילה (18-24 שעות) ב 4 ° C כדי לאפשר משקעים של פסולת תאים, RNA, וחלבון על ידי המלחה החוצה.
הערה: התערובת תהיה צמיגה מאוד, לכן יש להיזהר ולהתאזר בסבלנות בעת ערבוב טוב. - למחרת, הפרידו את הדנ"א מפסולת תאים, רנ"א וחלבון.
- צנטריפוגה את התערובת ב 29,500 × גרם במשך 45 דקות ב 4 ° C.
- מעבירים את הסופרנאטנט המכיל DNA, מוסיפים נפח אחד של פנול:כלורופורם:איזואמיל אלכוהול 25:24:1 (v/v), ומערבבים לזמן קצר עד להומוגניות.
זהירות: פנול:כלורופורם:אלכוהול איזואמיל הוא חומר מסוכן ויש לטפל בו עם PPE מתאים במכסה אדים כימי. - צנטריפוגה שוב ב 15,000 × גרם במשך 5 דקות בטמפרטורת החדר. מעבירים את השכבה המימית לצינור חרוטי חדש.
- לזרז ולשטוף את הדנ"א המבודד.
- מוסיפים נפח אחד של איזופרופנול טהור ודגרים בטמפרטורת החדר למשך 5 דקות לפחות. סובבו את הדנ"א ב-15,000 × גרם למשך 30 דקות ב-4°C.
- דקרו את הסופרנאטנט ושטפו את הגלולה באתנול קר 70% כדי להסיר עודפי מלח.
- יש לסחור פעם נוספת במהירות של 15,000 × גרם למשך 30 דקות ב-4°C, לייבש באוויר ולהשהות מחדש בעדינות את הגלולה במאגר Tris-EDTA (TE) (10 mM Tris-HCl, 1 mM EDTA).
הערה: ניתן להשהות את הניסוי כאן, וניתן להקפיא דגימות ב -20 ° C; עם זאת, יש להימנע ממחזור הקפאה/הפשרה מכיוון שהדבר עלול לפגוע באיכות מתווכי שכפול ה- DNA.
3. הכנת דגימה ואלקטרופורזה ג'ל דו מימדית
- לעכל את המתווכים המבודדים של שכפול פלסמיד.
- הוסיפו 100 יחידות של אנזימי הגבלה מתאימים לדגימה כדי לעכל את הדנ"א של הפלסמיד, ובמיוחד הניחו את הרצף המכיל חזרה על החצי המוצא-דיסטלי של המקטע הליניארי (איור 2). בנוסף, הוסף DpnI כדי לחתוך את ה- DNA שעבר מתילציה, ובכך להסיר כל DNA פלסמיד שלא שוכפל במלואו בתאים אנושיים בתרבית.
הערה: לקבלת התוצאות הטובות ביותר, אנזימי הגבלה צריכים להיות חותכים ייחודיים המניבים מקטע של 3-5 קילו-בתים הממקם את הרצף הנוטה למבנה על הזרוע היורדת של קשת Y. - לדגור את הדגימות ב 37 ° C במשך 6-10 שעות כדי לאפשר עיכול פלסמיד מלא.
- לזרז את הדנ"א עם 2.5 נפחים של אתנול טהור קר ולדגור ב -20 ° C במשך הלילה, או להוסיף נפח אחד של isopropanol ולדגור בטמפרטורת החדר במשך 5 דקות.
- צנטריפוגה הדגימות מעוכלות ומואצות ב 15,000 × גרם במשך 30 דקות ב 4 ° C.
- דקרו את הסופרנאטנט ושטפו את הדגימה באתנול קר 70%. יש לסחור שוב במהירות של 15,000 × גרם למשך 30 דקות ב-4°C.
- דקרו את הסופרנטנט, יבשו באוויר למשך 10 דקות, והשהו מחדש את הדגימות ב-15 מיקרוליטר של חיץ TE.
- הוסיפו 100 יחידות של אנזימי הגבלה מתאימים לדגימה כדי לעכל את הדנ"א של הפלסמיד, ובמיוחד הניחו את הרצף המכיל חזרה על החצי המוצא-דיסטלי של המקטע הליניארי (איור 2). בנוסף, הוסף DpnI כדי לחתוך את ה- DNA שעבר מתילציה, ובכך להסיר כל DNA פלסמיד שלא שוכפל במלואו בתאים אנושיים בתרבית.
- הכינו את ג'ל האגרוז מהממד הראשון ב-0.4-0.5% ב-1x Tris-borate-EDTA (TBE) (בסיס תריס 89 מילימול, 89 מ"מ חומצה בורית, 2 מ"מ EDTA). אפשר לתמיסה להתמצק במשך שעה אחת לפחות.
- התחל לטעון את הדגימות לממד הראשון.
- יש להעמיס את הסולם בתוך 3 הס"מ הראשונים ביחס לקצה השמאלי ביותר של הג'ל. לאחר מכן, לטעון את כל הדגימות מוכנות, להבטיח 3 ס"מ בין כל זוג.
- הפעל את הג'ל ב 1x TBE במשך 19-24 שעות ב 0.85 V / cm כדי להפריד את המתווכים ביחס לגודלם. ודא שהחדר מכוסה כדי להגן על הדגימות מפני אור, שעלול לגרום נזק לדנ"א.
- למחרת, הסר את הג'ל מהמאגר והערך את מיקום המקטע הליניארי המעוכל באמצעות סרגל.
- מוציאים את 3 הס"מ הראשונים של הג'ל המכיל את הסולם ומכתימים את קטע הג'ל ב-1x TBE המכיל 0.3 מיקרוגרם/מ"ל אתידיום ברומיד למשך 10-15 דקות. דמיינו את הסולם באמצעות מערכת תיעוד ג'ל.
- הוסף 1.3 ס"מ למיקום המשוער, מניב ערך a. לאחר מכן, החסרו 7.5 ס"מ מערך a, והניבו ערך b. יישר את הסרגל כנגד ג'ל הממד הראשון וחתוך אופקית לרוחב בערכים a ו - b. לאחר מכן, חתכו אנכית את החלל של 3 ס"מ השמור לכל דגימה. עיין באיור 3 לקבלת סכמה חזותית.
- במגש יציקה חדש, סובבו את המקטעים בכיוון השעון והניחו אותם במיקום של בארות הדגימה (איור 3).
- הכינו את ג'ל האגרוז מהממד השני בריכוז של 1-1.3% ב-1x TBE ב-0.3 מיקרוגרם/מ"ל אתידיום ברומיד.
- לאחר קירור לטמפרטורה של כ-55°C, שפכו את ג'ל הממד השני על המקטעים המסובבים של הממד הראשון ואפשרו לו להתמצק למשך שעה אחת לפחות.
- העבירו את הממד השני לתא עם 1x TBE ב-0.3 מיקרוגרם/מ"ל אתידיום ברומיד, ואפשרו לג'ל להתאזן למשך 30 דקות לפחות.
- הפעל את הג'ל, מכוסה שוב, במשך 9-10 שעות ב 4.23 V / cm ב 4 ° C כדי להפריד את המתווכים ביחס לצורתם.
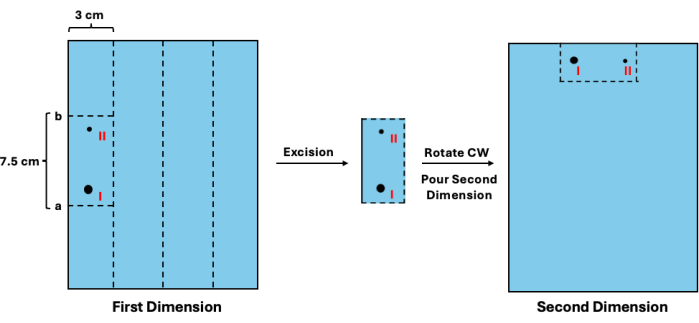
איור 3: כריתה של מתווכים מהממד הראשון לפני הפרדת הממד השני. לאחר הדמיה של הסולם, ניתן להעריך את הניידות של שברים לא משוכפלים. (I) לאחר מכן ניתן להשתמש בערך זה כדי לקבוע אתרי חיתוך מתאימים (a ו-b) כדי להחריג אותו ואת עמיתיו המשוכפלים (II). לאחר מכן יש לסובב את קטע הג'ל ולמקם אותו במצב של בארות להפרדת הממד השני. קיצור: CW = בכיוון השעון. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
4. כתם דרומי והכלאה עם גשושית מסומנת רדיו
- הסר את ג'ל הממד השני מהחדר ופרק את מקטעי ה- DNA למשך 10 דקות בתמיסת HCl של 0.24 M עם נדנוד עדין. שטפו את הג'ל במים נטולי יונים, והשרו אותו ב-0.4 M NaOH למשך 10-15 דקות.
אזהרה: HCl ו-NaOH הם קורוזיביים ויש לטפל בהם עם PPE מתאים במכסה אדים כימי. - התחל להרכיב את הכתם הדרומי כדי להקל על העברת המתווכים המופרדים מהג'ל לממברנה. עיין באיור 4 לקבלת סכמה מקיפה.
- מלא מיכל בגודל מספיק עם 1 L של 0.4 M NaOH.
- יישרו יריעת זכוכית ארוכה לרוחב המיכל וקפלו (לאורכו) שתי יריעות ארוכות של נייר כרומטוגרפיה בניצב לרוחב יריעת הזכוכית, הנמשכות לתוך המיכל של NaOH.
- הרטיבו את החלק העליון של הנייר עם NaOH והסירו בזהירות בועות אוויר מתחת לפני השטח שלו.
- השרו שלושה גיליונות נייר כרומטוגרפיה עם NaOH והניחו אותם על הנייר המקופל, שוב הסירו בועות.
- הפוך את ג'ל הממד השני והעבר אותו על הניירות.
- יש להרטיב קרום ניילון טעון חיובית (גודל נקבוביות של 0.45 מיקרומטר) במי DI ולהניח אותו מעל הג'ל.
- לבסוף, מוסיפים עוד שלושה גיליונות נייר, רטובים במי DI, מעל הממברנה.
- כסו את ה-NaOH החשוף במיכל התחתון בניילון נצמד למניעת אידוי. הניחו ערימה של מפיות או מגבות נייר על הכתם, וודאו שגובהו 0.3-0.5 מ'. מניחים משקולת על החלק העליון, דוחסים את הכתם כולו כדי להקל על פעולה נימים הדוקה. יש להמתין לפחות יומיים להעברת הדנ"א לממברנה.
הערה: האורך והרוחב של נייר הכרומטוגרפיה והממברנה תלויים בגודל ג'ל האגרוז המשמש לממד השני. להעברה היעילה ביותר, השתמש בנייר ובממברנה עם אותם ממדים כמו אלה של הג'ל.
- לאחר ההעברה, הצליבו את הדנ"א לממברנה באמצעות קרוסלינקר UV בגודל 120 μJ/cm2 למשך דקה אחת.
הערה: ניתן להשהות את הניסוי כאן על ידי הנחת הממברנה במגן יריעות אטום, יבש ונקי בטמפרטורת החדר. - שטפו את הממברנה 2x במשך 5 דקות עם 2x מלוחים נתרן ציטראט (מאגר SSC) (0.3 M NaCl, 0.03 M נתרן ציטראט).
- בצע הכלאה מוקדמת של הממברנה עם 0.18 מ"ל/ס"מ2 של חיץ ההכלאה של Church &; Gilbert [1 mM EDTA, אלבומין בסרום בקר 1% (BSA), 0.5 M נתרן פוספט, 7% SDS] ב-65°C, מסתובב באינקובטור הכלאה למשך שעתיים לפחות.
הערה: הממברנה עשויה לעבור הכלאה מוקדמת במשך מספר ימים. - הכן את הבדיקה עם תווית רדיו באמצעות α-32P, dATP או dCTP וערכת תיוג DNA בהתאם לפרוטוקול היצרן.
התראה: dNTPs עם תווית רדיו הם מסוכנים, ויש ללבוש PPE מתאים בעת הטיפול בהם. כל העבודה הרדיואקטיבית צריכה להתבצע מאחורי מיגון, ואנשים מאומנים צריכים להיות מנוטרים לספיגת קרינה באמצעות דוזימטרים.- תכננו מקטע דנ"א ליניארי של 400-900 bp המשלים את רצף ההגבלה המעוכל (איור 2) והגבירו את המקטע באמצעות תגובת שרשרת פולימראז (PCR).
הערה: אנו ממליצים להחזיק מקטע PCR מלאי של 50-100 ng/μL - שלב 100 ng של מקטע PCR משלים עם DNA Pol I, מקטע Klenow (3' 5' exo-) חיץ ואוליגוס decanucleotide אקראי.
- נטרל את השבר ב 100 ° C למשך 10 דקות.
- הוסף 5 יחידות של DNA Pol I, מקטע Klenow (3' 5' exo-), 50 μCi של α-32P dNTP, ו 30-50 μmol של תערובת dNTP חסר בסוג dNTP radiolabeled.
- יש לדגור בטמפרטורה של 37°C למשך 10 דקות כדי לאפשר פילמור ושילוב dNTP עם תווית רדיו.
- הוסף 30-50 μmol של סוג dNTP שנעדר בעבר לדגימה ודגור ב 37 ° C במשך 10 דקות.
- טהרו את הקטע המסומן ברדיו באמצעות עמוד ספין במשקל של 3,000 × למשך 2 דקות.
- תכננו מקטע דנ"א ליניארי של 400-900 bp המשלים את רצף ההגבלה המעוכל (איור 2) והגבירו את המקטע באמצעות תגובת שרשרת פולימראז (PCR).
- הוסף בדיקה רדיו-מסומנת ל-50 מ"ל של חיץ הכלאה ודגור עם הממברנה למשך הלילה ב-65 מעלות צלזיוס כשהיא מסתובבת באינקובטור הכלאה.
- למחרת, הסר את הבדיקה ושטוף את הממברנה 2x עם חיץ כביסה 1 (0.1x SSC, 0.1% SDS) ב 42 ° C ו 2x עם חיץ כביסה 2 (2x SSC, 0.1% SDS) ב 65 ° C.
הערה: יש לבצע את כל השטיפות בסיבובים מהירים למשך 15 דקות באינקובטור. - יבשו את הממברנה למשך 10 דקות והניחו אותה במגן יריעות דק ושקוף. אחסנו את הממברנה האטומה בקסטה מבוקרת ועמידה בפני קרינה עם מסך רגיש לזרחן. אפשרו לממברנה להיחשף למסך למשך 1-10 ימים.
- הצג את התוצאות באופן חזותי באמצעות מכשיר הדמיה ביומולקולרי המוגדר להדמיית זרחן. יש לשטוף ולחשוף מחדש במידת הצורך.
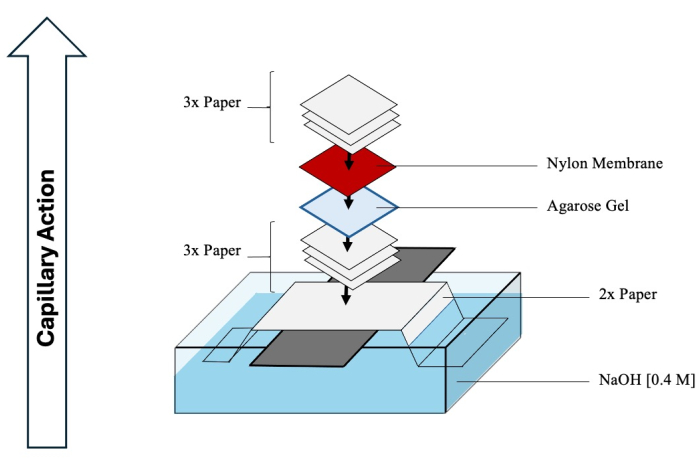
איור 4: הרכבה של כתם דרומי. סכמה מקיפה של מכשיר טיפוסי המשמש להעברת כתם דרומי של מתווכים מהממד השני לקרום ניילון. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
Access restricted. Please log in or start a trial to view this content.
תוצאות
אם ההדמיה מוצלחת, ניתן לראות קשת חדה של מזלגות שכפול הנמשכת מעלה והחוצה מהנקודה 1n המסיבית (איור 5A). גודלו של מקטע, או אחוזים המשוכפלים, קובע את ניידות המקטע בממד הראשון. ככל שהמתווכים מפתחים מבנה משותף יותר, הם יתחילו לנוע לאט יותר בממד השני. לכן, אם מתווך נע...
Access restricted. Please log in or start a trial to view this content.
Discussion
2DGE מספק תמונה כמותית ומקיפה למחצה של אוכלוסיות הביניים היחסיות המתעוררות במהלך שכפול של רצף מסוים. בהתחשב בכך שהמבנים המולקולריים השבירים של מזלגות השכפול חייבים להישמר לאורך הליך זה, יש ליישם זהירות רבה כדי למנוע גזירה פיזית ודנטורציה כימית. לכן, מומלץ מאוד להימנע מכל ...
Access restricted. Please log in or start a trial to view this content.
Disclosures
למחברים אין ניגודי עניינים לחשוף.
Acknowledgements
אנו מודים לחורחה סבריאן ואנסטסיה רסטוקינה שהחלו לפתח גישה זו במעבדה שלנו, למאסימו לופס על שסיפקו לנו פלסמיד pML113 ועצות שלא יסולא בפז, לילי דוקסאני על דיונים מעמיקים, ולחברי מעבדת מירקין על תמיכתם. העבודה במעבדת מירקין נתמכת על ידי המכון הלאומי למדעי הרפואה הכלליים [R35GM130322] ו- NSF-BSF [2153071].
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 10x TBE Buffer | Bio Rad | 1610733 | |
| 20x SSC Buffer | Fisher Scientific | BP1325-1 | |
| 293T cells | ATCC | CRL-3216 | |
| a-32P dATP, 3000 Ci/mmol | Revvity | BLU512H250UC | |
| Agarose | Fisher Scientific | BP160-500 | |
| Amersham Hybond-N+ | Fisher Scientific | RPN303B | |
| BAS Storage Phosphor Screens | Fisher Scientific | 28956482 | |
| Church and Gibert's hybriddization buffer | Fisher Scientific | 50-103-5408 | |
| DecaLabel DNA labeling kit | ThermoFisher Scientific | K0622 | |
| DMEM, high gluctose, GltaMAX Supplement, pyruvate | ThermoFisher Scientific | 10569010 | |
| DpnI | New England Biolabs | R0176S | Additional restriction enzymes will need to be purchased as well |
| EDTA 0.5 M, pH 8 | Fisher Scientific | BP2482500 | |
| Ethanol, 70% | Fisher Scientific | BP82031GAL | |
| Fetal Bovine Serum | VWR | 97068-085 | |
| Hydrochloric acid solution, 12 M | Millipore Sigma | 13-1683 | |
| Isopropanol | Fisher Scientific | BP26184 | |
| jetPRIME DNA and siRNA Transfection Reagent with Buffer | VWR | 101000027 | |
| MycoZap Plus-CL | VWR | 75870-448 | |
| NaCl | Millipore Sigma | 746398-500G | |
| Nalgene Oak Ridge High-Speed Centrifuge Tubes | ThermoFisher Scientific | 3139-0050 | |
| Phosphate Buffer Saline, pH 7.4 | ThermoFisher Scientific | 10010023 | |
| Phosphate Buffer Saline, pH 7.5 | ThermoFisher Scientific | 10010024 | |
| Proteinase K | ThermoFisher Scientific | EO0491 | |
| Proteinase K | ThermoFisher Scientific | EO0492 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-4 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-5 | |
| Ruler | Fisher Scientific | 09-016 | |
| Scalpel | Fisher Scientific | 12-460-451 | |
| Sodium dodecyl sulfate | Millipore Sigma | 436143-25G | |
| Sodium hydroxide | Fisher Scientific | S25548 | |
| Sorval LYNX 4000 Superspeed Centrifuge | ThermoFisher Scientific | 75006580 | |
| Sub-cell Horizontal Electrophoresis System | Bio Rad | 1704401 | |
| TH13-6 x 50 Swinging Bucket Rotor | ThermoFisher Scientific | 75003010 | |
| Tris-HCl 1 M, pH 7.5 | Fisher Scientific | BP1757-500 | |
| Trypsin-EDTA (0.25%), phenol red | ThermoFisher Scientific | 25200056 |
References
- Liao, X., et al. Repetitive DNA sequence detection and its role in the human genome. Commun Biol. 6 (1), 1-21 (2023).
- Fotsing, S. F., et al. The impact of short tandem repeat variation on gene expression. Nat Genet. 51 (11), 1652-1659 (2019).
- Fan, H., Chu, J. -Y. A brief review of short tandem repeat mutation. GPB. 5 (1), 7-14 (2007).
- Khristich, A. N., Mirkin, S. M. On the wrong DNA track: Molecular mechanisms of repeat-mediated genome instability. J Biol Chem. 295 (13), 4134-4170 (2020).
- Samadashwily, G. M., Raca, G., Mirkin, S. M. Trinucleotide repeats affect DNA replication in vivo. Nat Genet. 17 (3), 298-304 (1997).
- Khristich, A. N., Armenia, J. F., Matera, R. M., Kolchinski, A. A., Mirkin, S. M. Large-scale contractions of Friedreich's ataxia GAA repeats in yeast occur during DNA replication due to their triplex-forming ability. Proc Natl Acad Sci USA. 117 (3), 1628-1637 (2020).
- Shishkin, A. A., et al. Large-scale expansions of Friedreich's ataxia GAA repeats in yeast. Mol Cell. 35 (1), 82-92 (2009).
- Sundararajan, R., Gellon, L., Zunder, R. M., Freudenreich, C. H. Double-strand break repair pathways protect against CAG/CTG repeat expansions, contractions and repeat-mediated chromosomal fragility in Saccharomyces cerevisiae. Genetics. 184 (1), 65-77 (2010).
- Kim, H. -M., et al. Chromosome fragility at GAA tracts in yeast depends on repeat orientation and requires mismatch repair. EMBO J. 27 (21), 2896-2906 (2008).
- Polleys, E. J., House, N. C. M., Freudenreich, C. H. Role of recombination and replication fork restart in repeat instability. DNA Repair. 56, 156-165 (2017).
- Gold, M. A., et al. Restarted replication forks are error-prone and cause CAG repeat expansions and contractions. PLoS Genet. 17 (10), e1009863(2021).
- Lambert, S., et al. Homologous recombination restarts blocked replication forks at the expense of genome rearrangements by template exchange. Mol Cell. 39 (3), 346-359 (2010).
- Burssed, B., Zamariolli, M., Bellucco, F. T., Melaragno, M. I. Mechanisms of structural chromosomal rearrangement formation. Mol Cytogenet. 15 (1), 23(2022).
- Paulson, H. Repeat expansion diseases. Handb Clin Neurol. 147, 105-123 (2018).
- Malik, I., Kelley, C. P., Wang, E., Todd, P. Molecular mechanisms underlying nucleotide repeat expansion disorders. Nat Rev Mol Cell Biol. 22 (9), 589-607 (2021).
- Emanuel, B. S., Zackai, E. H., Medne, L. Emanuel Syndrome. GeneReviews®. Adam, M. P. , University of Washington. Seattle, Seattle, WA. (1993).
- Brewer, B. J., Fangman, W. L. The localization of replication origins on ARS plasmids in S. cerevisiae. Cell. 51 (3), 463-471 (1987).
- Bell, L., Byers, B. Separation of branched from linear DNA by two-dimensional gel electrophoresis. Anal Biochem. 130 (2), 527-535 (1983).
- Voineagu, I., Narayanan, V., Lobachev, K. S., Mirkin, S. M. Replication stalling at unstable inverted repeats: Interplay between DNA hairpins and fork stabilizing proteins. Proc Natl Acad Sci USA. 105 (29), 9936-9941 (2008).
- Nguyen, J. H. G., et al. Differential requirement of Srs2 helicase and Rad51 displacement activities in replication of hairpin-forming CAG/CTG repeats. Nucleic Acids Res. 45 (8), 4519-4531 (2017).
- Krasilnikova, M. M., Mirkin, S. M. Analysis of triplet repeat replication by two-dimensional gel electrophoresis. Trinucleotide Repeat Protocols. Kohwi, Y. , Humana Press. Totowa, NJ. (2004).
- Follonier, C., Oehler, J., Herrador, R., Lopes, M. Friedreich's ataxia-associated GAA repeats induce replication-fork reversal and unusual molecular junctions. Nat Struct Mol Biol. 20 (4), 486-494 (2013).
- Chandok, G. S., Patel, M. P., Mirkin, S. M., Krasilnikova, M. M. Effects of Friedreich's ataxia GAA repeats on DNA replication in mammalian cells. Nucleic Acids Res. 40 (9), 3964-3974 (2012).
- Rastokina, A., et al. Large-scale expansions of Friedreich's ataxia GAA•TTC repeats in an experimental human system: role of DNA replication and prevention by LNA-DNA oligonucleotides and PNA oligomers. Nucleic Acids Res. 51 (16), 8532-8549 (2023).
- Giannattasio, M., et al. Visualization of recombination-mediated damage-bypass by template switching. Nat Struct Mol Biol. 21 (10), 884-892 (2014).
- Hisey, J. A., et al. Pathogenic CANVAS (AAGGG)n repeats stall DNA replication due to the formation of alternative DNA structures. Nucleic Acids Res. 52 (8), 4361-4374 (2024).
- Kalejta, R. F., Lin, H. B., Dijkwel, P. A., Hamlin, J. L. Characterizing replication intermediates in the amplified CHO dihydrofolate reductase domain by two novel gel electrophoretic techniques. Mol Cell Biol. 16 (9), 4923-4931 (1996).
- Little, R. D., Platt, T. H., Schildkraut, C. L. Initiation and termination of DNA replication in human rRNA genes. Mol Cell Biol. 13 (10), 6600-6613 (1993).
- Fanning, E., Zhao, K. SV40 DNA replication: From the A gene to a nanomachine. Virology. 384 (2), 352-359 (2009).
- Sogo, J. M., Stahl, H., Koller, T., Knippers, R. Structure of replicating simian virus 40 minichromosomes: The replication fork, core histone segregation and terminal structures. J Mol Biol. 189 (1), 189-204 (1986).
- Weisshart, K., Taneja, P., Fanning, E. The replication protein A binding site in simian virus 40 (SV40) T antigen and its role in the initial steps of SV40 DNA replication. J Virol. 72 (12), 9771-9781 (1998).
- Sowd, G. A., Fanning, E. A Wolf in sheep's clothing: SV40 co-opts host genome maintenance proteins to replicate viral DNA. PLoS Pathog. 8 (11), e1002994(2012).
- National Library of Medicine: National Center for Biotechnology Information. BLAST: Basic Local Alignment Search Tool. , https://blast.ncbi.nlm.nih.gov/Blast.cgi (2024).
- Hirt, B. Selective extraction of polyoma DNA from infected mouse cell cultures. J Mol Biol. 26 (2), 365-369 (1967).
- Lopes, M., Foiani, M., Sogo, J. M. Multiple mechanisms control chromosome integrity after replication fork uncoupling and restart at irreparable UV lesions. Mol Cell. 21 (1), 15-27 (2006).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved