Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Электрофоретический анализ репликации через структурно-склонные повторы ДНК в эписоме человека, основанной на SV40
В этой статье
Резюме
Здесь мы описываем процедуру анализа прогрессии репликации через патогенные, склонные к структуре повторы с использованием 2-мерного гель-электрофореза.
Аннотация
Двумерный нейтральный/нейтральный гель-электрофорез (2DGE) стал эталонным методом анализа репликации ДНК через естественные препятствия. В этом протоколе описывается, как анализировать прогрессирование репликационной вилки через склонные к структуре, расширяемые повторы ДНК в эписоме на основе вируса обезьян 40 (SV40) в клетках человека. Вкратце, при трансфекции плазмиды в клетки человека промежуточные продукты репликации выделяют по модифицированному протоколу Hirt и обрабатывают ферментом рестрикции DpnI для удаления нереплицированной ДНК. Затем промежуточные продукты расщепляются соответствующими ферментами рестрикции, чтобы поместить интересующий их повтор в исходную дистальную половину фрагмента ДНК длиной 3-5 кб. Промежуточные продукты репликации разделены на два перпендикулярных измерения, сначала по размеру, а затем по форме. Следуя гибридизации методом Саузерн-блот, этот подход позволяет исследователям наблюдать остановку вилки при различных структурообразующих повторах на нисходящей половине Y-дуги репликации. Кроме того, такое расположение места остановки позволяет визуализировать различные результаты остановки вилки, опосредованной повторами, такие как реверсирование вилки, появление сходящейся вилки и рекомбинационный перезапуск вилки.
Введение
Короткие тандемные повторы (STR) представляют собой небольшие, обычно 2-9 пар оснований (.о.), повторяющиеся последовательности ДНК, которые составляют около 3% генома человека. STR играют важную роль в регуляции генов2; однако их повторяющийся состав делает их склонными к образованию неканонических вторичных структур ДНК и последующей генетической нестабильности 3,4. От левосторонних спиралей до шпилек/крестообразных, до трех- и четырехцепочечных спиралей, эти альтернативные структуры ДНК вызывают внутренние проблемы для репсомы. Естественной предпосылкой для формирования вторичной структуры является раскручивание ДНК, которое является предпосылкой для репликации ДНК. Это представляет собой уникальную головоломку для функционирования генома, поскольку многие из этих структур могут образовываться во время репликации, препятствуя прогрессированию репликосом и в конечном итоге вызывая остановку репликационной вилки 5,6,7 или, в тяжелых случаях, коллапс вилки и разрыв ДНК 8,9. Было показано, что как перезапуск застопорившихся форков, так и пути репарации ДНК приводят к повторной нестабильности, такой как повторные расширения10,11 и сложные перестройки генома (CGR)12,13. Эти события могут привести к развитию примерно 60 заболеваний человека, известных как расстройства повторного распространения, включая синдром ломкой X-хромосомы, болезнь Хантингтона, атаксию Фридрейха идругие14,15, а также заболевания CGR, такие как синдром Эммануэля16. Таким образом, чтобы лучше понять механизмы развития заболеваний человека, вызванных нестабильностью повторов, крайне важно изучить детали прогрессирования репликационной вилки через эти повторы.
Метод изучения прогрессии репликации появился в середине 1980-х годов, когда Брюэр и Фангман попытались предоставить прямые доказательства того, что инициация репликации у Saccharomyces cerevisiae происходитна элементах автономной репликации (широко известной как ARS). При этом они разделили структуры промежуточных продуктов репликации дрожжей в агарозе, адаптировав более ранний метод Белла и Байерса, известный как двумерный нейтральный/нейтральный гель-электрофорез (2DGE)18. Этот метод использовал тот факт, что нелинейная ДНК перемещается в агарозном геле иначе, чем ее линейный эквивалент той же массы. В частности, в 2DGE изолированная ДНК разделена в двух перпендикулярных измерениях, сначала по размеру, а затем преимущественно по форме, чтобы создать всеобъемлющую карту репликации в определенной области интереса. В своей оригинальной статье Брюэр и Фангман продемонстрировали это в виде дуги, состоящей из «простых» Y-образных структур или репликационных вилок, соединяющих нереплицированную ДНК с их реплицированными аналогами. Они также описывают другие наблюдаемые промежуточные продукты как «пузыри» и «двойные Y», представляющие начало репликации и сходящиеся вилки соответственно.
2DGE может быть использован для изучения относительных популяций промежуточных продуктов репликации ДНК в данный момент времени. Следовательно, если одна популяция промежуточных звеньев более распространена, чем другая, это будет очевидно при визуализации. Это делает 2DGE особенно полезным инструментом для изучения прогрессии репликации через сложные последовательности, такие как структурообразующие повторы. Например, если анализируемая область содержит последовательность, способную вызвать остановку репликационной вилки, она будет представлена в виде выпуклости на дуге (рис. 1A), указывающей на накопление репликационных вилок в этом локусе. Это можно увидеть на примере репликации как последовательностей повторяющихся шпилек, образующих шпильки, у дрожжей 19,20,21, так и триплексобразующих повторов в клетках человека 22,23,24. В дополнение к остановке, 2DGE может быть использован для наблюдения за структурами ДНК, которые не соответствуют стандартным простым Ys, образующимся при репликации, как в случае рекомбинантных промежуточных продуктов25. Эти промежуточные продукты имеют более тяжелую и разветвленную X-образную структуру и, следовательно, перемещаются медленнее как в первом, так и во втором измерениях, чем стандартные репликационные вилки. Аналогичные результаты также можно наблюдать в отношении реверсирования репликационной вилки 20,24,26. Было показано, что в ответ на сильный репликационный стресс эукариотические клетки используют реверсию репликационной вилки для спасения застрявших вилок. Эти перевернутые вилки имеют молекулярную массу, аналогичную застрявшим вилкам; тем не менее, их структура «куриная лапка» приводит к более медленной электрофоретической подвижности во втором измерении по сравнению с их Y-образными дополнениями, что приводит к удлинению дуги вверх и наружу.
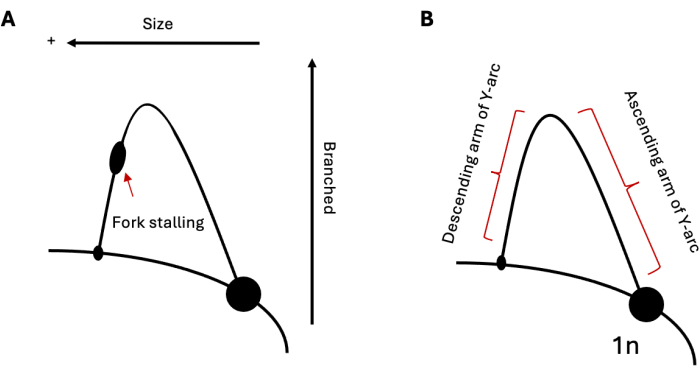
Рисунок 1: 2D гелевый электрофорезный анализ репликации ДНК. (A) Схема типичного 2DGE, изображающая репликацию через структурообразующий повтор, способный вызвать остановку вилки. Промежуточный размер и структура будут влиять на электрофоретическую подвижность. (B) Образец Y-дуги с соответственно восходящими и нисходящими рукавами. Аббревиатура: 2DGE = Двумерный нейтральный/нейтральный гель-электрофорез. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Естественно, что одним из наиболее важных аспектов 2DGE является качество и количество промежуточных продуктов репликации. Тем не менее, разрешение 2DGE-анализа репликации через эндогенные локусы в клетках млекопитающих является недостаточным для последовательности-мишени с одной копией в пределах диплоидного генома человека 6 × 109.н., хотя это было сделано для генов с несколькими копиями, таких как сильно амплифицированный DHFR локус27 или рибосомная РНК28. Репликация на основе SV40 является эффективным и хорошо охарактеризованным средством изучения репликации в эукариотических клетках29. Он обеспечивает надежную модель репликации эукариот, которая использует большую часть механизма репликации хозяина для репликации вирусного генома, который разделяется на нуклеосомы при инфицировании 30,31. Два заметных исключения из ответной реакции млекопитающих заключаются в том, что Т-антиген (Tag), вместо комплекса CMG хозяина, служит репликативной хеликазой ДНК, а ДНК-полимераза дельта синтезирует как ведущую, так и отстающую цепи ДНК32. Мы воспользовались преимуществами этой системы, поместив патогенные участки структурообразующих повторов ниже по потоку от источника репликации SV40 в плазмиде, которая была первоначально создана в лаборатории Массимо Лопеса22. Важно отметить, что эта плазмида также содержит ген, кодирующий саму Tag, что приводит к ее конститутивной и чрезвычайно мощной репликации при трансфекции в различные культивируемые клетки человека. Эта особенность обуславливает большое количество продуктов, идеально подходящих для 2DGE-анализа промежуточных продуктов, образующихся во время и в ответ на репликацию патогенных повторов в клетках человека. В данной статье мы подробно описываем метод визуализации репликации структурообразующих повторов в эписоме человека на основе SV40 с использованием двумерного гель-электрофореза.
Access restricted. Please log in or start a trial to view this content.
протокол
ПРИМЕЧАНИЕ: Плазмида, разработанная для нашего описанного нами анализа 2DGE в клетках млекопитающих, должна содержать источник репликации SV40 на несколько кб выше склонных к структуре повторов (рис. 2). Опережающий и запаздывающий синтез следует иметь в виду при выборе ориентации относительно происхождения повторов в плазмиду.

Рисунок 2: Расщепление плазмиды, содержащей повторы, для анализа 2DGE. Склонные к структуре повторы изображены в нескольких кб ниже по течению от правой, движущейся репликационной вилки. При разложении с помощью уникальных резцов 1 и 2 повторяющаяся последовательность будет помещена на нисходящее плечо Y-дуги, при условии, что последовательность находится за пределами середины расщепленного фрагмента. Аббревиатура: 2DGE = Двумерный нейтральный/нейтральный гель-электрофорез. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
1. Трансфекция плазмид в клетки млекопитающих
- Перед трансфекцией засейте 600 000 HEK293T клеток в 10-сантиметровый планшет для культуры тканей. Дайте клеткам восстановиться при температуре 37 °C в течение ночи.
ПРИМЕЧАНИЕ: Для этого эксперимента можно использовать множество клеточных линий, хотя для достижения оптимальных результатов рекомендуется использовать клетки, содержащие метку SV40. ВНИМАНИЕ: HEK293T клетки считаются BSL-2, и все работы по культивированию должны проводиться в шкафу биобезопасности с использованием соответствующей асептической техники и надлежащих СИЗ. - Когда клетки достигнут 60% конфлюенции, трансфектируйте 8 мкг плазмидной ДНК, содержащей повторы, в засеянные клетки с использованием соответствующих реагентов для трансфекции в соответствии с протоколом производителя.
- Если в данный момент времени промежуточные продукты не выделены, отсасывайте старую среду и замените ее 10 мл свежей среды через 24 часа.
- Начните забор клеток через 24-48 ч после трансфекции.
- Аспиратуйте среду и тщательно промойте 10 мл фосфатно-солевого буфера (PBS). Отделите и соберите клетки, используя 0,5 мл трипсина, и уменьшите до 340 × г в течение 4 минут.
- Отсадите надосадочную жидкость и промойте клеточные гранулы PBS. Снова вращайте при 340 × g в течение 4 минут и аспирируйте надосадочную жидкость.
ПРИМЕЧАНИЕ: Здесь эксперимент можно приостановить, заморозив гранулы клеток при -80 °C. Мы нашли наилучшее разрешение, изолируя промежуточные продукты репликации через 48 ч после трансфекции; Тем не менее, 24 часа дали жизнеспособные результаты.
2. Выделение промежуточных продуктов репликации
- Ресуспендируйте клетки в 1,5 мл модифицированного буфера для лизиса Хирта [10 мМ трис-HCl (pH 7,5), 10 мМ этилендиаминтетрауксусной кислоты (ЭДТА)] в конических пробирках объемом 50 мл и начните лизис клеток.
- Добавьте додецилсульфат натрия (SDS) до конечной концентрации 0,6% (приблизительно 650 мкл исходного 2% SDS) и протеиназу K до конечной концентрации 100 мкг/мл (приблизительно 10 мкл исходной протеиназы 20 мг/мл протеиназы K) для удаления нуклеаз.
- Аккуратно перемешайте с помощью пипетки до однородности и инкубируйте смесь при температуре 37 °C не менее 90 минут.
- Увеличьте концентрацию NaCl до 1 М (примерно 540 мкл исходного 5 М NaCl) и аккуратно перемешайте до однородности. Инкубируйте в течение ночи (18-24 часа) при температуре 4 °C, чтобы обеспечить осаждение клеточного мусора, РНК и белка путем высаживания.
ПРИМЕЧАНИЕ: Смесь будет очень вязкой, поэтому будьте осторожны и терпеливы при хорошем перемешивании. - На следующий день отделите ДНК от клеточного мусора, РНК и белка.
- Центрифугируйте смесь при 29 500 × г в течение 45 мин при 4 °C.
- Перенесите надосадочную жидкость, содержащую ДНК, добавьте один объем фенола:хлороформа:изоамилового спирта 25:24:1 (v/v) и быстро перемешайте до однородности.
ВНИМАНИЕ: Фенол: хлороформ: изоамиловый спирт является опасным материалом и должен обрабатываться с помощью соответствующих СИЗ в вытяжном шкафу для химикатов. - Снова центрифугируйте при 15 000 × г в течение 5 мин при комнатной температуре. Перенесите водный слой в новую коническую трубку.
- Осадите и промойте выделенную ДНК.
- Добавьте один объем чистого изопропанола и инкубируйте при комнатной температуре не менее 5 минут. Вращайте ДНК при 15 000 × г в течение 30 минут при 4 °C.
- Сцедите надосадочную жидкость и промойте гранулу холодным 70% этанолом, чтобы удалить излишки соли.
- Снова открутите при 15 000 × г в течение 30 минут при 4 °C, высушите на воздухе и осторожно ресуспендируйте гранулу в буфере Tris-EDTA (TE) (10 мМ Tris-HCl, 1 mM EDTA).
ПРИМЕЧАНИЕ: Здесь эксперимент может быть приостановлен, а образцы могут быть заморожены при температуре -20 °C; тем не менее, следует избегать циклов замораживания/размораживания, так как это может снизить качество промежуточных продуктов репликации ДНК.
3. Пробоподготовка и 2-мерный гель-электрофорез
- Расщепляйте выделенные промежуточные продукты репликации плазмид.
- Добавьте в образец 100 единиц соответствующих ферментов рестрикции для расщепления плазмидной ДНК, в частности, разместите последовательность, содержащую повтор, на исходной дистальной половине линейного фрагмента (рис. 2). Кроме того, добавьте DpnI, чтобы разрезать метилированную ДНК, тем самым удаляя любую плазмидную ДНК, которая не была полностью реплицирована в культивируемых клетках человека.
ПРИМЕЧАНИЕ: Для достижения наилучших результатов ферменты рестрикции должны быть уникальными резаками, которые дают фрагмент размером 3-5 кб, который размещает последовательность, подверженную структуре, на нисходящем плече Y-дуги. - Инкубируйте образцы при 37 °C в течение 6-10 часов, чтобы обеспечить полное плазмадное разложение.
- Осадите ДНК либо 2,5 объемами холодного чистого этанола и инкубируйте при температуре -20 °C в течение ночи, либо добавьте один объем изопропанола и инкубируйте при комнатной температуре в течение 5 минут.
- Центрифугируйте сброженные и осажденные образцы при давлении 15 000 × г в течение 30 минут при 4 °C.
- Сцедите надосадочную жидкость и промойте образец холодным 70% этанолом. Снова вращайте при 15 000 × g в течение 30 минут при температуре 4 °C.
- Сцедите надосадочную жидкость, высушите на воздухе в течение 10 минут и повторно суспендируйте образцы в 15 мкл TE-буфера.
- Добавьте в образец 100 единиц соответствующих ферментов рестрикции для расщепления плазмидной ДНК, в частности, разместите последовательность, содержащую повтор, на исходной дистальной половине линейного фрагмента (рис. 2). Кроме того, добавьте DpnI, чтобы разрезать метилированную ДНК, тем самым удаляя любую плазмидную ДНК, которая не была полностью реплицирована в культивируемых клетках человека.
- Приготовьте агарозный гель первого размера в концентрации 0,4-0,5% в 1x Трис-борат-ЭДТА (ТБЭ) (89 мМ трис-основание, 89 мМ борная кислота, 2 мМ ЭДТА). Дайте раствору застыть не менее 1 часа.
- Начните загрузку образцов в первое измерение.
- Загрузите лестницу в пределах первых 3 см относительно крайнего левого края геля. Затем загрузите полностью подготовленные образцы, обеспечивая расстояние 3 см между каждой парой.
- Запустите гель в 1x TBE в течение 19-24 часов при 0,85 В/см, чтобы разделить промежуточные продукты по их размеру. Убедитесь, что камера закрыта, чтобы защитить образцы от света, который может привести к повреждению ДНК.
- На следующий день извлеките гель из буфера и оцените расположение расваренного линейного фрагмента с помощью линейки.
- Удалите первые 3 см геля, содержащего лестницу, и окрасьте сегмент геля в 1x TBE, содержащий 0,3 мкг/мл бромида этидия, на 10-15 минут. Визуализируйте лестницу с помощью системы документирования геля.
- Прибавьте 1,3 см к предполагаемому местоположению, получив значение a. Затем вычтите 7,5 см из значения a, получив значение b. Выровняйте линейку относительно геля первого измерения и разрежьте горизонтально поперек на значениях a и b. Затем отрежьте вертикально 3 см пространства, отведенного для каждого образца. Визуальная схема приведена на рисунке 3 .
- В новом литейном лотке поверните сегменты по часовой стрелке и разместите их на месте лунок для образцов (рис. 3).
- Приготовьте агарозный гель второго измерения в концентрации 1-1,3% в 1x TBE с концентрацией 0,3 мкг/мл бромида этидия.
- После охлаждения примерно до 55 °C налейте гель второго измерения на вращающиеся сегменты первого размера и дайте ему застыть в течение не менее 1 часа.
- Перенесите второе измерение в камеру с 1x TBE при концентрации бромида этидия 0,3 мкг/мл и дайте гелю сбалансироваться в течение не менее 30 минут.
- Запустите гель, снова накрытый крышкой, в течение 9-10 часов при 4,23 В/см при 4 °C, чтобы отделить промежуточные продукты по их форме.
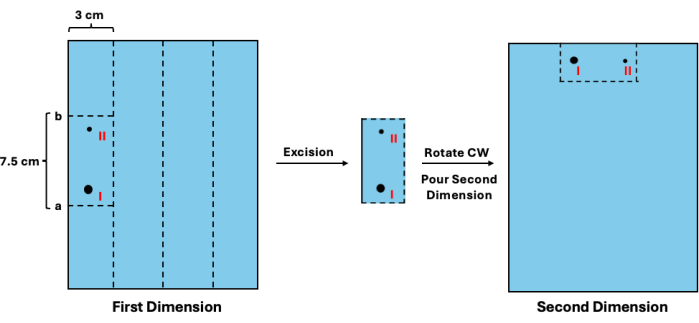
Рисунок 3: Иссечение промежуточных продуктов первого измерения перед разделением второго измерения. После визуализации лестницы можно оценить подвижность невоспроизведенных фрагментов. (I) Это значение затем может быть использовано для определения подходящих участков вырубки (a и b) для его удаления и его реплицированных аналогов (II). Затем секцию геля следует повернуть и поместить в положение лунок для разделения второго измерения. Сокращение: CW = по часовой стрелке. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
4. Саузерн-блоттинг и гибридизация с радиоактивно меченым зондом
- Удалите гель второго измерения из камеры и очистите фрагменты ДНК в течение 10 минут в 0,24 М растворе HCl с легким покачиванием. Смойте гель деионизированной водой и замочите его в 0,4 М NaOH на 10-15 минут.
ВНИМАНИЕ: HCl и NaOH вызывают коррозию и с ними следует обращаться с помощью соответствующих СИЗ в вытяжном шкафу для химических веществ. - Начните сборку Южного блота, чтобы облегчить перенос отделенных промежуточных продуктов из геля на мембрану. Полная схема приведена на рисунке 4 .
- Наполните достаточно большую емкость 1 л 0,4 М NaOH.
- Выровняйте длинный стеклянный лист поперек контейнера и сложите (по длине) два длинных листа хроматографической бумаги перпендикулярно стеклянному листу, расширяясь в контейнер NaOH.
- Намочите верхнюю часть бумаги NaOH и тщательно удалите любые пузырьки воздуха под ее поверхностью.
- Смочите три листа хроматографической бумаги с NaOH и положите их поверх сложенной бумаги, снова удалив любые пузыри.
- Переверните двухмерный гель вверх дном и перенесите его на бумагу.
- Смочите положительно заряженную нейлоновую мембрану (размер пор 0,45 мкм) деионизированной водой и поместите ее поверх геля.
- Наконец, добавьте еще три листа бумаги, смоченных в деионизированной воде, поверх мембраны.
- Накройте все открытые NaOH в нижней емкости полиэтиленовой пленкой, чтобы предотвратить испарение. Положите стопку салфеток или бумажных полотенец на кляксу, следя за тем, чтобы она была высотой 0,3-0,5 м. Поместите сверху груз, сжимая всю кляксу, чтобы облегчить плотное действие капилляров. Подождите не менее 2 дней для переноса ДНК на мембрану.
ПРИМЕЧАНИЕ: Длина и ширина хроматографической бумаги и мембраны зависят от размера агарозного геля, используемого для второго измерения. Для наиболее эффективного переноса используйте бумагу и мембрану с теми же размерами, что и у геля.
- После переноса сшивайте ДНК с мембраной с помощью УФ-сшивающего агента со скоростью 120 мкДж/см2 в течение 1 мин.
ПРИМЕЧАНИЕ: Эксперимент можно приостановить, поместив мембрану в герметичный, сухой и чистый протектор при комнатной температуре. - Промойте мембрану 2x в течение 5 минут 2x солевым цитратом натрия (буфер SSC) (0,3 М NaCl, 0,03 М цитрата натрия).
- Предварительно гибридизируйте мембрану с 0,18 мл/см2 гибридизационного буфера Church & Gilbert [1 мМ ЭДТА, 1% бычьего сывороточного альбумина (БСА), 0,5 М фосфата натрия, 7% SDS] при 65 °С, вращая в гибридизационном инкубаторе не менее 2 ч.
ПРИМЕЧАНИЕ: Мембрана может прегибридизоваться в течение нескольких дней. - Подготовьте меченый радиоактивным зондом с помощью α-32P dATP или dCTP и набора для мечения ДНК в соответствии с протоколом производителя.
ВНИМАНИЕ: ДНТП с радиоактивной маркировкой опасны, поэтому при обращении с ними следует носить соответствующие СИЗ. Все радиоактивные работы должны выполняться за экранированием, а обученные лица должны контролироваться на предмет поглощения радиации с помощью дозиметров.- Сконструируйте линейный фрагмент ДНК размером 400-900.о., комплементарный расщепленной последовательности (рис. 2), и амплифицируйте фрагмент с помощью полимеразной цепной реакции (ПЦР).
ПРИМЕЧАНИЕ: Мы рекомендуем иметь стоковый фрагмент ПЦР 50-100 нг/мкл - Объедините 100 нг комплементарного фрагмента ПЦР с ДНК Pol I, буфером фрагмента Кленова (3' 5' экзо-) и случайными олигонуклеотидами декануклеотидов.
- Денатурируйте фрагмент при 100 °C в течение 10 минут.
- Добавьте в образец 5 единиц ДНК Pol I, фрагмент Кленова (3' 5' экзо-), 50 μCi α-32P dNTP и 30-50 μмоль смеси dNTP с дефицитом радиоактивно меченого типа dNTP.
- Инкубировать при 37 °C в течение 10 мин для полимеризации и включения меченого радиоактивными веществами dNTP.
- Добавьте в образец 30-50 мкм ранее отсутствувшего типа dNTP и инкубируйте при 37 °С в течение 10 мин.
- Очистите меченый фрагмент с помощью спиновой колонки при давлении 3000 × г в течение 2 минут.
- Сконструируйте линейный фрагмент ДНК размером 400-900.о., комплементарный расщепленной последовательности (рис. 2), и амплифицируйте фрагмент с помощью полимеразной цепной реакции (ПЦР).
- Добавьте меченый радиоактивным зондом в 50 мл гибридизационного буфера и инкубируйте с мембраной в течение ночи при температуре 65 °С, вращаясь в гибридизационном инкубаторе.
- На следующий день снимите зонд и промойте мембрану 2x с буфером промывки 1 (0,1x SSC, 0,1% SDS) при 42 °C и 2x с буфером промывки 2 (2x SSC, 0,1% SDS) при 65 °C.
ПРИМЕЧАНИЕ: Все промывки следует выполнять с быстрыми вращениями в течение 15 минут в инкубаторе. - Просушите мембрану в течение 10 минут и поместите ее в тонкую прозрачную защитную пленку. Храните герметичную мембрану в контролируемой, радиационно-стойкой кассете с люминофорно-чувствительным экраном. Дайте мембране подвергнуться воздействию экрана в течение 1-10 дней.
- Визуализируйте результаты с помощью биомолекулярного тепловизора, настроенного на люминофорную визуализацию. Вымойте и при необходимости повторно выдержите.
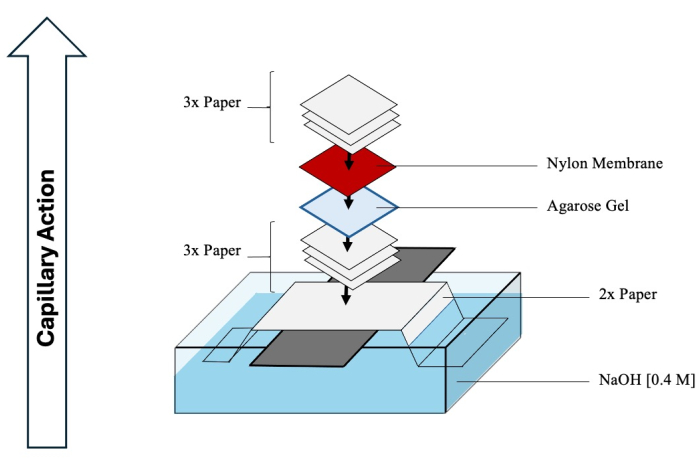
Рисунок 4: Сборка Южного блота. Комплексная схема типового аппарата, используемого для переноса промежуточных продуктов из второго измерения на нейлоновую мембрану. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Access restricted. Please log in or start a trial to view this content.
Результаты
В случае успеха, при визуализации, можно наблюдать острую дугу репликационных вилок, простирающуюся вверх и наружу от массивного пятна 1n (рисунок 5A). Размер фрагмента, или процент репликации, определяет подвижность фрагмента в первом измерении. По мере...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
2DGE обеспечивает полуколичественное и всеобъемлющее изображение относительных популяций промежуточных продуктов, возникающих при репликации той или иной последовательности. Учитывая, что хрупкие молекулярные структуры репликационных вилок должны поддерживаться ?...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
У авторов нет конфликта интересов, который можно было бы раскрыть.
Благодарности
Мы благодарим Хорхе Себриана и Анастасию Растокину, которые начали разрабатывать этот подход в нашей лаборатории, Массимо Лопеса за предоставленную нам плазмиду pML113 и бесценные советы, Илли Доксани за содержательные дискуссии и сотрудников лаборатории Миркина за их поддержку. Работа в лаборатории Миркина поддерживается Национальным институтом общих медицинских наук [R35GM130322] и NSF-BSF [2153071].
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| 10x TBE Buffer | Bio Rad | 1610733 | |
| 20x SSC Buffer | Fisher Scientific | BP1325-1 | |
| 293T cells | ATCC | CRL-3216 | |
| a-32P dATP, 3000 Ci/mmol | Revvity | BLU512H250UC | |
| Agarose | Fisher Scientific | BP160-500 | |
| Amersham Hybond-N+ | Fisher Scientific | RPN303B | |
| BAS Storage Phosphor Screens | Fisher Scientific | 28956482 | |
| Church and Gibert's hybriddization buffer | Fisher Scientific | 50-103-5408 | |
| DecaLabel DNA labeling kit | ThermoFisher Scientific | K0622 | |
| DMEM, high gluctose, GltaMAX Supplement, pyruvate | ThermoFisher Scientific | 10569010 | |
| DpnI | New England Biolabs | R0176S | Additional restriction enzymes will need to be purchased as well |
| EDTA 0.5 M, pH 8 | Fisher Scientific | BP2482500 | |
| Ethanol, 70% | Fisher Scientific | BP82031GAL | |
| Fetal Bovine Serum | VWR | 97068-085 | |
| Hydrochloric acid solution, 12 M | Millipore Sigma | 13-1683 | |
| Isopropanol | Fisher Scientific | BP26184 | |
| jetPRIME DNA and siRNA Transfection Reagent with Buffer | VWR | 101000027 | |
| MycoZap Plus-CL | VWR | 75870-448 | |
| NaCl | Millipore Sigma | 746398-500G | |
| Nalgene Oak Ridge High-Speed Centrifuge Tubes | ThermoFisher Scientific | 3139-0050 | |
| Phosphate Buffer Saline, pH 7.4 | ThermoFisher Scientific | 10010023 | |
| Phosphate Buffer Saline, pH 7.5 | ThermoFisher Scientific | 10010024 | |
| Proteinase K | ThermoFisher Scientific | EO0491 | |
| Proteinase K | ThermoFisher Scientific | EO0492 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-4 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-5 | |
| Ruler | Fisher Scientific | 09-016 | |
| Scalpel | Fisher Scientific | 12-460-451 | |
| Sodium dodecyl sulfate | Millipore Sigma | 436143-25G | |
| Sodium hydroxide | Fisher Scientific | S25548 | |
| Sorval LYNX 4000 Superspeed Centrifuge | ThermoFisher Scientific | 75006580 | |
| Sub-cell Horizontal Electrophoresis System | Bio Rad | 1704401 | |
| TH13-6 x 50 Swinging Bucket Rotor | ThermoFisher Scientific | 75003010 | |
| Tris-HCl 1 M, pH 7.5 | Fisher Scientific | BP1757-500 | |
| Trypsin-EDTA (0.25%), phenol red | ThermoFisher Scientific | 25200056 |
Ссылки
- Liao, X., et al. Repetitive DNA sequence detection and its role in the human genome. Commun Biol. 6 (1), 1-21 (2023).
- Fotsing, S. F., et al. The impact of short tandem repeat variation on gene expression. Nat Genet. 51 (11), 1652-1659 (2019).
- Fan, H., Chu, J. -Y. A brief review of short tandem repeat mutation. GPB. 5 (1), 7-14 (2007).
- Khristich, A. N., Mirkin, S. M. On the wrong DNA track: Molecular mechanisms of repeat-mediated genome instability. J Biol Chem. 295 (13), 4134-4170 (2020).
- Samadashwily, G. M., Raca, G., Mirkin, S. M. Trinucleotide repeats affect DNA replication in vivo. Nat Genet. 17 (3), 298-304 (1997).
- Khristich, A. N., Armenia, J. F., Matera, R. M., Kolchinski, A. A., Mirkin, S. M. Large-scale contractions of Friedreich's ataxia GAA repeats in yeast occur during DNA replication due to their triplex-forming ability. Proc Natl Acad Sci USA. 117 (3), 1628-1637 (2020).
- Shishkin, A. A., et al. Large-scale expansions of Friedreich's ataxia GAA repeats in yeast. Mol Cell. 35 (1), 82-92 (2009).
- Sundararajan, R., Gellon, L., Zunder, R. M., Freudenreich, C. H. Double-strand break repair pathways protect against CAG/CTG repeat expansions, contractions and repeat-mediated chromosomal fragility in Saccharomyces cerevisiae. Genetics. 184 (1), 65-77 (2010).
- Kim, H. -M., et al. Chromosome fragility at GAA tracts in yeast depends on repeat orientation and requires mismatch repair. EMBO J. 27 (21), 2896-2906 (2008).
- Polleys, E. J., House, N. C. M., Freudenreich, C. H. Role of recombination and replication fork restart in repeat instability. DNA Repair. 56, 156-165 (2017).
- Gold, M. A., et al. Restarted replication forks are error-prone and cause CAG repeat expansions and contractions. PLoS Genet. 17 (10), e1009863(2021).
- Lambert, S., et al. Homologous recombination restarts blocked replication forks at the expense of genome rearrangements by template exchange. Mol Cell. 39 (3), 346-359 (2010).
- Burssed, B., Zamariolli, M., Bellucco, F. T., Melaragno, M. I. Mechanisms of structural chromosomal rearrangement formation. Mol Cytogenet. 15 (1), 23(2022).
- Paulson, H. Repeat expansion diseases. Handb Clin Neurol. 147, 105-123 (2018).
- Malik, I., Kelley, C. P., Wang, E., Todd, P. Molecular mechanisms underlying nucleotide repeat expansion disorders. Nat Rev Mol Cell Biol. 22 (9), 589-607 (2021).
- Emanuel, B. S., Zackai, E. H., Medne, L. Emanuel Syndrome. GeneReviews®. Adam, M. P. , University of Washington. Seattle, Seattle, WA. (1993).
- Brewer, B. J., Fangman, W. L. The localization of replication origins on ARS plasmids in S. cerevisiae. Cell. 51 (3), 463-471 (1987).
- Bell, L., Byers, B. Separation of branched from linear DNA by two-dimensional gel electrophoresis. Anal Biochem. 130 (2), 527-535 (1983).
- Voineagu, I., Narayanan, V., Lobachev, K. S., Mirkin, S. M. Replication stalling at unstable inverted repeats: Interplay between DNA hairpins and fork stabilizing proteins. Proc Natl Acad Sci USA. 105 (29), 9936-9941 (2008).
- Nguyen, J. H. G., et al. Differential requirement of Srs2 helicase and Rad51 displacement activities in replication of hairpin-forming CAG/CTG repeats. Nucleic Acids Res. 45 (8), 4519-4531 (2017).
- Krasilnikova, M. M., Mirkin, S. M. Analysis of triplet repeat replication by two-dimensional gel electrophoresis. Trinucleotide Repeat Protocols. Kohwi, Y. , Humana Press. Totowa, NJ. (2004).
- Follonier, C., Oehler, J., Herrador, R., Lopes, M. Friedreich's ataxia-associated GAA repeats induce replication-fork reversal and unusual molecular junctions. Nat Struct Mol Biol. 20 (4), 486-494 (2013).
- Chandok, G. S., Patel, M. P., Mirkin, S. M., Krasilnikova, M. M. Effects of Friedreich's ataxia GAA repeats on DNA replication in mammalian cells. Nucleic Acids Res. 40 (9), 3964-3974 (2012).
- Rastokina, A., et al. Large-scale expansions of Friedreich's ataxia GAA•TTC repeats in an experimental human system: role of DNA replication and prevention by LNA-DNA oligonucleotides and PNA oligomers. Nucleic Acids Res. 51 (16), 8532-8549 (2023).
- Giannattasio, M., et al. Visualization of recombination-mediated damage-bypass by template switching. Nat Struct Mol Biol. 21 (10), 884-892 (2014).
- Hisey, J. A., et al. Pathogenic CANVAS (AAGGG)n repeats stall DNA replication due to the formation of alternative DNA structures. Nucleic Acids Res. 52 (8), 4361-4374 (2024).
- Kalejta, R. F., Lin, H. B., Dijkwel, P. A., Hamlin, J. L. Characterizing replication intermediates in the amplified CHO dihydrofolate reductase domain by two novel gel electrophoretic techniques. Mol Cell Biol. 16 (9), 4923-4931 (1996).
- Little, R. D., Platt, T. H., Schildkraut, C. L. Initiation and termination of DNA replication in human rRNA genes. Mol Cell Biol. 13 (10), 6600-6613 (1993).
- Fanning, E., Zhao, K. SV40 DNA replication: From the A gene to a nanomachine. Virology. 384 (2), 352-359 (2009).
- Sogo, J. M., Stahl, H., Koller, T., Knippers, R. Structure of replicating simian virus 40 minichromosomes: The replication fork, core histone segregation and terminal structures. J Mol Biol. 189 (1), 189-204 (1986).
- Weisshart, K., Taneja, P., Fanning, E. The replication protein A binding site in simian virus 40 (SV40) T antigen and its role in the initial steps of SV40 DNA replication. J Virol. 72 (12), 9771-9781 (1998).
- Sowd, G. A., Fanning, E. A Wolf in sheep's clothing: SV40 co-opts host genome maintenance proteins to replicate viral DNA. PLoS Pathog. 8 (11), e1002994(2012).
- National Library of Medicine: National Center for Biotechnology Information. BLAST: Basic Local Alignment Search Tool. , https://blast.ncbi.nlm.nih.gov/Blast.cgi (2024).
- Hirt, B. Selective extraction of polyoma DNA from infected mouse cell cultures. J Mol Biol. 26 (2), 365-369 (1967).
- Lopes, M., Foiani, M., Sogo, J. M. Multiple mechanisms control chromosome integrity after replication fork uncoupling and restart at irreparable UV lesions. Mol Cell. 21 (1), 15-27 (2006).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены