Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Elektrophoretische Analyse der Replikation durch strukturanfällige DNA-Wiederholungen innerhalb des SV40-basierten humanen Episoms
In diesem Artikel
Zusammenfassung
Im Folgenden wird das Verfahren zur Analyse des Replikationsverlaufs durch pathogene, strukturanfällige Wiederholungen mittels 2-dimensionaler Gelelektrophorese skizziert.
Zusammenfassung
Die zweidimensionale Neutral/Neutral-Gelelektrophorese (2DGE) hat sich als Benchmark-Technik zur Analyse der DNA-Replikation durch natürliche Impedimente herauskristallisiert. Dieses Protokoll beschreibt, wie die Progression der Replikationsgabel durch strukturanfällige, expandierbare DNA-Wiederholungen innerhalb des auf dem Affenvirus 40 (SV40) basierenden Episoms in menschlichen Zellen analysiert werden kann. Kurz gesagt, bei der Plasmidtransfektion in menschliche Zellen werden Replikationszwischenprodukte durch das modifizierte Hirt-Protokoll isoliert und mit dem DpnI-Restriktionsenzym behandelt, um nicht-replizierte DNA zu entfernen. Zwischenprodukte werden dann von geeigneten Restriktionsenzymen verdaut, um die Wiederholung des Interesses in der ursprungsdistalen Hälfte eines 3-5 kb langen DNA-Fragments zu platzieren. Die Replikationszwischenprodukte werden in zwei senkrechte Dimensionen unterteilt, zunächst nach Größe und dann nach Form. Nach der Southern-Blot-Hybridisierung ermöglicht dieser Ansatz den Forschern, das Abwürgen der Gabel bei verschiedenen strukturbildenden Wiederholungen in der absteigenden Hälfte des Replikations-Y-Bogens zu beobachten. Darüber hinaus ermöglicht diese Positionierung des Strömungsabrisses die Visualisierung verschiedener Ergebnisse des wiederholt vermittelten Gabelabwürgens, wie z. B. die Gabelumkehr, das Aufkommen einer konvergierenden Gabel und den rekombinatorischen Gabelneustart.
Einleitung
Short Tandem Repeats (STR) sind kleine, typischerweise 2-9 Basenpaare (bp), sich wiederholende DNA-Sequenzen, die etwa 3 % des menschlichen Genoms ausmachen1. STR spielen eine wichtige Rolle bei der Genregulation2; ihre repetitive Zusammensetzung macht sie jedoch anfällig für die Bildung nicht-kanonischer DNA-Sekundärstrukturen und die anschließende genetische Instabilität 3,4. Von linkshändigen Helices über Haarnadeln/Kreuzformen bis hin zu drei- und viersträngigen Helices stellen diese alternativen DNA-Strukturen intrinsische Herausforderungen für das Replisom dar. Eine natürliche Voraussetzung für die Bildung von Sekundärstrukturen ist die DNA-Abwicklung, die eine Voraussetzung für die DNA-Replikation ist. Dies stellt ein einzigartiges Rätsel für die Funktion des Genoms dar, da sich viele dieser Strukturen während der Replikation bilden können, was das Fortschreiten des Replizosoms behindert und letztendlich zum Stillstand der Replikationsgabelführt 5,6,7 oder in schweren Fällen zum Kollaps der Gabel und zum DNA-Bruch 8,9. Es wurde gezeigt, dass sowohl der Neustart von blockierten Gabeln als auch DNA-Reparaturwege zu wiederholter Instabilität führen, wie z. B. wiederholte Expansionen10,11 und komplexe Genomumlagerungen (CGR)12,13. Diese Ereignisse können zur Entwicklung von etwa 60 menschlichen Krankheiten führen, die als wiederholte Expansionsstörungen bekannt sind, darunter das Fragile-X-Syndrom, die Huntington-Krankheit, die Friedreich-Ataxie und andere14,15 sowie CGR-Erkrankungen wie das Emmanuel-Syndrom16. Um die Mechanismen menschlicher Krankheiten, die durch Wiederholungsinstabilität verursacht werden, besser zu verstehen, ist es daher unerlässlich, die Details der Progression der Replikationsgabel durch diese Wiederholungen zu untersuchen.
Mitte der 1980er Jahre entstand eine Technik zur Untersuchung des Replikationsverlaufs, als Brewer und Fangman versuchten, einen direkten Beweis dafür zu erbringen, dass die Replikationsinitiierung bei Saccharomyces cerevisiae an Elementen der autonomen Replikationssequenz (allgemein bekannt als ARS) stattfindet17. Dabei trennten sie die Strukturen von Hefereplikationszwischenprodukten in Agarose und adaptierten damit eine frühere Methode von Bell und Byers, die als 2-dimensionale Neutral/Neutral-Gelelektrophorese (2DGE)18 bekannt ist. Diese Technik nutzte die Tatsache, dass sich nichtlineare DNA in Agarosegel anders bewegt als ihr lineares Äquivalent der gleichen Masse. Genauer gesagt wird bei der 2DGE die isolierte DNA in zwei senkrechte Dimensionen getrennt, zunächst primär nach Größe und dann primär nach Form, um eine umfassende Karte der Replikation in einer bestimmten Region von Interesse zu erstellen. In ihrer ursprünglichen Arbeit zeigten Brewer und Fangman dies als einen Bogen, der aus "einfachen Y"-Strukturen oder Replikationsgabeln besteht, die nicht replizierte DNA mit ihren replizierten Gegenstücken verbinden. Sie beschreiben andere beobachtete Zwischenprodukte als "Blasen" und "doppelte Ys", die jeweils Replikationsursprünge und konvergierende Gabeln darstellen.
2DGE kann verwendet werden, um die relativen Populationen von DNA-Replikationszwischenprodukten zu einem bestimmten Zeitpunkt zu untersuchen. Wenn also eine Population von Zwischenstufen häufiger vorkommt als eine andere, würde dies bei der Visualisierung offensichtlich sein. Dies macht 2DGE zu einem besonders nützlichen Werkzeug für die Untersuchung des Replikationsfortschritts durch anspruchsvolle Sequenzen, wie z. B. strukturbildende Wiederholungen. Wenn die analysierte Region beispielsweise eine Sequenz enthält, die in der Lage ist, einen Strömungsabriss der Replikationsgabel zu induzieren, würde dies als Ausbuchtung auf dem Bogen dargestellt werden (Abbildung 1A), was auf eine Anhäufung von Replikationsgabeln an diesem Ort hinweist. Dies kann bei der Replikation sowohl haarnadelbildender Wiederholungssequenzen in Hefe 19,20,21 als auch von Triplex-bildenden Wiederholungen in menschlichen Zellen 22,23,24 beobachtet werden. Zusätzlich zum Stalling kann 2DGE verwendet werden, um DNA-Strukturen zu beobachten, die nicht mit den standardmäßigen einfachen Ys übereinstimmen, die während der Replikation gebildet werden, wie im Fall von rekombinanten Zwischenprodukten25. Diese Zwischenstufen haben eine schwerere und stärker verzweigte X-förmige Struktur und bewegen sich daher sowohl in der ersten als auch in der zweiten Dimension langsamer als Standard-Replikationsgabeln. Ähnliche Ergebnisse sind auch in Bezug auf die Replikationsgabelumkehr 20,24,26 zu beobachten. Als Reaktion auf starken Replikationsstress wurde gezeigt, dass eukaryotische Zellen die Umkehrung der Replikationsgabel nutzen, um blockierte Gabeln zu retten. Diese umgekehrten Gabeln haben ein ähnliches Molekulargewicht wie blockierte Gabeln; ihre Hühnerfußstruktur führt jedoch zu einer langsameren elektrophoretischen Beweglichkeit in der zweiten Dimension im Vergleich zu ihren Y-förmigen Komplementen, was zu einer Ausdehnung des Lichtbogens nach oben und außen führt.
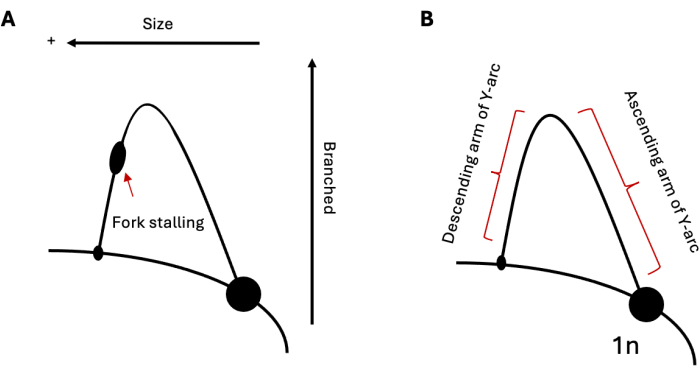
Abbildung 1: 2D-Gelelektrophorese-Analyse der DNA-Replikation. (A) Schematische Darstellung einer typischen 2DGE-Replikation, die die Replikation durch eine strukturbildende Wiederholung darstellt, die in der Lage ist, einen Gabelabriss zu induzieren. Die Zwischengröße und -struktur beeinflussen die elektrophoretische Mobilität. (B) Beispiel Y-Bogen mit aufsteigenden bzw. absteigenden Armen, beschriftet. Abkürzung: 2DGE = Zweidimensionale Neutral-/Neutral-Gel-Elektrophorese. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Einer der wichtigsten Aspekte von 2DGE betrifft natürlich die Qualität und Quantität der Replikationszwischenprodukte. Die Auflösung der 2DGE-Analyse der Replikation durch endogene Loci in Säugetierzellen ist jedoch für eine Einzelkopien-Zielsequenz innerhalb des 6 × 109 bp diploiden menschlichen Genoms unzureichend, obwohl dies für Multi-Copy-Gene wie stark amplifizierten DHFR-Locus27 oder ribosomale RNA28 durchgeführt wurde. Die SV40-basierte Replikation ist ein effizientes und gut charakterisiertes Mittel zur Untersuchung der Replikation in eukaryotischen Zellen29. Es bietet ein zuverlässiges Modell der eukaryotischen Replikation, das den größten Teil der Wirtsreplysomenmaschinerie nutzt, um das virale Genom zu replizieren, das bei der Infektion in Nukleosomen unterteilt wird30,31. Zwei bemerkenswerte Ausnahmen vom Replisom von Säugetieren sind, dass das T-Antigen (Tag) anstelle des CMG-Komplexes des Wirts als replikative DNA-Helikase dient und die DNA-Polymerase delta sowohl führende als auch nachlaufende DNA-Stränge synthetisiert32. Wir haben uns dieses System zunutze gemacht, indem wir pathogene Abschnitte strukturbildender Wiederholungen stromabwärts von einem SV40-Replikationsursprung in ein Plasmid platziert haben, das ursprünglich im Labor22 von Massimo Lopes entwickelt wurde. Wichtig ist, dass dieses Plasmid auch das Gen enthält, das für Tag selbst kodiert, was zu seiner konstitutiven und äußerst potenten Replikation bei der Transfektion in eine Vielzahl von kultivierten menschlichen Zellen führt. Diese Eigenschaft führt zu einer großen Menge von Produkten, die sich ideal für die 2DGE-Analyse der Zwischenprodukte eignen, die während und als Reaktion auf die Replikation pathogener Wiederholungen in menschlichen Zellen gebildet werden. In dieser Arbeit beschreiben wir eine detaillierte Methode zur Visualisierung der Replikation strukturbildender Wiederholungen innerhalb des SV40-basierten humanen Episoms mittels 2-dimensionaler Gelelektrophorese.
Access restricted. Please log in or start a trial to view this content.
Protokoll
HINWEIS: Das Plasmid, das für unsere skizzierte 2DGE-Analyse in Säugetierzellen entwickelt wurde, sollte einen SV40-Replikationsursprung mehrere kb stromaufwärts von strukturanfälligen Wiederholungen enthalten (Abbildung 2). Die führende und nachlaufende Synthese sollte bei der Wahl der Ausrichtung relativ zum Ursprung, in die die Wiederholungen in das Plasmid kloniert werden sollen, berücksichtigt werden.

Abbildung 2: Aufschluss von repeathaltigem Plasmid für die 2DGE-Analyse. Strukturanfällige Wiederholungen werden mehrere KB stromabwärts von der sich nach rechts bewegenden Replikationsgabel dargestellt. Beim Aufschluss mit den einzigartigen Fräsern 1 und 2 wird die Wiederholungssequenz auf dem absteigenden Arm des Y-Bogens platziert, wobei die Sequenz über die Hälfte des verdauten Fragments hinausgeht. Abkürzung: 2DGE = Zweidimensionale Neutral-/Neutral-Gel-Elektrophorese. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
1. Plasmidtransfektion in Säugetierzellen
- 600.000 HEK293T Zellen vor der Transfektion in einer 10 cm großen Gewebekulturplatte aussäen. Lassen Sie die Zellen über Nacht bei 37 °C sich erholen.
HINWEIS: Für dieses Experiment können viele Zelllinien verwendet werden, obwohl Zellen mit dem SV40-Tag für optimale Ergebnisse empfohlen werden. ACHTUNG: HEK293T Zellen gelten als BSL-2 und alle Kultivierungsarbeiten sollten in einer Biosicherheitswerkbank unter Verwendung einer geeigneten aseptischen Technik und geeigneter PSA durchgeführt werden. - Wenn die Zellen eine Konfluenz von 60 % erreichen, transfizieren Sie 8 μg repeathaltige Plasmid-DNA in die ausgesäten Zellen unter Verwendung geeigneter Transfektionsreagenzien gemäß dem Protokoll des Herstellers.
- Wenn zu diesem Zeitpunkt keine Zwischenprodukte isoliert werden, aspirieren Sie alte Medien und ersetzen Sie sie nach 24 Stunden durch 10 ml frisches Medium.
- Beginnen Sie 24-48 Stunden nach der Transfektion mit der Zellentnahme.
- Aspirieren Sie das Medium und waschen Sie es vorsichtig mit 10 ml phosphatgepufferter Kochsalzlösung (PBS). Lösen und sammeln Sie Zellen mit 0,5 ml Trypsin und schleudern Sie sie 4 Minuten lang bei 340 × g .
- Aspirieren Sie den Überstand und waschen Sie die Zellpellets mit PBS. Nochmals bei 340 × g 4 min schleudern und den Überstand ansaugen.
HINWEIS: Der Versuch kann hier unterbrochen werden, indem die Zellpellets bei -80 °C eingefroren werden. Wir haben die beste Auflösung bei der Isolierung von Replikationszwischenprodukten 48 Stunden nach der Transfektion gefunden; 24 h hat jedoch zu brauchbaren Ergebnissen geführt.
2. Isolierung von Replikationszwischenprodukten
- Resuspendieren Sie die Zellen in 1,5 mL modifiziertem Hirt-Lysepuffer [10 mM Tris-HCl (pH 7,5), 10 mM Ethylendiamintetraessigsäure (EDTA)] in konischen 50 mL-Röhrchen und beginnen Sie mit der Zelllyse.
- Natriumdodecylsulfat (SDS) wird bis zu einer Endkonzentration von 0,6 % (ca. 650 μl Stamm 2 % SDS) und Proteinase K bis zu einer Endkonzentration von 100 μg/ml (ca. 10 μl Stamm 20 mg/ml, Proteinase K) zugegeben, um Nukleasen zu entfernen.
- Vorsichtig durch Pipettieren mischen, bis eine homogene Masse entsteht, und die Mischung mindestens 90 Minuten lang bei 37 °C inkubieren.
- Die NaCl-Konzentration auf 1 M (ca. 540 μl 5 M NaCl) erhöhen und vorsichtig mischen, bis eine homogene Masse entsteht. Über Nacht (18-24 h) bei 4 °C inkubieren, um die Ausfällung von Zelltrümmern, RNA und Proteinen durch Aussalzen zu ermöglichen.
HINWEIS: Die Mischung wird hochviskos sein, also seien Sie vorsichtig und haben Sie Geduld, während Sie gut mischen. - Trennen Sie am nächsten Tag die DNA von Zelltrümmern, RNA und Proteinen.
- Die Mischung bei 29.500 × g für 45 min bei 4 °C zentrifugieren.
- Den DNA-haltigen Überstand wird übertragen, ein Volumen Phenol:Chloroform:Isoamylalkohol 25:24:1 (v/v) zugegeben und kurz gemischt, bis eine homogene Masse entsteht.
ACHTUNG: Phenol:Chloroform:Isoamylalkohol ist ein gefährliches Material und sollte mit geeigneter PSA in einem chemischen Abzug gehandhabt werden. - Erneut bei 15.000 × g für 5 min bei Raumtemperatur zentrifugieren. Übertragen Sie die wässrige Schicht in ein neues konisches Rohr.
- Die isolierte DNA ausfällen und waschen.
- Fügen Sie ein Volumen reines Isopropanol hinzu und inkubieren Sie es mindestens 5 Minuten lang bei Raumtemperatur. Schleudern Sie die DNA bei 15.000 × g für 30 min bei 4 °C herunter.
- Dekantieren Sie den Überstand und waschen Sie das Pellet mit kaltem 70%igem Ethanol, um überschüssiges Salz zu entfernen.
- Nochmals bei 15.000 × g für 30 min bei 4 °C schleudern, an der Luft trocknen lassen und das Pellet vorsichtig wieder in Tris-EDTA (TE)-Puffer (10 mM Tris-HCl, 1 mM EDTA) resuspendieren.
HINWEIS: Hier kann das Experiment pausiert werden, und die Proben können bei -20 °C eingefroren werden. Frost-Tau-Zyklen sollten jedoch vermieden werden, da dies die Qualität der DNA-Replikationszwischenprodukte beeinträchtigen kann.
3. Probenvorbereitung und 2-dimensionale Gelelektrophorese
- Verdauen Sie die isolierten Zwischenprodukte der Plasmidreplikation.
- Fügen Sie der Probe 100 Einheiten der entsprechenden Restriktionsenzyme hinzu, um die Plasmid-DNA zu verdauen, und platzieren Sie die repeathaltige Sequenz spezifisch auf der ursprungsdistalen Hälfte des linearen Fragments (Abbildung 2). Fügen Sie außerdem DpnI hinzu, um die methylierte DNA zu schneiden, wodurch alle Plasmid-DNA entfernt wird, die in kultivierten menschlichen Zellen nicht vollständig repliziert wurde.
HINWEIS: Um optimale Ergebnisse zu erzielen, sollten Restriktionsenzyme einzigartige Cutter sein, die ein 3-5 kb großes Fragment liefern, das die strukturanfällige Sequenz auf dem absteigenden Arm des Y-Bogens platziert. - Die Proben werden 6-10 Stunden lang bei 37 °C inkubiert, um einen vollständigen Plasmidaufschluss zu ermöglichen.
- Die DNA wird entweder mit 2,5 Volumen kaltem reinem Ethanol ausgefällt und über Nacht bei -20 °C inkubiert, oder es wird ein Volumen Isopropanol hinzugefügt und 5 Minuten lang bei Raumtemperatur inkubiert.
- Die aufgeschlossenen und gefällten Proben werden bei 15.000 × g für 30 min bei 4 °C zentrifugiert.
- Dekantieren Sie den Überstand und waschen Sie die Probe mit kaltem 70%igem Ethanol. Nochmals bei 15.000 × g für 30 min bei 4 °C schleudern.
- Dekantieren Sie den Überstand, trocknen Sie ihn 10 Minuten lang an der Luft und resuspendieren Sie die Proben in 15 μl TE-Puffer.
- Fügen Sie der Probe 100 Einheiten der entsprechenden Restriktionsenzyme hinzu, um die Plasmid-DNA zu verdauen, und platzieren Sie die repeathaltige Sequenz spezifisch auf der ursprungsdistalen Hälfte des linearen Fragments (Abbildung 2). Fügen Sie außerdem DpnI hinzu, um die methylierte DNA zu schneiden, wodurch alle Plasmid-DNA entfernt wird, die in kultivierten menschlichen Zellen nicht vollständig repliziert wurde.
- Bereiten Sie das Agarose-Gel der ersten Dimension mit 0,4-0,5% in 1x Tris-Borat-EDTA (TBE) (89 mM Trisbase, 89 mM Borsäure, 2 mM EDTA) vor. Lassen Sie die Lösung mindestens 1 h lang erstarren.
- Beginnen Sie mit dem Laden der Proben in die erste Dimension.
- Laden Sie die Leiter innerhalb der ersten 3 cm relativ zum linken Rand des Gels. Laden Sie dann die Gesamtheit der vorbereiteten Proben und stellen Sie sicher, dass zwischen jedem Paar ein Abstand von 3 cm besteht.
- Lassen Sie das Gel in 1x FSME für 19-24 h bei 0,85 V/cm laufen, um die Zwischenprodukte in Bezug auf ihre Größe zu trennen. Stellen Sie sicher, dass die Kammer abgedeckt ist, um die Proben vor Licht zu schützen, das die DNA beschädigen kann.
- Nehmen Sie am nächsten Tag das Gel aus dem Puffer und schätzen Sie die Position des verdauten linearen Fragments mit einem Lineal.
- Die ersten 3 cm des Gels, das die Leiter enthält, herausschneiden und das Gelsegment in 1x FSME mit 0,3 μg/ml Ethidiumbromid für 10-15 min färben. Visualisieren Sie die Leiter mit Hilfe eines Gel-Dokumentationssystems.
- Addieren Sie 1,3 cm zum geschätzten Standort, um den Wert a zu erhalten. Subtrahieren Sie dann 7,5 cm von Wert a, um Wert b zu erhalten. Richten Sie das Lineal gegen das Gel der ersten Dimension aus und schneiden Sie horizontal bei den Werten a und b durch. Schneiden Sie dann den für jede Probe reservierten Abstand von 3 cm vertikal ab. In Abbildung 3 finden Sie ein visuelles Schema.
- Drehen Sie die Segmente in einer neuen Gießschale im Uhrzeigersinn und platzieren Sie sie an der Position der Probenvertiefungen (Abbildung 3).
- Bereiten Sie das zweitdimensionale Agarosegel in einer Konzentration von 1-1,3% in 1x FSME bei 0,3 μg/mL Ethidiumbromid vor.
- Nach dem Abkühlen auf ca. 55 °C wird das Gel der zweiten Dimension über die gedrehten Segmente der ersten Dimension gegossen und mindestens 1 h lang fest werden gelassen.
- Die zweite Dimension wird in eine Kammer mit 1x FSME bei 0,3 μg/ml Ethidiumbromid überführt und das Gel mindestens 30 Minuten lang äquilibriert.
- Lassen Sie das Gel, wieder abgedeckt, 9-10 h bei 4,23 V/cm bei 4 °C laufen, um die Zwischenprodukte in Bezug auf ihre Form zu trennen.
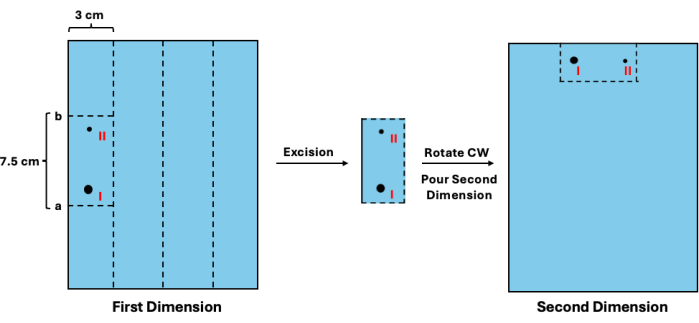
Abbildung 3: Exzision von Zwischenprodukten der ersten Dimension vor der Trennung der zweiten Dimension. Nach der Visualisierung der Leiter kann die Beweglichkeit von nicht replizierten Fragmenten abgeschätzt werden. (I) Dieser Wert kann dann verwendet werden, um die geeigneten Schnittstellen (a und b) für die Herausnahme und die replizierten Gegenstücke (II) zu bestimmen. Der Abschnitt des Gels sollte dann gedreht und in die Position von Vertiefungen für die zweitdimensionale Trennung gebracht werden. Abkürzung: CW = im Uhrzeigersinn. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
4. Southern-Blot und Hybridisierung mit radioaktiv markierter Sonde
- Nehmen Sie das zweitdimensionale Gel aus der Kammer und depurinieren Sie die DNA-Fragmente 10 Minuten lang in einer 0,24 M HCl-Lösung unter leichtem Schaukeln. Spülen Sie das Gel mit entionisiertem Wasser ab und weichen Sie es 10-15 Minuten lang in 0,4 M NaOH ein.
ACHTUNG: HCl und NaOH sind korrosiv und sollten mit geeigneter PSA in einem chemischen Abzug gehandhabt werden. - Beginnen Sie mit dem Zusammensetzen des Southern-Blots, um die Übertragung der getrennten Zwischenprodukte vom Gel auf die Membran zu erleichtern. In Abbildung 4 finden Sie ein umfassendes Schema.
- Füllen Sie einen ausreichend großen Behälter mit 1 L 0,4 M NaOH.
- Richten Sie eine lange Glasscheibe über den Behälter aus und falten Sie (entlang der Länge) zwei lange Blätter Chromatographiepapier senkrecht über die Glasscheibe, die in den Behälter mit NaOH hineinragen.
- Befeuchten Sie die Oberseite des Papiers mit NaOH und entfernen Sie vorsichtig alle Luftblasen unter der Oberfläche.
- Tränken Sie drei Blatt Chromatographiepapier mit NaOH und legen Sie sie auf das gefaltete Papier, wobei Sie alle Blasen wieder entfernen.
- Drehen Sie das Gel der zweiten Dimension auf den Kopf und übertragen Sie es über die Papiere.
- Befeuchten Sie eine positiv geladene Nylonmembran (0,45 μm Porengröße) mit DI-Wasser und legen Sie diese über das Gel.
- Legen Sie zum Schluss drei weitere Blätter Papier, angefeuchtet mit DI-Wasser, über die Membran.
- Decken Sie freiliegendes NaOH im unteren Behälter mit Plastikfolie ab, um eine Verdunstung zu verhindern. Lege einen Stapel Servietten oder Papiertücher über den Klecks und achte darauf, dass er 0,3-0,5 m hoch ist. Legen Sie ein Gewicht auf die Oberseite und drücken Sie den gesamten Blot zusammen, um eine straffe Kapillarwirkung zu ermöglichen. Warten Sie mindestens 2 Tage, bis die DNA auf die Membran übertragen wird.
HINWEIS: Die Länge und Breite des Chromatographiepapiers und der Membran hängen von der Größe des Agarosegels ab, das für die zweite Dimension verwendet wird. Für eine möglichst effiziente Übertragung verwenden Sie Papier und Membran mit den gleichen Abmessungen wie das Gel.
- Nach dem Transfer wird die DNA mit einem UV-Vernetzer bei 120 μJ/cm2 für 1 min mit der Membran vernetzt.
HINWEIS: Das Experiment kann hier unterbrochen werden, indem die Membran in einen luftdichten, trockenen und sauberen Lakenschutz bei Raumtemperatur gelegt wird. - Waschen Sie die Membran 2x für 5 min mit 2x Kochsalzlösung Natriumcitrat (SSC-Puffer) (0,3 M NaCl, 0,03 M Natriumcitrat).
- Prähybridisieren Sie die Membran mit 0,18 ml/cm2 des Hybridisierungspuffers von Church & Gilbert [1 mM EDTA, 1 % Rinderserumalbumin (BSA), 0,5 M Natriumphosphat, 7 % SDS] bei 65 °C und rotieren Sie mindestens 2 Stunden lang in einem Hybridisierungsinkubator.
HINWEIS: Die Membran kann mehrere Tage lang prähybridisieren. - Bereiten Sie die radioaktiv markierte Sonde mit α-32P, dATP oder dCTP und einem DNA-Markierungskit gemäß dem Protokoll des Herstellers vor.
VORSICHT: Radioaktiv markierte dNTPs sind gefährlich und sollten beim Umgang mit ihnen geeignete PSA getragen werden. Alle radioaktiven Arbeiten sollten hinter einer Abschirmung durchgeführt werden, und geschulte Personen sollten mit Dosimetern auf Strahlenaufnahme überwacht werden.- Entwerfen Sie ein lineares DNA-Fragment mit 400-900 bp, das komplementär zur restriktionsverdauten Sequenz ist (Abbildung 2), und amplifizieren Sie das Fragment mittels Polymerase-Kettenreaktion (PCR).
HINWEIS: Wir empfehlen ein Stamm-PCR-Fragment von 50-100 ng/μl - Kombinieren Sie 100 ng des komplementären PCR-Fragments mit DNA Pol I, Klenow-Fragment (3' 5' Exo-) Puffer und zufälligen Decanukleotid-Oligos.
- Denaturieren Sie das Fragment bei 100 °C für 10 min.
- Geben Sie der Probe 5 Einheiten DNA Pol I, Klenow-Fragment (3' 5' Exo-), 50 μCi α-32P dNTP und 30-50 μmol dNTP-Mix, der im radioaktiv markierten dNTP-Typ defizient ist.
- Inkubieren Sie 10 Minuten lang bei 37 °C, um die Polymerisation und den Einbau von radioaktiv markiertem dNTP zu ermöglichen.
- Geben Sie 30-50 μmol des zuvor fehlenden dNTP-Typs in die Probe und inkubieren Sie 10 Minuten lang bei 37 °C.
- Das radioaktiv markierte Fragment wird mit einer Spin-Säule bei 3.000 × g für 2 Minuten gereinigt.
- Entwerfen Sie ein lineares DNA-Fragment mit 400-900 bp, das komplementär zur restriktionsverdauten Sequenz ist (Abbildung 2), und amplifizieren Sie das Fragment mittels Polymerase-Kettenreaktion (PCR).
- Geben Sie eine radioaktiv markierte Sonde in 50 mL Hybridisierungspuffer und inkubieren Sie die Membran über Nacht bei 65 °C in einem Hybridisierungsinkubator.
- Am Folgetag die Sonde entfernen und die Membran 2x mit Waschpuffer 1 (0,1x SSC, 0,1 % SDS) bei 42 °C und 2x mit Waschpuffer 2 (2x SSC, 0,1 % SDS) bei 65 °C waschen.
HINWEIS: Alle Wäschen sollten mit schnellen Umdrehungen für 15 Minuten im Inkubator durchgeführt werden. - Trocknen Sie die Membran 10 Minuten lang und legen Sie sie in eine dünne, transparente Schutzfolie. Lagern Sie die versiegelte Membran in einer kontrollierten, strahlungsbeständigen Kassette mit einem phosphorempfindlichen Sieb. Lassen Sie die Membran 1-10 Tage lang dem Bildschirm aussetzen.
- Visualisieren Sie die Ergebnisse mit einem biomolekularen Imager, der auf Phosphor-Imaging eingestellt ist. Waschen und bei Bedarf erneut belichten.
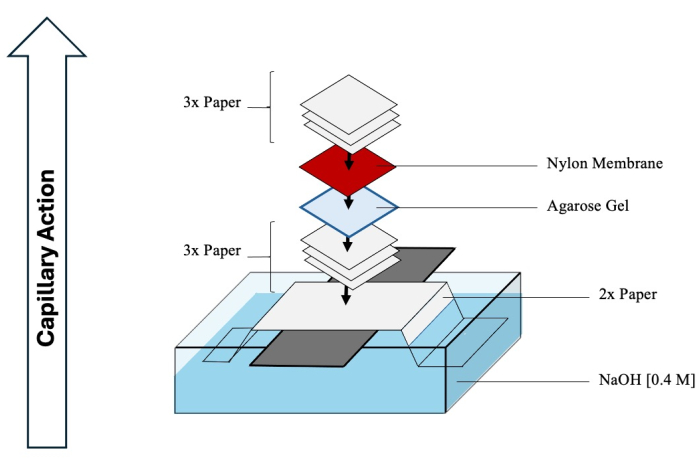
Abbildung 4: Assemblierung des Southern Blot. Umfassendes Schema einer typischen Apparatur, die für den Southern-Blot-Transfer von Zwischenprodukten aus der zweiten Dimension auf eine Nylonmembran verwendet wird. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Bei erfolgreicher Visualisierung kann ein scharfer Bogen von Replikationsgabeln beobachtet werden, der sich vom massiven 1n-Punkt nach oben und außen erstreckt (Abbildung 5A). Die Größe eines Fragments oder der Prozentsatz, der repliziert wird, bestimmt die Mobilität des Fragments in der ersten Dimension. Wenn die Zwischenstufen eine stärker gegliederte Struktur entwickeln, werden sie beginnen, sich langsamer in der zweiten Dimension zu bewegen. Wenn si...
Access restricted. Please log in or start a trial to view this content.
Diskussion
2DGE liefert ein semiquantitatives und umfassendes Bild der relativen Populationen von Zwischenstufen, die während der Replikation einer bestimmten Sequenz entstehen. Da die fragilen molekularen Strukturen der Replikationsgabeln während dieses Verfahrens erhalten bleiben müssen, sollte große Sorgfalt walten gelassen werden, um physikalisches Scheren und chemische Denaturierung zu verhindern. Daher wird dringend empfohlen, während der Plasmidisolierung auf eine alkalische Behandlung ...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Wir danken Jorge Cebrian und Anastasia Rastokina, die mit der Entwicklung dieses Ansatzes in unserem Labor begonnen haben, Massimo Lopes für die Bereitstellung von pML113-Plasmid und unschätzbaren Ratschlägen, Ylli Doksani für aufschlussreiche Diskussionen und Mitgliedern des Mirkin-Labors für ihre Unterstützung. Die Arbeit im Mirkin-Labor wird vom National Institute of General Medical Sciences [R35GM130322] und NSF-BSF [2153071] unterstützt.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 10x TBE Buffer | Bio Rad | 1610733 | |
| 20x SSC Buffer | Fisher Scientific | BP1325-1 | |
| 293T cells | ATCC | CRL-3216 | |
| a-32P dATP, 3000 Ci/mmol | Revvity | BLU512H250UC | |
| Agarose | Fisher Scientific | BP160-500 | |
| Amersham Hybond-N+ | Fisher Scientific | RPN303B | |
| BAS Storage Phosphor Screens | Fisher Scientific | 28956482 | |
| Church and Gibert's hybriddization buffer | Fisher Scientific | 50-103-5408 | |
| DecaLabel DNA labeling kit | ThermoFisher Scientific | K0622 | |
| DMEM, high gluctose, GltaMAX Supplement, pyruvate | ThermoFisher Scientific | 10569010 | |
| DpnI | New England Biolabs | R0176S | Additional restriction enzymes will need to be purchased as well |
| EDTA 0.5 M, pH 8 | Fisher Scientific | BP2482500 | |
| Ethanol, 70% | Fisher Scientific | BP82031GAL | |
| Fetal Bovine Serum | VWR | 97068-085 | |
| Hydrochloric acid solution, 12 M | Millipore Sigma | 13-1683 | |
| Isopropanol | Fisher Scientific | BP26184 | |
| jetPRIME DNA and siRNA Transfection Reagent with Buffer | VWR | 101000027 | |
| MycoZap Plus-CL | VWR | 75870-448 | |
| NaCl | Millipore Sigma | 746398-500G | |
| Nalgene Oak Ridge High-Speed Centrifuge Tubes | ThermoFisher Scientific | 3139-0050 | |
| Phosphate Buffer Saline, pH 7.4 | ThermoFisher Scientific | 10010023 | |
| Phosphate Buffer Saline, pH 7.5 | ThermoFisher Scientific | 10010024 | |
| Proteinase K | ThermoFisher Scientific | EO0491 | |
| Proteinase K | ThermoFisher Scientific | EO0492 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-4 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-5 | |
| Ruler | Fisher Scientific | 09-016 | |
| Scalpel | Fisher Scientific | 12-460-451 | |
| Sodium dodecyl sulfate | Millipore Sigma | 436143-25G | |
| Sodium hydroxide | Fisher Scientific | S25548 | |
| Sorval LYNX 4000 Superspeed Centrifuge | ThermoFisher Scientific | 75006580 | |
| Sub-cell Horizontal Electrophoresis System | Bio Rad | 1704401 | |
| TH13-6 x 50 Swinging Bucket Rotor | ThermoFisher Scientific | 75003010 | |
| Tris-HCl 1 M, pH 7.5 | Fisher Scientific | BP1757-500 | |
| Trypsin-EDTA (0.25%), phenol red | ThermoFisher Scientific | 25200056 |
Referenzen
- Liao, X., et al. Repetitive DNA sequence detection and its role in the human genome. Commun Biol. 6 (1), 1-21 (2023).
- Fotsing, S. F., et al. The impact of short tandem repeat variation on gene expression. Nat Genet. 51 (11), 1652-1659 (2019).
- Fan, H., Chu, J. -Y. A brief review of short tandem repeat mutation. GPB. 5 (1), 7-14 (2007).
- Khristich, A. N., Mirkin, S. M. On the wrong DNA track: Molecular mechanisms of repeat-mediated genome instability. J Biol Chem. 295 (13), 4134-4170 (2020).
- Samadashwily, G. M., Raca, G., Mirkin, S. M. Trinucleotide repeats affect DNA replication in vivo. Nat Genet. 17 (3), 298-304 (1997).
- Khristich, A. N., Armenia, J. F., Matera, R. M., Kolchinski, A. A., Mirkin, S. M. Large-scale contractions of Friedreich's ataxia GAA repeats in yeast occur during DNA replication due to their triplex-forming ability. Proc Natl Acad Sci USA. 117 (3), 1628-1637 (2020).
- Shishkin, A. A., et al. Large-scale expansions of Friedreich's ataxia GAA repeats in yeast. Mol Cell. 35 (1), 82-92 (2009).
- Sundararajan, R., Gellon, L., Zunder, R. M., Freudenreich, C. H. Double-strand break repair pathways protect against CAG/CTG repeat expansions, contractions and repeat-mediated chromosomal fragility in Saccharomyces cerevisiae. Genetics. 184 (1), 65-77 (2010).
- Kim, H. -M., et al. Chromosome fragility at GAA tracts in yeast depends on repeat orientation and requires mismatch repair. EMBO J. 27 (21), 2896-2906 (2008).
- Polleys, E. J., House, N. C. M., Freudenreich, C. H. Role of recombination and replication fork restart in repeat instability. DNA Repair. 56, 156-165 (2017).
- Gold, M. A., et al. Restarted replication forks are error-prone and cause CAG repeat expansions and contractions. PLoS Genet. 17 (10), e1009863(2021).
- Lambert, S., et al. Homologous recombination restarts blocked replication forks at the expense of genome rearrangements by template exchange. Mol Cell. 39 (3), 346-359 (2010).
- Burssed, B., Zamariolli, M., Bellucco, F. T., Melaragno, M. I. Mechanisms of structural chromosomal rearrangement formation. Mol Cytogenet. 15 (1), 23(2022).
- Paulson, H. Repeat expansion diseases. Handb Clin Neurol. 147, 105-123 (2018).
- Malik, I., Kelley, C. P., Wang, E., Todd, P. Molecular mechanisms underlying nucleotide repeat expansion disorders. Nat Rev Mol Cell Biol. 22 (9), 589-607 (2021).
- Emanuel, B. S., Zackai, E. H., Medne, L. Emanuel Syndrome. GeneReviews®. Adam, M. P. , University of Washington. Seattle, Seattle, WA. (1993).
- Brewer, B. J., Fangman, W. L. The localization of replication origins on ARS plasmids in S. cerevisiae. Cell. 51 (3), 463-471 (1987).
- Bell, L., Byers, B. Separation of branched from linear DNA by two-dimensional gel electrophoresis. Anal Biochem. 130 (2), 527-535 (1983).
- Voineagu, I., Narayanan, V., Lobachev, K. S., Mirkin, S. M. Replication stalling at unstable inverted repeats: Interplay between DNA hairpins and fork stabilizing proteins. Proc Natl Acad Sci USA. 105 (29), 9936-9941 (2008).
- Nguyen, J. H. G., et al. Differential requirement of Srs2 helicase and Rad51 displacement activities in replication of hairpin-forming CAG/CTG repeats. Nucleic Acids Res. 45 (8), 4519-4531 (2017).
- Krasilnikova, M. M., Mirkin, S. M. Analysis of triplet repeat replication by two-dimensional gel electrophoresis. Trinucleotide Repeat Protocols. Kohwi, Y. , Humana Press. Totowa, NJ. (2004).
- Follonier, C., Oehler, J., Herrador, R., Lopes, M. Friedreich's ataxia-associated GAA repeats induce replication-fork reversal and unusual molecular junctions. Nat Struct Mol Biol. 20 (4), 486-494 (2013).
- Chandok, G. S., Patel, M. P., Mirkin, S. M., Krasilnikova, M. M. Effects of Friedreich's ataxia GAA repeats on DNA replication in mammalian cells. Nucleic Acids Res. 40 (9), 3964-3974 (2012).
- Rastokina, A., et al. Large-scale expansions of Friedreich's ataxia GAA•TTC repeats in an experimental human system: role of DNA replication and prevention by LNA-DNA oligonucleotides and PNA oligomers. Nucleic Acids Res. 51 (16), 8532-8549 (2023).
- Giannattasio, M., et al. Visualization of recombination-mediated damage-bypass by template switching. Nat Struct Mol Biol. 21 (10), 884-892 (2014).
- Hisey, J. A., et al. Pathogenic CANVAS (AAGGG)n repeats stall DNA replication due to the formation of alternative DNA structures. Nucleic Acids Res. 52 (8), 4361-4374 (2024).
- Kalejta, R. F., Lin, H. B., Dijkwel, P. A., Hamlin, J. L. Characterizing replication intermediates in the amplified CHO dihydrofolate reductase domain by two novel gel electrophoretic techniques. Mol Cell Biol. 16 (9), 4923-4931 (1996).
- Little, R. D., Platt, T. H., Schildkraut, C. L. Initiation and termination of DNA replication in human rRNA genes. Mol Cell Biol. 13 (10), 6600-6613 (1993).
- Fanning, E., Zhao, K. SV40 DNA replication: From the A gene to a nanomachine. Virology. 384 (2), 352-359 (2009).
- Sogo, J. M., Stahl, H., Koller, T., Knippers, R. Structure of replicating simian virus 40 minichromosomes: The replication fork, core histone segregation and terminal structures. J Mol Biol. 189 (1), 189-204 (1986).
- Weisshart, K., Taneja, P., Fanning, E. The replication protein A binding site in simian virus 40 (SV40) T antigen and its role in the initial steps of SV40 DNA replication. J Virol. 72 (12), 9771-9781 (1998).
- Sowd, G. A., Fanning, E. A Wolf in sheep's clothing: SV40 co-opts host genome maintenance proteins to replicate viral DNA. PLoS Pathog. 8 (11), e1002994(2012).
- National Library of Medicine: National Center for Biotechnology Information. BLAST: Basic Local Alignment Search Tool. , https://blast.ncbi.nlm.nih.gov/Blast.cgi (2024).
- Hirt, B. Selective extraction of polyoma DNA from infected mouse cell cultures. J Mol Biol. 26 (2), 365-369 (1967).
- Lopes, M., Foiani, M., Sogo, J. M. Multiple mechanisms control chromosome integrity after replication fork uncoupling and restart at irreparable UV lesions. Mol Cell. 21 (1), 15-27 (2006).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten