È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Analisi elettroforetica della replicazione attraverso ripetizioni di DNA a struttura inclini all'episoma umano basato su SV40
In questo articolo
Riepilogo
Qui, descriviamo la procedura per analizzare la progressione della replicazione attraverso ripetizioni patogene e inclini alla struttura utilizzando l'elettroforesi su gel bidimensionale.
Abstract
La gel-elettroforesi bidimensionale neutra/neutra (2DGE) è emersa come tecnica di riferimento per analizzare la replicazione del DNA attraverso impedimenti naturali. Questo protocollo descrive come analizzare la progressione della forcella di replicazione attraverso ripetizioni di DNA espandibili e soggette a struttura all'interno dell'episoma basato sul virus delle scimmie 40 (SV40) nelle cellule umane. In breve, dopo la trasfezione del plasmide nelle cellule umane, gli intermedi di replicazione vengono isolati mediante il protocollo Hirt modificato e trattati con l'enzima di restrizione DpnI per rimuovere il DNA non replicato. Gli intermedi vengono quindi digeriti da appropriati enzimi di restrizione per collocare la ripetizione di interesse all'interno della metà distale di origine di un frammento di DNA lungo 3-5 kb. Gli intermedi di replicazione sono separati in due dimensioni perpendicolari, prima per dimensione e poi per forma. Dopo l'ibridazione Southern blot, questo approccio consente ai ricercatori di osservare lo stallo della forcella in varie ripetizioni di formazione della struttura sulla metà discendente dell'arco Y di replicazione. Inoltre, questo posizionamento del sito di stallo consente la visualizzazione di vari risultati dello stallo della forcella mediato dalla ripetizione, come l'inversione della forcella, l'avvento di una forcella convergente e il riavvio della forcella ricombinatrice.
Introduzione
Le brevi ripetizioni tandem (STR) sono piccole sequenze ripetitive di DNA che costituiscono circa il 3% del genoma umano1. Gli STR svolgono un ruolo importante nella regolazione genica2; tuttavia, la loro composizione ripetitiva li rende inclini alla formazione di strutture secondarie del DNA non canoniche e alla conseguente instabilità genetica 3,4. Dalle eliche sinistrorse alle forcine/cruciformi, alle eliche a tre e quattro filamenti, queste strutture alternative del DNA causano sfide intrinseche per il replosoma. Un prerequisito naturale per la formazione della struttura secondaria è lo svolgimento del DNA, che è un prerequisito per la replicazione del DNA. Ciò rappresenta un enigma unico per il funzionamento del genoma poiché molte di queste strutture possono formarsi durante la replicazione, ostacolando la progressione del reposoma e, infine, causando lo stallo della forcelladi replicazione 5,6,7 o, nei casi più gravi, il collasso della forcella e la rottura del DNA 8,9. È stato dimostrato che sia il riavvio delle forcelle in stallo che le vie di riparazione del DNA portano a instabilità ripetuta, come espansioni ripetute10,11 e riarrangiamenti genomici complessi (CGR)12,13. Questi eventi possono portare allo sviluppo di circa 60 malattie umane note come disturbi dell'espansione ripetuta, tra cui la sindrome dell'X fragile, la malattia di Huntington, l'atassia di Friedreich e altre14,15, nonché malattie CGR, come la sindrome di Emmanuel16. Pertanto, per comprendere meglio i meccanismi della malattia umana guidata dall'instabilità delle ripetizioni, è imperativo studiare i dettagli della progressione della forcella di replicazione attraverso tali ripetizioni.
Una tecnica per studiare la progressione della replicazione è emersa a metà degli anni '80, quando Brewer e Fangman hanno cercato di fornire prove dirette che l'inizio della replicazione in Saccharomyces cerevisiae avviene a livello di elementi di sequenza di replicazione autonoma (comunemente noti come ARS)17. In tal modo, hanno separato le strutture degli intermedi di replicazione del lievito nell'agarosio, adattando un metodo precedente di Bell e Byers noto come elettroforesi su gel neutro/neutro bidimensionale (2DGE)18. Questa tecnica ha sfruttato il fatto che il DNA non lineare viaggia in modo diverso nel gel di agarosio rispetto al suo equivalente lineare della stessa massa. Più specificamente, in 2DGE, il DNA isolato viene separato in due dimensioni perpendicolari, prima principalmente per dimensione e poi principalmente per forma, per creare una mappa completa della replicazione in una particolare regione di interesse. Nel loro articolo originale, Brewer e Fangman hanno dimostrato che questo è un arco composto da strutture a "Y semplici" o forcelle di replicazione che collegano il DNA non replicato alle loro controparti replicate. Descrivono inoltre altri intermedi osservati come "bolle" e "doppia Y", che rappresentano rispettivamente le origini di replicazione e le forcelle convergenti.
Il 2DGE può essere utilizzato per studiare le popolazioni relative di intermedi di replicazione del DNA in un dato momento. Pertanto, se una popolazione di intermedi è più prevalente di un'altra, ciò sarebbe evidente alla visualizzazione. Ciò rende 2DGE uno strumento particolarmente utile per studiare la progressione della replicazione attraverso sequenze impegnative, come le ripetizioni di formazione della struttura. Ad esempio, se la regione analizzata contiene una sequenza in grado di indurre lo stallo della forcella di replicazione, questo si presenterebbe come un rigonfiamento sull'arco (Figura 1A), indicando un accumulo di forcelle di replicazione in quel locus. Questo può essere visto con la replicazione di entrambe le sequenze di ripetizioni formanti forcine nel lievito 19,20,21 e ripetute triplex nelle cellule umane 22,23,24. Oltre allo stalling, il 2DGE può essere utilizzato per osservare strutture di DNA che non sono conformi allo standard di Ys semplice formato durante la replicazione, come nel caso degli intermedi ricombinanti25. Questi intermedi hanno una struttura a forma di X più pesante e ramificata e, quindi, viaggiano più lentamente sia nella prima che nella seconda dimensione rispetto alle forche di replica standard. Risultati simili possono essere osservati anche per quanto riguarda l'inversione della forcella di replicazione 20,24,26. In risposta a un forte stress di replicazione, è stato dimostrato che le cellule eucariotiche utilizzano l'inversione della forcella di replicazione per salvare le forcelle in stallo. Queste forcelle invertite hanno un peso molecolare simile a quelle delle forcelle in stallo; tuttavia, la loro struttura a zampa di pollo si traduce in una mobilità elettroforetica più lenta nella seconda dimensione rispetto ai loro complementi a forma di Y, con conseguente estensione verso l'alto e verso l'esterno dall'arco.
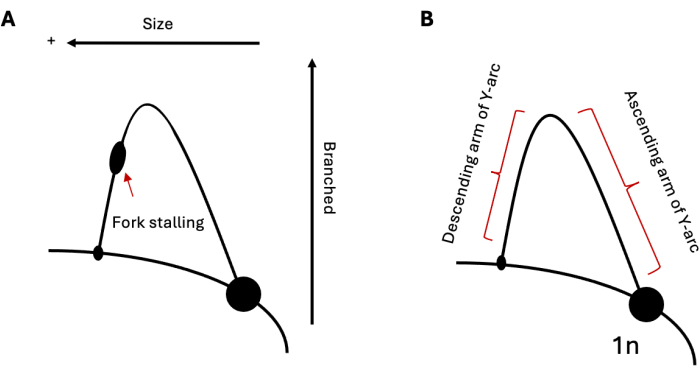
Figura 1: Analisi dell'elettroforesi su gel 2D della replicazione del DNA. (A) Schema di un tipico 2DGE che raffigura la replicazione attraverso una ripetizione di formazione della struttura in grado di indurre lo stallo della forcella. Le dimensioni e la struttura intermedie influenzeranno la mobilità elettroforetica. (B) Esempio di arco Y con bracci ascendenti e discendenti rispettivamente etichettati. Abbreviazione: 2DGE = Elettroforesi su gel bidimensionale neutro/neutro. Clicca qui per visualizzare una versione più grande di questa figura.
Naturalmente, uno degli aspetti più importanti di 2DGE riguarda la qualità e la quantità degli intermedi di replicazione. Tuttavia, la risoluzione dell'analisi 2DGE della replicazione attraverso loci endogeni in cellule di mammifero è insufficiente per una singola sequenza bersaglio di copia all'interno del genoma umano diploide 6 × 109 bp, sebbene sia stata fatta per geni multi-copia, come il locus27 DHFR fortemente amplificato o l'RNA ribosomiale28. La replicazione basata su SV40 è un mezzo efficiente e ben caratterizzato per studiare la replicazione nelle cellule eucariotiche29. Fornisce un modello affidabile di replicazione eucariotica che utilizza la maggior parte del meccanismo di replicazione dell'ospite per replicare il genoma virale, che viene parcellizzato in nucleosomi dopo l'infezione30,31. Due eccezioni degne di nota del replosoma dei mammiferi sono che l'antigene T (Tag), invece del complesso CMG ospite, funge da DNA elicasi replicativa, e la DNA polimerasi delta sintetizza sia i filamenti di DNA principali che quelli ritardati32. Abbiamo sfruttato questo sistema posizionando tratti patogeni di ripetizioni di formazione della struttura a valle di un'origine di replicazione SV40 all'interno di un plasmide che è stato originariamente creato nel laboratorio Massimo Lopes22. È importante sottolineare che questo plasmide contiene anche il gene che codifica per Tag stesso, determinando così la sua replicazione costitutiva ed estremamente potente dopo la trasfezione in una varietà di cellule umane in coltura. Questa caratteristica dà origine ad una grande quantità di prodotti, ideali per l'analisi 2DGE degli intermedi formati durante e in risposta alla replicazione di ripetizioni patogene nelle cellule umane. Qui, descriviamo un metodo dettagliato per visualizzare la replicazione delle ripetizioni che formano la struttura all'interno dell'episoma umano basato su SV40 utilizzando l'elettroforesi su gel bidimensionale.
Access restricted. Please log in or start a trial to view this content.
Protocollo
NOTA: Il plasmide progettato per la nostra analisi 2DGE delineata in cellule di mammifero dovrebbe contenere un'origine di replicazione SV40 diversi kb a monte delle ripetizioni inclini alla struttura (Figura 2). La sintesi leading e lagging dovrebbe essere tenuta a mente quando si sceglie quale orientamento rispetto all'origine le ripetizioni dovrebbero essere clonate nel plasmide.

Figura 2: Digestione di plasmidi contenenti ripetizioni per l'analisi 2DGE. Le ripetizioni soggette a struttura sono rappresentate diversi kb a valle della forcella di replicazione che si muove a destra. La digestione con le taglierine uniche 1 e 2 posizionerà la sequenza ripetuta sul braccio discendente dell'arco Y, data la sequenza oltre il punto di metà del frammento digerito. Abbreviazione: 2DGE = Elettroforesi su gel bidimensionale neutro/neutro. Clicca qui per visualizzare una versione più grande di questa figura.
1. Trasfezione plasmidica in cellule di mammifero
- Seminare 600.000 cellule HEK293T in una piastra di coltura tissutale di 10 cm prima della trasfezione. Lasciare che le celle si riprendano a 37 °C durante la notte.
NOTA: Molte linee cellulari possono essere utilizzate per questo esperimento, anche se si raccomandano celle contenenti il tag SV40 per ottenere risultati ottimali. ATTENZIONE: HEK293T cellule sono considerate BSL-2 e tutto il lavoro di coltura deve essere eseguito in una cabina di biosicurezza utilizzando un'appropriata tecnica asettica e DPI adeguati. - Quando le cellule raggiungono il 60% di confluenza, trasfettare 8 μg di DNA plasmidico contenente ripetizioni nelle cellule seminate utilizzando reagenti di trasfezione appropriati in conformità con il protocollo del produttore.
- Se in questo momento non sono isolanti intermedi, aspirare il vecchio terreno e sostituirlo con 10 ml di terreno fresco dopo 24 ore.
- Iniziare a raccogliere le cellule 24-48 ore dopo la trasfezione.
- Aspirare il terreno e lavare accuratamente con 10 mL di soluzione salina tamponata con fosfato (PBS). Staccare e raccogliere le cellule utilizzando 0,5 mL di tripsina e centrifugare a 340 × g per 4 minuti.
- Aspirare il surnatante e lavare i pellet cellulari con PBS. Centrifugare nuovamente a 340 × g per 4 minuti e aspirare il surnatante.
NOTA: L'esperimento può essere messo in pausa congelando i pellet di celle a -80 °C. Abbiamo trovato la migliore risoluzione isolando gli intermedi di replicazione 48 h dopo la trasfezione; Tuttavia, 24 ore ha prodotto risultati validi.
2. Isolamento degli intermedi di replicazione
- Risospendere le cellule in 1,5 mL di tampone di lisi Hirt modificato [10 mM di tris-HCl (pH 7,5), 10 mM di acido etilendiamminotetraacetico (EDTA)] in provette coniche da 50 mL e iniziare la lisi cellulare.
- Aggiungere il dodecil solfato di sodio (SDS) a una concentrazione finale dello 0,6% (circa 650 μL di SDS al 2%) e la proteinasi K a una concentrazione finale di 100 μg/mL (circa 10 μL di proteinasi K da 20 mg/mL) per rimuovere le nucleasi.
- Miscelare delicatamente pipettando fino a ottenere un composto omogeneo e incubare la miscela a 37 °C per almeno 90 minuti.
- Aumentare la concentrazione di NaCl a 1 M (circa 540 μL di NaCl 5 M) e mescolare delicatamente fino a ottenere un composto omogeneo. Incubare per una notte (18-24 ore) a 4 °C per consentire la precipitazione di detriti cellulari, RNA e proteine mediante salatura.
NOTA: La miscela sarà molto viscosa, quindi fai attenzione e sii paziente mentre mescoli bene. - Il giorno seguente, separa il DNA dai detriti cellulari, dall'RNA e dalle proteine.
- Centrifugare la miscela a 29.500 × g per 45 minuti a 4 °C.
- Trasferire il surnatante contenente DNA, aggiungere un volume di fenolo:cloroformio:alcol isoamilico 25:24:1 (v/v) e mescolare brevemente fino a ottenere un composto omogeneo.
ATTENZIONE: Fenolo:cloroformio:alcol isoamilico è un materiale pericoloso e deve essere maneggiato con DPI appropriati in una cappa chimica. - Centrifugare nuovamente a 15.000 × g per 5 min a temperatura ambiente. Trasferire lo strato acquoso in un nuovo tubo conico.
- Precipitare e lavare il DNA isolato.
- Aggiungere un volume di isopropanolo puro e incubare a temperatura ambiente per almeno 5 minuti. Centrifugare il DNA a 15.000 × g per 30 minuti a 4 °C.
- Decantare il surnatante e lavare il pellet con etanolo freddo al 70% per rimuovere il sale in eccesso.
- Centrifugare nuovamente a 15.000 × g per 30 minuti a 4 °C, asciugare all'aria e risospendere delicatamente il pellet nel tampone Tris-EDTA (TE) (10 mM Tris-HCl, 1 mM EDTA).
NOTA: L'esperimento può essere messo in pausa qui e i campioni possono essere congelati a -20 °C; tuttavia, il ciclo di congelamento/disgelo dovrebbe essere evitato in quanto ciò può ridurre la qualità degli intermedi di replicazione del DNA.
3. Preparazione del campione ed elettroforesi su gel bidimensionale
- Digerire gli intermedi isolati della replicazione plasmidica.
- Aggiungere 100 unità degli enzimi di restrizione appropriati al campione per digerire il DNA plasmidico, posizionando in particolare la sequenza contenente la ripetizione sulla metà distale di origine del frammento lineare (Figura 2). Inoltre, aggiungere DpnI per tagliare il DNA metilato, rimuovendo così il DNA plasmidico che non è stato completamente replicato nelle cellule umane in coltura.
NOTA: Per ottenere i migliori risultati, gli enzimi di restrizione dovrebbero essere taglierine uniche che producono un frammento di 3-5 kb che posiziona la sequenza incline alla struttura sul braccio discendente dell'arco Y. - Incubare i campioni a 37 °C per 6-10 ore per consentire la completa digestione del plasmide.
- Far precipitare il DNA con 2,5 volumi di etanolo puro freddo e incubare a -20 °C per una notte, oppure aggiungere un volume di isopropanolo e incubare a temperatura ambiente per 5 minuti.
- Centrifugare i campioni digeriti e precipitati a 15.000 × g per 30 minuti a 4 °C.
- Decantare il surnatante e lavare il campione con etanolo freddo al 70%. Centrifugare nuovamente a 15.000 × g per 30 minuti a 4 °C.
- Decantare il surnatante, asciugare all'aria per 10 minuti e risospendere i campioni in 15 μl di tampone TE.
- Aggiungere 100 unità degli enzimi di restrizione appropriati al campione per digerire il DNA plasmidico, posizionando in particolare la sequenza contenente la ripetizione sulla metà distale di origine del frammento lineare (Figura 2). Inoltre, aggiungere DpnI per tagliare il DNA metilato, rimuovendo così il DNA plasmidico che non è stato completamente replicato nelle cellule umane in coltura.
- Preparare il gel di agarosio di prima dimensione allo 0,4-0,5% in 1x Tris-borato-EDTA (TBE) (89 mM di tris base, 89 mM di acido borico, 2 mM di EDTA). Lasciare solidificare la soluzione per almeno 1 ora.
- Iniziare a caricare i campioni nella prima dimensione.
- Caricare la scala entro i primi 3 cm rispetto al bordo più a sinistra del gel. Quindi, caricare l'intero campione preparato, assicurandosi di 3 cm tra ogni coppia.
- Far scorrere il gel in 1x TBE per 19-24 ore a 0,85 V/cm per separare gli intermedi rispetto alle loro dimensioni. Assicurarsi che la camera sia coperta per proteggere i campioni dalla luce, che potrebbe causare danni al DNA.
- Il giorno seguente, rimuovere il gel dal tampone e stimare la posizione del frammento lineare digerito utilizzando un righello.
- Asportare i primi 3 cm di gel contenente la scala e colorare il segmento di gel in 1x TBE contenente 0,3 μg/mL di bromuro di etidio per 10-15 minuti. Visualizzare la scala utilizzando un sistema di documentazione su gel.
- Aggiungere 1,3 cm alla posizione stimata, ottenendo il valore a. Quindi, sottrai 7,5 cm dal valore a, ottenendo il valore b. Allineare il righello contro il gel di prima dimensione e tagliare orizzontalmente in corrispondenza dei valori a e b. Quindi, tagliare verticalmente lo spazio di 3 cm riservato a ciascun campione. Fare riferimento alla Figura 3 per uno schema visivo.
- In un nuovo vassoio di colata, ruotare i segmenti in senso orario e posizionarli nella posizione dei pozzetti del campione (Figura 3).
- Preparare il gel di agarosio di seconda dimensione a una concentrazione dell'1-1,3% in 1x TBE a 0,3 μg/mL di bromuro di etidio.
- Dopo il raffreddamento a circa 55 °C, versare il gel di seconda dimensione sui segmenti di prima dimensione ruotati e lasciarlo solidificare per almeno 1 ora.
- Trasferire la seconda dimensione in una camera con 1x TBE a 0,3 μg/mL di bromuro di etidio e lasciare che il gel si equilibri per almeno 30 minuti.
- Far scorrere il gel, sempre coperto, per 9-10 ore a 4,23 V/cm a 4 °C per separare gli intermedi rispetto alla loro forma.
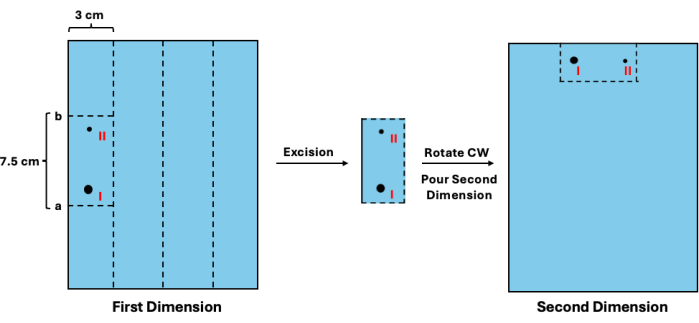
Figura 3: Escissione di intermedi di prima dimensione prima della separazione di seconda dimensione. Dopo la visualizzazione della scala, è possibile stimare la mobilità dei frammenti non replicati. (I) Questo valore può quindi essere utilizzato per determinare i siti di taglio appropriati (a e b) per asportarlo e le sue controparti replicate (II). La sezione del gel deve quindi essere ruotata e posta nella posizione dei pozzetti per la separazione di seconda dimensione. Abbreviazione: CW = senso orario. Clicca qui per visualizzare una versione più grande di questa figura.
4. Southern blotting e ibridazione con sonda radiomarcata
- Rimuovere il gel di seconda dimensione dalla camera e depurinare i frammenti di DNA per 10 minuti in una soluzione di HCl 0,24 M con un leggero oscillamento. Sciacquare il gel con acqua deionizzata e immergerlo in NaOH 0,4 M per 10-15 minuti.
ATTENZIONE: HCl e NaOH sono corrosivi e devono essere maneggiati con DPI appropriati in una cappa chimica. - Iniziare ad assemblare il Southern blot per facilitare il trasferimento degli intermedi separati dal gel alla membrana. Fare riferimento alla Figura 4 per uno schema completo.
- Riempire un contenitore di dimensioni sufficienti con 1 L di NaOH 0,4 M.
- Allineare una lunga lastra di vetro sul contenitore e piegare (per tutta la lunghezza) due lunghi fogli di carta per cromatografia perpendicolari sulla lastra di vetro, estendendosi nel contenitore di NaOH.
- Bagnare la parte superiore della carta con NaOH e rimuovere con cura eventuali bolle d'aria sotto la sua superficie.
- Immergere tre fogli di carta per cromatografia con NaOH e posizionarli sopra la carta piegata, rimuovendo nuovamente eventuali bolle.
- Capovolgere il gel di seconda dimensione e trasferirlo sulle carte.
- Inumidire una membrana di nylon caricata positivamente (dimensione dei pori di 0,45 μm) con acqua deionizzata e posizionarla sopra il gel.
- Infine, aggiungere altri tre fogli di carta, bagnati con acqua deionizzata, sopra la membrana.
- Coprire il NaOH esposto nel contenitore inferiore con pellicola trasparente per evitare l'evaporazione. Posiziona una pila di tovaglioli o tovaglioli di carta sopra la macchia, assicurandoti che sia alta 0,3-0,5 m. Posizionare un peso sulla parte superiore, comprimendo l'intera macchia per facilitare l'azione capillare stretta. Attendere almeno 2 giorni affinché il DNA si trasferisca sulla membrana.
NOTA: La lunghezza e la larghezza della carta e della membrana per cromatografia dipendono dalle dimensioni del gel di agarosio utilizzato per la seconda dimensione. Per un trasferimento più efficiente, utilizzare carta e membrana delle stesse dimensioni del gel.
- Dopo il trasferimento, reticolare il DNA alla membrana utilizzando un reticolante UV a 120 μJ/cm2 per 1 minuto.
NOTA: L'esperimento può essere messo in pausa qui posizionando la membrana in un foglio protettivo ermetico, asciutto e pulito a temperatura ambiente. - Lavare la membrana 2 volte per 5 minuti con 2 citrato di sodio salino (tampone SSC) (0,3 M NaCl, 0,03 M citrato di sodio).
- Preibridare la membrana con 0,18 mL/cm2 di tampone di ibridazione di Church & Gilbert [1 mM EDTA, 1% albumina sierica bovina (BSA), 0,5 M di fosfato di sodio, 7% SDS] a 65 °C, ruotandola in un incubatore di ibridazione per almeno 2 ore.
NOTA: La membrana può preibridarsi per diversi giorni. - Preparare la sonda radiomarcata utilizzando α-32P dATP o dCTP e un kit di marcatura del DNA in conformità con il protocollo del produttore.
ATTENZIONE: I dNTP radiomarcati sono pericolosi e durante la manipolazione è necessario indossare DPI adeguati. Tutto il lavoro radioattivo dovrebbe essere eseguito dietro una schermatura e le persone addestrate dovrebbero essere monitorate per l'assorbimento delle radiazioni utilizzando dosimetri.- Progettare un frammento di DNA lineare da 400-900 bp complementare alla sequenza digerita con restrizione (Figura 2) e amplificare il frammento utilizzando la reazione a catena della polimerasi (PCR).
NOTA: Si consiglia di avere un frammento PCR stock di 50-100 ng/μL - Combinare 100 ng del frammento PCR complementare con DNA Pol I, tampone del frammento di Klenow (3' 5' exo-) e oligo decanucleotidici casuali.
- Denaturare il frammento a 100 °C per 10 min.
- Aggiungere al campione 5 unità di DNA Pol I, frammento di Klenow (3' 5' exo-), 50 μCi di α-32P dNTP e 30-50 μmol di miscela di dNTP carente di tipo dNTP radiomarcato.
- Incubare a 37 °C per 10 minuti per consentire la polimerizzazione e l'incorporazione di dNTP radiomarcato.
- Aggiungere 30-50 μmol di tipo dNTP precedentemente assente al campione e incubare a 37 °C per 10 minuti.
- Purificare il frammento radiomarcato utilizzando una colonna di centrifuga a 3.000 × g per 2 minuti.
- Progettare un frammento di DNA lineare da 400-900 bp complementare alla sequenza digerita con restrizione (Figura 2) e amplificare il frammento utilizzando la reazione a catena della polimerasi (PCR).
- Aggiungere una sonda radiomarcata a 50 mL di tampone di ibridazione e incubare con la membrana per una notte a 65 °C rotante in un incubatore di ibridazione.
- Il giorno successivo, rimuovere la sonda e lavare la membrana 2 volte con il tampone di lavaggio 1 (0,1x SSC, 0,1% SDS) a 42 °C e 2 volte con il tampone di lavaggio 2 (2x SSC, 0,1% SDS) a 65 °C.
NOTA: Tutti i lavaggi devono essere eseguiti con rotazioni rapide per 15 minuti nell'incubatrice. - Asciugare la membrana per 10 minuti e metterla in un foglio protettivo sottile e trasparente. Conservare la membrana sigillata in una cassetta controllata e resistente alle radiazioni con uno schermo sensibile ai fosfori. Lasciare che la membrana sia esposta allo schermo per 1-10 giorni.
- Visualizzare i risultati utilizzando un imager biomolecolare impostato sull'imaging al fosforo. Lavare e riesporre se necessario.
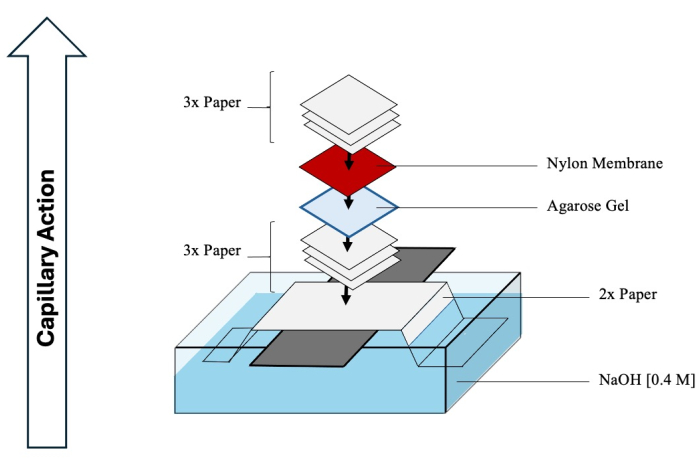
Figura 4: Assemblaggio di Southern blot. Schema completo di un tipico apparato utilizzato per il trasferimento Southern blot di intermedi dalla seconda dimensione su una membrana di nylon. Clicca qui per visualizzare una versione più grande di questa figura.
Access restricted. Please log in or start a trial to view this content.
Risultati
In caso di successo, dopo la visualizzazione, si può osservare un arco acuto di forcelle di replicazione che si estendono verso l'alto e verso l'esterno dal massiccio punto 1n (Figura 5A). La dimensione di un frammento, o percentuale replicata, determina la mobilità del frammento nella prima dimensione. Man mano che gli intermedi sviluppano una struttura più articolata, inizieranno a viaggiare più lentamente nella seconda dimensione. Pertanto, se un inte...
Access restricted. Please log in or start a trial to view this content.
Discussione
2DGE fornisce un'immagine semiquantitativa e completa delle popolazioni relative di intermedi che si formano durante la replicazione di una particolare sequenza. Dato che le fragili strutture molecolari delle forcelle di replicazione devono essere mantenute durante questa procedura, è necessario prestare molta attenzione per prevenire il taglio fisico e la denaturazione chimica. Pertanto, si raccomanda vivamente di evitare qualsiasi trattamento alcalino durante l'isolamento del plasmide...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Ringraziamo Jorge Cebrian e Anastasia Rastokina che hanno iniziato a sviluppare questo approccio nel nostro laboratorio, Massimo Lopes per averci fornito il plasmide pML113 e i suoi preziosi consigli, Ylli Doksani per le discussioni approfondite e i membri del laboratorio Mirkin per il loro supporto. Il lavoro nel laboratorio Mirkin è supportato dal National Institute of General Medical Sciences [R35GM130322] e NSF-BSF [2153071].
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 10x TBE Buffer | Bio Rad | 1610733 | |
| 20x SSC Buffer | Fisher Scientific | BP1325-1 | |
| 293T cells | ATCC | CRL-3216 | |
| a-32P dATP, 3000 Ci/mmol | Revvity | BLU512H250UC | |
| Agarose | Fisher Scientific | BP160-500 | |
| Amersham Hybond-N+ | Fisher Scientific | RPN303B | |
| BAS Storage Phosphor Screens | Fisher Scientific | 28956482 | |
| Church and Gibert's hybriddization buffer | Fisher Scientific | 50-103-5408 | |
| DecaLabel DNA labeling kit | ThermoFisher Scientific | K0622 | |
| DMEM, high gluctose, GltaMAX Supplement, pyruvate | ThermoFisher Scientific | 10569010 | |
| DpnI | New England Biolabs | R0176S | Additional restriction enzymes will need to be purchased as well |
| EDTA 0.5 M, pH 8 | Fisher Scientific | BP2482500 | |
| Ethanol, 70% | Fisher Scientific | BP82031GAL | |
| Fetal Bovine Serum | VWR | 97068-085 | |
| Hydrochloric acid solution, 12 M | Millipore Sigma | 13-1683 | |
| Isopropanol | Fisher Scientific | BP26184 | |
| jetPRIME DNA and siRNA Transfection Reagent with Buffer | VWR | 101000027 | |
| MycoZap Plus-CL | VWR | 75870-448 | |
| NaCl | Millipore Sigma | 746398-500G | |
| Nalgene Oak Ridge High-Speed Centrifuge Tubes | ThermoFisher Scientific | 3139-0050 | |
| Phosphate Buffer Saline, pH 7.4 | ThermoFisher Scientific | 10010023 | |
| Phosphate Buffer Saline, pH 7.5 | ThermoFisher Scientific | 10010024 | |
| Proteinase K | ThermoFisher Scientific | EO0491 | |
| Proteinase K | ThermoFisher Scientific | EO0492 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-4 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-5 | |
| Ruler | Fisher Scientific | 09-016 | |
| Scalpel | Fisher Scientific | 12-460-451 | |
| Sodium dodecyl sulfate | Millipore Sigma | 436143-25G | |
| Sodium hydroxide | Fisher Scientific | S25548 | |
| Sorval LYNX 4000 Superspeed Centrifuge | ThermoFisher Scientific | 75006580 | |
| Sub-cell Horizontal Electrophoresis System | Bio Rad | 1704401 | |
| TH13-6 x 50 Swinging Bucket Rotor | ThermoFisher Scientific | 75003010 | |
| Tris-HCl 1 M, pH 7.5 | Fisher Scientific | BP1757-500 | |
| Trypsin-EDTA (0.25%), phenol red | ThermoFisher Scientific | 25200056 |
Riferimenti
- Liao, X., et al. Repetitive DNA sequence detection and its role in the human genome. Commun Biol. 6 (1), 1-21 (2023).
- Fotsing, S. F., et al. The impact of short tandem repeat variation on gene expression. Nat Genet. 51 (11), 1652-1659 (2019).
- Fan, H., Chu, J. -Y. A brief review of short tandem repeat mutation. GPB. 5 (1), 7-14 (2007).
- Khristich, A. N., Mirkin, S. M. On the wrong DNA track: Molecular mechanisms of repeat-mediated genome instability. J Biol Chem. 295 (13), 4134-4170 (2020).
- Samadashwily, G. M., Raca, G., Mirkin, S. M. Trinucleotide repeats affect DNA replication in vivo. Nat Genet. 17 (3), 298-304 (1997).
- Khristich, A. N., Armenia, J. F., Matera, R. M., Kolchinski, A. A., Mirkin, S. M. Large-scale contractions of Friedreich's ataxia GAA repeats in yeast occur during DNA replication due to their triplex-forming ability. Proc Natl Acad Sci USA. 117 (3), 1628-1637 (2020).
- Shishkin, A. A., et al. Large-scale expansions of Friedreich's ataxia GAA repeats in yeast. Mol Cell. 35 (1), 82-92 (2009).
- Sundararajan, R., Gellon, L., Zunder, R. M., Freudenreich, C. H. Double-strand break repair pathways protect against CAG/CTG repeat expansions, contractions and repeat-mediated chromosomal fragility in Saccharomyces cerevisiae. Genetics. 184 (1), 65-77 (2010).
- Kim, H. -M., et al. Chromosome fragility at GAA tracts in yeast depends on repeat orientation and requires mismatch repair. EMBO J. 27 (21), 2896-2906 (2008).
- Polleys, E. J., House, N. C. M., Freudenreich, C. H. Role of recombination and replication fork restart in repeat instability. DNA Repair. 56, 156-165 (2017).
- Gold, M. A., et al. Restarted replication forks are error-prone and cause CAG repeat expansions and contractions. PLoS Genet. 17 (10), e1009863(2021).
- Lambert, S., et al. Homologous recombination restarts blocked replication forks at the expense of genome rearrangements by template exchange. Mol Cell. 39 (3), 346-359 (2010).
- Burssed, B., Zamariolli, M., Bellucco, F. T., Melaragno, M. I. Mechanisms of structural chromosomal rearrangement formation. Mol Cytogenet. 15 (1), 23(2022).
- Paulson, H. Repeat expansion diseases. Handb Clin Neurol. 147, 105-123 (2018).
- Malik, I., Kelley, C. P., Wang, E., Todd, P. Molecular mechanisms underlying nucleotide repeat expansion disorders. Nat Rev Mol Cell Biol. 22 (9), 589-607 (2021).
- Emanuel, B. S., Zackai, E. H., Medne, L. Emanuel Syndrome. GeneReviews®. Adam, M. P. , University of Washington. Seattle, Seattle, WA. (1993).
- Brewer, B. J., Fangman, W. L. The localization of replication origins on ARS plasmids in S. cerevisiae. Cell. 51 (3), 463-471 (1987).
- Bell, L., Byers, B. Separation of branched from linear DNA by two-dimensional gel electrophoresis. Anal Biochem. 130 (2), 527-535 (1983).
- Voineagu, I., Narayanan, V., Lobachev, K. S., Mirkin, S. M. Replication stalling at unstable inverted repeats: Interplay between DNA hairpins and fork stabilizing proteins. Proc Natl Acad Sci USA. 105 (29), 9936-9941 (2008).
- Nguyen, J. H. G., et al. Differential requirement of Srs2 helicase and Rad51 displacement activities in replication of hairpin-forming CAG/CTG repeats. Nucleic Acids Res. 45 (8), 4519-4531 (2017).
- Krasilnikova, M. M., Mirkin, S. M. Analysis of triplet repeat replication by two-dimensional gel electrophoresis. Trinucleotide Repeat Protocols. Kohwi, Y. , Humana Press. Totowa, NJ. (2004).
- Follonier, C., Oehler, J., Herrador, R., Lopes, M. Friedreich's ataxia-associated GAA repeats induce replication-fork reversal and unusual molecular junctions. Nat Struct Mol Biol. 20 (4), 486-494 (2013).
- Chandok, G. S., Patel, M. P., Mirkin, S. M., Krasilnikova, M. M. Effects of Friedreich's ataxia GAA repeats on DNA replication in mammalian cells. Nucleic Acids Res. 40 (9), 3964-3974 (2012).
- Rastokina, A., et al. Large-scale expansions of Friedreich's ataxia GAA•TTC repeats in an experimental human system: role of DNA replication and prevention by LNA-DNA oligonucleotides and PNA oligomers. Nucleic Acids Res. 51 (16), 8532-8549 (2023).
- Giannattasio, M., et al. Visualization of recombination-mediated damage-bypass by template switching. Nat Struct Mol Biol. 21 (10), 884-892 (2014).
- Hisey, J. A., et al. Pathogenic CANVAS (AAGGG)n repeats stall DNA replication due to the formation of alternative DNA structures. Nucleic Acids Res. 52 (8), 4361-4374 (2024).
- Kalejta, R. F., Lin, H. B., Dijkwel, P. A., Hamlin, J. L. Characterizing replication intermediates in the amplified CHO dihydrofolate reductase domain by two novel gel electrophoretic techniques. Mol Cell Biol. 16 (9), 4923-4931 (1996).
- Little, R. D., Platt, T. H., Schildkraut, C. L. Initiation and termination of DNA replication in human rRNA genes. Mol Cell Biol. 13 (10), 6600-6613 (1993).
- Fanning, E., Zhao, K. SV40 DNA replication: From the A gene to a nanomachine. Virology. 384 (2), 352-359 (2009).
- Sogo, J. M., Stahl, H., Koller, T., Knippers, R. Structure of replicating simian virus 40 minichromosomes: The replication fork, core histone segregation and terminal structures. J Mol Biol. 189 (1), 189-204 (1986).
- Weisshart, K., Taneja, P., Fanning, E. The replication protein A binding site in simian virus 40 (SV40) T antigen and its role in the initial steps of SV40 DNA replication. J Virol. 72 (12), 9771-9781 (1998).
- Sowd, G. A., Fanning, E. A Wolf in sheep's clothing: SV40 co-opts host genome maintenance proteins to replicate viral DNA. PLoS Pathog. 8 (11), e1002994(2012).
- National Library of Medicine: National Center for Biotechnology Information. BLAST: Basic Local Alignment Search Tool. , https://blast.ncbi.nlm.nih.gov/Blast.cgi (2024).
- Hirt, B. Selective extraction of polyoma DNA from infected mouse cell cultures. J Mol Biol. 26 (2), 365-369 (1967).
- Lopes, M., Foiani, M., Sogo, J. M. Multiple mechanisms control chromosome integrity after replication fork uncoupling and restart at irreparable UV lesions. Mol Cell. 21 (1), 15-27 (2006).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon