Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Analyse électrophorétique de la réplication à travers des répétitions d’ADN à structure variable dans l’épisome humain basé sur SV40
Dans cet article
Résumé
Ici, nous décrivons la procédure d’analyse de la progression de la réplication à travers des répétitions pathogènes et sujettes à la structure à l’aide de l’électrophorèse sur gel bidimensionnelle.
Résumé
L’électrophorèse bidimensionnelle sur gel neutre/neutre (2DGE) est apparue comme une technique de référence pour analyser la réplication de l’ADN à travers des obstacles naturels. Ce protocole décrit comment analyser la progression de la fourche de réplication à travers des répétitions d’ADN expansibles et susceptibles de structuration dans l’épisome basé sur le virus simien 40 (SV40) dans les cellules humaines. En bref, lors de la transfection du plasmide dans des cellules humaines, les intermédiaires de réplication sont isolés par le protocole Hirt modifié et traités avec l’enzyme de restriction DpnI pour éliminer l’ADN non répliqué. Les intermédiaires sont ensuite digérés par des enzymes de restriction appropriées pour placer la répétition d’intérêt dans la moitié distale d’origine d’un fragment d’ADN de 3 à 5 kb de long. Les intermédiaires de réplication sont séparés en deux dimensions perpendiculaires, d’abord par la taille, puis par la forme. Après l’hybridation par transfert de Southern, cette approche permet aux chercheurs d’observer le décrochage de la fourche à diverses répétitions structurelles sur la moitié descendante de l’arc Y de réplication. De plus, ce positionnement du site de décrochage permet de visualiser divers résultats du décrochage à médiation répétée, tels que l’inversion de la fourche, l’avènement d’une fourche convergente et le redémarrage de la fourche recombinatoire.
Introduction
Les courtes répétitions en tandem (STR) sont de petites paires de 2 à 9 paires de bases (pb), des séquences répétitives d’ADN qui constituent environ 3 % du génome humain1. Les STR jouent un rôle important dans la régulation des gènes2 ; cependant, leur composition répétitive les rend sujets à la formation d’une structure secondaire de l’ADN non canonique et à l’instabilité génétique ultérieure 3,4. Des hélices gauches aux épingles à cheveux/cruciformes, en passant par les hélices à trois et quatre brins, ces structures d’ADN alternatives posent des défis intrinsèques au réplisome. Une condition préalable naturelle à la formation d’une structure secondaire est le déroulement de l’ADN, qui est une condition préalable à la réplication de l’ADN. Cela présente une énigme unique pour le fonctionnement du génome, car bon nombre de ces structures peuvent se former pendant la réplication, entravant la progression du réplisome et provoquant finalement un blocage de la fourchede réplication 5,6,7 ou, dans les cas graves, un effondrement de la fourche et une rupture de l’ADN 8,9. Il a été démontré que le redémarrage des fourches bloquées et les voies de réparation de l’ADN entraînent des instabilités répétées, telles que des expansions répétées10,11 et des réarrangements génomiques complexes (CGR)12,13. Ces événements peuvent entraîner le développement d’environ 60 maladies humaines connues sous le nom de troubles d’expansion répétée, y compris le syndrome de l’X fragile, la maladie de Huntington, l’ataxie de Friedreich et d’autres14,15 ainsi que des maladies CGR, telles que le syndrome d’Emmanuel16. Par conséquent, pour mieux comprendre les mécanismes de la maladie humaine entraînée par l’instabilité des répétitions, il est impératif d’étudier les détails de la progression de la fourche de réplication à travers ces répétitions.
Une technique d’étude de la progression de la réplication est apparue au milieu des années 1980 lorsque Brewer et Fangman ont cherché à fournir des preuves directes que l’initiation de la réplication chez Saccharomyces cerevisiae se produit au niveau des éléments17 de la séquence de réplication autonome (communément appelée ARS). Ce faisant, ils ont séparé les structures des intermédiaires de réplication de la levure dans l’agarose, adaptant une méthode antérieure de Bell et Byers connue sous le nom d’électrophorèse sur gel neutre/neutre en 2 dimensions (2DGE)18. Cette technique a utilisé le fait que l’ADN non linéaire se déplace différemment dans le gel d’agarose que son équivalent linéaire de la même masse. Plus précisément, dans la 2DGE, l’ADN isolé est séparé en deux dimensions perpendiculaires, d’abord principalement par taille, puis principalement par forme, pour créer une carte complète de la réplication dans une région d’intérêt particulière. Dans leur article original, Brewer et Fangman ont démontré qu’il s’agissait d’un arc composé de structures en « Y simples » ou de fourches de réplication reliant l’ADN non répliqué à leurs homologues répliqués. Ils décrivent en outre d’autres intermédiaires observés comme des « bulles » et des « doubles Y », représentant respectivement les origines de réplication et les fourches convergentes.
La 2DGE peut être utilisée pour étudier les populations relatives d’intermédiaires de réplication de l’ADN à un moment donné. Par conséquent, si une population d’intermédiaires est plus répandue qu’une autre, cela serait évident à la visualisation. Cela fait de 2DGE un outil particulièrement utile pour étudier la progression de la réplication à travers des séquences difficiles, comme les répétitions formant la structure. Par exemple, si la région analysée contient une séquence capable d’induire un blocage de la fourche de réplication, cela se présenterait comme un renflement sur l’arc (Figure 1A), indiquant une accumulation de fourches de réplication à ce locus. Cela peut être observé avec la réplication des séquences de répétition formant des épingles à cheveux dans la levure 19,20,21 et des répétitions formant des triplex dans les cellules humaines 22,23,24. En plus du calage, la 2DGE peut être utilisée pour observer les structures de l’ADN qui ne sont pas conformes aux Y simples standard formés lors de la réplication, comme dans le cas des intermédiaires recombinants25. Ces intermédiaires ont une structure en forme de X plus lourde et plus ramifiée et, par conséquent, se déplacent plus lentement dans la première et la deuxième dimension que les fourches de réplication standard. Des résultats similaires peuvent également être observés en ce qui concerne l’inversion de la fourche de réplication 20,24,26. En réponse à un fort stress de réplication, il a été démontré que les cellules eucaryotes utilisent l’inversion de la fourche de réplication pour sauver les fourches bloquées. Ces fourches inversées ont un poids moléculaire similaire à celui des fourches bloquées ; cependant, leur structure en patte de poulet entraîne une mobilité électrophorétique plus lente dans la deuxième dimension par rapport à leurs compléments en forme de Y, ce qui entraîne une extension vers le haut et vers l’extérieur de l’arc.
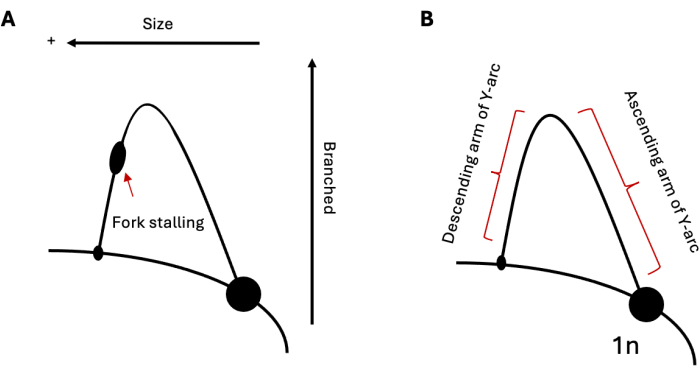
Figure 1 : Analyse par électrophorèse sur gel 2D de la réplication de l’ADN. (A) Schéma d’une 2DGE typique décrivant la réplication par une répétition formant une structure capable d’induire un décrochage de la fourche. La taille et la structure intermédiaires influenceront la mobilité électrophorétique. (B) Échantillon d’arc en Y avec les bras ascendant et descendant respectivement étiquetés. Abréviation : 2DGE = Électrophorèse sur gel neutre/neutre bidimensionnel. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Naturellement, l’un des aspects les plus importants de la 2DGE concerne la qualité et la quantité des intermédiaires de réplication. Cependant, la résolution de l’analyse 2DGE de la réplication à travers des loci endogènes dans des cellules de mammifères est insuffisante pour une séquence cible à copie unique dans le génome humain diploïde de 6 × 10à 9 pb, bien qu’elle ait été réalisée pour des gènes à copies multiples, tels que le locus27 DHFR fortement amplifié ou l’ARN ribosomique28. La réplication basée sur SV40 est un moyen efficace et bien caractérisé d’étudier la réplication dans les cellules eucaryotes29. Il fournit un modèle fiable de réplication eucaryote qui utilise la majeure partie de la machinerie du réplisome de l’hôte pour répliquer le génome viral, qui est divisé en nucléosomes lors de l’infection30,31. Deux exceptions notables au réplisome des mammifères sont que l’antigène T (Tag), au lieu du complexe CMG de l’hôte, sert d’ADN hélicase réplicatif, et que l’ADN polymérase delta synthétise à la fois les brins d’ADN principaux et retardés32. Nous avons tiré parti de ce système en plaçant des tronçons pathogènes de répétitions structurantes en aval d’une origine de réplication SV40 dans un plasmide créé à l’origine dans le laboratoire de Massimo Lopes22. Il est important de noter que ce plasmide contient également le gène codant pour Tag lui-même, ce qui entraîne sa réplication constitutive et extrêmement puissante lors de la transfection dans une variété de cellules humaines cultivées. Cette caractéristique donne lieu à une grande quantité de produits, idéale pour l’analyse 2DGE des intermédiaires formés pendant et en réponse à la réplication de répétitions pathogènes dans les cellules humaines. Ici, nous décrivons une méthode détaillée de visualisation de la réplication des répétitions formant la structure dans l’épisome humain basé sur SV40 à l’aide de l’électrophorèse sur gel bidimensionnelle.
Access restricted. Please log in or start a trial to view this content.
Protocole
REMARQUE : Le plasmide conçu pour notre analyse 2DGE décrite dans les cellules de mammifères doit contenir une origine SV40 de réplication plusieurs kb en amont des répétitions sujettes à la structure (Figure 2). Les synthèses en amont et en aval doivent être gardées à l’esprit lors du choix de l’orientation par rapport à l’origine des répétitions à cloner dans le plasmide.

Figure 2 : Digestion d’un plasmide contenant des répétitions pour l’analyse 2DGE. Les répétitions enclines à la structure sont représentées à plusieurs kb en aval de la fourche de réplication se déplaçant vers la droite. La digestion avec les couteaux uniques 1 et 2 placera la séquence répétée sur le bras descendant de l’arc en Y, étant donné que la séquence dépasse le point médian du fragment digéré. Abréviation : 2DGE = Électrophorèse sur gel neutre/neutre bidimensionnel. Veuillez cliquer ici pour voir une version agrandie de cette figure.
1. Transfection du plasmide dans des cellules de mammifères
- Ensemencez 600 000 cellules HEK293T dans une plaque de culture tissulaire de 10 cm avant la transfection. Laissez les cellules se rétablir à 37 °C pendant la nuit.
REMARQUE : De nombreuses lignées cellulaires peuvent être utilisées pour cette expérience, bien que les cellules contenant l’étiquette SV40 soient recommandées pour des résultats optimaux. ATTENTION : HEK293T cellules sont considérées comme BSL-2 et tous les travaux de culture doivent être effectués dans une enceinte de biosécurité en utilisant une technique d’asepsie appropriée et un EPI approprié. - Lorsque les cellules atteignent 60 % de confluence, transfectez 8 μg d’ADN plasmidique contenant des répétitions dans les cellules ensemencées à l’aide de réactifs de transfection appropriés conformément au protocole du fabricant.
- Si vous n’isolez pas les produits intermédiaires à ce stade, aspirez les anciens milieux et remplacez-les par 10 ml de produits frais après 24 heures.
- Commencer à prélever les cellules 24 à 48 h après la transfection.
- Aspirer le milieu et laver soigneusement avec 10 mL de solution saline tamponnée au phosphate (PBS). Détachez et prélevez les cellules à l’aide de 0,5 mL de trypsine et abaissez à 340 × g pendant 4 min.
- Aspirez le surnageant et lavez les pastilles cellulaires avec du PBS. Essorez à nouveau à 340 × g pendant 4 min et aspirez le surnageant.
REMARQUE : L’expérience peut être interrompue ici en congelant des pastilles de cellule à -80 °C. Nous avons trouvé la meilleure résolution en isolant les intermédiaires de réplication 48 h après la transfection ; Cependant, 24 h ont donné des résultats viables.
2. Isolement des intermédiaires de réplication
- Remettre les cellules en suspension dans 1,5 mL de tampon de lyse Hirt modifié [10 mM de tris-HCl (pH 7,5), 10 mM d’acide éthylènediaminetétraacétique (EDTA)] dans des tubes coniques de 50 mL et commencer la lyse cellulaire.
- Ajouter du dodécylsulfate de sodium (SDS) à une concentration finale de 0,6 % (environ 650 μL de SDS à 2 %) et de la protéinase K à une concentration finale de 100 μg/mL (environ 10 μL de protéinase K de 20 mg/mL) pour éliminer les nucléases.
- Mélangez délicatement par pipetage jusqu’à ce que l’homogénéité soit homogène et incubez le mélange à 37 °C pendant au moins 90 min.
- Augmenter la concentration de NaCl à 1 M (environ 540 μL de NaCl 5 M de matière première) et mélanger doucement jusqu’à ce que l’homogénéité soit homogène. Incuber pendant la nuit (18-24 h) à 4 °C pour permettre la précipitation des débris cellulaires, de l’ARN et des protéines par salage.
REMARQUE : Le mélange sera très visqueux, alors faites attention et soyez patient tout en mélangeant bien. - Le lendemain, séparez l’ADN des débris cellulaires, de l’ARN et des protéines.
- Centrifuger le mélange à 29 500 × g pendant 45 min à 4 °C.
- Transférez le surnageant contenant de l’ADN, ajoutez un volume de phénol :chloroforme :alcool isoamylique 25:24:1 (v/v), et mélangez brièvement jusqu’à ce que l’homogénéité soit homogène.
ATTENTION : L’alcool phénol :chloroforme :isoamylique est une matière dangereuse et doit être manipulé avec un EPI approprié dans une hotte chimique. - Centrifuger à nouveau à 15 000 × g pendant 5 min à température ambiante. Transférez la couche aqueuse dans un nouveau tube conique.
- Précipitez et lavez l’ADN isolé.
- Ajouter un volume d’isopropanol pur et incuber à température ambiante pendant au moins 5 min. Faites tourner l’ADN à 15 000 × g pendant 30 min à 4 °C.
- Décantez le surnageant et lavez la pastille avec de l’éthanol à froid à 70 % pour éliminer l’excès de sel.
- Essorer à nouveau à 15 000 × g pendant 30 min à 4 °C, sécher à l’air libre et remettre doucement la pastille en suspension dans un tampon Tris-EDTA (TE) (10 mM Tris-HCl, 1 mM EDTA).
REMARQUE : L’expérience peut être interrompue ici et les échantillons peuvent être congelés à -20 °C ; cependant, le cycle de gel/dégel doit être évité car cela peut diminuer la qualité des intermédiaires de réplication de l’ADN.
3. Préparation d’échantillons et électrophorèse sur gel en 2 dimensions
- Digérer les intermédiaires isolés de la réplication plasmidique.
- Ajoutez 100 unités des enzymes de restriction appropriées à l’échantillon pour digérer l’ADN plasmidique, en plaçant spécifiquement la séquence contenant la répétition sur la moitié distale d’origine du fragment linéaire (Figure 2). De plus, ajoutez DpnI pour couper l’ADN méthylé, éliminant ainsi tout ADN plasmidique qui n’a pas été entièrement répliqué dans les cellules humaines cultivées.
REMARQUE : Pour de meilleurs résultats, les enzymes de restriction doivent être des couteaux uniques qui produisent un fragment de 3 à 5 kb qui place la séquence sujette à la structure sur le bras descendant de l’arc en Y. - Incuber les échantillons à 37 °C pendant 6 à 10 h pour permettre une digestion complète du plasmide.
- Précipitez l’ADN avec 2,5 volumes d’éthanol pur froid et incubez à -20 °C pendant la nuit, ou ajoutez un volume d’isopropanol et incubez à température ambiante pendant 5 min.
- Centrifuger les échantillons digérés et précipités à 15 000 × g pendant 30 min à 4 °C.
- Décanter le surnageant et laver l’échantillon avec de l’éthanol à froid à 70 %. Faites tourner à nouveau à 15 000 × g pendant 30 min à 4 °C.
- Décanter le surnageant, le faire sécher à l’air libre pendant 10 minutes et remettre les échantillons en suspension dans 15 μL de tampon TE.
- Ajoutez 100 unités des enzymes de restriction appropriées à l’échantillon pour digérer l’ADN plasmidique, en plaçant spécifiquement la séquence contenant la répétition sur la moitié distale d’origine du fragment linéaire (Figure 2). De plus, ajoutez DpnI pour couper l’ADN méthylé, éliminant ainsi tout ADN plasmidique qui n’a pas été entièrement répliqué dans les cellules humaines cultivées.
- Préparez le gel d’agarose de première dimension à 0,4-0,5 % dans 1x Tris-borate-EDTA (TBE) (89 mM de base tris, 89 mM d’acide borique, 2 mM d’EDTA). Laisser la solution se solidifier pendant au moins 1 h.
- Commencez à charger les échantillons dans la première dimension.
- Chargez l’échelle dans les 3 premiers centimètres par rapport au bord le plus à gauche du gel. Ensuite, chargez l’intégralité des échantillons préparés, en veillant à ce qu’il y ait 3 cm entre chaque paire.
- Faites fonctionner le gel dans 1x TBE pendant 19 à 24 h à 0,85 V/cm pour séparer les intermédiaires par rapport à leur taille. Assurez-vous que la chambre est couverte pour protéger les échantillons de la lumière, qui pourrait endommager l’ADN.
- Le lendemain, retirez le gel du tampon et estimez l’emplacement du fragment linéaire digéré à l’aide d’une règle.
- Exciser les 3 premiers cm du gel contenant l’échelle et colorer le segment de gel dans 1 encéphalite à effet de serre contenant 0,3 μg/mL de bromure d’éthidium pendant 10 à 15 min. Visualisez l’échelle à l’aide d’un système de documentation en gel.
- Ajoutez 1,3 cm à l’emplacement estimé, ce qui donne la valeur a. Ensuite, soustrayez 7,5 cm de la valeur a, ce qui donne la valeur b. Alignez la règle contre le gel de première dimension et coupez horizontalement aux valeurs a et b. Ensuite, coupez verticalement l’espace de 3 cm réservé à chaque échantillon. Reportez-vous à la figure 3 pour un schéma visuel.
- Dans un nouveau plateau de coulée, faites pivoter les segments dans le sens des aiguilles d’une montre et placez-les à la position des puits d’échantillon (Figure 3).
- Préparer le gel d’agarose de deuxième dimension à une concentration de 1 à 1,3 % dans 1 TBE à 0,3 μg/mL de bromure d’éthidium.
- Une fois refroidi à environ 55 °C, versez le gel de deuxième dimension sur les segments de première dimension tournés et laissez-le se solidifier pendant au moins 1 h.
- Transférez la deuxième dimension dans une chambre avec 1x TBE à 0,3 μg/mL de bromure d’éthidium et laissez le gel s’équilibrer pendant au moins 30 min.
- Faites couler le gel, à nouveau couvert, pendant 9 à 10 h à 4,23 V/cm à 4 °C pour séparer les produits intermédiaires par rapport à leur forme.
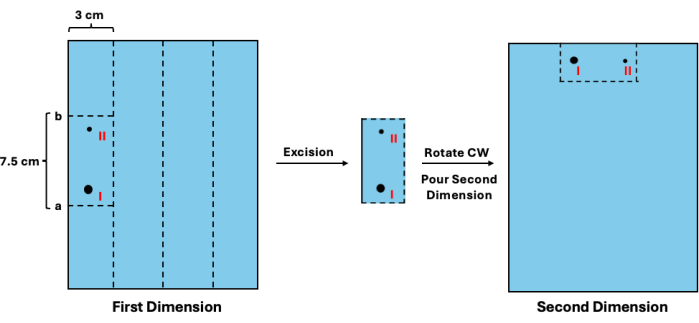
Figure 3 : Excision des intermédiaires de première dimension avant la séparation de la deuxième dimension. Après visualisation de l’échelle, la mobilité des fragments non répliqués peut être estimée. (I) Cette valeur peut ensuite être utilisée pour déterminer les sites de coupe appropriés (a et b) pour l’excision ainsi que ses homologues répliqués (II). La section du gel doit ensuite être tournée et placée dans la position des puits pour la séparation de la deuxième dimension. Abréviation : CW = dans le sens des aiguilles d’une montre. Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. Transfert de Southern et hybridation avec sonde radiomarquée
- Retirez le gel de deuxième dimension de la chambre et dépurinez les fragments d’ADN pendant 10 min dans une solution HCl de 0,24 M avec un léger balancement. Rincez le gel à l’eau déminéralisée et faites-le tremper dans 0,4 M de NaOH pendant 10 à 15 min.
ATTENTION : Le HCl et le NaOH sont corrosifs et doivent être manipulés avec un EPI approprié dans une hotte chimique. - Commencer à assembler le transfert de Southern pour faciliter le transfert des intermédiaires séparés du gel à la membrane. Reportez-vous à la figure 4 pour un schéma complet.
- Remplissez un récipient de taille suffisante avec 1 L de NaOH 0,4 M.
- Alignez une longue feuille de verre sur le récipient et pliez (sur toute la longueur) deux longues feuilles de papier de chromatographie perpendiculairement à la feuille de verre, en s’étendant dans le récipient de NaOH.
- Mouillez le haut du papier avec du NaOH et retirez soigneusement toutes les bulles d’air sous sa surface.
- Trempez trois feuilles de papier de chromatographie avec du NaOH et placez-les sur le papier plié, en enlevant à nouveau les bulles.
- Retournez le gel de deuxième dimension et transférez-le sur les papiers.
- Mouillez une membrane en nylon chargée positivement (taille de pores de 0,45 μm) avec de l’eau DI et placez-la sur le gel.
- Enfin, ajoutez trois autres feuilles de papier, mouillées avec de l’eau DI, sur la membrane.
- Couvrez tout NaOH exposé dans le récipient inférieur avec une pellicule plastique pour éviter l’évaporation. Placez une pile de serviettes ou d’essuie-tout sur la tache, en vous assurant qu’elle mesure 0,3 à 0,5 m de haut. Placez un poids sur le dessus, en comprimant tout le blot pour faciliter l’action capillaire serrée. Attendez au moins 2 jours pour que l’ADN soit transféré à la membrane.
REMARQUE : La longueur et la largeur du papier et de la membrane de chromatographie dépendent de la taille du gel d’agarose utilisé pour la deuxième dimension. Pour un transfert le plus efficace, utilisez du papier et une membrane ayant les mêmes dimensions que ceux du gel.
- Après le transfert, réticuler l’ADN à la membrane à l’aide d’un réticulant UV à 120 μJ/cm2 pendant 1 min.
REMARQUE : L’expérience peut être interrompue ici en plaçant la membrane dans un protecteur de feuille hermétique, sec et propre à température ambiante. - Laver la membrane 2x pendant 5 min avec 2x citrate de sodium salin (tampon SSC) (0,3 M NaCl, 0,03 M de citrate de sodium).
- Préhybrider la membrane avec 0,18 mL/cm2 de tampon d’hybridation de Church & Gilbert [1 mM d’EDTA, 1 % d’albumine sérique bovine (BSA), 0,5 M de phosphate de sodium, 7 % de SDS] à 65 °C, en alternant dans un incubateur d’hybridation pendant au moins 2 h.
REMARQUE : La membrane peut préhybrider pendant plusieurs jours. - Préparez la sonde radiomarquée à l’aide d’un α-32P dATP ou dCTP et d’un kit de marquage ADN conformément au protocole du fabricant.
ATTENTION : Les dNTP radiomarqués sont dangereux et un EPI approprié doit être porté lors de leur manipulation. Tous les travaux radioactifs doivent être effectués derrière le blindage, et les personnes formées doivent être surveillées pour l’absorption de rayonnement à l’aide de dosimètres.- Concevoir un fragment d’ADN linéaire de 400 à 900 pb complémentaire à la séquence digérée par restriction (Figure 2) et amplifier le fragment à l’aide de la réaction en chaîne par polymérase (PCR).
REMARQUE : Nous recommandons d’avoir un fragment PCR de stock de 50 à 100 ng/μL - Combinez 100 ng du fragment complémentaire de PCR avec de l’ADN Pol I, un fragment de Klenow (3' 5' exo-) tampon et des oligos décanucléotidiques aléatoires.
- Dénaturer le fragment à 100 °C pendant 10 min.
- Ajoutez à l’échantillon 5 unités d’ADN Pol I, fragment de Klenow (3' 5' exo-), 50 μCi de dNTP α-32P et 30-50 μmol de mélange dNTP déficient en type dNTP radiomarqué.
- Incuber à 37 °C pendant 10 min pour permettre la polymérisation et l’incorporation de dNTP radiomarqué.
- Ajouter 30 à 50 μmol de type dNTP précédemment absent à l’échantillon et incuber à 37 °C pendant 10 min.
- Purifier le fragment radiomarqué à l’aide d’une colonne de centrifugation à 3 000 × g pendant 2 min.
- Concevoir un fragment d’ADN linéaire de 400 à 900 pb complémentaire à la séquence digérée par restriction (Figure 2) et amplifier le fragment à l’aide de la réaction en chaîne par polymérase (PCR).
- Ajouter la sonde radiomarquée à 50 mL de tampon d’hybridation et incuber avec la membrane pendant la nuit à 65 °C en tournant dans un incubateur d’hybridation.
- Le lendemain, retirez la sonde et lavez la membrane 2x avec le tampon de lavage 1 (0,1x SSC, 0,1% SDS) à 42 °C et 2x avec le tampon de lavage 2 (2x SSC, 0,1% SDS) à 65 °C.
REMARQUE : Tous les lavages doivent être effectués avec des rotations rapides pendant 15 min dans l’incubateur. - Séchez la membrane pendant 10 min et placez-la dans un protecteur de feuille mince et transparent. Stockez la membrane scellée dans une cassette contrôlée et résistante aux radiations avec un écran sensible au phosphore. Laissez la membrane être exposée à l’écran pendant 1 à 10 jours.
- Visualisez les résultats à l’aide d’un imageur biomoléculaire réglé sur l’imagerie au phosphore. Laver et réexposer si nécessaire.
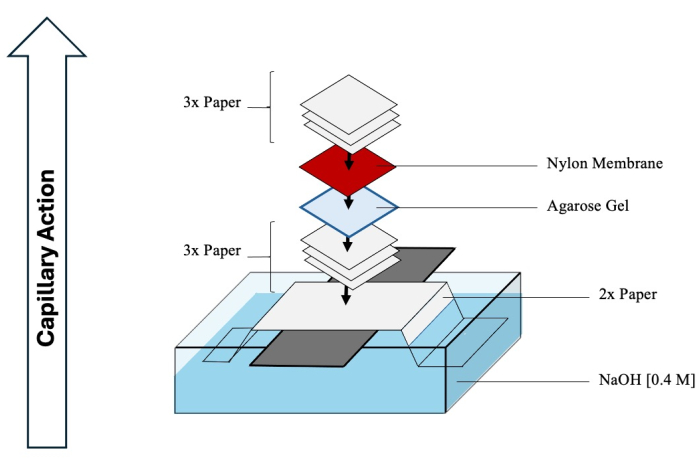
Figure 4 : Assemblage du transfert de Southern. Schéma complet d’un appareil typique utilisé pour le transfert par transfert de Southern d’intermédiaires de la deuxième dimension sur une membrane en nylon. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Résultats
En cas de succès, lors de la visualisation, on peut observer un arc aigu de fourches de réplication s’étendant vers le haut et vers l’extérieur à partir de l’énorme point 1n (Figure 5A). La taille d’un fragment, ou le pourcentage de réplication, détermine la mobilité du fragment dans la première dimension. Au fur et à mesure que les intermédiaires développent une structure plus articulée, ils commenceront à se déplacer plus lentement ...
Access restricted. Please log in or start a trial to view this content.
Discussion
2DGE fournit une image semi-quantitative et complète des populations relatives d’intermédiaires qui apparaissent lors de la réplication d’une séquence particulière. Étant donné que les structures moléculaires fragiles des fourches de réplication doivent être maintenues tout au long de cette procédure, une grande prudence doit être mise en œuvre pour éviter le cisaillement physique et la dénaturation chimique. Par conséquent, il est fortement recommandé d’éviter to...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Nous remercions Jorge Cebrian et Anastasia Rastokina qui ont commencé à développer cette approche dans notre laboratoire, Massimo Lopes pour nous avoir fourni le plasmide pML113 et des conseils inestimables, Ylli Doksani pour les discussions perspicaces, et les membres du laboratoire Mirkin pour leur soutien. Les travaux du laboratoire Mirkin sont soutenus par le National Institute of General Medical Sciences [R35GM130322] et la NSF-BSF [2153071].
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 10x TBE Buffer | Bio Rad | 1610733 | |
| 20x SSC Buffer | Fisher Scientific | BP1325-1 | |
| 293T cells | ATCC | CRL-3216 | |
| a-32P dATP, 3000 Ci/mmol | Revvity | BLU512H250UC | |
| Agarose | Fisher Scientific | BP160-500 | |
| Amersham Hybond-N+ | Fisher Scientific | RPN303B | |
| BAS Storage Phosphor Screens | Fisher Scientific | 28956482 | |
| Church and Gibert's hybriddization buffer | Fisher Scientific | 50-103-5408 | |
| DecaLabel DNA labeling kit | ThermoFisher Scientific | K0622 | |
| DMEM, high gluctose, GltaMAX Supplement, pyruvate | ThermoFisher Scientific | 10569010 | |
| DpnI | New England Biolabs | R0176S | Additional restriction enzymes will need to be purchased as well |
| EDTA 0.5 M, pH 8 | Fisher Scientific | BP2482500 | |
| Ethanol, 70% | Fisher Scientific | BP82031GAL | |
| Fetal Bovine Serum | VWR | 97068-085 | |
| Hydrochloric acid solution, 12 M | Millipore Sigma | 13-1683 | |
| Isopropanol | Fisher Scientific | BP26184 | |
| jetPRIME DNA and siRNA Transfection Reagent with Buffer | VWR | 101000027 | |
| MycoZap Plus-CL | VWR | 75870-448 | |
| NaCl | Millipore Sigma | 746398-500G | |
| Nalgene Oak Ridge High-Speed Centrifuge Tubes | ThermoFisher Scientific | 3139-0050 | |
| Phosphate Buffer Saline, pH 7.4 | ThermoFisher Scientific | 10010023 | |
| Phosphate Buffer Saline, pH 7.5 | ThermoFisher Scientific | 10010024 | |
| Proteinase K | ThermoFisher Scientific | EO0491 | |
| Proteinase K | ThermoFisher Scientific | EO0492 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-4 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-5 | |
| Ruler | Fisher Scientific | 09-016 | |
| Scalpel | Fisher Scientific | 12-460-451 | |
| Sodium dodecyl sulfate | Millipore Sigma | 436143-25G | |
| Sodium hydroxide | Fisher Scientific | S25548 | |
| Sorval LYNX 4000 Superspeed Centrifuge | ThermoFisher Scientific | 75006580 | |
| Sub-cell Horizontal Electrophoresis System | Bio Rad | 1704401 | |
| TH13-6 x 50 Swinging Bucket Rotor | ThermoFisher Scientific | 75003010 | |
| Tris-HCl 1 M, pH 7.5 | Fisher Scientific | BP1757-500 | |
| Trypsin-EDTA (0.25%), phenol red | ThermoFisher Scientific | 25200056 |
Références
- Liao, X., et al. Repetitive DNA sequence detection and its role in the human genome. Commun Biol. 6 (1), 1-21 (2023).
- Fotsing, S. F., et al. The impact of short tandem repeat variation on gene expression. Nat Genet. 51 (11), 1652-1659 (2019).
- Fan, H., Chu, J. -Y. A brief review of short tandem repeat mutation. GPB. 5 (1), 7-14 (2007).
- Khristich, A. N., Mirkin, S. M. On the wrong DNA track: Molecular mechanisms of repeat-mediated genome instability. J Biol Chem. 295 (13), 4134-4170 (2020).
- Samadashwily, G. M., Raca, G., Mirkin, S. M. Trinucleotide repeats affect DNA replication in vivo. Nat Genet. 17 (3), 298-304 (1997).
- Khristich, A. N., Armenia, J. F., Matera, R. M., Kolchinski, A. A., Mirkin, S. M. Large-scale contractions of Friedreich's ataxia GAA repeats in yeast occur during DNA replication due to their triplex-forming ability. Proc Natl Acad Sci USA. 117 (3), 1628-1637 (2020).
- Shishkin, A. A., et al. Large-scale expansions of Friedreich's ataxia GAA repeats in yeast. Mol Cell. 35 (1), 82-92 (2009).
- Sundararajan, R., Gellon, L., Zunder, R. M., Freudenreich, C. H. Double-strand break repair pathways protect against CAG/CTG repeat expansions, contractions and repeat-mediated chromosomal fragility in Saccharomyces cerevisiae. Genetics. 184 (1), 65-77 (2010).
- Kim, H. -M., et al. Chromosome fragility at GAA tracts in yeast depends on repeat orientation and requires mismatch repair. EMBO J. 27 (21), 2896-2906 (2008).
- Polleys, E. J., House, N. C. M., Freudenreich, C. H. Role of recombination and replication fork restart in repeat instability. DNA Repair. 56, 156-165 (2017).
- Gold, M. A., et al. Restarted replication forks are error-prone and cause CAG repeat expansions and contractions. PLoS Genet. 17 (10), e1009863(2021).
- Lambert, S., et al. Homologous recombination restarts blocked replication forks at the expense of genome rearrangements by template exchange. Mol Cell. 39 (3), 346-359 (2010).
- Burssed, B., Zamariolli, M., Bellucco, F. T., Melaragno, M. I. Mechanisms of structural chromosomal rearrangement formation. Mol Cytogenet. 15 (1), 23(2022).
- Paulson, H. Repeat expansion diseases. Handb Clin Neurol. 147, 105-123 (2018).
- Malik, I., Kelley, C. P., Wang, E., Todd, P. Molecular mechanisms underlying nucleotide repeat expansion disorders. Nat Rev Mol Cell Biol. 22 (9), 589-607 (2021).
- Emanuel, B. S., Zackai, E. H., Medne, L. Emanuel Syndrome. GeneReviews®. Adam, M. P. , University of Washington. Seattle, Seattle, WA. (1993).
- Brewer, B. J., Fangman, W. L. The localization of replication origins on ARS plasmids in S. cerevisiae. Cell. 51 (3), 463-471 (1987).
- Bell, L., Byers, B. Separation of branched from linear DNA by two-dimensional gel electrophoresis. Anal Biochem. 130 (2), 527-535 (1983).
- Voineagu, I., Narayanan, V., Lobachev, K. S., Mirkin, S. M. Replication stalling at unstable inverted repeats: Interplay between DNA hairpins and fork stabilizing proteins. Proc Natl Acad Sci USA. 105 (29), 9936-9941 (2008).
- Nguyen, J. H. G., et al. Differential requirement of Srs2 helicase and Rad51 displacement activities in replication of hairpin-forming CAG/CTG repeats. Nucleic Acids Res. 45 (8), 4519-4531 (2017).
- Krasilnikova, M. M., Mirkin, S. M. Analysis of triplet repeat replication by two-dimensional gel electrophoresis. Trinucleotide Repeat Protocols. Kohwi, Y. , Humana Press. Totowa, NJ. (2004).
- Follonier, C., Oehler, J., Herrador, R., Lopes, M. Friedreich's ataxia-associated GAA repeats induce replication-fork reversal and unusual molecular junctions. Nat Struct Mol Biol. 20 (4), 486-494 (2013).
- Chandok, G. S., Patel, M. P., Mirkin, S. M., Krasilnikova, M. M. Effects of Friedreich's ataxia GAA repeats on DNA replication in mammalian cells. Nucleic Acids Res. 40 (9), 3964-3974 (2012).
- Rastokina, A., et al. Large-scale expansions of Friedreich's ataxia GAA•TTC repeats in an experimental human system: role of DNA replication and prevention by LNA-DNA oligonucleotides and PNA oligomers. Nucleic Acids Res. 51 (16), 8532-8549 (2023).
- Giannattasio, M., et al. Visualization of recombination-mediated damage-bypass by template switching. Nat Struct Mol Biol. 21 (10), 884-892 (2014).
- Hisey, J. A., et al. Pathogenic CANVAS (AAGGG)n repeats stall DNA replication due to the formation of alternative DNA structures. Nucleic Acids Res. 52 (8), 4361-4374 (2024).
- Kalejta, R. F., Lin, H. B., Dijkwel, P. A., Hamlin, J. L. Characterizing replication intermediates in the amplified CHO dihydrofolate reductase domain by two novel gel electrophoretic techniques. Mol Cell Biol. 16 (9), 4923-4931 (1996).
- Little, R. D., Platt, T. H., Schildkraut, C. L. Initiation and termination of DNA replication in human rRNA genes. Mol Cell Biol. 13 (10), 6600-6613 (1993).
- Fanning, E., Zhao, K. SV40 DNA replication: From the A gene to a nanomachine. Virology. 384 (2), 352-359 (2009).
- Sogo, J. M., Stahl, H., Koller, T., Knippers, R. Structure of replicating simian virus 40 minichromosomes: The replication fork, core histone segregation and terminal structures. J Mol Biol. 189 (1), 189-204 (1986).
- Weisshart, K., Taneja, P., Fanning, E. The replication protein A binding site in simian virus 40 (SV40) T antigen and its role in the initial steps of SV40 DNA replication. J Virol. 72 (12), 9771-9781 (1998).
- Sowd, G. A., Fanning, E. A Wolf in sheep's clothing: SV40 co-opts host genome maintenance proteins to replicate viral DNA. PLoS Pathog. 8 (11), e1002994(2012).
- National Library of Medicine: National Center for Biotechnology Information. BLAST: Basic Local Alignment Search Tool. , https://blast.ncbi.nlm.nih.gov/Blast.cgi (2024).
- Hirt, B. Selective extraction of polyoma DNA from infected mouse cell cultures. J Mol Biol. 26 (2), 365-369 (1967).
- Lopes, M., Foiani, M., Sogo, J. M. Multiple mechanisms control chromosome integrity after replication fork uncoupling and restart at irreparable UV lesions. Mol Cell. 21 (1), 15-27 (2006).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon